JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
가스 크로마토그래피 - 경산호 시료의 질량분석법 기반 표적 대사체학
요약
여기에서는 가스 크로마토그래피-질량 분석 분석을 위해 산호 홀로비온트에서 극성 및 반극성 대사 산물의 추출 및 준비와 분리된 산호 숙주 조직 및 Symbiodiniaceae 세포 분획을 제시합니다.
초록
가스 크로마토그래피-질량분석법(GC-MS) 기반 접근법은 자포동물-와편모충 공생의 대사 기초와 산호가 스트레스에 어떻게 반응하는지(즉, 온도로 인한 표백 중)를 설명하는 데 강력한 것으로 입증되었습니다. 자포동물 숙주와 관련 미생물(Symbiodiniaceae 및 기타 원생생물, 박테리아, 고세균, 곰팡이 및 바이러스)로 구성된 산호 홀로비온트의 정상 상태 대사 산물 프로파일링은 산호의 전체적인 대사 상태를 특성화하기 위해 주변 및 스트레스 조건에서 성공적으로 적용되었습니다.
그러나 공생 상호 작용을 둘러싼 질문에 답하려면 산호 숙주와 조류 공생체의 대사 산물 프로필을 독립적으로 분석해야 하며, 이는 조직의 물리적 분리 및 분리에 의해서만 달성될 수 있으며 독립적인 추출 및 분석이 뒤따릅니다. 대사체학의 적용은 산호 분야에서 비교적 새로운 것이지만, 연구 그룹의 지속적인 노력으로 산호 숙주 조직과 조류 공생체의 분리를 포함하여 산호의 대사 산물을 분석하는 강력한 방법이 개발되었습니다.
이 백서는 고려해야 할 주요 최적화 단계를 포함하여 GC-MS 분석을 위한 홀로비온트 분리 및 대사 산물 추출에 대한 단계별 가이드를 제공합니다. 우리는 일단 독립적으로 분석되면 두 분획(산호 및 공생충과)의 결합된 대사 산물 프로필이 전체(홀로비온트)의 프로필과 유사하지만 조직을 분리함으로써 전체만으로는 얻을 수 없는 두 파트너의 대사 및 상호 작용에 대한 주요 정보를 얻을 수 있음을 보여줍니다.
서문
대사 산물은 세포 과정의 최종 산물을 나타내며, 대사체학(metabolomics)은 특정 유기체 또는 생태계에서 생산되는 대사 산물 모음에 대한 연구로 유기체 기능의 직접적인 측정을 제공할 수 있습니다1. 이는 생태계, 공생적 상호작용 및 복원 도구를 탐색하는 데 특히 중요한데, 대부분의 관리 전략의 목표는 특정 생태계 서비스 기능을 보존(또는 복원)하는 것이기 때문이다2. 산호초는 공생적 상호작용을 규명하고 산호의 생리적 반응을 군집 수준 및 생태계 수준의 영향과 연결하기 위한 대사체학의 잠재적 가치를 보여주는 하나의 수생 생태계이다3. 고처리량 가스 크로마토그래피-질량분석법(GC-MS)의 응용 분야는 높은 선택성과 감도로 광범위한 대사산물 클래스를 동시에 신속하게 분석하고, 스펙트럼 라이브러리를 사용할 수 있을 때 신속한 화합물 식별을 제공하며, 상대적으로 낮은 시료당 비용으로 높은 수준의 재현성과 정확도를 제공할 수 있기 때문에 특히 가치가 있습니다.
산호는 산호 동물, 광합성 와편모충 내공생체(과: Symbiodiniaceae4) 및 복잡한 마이크로바이옴 5,6으로 구성된 홀로비온트입니다. 전반적으로, 홀로비온트의 적합성은 주로 각 구성원의 대사 기능을 지원하기 위해 작은 분자와 요소의 교환을 통해 유지된다 7,8,9,10. 대사체학 접근법은 산호의 공생 특이성(symbiosis specificity)9,11, 열 스트레스에 대한 표백 반응(bleaching response)7,8,12,13, 질병 반응(disease response)14, 오염 노출 반응(pollution exposure response)15, 광순응(photo-aclimation)16 및 화학적 신호전달(chemical signalling)17의 대사 기초를 밝히고 바이오마커 발견(biomarker discovery)18을 돕는 데 특히 강력한 것으로 입증되었다,19. 또한, 대사체학은 DNA 및 RNA 기반 기술에서 추론된 결론에 대한 귀중한 확인을 제공할 수 있습니다 9,20. 따라서 스트레스의 대사 바이오마커 검출18,19 및 영양 보조금 21과 같은 적극적인 관리 전략의 잠재력을 조사하는 것과 같이 산호초의 건강을 평가하고 산호초 보존을 위한 도구를 개발하기 위해 대사체학을 사용할 수 있는 상당한 잠재력이 있다21.
숙주 세포와 공생 세포를 분리하고 홀로비온트로 함께 하지 않고 독립적으로 대사 산물 프로파일을 분석하면 파트너 상호 작용, 독립적인 생리학적 및 대사 상태, 적응을 위한 잠재적 분자 메커니즘에 대한 더 많은 정보를 얻을 수 있습니다 11,12,22,23,24. 산호와 공생충과(Symbiodiniaceae)를 분리하지 않고, 복잡한 게놈 재구성 및대사 모델링을 제외하고는 산호 및/또는 공생충과(Symbiodiniaceae)의 기여와 신진대사를 독립적으로 설명하는 것은 거의 불가능하지만, 이는 산호-와편모충 공생에 아직 적용되지 않았다. 또한, 홀로비온트의 대사 산물 프로필에서 숙주 또는 조류 공생체의 개별 대사에 대한 정보를 추출하려고 시도하면 오해가 발생할 수 있습니다.
예를 들어, 최근까지, 산호 및 홀로비온트 조직으로부터의 추출물에서 C18:3n-6, C18:4n-3 및 C16 고도불포화 지방산의 존재는 조류 공생체로부터 유래된 것으로 생각되었는데, 이는 산호가 오메가-3(ω3) 지방산의 생산에 필수적인 ωx 탈포화효소를 가지고 있지 않다고 가정했기 때문이다. 그러나, 최근의 게놈 증거에 의하면, 다수의 자포동물은 ω3 PUFA de novo를 생산하고 ω3 장쇄 PUFA26을 추가로 생합성할 수 있는 능력을 가지고 있다. GC-MS를 안정 동위원소 표지(예: 13C-중탄산염, NaH 13CO3)와결합하면 제어 조건과 외부 스트레스 요인에 대한 반응으로 산호 홀로비온트 대사 네트워크를 통해 광합성으로 고정된 탄소의 운명을 추적하는 데 사용할 수 있습니다27,28. 그러나, 13 C 운명의 추적에 있는 긴요한 단계는 조류 세포에서 산호 조직의 분리이다 그 때 산호 숙주 분획에 있는 13C 표지된 화합물의 존재는 산호에 전이된 Symbiodiniaceae 파생한 대사 산물 또는 전위된 레테르를 붙인 화합물의 하류 제품으로 명백하게 할당될 수 있다. 이 기술은 글리세롤이 광합성이 공생체에서 숙주로 전위되는 주요 형태라는 오랜 가정에 도전하고(29), 표백27,28 및 양립할 수 없는 Symbiodiniaceae 종11에 대한 반응으로 파트너 간 영양 플럭스가 어떻게 변하는지 설명함으로써 그 힘을 입증했습니다.
조직을 분리하는 결정은 주로 연구 질문에 의해 결정되지만, 이 접근법의 실용성, 신뢰성 및 잠재적인 대사 영향을 고려하는 것이 중요합니다. 여기에서는 홀로비온트에서 대사 산물을 추출하기 위한 상세하고 입증된 방법과 별도의 숙주 및 공생체 분획을 제공합니다. 우리는 숙주와 공생체의 대사 산물 프로필을 독립적으로 비교하고 이러한 프로필이 홀로비온트 대사 산물 프로필과 어떻게 비교되는지 비교합니다.
Access restricted. Please log in or start a trial to view this content.
프로토콜
참고: 실험 설계, 샘플 수집 및 보관은 2,30,31의 다른 곳에서 자세히 설명되었습니다. 야생 산호 채취에 대한 허가 승인은 채취 및 실험 전에 받아야 합니다. 이곳의 샘플은 바타비아 산호 농장(워싱턴 주 제럴턴)에서 수입된 몬티포라 몰리스(Montipora mollis, 녹색 형태) 군락에서 채취한 것으로, 원래는 양식 허가 AQ1643에 따라 수심 1m의 아브로로스 제도(Abrohlos Islands; 28°52'43.3"S 114°00'17.0"E)의 암초에서 채취한 것입니다. 샘플링 전에 콜로니는 35 PSU의 800 L 수족관에서, 150 μmol 광자·m−2·s−1의 청색광 및 백색광 아래에서, 그리고 25 °C ± 0.5 °C에서 3개월 동안 보관되었습니다. 산호 조각(~5cm2, N=6)을 액체 질소에서 급속 냉동하고 처리할 때까지 -80°C에서 보관하였다.
1. 추출 용액 및 장비의 준비
- 산호 조직 제거 최소 1일 전에 추출 용액과 장비를 준비하십시오.
- 깨끗하고 세제가 없는 유리 제품에 초순수를 4°C로 사전 식힙니다.
- 100% LC 등급 메탄올을 10μg·mL−1 최종 농도의 적절한 내부 표준물질(예: 13C6소르비톨)과 혼합합니다.
- 절반은 100% LC 등급 메탄올, 절반은 초순수를 사용하여 50% 메탄올 추출 용액을 만듭니다. 두 메탄올 용액을 모두 -20°C에서 보관합니다.
참고: 대사 산물의 분해를 방지하기 위해 한 번에 5개의 산호 조각 배치로 샘플 처리 단계를 수행하고 하나의 추가 생물학적(물만) 공백(총 샘플 N = 6)을 사용하는 것이 좋습니다. 각 산호 샘플이 두 개의 분획(산호 숙주 조직, 이하 "숙주", 미세조류 세포, 이하 "공생체")으로 분리되면 한 처리 배치의 총 샘플 수는 12개가 됩니다.
2. 산호 대사 담금질
참고: 실험 설계, 샘플 수집 및 보관은 2,30,31의 다른 곳에서 자세히 설명되었습니다. 그러나, 대사를 소멸시키는 데 걸리는 시간(즉, 산호 샘플 수집과 보존 사이의 시간)은 본래의 반응을 포착하는 데 중요하다는 것을 유의해야 한다(30). 채취 후 가능한 한 빨리 시료를 보존하여 시료 분해 또는 비표적 생리학적 반응으로 인한 대사산물 조성의 변화를 방지한다32.
- 산호 조각을 멸균 샘플 수집 백에 넣고 여분의 바닷물을 최대한 배출합니다. 샘플을 액체 질소에 최소 30초 동안 담그십시오. 샘플을 가능한 한 빨리 -80°C 냉동고로 옮겨 보관합니다.
참고: 샘플은 처리할 때까지 빛이 차단된 용기에서 -80°C로 동결할 수 있으므로 동결-해동 주기를 피할 수 있습니다.
3. 골격에서 산호 조직 제거
알림: 샘플은 지속적인 신진대사를 방지하면서 동시에 액체 형태가 되도록 항상 얼음(4°C) 위에 보관해야 합니다.
- 깨끗하고 멸균된 검체 채취 백을 얼음 위에 놓아 백이 안정적이고 얕은 우물의 얼음 위에서 열리지만 얼음에 잠기지 않도록 합니다. 10mL의 찬물(4°C) 초순수를 백에 넣습니다.
알림: 이렇게 하면 차갑고 가압된 공기와 주변 얼음으로 인한 산호 조각의 반복적인 동결-해동을 방지하는 데 도움이 됩니다. - 멸균된 핀셋으로 산호 조각을 선택하고 멸균 파스퇴르 피펫을 사용하여 해수 잔여물이 남지 않을 때까지 찬물(4°C) 초순수로 헹굽니다. 헹군 산호 조각을 초순수 10mL가 들어 있는 가방에 담그십시오.
참고: 이 헹굼은 다운스트림 분석을 방해할 수 있는 잔류 염을 제거하는 데 중요합니다. 샘플을 4°C로 유지하기 위해 백을 통해 물이나 산호 조각에 손이 닿지 않도록 하십시오. - 멸균 1mL 피펫 팁을 전기 테이프로 에어건 끝에 테이프로 붙이고 팁 끝을 ~5mm 잘라냅니다(그림 1A).
- 가방을 반쯤 밀봉하고 공기 흐름을 중저온으로 하여 산호 조각 위로 물이 원을 그리며 움직이도록 하여 조직을 부드럽게 제거하기 위해 산호 조각에 에어건을 조준합니다.
- ~3분 후 또는 골격에서 모든 조직이 제거된 것처럼 보이면 공기를 끄고 에어브러시를 제거합니다. 가방을 완전히 밀봉하십시오.
- 제거한 산호 조직을 모두 가방의 하단 모서리에 짜냅니다. 반대쪽 모서리를 잘라내고 봉지의 내용물을 얼음 위에 있는 15mL 튜브에 부드럽게 붓습니다.
4. 선택적인 균질화
알림: 일부 산호 종은 다른 종보다 점성이 더 높기 때문에 에어 브러싱으로 슬러리 대신 덩어리로 조직을 제거합니다. 에어 브러시 균질액에 조직 덩어리가 보이면 모든 샘플에 대해 4°C에서 균질화 단계를 추가할 수 있습니다.
- 기계식 톱니 균질화기를 4°C 70% 메탄올로 두 번 청소하고 마지막으로 4°C 초순수로 청소합니다.
- 샘플이 완전히 균질화되고 덩어리가 보이지 않을 때까지 15mL 튜브에서 산호 샘플을 ~1분 동안 균질화합니다.
- 각 샘플 사이의 4.1단계에서와 같이 균질기를 청소합니다. 시료 전체에서 균질화 시간을 일정하게 유지합니다.
5. 정규화를 위한 샘플 수집
- Symbiodiniaceae 세포 수, 산호 숙주 조직 단백질 함량 분석 및 엽록 소 추정을 위해 균질화된 조직에서 1,000μL 부분 표본을 수집합니다. 분석할 준비가 될 때까지 -20°C에서 보관합니다(섹션 10).
6. 선택적 산호 숙주 조직 - Symbiodiniaceae 세포 분리
- 냉장 원심분리기를 사용하여 2,500× g 에서 4°C에서 5분 동안 산호 균질액을 원심분리합니다.
참고: 이 속도는 상층액에 부유하는 숙주 조직에서 세포벽을 그대로 유지하면서 더 무거운 Symbiodiniaceae 세포를 분리하는 데 최적입니다. - 숙주 물질이 포함된 상층액을 제거하고 새 15mL 튜브에 넣습니다.
참고: 숙주 조직의 지질은 일반적으로 공생체 세포 위에 좁은 분홍색/흰색 층을 형성합니다. 이 층은 피펫팅을 통해 용해성 숙주 상층액과 함께 수집할 수 있습니다(그림 1B). - 정확히 1분 동안 호스트를 세게 소용돌이칩니다. 조류 펠릿 샘플과 호스트 상청액 샘플을 얼음 위에 보관하십시오.
- 조류 펠릿에 4°C에서 초순수 2mL를 추가합니다. 펠릿을 다시 부유시키기 위해 정확히 2분 동안 격렬하게 소용돌이칩니다.
참고: Symbiodiniaceae 유전형 분석을 위해 산호 콜로니에서 1cm의 개별 단편을 수집하지 않은 경우, Symbiodiniaceae 세포 현탁액의 200μL 분취액을 여기에서 수집하고, 선호하는 DNA 완충액에 보존하고, Symbiodiniaceae 유전형 분석에 대한 Thurber et al.30 에 설명된 대로 저장할 수 있습니다(예: González-Pech et al.12에 따름). - 6.1-6.4단계를 한 번 더 반복합니다.
알림: 숙주와 공생체의 신뢰할 수 있는 분리는 일부 종은 다른 종보다 점성이 더 높을 수 있기 때문에 산호 바이오매스와 종에 따라 다릅니다. 최소 3단계의 세척이 권장되지만 분리 성공 여부에 따라 늘릴 수 있습니다. 숙주 분획의 바닥에서 Symbiodiniaceae 세포가 보이지 않을 때까지, 그리고 Symbiodiniaceae 분획에 숙주 물질이 눈에 띄게 없어질 때까지(예: 상단에 흰색 층이 없음) 세척 단계 4.7-4.9를 반복합니다(그림 1). - 숙주 물질이 포함된 상층액을 제거하고 새 15mL 튜브에 넣습니다.
- 공생체 펠릿을 15mL 튜브에 보관합니다.
7. 샘플 건조
- holobiont 균질화 또는 분리된 숙주와 Symbiodiniaceae 분획을 모두 -80°C에서 ~120분 동안 동결합니다. -85°C에서 0.01mbar 진공으로 밤새 샘플을 동결건조합니다.
참고: 동결건조 중 샘플 손실을 방지하려면 멸균 2G 바늘을 사용하여 ~2mm 구멍이 조심스럽게 뚫린 다른 멸균 튜브 또는 멸균 파라필름에서 절단된 뚜껑을 사용하는 것이 좋습니다. - 건조 시 실험실 저울을 사용하여 다음 중 하나를 계량합니다: 1) 홀로비온트 25mg; 2) 공생체 분획 15mg; 또는 3) 각 샘플에서 30mg의 숙주 조직을 별도의 2mL 가소제가 없는 미세 원심분리기 튜브에 넣습니다.
참고: 중요 단계: 추출을 위한 바이오매스의 최적화는 충분한 신호를 보장하면서 GC-MS에 과부하가 걸리지 않도록 하는 데 필수적입니다. 말린 산호 물질은 매우 정적입니다. 샘플 손실을 방지하려면 정전기 방지 장치를 사용하여 샘플 및 칭량 용기에서 정전기 전하를 제거하십시오. 간단하고 비용 효율적인 대안은 샘플 튜브 아래에 세탁 건조기 시트를 놓는 것입니다. 건조된 공생체 펠릿은 멸균 블레이드를 사용하여 원하는 무게로 절단할 수 있습니다.
8. 세포내 대사산물 추출
- 동결건조된 홀로비온트에서 세포내 대사산물 추출:
- 내부 표준물질(IS; 13의C6 소르비톨 및/또는 13C5-15 N 발린, 10μM에서) 각 튜브에.
- 소량의 710-1,180μm 산 세척 유리 비드(~10mg)를 각 샘플에 추가합니다. 50Hz의 비드 밀에 3분 동안 사전 냉각된(-20°C) 비드 밀 인서트에 넣습니다.
- IS(13C6 소르비톨 및/또는 13C 5-15 N 발린, 10 μM에서)가 포함된 100% 저온(-20°C) 메탄올 600μL를 각 튜브에 추가합니다.
- 1분 동안 섞는 소용돌이. 4°C의 로티세리 셰이커에 30분간 올려 놓습니다.
- 분리된 동결건조된 Symbiodiniaceae 세포에서 세포내 대사산물 추출:
- 건조된 Symbiodiniaceae 재료에 IS(13C6 소르비톨 및/또는 13C5-15N 발린, 10μM에서)가 포함된 100% 저온(-20°C) 메탄올 200μL를 추가합니다.
- 소량의 710-1,180μm 산 세척 유리 비드(~10mg)를 추가합니다. 50Hz의 비드 밀에 3분 동안 사전 냉각된(-20°C) 비드 밀 인서트에 넣습니다.
- IS가 있는 800% 저온(-20°C) 메탄올 800μL를 추가하고 30초 동안 소용돌이를 추가합니다.
- 분리된 동결건조된 숙주 조직에서 세포내 대사산물 추출:
- IS(13C6 소르비톨 및/또는 13C5-15N 발린, 10μM에서)를 함유한 100% 저온(-20°C) LC 등급 메탄올 1mL를 건조된 호스트 물질에 추가합니다.
- 20초 동안 혼합하는 소용돌이. 플로팅 튜브 홀더에 넣고 4 ° C로 설정된 초음파 처리 수조에서 30 분 동안 놓습니다.
9. 대사 산물 추출물 정화
- 샘플(holobiont/host/symbiont)을 3,000× g 에서 4°C에서 30분 동안 원심분리합니다.
- 모든 상층액을 새로운 2mL 미세 원심분리기 튜브로 옮기고 세포 파편 펠릿을 방해하지 않도록 주의합니다.
참고 : 이들은 반 극성 추출물입니다. 이들은 일시적으로 얼음 위에 보관할 수 있지만 어둠 속에서 -80 °C에서 장기간 보관할 수 있습니다. - 나머지 세포 파편에 1,000μL의 50% 저온(-20°C) 메탄올을 추가합니다. 1분 동안 세게 소용돌이치면 다시 매립됩니다.
- 샘플을 3,000× g 에서 4°C에서 30분 동안 원심분리합니다.
- 동일한 샘플에서 상층액(극성 추출물)을 반극성 추출물과 함께 수집하고 합산합니다.
참고: 세포 파편은 -80°C에서 보관할 수 있으며 단백질 함량 정규화에 사용할 수 있습니다(섹션 11). - 고인 추출물을 16,100g에서 15분 동안 원심 분리하여 모든 침전물을 제거하고 상층액을 가소제가 없는 새로운 마이크로 원심분리기 튜브(2mL)로 옮깁니다.
참고: 샘플 추출물은 어두운 곳에서 -80°C에서 보관할 수 있습니다. - 분석할 준비가 되면 각 추출물 50μL를 유리 삽입물에 분주합니다. 진공 농축기를 사용하여 30°C에서 30분 동안 농축합니다. 4회 더 반복합니다(총 건조 추출물 250μL).
참고: 건조된 샘플은 분석할 때까지 건조제 조건에서 실온에 보관할 수 있습니다.
10. 대사 산물 유도체화
참고 : 극성 대사 산물의 메톡시메이션(methoximation) 및 트리메틸실릴화(trimethylsilylation)를 위해 2단계 온라인 유도체화 공정이 사용됩니다.
- 각 샘플에 25μL의 메톡시아민 염산염(피리딘 30mg/mL)을 추가합니다.
- 37°C에서 750rpm으로 설정된 오비탈 셰이커에서 2시간 동안 교반합니다.
- 각 샘플에 25μL의 N,O-bis(트리메틸실릴)트리플루오로아세트아미드 + 트리메틸클로로실란을 추가합니다.
- 37°C 및 750rpm에서 1시간 동안 다시 교반합니다.
- 시료를 실온에서 1시간 동안 평형화한 후 GC에 1:10 분할 비율로 1μL를 주입합니다.
11. 가스 크로마토그래피-질량 분석
알림: 질량 분석기는 트리스-(퍼플루오로부틸)-아민(CF43)을 사용하여 제조업체의 권장 사항에 따라 조정해야 합니다.
- 초고순도 헬륨을 1mL/분의 일정한 컬럼 유속으로 운반 가스로 사용합니다.
- 필름 두께가 1μm이고 내부 직경이 0.25mm인 30m DB-5 컬럼을 사용합니다.
- GC 오븐 프로그램
- 입구 온도를 280°C로 설정합니다.
- 오븐 온도 100°C로 주입을 시작하고 4분 동안 유지합니다.
- 온도를 10°C/분 320°C로 높인 다음 11분 동안 유지합니다.
- 질량분석기 파라미터
- MS 이송 라인을 280°C로 설정하고 이온 소스를 200°C로 조정합니다.
- 아르곤을 충돌 셀 가스로 사용하여 다중 반응 모니터링(MRM) 생성물 이온을 생성합니다.
- MRM 표적을 포함하는 시간 분할 MRM 라이브러리를 기준으로 대사체 검출을 달성합니다.
12. Symbiodiniaceae 세포 수, 산호 숙주 조직 단백질 함량 분석 및 엽록소 추정
- Symbiodiniaceae 세포 수:
- 산호 조직 균질액의 분취액을 취하십시오.
- 샘플을 2,000× g 으로 원심분리하여 조류를 펠릿화합니다.
- 조류 펠릿에서 ~200μL 상층액을 제거하고 새 미세 원심분리기 튜브에 넣습니다.
참고: 이것은 데이터를 정규화하는 데 사용되는 단백질 샘플입니다. 필요한 경우 분석하기 전에 -20 °C에서 보관하십시오. - 여과된 해수 1mL에 조류 펠릿을 위아래로 부드럽게 피펫팅하여 재현탁시킵니다. 필요한 경우 세포 계수를 용이하게 하기 위해 조류 현탁액을 희석합니다.
- 챔버 중 하나에 10μL를 추가하여 광학 현미경 아래에서 혈구계를 사용하여 세포 계수를 수행합니다. 샘플당 8-10개의 카운트를 완료합니다.
참고: 조류 세포를 계수하기 위한 대체 방법도 가능한 경우 적용할 수 있습니다(예: 유세포 분석, 고처리량 공초점 현미경 검사). - 사용된 희석 계수를 고려하여 공생체 세포의 농도(mL−1)를 계산합니다.
- 단백질 함량 분석
- 샘플 단백질 함량을 정량화합니다(예: Bradford et al.33에 의해 처음 기술된 Bradford의 비색 분석 또는 다른 곳에서 cnidarians에 대해 설명된 프로토콜인 Lowry 분석(34,35)을 통해).
- 엽록소 추출
- ~ 200, 000 세포, 냉동 또는 신선의 세포 펠릿을 사용하십시오.
- 각 조류 펠릿을 유리 섬광 바이알에 담긴 2mL의 디메틸포름아미드(DMF)에 옮기고 어두운 곳에서 48시간 동안 4°C에서 배양합니다.
참고: DMF는 독성이 있고 발암성이 있으므로 샘플 준비는 가능한 한 어두운 흄 후드 아래에서 얼음 위에서 완료해야 합니다. 셀이 <200,000개인 경우 DMF를 더 적게 사용합니다. - 16,000 × g에서 3분 동안 원심분리합니다.
- 측광 측정을 위해 200mL를 UV-96 웰 플레이트로 옮깁니다. DMF를 공백으로 사용하여 각 샘플을 삼중으로 실행합니다.
- 파장 (E) 663.8nm, 646nm 및 750nm에서 흡광도를 측정합니다. 다른 두 파장의 흡광도에서 750nm의 흡광도를 뺍니다.
참고: 750nm에서 측정하면 샘플의 산란 또는 탁도가 보정됩니다. - 방정식 (1)을 사용하여 엽록소 농도(μg/mL)를 계산합니다.
Chl 농도 (μg/mL) = (12.00 × E663.8) - (3.11 × E 646.8) (1)
13. 정상화를 위한 대사산물 추출 후 세포 바이오매스의 정량화
참고: 아래에 설명된 세포 바이오매스의 정량화에는 수정된 Bradford 비색법을 사용한 바이오매스와 관련된 단백질의 정량화와 세포 파편 건조 중량 측정의 두 가지 옵션이 있습니다. 두 방법 모두 세포 바이오매스의 정확한 정량화를 제공하므로 두 방법 모두 사용하기에 적합합니다.
- 세포 파편의 단백질 함량
- 동결된 세포 파편을 1mL의 0.2M NaOH로 재현탁시키고 샘플을 98°C에서 20분 동안 배양합니다.
- 샘플을 얼음 위에서 ~10분 동안 식히고 주변 온도에서 3,000× g 에서 5분 동안 원심분리합니다.
- 샘플 단백질 함량을 정량화합니다(예: Bradford et al.33에 의해 처음 설명되고 Smart et al.37에 의해 수정된 Bradford의 비색 분석을 통해).
- 세포 파편 건조 중량 측정
- 세포 내 대사 산물 추출에서 세포 파편을 이중 증류수(~10mL)에 재현탁시킵니다.
- 사전 계량된 멤브레인 필터(0.22μm 기공, 47mm)를 사용하여 진공 상태에서 용액을 여과합니다.
- 바이오매스가 포함된 튜브를 초순수로 두 번 세척하여 바이오매스가 멤브레인 필터로 완전히 전달되도록 합니다.
- 바이오매스가 포함된 멤브레인 필터를 제거하고 전자레인지(저전력, ~250W에서 20분)를 사용하여 건조시킵니다.
- 여과지를 데시케이터에 밤새 보관하십시오. 여과지의 건조 중량을 기록하고 총 중량에서 건식 멤브레인 필터(샘플 필터와 함께 건조된 깨끗한 건식 멤브레인 필터 사용)의 무게를 빼서 바이오매스의 건조 중량을 계산합니다.
14. 데이터 분석
- 각 표적이 정량자 및 한정자 MRM으로 구성된 대사체 데이터베이스를 사용하여 대사체 표적을 분석합니다.
- 검출된 대사산물 표적을 육안으로 검사하고 필요에 따라 수동으로 통합합니다.
- 대사산물 피크 면적을 사용하여 각 그룹에 대한 각 샘플의 상대적 존재도를 계산합니다. 값은 공백으로 수정되고 정규화되어 내부 표준 피크 영역을 샘플링한 다음 Smart et al.37에 따라 세포 파편 단백질 함량을 샘플링합니다.
- 모든 처리군에서 상대 표준 편차가 35%를 초과하는 대사 산물(N = 23 대사 산물)을 폐기합니다.
- 데이터(예: 세제곱근)를 변환하고 평균 중심화합니다. 분산의 정규 분포와 균질성을 확인합니다.
- 데이터 분석(분산 분석 및 히트맵 구성, 예: https://www.metaboanalyst.ca 사용)38을 수행합니다. 샘플을 군집화하여 "cluster", "factoextra" 및 "klustR" 패키지를 사용하여 처리 내 변동성을 조사합니다. 갭 통계량(최적의 군집 수를 결정하는 방법)을 R의 "clusGap" 함수를 사용하여계산하고, R 패키지 "tidyverse"를 사용하여 플롯한다. PERMANOVA를 수행하여 처리 대사 산물 프로파일 간의 분리에서 유의성을 조사합니다(예: Primer에서).
Access restricted. Please log in or start a trial to view this content.
결과
이 작업 중에 생성된 모든 데이터는 보충 정보에서 사용할 수 있습니다.
숙주-공생체 분리
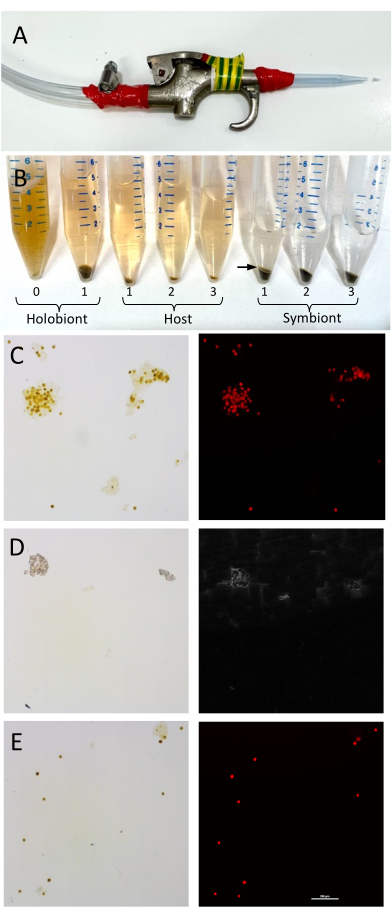
그림 1: 산호 숙주 조직과 Symbiodiniaceae 세포의 분리 설정 및 검증. (A) 산호 골격에서 산호 조직을 제거하기 ?...
Access restricted. Please log in or start a trial to view this content.
토론
숙주와 공생체의 분리는 간단한 원심분리를 통해 쉽고 빠르게 달성할 수 있으며, 여기의 결과는 분획을 분리하는 것이 특정 홀로비온트 구성원 기여를 나타내는 귀중한 정보를 제공할 수 있음을 보여주며, 이는 산호 건강의 기능적 분석에 기여할 수 있습니다. 성체 산호에서, 지질 합성은 주로 상주 조류 공생체(40)에 의해 수행되는데, 이 공생체는 지질(예를 들어, 트리아실글리세롤 및 인?...
Access restricted. Please log in or start a trial to view this content.
공개
저자는 공개할 이해 상충이 없습니다.
감사의 말
J.L.M.은 UTS Chancellor's Research Fellowship의 지원을 받았습니다.
Access restricted. Please log in or start a trial to view this content.
자료
| Name | Company | Catalog Number | Comments |
| 100% LC-grade methanol | Merck | 439193 | LC grade essential |
| 2 mL microcentrifuge tubes, PP | Eppendorf | 30121880 | Polypropylene provides high resistance to chemicals, mechanical stress and temperature extremes |
| 2030 Shimadzu gas chromatograph | Shimadzu | GC-2030 | |
| 710-1180 µm acid-washed glass beads | Merck | G1152 | This size is optimal for breaking the Symbiodiniaceae cells |
| AOC-6000 Plus Multifunctional autosampler | Shimadzu | AOC6000 | |
| Bradford reagent | Merck | B6916 | Any protein colourimetric reagent is acceptable |
| Compressed air gun | Ozito | 6270636 | Similar design acceptable. Having a fitting to fit a 1 mL tip over is critical. |
| DB-5 column with 0.25 mm internal diameter column and 1 µm film thickness | Agilent | 122-5013 | |
| DMF | Merck | RTC000098 | |
| D-Sorbitol-6-13C and/or 13C5–15N Valine | Merck | 605514/ 600148 | Either or both internal standards can be added to the methanol. |
| Flat bottom 96-well plate | Merck | CLS3614 | |
| Glass scintillation vials | Merck | V7130 | 20 mL, with non-plastic seal |
| Immunoglogin G | Merck | 56834 | if not availbe, Bovine Serum Albumin is acceptable |
| Primer | v4 | ||
| R | v4.1.2 | ||
| Shimadzu LabSolutions Insight software | v3.6 | ||
| Sodium Hydroxide | Merck | S5881 | Pellets to make 1 M solution |
| tidyverse | v1.3.1 | R package | |
| TissueLyser LT | Qiagen | 85600 | Or similar |
| TQ8050NX triple quadrupole mass spectrometer | Shimadzu | GCMS-TQ8050 NX | |
| UV-96 well plate | Greiner | M3812 | |
| Whirl-Pak sample bag | Merck | WPB01018WA | Sample collection bag; Size: big enough to house a ~5 cm coral fragment, but not too big that the water is too spread |
참고문헌
- Bundy, J. G., Davey, M. P., Viant, M. R. Environmental metabolomics: A critical review and future perspectives. Metabolomics. 5 (1), 3-21 (2008).
- Matthews, J. L., et al. The metabolic significance of symbiont community composition in the coral-algal symbiosis. Applied Environmental Metabolomics. Beale, D. J., Hillyer, K. E., Warden, A. C., Jones, O. A. H. , Academic Press. Cambridge, MA. 211-229 (2022).
- Lawson, C. A., et al. Informing coral reef conservation through metabolomic approaches. Coral Reef Conservation and Restoration in the Omics Age. Coral Reefs of the World. van Oppen, M. J. H., Aranda Lastra, M. , Springer. Cham, Switzerland. 179-202 (2022).
- LaJeunesse, T. C., et al. Systematic revision of Symbiodiniaceae highlights the antiquity and diversity of coral endosymbionts. Current Biology. 28 (16), 2570-2580 (2018).
- Rohwer, F., Seguritan, V., Azam, F., Knowlton, N. Diversity and distribution of coral-associated bacteria. Marine Ecology Progress Series. 243, 1-10 (2002).
- Maire, J., et al. Intracellular bacteria are common and taxonomically diverse in cultured and in hospite algal endosymbionts of coral reefs. The ISME Journal. 15 (7), 2028-2042 (2021).
- Hillyer, K. E., et al. Metabolite profiling of symbiont and host during thermal stress and bleaching in the coral Acropora aspera. Coral Reefs. 36, 105-118 (2016).
- Hillyer, K. E., Tumanov, S., Villas-Bôas, S., Davy, S. K. Metabolite profiling of symbiont and host during thermal stress and bleaching in a model cnidarian-dinoflagellate symbiosis. Journal of Experimental Biology. 219 (4), 516-527 (2016).
- Matthews, J. L., et al. Optimal nutrient exchange and immune responses operate in partner specificity in the cnidarian-dinoflagellate symbiosis. Proceedings of the National Academy of Sciences of the United States of America. 114 (50), 13194-13199 (2017).
- Rosset, S. L., et al. The molecular language of the cnidarian-dinoflagellate symbiosis. Trends in Microbiology. 29 (4), 320-333 (2020).
- Matthews, J. L., et al. Partner switching and metabolic flux in a model cnidarian-dinoflagellate symbiosis. Royal Society. 285 (1892), 20182336(2018).
- González-Pech, R. A., et al. Physiological factors facilitating the persistence of Pocillopora aliciae and Plesiastrea versipora in temperate reefs of south-eastern Australia under ocean warming. Coral Reefs. 41, 1239-1253 (2022).
- Williams, A., et al. Metabolomic shifts associated with heat stress in coral holobionts. Science Advances. 7 (1), (2021).
- Deutsch, J. M., et al. Metabolomics of healthy and stony coral tissue loss disease affected Montastraea cavernosa corals. Frontiers in Marine Science. 8, 1421(2021).
- Stien, D., et al. A unique approach to monitor stress in coral exposed to emerging pollutants. Scientific Reports. 10 (1), 9601(2020).
- Lohr, K. E., et al. Resolving coral photoacclimation dynamics through coupled photophysiological and metabolomic profiling. Journal of Experimental Biology. 222 (8), (2019).
- Jorissen, H., et al. Coral larval settlement preferences linked to crustose coralline algae with distinct chemical and microbial signatures. Scientific Reports. 11 (1), 14610(2021).
- Roach, T. N., Dilworth, J., Jones, A. D., Quinn, R. A., Drury, C. Metabolomic signatures of coral bleaching history. Nature Ecology & Evolution. 5 (4), 495-503 (2021).
- Parkinson, J. E., et al. Molecular tools for coral reef restoration: Beyond biomarker discovery. Conservation Letters. 13 (1), 12687(2020).
- Jiang, J., et al. How Symbiodiniaceae meets the challenges of life during coral bleaching. Coral Reefs. 40, 1339-1353 (2021).
- Guerra, F. D., Attia, M. F., Whitehead, D. C., Alexis, F. Nanotechnology for environmental remediation: materials and applications. Molecules. 23 (7), 1760(2018).
- Matthews, J. L., et al. Metabolite pools of the reef building coral Montipora capitata are unaffected by Symbiodiniaceae community composition. Coral Reefs. 39, 1727-1737 (2020).
- Papina, M., Meziane, T., van Woesik, R. Symbiotic zooxanthellae provide the host-coral Montipora digitata with polyunsaturated fatty acids. Comparative Biochemistry and Physiology Part B: Biochemistry and Molecular Biology. 135 (3), 533-537 (2003).
- Kellogg, R., Patton, J. Lipid droplets, medium of energy exchange in the symbiotic anemone Condylactis gigantea: A model coral polyp. Marine Biology. 75, 137-149 (1983).
- Ankrah, N. Y., Chouaia, B., Douglas, A. E. The cost of metabolic interactions in symbioses between insects and bacteria with reduced genomes. mBio. 9 (5), e01433(2018).
- Kabeya, N., et al. Genes for de novo biosynthesis of omega-3 polyunsaturated fatty acids are widespread in animals. Science Advances. 4 (5), (2018).
- Hillyer, K. E., Dias, D., Lutz, A., Roessner, U., Davy, S. K. 13C metabolomics reveals widespread change in carbon fate during coral bleaching. Metabolomics. 14 (1), 12(2018).
- Hillyer, K. E., Dias, D. A., Lutz, A., Roessner, U., Davy, S. K. Mapping carbon fate during bleaching in a model cnidarian symbiosis: the application of 13C metabolomics. New Phytologist. 214 (4), 1551-1562 (2017).
- Burriesci, M. S., Raab, T. K., Pringle, J. R. Evidence that glucose is the major transferred metabolite in dinoflagellate-cnidarian symbiosis. Journal of Experimental Biology. 215 (19), 3467-3477 (2012).
- Thurber, R. V., et al. Unified methods in collecting, preserving, and archiving coral bleaching and restoration specimens to increase sample utility and interdisciplinary collaboration. PeerJ. 10, 14176(2022).
- Grottoli, A. G., et al. Increasing comparability among coral bleaching experiments. Ecological Applications. 31 (4), 02262(2020).
- Mushtaq, M. Y., Choi, Y. H., Verpoorte, R., Wilson, E. G. Extraction for metabolomics: access to the metabolome. Phytochemical Analysis. 25 (4), 291-306 (2014).
- Bradford, M. M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Analytical Biochemistry. 72 (1), 248-254 (1976).
- Peterson, G. L., et al. A simplification of the protein assay method of Lowry et al. which is more generally applicable. Analytical Biochemistry. 83 (2), 346-356 (1977).
- Lowry, O. H., Rosebrough, N. J., Farr, A. L., Randall, R. J. Protein measurement with the Folin phenol reagent. Journal of Biological Chemistry. 193 (1), 265-275 (1951).
- Zamer, W. E., Shick, J. M., Tapley, D. W. Protein measurement and energetic considerations: Comparisons of biochemical and stoichiometric methods using bovine serum albumin and protein isolated from sea anemones. Limnology and Oceanography. 34 (1), 256-263 (1989).
- Smart, K. F., Aggio, R. B., Van Houtte, J. R., Villas-Boas, S. G. Analytical platform for metabolome analysis of microbial cells using methyl chloroformate derivatization followed by gas chromatography-mass spectrometry. Nature Protocols. 5 (10), 1709-1729 (2010).
- Pang, Z., et al. Using MetaboAnalyst 5.0 for LC-HRMS spectra processing, multi-omics integration and covariate adjustment of global metabolomics data. Nature Protocols. 17 (8), 1735-1761 (2022).
- Tibshirani, R., Walther, G., Hastie, T. Estimating the number of clusters in a data set via the gap statistic. Journal of the Royal Statistical Society: Series B (Statistical Methodology). 63 (2), 411-423 (2001).
- Chen, W. -N., et al. Diel rhythmicity of lipid-body formation in a coral-Symbiodinium endosymbiosis). Coral Reefs. 31 (2), 521-534 (2012).
- Imbs, A. Fatty acids and other lipids of corals: composition, distribution, and biosynthesis. Russian Journal of Marine Biology. 39 (3), 153-168 (2013).
- Rosset, S., et al. Lipidome analysis of Symbiodiniaceae reveals possible mechanisms of heat stress tolerance in reef coral symbionts. Coral Reefs. 38 (6), 1241-1253 (2019).
- Carreón-Palau, L., Parrish, C. C., Del Angel-Rodriguez, J. A., Perez-Espana, H. Seasonal shifts in fatty acids and sterols in sponges, corals, and bivalves, in a southern Gulf of Mexico coral reef under river influence. Coral Reefs. 40 (2), 571-593 (2021).
- Imbs, A. B., Dang, L. T. Seasonal dynamics of fatty acid biomarkers in the soft coral Sinularia flexibilis, a common species of Indo-Pacific coral reefs. Biochemical Systematics and Ecology. 96, 104246(2021).
- Oku, H., Yamashiro, H., Onaga, K., Sakai, K., Iwasaki, H. Seasonal changes in the content and composition of lipids in the coral Goniastrea aspera. Coral Reefs. 22 (1), 83-85 (2003).
- Weis, V. M. Cell biology of coral symbiosis: foundational study can inform solutions to the coral reef crisis. Integrative and Comparative Biology. 59 (4), 845-855 (2019).
- Oakley, C., Davy, S. Cell biology of coral bleaching. Coral Bleaching. van Oppen, M., Lough, J. , Springer. Cham, Switzerland. 189-211 (2018).
- Lu, W., et al. Metabolite measurement: Pitfalls to avoid and practices to follow. Annual Review of Biochemistry. 86, 277-304 (2017).
- Lawson, C. A., et al. Heat stress decreases the diversity, abundance and functional potential of coral gas emissions. Global Change Biology. 27 (4), 879-891 (2021).
- Olander, A., et al. Comparative volatilomics of coral endosymbionts from one-and comprehensive two-dimensional gas chromatography approaches. Marine Biology. 168 (5), 76(2021).
- Wuerz, M., et al. Symbiosis induces unique volatile profiles in the model cnidarian Aiptasia. Journal of Experimental Biology. 225 (19), (2022).
Access restricted. Please log in or start a trial to view this content.
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유