È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Methods Article
Gascromatografia-Metabolomica mirata basata sulla spettrometria di massa di campioni di corallo duro
In questo articolo
Riepilogo
Qui, presentiamo l'estrazione e la preparazione di metaboliti polari e semi-polari da un olobionte di corallo, nonché tessuto ospite di corallo separato e frazioni cellulari di Symbiodiniaceae, per l'analisi gascromatografica-spettrometria di massa.
Abstract
Gli approcci basati sulla gascromatografia-spettrometria di massa (GC-MS) si sono dimostrati potenti per chiarire le basi metaboliche della simbiosi cnidario-dinoflagellata e il modo in cui il corallo risponde allo stress (ad esempio, durante lo sbiancamento indotto dalla temperatura). Il profilo dei metaboliti allo stato stazionario dell'olobionte del corallo, che comprende l'ospite cnidariano e i suoi microbi associati (Symbiodiniaceae e altri protisti, batteri, archaea, funghi e virus), è stato applicato con successo in condizioni ambientali e di stress per caratterizzare lo stato metabolico olistico del corallo.
Tuttavia, per rispondere alle domande che circondano le interazioni simbiotiche, è necessario analizzare i profili dei metaboliti dell'ospite corallo e dei suoi simbionti algali in modo indipendente, il che può essere ottenuto solo mediante separazione fisica e isolamento dei tessuti, seguita da estrazione e analisi indipendenti. Mentre l'applicazione della metabolomica è relativamente nuova nel campo dei coralli, gli sforzi sostenuti dei gruppi di ricerca hanno portato allo sviluppo di metodi robusti per l'analisi dei metaboliti nei coralli, compresa la separazione del tessuto ospite del corallo e dei simbionti algali.
Questo documento presenta una guida passo-passo per la separazione degli olobionti e l'estrazione dei metaboliti per l'analisi GC-MS, comprese le principali fasi di ottimizzazione da prendere in considerazione. Dimostriamo come, una volta analizzato in modo indipendente, il profilo metabolitico combinato delle due frazioni (corallo e Symbiodiniaceae) sia simile al profilo dell'insieme (olobionte), ma separando i tessuti, possiamo anche ottenere informazioni chiave sul metabolismo e sulle interazioni tra i due partner che non possono essere ottenute dal solo tutto.
Introduzione
I metaboliti rappresentano i prodotti finali dei processi cellulari e la metabolomica - lo studio della serie di metaboliti prodotti da un determinato organismo o ecosistema - può fornire una misura direttadel funzionamento dell'organismo. Ciò è particolarmente importante per l'esplorazione degli ecosistemi, delle interazioni simbiotiche e degli strumenti di ripristino, poiché l'obiettivo della maggior parte delle strategie di gestione è quello di preservare (o ripristinare)specifiche funzioni dei servizi ecosistemici. Le barriere coralline sono un ecosistema acquatico che dimostra il valore potenziale della metabolomica per chiarire le interazioni simbiotiche e collegare le risposte fisiologiche dei coralli agli impatti a livello di comunità e di ecosistema3. L'applicazione della gascromatografia-spettrometria di massa ad alto rendimento (GC-MS) è particolarmente apprezzata per la sua capacità di analizzare rapidamente un'ampia gamma di classi di metaboliti contemporaneamente con elevata selettività e sensibilità, fornire una rapida identificazione dei composti quando sono disponibili librerie spettrali e fornire un elevato livello di riproducibilità e accuratezza, con un costo per campione relativamente basso.
I coralli sono olobionti costituiti dall'animale corallo, dagli endosimbionti dinoflagellati fotosintetici (famiglia: Symbiodiniaceae4) e da un microbioma complesso 5,6. Nel complesso, la fitness dell'olobionte viene mantenuta principalmente attraverso lo scambio di piccole molecole ed elementi per supportare il funzionamento metabolico di ciascun membro 7,8,9,10. Gli approcci metabolomici si sono dimostrati particolarmente efficaci per chiarire le basi metaboliche della specificità della simbiosi9,11, la risposta allo sbiancamento allo stress termico 7,8,12,13, le risposte alle malattie 14, le risposte all'esposizione all'inquinamento 15, la fotoacclimatazione16 e la segnalazione chimica 17 nei coralli, oltre a contribuire alla scoperta di biomarcatori 18,19. Inoltre, la metabolomica può fornire una preziosa conferma delle conclusioni dedotte dalle tecniche basate sul DNA e sull'RNA 9,20. Esiste, quindi, un notevole potenziale per l'uso della metabolomica per valutare la salute della barriera corallina e sviluppare strumenti per la conservazione della barriera corallina3, ad esempio attraverso l'individuazione di biomarcatori metabolici di stress18,19 e per esaminare il potenziale di strategie di gestione attiva come i sussidi nutrizionali21.
Separare le cellule ospite e simbionte e analizzare i loro profili metabolitici in modo indipendente, piuttosto che insieme come olobionte, può fornire maggiori informazioni sulle interazioni del partner, sugli stati fisiologici e metabolici indipendenti e sui potenziali meccanismi molecolari per l'adattamento 11,12,22,23,24. Senza separare il corallo dalle Symbiodiniaceae, è quasi impossibile chiarire il contributo e il metabolismo dei coralli e/o delle Symbiodiniaceae in modo indipendente, tranne che con la ricostruzione complessa del genoma e la modellazione metabolica25, ma questo deve ancora essere applicato alla simbiosi corallo-dinoflagellato. Inoltre, il tentativo di estrarre informazioni sul metabolismo individuale del simbionte ospite o algale dal profilo metabolitico dell'olobionte può portare a interpretazioni errate.
Ad esempio, fino a poco tempo fa, si pensava che la presenza di acidi grassi polinsaturi C18:3n-6, C18:4n-3 e C16 negli estratti di tessuti di corallo e olobionte derivasse dal simbionte algale, poiché si presumeva che i coralli non possedessero le desaturasi ωx essenziali per la produzione di acidi grassi omega-3 (ω3); tuttavia, recenti evidenze genomiche suggeriscono che più cnidari hanno la capacità di produrre ω3 PUFA de novo e di biosintetizzare ulteriormente ω3 PUFA26 a catena lunga. La combinazione di GC-MS con marcatura isotopica stabile (ad esempio, 13 C-bicarbonato, NaH13CO 3 ) può essere utilizzata per tracciare il destino del carbonio fissato fotosinteticamente attraverso le reti metaboliche degli olobionti corallini sia in condizioni di controllo che in risposta a fattori di stress esterni27,28. Tuttavia, un passo critico nel monitoraggio del destino del 13 C è la separazione del tessuto del corallo dalle cellule algali: solo allora la presenza di un composto marcato con 13C nella frazione ospite del corallo può essere inequivocabilmente assegnata come un metabolita derivato dalle Symbiodiniaceae traslocato nel corallo o un prodotto a valle di un composto marcato traslocato. Questa tecnica ha dimostrato la sua potenza sfidando l'ipotesi di lunga data che il glicerolo sia la forma primaria in cui il fotosintato, traslocato dal simbionte all'ospite29, oltre a chiarire come cambia il flusso nutrizionale tra i partner durante lo sbiancamento27,28 e in risposta a specie di Symbiodiniaceae incompatibili11.
Sebbene la decisione di separare i tessuti sia guidata principalmente dalla domanda di ricerca, la praticità, l'affidabilità e i potenziali impatti metabolici di questo approccio sono importanti da considerare. Qui, forniamo metodi dettagliati e dimostrati per l'estrazione dei metaboliti dall'olobionte, nonché dalle frazioni separate dell'ospite e del simbionte. Confrontiamo i profili dei metaboliti dell'ospite e del simbionte in modo indipendente e come questi profili si confrontano con il profilo del metabolita dell'olobionte.
Protocollo
NOTA: Il disegno sperimentale, la raccolta e la conservazione dei campioni sono stati descritti in dettaglio altrove 2,30,31. L'autorizzazione per la raccolta di coralli selvatici deve essere ottenuta prima della raccolta e della sperimentazione. I campioni qui riportati sono stati raccolti da colonie di Montipora mollis (colore verde) importate da Batavia Coral Farms (Geraldton, WA), originariamente raccolte da una barriera corallina al largo delle isole Abrohlos (Australia occidentale; 28°52'43.3"S 114°00'17.0"E) ad una profondità di 1 m nell'ambito della licenza di acquacoltura AQ1643. Prima del campionamento, le colonie sono state tenute in un acquario da 800 L a 35 PSU, sotto luce blu e bianca a 150 μmol di fotoni·m−2·s−1 e a 25 °C ± 0,5 °C per 3 mesi. I frammenti di corallo (~5 cm2, N = 6) sono stati congelati in azoto liquido e conservati a -80 °C fino alla lavorazione.
1. Preparazione delle soluzioni e delle attrezzature di estrazione
- Almeno 1 giorno prima della rimozione del tessuto corallino, preparare le soluzioni e le attrezzature per l'estrazione.
- Preraffreddare l'acqua ultrapura in vetreria pulita e priva di detergenti a 4 °C.
- Miscelare metanolo di grado LC al 100% con una concentrazione finale di 10 μg·mL−1 di uno o più standard interni appropriati (ad esempio, 13C6 sorbitolo).
- Creare una soluzione di estrazione del metanolo al 50% utilizzando metà metanolo di grado LC al 100% e metà acqua ultrapura. Conservare entrambe le soluzioni di metanolo a una temperatura di −20 °C.
NOTA: Per aiutare a prevenire la degradazione dei metaboliti, si consiglia di eseguire le fasi di lavorazione del campione in lotti di cinque frammenti di corallo alla volta, con un bianco biologico aggiuntivo (solo acqua) (campioni totali N = 6). Una volta che ogni campione di corallo è stato separato nelle due frazioni (tessuto ospite di corallo, d'ora in poi "Ospite", e cellule microalgali, d'ora in poi "Simbionte"), il numero totale di campioni in un lotto di lavorazione sarà 12.
2. Dissetamento del metabolismo dei coralli
NOTA: Il disegno sperimentale, la raccolta e la conservazione dei campioni sono stati descritti in dettaglio altrove 2,30,31. Tuttavia, va notato che il tempo impiegato per estinguere il metabolismo (cioè il tempo che intercorre tra la raccolta del campione di corallo e la conservazione) è fondamentale per catturare la risposta originale30. Conservare il campione il più rapidamente possibile dopo la raccolta per evitare cambiamenti nella composizione del metabolita dovuti alla degradazione del campione o a risposte fisiologiche non bersaglio32.
- Mettere il frammento di corallo in una sacca sterile per la raccolta dei campioni e scolare il più possibile l'acqua di mare in eccesso. Immergere il campione in azoto liquido per un minimo di 30 s. Spostare i campioni il prima possibile in un congelatore a -80 °C per la conservazione.
NOTA: I campioni possono essere congelati a -80°C in contenitori ostruiti dalla luce fino al trattamento, evitando cicli di gelo-scongelamento.
3. Rimozione del tessuto corallino dallo scheletro
NOTA: I campioni devono essere tenuti sempre in ghiaccio (4 °C) per garantire che siano contemporaneamente in forma liquida e prevenire il metabolismo in corso.
- Posizionare una sacca di raccolta del campione pulita e sterile sul ghiaccio in modo che la sacca sia stabile e aperta sopra il ghiaccio in un pozzetto poco profondo ma non sia immersa nel ghiaccio. Aggiungere 10 ml di acqua ultrapura fredda (4 °C) nel sacchetto.
NOTA: Ciò contribuirà a evitare ripetuti congelamenti del frammento di corallo a causa dell'aria fredda pressurizzata e del ghiaccio circostante. - Selezionare un frammento di corallo con una pinzetta sterilizzata e risciacquare con acqua ultrapura fredda (4 °C) utilizzando una pipetta Pasteur sterile fino a quando non rimangono residui di acqua di mare. Immergere il frammento di corallo risciacquato nel sacchetto contenente i 10 ml di acqua ultrapura.
NOTA: Questo risciacquo è fondamentale per rimuovere eventuali sali residui che interferirebbero con l'analisi a valle. Evitare il contatto delle mani con l'acqua o il frammento di corallo attraverso il sacchetto per mantenere il campione a 4 °C. - Fissare con nastro adesivo un puntale sterile per pipette da 1 mL sull'estremità di una pistola ad aria compressa con nastro isolante, tagliando ~5 mm all'estremità del puntale (Figura 1A).
- Puntare la pistola ad aria compressa sul frammento di corallo con il sacchetto semisigillato e il flusso d'aria su medio-basso per rimuovere delicatamente il tessuto incoraggiando un movimento circolare dell'acqua sul frammento di corallo.
- Dopo ~ 3 minuti, o quando tutto il tessuto sembra essere stato rimosso dallo scheletro, spegnere l'aria e rimuovere l'aerografo. Sigillare completamente il sacchetto.
- Spremere tutto il tessuto di corallo rimosso in un angolo inferiore del sacchetto. Tagliare l'angolo opposto e versare delicatamente il contenuto della busta in una provetta da 15 ml su ghiaccio.
4. Omogeneizzazione opzionale
NOTA: Alcune specie di corallo sono più viscose di altre, il che significa che l'aerografia rimuoverà il tessuto in ciuffi invece che in un liquame. Se nell'omogenato aerografato sono visibili grumi di tessuto, è possibile aggiungere una fase di omogeneizzazione a 4 °C per tutti i campioni.
- Pulire due volte un omogeneizzatore meccanico a dente di sega con metanolo al 70% a 4 °C e infine con acqua ultrapura a 4 °C.
- Omogeneizzare il campione di corallo in una provetta da 15 mL per ~1 minuto fino a quando il campione non è completamente omogeneizzato e non sono visibili grumi.
- Pulire l'omogeneizzatore come al punto 4.1 tra un campione e l'altro. Mantenere il tempo di omogeneizzazione costante tra i campioni.
5. Raccolta di campioni per la normalizzazione
- Raccogliere un'aliquota di 1.000 μL dal tessuto omogeneizzato per la conta delle cellule di Symbiodiniaceae, l'analisi del contenuto proteico del tessuto ospite del corallo e la stima della clorofilla. Conservare a −20 °C fino al momento dell'analisi (sezione 10).
6. Separazione facoltativa delle cellule del tessuto ospite del corallo-Symbiodiniaceae
- Centrifugare l'omogenato di corallo a 2.500 × g per 5 minuti a 4 °C utilizzando una centrifuga refrigerata.
NOTA: Questa velocità è ottimale per separare le cellule più pesanti delle Symbiodiniaceae, mantenendo intatte le loro pareti cellulari, dal tessuto ospite, che è sospeso nel surnatante. - Rimuovere il surnatante contenente il materiale ospite e metterlo in una nuova provetta da 15 ml.
NOTA: I lipidi del tessuto ospite formano tipicamente uno stretto strato rosa/bianco sopra le cellule simbionti. Questo strato può essere raccolto insieme al surnatante solubile dell'ospite tramite pipettaggio (Figura 1B). - Agitare vigorosamente l'ostia per esattamente 1 minuto. Conservare il campione di pellet di alghe e il campione di surnatante ospite sul ghiaccio.
- Aggiungere 2 mL di acqua ultrapura a 4 °C al pellet di alghe. Agitare energicamente per 2 minuti esatti per risospendere il pellet.
NOTA: Se singoli frammenti di 1 cm non sono stati raccolti dalla colonia di corallo per la genotipizzazione delle Symbiodiniaceae, un'aliquota di 200 μL della sospensione cellulare delle Symbiodiniaceae può essere raccolta qui, conservata nella soluzione tampone di DNA preferita e conservata come descritto in Thurber et al.30 per la genotipizzazione delle Symbiodiniaceae (ad esempio, come da González-Pech et al.12). - Ripetere ancora una volta i passaggi 6.1-6.4.
NOTA: La separazione affidabile dell'ospite e del simbionte dipende dalla biomassa e dalla specie del corallo, poiché alcune specie possono essere più viscose di altre. Si consiglia un minimo di tre fasi di lavaggio, ma questo può essere aumentato a seconda del successo della separazione. Ripetere le fasi di lavaggio 4.7-4.9 fino a quando non sono visibili cellule di Symbiodiniaceae nella parte inferiore della frazione ospite e fino a quando la frazione di Symbiodiniaceae è visibilmente priva di materiale ospite (ad esempio, nessuno strato bianco in cima) (Figura 1). - Rimuovere il surnatante contenente il materiale ospite e metterlo in una nuova provetta da 15 ml.
- Conservare il pellet simbionte nella provetta da 15 ml.
7. Essiccazione del campione
- Congelare l'omogenato olobionte o entrambe le frazioni separate dell'ospite e delle Symbiodiniaceae, a −80 °C per ~120 min. Liofilizzare i campioni durante la notte con un vuoto di 0,01 mbar a -85 °C.
NOTA: Per evitare la perdita di campione durante la liofilizzazione, si consiglia di utilizzare un coperchio tagliato da un'altra provetta sterile, o parafilm sterile, con un foro di ~2 mm praticato con cura utilizzando un ago sterile da 25 G. - Una volta asciutto, utilizzando una bilancia da laboratorio, pesare uno dei seguenti elementi: 1) 25 mg di olobionte; 2) 15 mg della frazione simbionte; o 3) 30 mg del tessuto ospite da ciascun campione in provette separate per microcentrifuga da 2 mL prive di plastificanti.
NOTA: Passaggio critico: L'ottimizzazione della biomassa per l'estrazione è essenziale per garantire che il GC-MS non venga sovraccaricato garantendo al contempo un segnale sufficiente. Il materiale di corallo essiccato è molto statico. Per evitare la perdita di campioni, utilizzare dispositivi antistatici per eliminare le cariche elettrostatiche dai campioni e dai recipienti di pesata. Un'alternativa semplice ed economica è quella di posizionare un foglio di asciugatrice sotto il tubo del campione. Il pellet simbionte essiccato può essere tagliato al peso desiderato utilizzando una lama sterile.
8. Estrazioni di metaboliti intracellulari
- Estrazione di metaboliti intracellulari da olobionte liofilizzato:
- Aggiungere 400 μL di metanolo freddo al 100% (−20 °C) con standard/i interno/i (IS; 13C6 sorbitolo e/o 13C5-15 N valina, a 10μM) a ciascuna provetta.
- Aggiungere un piccolo numero di perle di vetro lavate con acido da 710-1.180 μm (~10 mg) a ciascun campione. Mettere in un mulino a perline a 50 Hz per 3 minuti in un inserto per mulino a perline preraffreddato (−20 °C).
- Aggiungere altri 600 μL di metanolo freddo al 100% (−20 °C) con IS (13 C6 sorbitolo e/o 13C5-15 N valina, a 10μM) in ciascuna provetta.
- Frullare per mescolare per 1 min. Mettere su uno scuotitore girarrosto a 4 °C per 30 min.
- Estrazione di metaboliti intracellulari da cellule liofilizzate separate di Symbiodiniaceae:
- Aggiungere 200 μL di metanolo freddo al 100% (−20 °C) con IS (13 C6 sorbitolo e/o 13C 5-15 N valina, a 10μM) al materiale essiccato delle Symbiodiniaceae.
- Aggiungere un piccolo numero di perle di vetro lavate con acido da 710-1.180 μm (~10 mg). Mettere in un mulino a perline a 50 Hz per 3 minuti in un inserto per mulino a perline preraffreddato (−20 °C).
- Aggiungere altri 800 μL di metanolo freddo al 100% (−20 °C) con IS e vorticare per 30 s.
- Estrazione di metaboliti intracellulari da tessuto ospite liofilizzato separato:
- Aggiungere 1 mL di metanolo di grado LC freddo al 100% (−20 °C) contenente SI (13 C6 sorbitolo e/o 13C 5-15 N valina, a 10μM) al materiale ospite essiccato.
- Vortice da mescolare per 20 s. Mettere in un portaprovette galleggiante in un bagno di sonicazione a 4 °C per 30 min.
9. Purificazione dell'estratto di metaboliti
- Centrifugare i campioni (olobionte/ospite/simbionte) a 3.000 × g per 30 minuti a 4 °C.
- Trasferire tutto il surnatante in una nuova provetta per microcentrifuga da 2 mL, facendo attenzione a non disturbare il pellet di detriti cellulari.
NOTA: Questi sono gli estratti semipolari. Questi possono essere conservati temporaneamente sul ghiaccio, ma conservati a lungo termine a -80 °C al buio. - Ai detriti cellulari rimanenti, aggiungere 1.000 μL di metanolo freddo al 50% (−20 °C). Agitare energicamente per 1 minuto per risuspendere.
- Centrifugare i campioni a 3.000 × g per 30 minuti a 4 °C.
- Raccogliere e raggruppare il surnatante (estratti polari) con gli estratti semipolari dello stesso campione.
NOTA: I detriti cellulari possono essere conservati a -80 °C e utilizzati per la normalizzazione del contenuto proteico (sezione 11). - Centrifugare gli estratti aggregati a 16.100 g per 15 minuti per rimuovere tutti i precipitati e spostare il surnatante in una nuova provetta per microcentrifuga priva di plastificanti (2 mL).
NOTA: Gli estratti del campione possono essere conservati a -80 °C al buio. - Al termine dell'analisi, aliquotare 50 μL di ciascun estratto in un inserto di vetro. Concentrare per 30 minuti a 30 °C utilizzando un concentratore sottovuoto. Ripetere altre quattro volte (per 250 μL di estratto secco totale).
NOTA: I campioni essiccati possono essere conservati a temperatura ambiente in condizioni di essiccazione fino all'analisi.
10. Derivatizzazione dei metaboliti
NOTA : Un processo di derivatizzazione online in due fasi viene utilizzato per la metossimazione e la trimetilsililazione dei metaboliti polari.
- Aggiungere 25 μL di metossiamina cloridrato (30 mg/mL in piridina) a ciascun campione.
- Agitare a 37 °C su un agitatore orbitale tarato a 750 giri/min per 2 ore.
- Aggiungere 25 μL di N,O-bis (trimetilsilil)trifluoroacetammide + trimetilclorosilano a ciascun campione.
- Agitare nuovamente a 37 °C e 750 giri/min per 1 ora.
- Lasciare che i campioni si equilibrino a temperatura ambiente per 1 ora prima di iniettare 1 μL in un rapporto di divisione 1:10 sul GC.
11. Gascromatografia-spettrometria di massa
NOTA: Lo spettrometro di massa deve essere regolato secondo le raccomandazioni del produttore utilizzando tris-(perfluorobutil)-ammina (CF43).
- Utilizzare elio ad altissima purezza come gas di trasporto a una portata costante della colonna di 1 mL/min.
- Utilizzare una colonna DB-5 da 30 m con uno spessore del film di 1 μm e un diametro interno di 0,25 mm.
- Programma forno GC
- Impostare la temperatura di ingresso a 280 °C.
- Iniziare con l'iniezione con una temperatura del forno di 100 °C e tenere premuto per 4 minuti.
- Aumentare la temperatura di 10 °C/min a 320 °C, quindi mantenere la temperatura per 11 min.
- Parametri dello spettrometro di massa
- Impostare la linea di trasferimento MS a 280 °C e regolare la sorgente di ioni a 200 °C.
- Utilizzare l'argon come gas della cella di collisione per generare lo ione prodotto MRM (Multiple Reaction Monitoring).
- Ottenere il rilevamento dei metaboliti rispetto a una libreria di MRM segmentata nel tempo contenente target MRM.
12. Conta delle cellule di Symbiodiniaceae, analisi del contenuto proteico del tessuto ospite del corallo e stima della clorofilla
- Conta delle cellule delle Symbiodiniaceae:
- Prelevare un'aliquota dell'omogenato di tessuto corallino.
- Centrifugare i campioni a 2.000 × g per pellettare le alghe.
- Rimuovere il surnatante da ~200 μL dal pellet algale e inserirlo in una nuova provetta per microcentrifuga.
NOTA: Questo sarà il campione proteico che verrà utilizzato per normalizzare i dati; conservare a −20 °C prima dell'analisi, se necessario. - Risospendere il pellet di alghe in 1 mL di acqua di mare filtrata pipettando delicatamente su e giù. Se necessario, diluire la sospensione algale per facilitare la conta cellulare.
- Eseguire un conteggio delle cellule utilizzando un emocitometro al microscopio ottico aggiungendo 10 μL a una delle camere. Completare 8-10 conteggi per campione.
NOTA: Ove disponibili, possono essere applicati anche metodi alternativi per il conteggio delle cellule algali (ad es. citometria a flusso, microscopia confocale ad alto rendimento). - Calcolare la concentrazione delle cellule simbionti (mL−1), tenendo conto dei fattori di diluizione utilizzati.
- Dosaggio del contenuto proteico
- Quantificare il contenuto proteico del campione (ad esempio, tramite il test colorimetrico di Bradford, come inizialmente descritto da Bradford et al.33, o il test di Lowry34,35, il cui protocollo è stato descritto per gli cnidari altrove 36).
- Clorofilla un'estrazione
- Utilizzare un pellet cellulare di ~200.000 cellule, congelato o fresco.
- Trasferire ogni pellet di alghe in 2 mL di dimetilformammide (DMF) in un flaconcino di vetro a scintillazione e incubare al buio a 4 °C per 48 ore.
NOTA: Il DMF è tossico e cancerogeno, quindi la preparazione del campione deve essere completata sotto una cappa aspirante il più scura possibile e su ghiaccio. Se ci sono < 200.000 celle, usa meno DMF. - Centrifugare per 3 minuti a 16.000 × g.
- Trasferire 200 mL in una piastra a pozzetti UV-96 per misurazioni fotometriche. Eseguire ogni campione in triplice copia con DMF come pezzo grezzo.
- Misurare l'assorbanza alle lunghezze d'onda (E) 663,8 nm, 646 nm e 750 nm. Sottrarre l'assorbanza a 750 nm dall'assorbanza a entrambe le altre lunghezze d'onda.
NOTA: La misurazione a 750 nm corregge qualsiasi dispersione o torbidità nel campione. - Calcolare la concentrazione di clorofilla a (μg/mL) utilizzando l'equazione (1):
Concentrazione di Chl a (μg/mL) = (12.00 × E 663.8) - (3.11 × E646.8) (1)
13. Quantificazione della biomassa cellulare in seguito all'estrazione di metaboliti per la normalizzazione
NOTA: Ci sono due opzioni per la quantificazione della biomassa cellulare descritte di seguito: la quantificazione della proteina correlata alla biomassa utilizzando un metodo colorimetrico Bradford modificato e la misurazione del peso secco dei detriti cellulari. Entrambi i metodi sono appropriati da utilizzare, in quanto entrambi offrono una quantificazione accurata della biomassa cellulare.
- Contenuto proteico dei detriti cellulari
- Risospendere i detriti cellulari congelati con 1 mL di NaOH 0,2 M e incubare i campioni a 98 °C per 20 minuti.
- Raffreddare i campioni su ghiaccio per ~10 minuti e centrifugare a 3.000 × g per 5 minuti a temperatura ambiente.
- Quantificare il contenuto proteico del campione (ad esempio, tramite il saggio colorimetrico di Bradford, come inizialmente descritto da Bradford et al.33 e modificato da Smart et al.37).
- Misurazione del peso secco dei detriti cellulari
- Risospendere i detriti cellulari dall'estrazione del metabolita intracellulare in acqua bidistillata (~10 mL).
- Filtrare la soluzione sotto vuoto utilizzando un filtro a membrana prepesato (poro 0,22 μm, 47 mm).
- Lavare due volte i tubi contenenti biomassa con acqua ultrapura per garantire il completo trasferimento della biomassa al filtro a membrana.
- Rimuovere il filtro a membrana contenente la biomassa e asciugarlo con un forno a microonde (bassa potenza; ~250 W per 20 min).
- Conservare la carta da filtro in un essiccatore per una notte. Registrare il peso secco della carta da filtro e calcolare il peso secco della biomassa sottraendo il peso del filtro a membrana secca (utilizzando un filtro a membrana asciutto pulito ed essiccato insieme al filtro campione) dal peso totale.
14. Analisi dei dati
- Analizza i target dei metaboliti utilizzando database di metaboliti in cui ogni target è costituito da un quantificatore e da un qualificatore MRM.
- Ispezionare visivamente i metaboliti target rilevati e integrarli manualmente in base alle esigenze.
- Utilizzare un'area del picco del metabolita per calcolare l'abbondanza relativa di ciascun campione per ciascun gruppo. I valori vengono corretti in bianco e normalizzati per campionare l'area di picco standard interno e quindi per campionare il contenuto proteico dei detriti cellulari come da Smart et al.37.
- Scartare i metaboliti con una deviazione standard relativa superiore al 35% in tutti i gruppi di trattamento (N = 23 metaboliti).
- Trasformare i dati (ad esempio, radice cubica) e centrarli nella media; confermano una distribuzione normale e omogeneità della varianza.
- Eseguire l'analisi dei dati (costruzione di ANOVA e heatmap; ad esempio, utilizzando https://www.metaboanalyst.ca)38. Raggruppare i campioni per esaminare la variabilità all'interno del trattamento utilizzando i pacchetti "cluster", "factoextra" e "klustR". Calcola la statistica del gap (un metodo per determinare il numero ottimale di cluster39) usando la funzione "clusGap" in R e i grafici usando il pacchetto R "tidyverse". Eseguire PERMANOVA per esaminare il significato della separazione tra i profili dei metaboliti del trattamento (ad esempio, in Primer).
Risultati
Tutti i dati prodotti nel corso di questo lavoro sono disponibili nelle informazioni supplementari.
Separazione ospite-simbionte
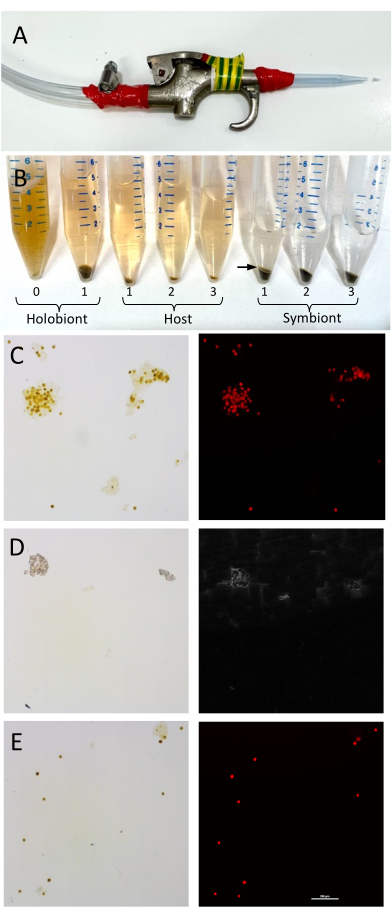
Figura 1: Messa a punto e validazione della separazione dei tessuti ospiti del corallo e delle cellule di Symbiodiniaceae. (A) La co...
Discussione
La separazione dell'ospite e del simbionte è facilmente e rapidamente ottenibile tramite una semplice centrifugazione, e i risultati qui mostrano che la separazione delle frazioni può fornire preziose informazioni indicative di specifici contributi dei membri olobionti, che possono contribuire all'analisi funzionale della salute dei coralli. Nei coralli adulti, la sintesi lipidica è eseguita principalmente dal simbionte algale residente 40, che fornisce lipidi (ad esempio, triacilglicerolo e fosfolipidi)41 e ...
Divulgazioni
Gli autori non hanno alcun conflitto di interessi da rivelare.
Riconoscimenti
J.L.M. è stato sostenuto da una borsa di studio di ricerca del Cancelliere dell'UTS.
Materiali
| Name | Company | Catalog Number | Comments |
| 100% LC-grade methanol | Merck | 439193 | LC grade essential |
| 2 mL microcentrifuge tubes, PP | Eppendorf | 30121880 | Polypropylene provides high resistance to chemicals, mechanical stress and temperature extremes |
| 2030 Shimadzu gas chromatograph | Shimadzu | GC-2030 | |
| 710-1180 µm acid-washed glass beads | Merck | G1152 | This size is optimal for breaking the Symbiodiniaceae cells |
| AOC-6000 Plus Multifunctional autosampler | Shimadzu | AOC6000 | |
| Bradford reagent | Merck | B6916 | Any protein colourimetric reagent is acceptable |
| Compressed air gun | Ozito | 6270636 | Similar design acceptable. Having a fitting to fit a 1 mL tip over is critical. |
| DB-5 column with 0.25 mm internal diameter column and 1 µm film thickness | Agilent | 122-5013 | |
| DMF | Merck | RTC000098 | |
| D-Sorbitol-6-13C and/or 13C5–15N Valine | Merck | 605514/ 600148 | Either or both internal standards can be added to the methanol. |
| Flat bottom 96-well plate | Merck | CLS3614 | |
| Glass scintillation vials | Merck | V7130 | 20 mL, with non-plastic seal |
| Immunoglogin G | Merck | 56834 | if not availbe, Bovine Serum Albumin is acceptable |
| Primer | v4 | ||
| R | v4.1.2 | ||
| Shimadzu LabSolutions Insight software | v3.6 | ||
| Sodium Hydroxide | Merck | S5881 | Pellets to make 1 M solution |
| tidyverse | v1.3.1 | R package | |
| TissueLyser LT | Qiagen | 85600 | Or similar |
| TQ8050NX triple quadrupole mass spectrometer | Shimadzu | GCMS-TQ8050 NX | |
| UV-96 well plate | Greiner | M3812 | |
| Whirl-Pak sample bag | Merck | WPB01018WA | Sample collection bag; Size: big enough to house a ~5 cm coral fragment, but not too big that the water is too spread |
Riferimenti
- Bundy, J. G., Davey, M. P., Viant, M. R. Environmental metabolomics: A critical review and future perspectives. Metabolomics. 5 (1), 3-21 (2008).
- Matthews, J. L., Beale, D. J., Hillyer, K. E., Warden, A. C., Jones, O. A. H., et al. The metabolic significance of symbiont community composition in the coral-algal symbiosis. Applied Environmental Metabolomics. , 211-229 (2022).
- Lawson, C. A., van Oppen, M. J. H., Aranda Lastra, M., et al. Informing coral reef conservation through metabolomic approaches. Coral Reef Conservation and Restoration in the Omics Age. Coral Reefs of the World. , 179-202 (2022).
- LaJeunesse, T. C., et al. Systematic revision of Symbiodiniaceae highlights the antiquity and diversity of coral endosymbionts. Current Biology. 28 (16), 2570-2580 (2018).
- Rohwer, F., Seguritan, V., Azam, F., Knowlton, N. Diversity and distribution of coral-associated bacteria. Marine Ecology Progress Series. 243, 1-10 (2002).
- Maire, J., et al. Intracellular bacteria are common and taxonomically diverse in cultured and in hospite algal endosymbionts of coral reefs. The ISME Journal. 15 (7), 2028-2042 (2021).
- Hillyer, K. E., et al. Metabolite profiling of symbiont and host during thermal stress and bleaching in the coral Acropora aspera. Coral Reefs. 36, 105-118 (2016).
- Hillyer, K. E., Tumanov, S., Villas-Bôas, S., Davy, S. K. Metabolite profiling of symbiont and host during thermal stress and bleaching in a model cnidarian-dinoflagellate symbiosis. Journal of Experimental Biology. 219 (4), 516-527 (2016).
- Matthews, J. L., et al. Optimal nutrient exchange and immune responses operate in partner specificity in the cnidarian-dinoflagellate symbiosis. Proceedings of the National Academy of Sciences of the United States of America. 114 (50), 13194-13199 (2017).
- Rosset, S. L., et al. The molecular language of the cnidarian-dinoflagellate symbiosis. Trends in Microbiology. 29 (4), 320-333 (2020).
- Matthews, J. L., et al. Partner switching and metabolic flux in a model cnidarian-dinoflagellate symbiosis. Royal Society. 285 (1892), 20182336 (2018).
- González-Pech, R. A., et al. Physiological factors facilitating the persistence of Pocillopora aliciae and Plesiastrea versipora in temperate reefs of south-eastern Australia under ocean warming. Coral Reefs. 41, 1239-1253 (2022).
- Williams, A., et al. Metabolomic shifts associated with heat stress in coral holobionts. Science Advances. 7 (1), (2021).
- Deutsch, J. M., et al. Metabolomics of healthy and stony coral tissue loss disease affected Montastraea cavernosa corals. Frontiers in Marine Science. 8, 1421 (2021).
- Stien, D., et al. A unique approach to monitor stress in coral exposed to emerging pollutants. Scientific Reports. 10 (1), 9601 (2020).
- Lohr, K. E., et al. Resolving coral photoacclimation dynamics through coupled photophysiological and metabolomic profiling. Journal of Experimental Biology. 222 (8), (2019).
- Jorissen, H., et al. Coral larval settlement preferences linked to crustose coralline algae with distinct chemical and microbial signatures. Scientific Reports. 11 (1), 14610 (2021).
- Roach, T. N., Dilworth, J., Jones, A. D., Quinn, R. A., Drury, C. Metabolomic signatures of coral bleaching history. Nature Ecology & Evolution. 5 (4), 495-503 (2021).
- Parkinson, J. E., et al. Molecular tools for coral reef restoration: Beyond biomarker discovery. Conservation Letters. 13 (1), 12687 (2020).
- Jiang, J., et al. How Symbiodiniaceae meets the challenges of life during coral bleaching. Coral Reefs. 40, 1339-1353 (2021).
- Guerra, F. D., Attia, M. F., Whitehead, D. C., Alexis, F. Nanotechnology for environmental remediation: materials and applications. Molecules. 23 (7), 1760 (2018).
- Matthews, J. L., et al. Metabolite pools of the reef building coral Montipora capitata are unaffected by Symbiodiniaceae community composition. Coral Reefs. 39, 1727-1737 (2020).
- Papina, M., Meziane, T., van Woesik, R. Symbiotic zooxanthellae provide the host-coral Montipora digitata with polyunsaturated fatty acids. Comparative Biochemistry and Physiology Part B: Biochemistry and Molecular Biology. 135 (3), 533-537 (2003).
- Kellogg, R., Patton, J. Lipid droplets, medium of energy exchange in the symbiotic anemone Condylactis gigantea: A model coral polyp. Marine Biology. 75, 137-149 (1983).
- Ankrah, N. Y., Chouaia, B., Douglas, A. E. The cost of metabolic interactions in symbioses between insects and bacteria with reduced genomes. mBio. 9 (5), e01433 (2018).
- Kabeya, N., et al. Genes for de novo biosynthesis of omega-3 polyunsaturated fatty acids are widespread in animals. Science Advances. 4 (5), (2018).
- Hillyer, K. E., Dias, D., Lutz, A., Roessner, U., Davy, S. K. 13C metabolomics reveals widespread change in carbon fate during coral bleaching. Metabolomics. 14 (1), 12 (2018).
- Hillyer, K. E., Dias, D. A., Lutz, A., Roessner, U., Davy, S. K. Mapping carbon fate during bleaching in a model cnidarian symbiosis: the application of 13C metabolomics. New Phytologist. 214 (4), 1551-1562 (2017).
- Burriesci, M. S., Raab, T. K., Pringle, J. R. Evidence that glucose is the major transferred metabolite in dinoflagellate-cnidarian symbiosis. Journal of Experimental Biology. 215 (19), 3467-3477 (2012).
- Thurber, R. V., et al. Unified methods in collecting, preserving, and archiving coral bleaching and restoration specimens to increase sample utility and interdisciplinary collaboration. PeerJ. 10, 14176 (2022).
- Grottoli, A. G., et al. Increasing comparability among coral bleaching experiments. Ecological Applications. 31 (4), 02262 (2020).
- Mushtaq, M. Y., Choi, Y. H., Verpoorte, R., Wilson, E. G. Extraction for metabolomics: access to the metabolome. Phytochemical Analysis. 25 (4), 291-306 (2014).
- Bradford, M. M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Analytical Biochemistry. 72 (1), 248-254 (1976).
- Peterson, G. L., et al. A simplification of the protein assay method of Lowry et al. which is more generally applicable. Analytical Biochemistry. 83 (2), 346-356 (1977).
- Lowry, O. H., Rosebrough, N. J., Farr, A. L., Randall, R. J. Protein measurement with the Folin phenol reagent. Journal of Biological Chemistry. 193 (1), 265-275 (1951).
- Zamer, W. E., Shick, J. M., Tapley, D. W. Protein measurement and energetic considerations: Comparisons of biochemical and stoichiometric methods using bovine serum albumin and protein isolated from sea anemones. Limnology and Oceanography. 34 (1), 256-263 (1989).
- Smart, K. F., Aggio, R. B., Van Houtte, J. R., Villas-Boas, S. G. Analytical platform for metabolome analysis of microbial cells using methyl chloroformate derivatization followed by gas chromatography-mass spectrometry. Nature Protocols. 5 (10), 1709-1729 (2010).
- Pang, Z., et al. Using MetaboAnalyst 5.0 for LC-HRMS spectra processing, multi-omics integration and covariate adjustment of global metabolomics data. Nature Protocols. 17 (8), 1735-1761 (2022).
- Tibshirani, R., Walther, G., Hastie, T. Estimating the number of clusters in a data set via the gap statistic. Journal of the Royal Statistical Society: Series B (Statistical Methodology). 63 (2), 411-423 (2001).
- Chen, W. -. N., et al. Diel rhythmicity of lipid-body formation in a coral-Symbiodinium endosymbiosis). Coral Reefs. 31 (2), 521-534 (2012).
- Imbs, A. Fatty acids and other lipids of corals: composition, distribution, and biosynthesis. Russian Journal of Marine Biology. 39 (3), 153-168 (2013).
- Rosset, S., et al. Lipidome analysis of Symbiodiniaceae reveals possible mechanisms of heat stress tolerance in reef coral symbionts. Coral Reefs. 38 (6), 1241-1253 (2019).
- Carreón-Palau, L., Parrish, C. C., Del Angel-Rodriguez, J. A., Perez-Espana, H. Seasonal shifts in fatty acids and sterols in sponges, corals, and bivalves, in a southern Gulf of Mexico coral reef under river influence. Coral Reefs. 40 (2), 571-593 (2021).
- Imbs, A. B., Dang, L. T. Seasonal dynamics of fatty acid biomarkers in the soft coral Sinularia flexibilis, a common species of Indo-Pacific coral reefs. Biochemical Systematics and Ecology. 96, 104246 (2021).
- Oku, H., Yamashiro, H., Onaga, K., Sakai, K., Iwasaki, H. Seasonal changes in the content and composition of lipids in the coral Goniastrea aspera. Coral Reefs. 22 (1), 83-85 (2003).
- Weis, V. M. Cell biology of coral symbiosis: foundational study can inform solutions to the coral reef crisis. Integrative and Comparative Biology. 59 (4), 845-855 (2019).
- Oakley, C., Davy, S., van Oppen, M., Lough, J. Cell biology of coral bleaching. Coral Bleaching. , 189-211 (2018).
- Lu, W., et al. Metabolite measurement: Pitfalls to avoid and practices to follow. Annual Review of Biochemistry. 86, 277-304 (2017).
- Lawson, C. A., et al. Heat stress decreases the diversity, abundance and functional potential of coral gas emissions. Global Change Biology. 27 (4), 879-891 (2021).
- Olander, A., et al. Comparative volatilomics of coral endosymbionts from one-and comprehensive two-dimensional gas chromatography approaches. Marine Biology. 168 (5), 76 (2021).
- Wuerz, M., et al. Symbiosis induces unique volatile profiles in the model cnidarian Aiptasia. Journal of Experimental Biology. 225 (19), (2022).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon