このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
生きているTrypanosoma bruceiの動的グリコソームpH変化の測定
* これらの著者は同等に貢献しました
要約
アフリカのトリパノソーマの血流型のグリコソームの環境的手がかりにpHがどのように応答するかを研究する方法について説明します。このアプローチでは、pH感受性遺伝性タンパク質センサーとフローサイトメトリーを組み合わせて、経時アッセイとハイスループットスクリーニングフォーマットの両方でpHダイナミクスを測定します。
要約
グルコース代謝は、アフリカのトリパノソーマ( Trypanosoma brucei)にとって、寄生虫発生の必須代謝プロセスおよび調節因子として重要です。環境中のグルコースレベルが変化したときに生じる細胞応答については、ほとんど知られていません。血流寄生虫とプロサイクリック型(昆虫期)寄生虫の両方で、グリコソームは解糖の大部分を収容しています。これらの細胞小器官は、グルコース欠乏に応答して急速に酸性化され、その結果、ヘキソキナーゼなどの解糖酵素のアロステリック調節が起こる可能性があります。以前の研究では、pH測定に使用する化学プローブの局所化が困難であり、他のアプリケーションでの有用性が制限されていました。
この論文では、遺伝性タンパク質pHバイオセンサーであるグリコソーム局在性pHorin2を発現する寄生虫の開発と使用について説明します。pHluorin2 はレシオメトリック pH バリアントであり、395 nm で pH(酸)依存的な励起の低下を示すと同時に、475 nm での励起の増加を示します。トランスジェニック寄生虫は、pHluorin2オープンリーディングフレームをトリパノソーマ発現ベクターpLEW100v5にクローニングすることによって生成され、どちらのライフサイクルステージでも誘導可能なタンパク質発現を可能にしました。免疫蛍光法を用いて、pHluorin2バイオセンサーのグリコソーム局在を確認し、バイオセンサーの局在をグリコソーム常在タンパク質アルドラーゼと比較しました。センサーの応答性は、以前にフルオレセインベースのpHセンサーのキャリブレーションに使用したアプローチである、pH 4 から 8 の範囲の一連のバッファーで細胞をインキュベートすることにより、異なる pH レベルでキャリブレーションしました。次に、フローサイトメトリーを用いて405 nmおよび488 nmのpHluorin2蛍光を測定し、グリコソームのpHを測定しました。PF寄生虫のグリコソーム酸性化の引き金として知られているグルコース欠乏に反応してpHを経時的にモニタリングし、生きたトランスジェニックpHluorin2発現寄生虫の性能を検証しました。このツールは、ハイスループット薬物スクリーニングに使用される可能性など、さまざまな用途が考えられます。グリコソームのpHだけでなく、このセンサーを他のオルガネラに適合させたり、他のトリパノソーマチドに使用したりして、生細胞環境におけるpHダイナミクスを理解することができます。
概要
寄生キネトプラスチドは、ほとんどの生物と同様に、中枢炭素代謝の基本的な構成要素としてグルコースに依存しています。このグループには、アフリカのトリパノソーマ、トリパノソーマ・ブルーセイなどの医学的に重要な生物が含まれます。アメリカのトリパノソーマ、T. cruzi; リーシュマニア属の寄生虫。グルコース代謝は、病原性ライフサイクル段階における寄生虫の増殖に不可欠です。例えば、グルコースが奪われると、アフリカトリパノソーマの血流型(BSF)は急速に死滅します。特に、解糖系は、感染のこの段階でATPの唯一の供給源として機能します1。リーシュマニア寄生虫も同様に宿主のグルコースに依存しており、宿主マクロファージに存在するアマスティゴートのライフサイクルステージは、成長のためにこの炭素源に依存しています2。
これらの寄生虫は、異なる昆虫媒介者を含む異なるライフスタイルを持っていますが、ブドウ糖への反応と消費方法には多くの共通点があります。例えば、これらの寄生虫は、ほとんどの解糖酵素をグリコソームと呼ばれる修飾ペルオキシソームに局在化させます。このキネトプラスト特異的細胞小器官は、保存された生合成メカニズムと形態に基づいて、哺乳類のペルオキシソームに関連しています3,4,5,6。
解糖系経路酵素のほとんどをグリコソームに区画化することで、寄生虫特異的な経路制御手段が提供されます。化学的pHプローブを用いて、栄養欠乏がプロサイクリック型(PF)グリコソームの急速な酸性化を引き起こし、主要な解糖酵素であるヘキソキナーゼ7,8のアロステリック調節因子結合部位の露出により解糖酵素活性を変化させることを立証しました。以前の研究では、化学プローブは使用するために一定の送達を必要とし、他の用途での有用性は限られていました。さらに、BSF中のグリコソーム内のプローブ分布を維持するという課題により、そのライフステージにおけるグリコソームのpHを調査するためのアプローチの有用性が制限されていました。
本研究では、蛍光タンパク質バイオセンサーpHluorin2を用いて、グルコース飢餓などの環境要因に応答したBSF T. brucei のグリコソームpH変化をモニターしました9 (図1)。この研究の結果は、BSF T. brucei が飢餓に応答してグリコソームを急速に酸性化することを示唆しています。このバイオセンサーにより、 T. brucei および関連寄生虫の解糖系制御に関する理解が深まることが期待されます。
プロトコル
単型寄生虫株である T. brucei brucei 90-13 BSFトリパノソーマは、バイオセーフティレベル2の施設で取り扱うべきリスクグループ2の生物とされているため、安全性への配慮が必要です。
1. トリパノソーマの培養とトランスフェクション
- HMI-9培地中の培養T. brucei brucei 90-13 BSFトリパノソーマに、5%CO2、10%、37°Cで10%熱不活化FBSおよび10%Nu-Serumを添加した。
注:培養を健康に保つには、細胞密度を2〜104 〜5〜10×6 細胞/mL×維持してください。 - pHluorin2-PTS1のpLEW100v5へのクローニング
- pHluorin2オープンリーディングフレームを商業的に合成して、2つのグリシンリンカーをコードする3'伸長とそれに続くPTS1局在タグであるトリペプチドAKLを有する遺伝子を産生する。
- 制限酵素消化により、このコンストラクトを誘導性トリパノソーマ発現ベクターpLEW100v5にクローニングします。pHluorin2-PTS1 と pLEW100v5 を含むクローニングベクターを、HindIII と BamHI11 を用いて二重消化します。クリーンアップステップを、好ましくはアガロースゲル精製により行い、制限酵素、望ましくないクローニングベクター骨格、および切除したルシフェラーゼ遺伝子含有断片を除去する。
- pHluorin2含有インサートを消化されたpLEW100v5にT4 DNAリガーゼ12でライゲーションします。(クローニングのスキームについては、 補足図S1 を参照)。
- 次世代全プラスミドシーケンシングによりプラスミドをシーケンシングし、インサートとベクターが正しくライゲーションされ、クローニングプロセス中にpHluorin2-PTS1遺伝子内に変異が導入されていないことを確認します。
注:完全なプラスミド配列は Addgene.org に提出され、#83680が割り当てられています。 - 20 μgのプラスミドを40単位のNotIで消化して直鎖化します。次に、参照したヒトT細胞キット( 材料表を参照)を使用して、ヌクレオフェクションを介してBSF 90-13寄生虫にトランスフェクションします。Burkard et al.13 によって記述されているように、安定した積分を選択します。
2. pHlourin2-PTS1の免疫蛍光共局在
- 顕微鏡スライドをポリ-L-リジン0.1%(w/v)含有H2Oで10分間処理して作製します。ポリ-L-リジン溶液を除去した後、スライドをPBSで一度洗浄します。
- pHluorin2-PTS1発現を誘導するには、採取の24時間前に2×105細胞/mLの細胞をテトラサイクリンまたはドキシサイクリン(1μg/mL)で処理します。遠心分離(室温[RT]、10分、1,000×g)により、親および誘導されたpLEWpHluorin2-PTS1(pHL)寄生虫を2×10 6ペレットし、PBSで1回洗浄する。
- 市販の16%EMグレード溶液から調製したPBSに調製した200 μLの2%パラホルムアルデヒドに細胞を再懸濁します。固定液中の細胞をスライドに塗布し、細胞を30分間沈降させます。接着した細胞を洗浄液(PBS中の0.1%正常ヤギ血清)で2回洗浄します。
- 透過処理溶液(0.5% Triton X-100 in PBS)を細胞に塗布し、正確に30分間インキュベートします。透過処理液を取り出し、十分な量の洗浄液で一度洗浄してください。
- ブロック溶液(10%正常ヤギ血清と0.1%Triton X-100 PBS溶液)を塗布し、30分間インキュベートします。
- T. brucei aldolase 1:500に対して提起した抗血清をブロック溶液で希釈し、細胞に塗布する14。室温で1時間インキュベートし、スライドを洗浄液で3〜5分間5回洗浄します。
- ヤギ抗ウサギAlexa fluor 568 1:1,000をブロック溶液で希釈し、細胞に塗布します。室温で1時間インキュベートし、スライドを洗浄液で3〜5分間5回洗浄します。
- 封入剤を塗布し、カバーガラスをスライドに密封します。
- 100倍の対物レンズ(NA 1.4-0.7)で細胞を画像化し、ImageJを使用して画像を解析します。ImageJ の 'Coloc 2' プラグインを使用して Pearson の共局在解析を実行します。代表的なセルのフィールドについては、 補足図S2 を参照してください。)
- これを完了するには、画像ファイルをフィジーに追加し、[ 画像] を選択します。
- 各チャンネルの明るさとコントラストを調整して、背景が見えないようにします。
- 画像を 16 ビットから 8 ビットに変更し、画像を結合してから、チャンネルが分割された 1 つのセルにトリミングします。
- 「解析と共局在化」で、「Coloc2」プラグインを選択します。チャンネル 1 としてアルドラーゼ(赤チャンネル)、チャンネル 2 として pHL(緑チャンネル)を選択し、[OK] をクリックして Pearson 相関の計算を開始します。
3. フローサイトメトリー用のサンプル調製
- テトラサイクリンまたはドキシサイクリン(1 μg/mL)のいずれかでpHL細胞を一晩誘導します。
- 遠心分離(RT、10分、1,000 × g)により細胞(~4 × 107 pHLおよび~1 × 106 pHL)をペレット化します。上清を除去し、サンプルが飢餓状態にあるか、飢餓状態になっていないかに応じて、10 mMグルコースの有無にかかわらず、細胞を1 mLのPBSに再懸濁します。経時アッセイでは、洗浄が完了するまで細胞が飢餓状態にならないように、10 mMグルコースを添加したPBSに再懸濁します。ハイスループットスクリーン(HTS)アッセイでは、グルコースのキャリーオーバーを最小限に抑えるために、グルコースを含まないPBSに再懸濁します。さらに2回洗浄を繰り返します。
- 細胞を4回目に遠心分離し、上清を除去し、処理に応じて、細胞ペレットをPBS、PBSと5 mMグルコース、またはPBSと10 mMグルコースのいずれかに再懸濁します。生死測定のために、1 μg/mL のヨウ化プロピジウム(PI)または 100 nM チアゾールレッド(TR)をサンプルに添加します。各サンプルをフローサイトメーターに適合する5 mLチューブに移します。
4. フローサイトメトリー
注:405 nm(紫色)、488 nm(青色)、561 nm(黄色)または638 nm(赤色)のレーザーを含むフローサイトメーターで実験を準備します。以下で説明するチャネルの一般名については、 補足表 S1 を参照してください。
- pHL蛍光を測定するには、チャンネルKO525(VL2-H、励起405 nm、発光542/27 nm)およびFITC(BL1-H、励起488 nm、発光530/30 nm)を使用します。生細胞と死細胞を区別するには、PIまたはTRを使用します。YL2-Hチャンネル(励起561 nm、発光620/25 nm)のPIを測定します。RL1-Hチャンネル(励起波長638 nm、660/10 BP)でTRを測定します。
- フローサイトメーターソフトウェアで実験をセットアップするには、1)YL2-Hチャネルヒストグラム(PIを使用する場合)またはRL1-Hチャネルヒストグラム(TRを使用する場合)、2)FSC-AとSSC-Aのドットプロット、3)FSC-AとFSC-Hのドットプロット、4)BL1-HとVL2-Hのチャネルのドットプロットを作成します。
- 最初に未染色のWT(親細胞株)コントロールをサンプル注入ポート(SIP)に置き、ステージを所定の位置に持ち上げます。サイトメーターで最低流速でデータの取得を開始し、必要な調整を行う時間を確保します。偽の破片や死細胞のスコアリングを回避するには、サンプル取り込み開始から 5 秒後にイベントの記録を開始します。
- YL2またはRL1電圧を調整して、メインピークが103-10 4 相対蛍光強度(RFI)単位以内になるようにします。イベントの>90%がドットプロットに収まるようにFSCとSSCの電圧を調整します。FSC閾値を調整して、デブリ集団を除外しますが、可能性の低い細胞は除外します。
- VL2 および BL1 チャンネルを調整して、一次ピークが 103 から 104 RFI 単位以内になるようにします。
- PIまたはTRで染色した誘導pHLを含む最初のサンプルをSIPに置き、ステージを所定の位置に上げます。最低流量でデータの集録を開始し、各プロットのイベントを注意深く観察します。イベントの>90%が各プロット内にあり、VL2-HおよびBL1-Hチャネルを飽和させるイベントがないことを確認します。
- サンプルの実行を続行します。サンプルごとに少なくとも 10,000 個のイベントを記録してください。
- サンプルのデータを .fcs ファイル形式で保存し、分析用にエクスポートします。
5. フローサイトメトリー結果のデータ解析
注:このデータ分析ワークフローでは、FlowJoソフトウェアを使用します。他のフローサイトメトリー解析ソフトウェアを使用する場合は、ソフトウェアに適したツールを使用して、以下に説明する主要な手順を引き続き実行してください。プロットとゲーティングを可視化するには、 補足図S3 および 補足図S4を参照してください。
- 新しいレイアウトを開き、手順 4.3 で取得した .fcs ファイルを開きます。.fcs ファイルをレイアウト ウィンドウにドラッグ アンド ドロップします。
- 生細胞用のゲート。
- 染色されていないWTコントロールをダブルクリックして、このサンプルのウィンドウを開きます。
- YL2-Hチャネル(PIを使用する場合)またはRL2-Hチャネル(TRを使用する場合)のいずれかのヒストグラムとしてデータを表示します。これと生存色素で染色したサンプルを切り替えて、生きている個体群と死んだ個体群を同定します。
注:このサンプルには生存率色素が添加されていないため、すべてのイベントは染色されていない必要があります。 - 生きている集団と死んだ人口を分ける二等分線ゲートを作成します。左のゲートに 「Live 」、右のゲートに 「Dead」という名前を付けます。このゲートをすべてのサンプルに適用し、サンプルを切り替えて、このゲートがすべてのサンプルに対して適切に描画されるようにします。必要に応じてゲートを調整します。
- セルのゲート。
- 染色されていない WT コントロールで、 ライブ ゲートをダブルクリックして、そのゲートのイベントを表示します。ドットプロットの x軸 を FSC-A に、 y軸 を SSC-Aに変更します。
- ポリゴン ゲート ツールを使用して、セル集団の周囲にゲートを描画し、「Cells」という名前を付けます。破片/死にかけている細胞(通常はドットプロットの左端と下部)と凝集体(ドットプロットの右端と上部)を除外するように注意してください。
- このゲートは、すべてのサンプルの ライブ ゲートの下に適用します。サンプルを切り替えて、ゲートがすべてのサンプルの可能性のある細胞集団を網羅していることを確認し、必要な調整を行います。ゲートを変更した後は、必ずすべてのサンプルにゲートを再適用してください。
注:細胞集団の分布は、FSCとSSCの飢餓状態と非飢餓状態の間で顕著に変化します。 セルゲート がすべての条件で細胞集団を包含していることを確認します。
- pH測定の品質を向上させるシングルセル用のゲート。
- 染色されていないWTコントロールで、 セル ゲートをダブルクリックして、そのゲートのイベントを表示します。ドットプロットの x軸 を FSC-A に、 y軸 を FSC-Hに変更します。
- このドットプロットで、シングルセルの対角分布を探し、シングレット集団の右下に2次母集団を形成しているダブレットを探します( 補足図S1 の3番目のプロットを参照)。 ポリゴン ゲート ツールを使用して、ダブレット母集団を除いたシングレット イベントの周囲にゲートを描画します。このゲートに 「シングレット」という名前を付けます。
- すべてのサンプルについて、Cellゲートの下にSingletsゲートを適用します。ここでも、サンプルを切り替えて、ゲートが一重項母集団を含めながらダブレット母集団を適切に除外していることを確認します。必要に応じて調整します。
- 蛍光pHL細胞用のゲート。
- 染色されていないWTコントロールサンプルで、 シングレット ゲートをダブルクリックして、その母集団のドットプロットを開きます。 x軸 を BL1-H に、 y軸 を VL2-Hに変更します。
- pHセンサpHluorin2は、VL2とBL1の両方で蛍光を発します。 ポリゴンゲートツールを使用して 、ドットプロットの左下の自家蛍光集団から上部と直後に伸びる斜めのゲートを描画します。このゲートに pHL+ という名前を付けます。
- すべてのサンプルについて、シングレットゲートの下にpHL+ゲートを適用します。pHLサンプルに切り替え、WTコントロールよりもVL2-HとBL1-Hの両方で蛍光強度が高いイベントが含まれるようにゲートを調整します。グリコソームのpHが変化すると、この集団の位置が変化するため、このゲートがすべてのサンプルでこの蛍光集団を包含するようにしてください。
注:pHL+ 集団におけるこの小さいながらも目に見える変化は、蛍光色素の励起スペクトルの pH 依存的な変化によるものです。
- 統計情報をエクスポートします。
- [テーブル エディター] |編集バー |[列の追加] をクリックして、エクスポートする新しい統計を追加します。
注: エクスポートする統計ごとに、それぞれの統計とエクスポート元の母集団を選択します。[中央値] など、該当する統計情報に適切なパラメーターを選択してください。サンプルは変更しないでおきます。 - 統計量として、 Total (ungated) Count、pHL+ Count、Live Freq. of Total (percentage based on total events)、pHL+ Freq. of Parent (percentage based on parent gate)、pHL+ Median VL2-H、pHL+ Median BL1-H の列を追加します。
- [テーブル エディタ] をクリックし、エクスポート設定の [表示先→ ファイルへ] と [テキスト → CSV] を変更し、ファイルの保存先と名前を選択します。[テーブルの作成] をクリックします。
- [テーブル エディター] |編集バー |[列の追加] をクリックして、エクスポートする新しい統計を追加します。
- 蛍光比を計算します。
- エクスポートした.csvファイルをスプレッドシートファイルとして保存します。
- 実験のすべてのサンプルで、サンプルあたりのイベント数、ライブイベントの割合、pHL+ イベントの割合を比較して、品質管理分析を実行します。実験に応じて、棒グラフまたは散布図でこれらを視覚的に比較します。
- 新しい列に pHL+ 中央値 VL2/BL1 というラベルを付けます。各サンプルについて、式(1)に示すように、VL2-H値の中央値をBL1-H値の中央値で割ります。
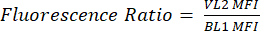 (1)
(1)
- 蛍光比を用いた統計解析を行うには、統計解析ソフトウェアを使用します。
6. pHバイオセンサーの校正
注:測定した蛍光比をpH単位に変換するには、ニゲリシンおよびバリノマイシンを使用してpHL発現細胞をキャリブレーションします。ニゲリシンはK+/H+アンチポーターであり、緩衝液中に十分なK+がある場合に膜を横切ってpHを平衡化できるイオノフォアである15。ニジェリシンは、pHluorinやその他のpHセンサーの校正に一般的に使用されています16,17。ペルオキシソームに局在するpHluorinは、10 μMのnigericin18を使用して以前に較正されていたため、その濃度で処理することを選択しました。バリノマイシンはカリウムイオノフォアであり、ミトコンドリア膜全体のpHを平衡化するために(4 μMで)使用されています19。10 μM のバリノマイシンを使用して、K+ イオンが膜全体で平衡化されるようにすることで、ニゲリシンの pH 平衡化活性を補助しました。ニゲリシンとバリノマイシンの組み合わせを使用しましたが、ニゲリシンはオルガネラのpHを平衡化するのに十分である可能性があります。
- それぞれ 5 〜 8.5 の範囲の異なる pH で、ユニバーサルキャリブレーションバッファー(UCB、15 mM MES、15 mM HEPES、および 130 mM KCl)の 8 つの溶液を調製します。
- 遠心分離機(RT、10分、800-1,000 × g)を、2 mLのpHL発現BSF培養物(各4~×4~106細胞)の8本の別々のチューブで遠心分離(RT、10分、800-1,000 g)します。
- 上清を除去し、各細胞ペレットを異なるpH値でUCBに再懸濁します。
- ニゲリシンとバリノマイシンをそれぞれ10 μMに導入します。 PI のスパイクを 1 μg/mL にします。
- 各溶液中の細胞を15分間インキュベートします。
- 各サンプルをフローサイトメーターで分析し、ステップ4〜5で説明したように蛍光比を測定します。
- この実験をさらに 2 回繰り返して、各 pH 値について 3 回の生物学的複製を取得します。データを .fcs 形式でエクスポートします。
- 手順 5.1 から 5.8 の説明に従って .fcs ファイルを分析します。各pHで測定された蛍光比を使用して、式(2)を使用する将来の実験でグリコソームのpHを補間します。
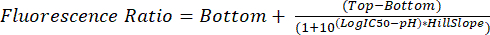 (2)
(2)
注: 補足図S3 は、ドットプロットとゲーティングスキームを示しています。その結果を 補足表S2に示す。GraphPad Prismを用いて蛍光比からpHを補間する方法は以下のとおりです。他の統計ソフトウェアを使用している場合は、同じ重要な手順に従ってください。- GraphPad Prismを開き、3つのy-反復をもつXY表を作成します。pH 値を x カラムに貼り付け、関連する蛍光比値を y レプリケートカラムに貼り付けます。
- テーブルに関連付けられているグラフをクリックします。グラフを表示しているときに、[分析]リボンの[分析]をクリックします。XY解析で検量線を補間します。
- シ グモイド、4PL、Xは対数(濃度) を選択してください。[上] パラメーターと [下限] パラメーターは、推定される上端と下端のプラトーです。[ 外れ値の特別な処理なし] を選択します。
注: ソフトウェアは、式 (2) およびステップ 6.9.3 で記述されたモデルにデータを適合させようとします。結果の表とグラフの曲線で適合不足の証拠を探します。 - 他の実験の蛍光比からpHを補間するには、pHLキャリブレーションデータの表を参照してください。蛍光比の値をキャリブレーションデータの下に y カラムに貼り付けます。各 y 値にタイトルを付けますが、x 値 (pH) は不明であるため空白のままにします。
- 結果ギャラリーで、補間分析をクリックし、補間X複製タブに移動します。入力した蛍光比値と一緒にリストされる補間されたpH値を探します。
注:ソフトウェアは、キャリブレーションデータから見つかったモデルと最適なパラメーター値を使用して、pH が不明な実験の蛍光比から pH を補間します。
7.グルコース飢餓とアドバックのタイムコース
- ステップ 1.1 で説明したように、1 μg/mL のドキシサイクリンを 37 °C でインキュベートした状態で、15 mL の BSF pHL 寄生虫を一晩誘導します。
- 15 mL の誘導 pHL 培養液を 10 mM グルコースを添加した PBS で洗浄します。この手順を3回繰り返します。
- 同時に、ステップ3.2の説明に従って、3 mLのWT培養物をPBS 3xで洗浄します。
- pHLサンプルを1 mLのPBSに再懸濁したときの最初の洗浄後、0.1 mLのpHL細胞懸濁液を除去して、飢餓状態のコントロールとして10 mMのグルコースを保持します。
- 最終洗浄後、pHL サンプルを 1 μg/mL の PI を添加した PBS に再懸濁します。
- フローサイトメトリーデータの取得
- 実験とサンプルに応じて、5分、10分、30分、または90分ごとにフローサイトメーターで各サンプルを測定することにより、飢餓サンプルと飢餓していないサンプルの両方のグリコソームpHのモニタリングを開始します。ステップ 4.1 から 4.3 の説明に従ってフローサイトメトリーデータを取得し、電圧とプロットが事前に準備されていることを確認します。
- 最初に未染色のWTコントロールを0分で実行します。
- 飢餓状態のコントロールサンプルをサイトメーターで、0分から90分ごとに実行します。飢餓時経過アッセイでは、飢餓サンプルを 0 分から 10 分ごとに分析します。グルコース アドバック アッセイでは、飢餓状態のサンプルを 0、5、10、20、30、60、および 90 分で分析します。その後、ブドウ糖を導入した後に繰り返します。
- サンプルを室温で、90 分間の飢餓時間コースと 180 分間のグルコース アドバック時間コースでインキュベートします。
- 手順 5.1 から 5.8 の説明に従って .fcs ファイルを分析します。
注: 補足図S4 は、ゲーティングの実行方法とドットプロットがどのように見えるかを示しています。 補足表S3 及び 補足表S4 は、それぞれ、グルコース飢餓時間経過アッセイ及びグルコース加算時間経過アッセイの結果を示す。
8. 薬物スクリーニングのためのアッセイの最適化
- 前述したように、約 32 mL の pHL 発現 BSF 寄生虫と 3 mL の WT 細胞を、どちらも約 2 × 106 cells/mL、2 倍の PBS で洗浄します。
注:測定された蛍光比のばらつきを最小限に抑え、Zファクター統計を正確に測定するには、ウェルごとに10,000を超えるイベントを記録する必要があります。これを達成するために必要な最小培養液は約26 mLですが、取り扱いを容易にするために32 mLを推奨します。- 最初の洗浄後、pHLサンプルを2本の微量遠心チューブに均等に分離します。
- 洗浄後、1 つの pHL サンプルを 5 mM グルコース、0.1% DMSO、TR を含む 18 mL の PBS に再懸濁し、もう 1 つの pHL サンプルを 18 mL の PBS と 0.1% DMSO および TR に再懸濁します。
注:化合物は通常DMSOに溶解するため、DMSOは薬物スクリーニングで緩衝液組成を模倣するために使用されます。 - これら 2 つの細胞溶液を 12 ウェルリザーバー(ウェルあたり 9 mL)に移します。
- ピペッティングロボットを使用して、細胞溶液を384ウェルプレートの別々の半分(ウェルあたり80 μL)にピペッティングします。
- プレートを室温で1.5時間インキュベートし、静かに振とうし、蛍光色素を光から保護するためにアルミホイルで包みます。
- 384ウェルプレートを分析できるフローサイトメーターでプレートを流動します。
注:次のワークフローは、Cytkick Max Auto Samplerを搭載したAttune NxTに適合しています。別のフローサイトメーターを使用する場合は、引き続き重要な手順に従ってください。- プレートについては、最も速い流速(1,000 uL/min)で分析し、 ブーストモードを有効にします。20 μL/ウェルを取得します。各ウェル間に 1 つの混合サイクルと 1 つのすすぎサイクルを含めます。
- ステップ4.1.3の説明に従ってプロットを作成します。
- 未染色のWTチューブコントロールを実行し、ステップ4.1.3および4.1.4の説明に従って電圧を最適化します。飢餓状態の pHL チューブサンプルを実行し、ステップ 4.1 の説明に従って VL2 および BL1 電圧を最適化します。
- フローサイトメーターでプレートの取り込みを開始します。プレートを水平に泳動させて、飢餓状態のサンプルウェルと飢餓状態のサンプルウェルの間に大きな取り込み時間差がないようにします。プレートの取得が~1.5時間で完了することを確認します。
- .fcs ファイルをエクスポートし、手順 5.1 から 5.8 の説明に従ってデータを分析します。グルコース(Glc)またはグルコースなし(飢餓)で処理したサンプルの蛍光比の平均(AVG)と標準偏差(SD)を求めます。
注:Zファクター実験のデータは、 補足表S5に記載されています。 - 式 (3) を使用して Z 係数20 の統計量を計算します。
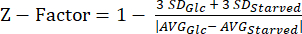 (3)
(3)
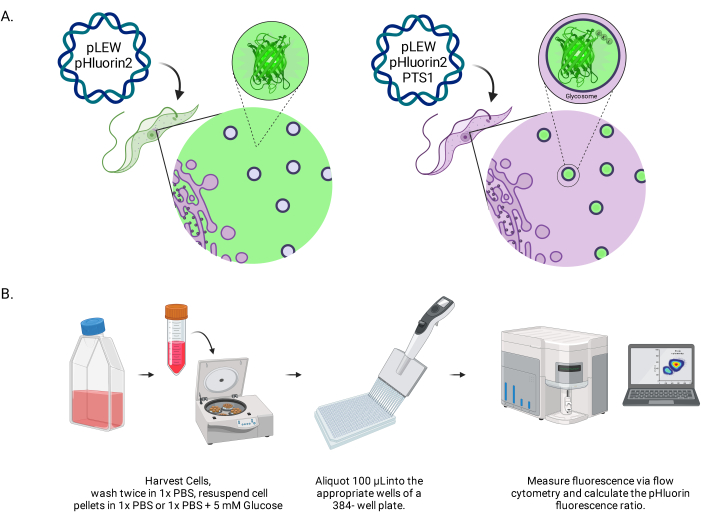
図1:生BSFトリパノソーマのグリコソームpHをスコアリングする方法の図。 (A)グリコソームに位置するpHluorin2センサーを発現する細胞株の描写。ペルオキシソーム標的配列を含めることで、局在化を制御することができます。注:PTS-1の除去は細胞質の局在化につながり、その細胞内コンパートメントのpHの将来の分析を可能にすると予想されます。(B)センサーバリデーションアッセイの描写。略語:BSF = bloodstream form。 この図の拡大版をご覧になるには、ここをクリックしてください。
注:Zファクター統計は、アッセイがHTSにどの程度適しているかを判断するために使用されます。0.5 から 1.0 の間の値は、一般に、アッセイ品質が HTS で許容できることを意味します。
結果
BSF T. bruceiにおけるグリコソームへのpHLuorin2-PTS1局在
pHluorin2-PTS1の細胞内局在を評価するために、寄生虫を免疫蛍光アッセイにかけました。グリコソーム常在タンパク質であるアルドラーゼ(TbAldolase)に対して抗血清と共局在した導入遺伝子からのシグナル(図2A)。抗TbアルドラーゼとpHluorin2-PTS1の共局在のピアソン相関係数の平均は0.895であり、pHluorin2-...
ディスカッション
アフリカのトリパノソーマにおける環境認識と応答機構は十分に理解されていない。栄養素の利用可能性の変化は、グリコソームの酸性化など、寄生虫の多様な応答を引き起こすことが知られています。ここでは、遺伝性タンパク質センサーpHluorin2とフローサイトメトリーを用いて、生細胞の環境摂動に対するグリコソームのpH応答を研究する方法について説明しました。
開示事項
著者は利益相反がないことを宣言します。
謝辞
pHluorin2-PTS1 は、Twist Bioscience 社によって pLEW100v5 にクローニングされ、高コピークローニングベクターでコンストラクトが提供されました。pLEW100v5はジョージ・クロス博士からの贈り物でした。 T. brucei アルドラーゼに対して提起された抗血清は、要求に応じてクレムソン大学のメレディス T. モリス博士から入手できます。JCMとKACの研究所の研究の一部は、米国国立衛生研究所(R01AI156382)からの助成金によって支援されました。SSP は NIH 3R01AI156382 でサポートされました。
資料
| Name | Company | Catalog Number | Comments |
| 50 mL Tissue Culture Flasks (Non-treated, sterile) | VWR | 10861-572 | |
| 75 cm2 Tissue Culture Flask (Non-Treated, sterile) | Corning | 431464U | |
| 80 µL flat-bottom 384-well plate | BrandTech | 781620 | |
| Amaxa Human T Cell Nucleofector Kit | Lonza | VPA-1002 | |
| Attune NxT Flow Cytometer | invitrogen by Thermo Fisher Scientific | A24858 | FlowJo software |
| BRANDplates 96-Well, flat bottom plate | Millipore Sigma | BR781662 | |
| Coloc 2 plugin of ImageJ | https://imagej.net/plugins/coloc-2 | ||
| CytKick Max Auto Sampler | invitrogen by Thermo Fisher Scientific | A42973 | |
| CytoFLEX Flow Cytometer | Beckman-Coulter | ||
| Electron Microscopy Sciences 16% Paraformaldehyde Aqueous Solution, EM Grade, 10 mL Ampoule | Fisher Scientific | 50-980-487 | |
| GraphPad Prism | statistical software | ||
| Nigericin (sodium salt) | Cayman Chemical | 11437 | |
| Nucleofector 2b | Lonza | Discontinued Product | |
| OP2 Liquid Handler | opentrons | OP2 | |
| poly-L-lysine, 0.1% (w/v) in H2O | Sigma Life Science | CAS:25988-63-0 | Pipetting robot for HTS assay |
| Thiazole Red (TO-PRO-3) | biotium | #40087 | We machined a custom acrylic plate stand so this brand of plate could be detected and used on our CytKick Max Auto Sampler |
| valinomycin | Cayman Chemical | 10009152 | Pipetting robot for HTS assay |
| For pH calibration | |||
| For pH calibration |
参考文献
- Coley, A. F., Dodson, H. C., Morris, M. T., Morris, J. C. Glycolysis in the African trypanosome: Targeting enzymes and their subcellular compartments for therapeutic development. Molecular Biology International. 2011, 123702 (2011).
- Mcconville, M. J., Saunders, E. C., Kloehn, J., Dagley, M. J. Leishmania carbon metabolism in the macrophage phagolysosome- feast or famine. F1000Res. 4, 938 (2015).
- Parsons, M. Glycosomes: Parasites and the divergence of peroxisomal purpose. Molecular Microbiology. 53 (3), 717-724 (2004).
- Parsons, M., Furuya, T., Pal, S., Kessler, P. Biogenesis and function of peroxisomes and glycosomes. Molecular and Biochemical Parasitology. 115 (1), 19-28 (2001).
- Haanstra, J. R., Gonzalez-Marcano, E. B., Gualdron-Lopez, M., Michels, P. A. Biogenesis, maintenance and dynamics of glycosomes in trypanosomatid parasites. Biochimica et Biophysica Acta. 1863 (5), 1038-1048 (2016).
- Allmann, S., Bringaud, F. Glycosomes: A comprehensive view of their metabolic roles in t. Brucei. International Journal of Biochemistry and Cell Biology. 85, 85-90 (2017).
- Dodson, H. C., Morris, M. T., Morris, J. C. Glycerol 3-phosphate alters Trypanosoma brucei hexokinase activity in response to environmental change. The Journal of Biological Chemistry. 286 (38), 33150-33157 (2011).
- Lin, S., Morris, M. T., Ackroyd, P. C., Morris, J. C., Christensen, K. A. Peptide targeted delivery of pH sensor for quantitative measurements of intraglycosomal pH in live Trypanosoma brucei. Biochemistry. 52 (21), 3629-3637 (2013).
- Mahon, M. J. Phluorin2: An enhanced, ratiometric, pH-sensitive green florescent protein. Advances in Bioscience and Biotechnology. 2 (3), 132-137 (2011).
- Wirtz, E., Leal, S., Ochatt, C., Cross, G. A. A tightly regulated inducible expression system for conditional gene knock-outs and dominant-negative genetics in Trypanosoma brucei. Molecular and Biochemical Parasitology. 99 (1), 89-101 (1999).
- . Restriction digest v.2 Available from: https://www.protocols.io/view/restriction-digest-nkqdg33pg25z/v2 (2018)
- . Ligation protocol with t4 DNA ligaase (m0202) v.3 Available from: https://www.protocols.io/view/ligation-protocol-with-t4-dna-ligase-m0202-95qpvorzv4o1/v3 (2021)
- Burkard, G., Fragoso, C. M., Roditi, I. Highly efficient stable transformation of bloodstream forms of Trypanosoma brucei. Molecular and Biochemical Parasitology. 153 (2), 220-223 (2007).
- Crowe, L. P., Wilkinson, C. L., Nicholson, K. R., Morris, M. T. Trypanosoma brucei pex13.2 is an accessory peroxin that functions in the import of peroxisome targeting sequence type 2 proteins and localizes to subdomains of the glycosome. mSphere. 5 (1), e00744 (2020).
- Kucejova, B., Kucej, M., Petrezselyova, S., Abelovska, L., Tomaska, L. A screen for nigericin-resistant yeast mutants revealed genes controlling mitochondrial volume and mitochondrial cation homeostasis. Genetics. 171 (2), 517-526 (2005).
- Huynh, M. H., Carruthers, V. B. Toxoplasma gondii excretion of glycolytic products is associated with acidification of the parasitophorous vacuole during parasite egress. PLoS Pathogens. 18 (5), e1010139 (2022).
- Lehoux, S., Abe, J., Florian, J. A., Berk, B. C. 14-3-3 binding to Na+/H+ exchanger isoform-1 is associated with serum-dependent activation of Na+/H+ exchange. TheJournal of Biological Chemistry. 276 (19), 15794-15800 (2001).
- Jankowski, A., et al. In situ measurements of the ph of mammalian peroxisomes using the fluorescent protein phluorin. The Journal of Biological Chemistry. 276 (52), 48748-48753 (2001).
- Jankowski, A., Grinstein, S. A. A noninvasive fluorimetric procedure for measurement of membrane potential. Quantification of the NADPH oxidase-induced depolarization in activated neutrophils. The Journal of Biological Chemistry. 274 (37), 26098-26104 (1999).
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A simple statistical parameter for use in evaluation and validation of high throughput screening assays. Journal of Biomolecular Screening. 4 (2), 67-73 (1999).
- Miesenbock, G., De Angelis, D. A., Rothman, J. E. Visualizing secretion and synaptic transmission with pH-sensitive green fluorescent proteins. Nature. 394 (6689), 192-195 (1998).
- Lin, S., et al. Ph regulation in glycosomes of procyclic form Trypanosoma brucei. The Journal of Biological Chemistry. 292 (19), 7795-7805 (2017).
- Ha, D. S., Schwarz, J. K., Turco, S. J., Beverley, S. M. Use of the green fluorescent protein as a marker in transfected Leishmania. Molecular and Biochemical Parasitology. 77 (1), 57-64 (1996).
- Kelly, J. M., Ward, H. M., Miles, M. A., Kendall, G. A shuttle vector which facilitates the expression of transfected genes in Trypanosoma cruzi and Leishmania. Nucleic Acids Research. 20 (15), 3963-3969 (1992).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved