JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
살아있는 트리파노소마 브루세이(Trypanosoma brucei)의 동적 당염색체 pH 변화 측정
* 이 저자들은 동등하게 기여했습니다
요약
우리는 아프리카 트리파노솜의 혈류 형태의 글리코솜에서 pH가 환경 신호에 어떻게 반응하는지 연구하는 방법을 설명합니다. 이 접근법에는 pH 민감성 유전성 단백질 센서와 유세포 분석이 결합되어 시간 경과 분석 및 고처리량 스크리닝 형식으로 pH 역학을 측정하는 것이 포함됩니다.
초록
포도당 대사는 아프리카 트리파노솜인 트리파노소마 브루세이(Trypanosoma brucei)에 중요한 역할을 하며, 이는 기생충 발달의 필수 대사 과정이자 조절자입니다. 환경 포도당 수치가 변할 때 생성되는 세포 반응에 대해서는 알려진 바가 거의 없습니다. 혈류와 순환 형태(곤충 단계) 기생충 모두에서 글리코솜은 해당과정의 대부분을 수용합니다. 이러한 세포 기관은 포도당 결핍에 대한 반응으로 빠르게 산성화되며, 이는 헥소키나아제(hexokinase)와 같은 해당효소(glycolytic enzyme)의 알로스테릭 조절(allosteric regulation)을 초래할 수 있습니다. 이전 연구에서는 pH 측정에 사용되는 화학 프로브의 위치를 파악하는 것이 어려웠기 때문에 다른 Application에서의 유용성이 제한되었습니다.
이 논문은 유전성 단백질 pH 바이오센서인 글리코솜 국소화 pHluorin2를 발현하는 기생충의 개발 및 사용에 대해 설명합니다. pHluorin2는 395nm에서 pH(산)에 따른 여기(excitation) 감소를 나타내는 동시에 475nm에서 여기(excitation)를 증가시키는 비율계량 pHluorin 변형체입니다. 형질전환 기생충은 pHluorin2 open reading frame을 트리파노솜 발현 벡터 pLEW100v5로 클로닝하여 생성되었으며, 두 수명 주기 단계에서 유도 가능한 단백질 발현을 가능하게 했습니다. 면역형광을 사용하여 pHluorin2 바이오센서의 글리코솜 국소화를 확인하고, 바이오센서의 국소화를 글리코솜 상주 단백질 알돌라아제와 비교하였다. 센서 응답성은 pH 4에서 8 사이의 일련의 완충액에서 세포를 배양하여 다양한 pH 수준에서 보정되었으며, 이는 이전에 플루오레세인 기반 pH 센서를 보정하는 데 사용한 접근 방식입니다. 그런 다음 글리코솜 pH를 측정하기 위해 유세포 분석을 사용하여 405nm 및 488nm에서 pHluorin2 형광을 측정했습니다. 우리는 PF 기생충에서 글리코솜 산성화의 알려진 원인인 포도당 결핍에 대한 반응으로 시간 경과에 따른 pH를 모니터링하여 살아있는 형질전환 pHluorin2 발현 기생충의 성능을 검증했습니다. 이 도구는 고처리량 약물 스크리닝에 잠재적으로 사용되는 것을 포함하여 다양한 잠재적 응용 분야를 가지고 있습니다. 글리코솜 pH 외에도 센서를 다른 소기관에 적용하거나 다른 트리파노소마티드에 사용하여 살아있는 세포 환경에서 pH 역학을 이해할 수 있습니다.
서문
대부분의 살아있는 유기체와 마찬가지로 기생 키네토플라스티드는 중앙 탄소 대사의 기본 구성 요소로 포도당에 의존합니다. 이 그룹에는 아프리카 트리파노솜, Trypanosoma brucei와 같은 의학적으로 중요한 유기체가 포함됩니다.미국 트리파노솜, T. cruzi; 그리고 Leishmania 속의 기생충. 포도당 대사는 병원성 수명 주기 단계에서 기생충 성장에 매우 중요합니다. 예를 들어, 포도당이 결핍되면 아프리카 트리파노솜의 혈류 형태(BSF)가 빠르게 죽습니다. 특히, 해당과정(glycolysis)은 이 감염 단계에서 ATP의 유일한 공급원 역할을 한다1. 리슈마니아 기생충도 마찬가지로 인간 숙주의 포도당에 의존하며, 숙주 대식세포에 상주하는 아마스티고테 수명 주기 단계는 성장을 위해 이 탄소원에 의존한다2.
이 기생충은 다양한 곤충 매개체와 관련된 독특한 생활 방식을 가지고 있지만 포도당에 반응하고 섭취하는 방식에서 많은 공통점을 공유합니다. 예를 들어, 이러한 기생충은 대부분의 해당작용 효소를 글리코솜이라고 하는 변형된 과산화소체로 국소화합니다. 이 키네토플라스티드 특이적 세포소기관은 보존된 생합성 메커니즘 및 형태 3,4,5,6을 기반으로 하는 포유류 과산화소체와 관련이 있습니다.
대부분의 해당과정 효소를 당염색체로 구획화하면 기생충 특이적 경로 조절 수단이 제공됩니다. 화학적 pH 프로브를 사용하여 영양 결핍이 프로사이클릭 형태(PF) 기생충 글리코좀의 급격한 산성화를 유발하여 주요 해당효소 헥소키나제 7,8에 대한 알로스테릭 조절자 결합 부위의 노출을 통해 해당효소 활성을 변화시킨다는 것을 확인했습니다. 이전 연구에서 화학 프로브는 사용을 위해 지속적인 전달이 필요했기 때문에 다른 응용 분야에서의 유용성이 제한되었습니다. 또한 BSF의 당체에서 프로브 분포를 유지하는 데 어려움이 있어 해당 수명 단계에서 당체 pH를 조사하기 위한 접근 방식의 유용성이 제한되었습니다.
이 연구에서는 형광 단백질 바이오센서 pHluorin2를 사용하여 포도당 결핍을 포함한 환경적 단서에 대한 반응으로 BSF T. brucei 의 당염색체 pH 변화를 모니터링했습니다9 (그림 1). 이 연구의 결과는 BSF T. brucei 가 PF 기생충에서 관찰된 반응과 유사한 가역적 방식으로 기아에 대한 반응으로 글리코솜을 빠르게 산성화한다는 것을 시사합니다. 우리는 이 바이오센서가 T. brucei 및 관련 기생충의 해당작용 조절에 대한 이해를 향상시킬 것으로 기대합니다.
프로토콜
단형 기생충 계통인 T. brucei brucei 90-13 BSF 트리파노솜을 사용하는 것은 생물안전 레벨 2 시설에서 취급해야 하는 위험 그룹 2 유기체로 간주되므로 안전성을 고려해야 합니다.
1. 트리파노솜 배양 및 형질주입
- HMI-9 배지의 배양 T. brucei brucei 90-13 BSF 트리파노솜은 37°C에서 5%CO210에서 10% 열-비활성화 FBS 및 10% Nu-혈청으로 보충되었습니다.
참고: 배양을 건강하게 유지하려면 세포 밀도를 2 × 104 와 5 × 106 cells/mL 사이로 유지하십시오. - pHluorin2-PTS1을 pLEW100v5로 클로닝
- pHluorin2 개방형 판독 프레임을 상업적으로 합성하여 2개의 글리신 링커를 인코딩하는 3' 확장을 가진 유전자와 PTS1 국소화 태그인 트리펩타이드 AKL을 생성합니다.
- 이 작제물을 제한 분해에 의해 유도성 트리파노솜 발현 벡터 pLEW100v5로 클로닝합니다. HindIII 및 BamHI11을 사용하여 pHluorin2-PTS1 및 pLEW100v5를 포함하는 클로닝 벡터를 모두 이중 분해합니다. 제한 효소, 바람직하지 않은 클로닝 벡터 골격 및 절제된 루시페라아제 유전자-함유 단편을 제거하기 위해, 바람직하게는 아가로스 겔 정제에 의한 클린업 단계를 수행한다.
- pHluorin2 함유 삽입물을 T4 DNA Ligase12로 분해된 pLEW100v5에 결찰합니다. (복제 계획에 대한 보충 그림 S1 참조).
- 차세대 전체 플라스미드 염기서열분석으로 플라스미드의 염기서열을 분석하여 삽입물과 벡터가 올바르게 ligation되었는지, 클로닝 과정에서 pHluorin2-PTS1 유전자 내에 돌연변이가 발생하지 않는지 확인합니다.
참고: 전체 플라스미드 염기서열은 Addgene.org 에 제출되었으며 #83680이 할당되었습니다. - 플라스미드 20 μg을 40 unit의 NotI로 분해하여 선형화합니다. 그런 다음 참조된 인간 T 세포 키트를 사용하여 핵을 통해 BSF 90-13 기생충으로 transfection합니다( 재료 표 참조). Burkard et al.13에 설명된 대로 안정적인 통합을 위해 선택합니다.
2. pHlourin2-PTS1의 면역형광 공동국소화
- 현미경 슬라이드를H2O에서 poly-L-lysine 0.1%(w/v)로 10분 동안 처리하여 준비합니다. 폴리-L-라이신 용액을 제거한 후 PBS로 슬라이드를 한 번 세척합니다.
- pHluorin2-PTS1 발현을 유도하려면 수확 24시간 전에 테트라사이클린 또는 독시사이클린(1μg/mL)으로 2 × 105 cells/mL의 세포를 처리합니다. 펠릿 2 × 106 부모 및 유도 pLEWpLuorin2-PTS1 (pHL) 기생충을 원심 분리 (실온 [RT], 10 분, 1,000 × g)하고 PBS로 한 번 세척합니다.
- 상업적으로 공급되는 16% EM 등급 용액으로 만든 PBS에서 갓 준비한 2% 파라포름알데히드 200μL에 세포를 재현탁시킵니다. 정착액의 세포를 슬라이드에 바르고 세포가 30분 동안 가라앉도록 합니다. 부착된 세포를 세척액(PBS의 0.1% 일반 염소 혈청)으로 2회 세척합니다.
- 투과화 용액(PBS의 0.5% Triton X-100)을 세포에 바르고 정확히 30분 동안 배양합니다. 투과성 용액을 제거하고 충분한 양의 세척액으로 한 번 세척하십시오.
- 블록 용액(10% 일반 염소 혈청 및 PBS의 0.1% Triton X-100)을 바르고 30분 동안 배양합니다.
- T. brucei aldolase 1:500에 대해 제기된 항혈청을 블록 용액에 희석하여 세포14에 적용한다. 상온에서 1시간 동안 배양합니다. 세척액으로 슬라이드를 5-3분 동안 5분 동안 세척합니다.
- 염소 토끼 방지 Alexa fluor 568 1:1,000을 블록 용액에 희석하여 세포에 적용합니다. 상온에서 1시간 동안 배양합니다. 세척액으로 슬라이드를 5-3분 동안 5번 세척합니다.
- 장착 매체를 적용하고 슬라이드에 커버슬립을 밀봉합니다.
- 100x 대물렌즈(NA 1.4-0.7)로 세포를 이미지화하고 ImageJ를 사용하여 이미지를 분석합니다. ImageJ용 'Coloc 2' 플러그인을 사용하여 Pearson의 공동 국소화 분석을 수행합니다. 대표적인 세포의 필드에 대해서는 보충 그림 S2 를 참조하십시오. )
- 이 작업을 완료하려면 피지에 이미지 파일을 추가하고 이미지를 선택합니다.
- 각 채널의 밝기와 대비를 배경이 보이지 않는 지점으로 조정합니다.
- 이미지를 16비트에서 8비트로 변경하고, 이미지를 병합한 다음, 분할 채널을 사용하여 단일 셀로 자릅니다.
- 분석 및 공동 국소화(Analyze and Colocalization)에서 Coloc2 플러그인을 선택합니다. 채널 1로 aldolase(빨간색 채널)를 선택하고 채널 2로 pHL(녹색 채널)을 선택하고 확인을 클릭하여 Pearson의 상관 계산을 시작합니다.
3. 유세포 분석을 위한 시료 전처리
- 하룻밤 동안 테트라사이클린 또는 독시사이클린(1μg/mL)으로 pHL 세포를 유도합니다.
- 원심분리(RT, 10분, 1,000×g)를 통해 세포(~4 × 107 pHL 및 ~1 × 106 부모)를 펠렛화합니다. 상층액을 제거하고 샘플이 굶주렸는지 또는 굶주리지 않았는지에 따라 10mM 포도당을 포함하거나 포함하지 않고 1mL의 PBS에 세포를 재현탁시킵니다. 시간 경과 분석의 경우, 세척이 완료될 때까지 세포가 굶주리는 것을 방지하기 위해 10mM 포도당이 보충된 PBS에 재현탁합니다. 고처리량 스크리닝(HTS) 분석의 경우 포도당 캐리오버를 최소화하기 위해 포도당 없이 PBS에 재현탁합니다. 세탁을 두 번 더 반복하십시오.
- 세포를 네 번째로 원심분리하고, 상층액을 제거하고, 처리에 따라 PBS, PBS 플러스 5mM 글루코스 또는 PBS 플러스 10mM 글루코스에 세포 펠릿을 재현탁시킵니다. 살아있는/죽은 측정을 위해 1μg/mL 프로피듐 요오드화물(PI) 또는 100nM 티아졸 레드(TR)로 샘플을 보충합니다. 각 샘플을 유세포 분석기와 호환되는 5mL 튜브로 옮깁니다.
4. 유세포 분석
참고: 405nm(보라색), 488nm(파란색), 561nm(노란색) 또는 638nm(빨간색) 레이저가 포함된 유세포 분석기에서 실험을 준비합니다. 아래에서 설명하는 채널의 공통 이름에 대해서는 보충 표 S1 을 참조하십시오.
- pHL 형광을 측정하려면 KO525(VL2-H, 여기 405nm, 방출 542/27nm) 및 FITC(BL1-H, 여기 488nm, 방출 530/30nm) 채널을 사용하십시오. 살아있는 세포와 죽은 세포를 구별하려면 PI 또는 TR을 사용하십시오. YL2-H 채널(여기 561nm, 방출 620/25nm)에서 PI를 측정합니다. RL1-H 채널(638nm 여기, 660/10 BP)에서 TR을 측정합니다.
- 유세포 분석기 소프트웨어에서 실험을 설정하려면 1) YL2-H 채널 히스토그램(PI를 사용하는 경우) 또는 RL1-H 채널 히스토그램(TR을 사용하는 경우), 2) FSC-A 대 SSC-A 도트 플롯, 3) FSC-A 대 FSC-H 도트 플롯, 4) BL1-H 대 VL2-H 채널 도트 플롯을 만듭니다.
- 염색되지 않은 WT(부모 세포주) 대조군을 샘플 주입 포트(SIP)에 먼저 놓고 스테이지를 제자리로 올립니다. 가장 낮은 유속으로 유세포분석기에서 데이터 수집을 시작하여 필요한 조정을 할 시간을 줍니다. 가짜 파편과 죽은 세포에 점수를 매기지 않으려면 샘플 수집이 시작된 후 5초 후에 이벤트 기록을 시작하십시오.
- YL2 또는 RL1 전압을 조정하여 메인 피크가 103-10 4 상대 형광 강도(RFI) 단위 내에 있도록 합니다. 사건의 >90%가 점도표에 맞도록 FSC 전압과 SSC 전압을 조정합니다. FSC 임계값을 조정하여 파편 모집단은 제외하지만 가능성이 있는 세포는 제외하지 않습니다.
- VL2 및 BL1 채널을 조정하여 1차 피크가 염색되지 않은 WT 제어를 위해 103-10 4 RFI 단위 내에 있도록 합니다.
- PI 또는 TR로 염색된 유도 pHL이 포함된 첫 번째 샘플을 SIP에 놓고 스테이지를 제자리로 올립니다. 가장 낮은 유속으로 데이터 수집을 시작하고 각 플롯에서 이벤트를 주의 깊게 관찰합니다. 이벤트의 >90%가 각 플롯 내에 있고 VL2-H 및 BL1-H 채널을 포화시키는 이벤트가 없는지 확인합니다.
- 샘플 실행을 계속합니다. 샘플당 최소 10,000개의 이벤트를 기록해야 합니다.
- 샘플의 데이터를 .fcs 파일 형식으로 저장하고 분석을 위해 내보냅니다.
5. 유세포 분석 결과의 데이터 분석
참고: 이 데이터 분석 워크플로는 FlowJo 소프트웨어를 사용합니다. 다른 유세포 분석 소프트웨어를 사용하는 경우, 소프트웨어에 적합한 도구를 사용하여 아래에 설명된 주요 단계를 계속 따르십시오. 플롯과 게이팅을 시각화하려면 보충 그림 S3 및 보충 그림 S4를 참조하십시오.
- 새 레이아웃을 열고 4.3단계에서 가져온 .fcs 파일을 엽니다. .fcs 파일을 레이아웃 창으로 끌어다 놓습니다.
- 살아있는 세포를 위한 게이트.
- 염색되지 않은 WT 컨트롤을 두 번 클릭하여 이 샘플에 대한 창을 엽니다.
- YL2-H 채널(PI를 사용하는 경우) 또는 RL2-H(TR을 사용하는 경우) 채널에서 데이터를 히스토그램으로 봅니다. 이것과 생존도 염료로 염색된 샘플 사이를 전환하여 살아있는 개체군과 죽은 개체군을 식별합니다.
참고: 이 샘플에 생존도 염료를 첨가하지 않았으므로 모든 이벤트는 염색되지 않아야 합니다. - 살아있는 인구와 죽은 인구를 나누는 이등분 게이트를 만듭니다. 왼쪽 게이트의 이름을 Live 로 지정하고 오른쪽 게이트의 이름을 Dead로 지정합니다. 이 게이트를 모든 샘플에 적용한 다음 샘플 사이를 전환하여 이 게이트가 모든 샘플에 적절하게 그려지도록 합니다. 필요에 따라 게이트를 조정합니다.
- 셀의 게이트입니다.
- 염색되지 않은 WT 컨트롤에서 라이브 게이트를 두 번 클릭하여 view 해당 게이트의 이벤트. 도트 플롯 x축 을 FSC-A 로 변경하고 y축 을 SSC-A로 변경합니다.
- 폴리곤 게이트 툴을 사용하여 셀 모집단 주위에 게이트를 그리고 이름을 Cells로 지정합니다. 파편/죽어가는 세포(일반적으로 점도표의 맨 왼쪽과 맨 아래)와 응집체(점도표의 맨 오른쪽 및 맨 위)를 제외하도록 주의하십시오.
- 모든 샘플에 대해 라이브 게이트 아래에 이 게이트를 적용합니다. 샘플 사이를 전환하여 게이트가 모든 샘플에 대해 가능한 세포 집단을 포함하는지 확인하고 필요한 조정을 수행합니다. 게이트를 변경한 후 모든 샘플에 다시 적용해야 합니다.
참고: 세포 집단 분포는 FSC 대 SSC에서 기아 상태와 비고갈된 조건 사이에서 눈에 띄게 변합니다. 셀 게이트가 모든 조건에서 셀 모집단을 포함하는지 확인합니다.
- pH 측정의 품질을 높이기 위한 단일 셀용 게이트.
- 염색되지 않은 WT 컨트롤에서 셀 게이트를 두 번 클릭하여 view 해당 게이트의 이벤트. 도트 플롯 x축 을 FSC-A 로 변경하고 y축 을 FSC-H로 변경합니다.
- 이 점도표에서 단일항 집단의 오른쪽 하단에 2차 집단을 형성하는 이중항이 있는 단세포의 대각선 분포를 찾습니다( 보충 그림 S1 세 번째 그림 참조). 폴리곤 게이트 도구를 사용하여 이중항 모집단을 제외한 단일항 이벤트 주위에 게이트를 그립니다. 이 게이트의 이름을 Singlets로 지정합니다.
- 모든 샘플에 대해 Cell 게이트 아래에 Singlets 게이트를 적용합니다. 다시 말하지만, 표본 간에 전환하여 게이트가 이중항 모집단을 적절하게 제외하는 동시에 단일항 모집단을 포함하는지 확인합니다. 필요에 따라 조정합니다.
- 형광 pHL 세포용 게이트.
- 염색되지 않은 WT 대조군 표본에서 Singlets 게이트를 두 번 클릭하여 해당 모집단에 대한 점도표를 엽니다. x축 을 BL1-H 로, y축 을 VL2-H로 변경합니다.
- pH 센서 pHluorin2는 VL2 및 BL1 모두에서 형광을 발합니다. 다각형 게이트 도구를 사용하여 점도표의 왼쪽 아래에 있는 자가형광 모집단에서 위쪽과 바로 떨어진 곳으로 확장되는 대각선 모양의 게이트를 그립니다. 이 게이트의 이름을 pHL+로 지정합니다.
- 모든 샘플에 대해 Singlets 게이트 아래에 pHL+ 게이트를 적용합니다. pHL 샘플로 전환하고 WT 컨트롤보다 VL2-H 및 BL1-H 모두에서 형광 강도가 더 큰 이벤트를 포함하도록 게이트를 조정합니다. 이 게이트가 모든 s에 대해 이 형광 집단을 포함하는지 확인하십시오.amp글리코솜 pH가 변함에 따라 이 집단의 위치가 이동하므로 les.
참고: pHL+ 집단의 이러한 작지만 눈에 띄는 변화는 형광단 여기 스펙트럼의 pH 의존적 변화 때문입니다.
- 통계를 내보냅니다.
- Table Editor(테이블 편집기) | 편집 막대 | Add Column(열 추가)을 클릭하여 내보낼 새 통계를 추가합니다.
참고: 내보낼 각 통계에 대해 해당 통계와 내보낼 모집단을 선택합니다. 적용 가능한 통계에 대해 적절한 매개 변수(예: 중앙값)를 선택해야 합니다. 샘플을 변경하지 않고 그대로 둡니다. - 총계(unated) 개수, pHL+ 개수, 총계의 라이브 빈도(총 이벤트에 대한 백분율), 상위의 pHL+ 빈도(상위 게이트에 대한 백분율), pHL+ 중앙값 VL2-H 및 pHL+ 중앙값 BL1-H 통계가 있는 열을 추가합니다.
- 테이블 편집기를 클릭하고 내보내기 설정을 [파일로] 및 [텍스트]를 [CSV로]로 변경한 다음 파일 대상과 이름을 선택합니다. Create Table(테이블 만들기)을 클릭합니다.
- Table Editor(테이블 편집기) | 편집 막대 | Add Column(열 추가)을 클릭하여 내보낼 새 통계를 추가합니다.
- 형광 비율을 계산합니다.
- 내보낸 .csv 파일을 스프레드시트 파일로 저장합니다.
- 실험의 모든 샘플에서 샘플당 이벤트 수, 라이브 이벤트 백분율 및 pHL+ 이벤트 백분율을 비교하여 품질 관리 분석을 수행합니다. 실험에 따라 막대 또는 산점도에서 시각적으로 비교합니다.
- 새 열에 pHL+ 중앙값 VL2/BL1로 레이블을 지정합니다. 각 샘플에 대해 등식 (1)과 같이 VL2-H 중앙값을 BL1-H 중앙값으로 나눕니다.
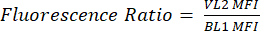 (1)
(1)
- 통계 분석 소프트웨어를 사용하여 형광 비율을 사용하여 통계 분석을 수행합니다.
6. pH 바이오센서 교정
참고: 측정된 형광 비율을 pH 단위로 변환하려면 니게리신과 발리노마이신을 사용하여 pHL 발현 세포를 보정합니다. 니제리신은 완충액(15)에 충분한 K+가 있을 때 멤브레인을 가로질러 pH를 평형화할 수 있는 이온단인 K+/H+ 항운반체이다. 니제리신은 일반적으로 pHluorin 및 기타 pH 센서를 교정하는 데 사용되어 왔습니다(16,17). peroxisomally localized pHluorin은 이전에 10μM nigericin18을 사용하여 보정되었으므로 해당 농도로 처리하기로 결정했습니다. 발리노마이신(Valinomycin)은 칼륨 이오단단(potassium ionophore)이며 미토콘드리아 막의 pH를 평형화하기 위해(4μM에서) 사용되어 왔다19. 10μM 발리노마이신을 사용하여 K+ 이온이 멤브레인 전체에서 평형을 이루도록 하여 니제리신의 pH 평형 활성을 지원했습니다. 니제리신-발리노마이신 조합을 사용했지만, 니게리신은 세포 소기관 pH를 평형화하는 데 충분할 수 있습니다.
- 범용 보정 완충액(UCB, 15mM MES, 15mM HEPES 및 130mM KCl)의 8가지 용액을 각각 5에서 8.5 사이의 서로 다른 pH에서 준비합니다.
- 원심분리기(RT, 10분, 800-1,000 × g) 2mL의 pHL 발현 BSF 배양액의 개별 튜브 8개(각각 ~4 × 106 개 세포).
- 상층액을 제거한 다음 각 세포 펠릿을 다른 pH 값으로 UCB에 재현탁시킵니다.
- 니제리신과 발리노마이신을 각각 10μM로 도입합니다. PI를 1μg/mL로 급증시킵니다.
- 각 용액에서 세포를 15분 동안 배양합니다.
- 유세포 분석기에서 각 샘플을 실행하여 4-5단계에 설명된 대로 형광 비율을 측정합니다.
- 이 실험을 두 번 더 반복하여 각 pH 값에 대해 세 번의 생물학적 반복을 얻습니다. 데이터를 .fcs 형식으로 내보냅니다.
- 5.1-5.8단계에 설명된 대로 .fcs 파일을 분석합니다. 각 pH에 대해 측정된 형광 비율을 사용하여 방정식 (2)를 사용한 향후 실험에서 글리코솜 pH를 보간합니다.
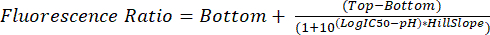 (2)
(2)
참고: 보충 그림 S3 은 점도표와 게이팅 구성표를 보여줍니다. 결과는 보충표 S2에서 확인할 수 있습니다. 다음은 GraphPad Prism을 사용하여 형광 비율에서 pH를 보간하는 방법을 설명합니다. 다른 통계 소프트웨어를 사용하는 경우 동일한 주요 단계를 따르십시오.- GraphPad Prism을 열고 세 개의 y-반복실험이 있는 XY 테이블을 만듭니다. x-컬럼의 pH 값과 y-반복실험 컬럼의 관련 형광 비율 값을 붙여넣습니다.
- 테이블과 연결된 그래프를 클릭합니다. 그래프를 볼 때 Analysis(분석) 리본 아래에서 Analyze(분석) | XY 해석에서 표준 곡선을 보간합니다.
- 시그모이드, 4PL을 선택하고, pH 단위는 로그 스케일이므로 X는 로그(농도)입니다. 위쪽 및 아래쪽 매개변수는 추정된 위쪽 및 아래쪽 고원입니다. 이상치에 대한 특별한 처리 없음을 선택합니다.
알림: 소프트웨어는 방정식 (2) 및 단계 6.9.3에 설명된 모델에 데이터를 맞추려고 합니다. 결과 표와 그래프의 곡선에서 적합치 부족의 증거를 찾습니다. - 다른 실험의 형광 비율에서 pH를 보간하려면 pHL 보정 데이터가 있는 표로 이동하십시오. 형광 비율 값을 y-column(s)의 보정 데이터 아래에 붙여넣습니다. 각 y-값에 제목을 지정하되 x-값(pH)은 알 수 없으므로 비워 둡니다.
- 결과 갤러리에서 보간 해석을 클릭한 다음 보간된 X 반복실험 탭으로 이동합니다. 입력된 형광 비율 값과 함께 나열될 보간된 pH 값을 찾습니다.
참고: 이 소프트웨어는 보정 데이터에서 찾은 모델 및 최적 파라미터 값을 사용하여 pH를 알 수 없는 실험의 형광 비율에서 pH를 보간합니다.
7. 포도당 결핍 및 추가 시간 과정
- 1.1단계에서 설명한 대로 37°C에서 배양한 1μg/mL 독시사이클린으로 밤새 15mL의 BSF pHL 기생충을 유도합니다.
- 10mM 포도당이 보충된 PBS에서 유도된 pHL 배양 15mL를 세척합니다. 이 단계를 3번 반복합니다.
- 동시에 3.2단계에서 설명한 대로 PBS 3x에서 WT 배양액 3mL를 세척합니다.
- pHL 샘플이 1mL의 PBS에 재현양될 때 첫 번째 세척 후 pHL 세포 현탁액 0.1mL를 제거하여 10mM 포도당을 굶주리지 않은 대조군으로 유지합니다.
- 최종 세척 후 pHL 샘플을 1μg/mL PI가 보충된 PBS에 재현탁시킵니다.
- 유세포 분석 데이터 수집
- 실험 및 샘플에 따라 5분, 10분, 30분 또는 90분마다 유세포 분석기에서 각 샘플을 측정하여 결핍된 샘플과 굶주리지 않은 샘플 모두에 대한 글리코솜 pH 모니터링을 시작합니다. 4.1 - 4.3 단계에서 설명한 대로 유세포 분석 데이터를 획득하여 전압과 플롯이 미리 준비되었는지 확인합니다.
- 0분에 염색되지 않은 WT 컨트롤을 먼저 실행합니다.
- 0분부터 시작하여 90분마다 세포분석기에서 굶주리지 않은 대조군 검체를 실행합니다. 굶주림 시간 경과 분석의 경우 0분부터 10분마다 굶주린 샘플을 실행합니다. Glucose Addback 분석의 경우 0, 5, 10, 20, 30, 60 및 90분에 굶주린 샘플을 실행합니다. 그런 다음 포도당을 주입한 후 반복하십시오.
- 90분 결핍 시간 과정과 180분 포도당 Addback 시간 과정 동안 실온에서 샘플을 배양합니다.
- 5.1-5.8단계에 설명된 대로 .fcs 파일을 분석합니다.
참고: 보충 그림 S4 는 게이팅을 수행하는 방법과 점도표가 어떻게 생겼는지 보여줍니다. 보충표 S3 및 보충표 S4 는 각각 포도당 결핍 시간 경과 분석 및 포도당 추가 시간 경과 분석의 결과를 나타낸다.
8. 약물 스크리닝을 위한 분석 최적화
- 약 32mL의 pHL 발현 BSF 기생충과 3mL의 WT 세포를 각각 약 2 × 106 cells/mL, PBS에서 2배로 세척합니다.
참고: 측정된 형광 비율의 변동성을 최소화하고 Z 인자 통계를 정확하게 측정하려면 웰당 10,000개 이상의 이벤트를 기록해야 합니다. 이를 위해 필요한 최소 배양량은 약 26mL이지만 취급 용이성을 위해 32mL를 권장합니다.- 첫 번째 세척 후 pHL 샘플을 두 개의 마이크로 원심분리기 튜브로 균등하게 분리합니다.
- 세척 후, 5mM 글루코스, 0.1% DMSO 및 TR을 함유하는 18mL의 PBS에 하나의 pHL 샘플을 재현탁시킵니다. 다른 pHL 샘플을 18mL의 PBS와 0.1% DMSO 및 TR에 재현탁시킵니다.
참고: DMSO는 화합물이 일반적으로 DMSO에 용해되기 때문에 약물 스크리닝에서 완충액 조성을 모방하는 데 사용됩니다. - 이 두 세포 용액을 12웰 저장소(웰당 9mL)로 옮깁니다.
- 피펫팅 로봇을 사용하여 세포 용액을 웰당 80μL의 384웰 플레이트의 별도 반으로 피펫팅합니다.
- 플레이트를 실온에서 1.5시간 동안 배양하고 부드럽게 흔들고 알루미늄 호일로 싸서 빛으로부터 형광단을 보호합니다.
- 384웰 플레이트를 실행할 수 있는 유세포 분석기에서 플레이트를 실행합니다.
참고: 다음 워크플로우는 Cytkick Max Auto Sampler가 포함된 Attune NxT에 맞게 조정되었습니다. 다른 유세포 분석기를 사용하는 경우 주요 단계를 계속 따르십시오.- 플레이트의 경우 가장 빠른 유속(1,000uL/min)으로 실행하고 부스트 모드를 활성화합니다. 20 μL/웰을 획득합니다. 각 웰 사이에 하나의 혼합 주기와 하나의 헹굼 과정을 포함합니다.
- 4.1.3단계에서 설명한 대로 플롯을 만듭니다.
- 염색되지 않은 WT 튜브 제어를 실행하고 vol을 최적화합니다.tag4.1.3 및 4.1.4단계에 설명된 대로. 굶주린 pHL 튜브 샘플을 실행하고 4.1단계에서 설명한 대로 VL2 및 BL1 전압을 최적화합니다.
- 유세포 분석기에서 플레이트를 획득하기 시작합니다. 플레이트를 수평으로 실행하여 결핍된 샘플 웰과 결핍되지 않은 샘플 웰 사이에 큰 획득 시간 차이가 없도록 합니다. 플레이트 획득이 ~1.5시간 내에 완료되었는지 확인합니다.
- .fcs 파일을 내보내고 5.1-5.8단계에 설명된 대로 데이터를 분석합니다. 포도당(Glc) 또는 포도당이 없는(Starved) 처리된 샘플에 대한 형광 비율의 평균(AVG) 및 표준 편차(SD)를 구합니다.
참고: Z 인자 실험의 데이터는 보충 표 S5에서 찾을 수 있습니다. - 방정식 (3)을 사용하여 Z-계수20 통계량을 계산합니다.
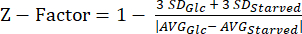 (3)
(3)
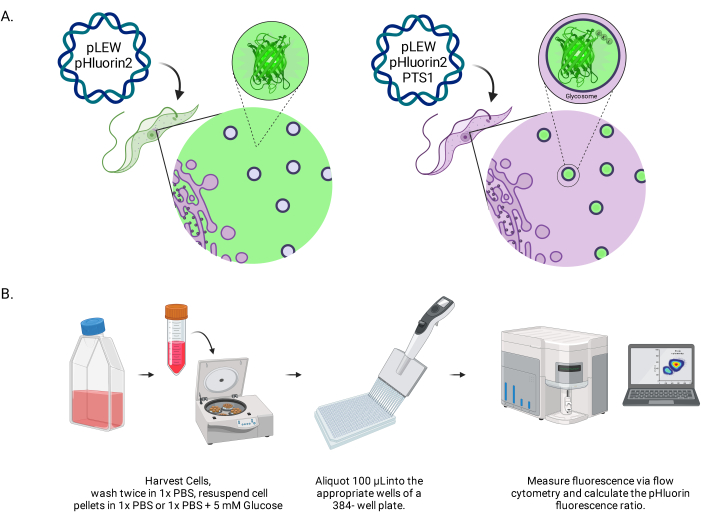
그림 1: 살아있는 BSF 트리파노솜에서 글리코솜 pH를 채점하는 방법의 다이어그램. (A) 글리코솜에 위치한 pHluorin2 센서를 발현하는 세포주의 묘사. 퍼옥시솜 표적화 염기서열을 포함하면 국소화를 제어할 수 있습니다. 참고: PTS-1을 제거하면 세포질 국소화가 가능하여 해당 세포 내 구획의 pH를 향후 분석할 수 있을 것으로 예상합니다. (B) 센서 검증 분석의 묘사. 약어: BSF = 혈류 형태. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
참고: Z-인자 통계량은 분석이 HTS에 얼마나 적합한지 결정하는 데 사용됩니다. 0.5에서 1.0 사이의 값은 일반적으로 분석 품질이 HTS에 적합함을 의미합니다.
결과
BSF T. brucei의 당체에 대한 pHLuorin2-PTS1 국소화
pHluorin2-PTS1의 세포 내 국소화를 평가하기 위해 기생충을 면역형광 분석으로 실시했습니다. 글리코솜 상주 단백질인 알돌라아제(TbAldolase)에 대해 제기된 항혈청과 함께 국소화된 전이유전자의 신호(그림 2A). 항-TbAldolase와 pHluorin2-PTS1 사이의 평균 Pearson's colocalization 상관계수는 0.895였으며, 이는 pHluorin2-PTS1이 주...
토론
아프리카 트리파노솜의 환경 인식과 반응 메커니즘은 잘 알려져 있지 않습니다. 영양소 가용성의 변화는 글리코솜의 산성화를 포함하여 기생충의 다양한 반응을 유발하는 것으로 알려져 있습니다. 여기에서는 유전성 단백질 센서, pHluorin2 및 유세포 분석을 사용하여 살아있는 세포의 환경 섭동에 대한 글리코솜 pH 반응을 연구하는 방법을 설명했습니다.
센서 사용에는 몇 가?...
공개
저자는 이해 상충이 없음을 선언합니다.
감사의 말
pHluorin2-PTS1은 고-카피 클로닝 벡터에서 작제물을 제공한 Twist Bioscience에 의해 pLEW100v5로 클로닝되었습니다. pLEW100v5는 조지 크로스 박사의 선물이었습니다. T. brucei aldolase에 대해 제기된 항혈청은 요청 시 Clemson University의 Meredith T. Morris 박사로부터 구할 수 있습니다. JCM과 KAC 실험실의 연구는 미국 국립보건원(National Institutes of Health, R01AI156382)의 상에 의해 부분적으로 지원되었습니다. SSP는 NIH 3R01AI156382에 의해 지원되었습니다.
자료
| Name | Company | Catalog Number | Comments |
| 50 mL Tissue Culture Flasks (Non-treated, sterile) | VWR | 10861-572 | |
| 75 cm2 Tissue Culture Flask (Non-Treated, sterile) | Corning | 431464U | |
| 80 µL flat-bottom 384-well plate | BrandTech | 781620 | |
| Amaxa Human T Cell Nucleofector Kit | Lonza | VPA-1002 | |
| Attune NxT Flow Cytometer | invitrogen by Thermo Fisher Scientific | A24858 | FlowJo software |
| BRANDplates 96-Well, flat bottom plate | Millipore Sigma | BR781662 | |
| Coloc 2 plugin of ImageJ | https://imagej.net/plugins/coloc-2 | ||
| CytKick Max Auto Sampler | invitrogen by Thermo Fisher Scientific | A42973 | |
| CytoFLEX Flow Cytometer | Beckman-Coulter | ||
| Electron Microscopy Sciences 16% Paraformaldehyde Aqueous Solution, EM Grade, 10 mL Ampoule | Fisher Scientific | 50-980-487 | |
| GraphPad Prism | statistical software | ||
| Nigericin (sodium salt) | Cayman Chemical | 11437 | |
| Nucleofector 2b | Lonza | Discontinued Product | |
| OP2 Liquid Handler | opentrons | OP2 | |
| poly-L-lysine, 0.1% (w/v) in H2O | Sigma Life Science | CAS:25988-63-0 | Pipetting robot for HTS assay |
| Thiazole Red (TO-PRO-3) | biotium | #40087 | We machined a custom acrylic plate stand so this brand of plate could be detected and used on our CytKick Max Auto Sampler |
| valinomycin | Cayman Chemical | 10009152 | Pipetting robot for HTS assay |
| For pH calibration | |||
| For pH calibration |
참고문헌
- Coley, A. F., Dodson, H. C., Morris, M. T., Morris, J. C. Glycolysis in the African trypanosome: Targeting enzymes and their subcellular compartments for therapeutic development. Molecular Biology International. 2011, 123702 (2011).
- Mcconville, M. J., Saunders, E. C., Kloehn, J., Dagley, M. J. Leishmania carbon metabolism in the macrophage phagolysosome- feast or famine. F1000Res. 4, 938 (2015).
- Parsons, M. Glycosomes: Parasites and the divergence of peroxisomal purpose. Molecular Microbiology. 53 (3), 717-724 (2004).
- Parsons, M., Furuya, T., Pal, S., Kessler, P. Biogenesis and function of peroxisomes and glycosomes. Molecular and Biochemical Parasitology. 115 (1), 19-28 (2001).
- Haanstra, J. R., Gonzalez-Marcano, E. B., Gualdron-Lopez, M., Michels, P. A. Biogenesis, maintenance and dynamics of glycosomes in trypanosomatid parasites. Biochimica et Biophysica Acta. 1863 (5), 1038-1048 (2016).
- Allmann, S., Bringaud, F. Glycosomes: A comprehensive view of their metabolic roles in t. Brucei. International Journal of Biochemistry and Cell Biology. 85, 85-90 (2017).
- Dodson, H. C., Morris, M. T., Morris, J. C. Glycerol 3-phosphate alters Trypanosoma brucei hexokinase activity in response to environmental change. The Journal of Biological Chemistry. 286 (38), 33150-33157 (2011).
- Lin, S., Morris, M. T., Ackroyd, P. C., Morris, J. C., Christensen, K. A. Peptide targeted delivery of pH sensor for quantitative measurements of intraglycosomal pH in live Trypanosoma brucei. Biochemistry. 52 (21), 3629-3637 (2013).
- Mahon, M. J. Phluorin2: An enhanced, ratiometric, pH-sensitive green florescent protein. Advances in Bioscience and Biotechnology. 2 (3), 132-137 (2011).
- Wirtz, E., Leal, S., Ochatt, C., Cross, G. A. A tightly regulated inducible expression system for conditional gene knock-outs and dominant-negative genetics in Trypanosoma brucei. Molecular and Biochemical Parasitology. 99 (1), 89-101 (1999).
- . Restriction digest v.2 Available from: https://www.protocols.io/view/restriction-digest-nkqdg33pg25z/v2 (2018)
- . Ligation protocol with t4 DNA ligaase (m0202) v.3 Available from: https://www.protocols.io/view/ligation-protocol-with-t4-dna-ligase-m0202-95qpvorzv4o1/v3 (2021)
- Burkard, G., Fragoso, C. M., Roditi, I. Highly efficient stable transformation of bloodstream forms of Trypanosoma brucei. Molecular and Biochemical Parasitology. 153 (2), 220-223 (2007).
- Crowe, L. P., Wilkinson, C. L., Nicholson, K. R., Morris, M. T. Trypanosoma brucei pex13.2 is an accessory peroxin that functions in the import of peroxisome targeting sequence type 2 proteins and localizes to subdomains of the glycosome. mSphere. 5 (1), e00744 (2020).
- Kucejova, B., Kucej, M., Petrezselyova, S., Abelovska, L., Tomaska, L. A screen for nigericin-resistant yeast mutants revealed genes controlling mitochondrial volume and mitochondrial cation homeostasis. Genetics. 171 (2), 517-526 (2005).
- Huynh, M. H., Carruthers, V. B. Toxoplasma gondii excretion of glycolytic products is associated with acidification of the parasitophorous vacuole during parasite egress. PLoS Pathogens. 18 (5), e1010139 (2022).
- Lehoux, S., Abe, J., Florian, J. A., Berk, B. C. 14-3-3 binding to Na+/H+ exchanger isoform-1 is associated with serum-dependent activation of Na+/H+ exchange. TheJournal of Biological Chemistry. 276 (19), 15794-15800 (2001).
- Jankowski, A., et al. In situ measurements of the ph of mammalian peroxisomes using the fluorescent protein phluorin. The Journal of Biological Chemistry. 276 (52), 48748-48753 (2001).
- Jankowski, A., Grinstein, S. A. A noninvasive fluorimetric procedure for measurement of membrane potential. Quantification of the NADPH oxidase-induced depolarization in activated neutrophils. The Journal of Biological Chemistry. 274 (37), 26098-26104 (1999).
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A simple statistical parameter for use in evaluation and validation of high throughput screening assays. Journal of Biomolecular Screening. 4 (2), 67-73 (1999).
- Miesenbock, G., De Angelis, D. A., Rothman, J. E. Visualizing secretion and synaptic transmission with pH-sensitive green fluorescent proteins. Nature. 394 (6689), 192-195 (1998).
- Lin, S., et al. Ph regulation in glycosomes of procyclic form Trypanosoma brucei. The Journal of Biological Chemistry. 292 (19), 7795-7805 (2017).
- Ha, D. S., Schwarz, J. K., Turco, S. J., Beverley, S. M. Use of the green fluorescent protein as a marker in transfected Leishmania. Molecular and Biochemical Parasitology. 77 (1), 57-64 (1996).
- Kelly, J. M., Ward, H. M., Miles, M. A., Kendall, G. A shuttle vector which facilitates the expression of transfected genes in Trypanosoma cruzi and Leishmania. Nucleic Acids Research. 20 (15), 3963-3969 (1992).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유