É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Medindo as alterações dinâmicas do pH glicossomal em Trypanosoma brucei vivo
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Descrevemos um método para estudar como o pH responde a pistas ambientais nos glicossomas da forma sanguínea de tripanossomas africanos. Essa abordagem envolve um sensor de proteína hereditária sensível ao pH em combinação com citometria de fluxo para medir a dinâmica do pH, tanto como um ensaio de curso de tempo quanto em um formato de tela de alto rendimento.
Resumo
O metabolismo da glicose é crítico para o tripanossoma africano, Trypanosoma brucei, como um processo metabólico essencial e regulador do desenvolvimento do parasita. Pouco se sabe sobre as respostas celulares geradas quando os níveis de glicose ambiental se alteram. Tanto na corrente sanguínea quanto na forma pró-cíclica (estágio de inseto) parasitas, os glicossomas abrigam a maior parte da glicólise. Essas organelas são rapidamente acidificadas em resposta à privação de glicose, o que provavelmente resulta na regulação alostérica de enzimas glicolíticas como a hexoquinase. Em trabalhos anteriores, localizar a sonda química usada para fazer medições de pH foi um desafio, limitando sua utilidade em outras aplicações.
Este trabalho descreve o desenvolvimento e o uso de parasitas que expressam pHluorina glicossomicamente localizada2, um biossensor de pH de proteína hereditária. A pHluorina2 é uma variante ratiométrica da pHluorina que apresenta uma diminuição da excitação dependente do pH (ácido) a 395 nm, ao mesmo tempo que produz um aumento da excitação a 475 nm. Parasitas transgênicos foram gerados pela clonagem do quadro de leitura aberta pHluorin2 no vetor de expressão de tripanossomas pLEW100v5, permitindo a expressão de proteínas induzíveis em ambos os estágios do ciclo de vida. A imunofluorescência foi utilizada para confirmar a localização glicossomal do biossensor pHluorin2, comparando a localização do biossensor com a aldolase da proteína residente no glicossomo. A responsividade do sensor foi calibrada em diferentes níveis de pH incubando as células em uma série de tampões que variaram em pH de 4 a 8, uma abordagem que usamos anteriormente para calibrar um sensor de pH baseado em fluoresceína. Em seguida, medimos a fluorescência da pHluorina2 a 405 nm e 488 nm usando citometria de fluxo para determinar o pH glicossomal. Validamos o desempenho dos parasitas transgênicos vivos que expressam pHluorina2, monitorando o pH ao longo do tempo em resposta à privação de glicose, um conhecido gatilho da acidificação glicossomal em parasitas PF. Esta ferramenta tem uma gama de aplicações potenciais, incluindo potencialmente ser usada na triagem de drogas de alto rendimento. Além do pH glicossomal, o sensor pode ser adaptado a outras organelas ou usado em outros tripanosomatídeos para entender a dinâmica do pH no ambiente de células vivas.
Introdução
Os cinetoplastídeos parasitas, como a maioria dos organismos vivos, dependem da glicose como um componente fundamental do metabolismo central do carbono. Este grupo inclui organismos medicamente importantes, como o tripanossoma africano, Trypanosoma brucei; o tripanossoma americano, T. cruzi; e parasitas do gênero Leishmania. O metabolismo da glicose é crítico para o crescimento do parasita nos estágios do ciclo de vida patogênico. Por exemplo, quando privado de glicose, a forma sanguínea (BSF) do tripanossoma africano morre rapidamente. Notadamente, a glicólise serve como única fonte de ATP durante esse estágio da infecção1. Os parasitas de Leishmania também são dependentes de glicose no hospedeiro humano, sendo o estágio do ciclo de vida amastigota que reside nos macrófagos do hospedeiro dependente dessa fonte de carbono para o crescimento2.
Embora esses parasitas tenham estilos de vida distintos envolvendo diferentes insetos vetores, eles compartilham muitas semelhanças em como respondem e consomem glicose. Por exemplo, esses parasitas localizam a maioria das enzimas glicolíticas em peroxissomas modificados chamados glicossomas. Esta organela cinetoplastídica-específica está relacionada aos peroxissomas de mamíferos com base em mecanismos biossintéticos conservadose morfologia 3,4,5,6.
A compartimentalização da maioria das enzimas da via glicolítica no glicossoma oferece meios parasito-específicos de regulação da via. Usando uma sonda química de pH, estabelecemos que a privação de nutrientes desencadeia uma rápida acidificação dos glicossomas parasitas da forma pró-cíclica (PF) que resulta em alteração da atividade enzimática glicolítica através da exposição de um sítio de ligação do regulador alostérico na enzima glicolítica chave hexoquinase 7,8. Em nosso trabalho anterior, a sonda química exigia entrega constante para uso, limitando sua utilidade em outras aplicações. Além disso, desafios na manutenção da distribuição da sonda no glicossomo na BSF limitaram a utilidade da abordagem para a investigação do pH glicossomal nessa fase da vida.
Neste estudo, usamos o biossensor de proteína fluorescente pHluorin2 para monitorar a mudança de pH glicossomal em BSF T. brucei em resposta a pistas ambientais, incluindo a falta de glicose9 (Figura 1). Os resultados deste trabalho sugerem que o FSB T. brucei acidifica glicossomas rapidamente em resposta à inanição de forma reversível, semelhante às respostas que observamos em parasitas da FP. Esperamos que este biossensor melhore nossa compreensão da regulação glicolítica em T. brucei e parasitas relacionados.
Protocolo
O uso de tripanossomas de T. brucei brucei 90-13 BSF, uma linhagem de parasitas monomórficos, requer consideração de segurança, pois são considerados organismos do Grupo de Risco 2 que devem ser manuseados em instalações de nível de biossegurança 2.
1. Cultura e transfecção de tripanossomas
- Cultura de tripanossomas de T. brucei brucei 90-13 BSF em meio HMI-9 suplementado com 10% de SFB inativado pelo calor e 10% de Nu-soro a 37 °C em 5% de CO2 10.
NOTA: Para manter a cultura saudável, mantenha a densidade celular entre 2 × 104 e 5 × 106 células/mL. - Clonagem de pHluorin2-PTS1 em pLEW100v5
- Sintetizar comercialmente o quadro de leitura aberto pHluorin2 para produzir o gene com uma extensão de 3' que codifica um ligante de duas glicinas seguido pelo tripeptídeo AKL, uma etiqueta de localização PTS1.
- Clone esta construção no vetor de expressão induzível de tripanossomas pLEW100v5 por digesto de restrição. Digestão dupla do vetor de clonagem contendo pHluorin2-PTS1 e pLEW100v5 usando HindIII e BamHI11. Execute uma etapa de limpeza, de preferência por purificação em gel de agarose, para remover as enzimas de restrição, a espinha dorsal do vetor de clonagem indesejado e o fragmento contendo o gene da luciferase excisado.
- Ligate a pastilha de rolamento de pHluorin2 em pLEW100v5 digerido com T4 DNA Ligase12. (ver Figura suplementar S1 para um esquema de clonagem).
- Sequenciar o plasmídeo por sequenciamento de plasmídios inteiros de próxima geração para verificar se a inserção e o vetor estão ligados corretamente e se nenhuma mutação é introduzida no gene pHluorin2-PTS1 durante o processo de clonagem.
NOTA: A sequência completa do plasmídeo foi submetida a Addgene.org e recebeu o #83680. - Linearizar 20 μg do plasmídeo digerindo com 40 unidades de NotI; em seguida, transfectar em parasitas BSF 90-13 via nucleofeção usando o kit de células T humanas referenciadas (ver Tabela de Materiais). Selecione para integração estável conforme descrito por Burkard et al.13.
2. Colocalização por imunofluorescência da pHlourin2-PTS1
- Preparar lâminas de microscopia tratando-as com poli-L-lisina 0,1% (p/v) em H2O por 10 min. Após a remoção da solução de poli-L-lisina, lave a lâmina uma vez com PBS.
- Para induzir a expressão de pHluorina2-PTS1, tratar as células em 2 × 105 células/mL com tetraciclina ou doxiciclina (1 μg/mL) 24 h antes da colheita. Pellet 2 × 106 parasitas parentais e induzidos pLEWpHluorin2-PTS1 (pHL) por centrifugação (temperatura ambiente [TR], 10 min, 1.000 × g) e lavar uma vez com PBS.
- Ressuspender as células em 200 μL de paraformaldeído a 2% recém-preparado em PBS feito a partir de solução de grau EM a 16% fornecida comercialmente. Aplique as células do fixador na lâmina e deixe que as células se acomodem por 30 min. Lavar as células aderidas 2x com solução de lavagem (0,1% de soro normal de cabra em PBS).
- Aplicar a solução de permeabilização (Triton X-100 a 0,5% em PBS) nas células e incubar por exatos 30 min. Retire a solução de permeabilização e lave uma vez com grandes quantidades de solução de lavagem.
- Aplicar a solução de bloqueio (soro normal de cabra a 10% e Triton X-100 a 0,1% em PBS) e incubar durante 30 minutos.
- Diluir os antissoros levantados contra T. brucei aldolase 1:500 em solução de bloco e aplicá-lo nas células14. Incube por 1 h em RT. Lave as lâminas por 5x por 3-5 min com solução de lavagem.
- Diluir cabra anti-coelho Alexa fluor 568 1:1.000 em solução de bloco e aplicar nas células. Incubar por 1 h em RT. Lave as lâminas por 5x por 3-5 min com solução de lavagem.
- Aplique o meio de montagem e sele uma lamínula na corrediça.
- Fotografe as células com uma objetiva de 100x (NA 1.4-0.7) e analise as imagens usando o ImageJ. Execute a análise de colocalização da Pearson usando o plug-in 'Coloc 2' para ImageJ. Consulte a Figura suplementar S2 para obter um campo de células representativas. )
- Para concluir isso, adicione o arquivo de imagem a Fiji e selecione Imagens.
- Ajuste o brilho e o contraste de cada canal até um ponto em que nenhum plano de fundo esteja visível.
- Altere as imagens de 16 bits para 8 bits, mescle as imagens e corte em uma única célula com canais divididos.
- Em Analisar e Colocalização, selecione o plug-in Coloc2. Selecione aldolase (o canal vermelho) como Canal 1 e pHL (o canal verde) como Canal 2 e clique em OK para iniciar o cálculo da correlação de Pearson.
3. Preparo da amostra para citometria de fluxo
- Induzir células pHL com tetraciclina ou doxiciclina (1 μg/mL) durante a noite.
- Pelotizar as células (~4 × 107 pHL e ~1 × 106 parental) por centrifugação (RT, 10 min, 1.000 × g). Remover o sobrenadante e ressuspender as células em 1 mL de PBS com ou sem glicose 10 mM, dependendo se a amostra está passando fome ou não. Para ensaios de curso de tempo, ressuspenda em PBS suplementado com glicose de 10 mM para evitar que as células passem fome até que as lavagens estejam completas. Para o ensaio de tela de alto rendimento (HTS), ressuspender em PBS sem glicose para minimizar o transporte de glicose; Repita as lavagens mais duas vezes.
- Centrifugar as células pela quarta vez, remover o sobrenadante e ressuspender a pastilha celular em PBS, PBS mais 5 mM de glicose ou PBS mais 10 mM de glicose, dependendo do tratamento. Completar as amostras com 1 μg/ml de iodeto de propídio (PI) ou 100 nM de vermelho de tiazol (TR) para determinação de vivos/mortos. Transfira cada amostra para tubos de 5 mL compatíveis com o citômetro de fluxo.
4. Citometria de fluxo
NOTA: Preparar o experimento em um citômetro de fluxo contendo os seguintes lasers: 405 nm (violeta), 488 nm (azul) e 561 nm (amarelo) ou 638 nm (vermelho). Consulte a Tabela Suplementar S1 para obter nomes comuns para canais discutidos abaixo.
- Para medir a fluorescência pHL, utilizar os canais KO525 (VL2-H, excitação 405 nm, emissão 542/27 nm) e FITC (BL1-H, excitação 488 nm, emissão 530/30 nm). Para diferenciar células vivas de células mortas, use PI ou TR; medir IP no canal YL2-H (excitação 561 nm, emissão 620/25 nm). Meça TR no canal RL1-H (excitação de 638 nm, 660/10 BP).
- Para configurar o experimento no software do citômetro de fluxo, crie os seguintes gráficos: 1) histograma do canal YL2-H (se estiver usando PI) ou um histograma do canal RL1-H (se estiver usando TR), 2) gráfico de pontos FSC-A vs SSC-A, 3) gráfico de pontos FSC-A vs FSC-H e 4) gráfico de pontos do canal BL1-H vs VL2-H.
- Coloque o controle WT (linha celular parental) não manchado na porta de injeção da amostra (SIP) primeiro e eleve o estágio para a posição. Comece a adquirir dados no citômetro na menor vazão para dar tempo de fazer os ajustes necessários. Para evitar a pontuação de detritos espúrios e células mortas, comece a registrar os eventos 5 s após o início da aquisição da amostra.
- Ajuste a tensão YL2 ou RL1 para que o pico principal esteja dentro de 103-10 4 unidades de intensidade relativa de fluorescência (RFI). Ajuste as tensões FSC e SSC para que >90% dos eventos caibam no gráfico de pontos. Ajuste o limite do FSC para excluir a população de detritos, mas não as células prováveis.
- Ajuste os canais VL2 e BL1 para que o pico primário esteja dentro de 103-10 4 unidades RFI para o controle WT sem manchas.
- Colocar a primeira amostra contendo pHL induzida corada com IP ou TR no SIP e elevar o estágio à posição. Comece a adquirir dados na menor vazão e observe cuidadosamente os eventos em cada parcela. Certifique-se de que >90% dos eventos estejam dentro de cada gráfico e que nenhum evento esteja saturando os canais VL2-H e BL1-H.
- Continue com a execução das amostras. Certifique-se de registrar pelo menos 10.000 eventos por amostra.
- Salve os dados das amostras no formato de arquivo .fcs e exporte para análise.
5. Análise dos dados dos resultados da citometria de fluxo
NOTA: Este fluxo de trabalho de análise de dados usa o software FlowJo. Se outro software de análise de citometria de fluxo for usado, continue a seguir as principais etapas descritas abaixo, usando ferramentas apropriadas ao software. Para visualizar os gráficos e o gating, consulte a Figura Suplementar S3 e a Figura Suplementar S4.
- Abra um novo layout e abra os arquivos .fcs adquiridos na etapa 4.3. Arraste e solte os arquivos .fcs na janela de layout.
- Portão para celas vivas.
- Clique duas vezes no controle WT sem manchas para abrir uma janela para este exemplo.
- Exiba os dados como um histograma no canal YL2-H (se estiver usando PI) ou RL2-H (se estiver usando TR). Alterne entre este e amostras coradas com o corante de viabilidade para identificar as populações vivas e mortas.
NOTA: Todos os eventos devem ser desmanchados, uma vez que um corante de viabilidade não foi adicionado a esta amostra. - Criar um portão de bissetriz dividindo as populações vivas e mortas; nomeie o portão esquerdo Live e o portão direito Dead. Aplique este portão a todas as amostras e, em seguida, alterne entre as amostras para garantir que este portão seja desenhado adequadamente para todas as amostras. Ajuste o portão conforme necessário.
- Portão para as celas.
- No controle WT sem manchas, clique duas vezes no portão ao vivo para exibir os eventos nesse portão. Altere o eixo x do gráfico de pontos para FSC-A e o eixo y para SSC-A.
- Use a ferramenta de porta de polígono para desenhar uma porta ao redor da população de células e nomeá-la como Células. Tome cuidado para excluir detritos/células moribundas (normalmente na extrema esquerda e na parte inferior do gráfico de pontos) e agregados (extrema direita e superior do gráfico de pontos).
- Aplique este portão sob o portão Live para todas as amostras. Alterne entre as amostras para garantir que o portão abranja a população de células provável para todas as amostras e faça os ajustes necessários. Certifique-se de reaplicar o portão em todas as amostras depois de alterá-lo.
NOTA: A distribuição da população celular muda visivelmente entre as condições de fome e não faminto em FSC vs SSC; garantir que o portão da célula abranja a população celular em todas as condições.
- Portão para células individuais para aumentar a qualidade das medições de pH.
- No controle WT sem manchas, clique duas vezes no portão Cell para exibir eventos nesse gate. Altere o eixo x do gráfico de pontos para FSC-A e o eixo y para FSC-H.
- Procure uma distribuição diagonal de células únicas neste gráfico de pontos com duplos formando uma população secundária à direita inferior da população singlete (ver Figura Suplementar S1 terceiro gráfico). Usando a ferramenta de porta de polígono, desenhe um portão em torno dos eventos singlet excluindo a população doublet. Nomeie este portão como Singlets.
- Aplique o portão Singlets sob o portão Cell para todas as amostras. Novamente, alterne entre as amostras para garantir que o portão esteja excluindo adequadamente a população de duplos, incluindo a população de singletes. Ajuste conforme necessário.
- Portão para células pHL fluorescentes.
- Na amostra de controle WT não manchada, clique duas vezes no portão de Singlets para abrir um gráfico de pontos para essa população. Altere o eixo x para BL1-H e o eixo y para VL2-H.
- O sensor de pH pHluorin2 é fluorescente tanto em VL2 quanto em BL1. Use a ferramenta de portão de polígono para desenhar um portão em forma de diagonal que se estende até o topo e à direita da população autofluorescente no canto inferior esquerdo do gráfico de pontos. Nomeie este portão pHL+.
- Aplique o portão pHL+ sob o portão Singlets para todas as amostras. Alterne para uma amostra de pHL e ajuste a porta para incluir eventos com maior intensidade de fluorescência em VL2-H e BL1-H do que o controle WT. Certifique-se de que esta porta abranja esta população fluorescente para todas as amostras, pois a posição desta população mudará à medida que o pH glicossomal muda.
NOTA: Esta mudança pequena, mas visível, na população pHL+ é devida a mudanças dependentes do pH no espectro de excitação do fluoróforo.
- Exporte as estatísticas.
- Clique em Editor de Tabelas | Barra de edição | Adicionar Coluna para adicionar novas estatísticas a serem exportadas.
NOTA: Para cada estatística a ser exportada, escolha a respectiva estatística e de qual população exportá-la. Certifique-se de escolher o parâmetro apropriado para estatísticas aplicáveis, como Mediana. Deixe a amostra inalterada. - Adicione colunas com as seguintes estatísticas: Contagem Total (unada), Contagem pHL+, Freq ao vivo do Total (porcentagem baseada no total de eventos), Freq pHL+ do Pai (porcentagem baseada na porta pai), pHL+ Mediana VL2-H e pHL+ Mediana BL1-H.
- Clique no Editor de Tabela e altere as seguintes configurações de exportação: Exibir para arquivo e Texto para CSV e, em seguida, escolha o destino e o nome do arquivo. Clique em Criar tabela.
- Clique em Editor de Tabelas | Barra de edição | Adicionar Coluna para adicionar novas estatísticas a serem exportadas.
- Calcule a razão de fluorescência.
- Salve o arquivo de .csv exportado como um arquivo de planilha.
- Realize a análise de controle de qualidade comparando o seguinte em todas as amostras do experimento: número de eventos por amostra, porcentagem de eventos ao vivo e porcentagem de eventos pHL+. Compare-os visualmente em gráficos de barras ou dispersão, dependendo do experimento.
- Rotule uma nova coluna como pHL+ Mediana VL2/BL1. Para cada amostra, divida o valor mediano de VL2-H pelo valor mediano de BL1-H, conforme mostrado na equação (1).
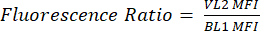 (1º)
(1º)
- Utilizar software de análise estatística para realizar análises estatísticas utilizando a razão de fluorescência.
6. Calibração do biossensor de pH
NOTA: Para converter as razões de fluorescência medidas em unidades de pH, calibre as células que expressam pHL usando nigericina e valinomicina. A nigericina é um antiporter K+/H+, um ionóforo que pode equilibrar o pH através das membranas quando há K+ suficiente no tampão15. A nigericina tem sido comumente utilizada para calibrar a pHluorina e outros sensores de pH16,17. Como a pHluorina peroxissomalmente localizada foi previamente calibrada com 10 μM de nigericina18, optou-se por tratar com essa concentração. A valinomicina é um ionóforo de potássio e tem sido utilizada (a 4 μM) para equilibrar o pH entre as membranas mitocondriais19. Usamos 10 μM de valinomicina para auxiliar a atividade de equilíbrio do pH da nigericina, garantindo que os íons K+ estivessem equilibrados através das membranas. Embora tenhamos usado uma combinação de nigericina e valinomicina, a nigericina pode ser suficiente para equilibrar o pH organelar.
- Preparar oito soluções de tampão de calibração universal (UCB; 15 mM MES, 15 mM HEPES e 130 mM KCl) cada uma a um pH diferente que varia de 5 a 8,5.
- Centrífuga (RT, 10 min, 800-1.000 × g) oito tubos separados de 2 mL de cultura BSF expressando pHL (~4 × 106 células cada).
- Remova o sobrenadante e, em seguida, ressuspenda cada pellet de célula em UCB em diferentes valores de pH.
- Introduzir nigericina e valinomicina, cada uma a 10 μM. Spike no IP a 1 μg/mL.
- Incubar as células em cada solução durante 15 min.
- Execute cada amostra em um citômetro de fluxo para medir a razão de fluorescência, conforme descrito nas etapas 4-5.
- Repetir este experimento mais duas vezes para obter três réplicas biológicas para cada valor de pH. Exporte dados no formato .fcs.
- Analise os arquivos .fcs conforme descrito nas etapas 5.1 a 5.8. Use a razão de fluorescência medida para cada pH para interpolar o pH glicossomal em experimentos futuros usando a equação (2).
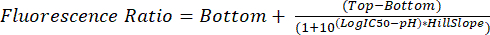 (2)
(2)
NOTA: A Figura suplementar S3 mostra os gráficos de pontos e o esquema de limitação. Os resultados podem ser encontrados na Tabela Suplementar S2. A seguir descreve-se como interpolar o pH da razão de fluorescência usando o GraphPad Prism. Se estiver usando outro software estatístico, siga os mesmos passos-chave.- Abra o GraphPad Prism e crie uma tabela XY com três réplicas y. Cole os valores de pH na coluna x e seus valores de razão de fluorescência associados nas colunas de replicação y.
- Clique no gráfico associado à tabela. Ao visualizar o gráfico, clique em Analisar na faixa de opções Análise | Interpolar uma curva padrão sob análises XY.
- Escolha Sigmoidal, 4PL, X é log(concentração) uma vez que as unidades de pH estão em uma escala logarítmica. Os parâmetros Superior e Inferior são os platôs superiores e inferiores estimados. Escolha Sem manipulação especial de outliers.
OBS: O software buscará adequar os dados ao modelo descrito na equação (2) e passo 6.9.3. Procure evidências de falta de ajuste na Tabela de resultados e na curva no gráfico. - Para interpolar o pH a partir de razões de fluorescência em outros experimentos, vá para a tabela com os dados de calibração do pHL. Cole os valores da razão de fluorescência abaixo dos dados de calibração na(s) coluna(s) y. Dê a cada valor y um título, mas deixe o valor x (o pH) em branco, pois ele é desconhecido.
- Na galeria de resultados, clique na análise de interpolação e, em seguida, vá para a guia Replicações X interpoladas . Procure os valores de pH interpolados que serão listados ao lado dos valores de razão de fluorescência inseridos.
NOTA: O software usa o modelo e os valores de parâmetro de melhor ajuste encontrados a partir dos dados de calibração para interpolar o pH das razões de fluorescência para experimentos onde o pH é desconhecido.
7. Falta de glicose e cursos de tempo de aditivo
- Induzir 15 ml de parasitas BSF pHL durante a noite com 1 μg/ml de doxiciclina incubada a 37 °C, conforme descrito no passo 1.1.
- Lavar 15 mL de cultura de LHp induzida em PBS suplementado com glicose 10 mM. Repita esta etapa 3x.
- Simultaneamente, lavar 3 mL de cultura WT em PBS 3x conforme descrito no passo 3.2.
- Após a primeira lavagem, quando a amostra de pHL for ressuspensa em 1 mL de PBS, remova uma alíquota de 0,1 mL de suspensão de células de pHL para manter em 10 mM glicose como um controle sem fome.
- Após a lavagem final, ressuspender a amostra de pHL em PBS suplementado com 1 μg/mL de IP.
- Aquisição de dados de citometria de fluxo
- Comece a monitorar o pH glicossomal para as amostras famintas e não famintas medindo cada amostra em um citômetro de fluxo a cada 5, 10, 30 ou 90 min, dependendo do experimento e da amostra. Adquira dados de citometria de fluxo conforme descrito nas etapas 4.1 a 4.3, garantindo que as tensões e gráficos sejam preparados previamente.
- Execute o controle WT sem manchas primeiro em 0 min.
- Execute a amostra de controle sem fome no citômetro a cada 90 min, começando em 0 min. Para o ensaio de curso de tempo de fome, execute a amostra faminta a cada 10 minutos a partir de 0 min. Para o ensaio de Glucose Addback, executar a amostra faminta em 0, 5, 10, 20, 30, 60 e 90 min; em seguida, repita após a introdução de glicose.
- Incubar as amostras à temperatura ambiente durante o curso de tempo de inanição de 90 min e o curso de tempo de Glucose Addback de 180 min.
- Analise os arquivos .fcs conforme descrito nas etapas 5.1 a 5.8.
Observação : figura suplementar S4 mostra como executar o gating e como os gráficos de pontos devem parecer. A Tabela Suplementar S3 e a Tabela Suplementar S4 mostram os resultados do ensaio do curso do tempo de inanição de glicose e do ensaio do curso do tempo de retorno da glicose, respectivamente.
8. Otimização do ensaio para triagem de drogas
- Lavar aproximadamente 32 mL de parasitas BSF expressando pHL e 3 mL de células WT, ambos em aproximadamente 2 × 106 células/mL, 2x em PBS, conforme descrito anteriormente.
NOTA: Mais de 10.000 eventos precisam ser registrados por poço para minimizar a variabilidade das razões de fluorescência medidas e medir com precisão a estatística do fator Z. A cultura mínima necessária para isso é de aproximadamente 26 mL, mas recomendamos 32 mL para facilitar o manuseio.- Após a primeira lavagem, separar igualmente a amostra de pHL em dois tubos de microcentrífuga.
- Após as lavagens, ressuspender uma amostra de pHL em 18 mL de PBS contendo 5 mM de glicose, 0,1% de DMSO e TR. Ressuspender a outra amostra de pHL em 18 mL de PBS mais 0,1% de DMSO e TR.
NOTA: O DMSO é usado para imitar a composição do tampão em uma triagem de drogas como compostos são tipicamente dissolvidos em DMSO. - Transfira essas duas soluções celulares para um reservatório de 12 poços, 9 mL por poço.
- Use um robô pipetador para pipetar as soluções celulares em metades separadas de uma placa de 384 poços, 80 μL por poço.
- Incubar a placa à temperatura ambiente durante 1,5 h, agitando suavemente e enrolada em papel alumínio para proteger os fluoróforos da luz.
- Execute a placa em um citômetro de fluxo capaz de executar placas de 384 poços.
NOTA: O fluxo de trabalho a seguir é adaptado para um Attune NxT com Cytkick Max Auto Sampler. Se outro citômetro de fluxo for usado, continue a seguir as etapas principais.- Para a placa, execute na taxa de fluxo mais rápida (1.000 uL/min) e ative o modo boost. Adquira 20 μL/poço. Inclua um ciclo de mistura e um ciclo de enxágue entre cada poço.
- Crie gráficos conforme descrito na etapa 4.1.3.
- Execute um controle de tubo WT sem manchas e otimize as tensões conforme descrito nas etapas 4.1.3 e 4.1.4. Execute uma amostra de tubo pHL sem fome e otimize as tensões VL2 e BL1 conforme descrito na etapa 4.1.
- Comece a adquirir a placa no citômetro de fluxo. Execute a placa horizontalmente para que não haja diferença significativa no tempo de aquisição entre os poços de amostra famintos e não famintos. Certifique-se de que a aquisição da placa seja concluída em ~1,5 h.
- Exporte os arquivos .fcs e analise os dados conforme descrito nas etapas 5.1 a 5.8. Encontre a média (AVG) e o desvio padrão (DP) das razões de fluorescência para as amostras tratadas com glicose (Glc) ou sem glicose (Starved).
NOTA: Os dados de nossos experimentos com fator Z podem ser encontrados na Tabela Suplementar S5. - Calcule a estatística do fatorZ 20 usando a equação (3).
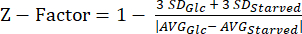 (3º)
(3º)
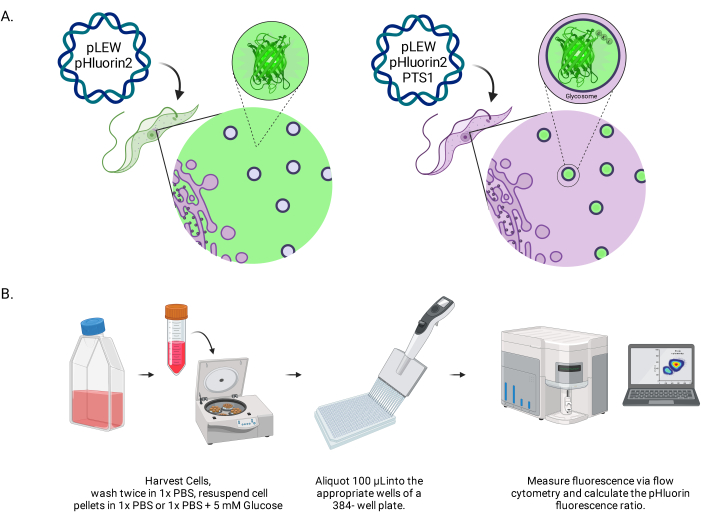
Figura 1: Diagrama do método de pontuação do pH glicossomal em tripanossomas BSF vivos. (A) Representação de linhagens celulares expressando o sensor pHluorin2 glicossomicamente localizado. A inclusão de uma sequência de direcionamento peroxissomal fornece controle sobre a localização. NOTA: Prevemos que a eliminação do PTS-1 levaria à localização citosólica, permitindo análise futura do pH nesse compartimento subcelular. (B) Representação do ensaio de validação do sensor. Abreviação: BSF = forma da corrente sanguínea. Clique aqui para ver uma versão maior desta figura.
NOTA: A estatística do fator Z é usada para determinar o quão adequado um ensaio é para HTS. Valores entre 0,5 e 1,0 geralmente significam que a qualidade do ensaio é aceitável para HTS.
Resultados
Localização de pHLuorin2-PTS1 em glicossomas em FSB de T. brucei
Para avaliar a localização subcelular da pHluorina2-PTS1, os parasitas foram submetidos a ensaios de imunofluorescência. Sinal do transgene colocalizado com antissoros levantados contra uma proteína residente em glicossoma, aldolase (TbAldolase) (Figura 2A). O coeficiente de correlação médio de Pearson de colocalização entre anti-TbAldolase e pHluorina2-PTS1 foi de 0,895, indicando que a ...
Discussão
A percepção ambiental e os mecanismos de resposta no tripanossoma africano são pouco compreendidos. Sabe-se que alterações na disponibilidade de nutrientes desencadeiam diversas respostas no parasita, incluindo acidificação de glicossomas. Aqui, descrevemos um método para estudar a resposta do pH glicossomal a perturbações ambientais em células vivas usando um sensor de proteína hereditária, pHluorin2, e citometria de fluxo.
Existem várias etapas críticas no uso do sensor. Prime...
Divulgações
Os autores declaram não haver conflitos de interesse.
Agradecimentos
pHluorin2-PTS1 foi clonado em pLEW100v5 pela Twist Bioscience que forneceu a construção em um vetor de clonagem de alta cópia; pLEW100v5 foi um presente do Dr. George Cross. Antissoro levantado contra T. brucei aldolase está disponível na Dr. Meredith T. Morris, Clemson University, mediante solicitação. O trabalho dos laboratórios JCM e KAC foi parcialmente apoiado por um prêmio do National Institutes of Health (R01AI156382). O SSP foi suportado pelo NIH 3R01AI156382.
Materiais
| Name | Company | Catalog Number | Comments |
| 50 mL Tissue Culture Flasks (Non-treated, sterile) | VWR | 10861-572 | |
| 75 cm2 Tissue Culture Flask (Non-Treated, sterile) | Corning | 431464U | |
| 80 µL flat-bottom 384-well plate | BrandTech | 781620 | |
| Amaxa Human T Cell Nucleofector Kit | Lonza | VPA-1002 | |
| Attune NxT Flow Cytometer | invitrogen by Thermo Fisher Scientific | A24858 | FlowJo software |
| BRANDplates 96-Well, flat bottom plate | Millipore Sigma | BR781662 | |
| Coloc 2 plugin of ImageJ | https://imagej.net/plugins/coloc-2 | ||
| CytKick Max Auto Sampler | invitrogen by Thermo Fisher Scientific | A42973 | |
| CytoFLEX Flow Cytometer | Beckman-Coulter | ||
| Electron Microscopy Sciences 16% Paraformaldehyde Aqueous Solution, EM Grade, 10 mL Ampoule | Fisher Scientific | 50-980-487 | |
| GraphPad Prism | statistical software | ||
| Nigericin (sodium salt) | Cayman Chemical | 11437 | |
| Nucleofector 2b | Lonza | Discontinued Product | |
| OP2 Liquid Handler | opentrons | OP2 | |
| poly-L-lysine, 0.1% (w/v) in H2O | Sigma Life Science | CAS:25988-63-0 | Pipetting robot for HTS assay |
| Thiazole Red (TO-PRO-3) | biotium | #40087 | We machined a custom acrylic plate stand so this brand of plate could be detected and used on our CytKick Max Auto Sampler |
| valinomycin | Cayman Chemical | 10009152 | Pipetting robot for HTS assay |
| For pH calibration | |||
| For pH calibration |
Referências
- Coley, A. F., Dodson, H. C., Morris, M. T., Morris, J. C. Glycolysis in the African trypanosome: Targeting enzymes and their subcellular compartments for therapeutic development. Molecular Biology International. 2011, 123702 (2011).
- Mcconville, M. J., Saunders, E. C., Kloehn, J., Dagley, M. J. Leishmania carbon metabolism in the macrophage phagolysosome- feast or famine. F1000Res. 4, 938 (2015).
- Parsons, M. Glycosomes: Parasites and the divergence of peroxisomal purpose. Molecular Microbiology. 53 (3), 717-724 (2004).
- Parsons, M., Furuya, T., Pal, S., Kessler, P. Biogenesis and function of peroxisomes and glycosomes. Molecular and Biochemical Parasitology. 115 (1), 19-28 (2001).
- Haanstra, J. R., Gonzalez-Marcano, E. B., Gualdron-Lopez, M., Michels, P. A. Biogenesis, maintenance and dynamics of glycosomes in trypanosomatid parasites. Biochimica et Biophysica Acta. 1863 (5), 1038-1048 (2016).
- Allmann, S., Bringaud, F. Glycosomes: A comprehensive view of their metabolic roles in t. Brucei. International Journal of Biochemistry and Cell Biology. 85, 85-90 (2017).
- Dodson, H. C., Morris, M. T., Morris, J. C. Glycerol 3-phosphate alters Trypanosoma brucei hexokinase activity in response to environmental change. The Journal of Biological Chemistry. 286 (38), 33150-33157 (2011).
- Lin, S., Morris, M. T., Ackroyd, P. C., Morris, J. C., Christensen, K. A. Peptide targeted delivery of pH sensor for quantitative measurements of intraglycosomal pH in live Trypanosoma brucei. Biochemistry. 52 (21), 3629-3637 (2013).
- Mahon, M. J. Phluorin2: An enhanced, ratiometric, pH-sensitive green florescent protein. Advances in Bioscience and Biotechnology. 2 (3), 132-137 (2011).
- Wirtz, E., Leal, S., Ochatt, C., Cross, G. A. A tightly regulated inducible expression system for conditional gene knock-outs and dominant-negative genetics in Trypanosoma brucei. Molecular and Biochemical Parasitology. 99 (1), 89-101 (1999).
- . Restriction digest v.2 Available from: https://www.protocols.io/view/restriction-digest-nkqdg33pg25z/v2 (2018)
- . Ligation protocol with t4 DNA ligaase (m0202) v.3 Available from: https://www.protocols.io/view/ligation-protocol-with-t4-dna-ligase-m0202-95qpvorzv4o1/v3 (2021)
- Burkard, G., Fragoso, C. M., Roditi, I. Highly efficient stable transformation of bloodstream forms of Trypanosoma brucei. Molecular and Biochemical Parasitology. 153 (2), 220-223 (2007).
- Crowe, L. P., Wilkinson, C. L., Nicholson, K. R., Morris, M. T. Trypanosoma brucei pex13.2 is an accessory peroxin that functions in the import of peroxisome targeting sequence type 2 proteins and localizes to subdomains of the glycosome. mSphere. 5 (1), e00744 (2020).
- Kucejova, B., Kucej, M., Petrezselyova, S., Abelovska, L., Tomaska, L. A screen for nigericin-resistant yeast mutants revealed genes controlling mitochondrial volume and mitochondrial cation homeostasis. Genetics. 171 (2), 517-526 (2005).
- Huynh, M. H., Carruthers, V. B. Toxoplasma gondii excretion of glycolytic products is associated with acidification of the parasitophorous vacuole during parasite egress. PLoS Pathogens. 18 (5), e1010139 (2022).
- Lehoux, S., Abe, J., Florian, J. A., Berk, B. C. 14-3-3 binding to Na+/H+ exchanger isoform-1 is associated with serum-dependent activation of Na+/H+ exchange. TheJournal of Biological Chemistry. 276 (19), 15794-15800 (2001).
- Jankowski, A., et al. In situ measurements of the ph of mammalian peroxisomes using the fluorescent protein phluorin. The Journal of Biological Chemistry. 276 (52), 48748-48753 (2001).
- Jankowski, A., Grinstein, S. A. A noninvasive fluorimetric procedure for measurement of membrane potential. Quantification of the NADPH oxidase-induced depolarization in activated neutrophils. The Journal of Biological Chemistry. 274 (37), 26098-26104 (1999).
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A simple statistical parameter for use in evaluation and validation of high throughput screening assays. Journal of Biomolecular Screening. 4 (2), 67-73 (1999).
- Miesenbock, G., De Angelis, D. A., Rothman, J. E. Visualizing secretion and synaptic transmission with pH-sensitive green fluorescent proteins. Nature. 394 (6689), 192-195 (1998).
- Lin, S., et al. Ph regulation in glycosomes of procyclic form Trypanosoma brucei. The Journal of Biological Chemistry. 292 (19), 7795-7805 (2017).
- Ha, D. S., Schwarz, J. K., Turco, S. J., Beverley, S. M. Use of the green fluorescent protein as a marker in transfected Leishmania. Molecular and Biochemical Parasitology. 77 (1), 57-64 (1996).
- Kelly, J. M., Ward, H. M., Miles, M. A., Kendall, G. A shuttle vector which facilitates the expression of transfected genes in Trypanosoma cruzi and Leishmania. Nucleic Acids Research. 20 (15), 3963-3969 (1992).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados