Method Article
更新されたマルチプレックスLN34リアルタイム逆転写ポリメラーゼ連鎖反応アッセイによる動物の狂犬病の死後診断
要約
このプロトコルは、一部の非狂犬病リッサウイルスおよびラゴモルフのアッセイ性能を改善するためのプライマー配列および製剤の更新を含む、組織収集から結果の解釈までの汎リッサウイルスLN34リアルタイム逆転写ポリメラーゼ連鎖反応(RT-PCR)アッセイを実証しています。また、シングルウェルLN34マルチプレックス(LN34M)フォーマットのアッセイセットアップのデモも行います。
要約
狂犬病は、リッサウイルス狂犬病(RABV)および リッサウイルス 属( ラブドウイルス科)の関連陰性鎖RNAウイルスによって引き起こされる致命的な人獣共通感染症です。LN34 アッセイは、リッサウイルスゲノムの高度に保存されたリーダー領域と核タンパク質遺伝子を標的とし、縮退プライマーとロックされたヌクレオチドを含む TaqMan プローブを利用して、多様な リッサウイルス 属の RNA を検出します。狂犬病の陰性所見は、脳幹の完全な断面と小脳の3つの葉が検査された場合にのみ行われるべきです。しかし、あらゆる組織におけるリッサウイルスRNAの同定は狂犬病感染の診断です。組織はTRIzol試薬で収集および均質化され、ウイルスも不活性化されます。RNA抽出は、スピンカラムベースの市販の抽出キットを使用して行います。マスターミックスはクリーンなスペースで調製され、サンプルRNAを添加する前に96ウェルプレートに分注されます。臨床現場では、各サンプルはリアルタイムRT-PCRによって、リサウイルスRNAの存在をトリプリケートで、宿主β-actin mRNAを単独でテストします。ポジティブコントロールとネガティブコントロールは、プロトコルの抽出ステップとリアルタイムRT-PCRステップに含まれます。データ解析では、閾値を手動で調整して、装置の実行全体でCt値を標準化します。陽性の結果は、汎リッサウイルスアッセイにおける典型的な増幅の存在によって決定されます(Ct ≤ 35)。陰性の結果は、汎リッサウイルスアッセイにおける典型的な増幅の欠如と、宿主のβ-アクチンmRNA(Ct ≤ 33)の検出によって決定されます。これらの範囲外の値を観察したり、アッセイコントロールが失敗したりすると、分析が無効になったり、検体の結果が不確定になったりする可能性があります。高いアッセイ感度と特異性を確保するために、プロトコールに厳密に従う必要があります。手続き上の変更は、アッセイの性能に影響を与え、偽陽性、偽陰性、または解釈不能な結果につながる可能性があります。
概要
このプロトコルは、サンプル収集から結果の解釈まで、LN34 汎リッサウイルスリアルタイム逆転写ポリメラーゼ連鎖反応 (RT-PCR) アッセイを使用した狂犬病診断検査の手順を説明しています。手順は、LN34 アッセイに関連する脳サンプルの収集 (セクション 1)、Direct-zol RNA Miniprep キット (Zymo Research R2051) を使用した手動カラムベースの RNA 抽出 (セクション 2)、AgPath-ID One-Step RT-PCR キット (ThermoFisher Scientific AM1005) を使用した LN34 リアルタイム RT-PCR アッセイ (セクション 3) の 3 つのセクションに分かれています。RNA抽出とRT-PCRは他の製品を使用して実施できますが、リッサウイルスRNAが適切に抽出および増幅されていることを確認するために、キットを使用する前にバリデーションする必要があります。
セクション1では、LN34リアルタイムRT-PCRアッセイで使用する適切な脳組織の収集について説明します。動物の剖検、斬首、脳の摘出に関する記述は含まれていません。サンプルには感染性病原体が含まれている場合があります。リスクを軽減するために、Biosafety in Microbiological and Biomedical Laboratories 6th Edition1 に詳述されているバイオセーフティ手順に従う必要があります。サンプルは、不活化が完了するまで感染性と見なす必要があります。.ウイルスの不活化とアッセイの検証は、その機関の基準に従って各ラボで実施する必要があります。検査室は、新しい診断テストを実施する際に、所属機関が定めた標準的な安全性と品質の手順に従う必要があります。
感染時の狂犬病ウイルスの蔓延について知られていることに基づいて、脳幹と小脳は狂犬病診断に最適な組織であり、これらの組織は世界保健機関および国際獣疫事務局による狂犬病検査に推奨されています2,3,4,5。ウイルスの拡散は、特に大型動物では片側性である可能性があるため(図1)、狂犬病の除外については、脳幹の完全な断面と小脳の3つの葉を検査する必要があります。これらの最低基準を満たさないサンプルの場合、検査室はサンプルを検査に不十分であるとして拒否するか、監視またはルールイン目的で検査することを選択する場合があります。必要な組織を受け取らなかったが、検査室がサンプルの検査を選択した場合、他の組織でのウイルスRNAの存在が遅れたり、存在量が少なく、断続的であったり、存在しない可能性があるため、陰性の検査結果はその動物の狂犬病にとって決定的ではないと解釈されるべきです。その場合、狂犬病を除外するには、必要なサンプルの収集または追加の検査が必要です。しかし、任意の組織におけるリッサウイルスRNAの同定は、狂犬病感染の診断である3,6。ルールインまたはサーベイランス(ただし除外しない)のために狂犬病ウイルスRNAを検査できるサンプルの例としては、皮質、海馬、脊髄、分解サンプル、皮膚、唾液、角膜などがあります。各サンプルの状態の定性的評価は、ラボに到着したときに行う必要があります。冷蔵はサンプルを少なくとも72時間保存できますが、長期間使用しないでください。凍結融解サイクルを繰り返すと、テスト感度が低下する可能性があるため、5回を超える凍結融解サイクルは避ける必要があります。組織の状態が脳構造の確実な同定を妨げる場合、サンプルは不十分であると特定する必要があります。検体が不十分な場合でも、狂犬病を除外する(ただし除外しない)ための検査を行うことができます。陽性の検査結果はそのように報告されます。不十分組織に対する陰性または決定的でない結果は、陰性診断と誤解されるのを防ぐために、決定的でないと報告する必要があります。
このプロトコルは、公開された手順7、8、9 から開発され、リッサウイルスゲノムリーダー領域と核タンパク質コード配列を標的とする更新されたプライマーが含まれています。このプローブは、短く、高度に保存された配列を標的とし、ロックされたヌクレオチドを使用して広範な検出を可能にします。このアッセイは、さまざまな濃度の多様なリッサウイルスからRNAを検出します8。このプロトコルは、LN34リアルタイムPCRアッセイを実施するための実験室手順を示していますが、リッサウイルスRNAの正確で高感度な検出は、このプロトコルで広くカバーされていない他の要素、例えば、検体保管、記録保持、人員のトレーニング/能力、結果追跡、結果の解釈、品質保証、実験室の安全対策、トラブルシューティングなどに依存します。PCRベースのアッセイは、その高感度のためにクロスコンタミネーションが発生しやすいです。クロスコンタミネーションは、手袋を頻繁に交換する、一度に1つのサンプルを取り扱う、サンプル間で効果的な除染剤で作業面を消毒する、チューブを閉じてサンプルをPCR試薬から分離するなど、ラボの優れた慣行に従うことで回避できます。PCR試薬とサンプルは、片側のワークフローを採用し、増幅前と増幅後の作業領域を分離することで、簡単に分離できます。例えば、PCRマスターミックスは、サンプルが処理される場所とは物理的に離れた場所で調製します。PCR試薬がサンプル、破片、またはポジティブコントロールRNAで汚染されないように、手袋を頻繁に交換してください。PCRプレートまたはチューブは、マスターミックス添加後、サンプルおよびコントロールRNAを追加できる2番目の場所に移動する必要があります。重要なことは、PCR産物は、サンプルやマスターミックスが調製される領域で操作してはならないということです。
診断テストを実施する際の実践的な実践と経験に代わるものはありません。すべての新入社員は訓練を受けるべきであり、試験要員は、関連する研究所長の要件に従って、少なくとも年に1回は能力評価を受けるべきです。異常な結果やアッセイの失敗が観察された場合は、直ちに注意し、調査し、修正する必要があります。新しい試薬の各ロットは、既知のCt値を持つサンプル(ポジティブコントロールやアーカイブサンプルなど)を使用してバリデーションする必要があります。すべての機器は、メーカーが提案するように定期的なメンテナンスを受ける必要があり、メンテナンスや修理後にアッセイ性能を検証する必要があります。診断テストで使用される試薬の許容温度範囲に設定された基準内に冷蔵庫と冷凍庫が収まるように、該当する機器の温度レベルを監視する必要があります。
手続き上の変更はアッセイの性能に影響を与える可能性があり、偽陽性、偽陰性、または解釈不能な結果につながる可能性があります。高いアッセイ感度と特異性を確保するために、推奨事項に厳密に従う必要があります。このプロトコルの変更を取り入れることを希望する研究室は、CDCと協議して、変更された方法を検証し、確認する必要があります。
プロトコル
死後の脳組織サンプルは、ポックスウイルスおよび狂犬病支部(CDC;アトランタ、ジョージア州、米国)。
1. LN34汎リッサウイルスリアルタイムRT-PCRアッセイによる動物の狂犬病死後診断のための脳組織の採取
注:サンプルには感染性病原体が含まれている場合があります。適切な個人用保護具(PPE)(厚手のゴム手袋またはその他の耐切創手袋、実験用ガウン、防水エプロン、サージカルマスク、ブーツ、保護スリーブ、フェイスシールド)を着用し、サンプルの使用、保管、廃棄に必要な安全規制に従ってください。曝露前の狂犬病ワクチン接種、定期的な血清学的検査、およびブースター予防接種(必要に応じて)は、リッサウイルスまたは既知または感染する可能性のある検体2、3、4、6、10を取り扱う、試験、生産、または研究活動を行う前に必要です。
- サンプルごとに1つのサンプル収集チューブにアクセッションラベルを貼付します。各サンプル採取チューブに、1 mLのTRIzol試薬またはその他の均質化バッファーとMagNA Lyserビーズ(以下「セラミックビーズ」)の一部を満たします。セラミックビーズを追加するには、ビーズのチューブからサンプル収集チューブに慎重に注ぎます。セラミックビーズのチューブには、通常、サンプルごとに直径1.4mmのビーズが少なくとも20個使用されており、2〜5個のサンプルに十分なビーズが含まれています。
注意:トリゾール試薬(以下「ホモジナイズバッファー」)は危険な化学物質です。酸や漂白剤と接触すると、有毒ガスが放出されます。適切な換気を確保します。詳細については、安全データシートを参照してください。ユーザーがTRIzol試薬またはTRI試薬を別のホモジナイズバッファーに置き換える場合は、追加のバリデーションが必要です。TRIzolは、このプロトコルのサンプルホモジナイゼーション/溶解バッファー、リッサウイルス不活化バッファー、およびRNA安定性バッファーとして機能します。代替の均質化バッファーを使用するには、抽出効率、不活性化、および安定性を制御された並べて比較することで検証する必要があります。 - 作業面を第四級アンモニウム化合物(QAC)消毒剤で2分間洗浄および消毒し、プラスチックで裏打ちされた吸収パッドをレイアウトします。最初のサンプルの試薬と消耗品のみを、部屋の外に危険なガスを排出する機能を備えたクラスII生物学的安全キャビネット(BSC)に入れます。
注:希釈QACの保管制限については、製造元のガイダンスを参照してください。プラスチックで裏打ちされたパッドが生物学的安全キャビネットの空気の流れを妨げないことを確認してください。空気の流れが乱れる場合は、パッドを使用しないでください。 - 脳幹と小脳の全断面を表す組織を、清潔な使い捨てメスで採取します。
注:組織の操作は、液体をエアロゾル化したり、空気中の粒子を生成したりしない方法で行う必要があります。ヒュームフードやバイオセーフティキャビネットは必要ありませんが、臭気、煙、外部寄生虫、骨片からの保護を強化するため、通気式バイオセーフティキャビネットをお勧めします。
注意: リッサウイルスに感染する可能性のある物質を含むメスの使用は危険であり、ユーザーは適切な安全対策を講じる必要があります。使い捨て鉗子の使用をお勧めします。- 小動物(コウモリなど)の場合、脳幹と小脳全体を採取することがあります。
- より大きな動物の場合は、小脳の3つの葉のそれぞれから脳幹と組織の全断面を採取します。
- オプション:直接蛍光抗体(DFA)テストを実行する場合は、この時点で脳の印象を収集します。脳の印象を収集した後に残った組織をDFAに使用し、RNA抽出とLN34アッセイによる試験を行います。
注:TRIzolをサンプルに添加すると、サンプルは抗原ベースの検出法やウイルスの分離に使用できなくなります。
- 均質化とRNA抽出のためのサンプルを調製します。
注:RNA抽出とウイルスの不活化の効率は、組織を使いすぎると影響を受ける可能性があります。組織の量は、使用する均質化緩衝液の体積の約1/10を超えてはなりません。より多くの組織を使用する場合は、それに応じて均質化バッファーの量を増やし、効率的で成功したRNA抽出を確保します。- 小動物の場合は、必要なすべての組織をホモジナイズバッファーとビーズが入ったチューブに入れて抽出します。1 mLのホモジナイズバッファー中のサンプル100 mgを超えないようにしてください。より大きなサンプルの場合は、ホモジナイズバッファーの容量を増やすか、複数のチューブを使用して1:10の比率のサンプル:バッファーを反映させます。
- より大きな動物の場合は、組織を完全にミンチして均質化し、ホモジナイゼーションバッファーとビーズをあらかじめ充填したチューブに代表的な部分を取り出します。1 mLのホモジナイズバッファー中のサンプル100 mgを超えないようにしてください。より大きなサンプルの場合は、ホモジナイズバッファーの容量を増やすか、複数のチューブを使用して1:10の比率のサンプル:バッファーを反映させます。
- オプション1(ビーズビーター):ビーズビーター、1 mLのバッファー、およびセラミックビーズを使用して組織をホモジナイズします。2 mL以上のチューブを数本使用する必要がある場合があります。
- オプション1(ビーズビーター):ワークステーション、機器、および外部サンプルチューブをQAC消毒剤で洗浄および消毒します(1:256)。2分間放置します。
- オプション1(ビーズビーター):BSC内に、均質化されたサンプルを遠心分離機ローターにロードします。すべてのサンプルを10,000〜16,000 × g で卓上型マイクロ遠心分離機で2分間遠心分離します。BSC内の遠心分離機ローターを降ろします。
- オプション1(ビーズビーター):2分間放置します。
- オプション1(ビーズビーター):120 μLのホモジネートを、1 mLのホモジナイゼーションバッファーをあらかじめ充填したチューブに移します。
注意:均質化はエアロゾルを生成する可能性があるため、BSCで実行する必要があります。 - オプション2(メス):使い捨てメスを使用して必要な組織を細かく刻み、綿棒で塗抹標本を塗り、均質化バッファーとビーズをあらかじめ充填したチューブに綿棒を移します。1 mLのホモジナイズバッファー中のサンプル100 mgを超えないようにしてください。より大きなサンプルの場合は、ホモジナイズバッファーの容量を増やすか、複数のチューブを使用して1:10の比率のサンプル:バッファーを反映させます。
注意: リッサウイルスに感染する可能性のある物質を含むメスの使用は危険であり、ユーザーは適切な安全対策を講じる必要があります。
- 残っている組織を元の容器に集めるか、アクセッションラベルでラベル付けされた新しい空の容器に集めます。この組織は、再試験や追加の特性評価が必要な場合に備えて保管してください。
- ワークステーション、機器、およびサンプルチューブの外側をQAC消毒剤1:256で洗浄および消毒します。2分間放置します。
- 残りのすべてのサンプルについて、手順1.2〜1.5を繰り返します。
- ミニビーズビーターでサンプルを少なくとも60秒間均質化します。チューブを目視検査します。大きなティッシュ片が残っている場合は、ビーズビーターをさらに60秒間繰り返します。この手順は、上記の手順 1.4.2.1 で組織が完全に均質化されている場合、オプションです。
注:組織が完全に均質化されていることを確認することが重要です。不完全な均質化はRNAの収量を減少させます。 - 室温(RT)で少なくとも5分間放置します。
- ワークステーション、機器、およびサンプルチューブの外側をQAC消毒剤(1:256)で洗浄および消毒します。
注:現時点では、サンプルは非感染性と見なされており、狂犬病研究所から取り外すことができます。 - RNA抽出のためにホモジナイゼーションバッファーでサンプルをすぐに処理し、RT(20°C〜25°C)または冷蔵(4°C〜8°C)で数日間保存するか、-16°C以下で長期保存します。
2. RNA MiniPrepキットを用いたRNA抽出のプロトコール
- BSC の下にワークスペースを設定します。
- 作業を開始する前に、70%エタノールを使用してBSC作業面を清掃し、ほこりやその他の環境汚染物質を除去してください。QAC消毒剤(1:256)、RNase AWAY、またはRNaseZap(メーカーの推奨による)を使用して、追加の表面除染を行います。
- プラスチックで裏打ちされた吸収性ワークパッドをレイアウトし、試薬、消耗品、およびサンプルをBSCに置きます。
注意: プラスチックで裏打ちされたパッドがBSCの空気の流れを妨げないようにしてください。空気の流れが乱れる場合は、パッドを使用しないでください。 - すべての収集チューブをマイクロ遠心チューブ用のきれいなラックにレイアウトします。1.5 mLの微量遠心チューブ1本に、コウモリ以外の脳サンプルごとに300 μLの100%エタノールをあらかじめ充填します。組織が少ないサンプル(コウモリの脳サンプル、非脳サンプル、または劣化したサンプル)の場合は、1.5 mLの微量遠心チューブ1本にそれぞれ600 μLの100%エタノールを事前に充填します。
- サンプル調製
- セクション1で調製したすべてのサンプルをBSCのチューブラックに収集します。凍結したサンプルは、テストの直前に解凍します。
- 抽出コントロールを解凍します。
注:リッサウイルスRNAを含まないサンプルを選択することをお勧めします。サンプルは、ベータアクチンアッセイで予想されるCt値範囲で事前にテストする必要があります。例えば、事前に分注した組織培養細胞や、以前に検査された陰性の狂犬病症例(ヒトまたは動物)などです。
- 卓上型微量遠心分離機で、すべてのサンプルを10,000〜16,000 × g で2 m遠心分離します。
- 上清を100%エタノールを含む新しい滅菌微量遠心チューブに移します。上清が透明で、明らかな脂質や固形組織がないことを確認してください。脂質や固形組織の収集は避けてください。
- 非コウモリ脳組織の場合:上清300μLを移します。
- 組織が少ないサンプル(コウモリの脳サンプル、非脳サンプル、または劣化したサンプル)の場合は、上清600μLを移します。
- 残りのホモジネートをスクリュートップマイクロ遠心チューブに≤〜16°Cで保存します。
- ピペットで10回上下させて混ぜます。
- 各サンプルについて、600 μLのエタノール-上清混合物をコレクションチューブ内のスピンカラムに移します。
- 液体がカラムを通過するまで遠心分離します(10,000〜16,000 × gで1分間)。フロースルーを破棄します。
- サンプルに600 μLを超えるエタノール-ホモジナイズバッファー混合物がある場合は、この手順を繰り返します。
- 各カラムを新しいコレクションチューブに移します。
- 各カラムに400 μLのRNAプレウォッシュバッファーを添加し、10,000〜16,000 × g で30秒間遠心分離します。
- フロースルーを廃棄し、各カラムを同じ収集チューブに戻します。
- 手順2.10〜2.11を繰り返します。
- 各カラムに700 μLのRNA洗浄バッファーを加え、10,000-16,000 × g で2分間遠心分離します。洗浄バッファーが各カラムを完全に通過していることを確認します。
- 各カラムをRNaseフリーチューブに慎重に移します。
- 2.13からフロースルーと収集チューブを廃棄します。
- 50 μL の DNase/RNase-Free 水をカラムマトリックスに直接加えて RNA を溶出します。
注意:ピペットチップでカラムマトリックスに触れないでください。 - RTで30秒間インキュベートし、その後、10,000-16,000 g×1分間遠心 分離します。
- RNAを新しいスクリュートップフラットボトムアクセッションに慎重に移し、ラベル付けされた微量遠心チューブに移します。抽出したRNAを氷に移し、すぐにテストします。-70°C以下で長期保存してください。
注:高温での保存や凍結融解の繰り返しは、RNAの分解につながり、診断結果に影響を与える可能性があります。
3. LN34汎リッサウイルスリアルタイムRT-PCRアッセイのプロトコール
- 試薬を準備します。
- 人工ポジティブコントロール
- CDC8によって人工ポジティブコントロールRNAが産生された場合は、保存、再構成、および分注のためにパッケージの指示に従ってください。ポジティブコントロールRNAのシングルユースアリコートがすでに手元にある場合は、このステップをスキップしてください。
注:使用濃度でのポジティブコントロールRNAは、マスターミックス調製と同じ領域ではなく、テンプレート添加領域で取り扱う必要があります。ポジティブコントロールRNAは、特定のロットで決定された予想範囲内でサイクル閾値(Ct)を生成する必要があります。分析間で、ポジティブコントロールRNAのLN34 Ct値は、±1.5 Ct値を超えて差があってはなりません。 - -70°Cで保存した≤で使い捨てのアリコートを解凍し、氷または氷の塊で使用してください。凍結融解したり、冷蔵温度で長時間保管したアリコートは廃棄しないでください。
注:ポジティブコントロールは、LN34アッセイでトリプリケートに実行する必要があります。人工ポジティブコントロール8 は、β-アクチンアッセイでは増幅しない。
- CDC8によって人工ポジティブコントロールRNAが産生された場合は、保存、再構成、および分注のためにパッケージの指示に従ってください。ポジティブコントロールRNAのシングルユースアリコートがすでに手元にある場合は、このステップをスキップしてください。
- 抽出制御とサンプル:抽出したばかりのサンプルを氷(または氷ブロック)に置くか、-70°C≤保存したサンプルを氷(または氷ブロック)で解凍します。
注:RNAは、PCR産物または大量のウイルス物質(ポジティブコントロールの生成、ウイルス増殖など)のマスターミックス調製または操作に使用される領域とは別の、サンプルまたはテンプレートの添加用に指定された領域で解凍および処理する必要があります
- 人工ポジティブコントロール
- マスターミックス調製エリアでマスターミックス試薬を調製します。
- シングルプレックスLN34 RT-PCRアッセイのマスターミックス調製
注:ユーザーは、シングルプレックス(ステップ3.2.1)またはマルチプレックス(ステップ3.2.2)形式でサンプルをテストできます。3.2.1 と 3.3.2 の両方を実行する必要はありません。マスターミックスの調製、プライマーとプローブの分注、およびテンプレートコントロール試薬は、サンプル処理、剖検、PCR、およびウイルス材料が操作される他の領域とは別のクリーンな領域で解凍および操作する必要があります。これは、別々の部屋または片側のサンプルフローを備えたキャビネットシステムによって達成できます。- プライマーとプローブの混合物を 、表1 および 表2に示す使用濃度で生成します。プライマーやプローブの希釈アリコートがすでに手元にある場合は、このステップをスキップしてください。
- プライマーとプローブを1.5 mLの保存ストックと50 μLのワーキングストックに分注し、暗所で≤-16°Cで保存します。プライマーやプローブの希釈アリコートがすでに手元にある場合は、このステップをスキップしてください。
メモ: 手順 3.2.1.2 を実行することをお勧めします。 - -16°Cで≤保存したワンステップRT-PCRバッファー、テンプレートコントロールなし、プライマー、およびプローブを氷上またはマスターミックス調製エリアの氷上または氷ブロックで解凍します。
注意: 試薬は、有効期限またはパフォーマンス障害のいずれか早い方まで使用してください。 - 使用前に、すべてのバッファー、プライマー、プローブを短時間ボルテックスし、遠心分離します。
- ワンステップRT-PCR酵素は、使用するまで氷上または氷ブロックに保管してください。
- RNAサンプル
注:凍結融解は性能に影響を与える可能性があるため、可能な限り抽出したばかりのRNAを使用してください - RNAサンプルは、使用するまで氷上または氷塊上に保存します。
- 凍結したRNAサンプルを氷上または氷の塊で解凍します。
- アッセイごとに1本の微量遠心チューブ(LN34およびβA)を標識します。
- アッセイごとに設定する反応数(N)を決定します。
- LN34アッセイの反応数を計算するには、サンプル数に3を掛け、コントロール反応ウェルの場合は6を加算し、さらにピペッティング中に失われる容量を考慮して反応を10%追加します。(例:10サンプルの場合:(10 x 3)+ 6 = 36反応、過剰反応:(36 x 0.1)+ 36 = 3.6 + 36 = 39.6合計反応、または40反応を切り上げ)
注:臨床試験では、LN34のすべてのサンプルをトリプリケートでテストすることをお勧めします。監視の目的で、各サンプルを重複してテストする場合があります。最初のアッセイオンボーディングでは、反復間のばらつきを低く抑え、良好な技術を確保するために、トリプリケートを使用することをお勧めします。 - サンプル数と4つのコントロール反応ウェルを加算し、ピペッティング中に失われる体積を考慮して10%の追加反応を行うことにより、βAアッセイの反応数を計算します。
- 表2を使用して、LN34およびβAマスターミックスの各試薬の容量を決定します。
- LN34 アッセイでは各サンプルのウェルを 3 倍に、βA アッセイでは 96 ウェルプレートマップを使用してシングルレートを指定します。
- 23 μLのLN34アッセイマスターミックスを、卓上型微量遠心分離機を使用して30秒間短時間ボルテックスしてスピンダウンした後、チューブの底に液体を回収した後、割り当てられた各LN34に分注します。気泡の導入は避けてください。
- 23 μLのβAアッセイマスターミックスを、卓上型微量遠心分離機で短時間ボルテックスして30秒間スピンダウンした後、割り当てられた各βA標識に試薬を分注し、底部に液体を収集します。
- 各NTCウェルに2 μLのPCRグレードの水をピペットで移すことにより、テンプレートなしコントロール(NTC)反応を設定します。
- ウェルを覆い、プレートをテンプレート追加エリアに移します。
- RNAサンプルを含むチューブを短時間ボルテックスし、遠心分離します。
- 最初のサンプルから抽出した 2 μL の RNA を、そのサンプル用に標識された各ウェルにピペットで移します。気泡の導入は避けてください。
- 可視化により、RNAがピペットに引き込まれたことを確認します。
- サンプルが正しいウェルに追加されることを確認するために、ウェルの側面にピペットを取り付けます。
- RNAを含むピペットチップをオープンウェル上で振ることはできるだけ避けてください。
- 残りのサンプルとポジティブコントロールRNAについて、ステップ3.2.10を繰り返します。
- すべてのサンプルとコントロールを追加した後、光学接着剤カバーをウェルの上に置きます。すべてのウェルを覆い、完全に密封するように注意してください。
- 卓上遠心分離機で500 × g でRTで1分間遠心分離するか、サラダスピナータイプのプレートスピナーを使用します。
- 密封されたプレートを、FAMおよびVIC / HEXレポーター色素用に較正されたリアルタイムPCR装置に入れ、 表3に示すサイクリングパラメータに設定します。
- LN34マルチプレックス(LN34M)アッセイ用のマスターミックスを調製します。
- 表2に従って、1本の微量遠心チューブLN34Mにラベルを付けます。
- アッセイごとに設定する反応数(N)を決定します。
- LN34Mアッセイの反応数を計算するには、サンプル数に3を掛け、コントロール反応ウェルの場合は6を加算し、さらにピペッティング中に失われる量を考慮して10%の反応を追加します。(例:10サンプルの場合:(10 x 3)+ 6 = 36反応;過剰反応:(36 x 0.1)+ 36 = 3.6 + 36 = 39.6合計反応、または40反応を切り上げ)
- 25 μLまたは12.5 μLのフォーマットをお選びください。 表2を使用して、LN34Mマスターミックスの各試薬の容量を決定します。
- LN34Mアッセイで3回試験する各サンプルのウェルを、96ウェルプレートマップを使用して指定します。
- LN34Mアッセイ用の試薬をウェルに分注します。チューブを短時間ボルテックスしてスピンダウンし、底部に液体を集めてから、23 μL(25 μL反応の場合)または10.5 μL(12.5 μL反応の場合)のマスターミックスを割り当てられた各ウェルに分注します。気泡の導入は避けてください。
- 各NTCウェルに2 μLのPCRグレードの水をピペットで移し、NTC反応をセットアップします。
- ウェルを覆い、プレートをテンプレート追加エリアに移します。
- RNAサンプルを含むチューブを短時間ボルテックスしてスピンダウンし、下部に液体を収集します。
- 最初のサンプルから抽出した 2 μL の RNA を、そのサンプル用に標識された各ウェルにピペットで移します。気泡の導入は避けてください。
- RNAがピペットの可視化に引き込まれていることを確認します。
- サンプルが正しいウェルに追加されることを確認するために、ウェルの側面にピペットを取り付けます。
- RNAを含むピペットチップをオープンウェル上で振ることはできるだけ避けてください。
- 残りのサンプルとポジティブコントロールRNAについて、ステップ3.3.8を繰り返します。
- 最後のサンプル/コントロールを追加した後、光学接着剤カバーをウェルに置き、すべてのウェルが覆われて完全に密封されていることを確認します。
- 卓上型遠心分離機でRTで500 × g で1分間遠心分離するか、サラダスピナータイプのプレートスピナーを使用します。
- 密封されたプレートを、FAMおよびVIC / HEXレポーター色素用に較正されたリアルタイムPCR装置に入れ、 表3に示すようにサイクリングパラメータに設定します。パッシブリファレンス色素をROXに設定し、 標準モードで 実行します(高速モードでは実行しないでください)
注:この設定は、このプロトコルに記載されている機器に固有であり、不動態色素としてROXを含むワンステップRT-PCR試薬を使用する必要があります。代替の装置では、最適な分析設定を決定するために異なるアプローチが必要です。最高のパフォーマンスを得るには、メーカーごとに通常の機器のメンテナンスを確保してください。
- シングルプレックスLN34 RT-PCRアッセイのマスターミックス調製
4. 結果の解釈
- LN34/FAM では 0.2、βA/HEX/VIC では 0.05 の値を使用して、自動ベースラインと手動しきい値計算を設定します。
注:この設定は、このプロトコルに記載されている機器に固有であり、不動態色素としてROXを含むワンステップRT-PCR試薬を使用する必要があります。別の手段では、ベースライン値としきい値を計算するために異なるアプローチが必要です。 - 表 4 のガイダンスを使用して、すべてのコントロールが期待どおりに実行された場合は、診断結果を確認します (表 5)。
- 増幅プロットを表示して、すべてのCtまたはCq値を確認します。
- 推奨されるように、異常な結果を調査します。
5. サンプルの保持と保存
- すべてのサンプルは、試験が完了して結果が報告されるまで、-16°C以下で凍結して保存してください。結果を確認するため、または異常なテスト結果の場合に宿主動物を種に識別するために、元の組織を保持します。
- 一意のサンプル識別子を使用します。すべてのチューブ、レポート、書類に、完全に固有のサンプル識別子をラベル付けします。
- 繰り返しテストが必要な場合に備えて、中間サンプル(短期)を保持してください。
- 必要に応じて、コントロール、疫学タイピング、およびその他の目的で使用するために、代表的な陽性サンプルを保持します。
- RNAは≤〜70°Cで保存し、長期保存してください。
結果
図 2に、ABI ViiA7リアルタイムPCR装置で成功したLN34アッセイの実行から得られた代表的な画像を示します。対数スケールでプロットされた結果を表示すると、曲線がしきい値線を横切る点であるCt値を簡単に表示できます(図2A、C)。線形スケールでプロットすると、成功した増幅はシグモイド(または「S」字型)曲線(図2B、D)として表示され、否定的な結果は直線で平らな線として表示されます。線形スケール ビューと対数スケール ビューの両方で結果を表示することは、考えられる異常やエラーを特定するために推奨されます。多成分プロットビューの典型的な陽性および陰性の結果をそれぞれ 図2E、Fに示すことができ、プローブを標識する色素の蛍光レベル(LN34の場合はFAC、βAの場合はVIC/HEX)を反応バッファー(ROX)中の不動態色素と比較して観察できます。
異常な結果の例を 図 3 に示します。成功したランのグラフ(図 2)と異常なグラフ(図 3)の比較を使用して、非定型ランと装置の問題を特定できます。 図3A は、信号がスレッショルドを横切り、LN34のCt値を生成することを示していますが、増幅曲線は非常に非典型的であり、直線的に増加します。多成分プロット(図3B)には、正のサンプルに特有ではない波線も示されています。この例では、単に Ct 値をコピーするのではなく、増幅プロットを表示することの重要性を強調しています。増幅曲線がすべてのサンプルで正常に見えることを常に確認してください。多成分プロットを表示して、不規則性が存在しないことを確認することもお勧めします。場合によっては、乱雑なベースライン信号が、増幅が発生していない場合にCt値を生成することがあります。増幅信号が線形に見える場合は、ベースラインを調整して、曲線が消えるかどうかを確認することをお勧めします。異常な信号が発生した場合は、実行全体を繰り返す必要があります。問題が解決しない場合は、リアルタイムPCR装置のバックグラウンドプレートをクリーニングして実行することをお勧めします。利用可能な場合、PCR産物をアガロースゲル上で実行したり、配列決定して異常な結果をトラブルシューティングしたりできます。ゲル電気泳動やシーケンシングの結果を使用して診断結果を決定することはお勧めしません。
これまでの研究では、LN34 アッセイ7 の反復、アッセイ実行、オペレーター、およびラボ間のばらつきが少ないことが示されています。同じサンプルの繰り返し間で高いばらつき(>±1.5 Ct の差)が観察された場合は、その RNA を再試験する必要があります。高いばらつきは、ピペットの問題、実験室での作業、ピペッティングの誤り、またはリアルタイムPCRマシンの問題によって引き起こされる可能性があります。複数のサンプル間またはアッセイラン間で高いばらつきが繰り返し観察される場合は、全身の問題を示している可能性があります。陽性サンプル(Ct 35)のアッセイ閾値に近づく低 RNA のサンプルは、レプリケート間で Ct 値の変動が大きくなる可能性があります。CDCとの協議とトラブルシューティングは、持続的なばらつき、一貫性のない結果、またはアッセイの失敗の原因に対処するために必要になる場合があります。
PCRベースのアッセイは感度が高いため、本質的にコンタミネーションの影響を受けやすくなります。優れたラボラトリープラクティスを厳守することが、クロスコンタミネーションを軽減する最善の方法です。潜在的な汚染を特定する方法を知ることは重要です。テンプレートコントロールがなく、アッセイの実行で陰性が疑われるサンプルウェルがすべて同様のCt値を生成する場合は、試薬の汚染を疑う必要があります。PCR試薬(バッファー、水、プライマー、酵素)と同じRNAの新しいアリコートで試験を繰り返します。すべてのサンプルと抽出コントロールで同様の CT 値が得られるが、NTC が陰性の場合は、抽出試薬の汚染を調査し、新しい試薬を使用して抽出を繰り返す必要があります。汚染のリスクを減らし、高価な試薬を大量に廃棄する可能性を避けるために、試薬の少量のアリコートを作成することをお勧めします。サンプルのクロスコンタミネーションは、特定がより困難です。サンプルの汚染が疑われる場合は、元の組織からサンプル採取を繰り返します。場合によっては、ウイルスRNAのシーケンシングにより、特に汚染されたRNAが予想されるウイルス変異体(実験室で使用されるコントロールウイルスなど)と大きく異なる場合に、汚染を確認できることがあります。同時に処理された2つのサンプルのシーケンシングにより、ウイルスの配列が同一であるかどうかを判断できますが、シーケンシングが非常に類似していると予想される場合(たとえば、同じ郡で収集された同じ変異体)は情報が得られない可能性があります。ポジティブコントロールRNAによるサンプルの汚染が疑われる場合は、アガロースゲル上でLN34アッセイアンプリコンを泳動し、リッサウイルスRNA(165 bp)とポジティブコントロールRNA(99 bp)を区別することができます。CDC8 によって提供されるポジティブコントロール RNA の生成に使用されるテンプレートの配列。
他の病原体については、 図4のシアンで示されている弱い増幅などの「ノイズ」を取り除くために、ラボラトリアンを使用して手動でしきい値を設定することができます。狂犬病はほぼ100%致命的であるため、狂犬病の診断には推奨されません。閾値を手動で変更して、弱い増幅サンプルや遅延増幅サンプルに対して否定的な結果を生成しないでください。これらのサンプルは、狂犬病を除外するために再抽出および/または再テストする必要があります。
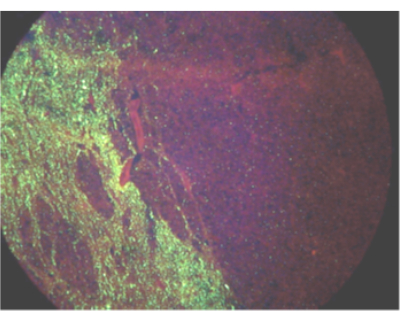
図1:直接蛍光抗体試験による感染したロバにおける狂犬病ウイルス抗原の一方的な広がりを示す視野。 この 図の拡大版を見るには、ここをクリックしてください。
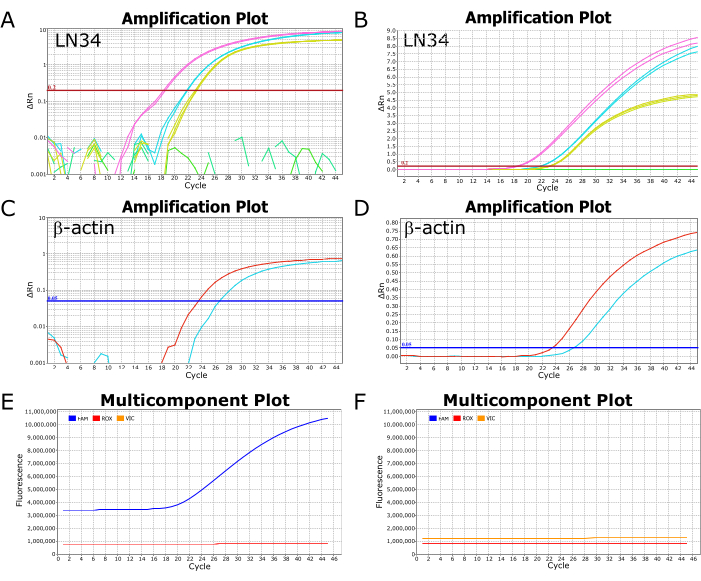
図2:成功したLN34アッセイの実行からの増幅および多成分プロット。(A-D)結果データは、LN34およびβAアッセイの(A,C)logスケールおよび(B,D)線形スケールにプロットされます。パネルAとBは、2つのサンプル(ピンクとシアン)からのLN34の結果とポジティブコントロール(黄色)の比較を示しています。パネル B には、分析中の追加のネガティブサンプルを示す平らな緑色の線があります。Aでは、緑色の線は増幅を示さず、壊れたセグメントとして描かれています。LN34アッセイの閾値は手動で0.2に設定され、赤い水平線で示されています。(C,D)2つのサンプル(赤とシアン)のβAアッセイの結果。βAアッセイの閾値は手動で0.05に設定しました。(E,F)多成分プロットは、FAM(LN34)、VIC(βA)、およびROX(AgPath-IDバッファーに存在する不動態色素)の各サイクルにおける蛍光(RFU)を示しています。ROXレベルは、すべてのサイクルで横ばいに保たれる必要があります。典型的な陽性サンプルをパネルEに示します。FAM蛍光は、このサンプルのサイクル18から始まるシグモイド曲線として増加します。典型的なネガティブサンプルをパネルFに示し、FAMレベルはすべてのサイクルでROXレベルと平行に保たれています。データはABI ViiA7リアルタイムPCR装置からのものです。この図の拡大版を表示するには、ここをクリックしてください。

図3:LN34アッセイで観察されたまれな非定型シグナルの代表的な画像を、ViiA7リアルタイムPCR装置で実行。(A-F)坑井汚染により生成された増幅(A、C、E)および多成分(B、D、F)プロット。FAM蛍光の線形増加(A、C)と波状揺らぎ(B、D)は、曲線の形状と蛍光変化の大きさに基づく真の増幅を表すものではありません。パネルAからDは、パネルAとBに示されている反復に対してCt値が生成されている場合でも、ネガティブサンプルを表している可能性があります。パネル E と F は、多成分プロットでより簡単に確認できる奇妙な波状信号を示しています。このタイプの信号は調査する必要があり、この実行ですべてのコントロールが期待どおりに実行された場合でも、機器の問題を示している可能性があります。この図の拡大版を表示するには、ここをクリックしてください。
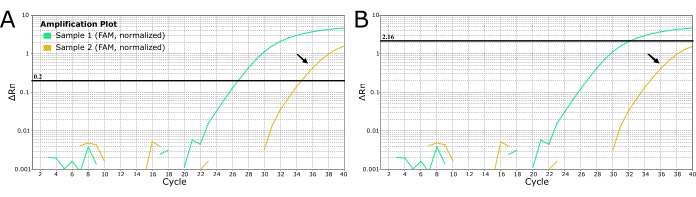
図4:狂犬病疑いサンプル2体からのLN34リアルタイムRT-PCR曲線は、閾値を設定する2つの方法を示しています。 (A)LN34の閾値は0.2に設定されました(すべてのランで推奨)。(B)「ノイズ」(遅延増幅信号)と判断された信号をマスクするために、実行ごとに異なるしきい値を手動で決定します。パネル B で使用されている方法は、真陽性の結果を見逃すことによる深刻な結果のため、狂犬病には推奨されません。増幅の遅延は、サンプルが弱い、PCR阻害、または陽性の場合の抽出の失敗を示している可能性があります。また、交差汚染を示している可能性もあります。金色のサンプル(黒色の矢印で示されている)は、アッセイのカットオフでCt値を生成するため、陰性と見なすべきではありません。遅延増幅を伴うサンプルは、再抽出して再試験する必要があります。 この図の拡大版を表示するには、ここをクリックしてください。
表1:LN34lys(シングルプレックスLN34)、LN34M(LN34およびβAマルチプレックス)リアルタイムRT-PCRアッセイで使用されるプライマーおよびプローブの配列と濃度。 LN34プローブは、5末端に蛍光FAM色素、3末端にブラックホールクエンチャー(BHQ1)で標識されています。βAプローブは、5末端に蛍光HEX色素、3末端にブラックホールクエンチャー(BHQ1)で標識されています。ロックされたヌクレオチド修飾塩基は、配列中の塩基の前にプラス記号で示されます。 この表をダウンロードするには、ここをクリックしてください。
表2:LN34lys、Actin3、およびLN34Mアッセイ用のアッセイセットアップ。 プライマーとプローブの名称、配列、および濃度は 、表1に記載されています。LN34_F1 ACGCTTAACAACCAGATCAAAGAA7に対応します。 この表をダウンロードするには、ここをクリックしてください。
表3:ABI機器のサイクリングパラメータ。 重要: FASTモードではなく、STANDARDモードで実行してください。受動参照色素としてROXを選択する必要があります。この表をダウンロードするには、ここをクリックしてください。
表4:シングルプレックス(上、青のテーブル)およびマルチプレックス(下、赤いテーブル)フォーマットのLN34リアルタイムRT-PCR結果を解釈するためのアルゴリズム。 LN34の結果が陽性であれば、βAの結果が陰性であったり、決定的ではない場合でも、陽性と見なすべきです。LN34アンプリコンが検出されない場合、βA Ctがネガティブとみなされるためには、リストされているCt値のカットオフ≤必要があります。βA Ct値は、試験対象のサンプルの品質を示し、阻害の可能性を特定します。元の臨床検体中の低濃度は、βAの成長曲線に影響を与え、識別可能な増幅につながらない可能性があります。β-アクチンの検出に失敗するその他の要因には、RNAの損失やPCR阻害剤のキャリーオーバーによるRNAの抽出不良、アッセイのセットアップや技術の誤り、サンプルの種類や品質の不満足、試薬や機器の誤動作などがあります。 この表をダウンロードするには、ここをクリックしてください。
表5:LN34アッセイコントロールの一般的な結果の作用と解釈。 3 つのコントロールすべて (狂犬病陽性対照 RNA、狂犬病陰性抽出コントロール、およびテンプレートなしコントロール) は、ランが合格するために期待される結果を生成する必要があります。ポジティブコントロールの失敗またはテンプレートコントロールなしは、ピペッティングの誤り、試薬、または機器の故障を示している可能性があります。試験したすべての RNA サンプルを含む全ランを繰り返す必要があります。抽出制御の失敗は、試薬の失敗、ピペッティングの誤り、交差汚染など、抽出中の問題を示している可能性があります。すべてのサンプルの抽出を繰り返す必要があります。制御の失敗は、経験豊富な研究所の担当者にとってまれなはずです。 この表をダウンロードするには、ここをクリックしてください。
ディスカッション
LN34アッセイを正常に実行するには、ポジティブコントロール、抽出コントロールが必要であり、各アッセイランでテンプレートコントロール反応が期待どおりに機能しないか、ランを無効にして繰り返す必要があります。LN34ポジティブコントロールのレプリケート反応3つすべてが、指定された範囲内で閾値を超えるか、ランを繰り返す必要があります。以前の出版物7,8に記載されているポジティブコントロールRNAは、βAアッセイでは増幅しません。テンプレートなしのコントロール反応は、LN34またはβAアッセイのいずれかの閾値ラインを横切る増幅曲線を示さないようにする必要があります。抽出制御は、LN34の増幅を示さないようにする必要があります。NTCまたは抽出コントロールで予期しない増幅が観察された場合、汚染を示し、すべてのサンプルの実行および再試験が無効になる可能性があります(表5を参照)。ユーザーは、抽出試薬のホストβA汚染を監視するために、ノープロセスコントロールやノーサンプル抽出コントロールなどの追加のコントロールを追加することを検討できます。
狂犬病の致死率が100%に近づくと、Ct値を生成しない場合でも、弱いまたは異常な増幅をさらに調査することをお勧めします。陰性反応やNTC反応では増幅を示さず、蛍光は多成分ビューでROX蛍光に平行な平坦な線として現れる必要があります。曲線の観察、特に複数回の反復での観察は、交差汚染または弱い陽性の結果を示している可能性があります。陽性サンプルのすべての反復は、有効な陽性結果を得るために増幅する必要があります。いずれかのアッセイで反復の一部のみが増幅される場合は、サンプルを再試験する必要があります。さらに、結果が大きく変動するサンプル(反復間のCt値の差> ±1.5)は無効とみなし、サンプルを再試験する必要があります。問題が解決しない場合は、サンプルを再抽出する必要があります。
適切に収集および保存された脳幹および小脳組織から抽出された狂犬病陽性サンプルは、LN34アッセイのCt値が35サイクル未満であると予想されます。すべての決定的でないサンプルは、LN34リアルタイムRT-PCRで再テストする必要があります。再試験を行い、すべてのコントロールを期待どおりに実施した結果、サンプルが決定的でない場合は、RNAの再抽出をお勧めします。ウイルス RNA が少ないサンプル(LN34 Ct > 35)は、コンタミネーション、ウイルス負荷の低下、PCR 阻害、抽出の失敗などの潜在的な問題を示している可能性があります。元の組織から脳の新鮮な断片を採取し、RNA抽出を行い、サンプルを再試験します。同様に、βAアッセイでのCt値が33(シングルプレックス)、37(LN34M)>、または増幅がない場合、RNA抽出が失敗したことを示している可能性があります。そのようなサンプルについて抽出を繰り返し、次にLN34とアクチンの両方についてテストを繰り返します。テストを繰り返してもサンプルの結果が再び決定的ではない場合は、DFA(FAT)テスト、DRIT、ウイルス分離などの二次的な方法を使用してください。不一致の結果が続く場合や決定的でない結果が観察される場合は、狂犬病参照検査室で確認試験を受けてください。
RNA抽出にインヒビターが存在する場合、PCRアッセイでは偽陰性の結果が生じる可能性があります。特定のサンプルで阻害が疑われる場合、またはβA制御反応(Ct値>33やCt値>37など)の阻害が認められた場合は、抽出したRNAを2回以上の希釈(ヌクレアーゼフリー水で1:10および1:100など)で試験し、潜在的なPCR阻害剤を希釈する必要があります。難しいサンプルの場合、RT-PCR反応では、水を一切加えないことで、RNAインプットを8.5μLに増やすことができます。これにより、阻害の増加(2 μLインプットRNAと比較してCt値が遅い)または元のサンプルのRNAレベルが低い(2 μLインプットRNAと比較して8.5 μLを使用した場合のCt値が早い)ことが明らかになる場合があります。
LN34アッセイは、リッサウイルスを区別したり、狂犬病ウイルスの変異体を決定したりしません。LN34アッセイアンプリコンは、低分解能狂犬病ウイルス変異体タイピングまたはリッサウイルス種の同定のためにシーケンシングできます11。
開示事項
開示するものなし
謝辞
私たちは、オープンなデータ共有とフィードバックを通じてLN34アッセイの実施、検証、最適化に貢献してきた多くの狂犬病診断検査研究所の努力と協力に感謝します。商号および商業的な情報源の使用は、識別のみを目的としており、疾病管理予防センター、米国保健社会福祉省、または著者の関連機関による承認を意味するものではありません。著者が表明した結論、調査結果、意見は、必ずしも米国保健社会福祉省、疾病管理予防センター、または著者の関連機関の公式な立場を反映しているわけではありません。
資料
| Name | Company | Catalog Number | Comments |
| 7500 Fast | Applied Biosystems | N/A | Do not substitute without validation |
| 7500 Fast Dx | Applied Biosystems | N/A | Do not substitute without validation |
| ABI ViiA 7 | Applied Biosystems | N/A | Do not substitute without validation |
| AgPath-ID One-Step RT-PCR Kit | ThermoFisher Scientific | AM1005 | Do not substitute without validation |
| Beadbug6 | Benchmark Scientific | D1036 | |
| Direct-zol RNA MiniPrep kit | Zymo Research | R2052 | |
| MagNA Lyser green beads | Roche | 3358941001 | |
| Microcentrifuge | Eppendorf | 5425 R | |
| Optical 96-well Reaction Plates | ThermoFisher Scientific | 4346907 | |
| Optical Adhesive covers | ThermoFisher Scientific | 4311971 | Alternative: caps |
| Polyester fiber-tipped applicator swabs | BD BBL Polyester Fiber Tipped Application Swab | 220690 | |
| QuantStudio 6Flex | Applied Biosystems | 4485691 | Do not substitute without validation |
| Quaternary ammonium disinfectant (1:256) | LYSOL | WBB56939 | Do not substitute without validation |
| RNase AWAY | ThermoFisher Scientific | 7002PK | |
| RNaseZap | ThermoFisher Scientific | AM9780 | |
| Single-use scalpel, a scalpel with a safety mechanism | Integra Miltex | 4-510 | |
| Sterile polyproylene microcentrifuge tubes (1.5 mL), nuclease free | Sarstedt | 72.692.405 | |
| Sterile polyproylene microcentrifuge tubes (2 mL), nuclease free | Sarstedt | 72.694.600 | |
| TRIzol Reagent | ThermoFisher Scientific | 15596026 | Do not substitute without validation |
参考文献
- Meechan, P. J., Potts, J. . Biosafety in Microbiological and Biomedical Laboratories. , (2020).
- . Terrestrial Manual 2023 Available from: https://www.woah.org/en/what-we-do/standards/codes-and-manuals (2023)
- . Laboratory Techniques in Rabies Available from: https://iris.who.int/handle/10665/310836 (2018)
- . Laboratory Techniques in Rabies Available from: https://iris.who.int/bitstream/handle/10665/310837/9789241515306-eng.pdf?ua=1 (2019)
- . Protocol for postmortem diagnosis of rabies in animals by direct fluorescent antibody testing: A minimum standard for rabies diagnosis in the United States Available from: https://www.cdc.gov/rabies/pdf/RabiesDFASPv2.pdf (2003)
- World Health Organization. . WHO Expert Consultation on Rabies: Third Report. , (2018).
- Gigante, C. M., et al. Multi-site evaluation of the LN34 pan-lyssavirus real-time RT-PCR assay for postmortem rabies diagnostics. PLoS One. 13 (5), e0197074 (2018).
- Wadhwa, A., et al. A Pan-Lyssavirus Taqman Real-Time RT-PCR assay for the detection of highly variable rabies virus and other lyssaviruses. PLoS Negl Trop Dis. 11 (1), e0005258 (2017).
- Gigante, C. M., Wicker, V., Wilkins, K., Seiders, M., Zhao, H., Patel, P., Orciari, L., Condori, R. E., Dettinger, L., Yager, P., Xia, D., Li, Y., et al. Optimization of pan-lyssavirus LN34 assay for streamlined rabies diagnostics by real-time RT-PCR. Journal Virological Methods. , (2024).
- Rao, A. K., et al. Use of a modified preexposure prophylaxis vaccination schedule to prevent human rabies: recommendations of the advisory committee on immunization practices-United States, 2022. Morbidity and Mortality Weekly Report. 71 (18), 619 (2022).
- Condori, R. E., et al. Using the LN34 Pan-Lyssavirus Real-Time RT-PCR assay for rabies diagnosis and rapid genetic typing from formalin-fixed human brain tissue. Viruses. 12 (1), 120 (2020).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved