Method Article
Diagnosi post-mortem della rabbia negli animali mediante il test di reazione a catena della polimerasi a trascrizione inversa in tempo reale LN34 aggiornato e multiplexato
In questo articolo
Riepilogo
Questo protocollo dimostra il test della reazione a catena della polimerasi (RT-PCR) in tempo reale della trascrizione inversa del pan-lyssavirus LN34 dalla raccolta dei tessuti all'interpretazione dei risultati, compresi gli aggiornamenti alle sequenze e alle formulazioni dei primer per migliorare le prestazioni del test per alcuni lissavirus e lagomorfi non antirabbici. Dimostriamo anche la configurazione del saggio per un formato multiplex LN34 a pozzetto singolo (LN34M).
Abstract
La rabbia è una malattia zoonotica fatale causata dal Lyssavirus rabies (RABV) e dai virus a RNA a filamento negativo correlati del genere Lyssavirus (famiglia Rhabdoviridae). Il test LN34 ha come bersaglio la regione leader altamente conservata e il gene nucleoproteico del genoma del lyssavirus e utilizza primer degeneri e una sonda TaqMan contenente nucleotidi bloccati per rilevare l'RNA nel variegato genere Lyssavirus . Un risultato negativo per la rabbia dovrebbe essere fatto solo se vengono esaminati una sezione trasversale completa del tronco encefalico e tre lobi del cervelletto; tuttavia, l'identificazione dell'RNA del lyssavirus in qualsiasi tessuto è diagnostica dell'infezione da rabbia. Il tessuto viene raccolto e omogeneizzato nel reagente TRIzol, che inattiva anche il virus. L'estrazione dell'RNA viene eseguita utilizzando un kit di estrazione commerciale basato su colonna di rotazione. Le miscele master vengono preparate in uno spazio pulito e aliquotate in una piastra a 96 pozzetti prima di aggiungere l'RNA campione. In ambito clinico, ogni campione viene testato mediante RT-PCR in tempo reale per la presenza di RNA del lyssavirus in triplicato e singolarmente per l'β dell'ospite -actina mRNA. I controlli positivi e negativi sono inclusi nelle fasi di estrazione e RT-PCR in tempo reale del protocollo. L'analisi dei dati comporta la regolazione manuale delle soglie per standardizzare i valori Ct tra le esecuzioni dello strumento. I risultati positivi sono determinati dalla presenza di amplificazione tipica nel test del pan-lyssavirus (Ct ≤ 35). I risultati negativi sono determinati dall'assenza di amplificazione tipica nel test del pan-lyssavirus e dal rilevamento dell'mRNA dell'ospite β-actina (Ct ≤ 33). L'osservazione di valori al di fuori di questi intervalli o il fallimento dei controlli del saggio possono invalidare la corsa o portare a risultati inconcludenti per un campione. Il protocollo deve essere seguito attentamente per garantire un'elevata sensibilità e specificità del test. Le modifiche procedurali possono influire sulle prestazioni del test e portare a risultati falsi positivi, falsi negativi o non interpretabili.
Introduzione
Questo protocollo descrive la procedura per i test diagnostici della rabbia utilizzando il test LN34 pan-lyssavirus in tempo reale della reazione a catena della polimerasi a trascrizione inversa (RT-PCR) dalla raccolta del campione all'interpretazione dei risultati. La procedura sarà suddivisa in tre sezioni: raccolta del campione cerebrale pertinente al test LN34 (Sezione 1), estrazione manuale dell'RNA basata su colonna utilizzando il kit Direct-zol RNA Miniprep (Zymo Research R2051) (Sezione 2) e test RT-PCR in tempo reale LN34 impostato utilizzando il kit AgPath-ID One-Step RT-PCR (ThermoFisher Scientific AM1005) (Sezione 3). L'estrazione dell'RNA e la RT-PCR possono essere eseguite utilizzando altri prodotti, ma i kit devono essere convalidati prima dell'uso per garantire che l'RNA del Lyssavirus venga estratto e amplificato in modo appropriato.
La sezione 1 descrive la raccolta dei tessuti cerebrali appropriati da utilizzare nel test RT-PCR in tempo reale LN34. La descrizione della necroscopia animale, la decapitazione e la rimozione del cervello non sono incluse. I campioni possono contenere agenti infettivi. Per mitigare il rischio, è necessario seguire le procedure di biosicurezza descritte nella sezione Biosicurezza nei laboratori microbiologici e biomedici 6a edizione1 . I campioni devono essere considerati infettivi fino al completamento dell'inattivazione. L'inattivazione virale e la convalida del test devono essere eseguite in ciascun laboratorio secondo gli standard di tale istituto. I laboratori devono seguire le procedure standard di sicurezza e qualità determinate dal loro istituto quando implementano un nuovo test diagnostico.
Sulla base di ciò che è noto sulla diffusione del virus della rabbia durante l'infezione, il tronco encefalico e il cervelletto sono i migliori tessuti per la diagnosi della rabbia e questi tessuti sono raccomandati per i test della rabbia dall'Organizzazione Mondiale della Sanità e dall'Organizzazione Mondiale per la Salute Animale 2,3,4,5. Poiché la diffusione del virus può essere unilaterale (Figura 1), specialmente negli animali più grandi, una sezione trasversale completa del tronco encefalico e tre lobi del cervelletto dovrebbero essere esaminati per escludere la rabbia. Per i campioni che non soddisfano questi criteri minimi, il laboratorio può rifiutare il campione in quanto insufficiente per l'analisi o scegliere di eseguire l'analisi a fini di sorveglianza o di regolamentazione. Se i tessuti richiesti non vengono ricevuti, ma il laboratorio sceglie di testare il campione, un risultato negativo del test deve essere interpretato come inconcludente per la rabbia per quell'animale perché la presenza di RNA virale in altri tessuti può essere ritardata, bassa abbondanza, intermittente o inesistente. In questo caso è necessaria la raccolta dei campioni richiesti o ulteriori test per escludere la rabbia. Tuttavia, l'identificazione dell'RNA del lyssavirus in qualsiasi tessuto è diagnostica dell'infezione da rabbia 3,6. Esempi di campioni che possono essere testati per l'RNA del virus della rabbia per l'infezione da rabbia per il rule-in o la sorveglianza (ma non l'esclusione) sono la corteccia, l'ippocampo, il midollo spinale, i campioni degradati, la pelle, la saliva e la cornea. Una valutazione qualitativa delle condizioni di ciascun campione deve essere effettuata all'arrivo in laboratorio. La refrigerazione può conservare un campione per almeno 72 ore, ma non deve essere utilizzata a lungo termine. Cicli ripetuti di congelamento-scongelamento possono ridurre la sensibilità del test e dovrebbero essere evitati più di cinque cicli di congelamento-scongelamento. Se le condizioni del tessuto impediscono l'identificazione sicura delle strutture cerebrali, il campione deve essere identificato come insoddisfacente. Nel caso di un campione insoddisfacente, i test possono ancora essere eseguiti per stabilire (ma non escludere) la rabbia. I risultati positivi del test sono riportati come tali. I risultati negativi o inconcludenti su tessuto insoddisfacente devono essere riportati come non conclusivi per evitare interpretazioni errate come diagnosi negativa.
Questo protocollo è stato sviluppato a partire dalle procedure pubblicate 7,8,9 e include primer aggiornati che mirano alla regione leader del genoma del lyssavirus e alla sequenza codificante delle nucleoproteine. La sonda si rivolge a una sequenza corta e altamente conservata e utilizza nucleotidi bloccati per consentire un'ampia rivelazione. Il test rileva l'RNA di diversi lyssavirus a concentrazioni variabili8. Questo protocollo dimostra le procedure di laboratorio per eseguire il test PCR in tempo reale LN34, ma il rilevamento accurato e sensibile dell'RNA del lyssavirus dipende da altri elementi che non sono ampiamente trattati in questo protocollo, come la conservazione dei campioni, la tenuta dei registri, la formazione/competenza del personale, il monitoraggio dei risultati, l'interpretazione dei risultati, la garanzia della qualità, le misure di sicurezza del laboratorio e la risoluzione dei problemi. I saggi basati sulla PCR sono soggetti a contaminazione incrociata a causa della loro elevata sensibilità. La contaminazione incrociata può essere evitata aderendo alle buone pratiche di laboratorio, come cambiare frequentemente i guanti, maneggiare un campione alla volta, disinfettare le superfici di lavoro con agenti decontaminanti efficaci tra i campioni e mantenere le provette chiuse e i campioni separati dai reagenti PCR. I reagenti e i campioni per PCR possono essere facilmente separati utilizzando un flusso di lavoro unilaterale e separando le aree di lavoro di pre-amplificazione e post-amplificazione. Ad esempio, è possibile preparare le mastermix PCR in un luogo fisicamente separato da quello in cui vengono manipolati i campioni. Cambiare spesso i guanti per evitare la contaminazione dei reagenti PCR con campioni, detriti o RNA di controllo positivo. La piastra o le provette per PCR devono essere spostate dopo l'aggiunta della mastermix in una seconda posizione in cui è possibile aggiungere il campione e l'RNA di controllo. È importante sottolineare che i prodotti PCR non devono essere manipolati nelle aree in cui vengono preparati campioni o mastermix.
Non c'è nulla che possa sostituire la pratica e l'esperienza nell'esecuzione dei test diagnostici. Tutti i nuovi dipendenti devono essere formati e il personale addetto ai test deve essere valutato per la competenza almeno una volta all'anno in base ai requisiti del direttore di laboratorio competente. Eventuali osservazioni di risultati insoliti o di errori di analisi devono essere annotate, esaminate e corrette immediatamente. Ogni nuovo lotto di reagenti deve essere convalidato utilizzando campioni con valori Ct noti (come un campione di controllo positivo o un campione archiviato). Tutte le apparecchiature devono essere sottoposte a manutenzione ordinaria, come suggerito dal produttore, e le prestazioni del test devono essere verificate dopo qualsiasi manutenzione o riparazione. I livelli di temperatura devono essere monitorati sulle apparecchiature applicabili per garantire che i frigoriferi e i congelatori rimangano entro i criteri stabiliti per un intervallo di temperatura accettabile per i reagenti utilizzati nei test diagnostici.
Le modifiche procedurali possono influire sulle prestazioni del test e possono portare a risultati falsi positivi, falsi negativi o non interpretabili. Le raccomandazioni devono essere seguite attentamente per garantire un'elevata sensibilità e specificità del test. Un laboratorio che desideri incorporare modifiche a questo protocollo dovrebbe convalidare e confermare i metodi modificati in consultazione con il CDC.
Protocollo
I campioni di tessuto cerebrale post mortem sono stati ottenuti tramite la sorveglianza di routine o le attività diagnostiche del Poxvirus and Rabies Branch (CDC; Atlanta, GA, USA).
1. Raccolta di tessuto cerebrale per la diagnosi post mortem della rabbia negli animali mediante il test RT-PCR in tempo reale LN34 pan-lyssavirus
NOTA: I campioni possono contenere agenti infettivi. Indossare dispositivi di protezione individuale (DPI) adeguati (guanti di gomma pesanti o altri guanti resistenti al taglio, camice da laboratorio, grembiule impermeabile, maschera chirurgica, stivali, maniche protettive e visiera) e seguire le norme di sicurezza richieste per l'uso, la conservazione e lo smaltimento dei campioni. La vaccinazione antirabbica pre-esposizione, i test sierologici regolari e le vaccinazioni di richiamo (se necessario) sono richiesti per chiunque prima di lavorare, testare, produrre o eseguire attività di ricerca con lyssavirus o campioni noti o potenzialmente infetti 2,3,4,6,10.
- Etichettare una provetta di raccolta per campione con un'etichetta di adesione. Riempire ogni provetta di raccolta del campione con 1 mL di reagente TRIzol o altro tampone di omogeneizzazione e una porzione di perle MagNA Lyser (di seguito "perle di ceramica"). Per aggiungere le perle di ceramica, versare con cura dal tubo di perline nella provetta di raccolta del campione. Le provette di perle di ceramica contengono generalmente abbastanza perle per 2-5 campioni, utilizzando almeno 20 perle di 1,4 mm di diametro per campione.
ATTENZIONE: Il reagente TRIzol (di seguito "tampone di omogeneizzazione") è una sostanza chimica pericolosa; il contatto con acidi o candeggina libera gas tossici; garantire un'adeguata ventilazione; Per ulteriori informazioni, fare riferimento alla scheda di dati di sicurezza. Se gli utenti sostituiscono il reagente TRIzol o il reagente TRI con un altro tampone di omogeneizzazione, è necessaria un'ulteriore convalida. TRIzol funge da tampone di omogeneizzazione/lisi del campione, tampone di inattivazione del lissavirus e tampone di stabilità dell'RNA per questo protocollo. L'uso di un tampone di omogeneizzazione alternativo richiederà la convalida dell'efficienza di estrazione, dell'inattivazione e della stabilità in un confronto controllato fianco a fianco. - Pulire e disinfettare la superficie di lavoro con disinfettante a base di composti di ammonio quaternario (QAC) per 2 minuti e stendere un tampone assorbente rivestito di plastica. Collocare solo i reagenti e le forniture per il primo campione in una cabina di sicurezza biologica (BSC) di Classe II con caratteristiche per l'evacuazione dei fumi pericolosi all'esterno della stanza.
NOTA: Fare riferimento alle linee guida del produttore per i limiti di conservazione del QAC diluito. Assicurarsi che il cuscinetto rivestito in plastica non ostruisca il flusso d'aria della cabina di sicurezza biologica. Se il flusso d'aria viene interrotto, non utilizzare un tampone. - Raccogli il tessuto che rappresenta una sezione trasversale completa del tronco encefalico e del cervelletto utilizzando un bisturi monouso pulito.
NOTA: La manipolazione dei tessuti deve essere condotta in modo da non aerosolizzare liquidi o produrre particelle sospese nell'aria. Non sono necessarie cappe chimiche o cappe di biosicurezza, ma si consigliano cappe di biosicurezza ventilate poiché forniscono una protezione aggiuntiva da odori, fumi, ectoparassiti e frammenti ossei.
ATTENZIONE: L'uso di un bisturi con materiale potenzialmente infetto da lyssavirus è pericoloso e gli utenti devono prendere le opportune precauzioni di sicurezza. Si raccomanda l'uso di pinze monouso.- Per i piccoli animali (come i pipistrelli), l'intero tronco encefalico e il cervelletto possono essere raccolti.
- Per gli animali più grandi, raccogli una sezione trasversale completa del tronco encefalico e del tessuto da ciascuno dei tre lobi del cervelletto.
- FACOLTATIVO: Se si esegue il test degli anticorpi fluorescenti diretti (DFA), raccogliere le impronte cerebrali a questo punto. Utilizzare il tessuto rimanente dopo aver raccolto le impronte cerebrali per la DFA per l'estrazione dell'RNA e il test mediante il test LN34.
NOTA: Se il TRIzol viene aggiunto ai campioni, i campioni non possono più essere utilizzati per i metodi di rilevamento basati sull'antigene o per l'isolamento del virus.
- Preparare i campioni per l'omogeneizzazione e l'estrazione dell'RNA.
NOTA: L'efficienza dell'estrazione dell'RNA e l'inattivazione del virus possono essere influenzate dall'utilizzo di una quantità eccessiva di tessuto. La quantità di tessuto non deve superare circa 1/10 del volume del tampone di omogeneizzazione utilizzato. Se viene utilizzato più tessuto, aumentare di conseguenza la quantità di tampone di omogeneizzazione per garantire un'estrazione efficiente e di successo dell'RNA.- Per gli animali di piccola taglia, posizionare tutti i tessuti necessari in una provetta contenente un tampone di omogeneizzazione e perline per l'estrazione. Non superare i 100 mg di campione in 1 mL di tampone di omogeneizzazione; Per campioni più grandi, aumentare il volume del tampone di omogeneizzazione o utilizzare più provette per riflettere un rapporto campione: tampone con rapporto 1:10.
- Per gli animali più grandi, tritare e omogeneizzare accuratamente il tessuto e rimuovere una parte rappresentativa in una provetta preriempita con tampone di omogeneizzazione e perline. Non superare i 100 mg di campione in 1 mL di tampone di omogeneizzazione; Per campioni più grandi, aumentare il volume del tampone di omogeneizzazione o utilizzare più provette per riflettere un rapporto campione: tampone con rapporto 1:10.
- Opzione 1 (battitore di microsfere): omogeneizzare il tessuto utilizzando un battitore di perline, 1 ml di tampone e perle di ceramica. Potrebbe essere necessario utilizzare diverse provette da 2 mL o provette più grandi.
- Opzione 1 (Battitore di talloni): pulire e disinfettare la postazione di lavoro, l'attrezzatura e le provette esterne con disinfettante QAC (1:256). Lasciate riposare per 2 min.
- Opzione 1 (Battitore di perline): all'interno del BSC, caricare un rotore da centrifuga con campioni omogeneizzati. Centrifugare tutti i campioni a 10.000-16.000 × g per 2 minuti in una microcentrifuga da tavolo. Scaricare il rotore della centrifuga all'interno del BSC.
- Opzione 1 (Sbattitore per talloni): Lasciare riposare per 2 minuti.
- Opzione 1 (Battitore di microerline): trasferire 120 μL di omogeneizzato in una provetta preriempita con 1 mL di tampone di omogeneizzazione.
ATTENZIONE: L'omogeneizzazione può produrre aerosol e deve essere eseguita in un BSC. - Opzione 2 (bisturi): tritare finemente i tessuti necessari utilizzando un bisturi monouso, spalmare con un tampone e trasferire il tampone in una provetta preriempita con tampone di omogeneizzazione e perline. Non superare i 100 mg di campione in 1 mL di tampone di omogeneizzazione; Per campioni più grandi, aumentare il volume del tampone di omogeneizzazione o utilizzare più provette per riflettere un rapporto campione:tampone 1:10.
ATTENZIONE: L'uso di un bisturi con materiale potenzialmente infetto da lyssavirus è pericoloso e gli utenti devono prendere le opportune precauzioni di sicurezza.
- Raccogli i fazzoletti rimanenti nel contenitore originale o in un nuovo contenitore vuoto etichettato con un'etichetta di accesso. Conservare questo tessuto nel caso in cui sia necessario ripetere il test o caratterizzare ulteriormente.
- Pulire e disinfettare la postazione di lavoro, l'attrezzatura e l'esterno delle provette del campione con il disinfettante QAC 1:256. Lasciate riposare per 2 min.
- Ripetere i passaggi 1.2-1.5 per tutti i campioni rimanenti.
- Omogeneizzare i campioni con un mini battitore per microsfere per almeno 60 s. Ispezionare visivamente i tubi. Ripetere il frusta per altri 60 s se rimangono pezzi di tessuto di grandi dimensioni. Questo passaggio è facoltativo se il tessuto è completamente omogeneizzato nel passaggio 1.4.2.1 di cui sopra.
NOTA: È importante assicurarsi che il tessuto sia completamente omogeneizzato. L'omogeneizzazione incompleta diminuirà la resa dell'RNA. - Lasciare riposare per almeno 5 minuti a temperatura ambiente (RT).
- Pulire e disinfettare la postazione di lavoro, l'attrezzatura e l'esterno delle provette con il disinfettante QAC (1:256).
NOTA: Il campione è considerato non infettivo in questo momento e può essere rimosso dal laboratorio di rabbia. - Processare immediatamente i campioni nel tampone di omogeneizzazione per l'estrazione dell'RNA, conservare a RT (da 20 °C a 25 °C) o in frigorifero (da 4 °C a 8 °C) per diversi giorni, oppure conservare a -16 °C o più freddo per una conservazione a lungo termine.
2. Protocollo per l'estrazione dell'RNA utilizzando il kit RNA MiniPrep
- Configura l'area di lavoro sotto il BSC.
- Pulire la superficie di lavoro BSC utilizzando etanolo al 70% prima di iniziare il lavoro per rimuovere polvere o altri contaminanti ambientali. Eseguire un'ulteriore decontaminazione delle superfici con disinfettante QAC (1:256), RNase AWAY o RNaseZap (secondo le raccomandazioni del produttore).
- Stendere un tampone di lavoro assorbente rivestito di plastica e posizionare i reagenti, i materiali di consumo e il campione nel BSC.
NOTA: Assicurarsi che il cuscinetto rivestito in plastica non ostruisca il flusso d'aria del BSC. Se il flusso d'aria viene interrotto, non utilizzare un tampone. - Disporre tutte le provette di raccolta in un rack pulito per provette da microcentrifuga. Preriempire una provetta per microcentrifuga da 1,5 mL con 300 μL di etanolo al 100% per ogni campione di cervello non di pipistrello. Per i campioni con poco tessuto (campione di cervello di pipistrello, campione non cerebrale o campione deteriorato), preriempire una provetta da microcentrifuga da 1,5 ml con 600 μl di etanolo al 100% per ciascuno.
- Preparazione del campione
- Raccogliere tutti i campioni preparati nella sezione 1 in un rack per provette nel BSC. Scongelare i campioni congelati appena prima del test.
- Scongelare un controllo di estrazione.
NOTA: Si raccomanda di scegliere un campione privo di RNA di lyssavirus; il campione deve essere precedentemente testato con un intervallo di valori Ct previsto per il test della beta-actina. Ad esempio, cellule di coltura tissutale pre-aliquotate o un caso di rabbia negativo precedentemente testato (umano o animale).
- Centrifugare tutti i campioni a 10.000-16.000 × g per 2 m in una microcentrifuga da tavolo.
- Trasferire il surnatante in una nuova provetta sterile per microcentrifuga contenente etanolo al 100%. Assicurarsi che il surnatante sia limpido, senza lipidi evidenti o tessuti solidi. Evitare la raccolta di lipidi e tessuti solidi.
- Per il tessuto cerebrale non bat-bat: trasferire 300 μl di surnatante.
- Per i campioni con poco tessuto (campione di cervello di pipistrello, campione non cerebrale o campione deteriorato), trasferire 600 μl di surnatante.
- Conservare l'omogeneizzato rimanente in una provetta da microcentrifuga con tappo a vite a ≤-16 °C.
- Pipettare su e giù 10 volte per mescolare.
- Per ogni campione, trasferire 600 μl della miscela etanolo-surnatante in una colonna di centrifuga in una provetta di raccolta.
- Centrifugare fino a quando il liquido non è passato attraverso la colonna (1 min a 10.000-16.000 × g). Scartare il flusso.
- Ripetere se sono presenti più di 600 μl di miscela tampone per omogeneizzazione dell'etanolo per un campione.
- Trasferire ogni colonna in una nuova provetta di raccolta.
- Aggiungere 400 μl di tampone di prelavaggio RNA a ciascuna colonna e centrifugare a 10.000-16.000 × g per 30 s.
- Scartare il flusso di passaggio e riportare ogni colonna nella stessa provetta di raccolta.
- Ripetere i passaggi 2.10-2.11.
- Aggiungere 700 μl di tampone di lavaggio dell'RNA a ciascuna colonna e centrifugare a 10.000-16.000 × g per 2 minuti. Assicurarsi che il tampone di lavaggio sia passato completamente attraverso ciascuna colonna.
- Trasferire con cura ogni colonna in una provetta priva di RNasi.
- Eliminare il flusso di passaggio e il tubo di raccolta da 2.13.
- Aggiungere 50 μL di acqua priva di DNasi/RNasi direttamente nella matrice della colonna per eluire l'RNA.
NOTA: Non toccare la matrice della colonna con il puntale della pipetta. - Incubare per 30 s a RT, quindi centrifugare a 10.000-16.000 × g per 1 min.
- Trasferire con cura l'RNA in una nuova provetta per microcentrifuga con etichetta di adesione a fondo piatto con tappo a vite. Spostare l'RNA estratto nel ghiaccio per un test immediato. Conservare a lungo termine a -70 °C o più freddo.
NOTA: La conservazione a temperature più calde o ripetuti scongelamenti da gelo possono portare alla degradazione dell'RNA e influire sui risultati diagnostici.
3. Protocollo per il test RT-PCR in tempo reale del pan-lyssavirus LN34
- Preparare i reagenti.
- Controllo positivo artificiale
- Se un RNA di controllo positivo artificiale viene prodotto da CDC8, seguire le istruzioni sulla confezione per la conservazione, la ricostituzione e l'aliquotazione. Saltare questo passaggio se sono già disponibili aliquote monouso di RNA di controllo positivo.
NOTA: L'RNA di controllo positivo alle concentrazioni di lavoro deve essere maneggiato nell'area di aggiunta del modello e non nella stessa area della preparazione del mastermix. L'RNA di controllo positivo dovrebbe produrre un valore di soglia del ciclo (Ct) entro l'intervallo previsto determinato per un dato lotto. Tra una corsa e l'altra, il valore LN34 Ct per l'RNA di controllo positivo non deve differire di oltre ±1,5 Ct valori. - Scongelare un'aliquota monouso da ≤ -70 °C appena prima dell'uso su ghiaccio o blocco di ghiaccio. Non congelare-scongelare e gettare le aliquote conservate per un periodo prolungato a temperature refrigerate.
NOTA: Un controllo positivo deve essere eseguito in triplicato nel saggio LN34; Il controllo positivo artificiale8 non si amplificherà nel saggio della beta-actina.
- Se un RNA di controllo positivo artificiale viene prodotto da CDC8, seguire le istruzioni sulla confezione per la conservazione, la ricostituzione e l'aliquotazione. Saltare questo passaggio se sono già disponibili aliquote monouso di RNA di controllo positivo.
- Controllo dell'estrazione e campioni: Posizionare i campioni appena estratti sul ghiaccio (o blocco di ghiaccio) o scongelare i campioni da ≤ -70 °C di conservazione su ghiaccio (o blocco di ghiaccio) immediatamente prima dell'uso.
NOTA: L'RNA deve essere scongelato e processato in un'area designata per l'aggiunta di campioni o mascherine, separata dalle aree utilizzate per la preparazione del mastermix o la manipolazione di prodotti PCR o grandi quantità di materiale virale (ad esempio, generazione di controlli positivi, propagazione virale)
- Controllo positivo artificiale
- Preparare i reagenti mastermix nell'area di preparazione mastermix.
- Preparazione Mastermix del test RT-PCR singleplex LN34
NOTA: Gli utenti possono testare i campioni in formato singleplex (passaggio 3.2.1) o multiplex (passaggio 3.2.2). Non è necessario eseguire sia la versione 3.2.1 che la versione 3.3.2. La preparazione del mastermix, l'aliquotazione del primer e della sonda e l'assenza di reagenti di controllo del modello devono essere scongelati e manipolati in un'area pulita separata dal trattamento dei campioni, dalla necroscopia, dalla PCR e da altre aree in cui vengono manipolati i materiali virali. Ciò può essere ottenuto attraverso stanze separate o un sistema di armadi con flusso di campioni unilaterale.- Generare miscele di primer e sonde alle concentrazioni di lavoro indicate nella Tabella 1 e nella Tabella 2. Saltare questo passaggio se le aliquote di diluizione di lavoro di primer e sonde sono già in mano.
- Aliquotare i primer e le sonde in scorte di stoccaggio da 1,5 ml e in scorte di lavoro da 50 μl e conservare a ≤ -16 °C al buio. Saltare questo passaggio se le aliquote di diluizione di lavoro di primer e sonde sono già in mano.
NOTA: Si consiglia di eseguire il passaggio 3.2.1.2. - Scongelare il tampone RT-PCR in un'unica fase, nessun controllo del modello, primer e sonde da ≤ -16 °C Conservazione su ghiaccio o blocco di ghiaccio nell'area di preparazione del mastermix.
NOTA: Utilizzare i reagenti fino alla data di scadenza o all'insuccesso delle prestazioni, a seconda dell'evento che si verifica per primo. - Agitare brevemente e centrifugare tutti i tamponi, i primer e le sonde prima dell'uso.
- Conservare l'enzima RT-PCR in una sola fase su ghiaccio o in un blocco di ghiaccio fino all'uso.
- Campioni di RNA
NOTA: Utilizzare l'RNA appena estratto quando possibile, poiché il congelamento e lo scongelamento possono influire sulle prestazioni - Conservare i campioni di RNA su ghiaccio o blocco di ghiaccio fino al momento dell'uso.
- Scongelare eventuali campioni di RNA congelati su ghiaccio o un blocco di ghiaccio.
- Etichettare una provetta per microcentrifuga per saggio (LN34 e βA).
- Determinare il numero di reazioni (N) da impostare per ogni saggio.
- Calcolare il numero di reazioni per il test LN34 moltiplicando il numero di campioni per 3 e aggiungendo 6 per i pozzetti di reazione di controllo più il 10% di reazioni extra per tenere conto del volume perso durante il pipettaggio. (ad es. per 10 campioni: (10 x 3) + 6 = 36 reazioni; reazioni eccessive: (36 x 0,1) + 36 = 3,6 + 36 = 39,6 reazioni totali, o 40 reazioni arrotondate per eccesso)
NOTA: Per i test clinici, si consiglia di testare tutti i campioni in triplicato per LN34. A fini di sorveglianza, ogni campione può essere analizzato in duplice copia. Si raccomanda di utilizzare il triplicato durante l'onboarding iniziale del saggio per garantire una bassa variabilità tra le repliche e una buona tecnica. - Calcolare il numero di reazioni per il saggio βA sommando il numero di campioni più 4 pozzetti di reazione di controllo più il 10% di reazioni extra per tenere conto del volume perso durante il pipettaggio.
- Determinare il volume di ciascun reagente per le mastermix LN34 e βA utilizzando la Tabella 2.
- Designare i pozzetti per ciascun campione da analizzare in triplicato nel saggio LN34 e il singolo per il saggio βA utilizzando una mappa della piastra a 96 pozzetti.
- Erogare 23 μl di mastermix per saggi LN34 in ciascun pozzetto LN34 assegnato dopo aver brevemente agitato e girato per 30 s utilizzando una microcentrifuga da tavolo per raccogliere il liquido sul fondo della provetta. Evitare di introdurre bolle.
- Erogare i reagenti 23 μL di mastermix per saggi βA in ciascun metodo βA assegnato dopo aver brevemente agitato e girato per 30 s in una microcentrifuga da tavolo per raccogliere il liquido sul fondo.
- Impostare le reazioni NTC (No Template Control) pipettando 2 μL di acqua di grado PCR in ciascun pozzetto NTC.
- Coprire i pozzetti e trasferire la piastra nell'area di aggiunta della dima.
- Agitare brevemente e centrifugare le provette contenenti i campioni di RNA.
- Pipettare 2 μL di RNA estratto dal primo campione in ciascun pozzetto etichettato per quel campione. Evitare di introdurre bolle.
- Assicurarsi che l'RNA sia stato aspirato nella pipetta mediante visualizzazione.
- Pipettare sul lato del pozzetto per assicurarsi che il campione venga aggiunto al pozzetto corretto.
- Evitare il più possibile di agitare i puntali delle pipette contenenti RNA sui pozzetti aperti.
- Ripetere il passaggio 3.2.10 per i campioni rimanenti e l'RNA di controllo positivo.
- Posizionare la copertura adesiva ottica sui pozzetti dopo aver aggiunto tutti i campioni e i controlli. Fare attenzione a coprire tutti i pozzetti e sigillare completamente.
- Centrifugare a 500 × g per 1 minuto in RT in una centrifuga da tavolo o utilizzare una centrifuga per insalata tipo centrifuga.
- Posizionare la piastra sigillata in uno strumento PCR in tempo reale calibrato per i coloranti reporter FAM e VIC/HEX e impostarla sui parametri ciclici mostrati nella Tabella 3.
- Preparare le miscele master per il saggio LN34 Multiplexed (LN34M).
- Etichettare una provetta per microcentrifuga LN34M secondo la Tabella 2.
- Determinare il numero di reazioni (N) da impostare per ogni saggio.
- Calcolare il numero di reazioni per il test LN34M moltiplicando il numero di campioni per 3 e aggiungere 6 per i pozzetti di reazione di controllo più il 10% di reazioni extra per tenere conto del volume perso durante il pipettaggio. (ad esempio, per 10 campioni: (10 x 3) + 6 = 36 reazioni; reazioni in eccesso: (36 x 0,1) + 36 = 3,6 + 36 = 39,6 reazioni totali o 40 reazioni arrotondate per eccesso)
- Scegli il formato da 25 μl o 12,5 μl. Determinare il volume di ciascun reagente per la mastermix LN34M utilizzando la Tabella 2.
- Designare i pozzetti per ogni campione da analizzare in triplicato nel saggio LN34M utilizzando una mappa della piastra a 96 pozzetti.
- Erogare i reagenti per il saggio LN34M nei pozzetti. Agitare brevemente le provette e farle girare verso il basso per raccogliere il liquido sul fondo prima di erogare 23 μl (per una reazione da 25 μl) o 10,5 μl (per una reazione da 12,5 μl) di miscela master in ciascun pozzetto assegnato. Evitare di introdurre bolle.
- Impostare le reazioni NTC pipettando 2 μL di acqua di grado PCR in ciascun pozzetto NTC.
- Coprire i pozzetti e trasferire la piastra nell'area di aggiunta della dima.
- Agitare brevemente e far girare i tubi contenenti i campioni di RNA per raccogliere il liquido sul fondo.
- Pipettare 2 μL di RNA estratto dal primo campione in ciascun pozzetto etichettato per quel campione. Evitare di introdurre bolle.
- Assicurarsi che l'RNA sia aspirato nella visualizzazione della pipetta.
- Pipettare sul lato del pozzetto per assicurarsi che il campione venga aggiunto al pozzetto corretto.
- Evitare il più possibile di agitare i puntali delle pipette contenenti RNA sui pozzetti aperti.
- Ripetere il passaggio 3.3.8 per i campioni rimanenti e l'RNA di controllo positivo.
- Dopo aver aggiunto l'ultimo campione/controllo, posizionare la copertura adesiva ottica sui pozzetti, assicurandosi che tutti i pozzetti siano coperti e sigillati completamente.
- Centrifugare a 500 × g per 1 minuto in RT in una centrifuga da tavolo o utilizzare una centrifuga per insalata.
- Posizionare la piastra sigillata in uno strumento di PCR in tempo reale calibrato per coloranti reporter FAM e VIC/HEX e impostarlo sui parametri del ciclo, come mostrato nella Tabella 3. Imposta il colorante di riferimento passivo su ROX ed esegui in modalità standard (non eseguire in modalità veloce)
NOTA: Questa impostazione è specifica per gli strumenti menzionati in questo protocollo e richiede l'uso di un reagente RT-PCR in un'unica fase contenente ROX come colorante passivo. Gli strumenti alternativi richiedono approcci diversi per determinare le impostazioni ottimali di esecuzione. Garantire la normale manutenzione dello strumento secondo il produttore per ottenere le migliori prestazioni.
- Preparazione Mastermix del test RT-PCR singleplex LN34
4. Interpretazione dei risultati
- Imposta una linea di base automatica e calcoli manuali della soglia utilizzando un valore di 0,2 per LN34/FAM e 0,05 per βA/HEX/VIC.
NOTA: Questa impostazione è specifica per gli strumenti menzionati in questo protocollo e richiede l'uso di un reagente RT-PCR in un'unica fase contenente ROX come colorante passivo. Gli strumenti alternativi richiedono approcci diversi per calcolare i valori di base e di soglia. - Determinare il risultato diagnostico utilizzando le indicazioni riportate nella Tabella 4 se tutti i controlli sono stati eseguiti come previsto (Tabella 5).
- Confermare tutti i valori Ct o Cq visualizzando i grafici di amplificazione.
- Esaminare eventuali risultati insoliti come consigliato.
5. Conservazione e conservazione dei campioni
- Conservare tutti i campioni congelati a una temperatura pari o inferiore a -16 °C fino al completamento del test e alla comunicazione dei risultati. Conservare i tessuti originali per confermare i risultati o identificare l'animale ospite della specie in caso di risultati insoliti dei test.
- Utilizzare identificatori univoci del campione; Etichettare tutte le provette, i rapporti e i documenti con identificatori univoci completi del campione.
- Conservare i campioni intermedi (a breve termine) nel caso in cui sia necessario ripetere i test.
- Conservare i campioni positivi rappresentativi necessari per l'uso come controlli, tipizzazione epidemiologica e altri scopi.
- Conservare l'RNA a ≤-70 °C per la conservazione a lungo termine.
Risultati
Nella Figura 2 sono mostrate le immagini rappresentative di un test LN34 eseguito con successo su uno strumento di PCR in tempo reale ABI ViiA7. La visualizzazione dei risultati tracciati su una scala logaritmica consente una facile visualizzazione del valore Ct, il punto in cui la curva attraversa la linea di soglia (Figura 2A, C). Se tracciata su una scala lineare, l'amplificazione riuscita apparirà come una curva sigmoidale (o a forma di "S") (Figura 2B, D), mentre i risultati negativi dovrebbero apparire come una linea retta e piatta. Si consiglia di visualizzare i risultati sia in visualizzazione lineare che in scala logaritmica per identificare possibili anomalie o errori. I tipici risultati positivi e negativi nella vista del grafico multicomponente possono essere visti rispettivamente nella Figura 2E, F, dove il livello di fluorescenza del colorante che marca la sonda (FAM per LN34, VIC/HEX per βA) può essere osservato rispetto al colorante passivo nel tampone di reazione (ROX).
Esempi di risultati anomali sono mostrati nella Figura 3. I confronti tra i grafici delle esecuzioni riuscite (Figura 2) e i grafici anomali (Figura 3) possono essere utilizzati per isolare le esecuzioni atipiche e i problemi dello strumento. La Figura 3A mostra un segnale che attraversa la soglia, producendo un valore Ct per LN34, ma la curva di amplificazione è molto atipica, aumentando linearmente. Il grafico multicomponente (Figura 3B) mostra anche una linea ondulata che non è tipica di un campione positivo. Questo esempio evidenzia l'importanza di visualizzare i grafici di amplificazione e non semplicemente copiare i valori Ct. Assicurarsi sempre che le curve di amplificazione appaiano normali per tutti i campioni. Si consiglia inoltre di visualizzare il grafico multicomponente per assicurarsi che non siano presenti irregolarità. A volte, segnali di base disordinati possono generare valori Ct nei casi in cui non si è verificata alcuna amplificazione. Se i segnali di amplificazione appaiono lineari, si consiglia di regolare la linea di base per vedere se la curva scompare. In caso di segnali insoliti, l'intera corsa deve essere ripetuta. Si consiglia di pulire ed eseguire una piastra di sfondo sullo strumento PCR in tempo reale se i problemi persistono. Se disponibili, i prodotti PCR possono essere eseguiti su un gel di agarosio e/o sequenziati per risolvere eventuali risultati insoliti. Non è consigliabile utilizzare i risultati dell'elettroforesi su gel o del sequenziamento per determinare i risultati diagnostici.
Studi precedenti hanno mostrato una bassa variabilità tra le repliche, l'esecuzione del test, l'operatore e il laboratorio per il test LN347. Se si osserva un'elevata variabilità (differenza di >±1,5 Ct) tra le repliche dello stesso campione, l'RNA deve essere riesaminato. L'elevata variabilità può essere causata da problemi con pipette, pratiche di laboratorio, pipettaggio errato o macchine per PCR in tempo reale. L'osservazione ripetuta di un'elevata variabilità tra diversi campioni o tra le esecuzioni dei saggi può indicare problemi sistemici. I campioni con basso RNA, che si avvicinano alla soglia di saggio per un campione positivo (Ct 35), possono mostrare una maggiore variabilità nei valori di Ct tra le repliche. La consultazione con il CDC e la risoluzione dei problemi possono essere necessarie per affrontare la causa della variabilità persistente, dei risultati incoerenti o del fallimento del test.
L'elevata sensibilità dei test basati sulla PCR li rende intrinsecamente suscettibili alla contaminazione. Il rigoroso rispetto delle buone pratiche di laboratorio è il modo migliore per mitigare la contaminazione incrociata. Saper identificare una potenziale contaminazione è importante. La contaminazione del reagente deve essere sospettata se non c'è un controllo del modello e i pozzetti del campione sospetti negativi in un test producono tutti valori Ct simili. Ripetere il test con nuove aliquote di reagenti PCR (tampone, acqua, primer ed enzima) e lo stesso RNA. Se tutti i campioni e il controllo dell'estrazione producono valori CT simili ma NTC è negativo, è necessario indagare sulla contaminazione dei reagenti di estrazione e ripetere l'estrazione utilizzando nuovi reagenti. È buona norma fare piccole aliquote di reagenti per ridurre il rischio di contaminazione ed evitare la possibilità di scartare grandi volumi di reagenti costosi. La contaminazione incrociata del campione è più difficile da identificare. Se si sospetta la contaminazione del campione, ripetere la raccolta del campione partendo dai tessuti originali. In alcuni casi, il sequenziamento dell'RNA virale può confermare la contaminazione, soprattutto quando l'RNA contaminante è molto diverso dalla variante virale prevista (come un virus di controllo utilizzato in laboratorio). Il sequenziamento di due campioni processati contemporaneamente può determinare se le sequenze virali sono identiche, ma può essere poco informativo se si prevede che le sequenze siano molto simili (ad esempio, la stessa variante raccolta nella stessa contea). Se si sospetta la contaminazione del campione con l'RNA di controllo positivo, è possibile eseguire gli ampliconi del saggio LN34 su un gel di agarosio per differenziare l'RNA del lyssavirus (165 bp) dall'RNA di controllo positivo (99 bp). La sequenza del modello utilizzata per generare l'RNA di controllo positivo fornito dal CDC8.
Per altri agenti patogeni, i laboratoristi possono essere utilizzati per impostare manualmente la soglia per eliminare il "rumore" come la debole amplificazione mostrata in ciano nella Figura 4. Questa pratica NON è raccomandata per la diagnosi della rabbia perché può portare a risultati falsi negativi con conseguenze disastrose poiché la rabbia è quasi al 100% fatale. NON modificare manualmente la soglia per produrre risultati negativi per campioni di amplificazione deboli o tardivi. Questi campioni devono essere estratti e/o ritestati per escludere la rabbia.
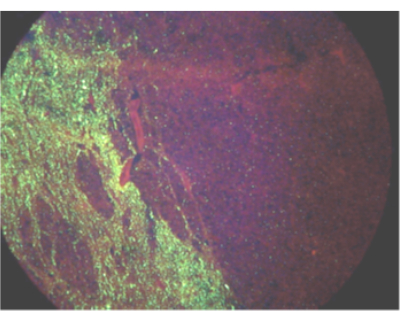
Figura 1: Campo visivo che mostra la diffusione unilaterale dell'antigene del virus della rabbia in un asino infetto mediante test diretto degli anticorpi fluorescenti. Fare clic qui per visualizzare una versione più grande di questa figura.
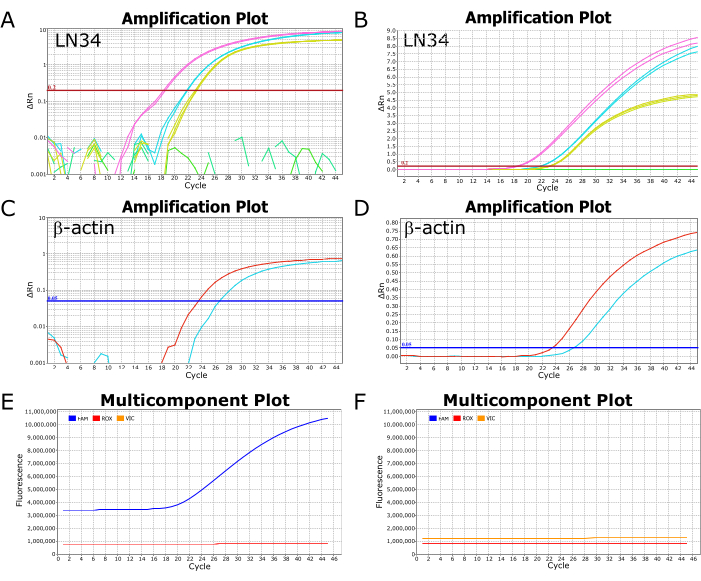
Figura 2: Amplificazione e grafici multicomponente da un test LN34 eseguito con successo. (A-D) I dati dei risultati sono tracciati su una scala logaritmica (A,C) e su una scala lineare (B,D) per il saggio LN34 e βA. I pannelli A e B mostrano i risultati di LN34 provenienti da due campioni (in rosa e ciano) rispetto al controllo positivo (in giallo). Nel pannello B, c'è una linea verde piatta che raffigura un ulteriore campione negativo nella corsa. In A, le linee verdi non mostrano alcuna amplificazione e sono rappresentate come segmenti spezzati. La soglia per il test LN34 è stata impostata manualmente su 0,2 ed è indicata dalla linea orizzontale rossa. (C, D) Risultati del test βA per due campioni (rosso e ciano). La soglia per il saggio βA è stata impostata manualmente a 0,05. (E,F) I grafici multicomponente rappresentano la fluorescenza (RFU) ad ogni ciclo per FAM (LN34), VIC (βA) e ROX (colorante passivo presente nel tampone AgPath-ID). I livelli di ROX dovrebbero rimanere piatti in tutti i cicli. Un tipico campione positivo è mostrato nel pannello E; La fluorescenza FAM aumenta come curva sigmoidale a partire dal ciclo 18 per questo campione. Un tipico campione negativo è mostrato nel pannello F, dove il livello FAM rimane parallelo al livello ROX in tutti i cicli. I dati provengono da uno strumento di PCR in tempo reale ABI ViiA7. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 3: Immagini rappresentative del segnale raro e atipico osservato nelle esecuzioni del saggio LN34 su uno strumento di PCR in tempo reale ViiA7. (A-F) Grafici di amplificazione (A,C,E) e multicomponente (B,D,F) prodotti a causa della contaminazione del pozzo. L'aumento lineare (A,C) e le fluttuazioni ondulate (B,D) nella fluorescenza FAM non rappresentano una vera amplificazione basata sulla forma delle curve e sull'entità della variazione di fluorescenza. I pannelli da A a D rappresentano probabilmente campioni negativi anche se è stato prodotto un valore Ct per la replica mostrata nei pannelli A e B. I pannelli E e F mostrano uno strano segnale ondulato che è più facilmente visibile nel grafico multicomponente. Questo tipo di segnale deve essere esaminato e può indicare problemi allo strumento, anche se tutti i controlli sono stati eseguiti come previsto in questa corsa. Clicca qui per visualizzare una versione più grande di questa figura.
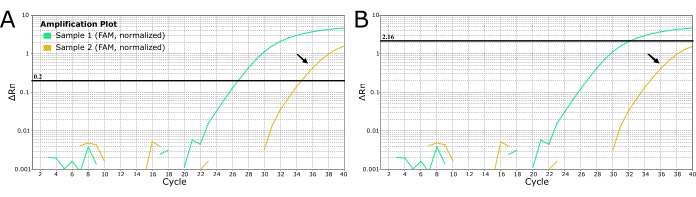
Figura 4: Curve RT-PCR in tempo reale LN34 da 2 campioni sospetti di rabbia che mostrano due metodi di impostazione dei valori di soglia. (A) La soglia LN34 è stata impostata su 0,2 (consigliata per tutte le esecuzioni). (B) Determinazione manuale di una soglia diversa per ogni corsa per mascherare il segnale determinato come "rumore" (segnale di amplificazione tardiva). Il metodo utilizzato nel pannello B NON è raccomandato per la rabbia a causa delle gravi conseguenze del mancato raggiungimento di un vero risultato positivo. L'amplificazione tardiva potrebbe indicare un campione debole positivo, l'inibizione della PCR o la mancata estrazione in un caso positivo. Potrebbe anche indicare una contaminazione incrociata. Il campione d'oro (indicato da frecce nere) produce un valore Ct al cutoff del test e non deve essere considerato negativo. I campioni con amplificazione tardiva devono essere nuovamente estratti e ritestati. Clicca qui per visualizzare una versione più grande di questa figura.
Tabella 1: Sequenze di primer e sonda e concentrazioni utilizzate nei saggi di RT-PCR in tempo reale LN34lys (singleplex LN34), LN34M (LN34 e βA multiplexed). Le sonde LN34 sono marcate con il colorante fluorescente FAM all'estremità 5' e il quencher Black Hole (BHQ1) all'estremità 3'. La sonda βA è marcata con il colorante fluorescente HEX all'estremità 5' e il quencher Black Hole (BHQ1) all'estremità 3'. Le basi modificate con nucleotidi bloccati sono indicate da un segno più che precede la base nella sequenza. Clicca qui per scaricare questa tabella.
Tabella 2: Impostazione del saggio per i saggi LN34lys, Actin3 e LN34M. I nomi, le sequenze e le concentrazioni dei primer e delle sonde sono riportati nella Tabella 1. LN34_F1 corrisponde a ACGCTTAACAACCAGATCAAAGAA7. Clicca qui per scaricare questa tabella.
Tabella 3: Parametri di ciclo per gli strumenti ABI. IMPORTANTE: Assicurati di eseguire in modalità STANDARD , non in modalità VELOCE . ROX deve essere selezionato come colorante di riferimento passivo. Clicca qui per scaricare questa tabella.
Tabella 4: Algoritmo per l'interpretazione dei risultati della RT-PCR in tempo reale LN34 per i formati singleplex (in alto, tabella blu) e multiplex (in basso, tabella rossa). Un risultato LN34 positivo deve essere considerato positivo, anche se il risultato βA è negativo o non conclusivo. Se l'amplicone LN34 non viene rilevato, il βA Ct deve essere ≤ il valore di cutoff Ct elencato per essere considerato negativo. I valori di βA Ct indicano la qualità del campione da testare e identificano una possibile inibizione. Una bassa concentrazione nel campione clinico originale può influire sulle curve di crescita βA, portando a un'amplificazione non distinguibile. Ulteriori fattori che contribuiscono alla mancata rilevazione della β-actina includono una scarsa estrazione dell'RNA a causa della perdita di RNA o del carryover di inibitori della PCR, impostazione e tecnica del test errate, tipo o qualità del campione insoddisfacenti e malfunzionamento dei reagenti o delle apparecchiature. Clicca qui per scaricare questa tabella.
Tabella 5: Azioni e interpretazioni dei risultati comuni per i controlli del saggio LN34. Tutti e tre i controlli (RNA di controllo positivo della rabbia, controllo di estrazione negativo della rabbia e nessun controllo del modello devono produrre i risultati attesi affinché una corsa possa essere superata. Il mancato controllo del modello o l'assenza di controllo del modello possono indicare un errore di pipettaggio, reagente o guasto dell'apparecchiatura. L'intera corsa, compresi tutti i campioni di RNA testati, deve essere ripetuta. Il mancato controllo dell'estrazione può indicare un problema durante l'estrazione, come guasto del reagente, pipettaggio errato o contaminazione incrociata. L'estrazione di tutti i campioni deve essere ripetuta. Il fallimento dei controlli dovrebbe essere raro per il personale di laboratorio esperto. Clicca qui per scaricare questa tabella.
Discussione
Una serie di test LN34 riuscita richiede un controllo positivo, un controllo di estrazione e nessuna reazione di controllo del modello viene eseguita come previsto in ogni sessione di analisi, altrimenti la corsa deve essere invalidata e ripetuta. Tutte e tre le reazioni di replica del controllo positivo LN34 devono superare la soglia entro l'intervallo specificato, oppure l'esecuzione deve essere ripetuta. L'RNA di controllo positivo descritto nelle precedenti pubblicazioni 7,8 non si amplificherà nel saggio βA. Le reazioni di controllo senza stampo non devono presentare curve di amplificazione che superano la linea di soglia per il saggio LN34 o βA. Il controllo dell'estrazione non deve mostrare amplificazione per LN34. Se si osserva un'amplificazione inaspettata nel controllo NTC o nell'estrazione, può indicare una contaminazione e invalidare l'esecuzione e la ripetizione del test per tutti i campioni (vedere la Tabella 5). Gli utenti possono prendere in considerazione l'aggiunta di ulteriori controlli, tra cui un controllo senza processo o un controllo senza estrazione del campione, per monitorare la contaminazione da βA ospite dei reagenti di estrazione.
Poiché la mortalità per rabbia si avvicina al 100%, si raccomanda di indagare ulteriormente su qualsiasi amplificazione debole o anormale, anche se non produce un valore Ct. Le reazioni negative o NTC non dovrebbero mostrare alcuna amplificazione e la fluorescenza dovrebbe apparire come una linea piatta parallela alla fluorescenza ROX nella vista multicomponente. Le osservazioni delle curve, specialmente in più repliche, possono indicare una contaminazione incrociata o un risultato debole positivo. Tutte le repliche per un campione positivo devono essere amplificate per un risultato positivo valido. Se in uno dei due test si amplifica solo un sottogruppo di repliche, il campione deve essere riesaminato. Inoltre, qualsiasi campione che produca risultati altamente variabili (differenze di valore Ct > ±1,5 tra le repliche) deve essere considerato non valido e il campione deve essere riesaminato. Se il problema persiste, il campione deve essere estratto nuovamente.
Si prevede che un campione positivo alla rabbia estratto dal tessuto del tronco encefalico e del cervelletto correttamente raccolto e conservato abbia un valore Ct inferiore a 35 cicli per il test LN34. Tutti i campioni non conclusivi devono essere nuovamente analizzati mediante RT-PCR in tempo reale LN34. Se il campione non risulta conclusivo dopo aver ripetuto il test e tutti i controlli sono stati eseguiti come previsto, si raccomanda la riestrazione dell'RNA. I campioni con basso RNA virale (LN34 Ct > 35) possono indicare potenziali problemi come contaminazione, bassa carica virale, inibizione della PCR o mancata estrazione. Raccogli pezzi freschi del cervello dal tessuto originale, esegui l'estrazione dell'RNA e riesamina il campione. Allo stesso modo, valori di Ct > 33 (singleplex), 37 (LN34M) o nessuna amplificazione nel test βA possono indicare una mancata estrazione dell'RNA. Ripetere l'estrazione per tali campioni, quindi ripetere il test sia per LN34 che per l'actina. Se un campione produce nuovamente un risultato inconcludente dopo test ripetuti, utilizzare un metodo secondario come il test DFA (chiamato anche FAT), DRIT o l'isolamento del virus. Se si osservano risultati discordanti o inconcludenti, consultare un laboratorio di riferimento per la rabbia per i test di conferma.
Se in un'estrazione di RNA sono presenti inibitori, i test PCR possono produrre un risultato falso negativo. Se si sospetta un'inibizione o si nota un'inibizione delle reazioni di controllo βA (come il valore Ct > 33 o il valore Ct > 37) per un particolare campione, l'RNA estratto deve essere testato a 2 o più diluizioni (ad esempio, 1:10 e 1:100 in acqua priva di nucleasi) per diluire eventuali potenziali inibitori della PCR. Per i campioni difficili, l'input di RNA può essere aumentato a 8,5 μl nella reazione RT-PCR senza aggiungere acqua. Ciò può rivelare un aumento dell'inibizione (valore Ct successivo rispetto a 2 μL di RNA in ingresso) o un basso livello di RNA nel campione originale (valore Ct precedente quando si utilizzano 8,5 μL rispetto a 2 μL di RNA in ingresso).
Il test LN34 non distingue tra i lyssavirus né determina le varianti del virus della rabbia. L'amplicone del saggio LN34 può essere sequenziato per la tipizzazione di varianti del virus della rabbia a bassa risoluzione o per l'identificazione della specie11 di lyssavirus.
Divulgazioni
Nessuno da rivelare
Riconoscimenti
Riconosciamo gli sforzi e la collaborazione di molti laboratori di test diagnostici per la rabbia che hanno contribuito all'implementazione, alla convalida e all'ottimizzazione del test LN34 attraverso la condivisione di dati aperti e il feedback. L'uso di nomi commerciali e fonti commerciali è solo per l'identificazione e non implica l'approvazione da parte dei Centers for Disease Control and Prevention, del Dipartimento della Salute e dei Servizi Umani degli Stati Uniti o delle istituzioni affiliate agli autori. Le conclusioni, i risultati e le opinioni espresse dagli autori non riflettono necessariamente la posizione ufficiale del Dipartimento della Salute e dei Servizi Umani degli Stati Uniti, dei Centri per il Controllo e la Prevenzione delle Malattie o delle istituzioni affiliate agli autori.
Materiali
| Name | Company | Catalog Number | Comments |
| 7500 Fast | Applied Biosystems | N/A | Do not substitute without validation |
| 7500 Fast Dx | Applied Biosystems | N/A | Do not substitute without validation |
| ABI ViiA 7 | Applied Biosystems | N/A | Do not substitute without validation |
| AgPath-ID One-Step RT-PCR Kit | ThermoFisher Scientific | AM1005 | Do not substitute without validation |
| Beadbug6 | Benchmark Scientific | D1036 | |
| Direct-zol RNA MiniPrep kit | Zymo Research | R2052 | |
| MagNA Lyser green beads | Roche | 3358941001 | |
| Microcentrifuge | Eppendorf | 5425 R | |
| Optical 96-well Reaction Plates | ThermoFisher Scientific | 4346907 | |
| Optical Adhesive covers | ThermoFisher Scientific | 4311971 | Alternative: caps |
| Polyester fiber-tipped applicator swabs | BD BBL Polyester Fiber Tipped Application Swab | 220690 | |
| QuantStudio 6Flex | Applied Biosystems | 4485691 | Do not substitute without validation |
| Quaternary ammonium disinfectant (1:256) | LYSOL | WBB56939 | Do not substitute without validation |
| RNase AWAY | ThermoFisher Scientific | 7002PK | |
| RNaseZap | ThermoFisher Scientific | AM9780 | |
| Single-use scalpel, a scalpel with a safety mechanism | Integra Miltex | 4-510 | |
| Sterile polyproylene microcentrifuge tubes (1.5 mL), nuclease free | Sarstedt | 72.692.405 | |
| Sterile polyproylene microcentrifuge tubes (2 mL), nuclease free | Sarstedt | 72.694.600 | |
| TRIzol Reagent | ThermoFisher Scientific | 15596026 | Do not substitute without validation |
Riferimenti
- Meechan, P. J., Potts, J. . Biosafety in Microbiological and Biomedical Laboratories. , (2020).
- . Terrestrial Manual 2023 Available from: https://www.woah.org/en/what-we-do/standards/codes-and-manuals (2023)
- . Laboratory Techniques in Rabies Available from: https://iris.who.int/handle/10665/310836 (2018)
- . Laboratory Techniques in Rabies Available from: https://iris.who.int/bitstream/handle/10665/310837/9789241515306-eng.pdf?ua=1 (2019)
- . Protocol for postmortem diagnosis of rabies in animals by direct fluorescent antibody testing: A minimum standard for rabies diagnosis in the United States Available from: https://www.cdc.gov/rabies/pdf/RabiesDFASPv2.pdf (2003)
- World Health Organization. . WHO Expert Consultation on Rabies: Third Report. , (2018).
- Gigante, C. M., et al. Multi-site evaluation of the LN34 pan-lyssavirus real-time RT-PCR assay for postmortem rabies diagnostics. PLoS One. 13 (5), e0197074 (2018).
- Wadhwa, A., et al. A Pan-Lyssavirus Taqman Real-Time RT-PCR assay for the detection of highly variable rabies virus and other lyssaviruses. PLoS Negl Trop Dis. 11 (1), e0005258 (2017).
- Gigante, C. M., Wicker, V., Wilkins, K., Seiders, M., Zhao, H., Patel, P., Orciari, L., Condori, R. E., Dettinger, L., Yager, P., Xia, D., Li, Y., et al. Optimization of pan-lyssavirus LN34 assay for streamlined rabies diagnostics by real-time RT-PCR. Journal Virological Methods. , (2024).
- Rao, A. K., et al. Use of a modified preexposure prophylaxis vaccination schedule to prevent human rabies: recommendations of the advisory committee on immunization practices-United States, 2022. Morbidity and Mortality Weekly Report. 71 (18), 619 (2022).
- Condori, R. E., et al. Using the LN34 Pan-Lyssavirus Real-Time RT-PCR assay for rabies diagnosis and rapid genetic typing from formalin-fixed human brain tissue. Viruses. 12 (1), 120 (2020).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon