Method Article
Diagnostic post-mortem de la rage chez les animaux par le test actualisé et multiplexé LN34 Real-time Reverse Transcription-Polymerase Chain Reaction
Dans cet article
Résumé
Ce protocole fait la démonstration de l’essai RT-PCR (transcription inverse en chaîne par polymérase en temps réel du pan-lyssavirus LN34), de la collecte de tissus à l’interprétation des résultats, y compris les mises à jour des séquences d’amorces et des formulations afin d’améliorer les performances des tests pour certains lyssavirus et lagomorphes non enragés. Nous démontrons également la configuration de l’essai pour un format LN34 multiplexé à puits unique (LN34M).
Résumé
La rage est une zoonose mortelle causée par la rage à Lyssavirus (RABV) et les virus à ARN à brin négatif apparentés du genre Lyssavirus (famille des Rhabdoviridae). Le test LN34 cible la région leader et le gène de la nucléoprotéine hautement conservés du génome du lyssavirus et utilise des amorces dégénérées et une sonde TaqMan contenant des nucléotides verrouillés pour détecter l’ARN dans le genre diversifié de Lyssavirus . Un résultat négatif pour la rage ne devrait être fait que si une coupe transversale complète du tronc cérébral et des trois lobes du cervelet est examinée ; Cependant, l’identification de l’ARN du lyssavirus dans n’importe quel tissu permet de diagnostiquer une infection par la rage. Les tissus sont collectés et homogénéisés dans le réactif TRIzol, qui inactive également le virus. L’extraction de l’ARN est réalisée à l’aide d’un kit d’extraction commercial basé sur une colonne de spin. Les mélanges maîtres sont préparés dans un espace propre et aliquotes dans une plaque à 96 puits avant d’ajouter l’ARN de l’échantillon. En milieu clinique, chaque échantillon est testé par RT-PCR en temps réel pour détecter la présence d’ARN du lyssavirus en trois exemplaires et individuellement pour l’ARNm de l’β de l’hôte. Des contrôles positifs et négatifs sont inclus aux étapes d’extraction et de RT-PCR en temps réel du protocole. L’analyse des données implique un ajustement manuel des seuils afin de normaliser les valeurs Ct entre les cycles d’instruments. Les résultats positifs sont déterminés par la présence d’une amplification typique dans le dosage du pan-lyssavirus (Ct ≤ 35). Les résultats négatifs sont déterminés par l’absence d’amplification typique dans le dosage du pan-lyssavirus et la détection de l’ARNm de l’β-actine de l’hôte (Ct ≤ 33). L’observation de valeurs à l’extérieur de ces plages ou l’échec des contrôles d’essai peut invalider l’exécution ou entraîner des résultats non concluants pour un échantillon. Le protocole doit être suivi de près pour garantir une sensibilité et une spécificité élevées du dosage. Les modifications procédurales peuvent affecter les performances du test et entraîner des résultats faussement positifs, faux négatifs ou ininterprétables.
Introduction
Ce protocole décrit la procédure de dépistage de la rage à l’aide du test RT-PCR (Real Time Reverse Transcription-Polymerase Chain Action) LN34 pan-lyssavirus, depuis le prélèvement de l’échantillon jusqu’à l’interprétation des résultats. La procédure sera divisée en trois sections : la collecte d’échantillons de cerveau en fonction du test LN34 (section 1), l’extraction manuelle de l’ARN sur colonne à l’aide du kit Direct-zol RNA Miniprep (Zymo Research R2051) (section 2) et la configuration du test RT-PCR en temps réel LN34 à l’aide du kit RT-PCR en une étape AgPath-ID (ThermoFisher Scientific AM1005) (section 3). L’extraction de l’ARN et la RT-PCR peuvent être effectuées à l’aide d’autres produits, mais les trousses doivent être validées avant utilisation pour s’assurer que l’ARN du lyssavirus est extrait et amplifié de manière appropriée.
La section 1 décrit la collecte des tissus cérébraux appropriés à utiliser dans le test RT-PCR en temps réel LN34. La description de l’autopsie animale, de la décapitation et de l’ablation du cerveau n’est pas incluse. Les échantillons peuvent contenir des agents infectieux. Les procédures de biosécurité détaillées dans la 6eédition de Biosécurité dans les laboratoires microbiologiques et biomédicaux 1 doivent être suivies pour atténuer les risques. Les échantillons doivent être considérés comme infectieux jusqu’à la fin de l’inactivation. L’inactivation virale et la validation des tests doivent être effectuées dans chaque laboratoire conformément aux normes de cet établissement. Les laboratoires doivent suivre les procédures standard de sécurité et de qualité déterminées par leur établissement lorsqu’ils mettent en œuvre un nouveau test de diagnostic.
D’après ce que l’on sait de la propagation du virus de la rage pendant l’infection, le tronc cérébral et le cervelet sont les meilleurs tissus pour le diagnostic de la rage, et ces tissus sont recommandés pour le dépistage de la rage par l’Organisation mondiale de la santé et l’Organisation mondiale de la santé animale 2,3,4,5. Étant donné que la propagation du virus peut être unilatérale (Figure 1), en particulier chez les animaux de grande taille, une coupe transversale complète du tronc cérébral et des trois lobes du cervelet doit être examinée pour exclure la rage. Pour les échantillons qui ne répondent pas à ces critères minimaux, le laboratoire peut rejeter l’échantillon parce qu’il est insuffisant pour l’analyse ou choisir de l’effectuer à des fins de surveillance ou d’interdiction. Si les tissus requis ne sont pas reçus, mais que le laboratoire choisit d’analyser l’échantillon, un résultat de test négatif doit être interprété comme non concluant pour la rage chez cet animal, car la présence d’ARN viral dans d’autres tissus peut être retardée, en faible abondance, intermittente ou inexistante. Le prélèvement des échantillons requis ou des tests supplémentaires sont nécessaires pour exclure la rage dans ce cas. Cependant, l’identification de l’ARN du lyssavirus dans n’importe quel tissu permet de diagnostiquer l’infection par la rage 3,6. Des exemples d’échantillons qui peuvent être analysés pour l’ARN du virus de la rage à des fins d’inclusion ou de surveillance (mais pas d’exclusion) de l’infection par la rage sont le cortex, l’hippocampe, la moelle épinière, les échantillons dégradés, la peau, la salive et la cornée. Une évaluation qualitative de l’état de chaque échantillon doit être effectuée à l’arrivée au laboratoire. La réfrigération permet de conserver un échantillon pendant au moins 72 h, mais ne doit pas être utilisée à long terme. Des cycles répétés de gel-dégel peuvent réduire la sensibilité de l’essai, et plus de cinq cycles de gel-dégel doivent être évités. Si l’état du tissu empêche l’identification fiable des structures cérébrales, l’échantillon doit être identifié comme insatisfaisant. Dans le cas d’un échantillon insatisfaisant, des tests peuvent toujours être effectués pour exclure (mais pas exclure) la rage. Les résultats positifs des tests sont signalés comme tels. Les résultats négatifs ou non concluants sur des tissus insatisfaisants doivent être signalés comme non concluants afin d’éviter toute interprétation erronée comme un diagnostic négatif.
Ce protocole a été élaboré à partir des procédures publiées7, 8 et 9 et comprend des amorces mises à jour ciblant la région leader du génome du lyssavirus et la séquence codante de la nucléoprotéine. La sonde cible une séquence courte et hautement conservée et utilise des nucléotides verrouillés pour permettre une détection à grande échelle. Le test détecte l’ARN de divers lyssavirus à des concentrations variables8. Ce protocole décrit les procédures de laboratoire pour effectuer le test PCR en temps réel LN34, mais la détection précise et sensible de l’ARN du lyssavirus dépend d’autres éléments qui ne sont pas largement couverts dans ce protocole, tels que l’entreposage des échantillons, la tenue de registres, la formation et les compétences du personnel, le suivi des résultats, l’interprétation des résultats, l’assurance de la qualité, les mesures de sécurité en laboratoire et le dépannage. Les tests basés sur la PCR sont sujets à la contamination croisée en raison de leur grande sensibilité. La contamination croisée peut être évitée en adhérant aux bonnes pratiques de laboratoire, telles que le changement fréquent de gants, la manipulation d’un échantillon à la fois, la désinfection des surfaces de travail avec des agents décontaminants efficaces entre les échantillons, et le maintien des tubes fermés et des échantillons séparés des réactifs PCR. Les réactifs et les échantillons de PCR peuvent être facilement séparés en utilisant un flux de travail unilatéral et en séparant les zones de travail de pré-amplification et de post-amplification. Par exemple, préparez des mastermixes PCR dans un endroit physiquement séparé de l’endroit où les échantillons sont manipulés. Changez souvent de gants pour éviter de contaminer les réactifs de PCR par des échantillons, des débris ou de l’ARN de contrôle positif. La plaque ou les tubes de PCR doivent être déplacés après l’ajout du mastermix vers un deuxième endroit où l’échantillon et l’ARN de contrôle peuvent être ajoutés. Il est important de noter que les produits PCR ne doivent pas être manipulés dans les zones où des échantillons ou des mélanges maîtres sont préparés.
Rien ne remplace la pratique et l’expérience lors de la réalisation de tests de diagnostic. Tous les nouveaux employés devraient être formés, et le personnel d’essai devrait être évalué pour ses compétences au moins une fois par an, conformément aux exigences du directeur de laboratoire concerné. Toute observation de résultats inhabituels ou d’échec de test doit être notée, étudiée et corrigée immédiatement. Chaque nouveau lot de réactifs doit être validé à l’aide d’échantillons dont les valeurs Ct sont connues (tels qu’un témoin positif ou un échantillon archivé). Tout l’équipement doit faire l’objet d’un entretien régulier, tel que suggéré par le fabricant, et les performances du test doivent être vérifiées après tout entretien ou réparation. Les niveaux de température doivent être surveillés sur l’équipement applicable pour s’assurer que les réfrigérateurs et les congélateurs restent dans les limites des critères établis pour une plage de température acceptable pour les réactifs utilisés dans les tests de diagnostic.
Les modifications procédurales peuvent affecter les performances du test et peuvent conduire à des résultats faussement positifs, faux négatifs ou ininterprétables. Les recommandations doivent être suivies de près pour garantir une sensibilité et une spécificité élevées du dosage. Un laboratoire qui souhaite incorporer des modifications à ce protocole doit valider et confirmer les méthodes modifiées en consultation avec les CDC.
Protocole
Des échantillons de tissu cérébral post-mortem ont été obtenus dans le cadre d’activités de surveillance ou de diagnostic de routine de la Direction des poxvirus et de la rage (CDC ; Atlanta, GA, États-Unis).
1. Prélèvement de tissu cérébral pour le diagnostic post-mortem de la rage chez les animaux par le test RT-PCR en temps réel du pan-lyssavirus LN34
REMARQUE : Les échantillons peuvent contenir des agents infectieux. Portez l’équipement de protection individuelle (EPI) approprié (gants en caoutchouc épais ou autres gants résistants aux coupures, blouse de laboratoire, tablier imperméable, masque chirurgical, bottes, manchons de protection et écran facial) et respectez les règles de sécurité requises pour l’utilisation, l’entreposage et l’élimination des échantillons. La vaccination antirabique pré-exposition, des tests sérologiques réguliers et des vaccinations de rappel (au besoin) sont exigés de toute personne avant de travailler avec des lyssavirus ou des échantillons connus ou potentiellement infectés, de les tester, de les produire ou d’effectuer des activités de recherche avec eux 2,3,4,6,10.
- Étiqueter un tube de prélèvement d’échantillon par échantillon avec une étiquette d’adhésion. Remplissez chaque tube de prélèvement d’échantillon avec 1 mL de réactif TRIzol ou un autre tampon d’homogénéisation et une portion de billes de lyser MagNA (ci-après « billes de céramique »). Pour ajouter des billes de céramique, versez délicatement du tube de perles dans le tube de prélèvement d’échantillon. Les tubes de billes de céramique contiennent généralement suffisamment de billes pour 2 à 5 échantillons, en utilisant au moins 20 billes de 1,4 mm de diamètre par échantillon.
ATTENTION : Le réactif TRIzol (ci-après « tampon d’homogénéisation ») est un produit chimique dangereux ; le contact avec des acides ou de l’eau de Javel libère des gaz toxiques ; assurer une ventilation adéquate ; Veuillez vous référer à la fiche de données de sécurité pour plus d’informations. Si les utilisateurs remplacent le réactif TRIzol ou le réactif TRI par un autre tampon d’homogénéisation, une validation supplémentaire est nécessaire. TRIzol agit comme un tampon d’homogénéisation/lyse d’échantillon, un tampon d’inactivation du lyssavirus et un tampon de stabilité de l’ARN pour ce protocole. L’utilisation d’un autre tampon d’homogénéisation nécessitera la validation de l’efficacité de l’extraction, de l’inactivation et de la stabilité dans une comparaison côte à côte contrôlée. - Nettoyez et désinfectez la surface de travail avec un désinfectant à base de composés d’ammonium quaternaire (QAC) pendant 2 minutes et disposez un tampon absorbant doublé de plastique. Ne placez que les réactifs et les fournitures du premier échantillon dans une enceinte de sécurité biologique (ESB) de classe II dotée de fonctions permettant d’évacuer les fumées dangereuses à l’extérieur de la pièce.
REMARQUE : Reportez-vous aux directives du fabricant pour connaître les limites de stockage du CAQ dilué. Assurez-vous que le tampon doublé de plastique ne bloque pas le flux d’air de l’enceinte de sécurité biologique. Si le flux d’air est perturbé, n’utilisez pas de tampon. - Prélevez du tissu représentant une coupe transversale complète du tronc cérébral et du cervelet à l’aide d’un scalpel propre à usage unique.
REMARQUE : La manipulation des tissus doit être effectuée de manière à ne pas aérosoliser les liquides ou produire des particules en suspension dans l’air. Les hottes ou les enceintes de biosécurité ne sont pas obligatoires, mais les enceintes de biosécurité ventilées sont recommandées car elles offrent une protection supplémentaire contre les odeurs, les fumées, les ectoparasites et les fragments d’os.
ATTENTION : L’utilisation d’un scalpel avec du matériel potentiellement infecté par le lyssavirus est dangereuse et les utilisateurs doivent prendre les précautions de sécurité appropriées. L’utilisation d’une pince à usage unique est recommandée.- Pour les petits animaux (comme les chauves-souris), tout le tronc cérébral et le cervelet peuvent être collectés.
- Pour les animaux plus gros, prélevez une coupe transversale complète du tronc cérébral et des tissus de chacun des trois lobes du cervelet.
- FACULTATIF : Si vous effectuez le test d’anticorps fluorescents directs (DFA), recueillez des empreintes cérébrales à ce stade. Utilisez le tissu restant après la collecte d’empreintes cérébrales pour l’AFD pour l’extraction et le test de l’ARN par le test LN34.
REMARQUE : Si TRIzol est ajouté aux échantillons, ceux-ci ne peuvent plus être utilisés pour des méthodes de détection basées sur l’antigène ou l’isolement du virus.
- Préparer les échantillons pour l’homogénéisation et l’extraction de l’ARN.
REMARQUE : L’efficacité de l’extraction de l’ARN et de l’inactivation du virus peut être affectée par l’utilisation d’une trop grande quantité de tissu. La quantité de tissu ne doit pas dépasser environ 1/10 du volume du tampon d’homogénéisation utilisé. Si plus de tissu est utilisé, augmentez la quantité de tampon d’homogénéisation en conséquence pour assurer une extraction efficace et réussie de l’ARN.- Pour les petits animaux, placez tous les tissus requis dans un tube contenant un tampon d’homogénéisation et des billes pour l’extraction. Ne pas dépasser 100 mg d’échantillon dans 1 mL de tampon d’homogénéisation ; Pour les échantillons plus grands, augmentez le volume du tampon d’homogénéisation ou utilisez plusieurs tubes pour refléter un rapport 1:10 : Tampon.
- Pour les animaux plus grands, hachez et homogénéisez soigneusement le tissu et prélevez une partie représentative dans un tube prérempli de tampon d’homogénéisation et de billes. Ne pas dépasser 100 mg d’échantillon dans 1 mL de tampon d’homogénéisation ; Pour les échantillons plus grands, augmentez le volume du tampon d’homogénéisation ou utilisez plusieurs tubes pour refléter un rapport 1:10 : Tampon.
- Option 1 (batteur à billes) : Homogénéiser les tissus à l’aide d’un batteur à billes, de 1 mL de tampon et de billes de céramique. Il peut être nécessaire d’utiliser plusieurs tubes de 2 ml ou des tubes plus grands.
- Option 1 (batteur à billes) : Nettoyez et désinfectez le poste de travail, l’équipement et les tubes d’échantillon extérieurs avec le désinfectant QAC (1:256). Laissez reposer 2 min.
- Option 1 (batteur à billes) : À l’intérieur de l’ESB, chargez un rotor de centrifugeuse avec un ou plusieurs échantillons homogénéisés. Centrifuger tous les échantillons à 10 000-16 000 × g pendant 2 min dans une microcentrifugeuse de table. Déchargez le rotor de la centrifugeuse à l’intérieur de la BSC.
- Option 1 (Batteur à perles) : Laisser reposer 2 min.
- Option 1 (batteur à billes) : Transférer 120 μL d’homogénat dans un tube prérempli de 1 mL de tampon d’homogénéisation.
ATTENTION : L’homogénéisation peut produire des aérosols et doit être effectuée dans une ESB. - Option 2 (scalpel) : Hachez finement les tissus requis à l’aide d’un scalpel à usage unique, frottez avec un écouvillon et transférez l’écouvillon dans un tube prérempli de tampon d’homogénéisation et de billes. Ne pas dépasser 100 mg d’échantillon dans 1 mL de tampon d’homogénéisation ; Pour les échantillons plus grands, augmentez le volume du tampon d’homogénéisation ou utilisez plusieurs tubes pour refléter un rapport échantillon :tampon de 1:10.
ATTENTION : L’utilisation d’un scalpel avec du matériel potentiellement infecté par le lyssavirus est dangereuse et les utilisateurs doivent prendre les précautions de sécurité appropriées.
- Recueillir les tissus restants dans le contenant d’origine ou dans un nouveau contenant vide étiqueté avec une étiquette d’adhésion. Conservez ce tissu au cas où un nouveau test ou une caractérisation supplémentaire serait nécessaire.
- Nettoyez et désinfectez le poste de travail, l’équipement et l’extérieur des tubes d’échantillon avec le désinfectant QAC 1:256. Laissez reposer 2 min.
- Répétez les étapes 1.2 à 1.5 pour tous les échantillons restants.
- Homogénéiser les échantillons à l’aide d’un mini batteur à billes pendant au moins 60 s. Inspectez visuellement les tubes. Répétez l’opération avec le batteur de perles pendant 60 secondes supplémentaires s’il reste de gros morceaux de tissu. Cette étape est facultative si le tissu est complètement homogénéisé à l’étape 1.4.2.1 ci-dessus.
REMARQUE : Il est important de s’assurer que les tissus sont bien homogénéisés. Une homogénéisation incomplète diminuera le rendement en ARN. - Laisser reposer au moins 5 min à température ambiante (RT).
- Nettoyez et désinfectez le poste de travail, l’équipement et l’extérieur des tubes d’échantillonnage avec le désinfectant QAC (1:256).
REMARQUE : L’échantillon est considéré comme non infectieux pour le moment et peut être retiré du laboratoire de lutte contre la rage. - Traiter immédiatement les échantillons dans un tampon d’homogénéisation pour l’extraction de l’ARN, les stocker à RT (20 °C à 25 °C) ou au réfrigérateur (4 °C à 8 °C) pendant plusieurs jours, ou les stocker à -16 °C ou moins pour un stockage à long terme.
2. Protocole d’extraction d’ARN à l’aide du kit RNA MiniPrep
- Configurez l’espace de travail sous le BSC.
- Nettoyez la surface de travail de l’ESB avec de l’éthanol à 70 % avant de commencer le travail pour enlever la poussière ou d’autres contaminants environnementaux. Effectuez une décontamination supplémentaire des surfaces avec le désinfectant QAC (1:256), RNase AWAY ou RNaseZap (selon les recommandations du fabricant).
- Disposez un tampon absorbant doublé de plastique et placez les réactifs, les fournitures et l’échantillon dans l’ESB.
REMARQUE : Assurez-vous que le tampon doublé de plastique ne bloque pas le flux d’air du BSC. Si le flux d’air est perturbé, n’utilisez pas de tampon. - Disposez tous les tubes de collecte dans un rack propre pour les tubes de microcentrifugation. Préremplissez un tube de microcentrifugation de 1,5 mL avec 300 μL d’éthanol à 100 % pour chaque échantillon de cerveau de non-chauve-souris. Pour les échantillons contenant peu de tissu (échantillon de cerveau de chauve-souris, échantillon non cérébral ou échantillon détérioré), préremplissez un tube de microcentrifugation de 1,5 mL avec 600 μL d’éthanol à 100 % pour chacun.
- Préparation des échantillons
- Prélever tous les échantillons préparés à la section 1 dans un support à tubes dans l’ESB. Décongelez tous les échantillons congelés juste avant l’analyse.
- Décongeler un contrôle d’extraction.
REMARQUE : Il est recommandé de choisir un échantillon sans ARN du lyssavirus ; l’échantillon doit être préalablement testé avec une plage de valeurs Ct attendue pour le dosage de la bêta-actine. Par exemple, des cellules de culture tissulaire pré-alicitées ou un cas de rage précédemment testé négatif (humain ou animal).
- Centrifuger tous les échantillons à 10 000-16 000 × g pendant 2 m dans une microcentrifugeuse de table.
- Transférez le surnageant dans un nouveau tube de microcentrifugation stérile contenant 100 % d’éthanol. Assurez-vous que le surnageant est clair, sans lipides ni tissus solides évidents. Évitez de collecter des lipides et des tissus solides.
- Pour les tissus cérébraux autres que ceux d’une chauve-souris : transférer 300 μL de surnageant.
- Pour les échantillons contenant peu de tissu (échantillon de cerveau de chauve-souris, échantillon non cérébral ou échantillon détérioré), transférez 600 μL de surnageant.
- Stockez l’homogénat restant dans un tube de microcentrifugation à vis à ≤-16 °C.
- Pipette de haut en bas 10 fois pour mélanger.
- Pour chaque échantillon, transférez 600 μL du mélange éthanol-surnageant dans une colonne de centrifugation dans un tube de collecte.
- Centrifuger jusqu’à ce que le liquide soit passé dans la colonne (1 min à 10 000-16 000 × g). Jetez le flux continu.
- Répéter l’opération s’il y a plus de 600 μL de mélange tampon d’homogénéisation de l’éthanol pour un échantillon.
- Transférez chaque colonne dans un nouveau tube de collecte.
- Ajouter 400 μL de tampon de prélavage de l’ARN dans chaque colonne et centrifuger à 10 000-16 000 × g pendant 30 s.
- Jetez le flux continu et retournez chaque colonne dans le même tube de collecte.
- Répétez les étapes 2.10-2.11.
- Ajouter 700 μL de tampon de lavage d’ARN dans chaque colonne et centrifuger à 10 000-16 000 × g pendant 2 min. Assurez-vous que le tampon de lavage a complètement traversé chaque colonne.
- Transférez soigneusement chaque colonne dans un tube sans RNase.
- Jetez le flux et le tube de collecte de la version 2.13.
- Ajoutez 50 μL d’eau exempte de DNase/RNase directement dans la matrice de la colonne pour éluer l’ARN.
REMARQUE : Ne touchez pas la matrice de colonne avec la pointe de la pipette. - Incuber pendant 30 s à RT, puis centrifuger à 10 000-16 000 × g pendant 1 min.
- Transférez soigneusement l’ARN dans un nouveau tube de microcentrifugation à accession à vis à fond plat. Déplacez l’ARN extrait dans la glace pour un test immédiat. Conserver à long terme à -70 °C ou moins.
REMARQUE : L’entreposage à des températures plus chaudes ou des gels-dégels répétés peuvent entraîner une dégradation de l’ARN et affecter les résultats du diagnostic.
3. Protocole pour le test RT-PCR en temps réel du pan-lyssavirus LN34
- Préparez les réactifs.
- Contrôle positif artificiel
- Si un ARN de contrôle positif artificiel est produit par le CDC8, suivez les instructions sur l’emballage pour le stockage, la reconstitution et l’aliquotage. Sautez cette étape si des aliquotes à usage unique d’ARN de contrôle positif sont déjà en main.
REMARQUE : L’ARN témoin positif aux concentrations de travail doit être manipulé dans la zone d’ajout de la matrice et non dans la même zone que la préparation du mastermix. L’ARN témoin positif doit produire une valeur de seuil de cycle (Ct) dans la plage attendue déterminée pour un lot donné. Entre les cycles, la valeur LN34 Ct pour l’ARN témoin positif ne doit pas différer de plus de ±1,5 Ct valeurs Ct. - Décongeler une aliquote à usage unique d'≤ stockage à -70 °C juste avant de l’utiliser sur de la glace ou un bloc de glace. Ne pas congeler-décongeler et jeter les aliquotes conservées pendant une période prolongée à des températures réfrigérées.
REMARQUE : Un témoin positif doit être analysé en trois exemplaires dans le dosage du LN34 ; Le contrôle positif artificiel8 ne s’amplifiera pas dans le dosage de la bêta-actine.
- Si un ARN de contrôle positif artificiel est produit par le CDC8, suivez les instructions sur l’emballage pour le stockage, la reconstitution et l’aliquotage. Sautez cette étape si des aliquotes à usage unique d’ARN de contrôle positif sont déjà en main.
- Contrôle de l’extraction et échantillons : Placez les échantillons fraîchement extraits sur de la glace (ou un bloc de glace) ou décongelez les échantillons de ≤ stockage à -70 °C sur de la glace (ou un bloc de glace) immédiatement avant utilisation.
REMARQUE : L’ARN doit être décongelé et traité dans une zone désignée pour l’ajout d’échantillons ou de matrices qui est distincte des zones utilisées pour la préparation du mastermix ou la manipulation de produits PCR ou de grandes quantités de matériel viral (p. ex. génération de témoins positifs, propagation virale)
- Contrôle positif artificiel
- Préparez les réactifs de mastermix dans la zone de préparation du mastermix.
- Préparation Mastermix du test RT-PCR monoplexe LN34
REMARQUE : Les utilisateurs peuvent tester des échantillons en format singleplex (étape 3.2.1) ou multiplex (étape 3.2.2). Il n’est pas nécessaire d’effectuer à la fois les versions 3.2.1 et 3.3.2. La préparation du Mastermix, l’aliquotage de l’amorce et de la sonde, ainsi que l’absence de réactif de contrôle de la matrice doivent être décongelés et manipulés dans une zone propre séparée du traitement des échantillons, de la nécropsie, de la PCR et d’autres zones où le matériel viral est manipulé. Cela peut se faire par le biais de pièces séparées ou d’un système d’armoire avec un flux d’échantillon unilatéral.- Produire des mélanges d’amorces et de sondes aux concentrations de travail indiquées dans les tableaux 1 et 2. Sautez cette étape si les aliquotes de dilution de travail des amorces et des sondes sont déjà en main.
- Aliquote des amorces et des sondes dans des supports de stockage de 1,5 mL et des supports de travail de 50 μL et stockables à ≤ -16 °C dans l’obscurité. Sautez cette étape si les aliquotes de dilution de travail des amorces et des sondes sont déjà en main.
REMARQUE : Il est recommandé d’effectuer l’étape 3.2.1.2. - Décongélation du tampon RT-PCR en une étape, pas de contrôle de modèle, amorces et sondes à partir d'≤ stockage à -16 °C sur de la glace ou un bloc de glace dans la zone de préparation du mastermix.
REMARQUE : Utilisez les réactifs jusqu’à la date d’expiration ou jusqu’à la défaillance des performances, selon la première éventualité. - Vortex et centrifugez brièvement tous les tampons, amorces et sondes avant utilisation.
- Conservez l’enzyme RT-PCR en une étape sur de la glace ou dans un bloc de glace jusqu’à utilisation.
- Échantillons d’ARN
REMARQUE : Utilisez de l’ARN fraîchement extrait dans la mesure du possible, car les gels-dégels peuvent affecter les performances - Conservez les échantillons d’ARN sur de la glace ou un bloc de glace jusqu’à utilisation.
- Décongelez tous les échantillons d’ARN congelés sur de la glace ou un bloc de glace.
- Étiqueter un tube de microcentrifugation par test (LN34 et βA).
- Déterminez le nombre de réactions (N) à mettre en place par test.
- Calculez le nombre de réactions pour le test LN34 en multipliant le nombre d’échantillons par 3 et en ajoutant 6 pour les puits de réaction de contrôle, plus 10 % de réactions supplémentaires pour tenir compte du volume perdu pendant le pipetage. (par exemple, pour 10 échantillons : (10 x 3) + 6 = 36 réactions ; réactions excédentaires : (36 x 0,1) + 36 = 3,6 + 36 = 39,6 réactions totales, ou 40 réactions arrondies à l’unité supérieure)
REMARQUE : Pour les essais cliniques, il est suggéré d’analyser tous les échantillons en trois exemplaires pour le LN34. À des fins de surveillance, chaque échantillon peut être analysé en double. Il est recommandé d’utiliser le triplicate lors de l’intégration initiale du test afin d’assurer une faible variabilité entre les répétitions et une bonne technique. - Calculez le nombre de réactions pour le test βA en additionnant le nombre d’échantillons plus 4 puits de réaction de contrôle plus 10 % de réactions supplémentaires pour tenir compte du volume perdu pendant le pipetage.
- Déterminer le volume de chaque réactif pour les mélanges maîtres LN34 et βA à l’aide du tableau 2.
- Désigner les puits pour chaque échantillon à analyser en trois exemplaires dans l’essai LN34 et en un seul pour l’essai βA à l’aide d’une carte de plaque à 96 puits.
- Distribuer 23 μL de mélange principal de dosage LN34 dans chaque puits LN34 assigné après un bref vortex et une rotation pendant 30 s à l’aide d’une microcentrifugeuse de table pour recueillir le liquide au fond du tube. Évitez d’introduire des bulles.
- Distribuez 23 μL de réactifs de dosage βA dans chaque puits attribué marqué au βA, après avoir brièvement tourbillonné et essoré pendant 30 s dans une microcentrifugeuse de table pour recueillir le liquide au fond.
- Configurez les réactions sans contrôle de matrice (NTC) en pipetant 2 μL d’eau de qualité PCR dans chaque puits NTC.
- Couvrir les puits et la plaque de transfert vers la zone d’ajout du modèle.
- Vortex et centrifugez brièvement les tubes contenant les échantillons d’ARN.
- Pipette de 2 μL de l’ARN extrait du premier échantillon dans chaque puits étiqueté pour cet échantillon. Évitez d’introduire des bulles.
- Assurez-vous que l’ARN a été aspiré dans la pipette par visualisation.
- Pipette sur le côté du puits pour s’assurer que l’échantillon est ajouté au bon puits.
- Évitez autant que possible d’agiter les pointes de pipette contenant de l’ARN au-dessus des puits ouverts.
- Répétez l’étape 3.2.10 pour les échantillons restants et l’ARN témoin positif.
- Placez le couvercle adhésif optique sur les puits après avoir ajouté tous les échantillons et les témoins. Veillez à couvrir tous les puits et à sceller complètement.
- Centrifuger à 500 × g pendant 1 min à RT dans une centrifugeuse de table ou utiliser une essoreuse à assiette de type essoreuse à salade.
- Placez la plaque scellée dans un instrument de PCR en temps réel calibré pour les colorants rapporteurs FAM et VIC/HEX et réglez-la sur les paramètres de cyclage indiqués dans le tableau 3.
- Préparez des mélanges maîtres pour le dosage LN34 Multiplexed (LN34M).
- Étiqueter un tube de microcentrifugation LN34M conformément au tableau 2.
- Déterminez le nombre de réactions (N) à mettre en place par test.
- Calculez le nombre de réactions pour le test LN34M en multipliant le nombre d’échantillons par 3 et ajoutez 6 pour les puits de réaction de contrôle plus 10 % de réactions supplémentaires pour tenir compte du volume perdu lors du pipetage. (p. ex., pour 10 échantillons : (10 x 3) + 6 = 36 réactions ; réactions en excès : (36 x 0,1) + 36 = 3,6 + 36 = 39,6 réactions au total, ou 40 réactions arrondies à la hausse)
- Choisissez le format 25 μL ou 12,5 μL. Déterminez le volume de chaque réactif pour le mélange maître LN34M à l’aide du Tableau 2.
- Désigner des puits pour chaque échantillon à analyser en trois exemplaires dans l’analyse LN34M à l’aide d’une carte de plaque de 96 puits.
- Distribuez les réactifs pour le test LN34M dans les puits. Agiter brièvement les tubes au tourbillon et les faire pivoter pour recueillir le liquide au fond avant de distribuer 23 μL (pour une réaction de 25 μL) ou 10,5 μL (pour une réaction de 12,5 μL) de mélange maître dans chaque puits assigné. Évitez d’introduire des bulles.
- Configurez les réactions NTC en pipetant 2 μL d’eau de qualité PCR dans chaque puits NTC.
- Couvrir les puits et la plaque de transfert vers la zone d’ajout du modèle.
- Manipulez brièvement et faites tourner les tubes contenant les échantillons d’ARN pour recueillir le liquide au fond.
- Pipette de 2 μL de l’ARN extrait du premier échantillon dans chaque puits étiqueté pour cet échantillon. Évitez d’introduire des bulles.
- Assurez-vous que l’ARN est aspiré dans la visualisation de la pipette.
- Pipette sur le côté du puits pour s’assurer que l’échantillon est ajouté au bon puits.
- Évitez autant que possible d’agiter les pointes de pipette contenant de l’ARN au-dessus des puits ouverts.
- Répétez l’étape 3.3.8 pour les échantillons restants et l’ARN témoin positif.
- Après avoir ajouté le dernier échantillon/contrôle, placez le couvercle adhésif optique sur les puits, en vous assurant que tous les puits sont couverts et complètement scellés.
- Centrifuger à 500 × g pendant 1 min à RT dans une centrifugeuse de table ou utiliser une essoreuse à assiette de type essoreuse à salade.
- Placez la plaque scellée dans un instrument de PCR en temps réel calibré pour les colorants rapporteurs FAM et VIC/HEX et réglez-la sur les paramètres de cyclage, comme indiqué dans le tableau 3. Réglez le colorant de référence passif sur ROX et exécutez-le en mode standard (ne l’exécutez pas en mode rapide)
REMARQUE : Ce réglage est spécifique aux instruments mentionnés dans ce protocole et nécessite l’utilisation d’un réactif RT-PCR en une étape contenant ROX comme colorant passif. D’autres instruments nécessitent des approches différentes pour déterminer les paramètres de fonctionnement optimaux. Assurez l’entretien normal de l’instrument selon le fabricant pour de meilleures performances.
- Préparation Mastermix du test RT-PCR monoplexe LN34
4. Interprétation des résultats
- Définissez une référence automatique et des calculs de seuil manuels à l’aide d’une valeur de 0,2 pour LN34/FAM et de 0,05 pour βA/HEX/VIC.
REMARQUE : Ce réglage est spécifique aux instruments mentionnés dans ce protocole et nécessite l’utilisation d’un réactif RT-PCR en une étape contenant ROX comme colorant passif. D’autres instruments nécessitent des approches différentes pour calculer les valeurs de référence et les valeurs seuils. - Déterminez le résultat du diagnostic à l’aide des instructions du Tableau 4 si toutes les commandes ont fonctionné comme prévu (Tableau 5).
- Confirmez toutes les valeurs Ct ou Cq en consultant les graphiques d’amplification.
- Examinez tout résultat inhabituel comme recommandé.
5. Conservation et stockage des échantillons
- Conservez tous les échantillons congelés à -16 °C ou moins jusqu’à ce que les analyses soient terminées et que les résultats soient communiqués. Conservez les tissus originaux pour confirmer les résultats ou identifier l’animal hôte à l’espèce en cas de résultats de test inhabituels.
- Utiliser des identificateurs d’échantillon uniques ; Étiquetez tous les tubes, rapports et documents avec des identificateurs d’échantillon uniques complets.
- Conserver les échantillons intermédiaires (à court terme) au cas où des essais répétés seraient nécessaires.
- Conserver au besoin des échantillons positifs représentatifs pour les utiliser comme témoins, pour le typage épidémiologique et à d’autres fins.
- Stocker l’ARN à ≤-70 °C pour un stockage à long terme.
Résultats
La figure 2 présente des images représentatives d’un test LN34 réussi sur un instrument de PCR en temps réel ABI ViiA7. L’affichage des résultats tracés sur une échelle logarithmique permet de visualiser facilement la valeur Ct, le point où la courbe franchit la ligne de seuil (Figure 2A,C). Lorsqu’elle est tracée sur une échelle linéaire, l’amplification réussie apparaîtra sous la forme d’une courbe sigmoïdale (ou en forme de « S ») (Figure 2B, D), tandis que les résultats négatifs devraient apparaître sous la forme d’une ligne droite et plate. Il est recommandé d’afficher les résultats à la fois à l’échelle linéaire et à l’échelle logarithmique afin d’identifier les anomalies ou les erreurs possibles. Des résultats positifs et négatifs typiques dans la vue du tracé à plusieurs composants peuvent être observés dans les figures 2E, F, respectivement, où le niveau de fluorescence du colorant marquant la sonde (FAM pour LN34, VIC/HEX pour βA) peut être observé par rapport au colorant passif dans le tampon de réaction (ROX).
La figure 3 présente des exemples de résultats anormaux. Les comparaisons entre les graphiques d’exécutions réussies (Figure 2) et les graphiques anormaux (Figure 3) peuvent être utilisées pour isoler les exécutions atypiques et les problèmes d’instruments. La figure 3A montre un signal franchissant le seuil, produisant une valeur Ct pour LN34, mais la courbe d’amplification est très atypique, augmentant linéairement. Le graphique à composantes multiples (figure 3B) montre également une ligne ondulée qui n’est pas typique d’un échantillon positif. Cet exemple souligne l’importance de visualiser les graphiques d’amplification et de ne pas se contenter de copier les valeurs Ct. Assurez-vous toujours que les courbes d’amplification sont normales pour tous les échantillons. Il est également conseillé de visualiser le graphique à plusieurs composants pour s’assurer qu’il n’y a pas d’irrégularités. À l’occasion, des signaux de base désordonnés peuvent générer des valeurs Ct dans les cas où aucune amplification n’a eu lieu. Si les signaux d’amplification semblent linéaires, il est suggéré d’ajuster la ligne de base pour voir si la courbe disparaît. Dans le cas d’un signal inhabituel, l’ensemble du parcours doit être répété. Il est recommandé de nettoyer et d’exécuter une plaque d’arrière-plan sur votre instrument de PCR en temps réel si les problèmes persistent. S’ils sont disponibles, les produits PCR peuvent être exécutés sur un gel d’agarose et/ou séquencés pour résoudre tout résultat inhabituel. Il n’est pas recommandé d’utiliser les résultats de l’électrophorèse sur gel ou du séquençage pour déterminer les résultats diagnostiques.
Des études antérieures ont montré une faible variabilité entre les répétitions, l’analyse, l’opérateur et le laboratoire pour l’essai LN347. Si une variabilité élevée (différence de >±1,5 Cat) entre les répétitions d’un même échantillon est observée, cet ARN doit être testé à nouveau. Une grande variabilité peut être causée par des problèmes avec les pipettes, les pratiques de laboratoire, les erreurs de pipetage ou les machines de PCR en temps réel. L’observation répétée d’une variabilité élevée entre plusieurs échantillons ou d’une série d’essais peut indiquer des problèmes systémiques. Les échantillons à faible ARN, approchant du seuil de dosage pour un échantillon positif (Ct 35), peuvent présenter une variabilité plus élevée des valeurs de Ct entre les répétitions. Une consultation avec le CDC et un dépannage peuvent être nécessaires pour s’attaquer à la cause de la variabilité persistante, des résultats incohérents ou de l’échec du test.
La grande sensibilité des tests basés sur la PCR les rend intrinsèquement sensibles à la contamination. Le respect strict des bonnes pratiques de laboratoire est le meilleur moyen d’atténuer la contamination croisée. Il est important de savoir identifier les contaminations potentielles. Une contamination par le réactif doit être suspectée si aucun contrôle de matrice et des puits d’échantillon négatifs suspects dans un cycle d’essai produisent tous des valeurs Ct similaires. Répétez les tests avec de nouvelles aliquotes de réactifs PCR (tampon, eau, amorces et enzyme) et le même ARN. Si tous les échantillons et le contrôle d’extraction produisent des valeurs CT similaires mais que le NTC est négatif, la contamination des réactifs d’extraction doit être étudiée et l’extraction doit être répétée à l’aide de nouveaux réactifs. Il est recommandé de faire de petites aliquotes de réactifs afin de réduire le risque de contamination et d’éviter la possibilité de jeter de gros volumes de réactifs coûteux. La contamination croisée des échantillons est plus difficile à identifier. Si l’on soupçonne une contamination de l’échantillon, répétez le prélèvement de l’échantillon en commençant par les tissus d’origine. Dans certains cas, le séquençage de l’ARN viral peut confirmer la contamination, en particulier lorsque l’ARN contaminant est très différent de la variante virale attendue (comme un virus témoin utilisé en laboratoire). Le séquençage de deux échantillons traités en même temps peut déterminer si les séquences virales sont identiques, mais peut être peu informatif si l’on s’attend à ce que les séquences soient très similaires (par exemple, le même variant collecté dans le même comté). Si l’on soupçonne une contamination de l’échantillon par l’ARN de contrôle positif, on peut exécuter les amplicons de test LN34 sur un gel d’agarose pour différencier l’ARN du lyssavirus (165 pb) de l’ARN de contrôle positif (99 pb). Séquence de la matrice utilisée pour générer l’ARN de contrôle positif fourni par CDC8.
Pour d’autres agents pathogènes, les laboratoires peuvent être utilisés pour régler le seuil manuellement afin de se débarrasser du « bruit » tel que la faible amplification montrée en cyan sur la figure 4. Cette pratique n’est PAS recommandée pour le diagnostic de la rage car elle peut conduire à des résultats faussement négatifs avec des conséquences désastreuses puisque la rage est presque mortelle à 100 %. Ne modifiez PAS manuellement le seuil pour produire des résultats négatifs pour des échantillons d’amplification faibles ou tardifs. Ces échantillons doivent être extraits de nouveau et/ou analysés à nouveau pour exclure la rage.
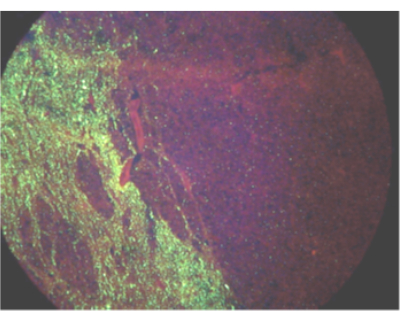
Figure 1 : Champ de vision montrant la propagation unilatérale de l’antigène du virus de la rage chez un âne infecté par un test d’anticorps fluorescent direct. Veuillez cliquer ici pour voir une version agrandie de cette figure.
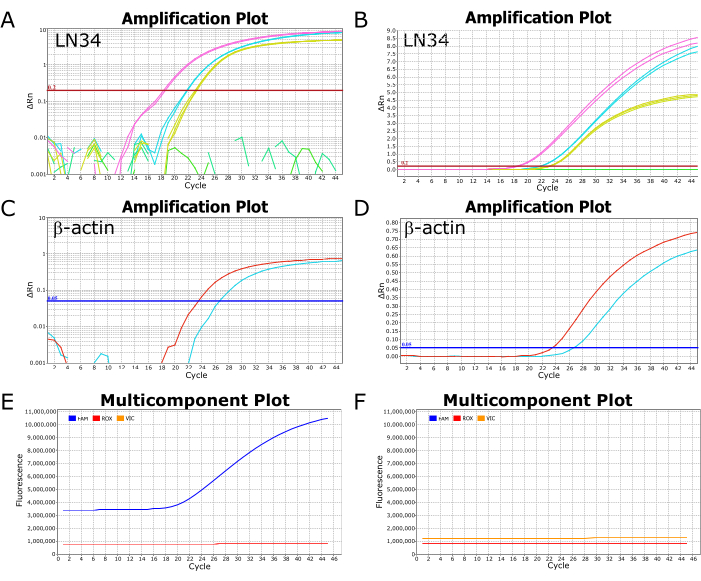
Figure 2 : Graphiques d’amplification et à composantes multiples d’un essai LN34 réussi. (A-D) Les données de résultat sont tracées sur une échelle logarithmique (A,C) et une échelle linéaire (B,D) pour les essais LN34 et βA. Les panneaux A et B représentent les résultats du LN34 à partir de deux échantillons (en rose et en cyan) par rapport au témoin positif (en jaune). Dans le panneau B, il y a une ligne verte plate qui représente un échantillon négatif supplémentaire dans le cycle. Dans A, la ou les lignes vertes ne montrent aucune amplification et sont représentées comme des segments brisés. Le seuil pour le dosage du LN34 a été fixé manuellement à 0,2 et est indiqué par la ligne horizontale rouge. (C, D) Résultats de l’analyse βA pour deux échantillons (rouge et cyan). Le seuil pour le dosage βA a été fixé manuellement à 0,05. (E, F) Les graphiques à plusieurs composants représentent la fluorescence (RFU) à chaque cycle pour FAM (LN34), VIC (βA) et ROX (colorant passif présent dans le tampon AgPath-ID). Les niveaux de ROX doivent rester stables sur tous les cycles. Un échantillon positif typique est présenté dans le panneau E ; La fluorescence de FAM augmente sous la forme d’une courbe sigmoïdale à partir du cycle 18 pour cet échantillon. Un échantillon négatif typique est montré dans le panneau F, où le niveau FAM reste parallèle au niveau ROX sur tous les cycles. Les données proviennent d’un instrument de PCR en temps réel ABI ViiA7. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 3 : Images représentatives du signal rare et atypique observé dans les analyses de test LN34 sur un instrument de PCR en temps réel ViiA7. (A-F) Graphiques d’amplification (A, C, E) et à composantes multiples (B, D, F) produits en raison de la contamination des puits. L’augmentation linéaire (A,C) et les fluctuations ondulatoires (B,D) de la fluorescence FAM ne représentent pas une véritable amplification basée sur la forme des courbes et l’amplitude du changement de fluorescence. Les panneaux A à D représentent probablement des échantillons négatifs même si une valeur Ct a été produite pour la répétition montrée dans les panneaux A et B. Les panneaux E et F montrent un signal ondulé étrange qui est plus facilement visible dans le graphique à plusieurs composantes. Ce type de signal doit être examiné et peut indiquer des problèmes d’instrument, même si toutes les commandes ont fonctionné comme prévu dans cette exécution. Veuillez cliquer ici pour voir une version agrandie de cette figure.
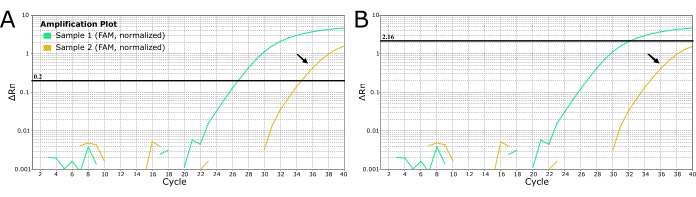
Figure 4 : Courbes RT-PCR en temps réel du LN34 à partir de 2 échantillons suspects de rage montrant deux méthodes de réglage des valeurs seuils. (A) Le seuil LN34 a été fixé à 0,2 (recommandé pour tous les cycles). (B) Détermination manuelle d’un seuil différent pour chaque passage afin de masquer le signal déterminé comme étant du « bruit » (signal d’amplification tardive). La méthode utilisée dans le panneau B n’est PAS recommandée pour la rage en raison des conséquences graves de l’absence d’un résultat vraiment positif. Une amplification tardive pourrait indiquer un échantillon positif faible, une inhibition de la PCR ou un échec de l’extraction dans un cas positif. Cela pourrait également indiquer une contamination croisée. L’échantillon doré (indiqué par des flèches noires) produit une valeur Ct à la limite du test et ne doit pas être considéré comme négatif. Les échantillons avec amplification tardive doivent être réextraits et testés à nouveau. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Tableau 1 : Séquences et concentrations d’amorces et de sondes utilisées dans les tests RT-PCR en temps réel LN34lys (singleplex LN34), LN34M (LN34 et βA multiplexés). Les sondes LN34 sont marquées avec le colorant fluorescent FAM à l’extrémité 5 et le quencher Black Hole (BHQ1) à l’extrémité 3. La sonde βA est marquée avec le colorant fluorescent HEX à l’extrémité 5 et le quincher de trou noir (BHQ1) à l’extrémité 3. Les bases modifiées par des nucléotides verrouillées sont indiquées par un plus précédant la base dans la séquence. Veuillez cliquer ici pour télécharger ce tableau.
Tableau 2 : Configuration des dosages LN34lys, Actin3 et LN34M. Les noms, les séquences et les concentrations des amorces et des sondes se trouvent dans le tableau 1. LN34_F1 correspond à ACGCTTAACAACAACAGATCAAAGAA7. Veuillez cliquer ici pour télécharger ce tableau.
Tableau 3 : Paramètres de cyclage des instruments ABI. IMPORTANT : Assurez-vous de fonctionner en mode STANDARD et non en mode RAPIDE . ROX doit être sélectionné comme colorant de référence passif. Veuillez cliquer ici pour télécharger ce tableau.
Tableau 4 : Algorithme d’interprétation des résultats de RT-PCR en temps réel LN34 pour les formats singleplex (en haut, tableau bleu) et multiplex (en bas, tableau rouge). Un résultat LN34 positif doit être considéré comme positif, même si le résultat βA est négatif ou non concluant. Si l’amplicon LN34 n’est pas détecté, le Ct βA doit être ≤ la valeur seuil Ct indiquée pour être considéré comme négatif. Les valeurs Ct de βA indiquent la qualité de l’échantillon testé et identifient une éventuelle inhibition. Une faible concentration dans l’échantillon clinique d’origine peut avoir un impact sur les courbes de croissance de βA, entraînant l’absence d’amplification discernable. D’autres facteurs contributifs à l’échec de la détection de la β-actine comprennent une mauvaise extraction de l’ARN en raison de la perte d’ARN ou du transfert d’inhibiteurs de PCR, une configuration et une technique de test incorrectes, un type ou une qualité d’échantillon insatisfaisants et un dysfonctionnement des réactifs ou de l’équipement. Veuillez cliquer ici pour télécharger ce tableau.
Tableau 5 : Actions et interprétations des résultats communs pour les contrôles d’essai LN34. Les trois témoins (contrôle positif de la rage, ARN, contrôle négatif de l’extraction de la rage et aucun contrôle de matrice doivent produire les résultats attendus pour qu’une série réussisse. L’échec du témoin positif ou l’absence de contrôle de la matrice peut indiquer une erreur de pipetage, une défaillance du réactif ou de l’équipement. L’ensemble de l’analyse, y compris tous les échantillons d’ARN analysés, doit être répété. L’échec du contrôle d’extraction peut indiquer un problème pendant l’extraction, tel qu’une défaillance du réactif, un mauvais pipetage ou une contamination croisée. L’extraction de tous les échantillons doit être répétée. L’échec des contrôles devrait être rare pour le personnel de laboratoire expérimenté. Veuillez cliquer ici pour télécharger ce tableau.
Discussion
Une analyse réussie du LN34 nécessite un contrôle positif, un contrôle d’extraction, et aucune réaction de contrôle de matrice ne fonctionne comme prévu dans chaque série d’essais, sinon l’exécution doit être invalidée et répétée. Les trois réactions de réplication du témoin positif LN34 doivent franchir le seuil dans la plage spécifiée, ou l’exécution doit être répétée. L’ARN témoin positif décrit dans les publications précédentes 7,8 ne s’amplifiera pas dans le dosage βA. Les réactions sans témoin ne doivent pas présenter de courbes d’amplification qui franchissent la ligne de seuil pour le test LN34 ou βA. Le témoin d’extraction ne doit pas présenter d’amplification pour le LN34. Si une amplification inattendue est observée dans le NTC ou le témoin d’extraction, cela peut indiquer une contamination et invalider l’essai et répéter l’essai pour tous les échantillons (voir le tableau 5). Les utilisateurs peuvent envisager d’ajouter des contrôles supplémentaires, y compris un contrôle sans procédé ou un contrôle sans extraction d’échantillon, pour surveiller la contamination βA de l’hôte des réactifs d’extraction.
Alors que la mortalité de la rage approche les 100 %, il est recommandé d’enquêter davantage sur toute amplification faible ou anormale, même si elle ne produit pas de valeur Ct. Les réactions négatives ou NTC ne doivent présenter aucune amplification, et la fluorescence doit apparaître comme une ligne plate parallèle à la fluorescence ROX dans la vue multicomposante. L’observation des courbes, en particulier dans les répétitions multiples, peut indiquer une contamination croisée ou un résultat faiblement positif. Toutes les répétitions d’un échantillon positif doivent être amplifiées pour obtenir un résultat positif valide. Si seulement un sous-ensemble de répétitions s’amplifie dans l’un ou l’autre des essais, l’échantillon doit être testé à nouveau. De plus, tout échantillon produisant des résultats très variables (différences de valeurs Ct > ±1,5 entre les répétitions) doit être considéré comme invalide et l’échantillon doit être testé à nouveau. Si le problème persiste, l’échantillon doit être réextrait.
Un échantillon positif pour la rage extrait de tissus du tronc cérébral et du cervelet correctement collectés et entreposés devrait avoir une valeur Ct inférieure à 35 cycles pour le test LN34. Tous les échantillons non concluants doivent être testés à nouveau par RT-PCR en temps réel LN34. Si l’échantillon n’est pas concluant après des tests répétés et que tous les contrôles ont été effectués comme prévu, une nouvelle extraction de l’ARN est recommandée. Les échantillons à faible teneur en ARN viral (LN34 Ct > 35) peuvent indiquer des problèmes potentiels tels qu’une contamination, une faible charge virale, une inhibition de la PCR ou un échec de l’extraction. Prélevez des morceaux frais du cerveau du tissu d’origine, effectuez l’extraction de l’ARN et testez à nouveau l’échantillon. De même, des valeurs Ct > 33 (singleplex), 37 (LN34M) ou l’absence d’amplification dans le test βA peuvent indiquer un échec de l’extraction de l’ARN. Répétez l’extraction pour ces échantillons, puis répétez les tests pour le LN34 et l’actine. Si un échantillon produit à nouveau un résultat non concluant après des tests répétés, utilisez une méthode secondaire telle que le test DFA (également appelé test FAT), DRIT ou l’isolement du virus. Si des résultats discordants persistants ou des résultats non concluants sont observés, veuillez consulter un laboratoire de référence pour la rage pour un test de confirmation.
Si des inhibiteurs sont présents dans une extraction d’ARN, les tests PCR peuvent produire un résultat faussement négatif. Si l’on soupçonne une inhibition ou si l’on observe une inhibition des réactions de contrôle βA (comme une valeur Ct > 33 ou une valeur Ct > 37) pour un échantillon particulier, l’ARN extrait doit être testé à 2 dilutions ou plus (p. ex., 1:10 et 1:100 dans de l’eau exempte de nucléases) afin de diluer tout inhibiteur potentiel de la PCR. Pour les échantillons difficiles, l’apport d’ARN peut être augmenté à 8,5 μL dans la réaction RT-PCR en n’ajoutant pas d’eau. Cela peut révéler une inhibition accrue (valeur Ct plus tardive par rapport à l’ARN d’entrée de 2 μL) ou un faible niveau d’ARN dans l’échantillon d’origine (valeur Ct plus précoce lors de l’utilisation de 8,5 μL par rapport à 2 μL d’ARN d’entrée).
Le test LN34 ne fait pas la différence entre les lyssavirus et ne détermine pas les variantes du virus de la rage. L’amplicon du test LN34 peut être séquencé pour le typage à basse résolution des variants du virus de la rage ou l’identification de l’espèce11 du lyssavirus.
Déclarations de divulgation
Aucune divulgation
Remerciements
Nous reconnaissons les efforts et la collaboration de nombreux laboratoires de diagnostic de la rage qui ont contribué à la mise en œuvre, à la validation et à l’optimisation du test LN34 grâce à leur partage de données ouvertes et à leurs commentaires. L’utilisation des noms commerciaux et des sources commerciales n’est fournie qu’à des fins d’identification et n’implique pas l’approbation des Centers for Disease Control and Prevention, du ministère américain de la Santé et des Services sociaux ou des institutions affiliées aux auteurs. Les conclusions, les résultats et les opinions exprimés par les auteurs ne reflètent pas nécessairement la position officielle du ministère américain de la Santé et des Services sociaux, des Centers for Disease Control and Prevention ou des institutions affiliées aux auteurs.
matériels
| Name | Company | Catalog Number | Comments |
| 7500 Fast | Applied Biosystems | N/A | Do not substitute without validation |
| 7500 Fast Dx | Applied Biosystems | N/A | Do not substitute without validation |
| ABI ViiA 7 | Applied Biosystems | N/A | Do not substitute without validation |
| AgPath-ID One-Step RT-PCR Kit | ThermoFisher Scientific | AM1005 | Do not substitute without validation |
| Beadbug6 | Benchmark Scientific | D1036 | |
| Direct-zol RNA MiniPrep kit | Zymo Research | R2052 | |
| MagNA Lyser green beads | Roche | 3358941001 | |
| Microcentrifuge | Eppendorf | 5425 R | |
| Optical 96-well Reaction Plates | ThermoFisher Scientific | 4346907 | |
| Optical Adhesive covers | ThermoFisher Scientific | 4311971 | Alternative: caps |
| Polyester fiber-tipped applicator swabs | BD BBL Polyester Fiber Tipped Application Swab | 220690 | |
| QuantStudio 6Flex | Applied Biosystems | 4485691 | Do not substitute without validation |
| Quaternary ammonium disinfectant (1:256) | LYSOL | WBB56939 | Do not substitute without validation |
| RNase AWAY | ThermoFisher Scientific | 7002PK | |
| RNaseZap | ThermoFisher Scientific | AM9780 | |
| Single-use scalpel, a scalpel with a safety mechanism | Integra Miltex | 4-510 | |
| Sterile polyproylene microcentrifuge tubes (1.5 mL), nuclease free | Sarstedt | 72.692.405 | |
| Sterile polyproylene microcentrifuge tubes (2 mL), nuclease free | Sarstedt | 72.694.600 | |
| TRIzol Reagent | ThermoFisher Scientific | 15596026 | Do not substitute without validation |
Références
- Meechan, P. J., Potts, J. . Biosafety in Microbiological and Biomedical Laboratories. , (2020).
- . Terrestrial Manual 2023 Available from: https://www.woah.org/en/what-we-do/standards/codes-and-manuals (2023)
- . Laboratory Techniques in Rabies Available from: https://iris.who.int/handle/10665/310836 (2018)
- . Laboratory Techniques in Rabies Available from: https://iris.who.int/bitstream/handle/10665/310837/9789241515306-eng.pdf?ua=1 (2019)
- . Protocol for postmortem diagnosis of rabies in animals by direct fluorescent antibody testing: A minimum standard for rabies diagnosis in the United States Available from: https://www.cdc.gov/rabies/pdf/RabiesDFASPv2.pdf (2003)
- World Health Organization. . WHO Expert Consultation on Rabies: Third Report. , (2018).
- Gigante, C. M., et al. Multi-site evaluation of the LN34 pan-lyssavirus real-time RT-PCR assay for postmortem rabies diagnostics. PLoS One. 13 (5), e0197074 (2018).
- Wadhwa, A., et al. A Pan-Lyssavirus Taqman Real-Time RT-PCR assay for the detection of highly variable rabies virus and other lyssaviruses. PLoS Negl Trop Dis. 11 (1), e0005258 (2017).
- Gigante, C. M., Wicker, V., Wilkins, K., Seiders, M., Zhao, H., Patel, P., Orciari, L., Condori, R. E., Dettinger, L., Yager, P., Xia, D., Li, Y., et al. Optimization of pan-lyssavirus LN34 assay for streamlined rabies diagnostics by real-time RT-PCR. Journal Virological Methods. , (2024).
- Rao, A. K., et al. Use of a modified preexposure prophylaxis vaccination schedule to prevent human rabies: recommendations of the advisory committee on immunization practices-United States, 2022. Morbidity and Mortality Weekly Report. 71 (18), 619 (2022).
- Condori, R. E., et al. Using the LN34 Pan-Lyssavirus Real-Time RT-PCR assay for rabies diagnosis and rapid genetic typing from formalin-fixed human brain tissue. Viruses. 12 (1), 120 (2020).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon