Method Article
Diagnóstico post-mortem de raiva em animais pelo ensaio de reação em cadeia da polimerase com transcrição reversa em tempo real LN34 atualizado e multiplexado
Neste Artigo
Resumo
Este protocolo demonstra o ensaio de reação em cadeia da polimerase com transcrição reversa em tempo real (RT-PCR) pan-lyssavirus LN34 desde a coleta do tecido até a interpretação dos resultados, incluindo atualizações nas sequências e formulações de primers para melhorar o desempenho do ensaio para alguns lyssavirus e lagomorfos não anti-rábicos. Também demonstramos a configuração do ensaio para um formato multiplexado LN34 de poço único (LN34M).
Resumo
A raiva é uma doença zoonótica fatal causada pela raiva por Lyssavirus (RABV) e vírus de RNA de fita negativa relacionados do gênero Lyssavirus (família Rhabdoviridae). O ensaio LN34 tem como alvo a região líder altamente conservada e o gene da nucleoproteína do genoma do lyssavirus e utiliza primers degenerados e uma sonda TaqMan contendo nucleotídeos bloqueados para detectar RNA em todo o gênero Lyssavirus diverso. Um achado negativo para raiva deve ser feito apenas se um corte transversal completo do tronco cerebral e três lobos do cerebelo forem examinados; no entanto, a identificação do RNA do lyssavirus em qualquer tecido é diagnóstica de infecção por raiva. O tecido é coletado e homogeneizado no reagente TRIzol, que também inativa o vírus. A extração de RNA é realizada usando um kit de extração comercial baseado em coluna giratória. As misturas master são preparadas em um espaço limpo e aliquotadas em uma placa de 96 poços antes de adicionar a amostra de RNA. Em ambientes clínicos, cada amostra é testada por RT-PCR em tempo real para a presença de RNA de lissavírus em triplicado e isoladamente para mRNA de β actina do hospedeiro. Os controles positivos e negativos são incluídos nas etapas de extração e RT-PCR em tempo real do protocolo. A análise de dados envolve o ajuste manual dos limites para padronizar os valores de Ct nas execuções do instrumento. Os resultados positivos são determinados pela presença de amplificação típica no ensaio de pan-lyssavirus (Ct ≤ 35). Os resultados negativos são determinados pela ausência de amplificação típica no ensaio de pan-lyssavirus e detecção de mRNA de β-actina do hospedeiro (Ct ≤ 33). A observação de valores fora desses intervalos ou a falha dos controles do ensaio podem invalidar a execução ou resultar em resultados inconclusivos para uma amostra. O protocolo deve ser seguido de perto para garantir alta sensibilidade e especificidade do ensaio. As modificações processuais podem afetar o desempenho do ensaio e levar a resultados falsos positivos, falsos negativos ou não interpretáveis.
Introdução
Este protocolo descreve o procedimento para testes de diagnóstico da raiva usando o ensaio de reação em cadeia da polimerase com transcrição reversa em tempo real (RT-PCR) do pan-lyssavirus LN34 desde a coleta da amostra até a interpretação dos resultados. O procedimento será dividido em três seções: coleta de amostras cerebrais conforme relevante para o ensaio LN34 (Seção 1), extração manual de RNA baseada em coluna usando o kit Direct-zol RNA Miniprep (Zymo Research R2051) (Seção 2) e ensaio RT-PCR em tempo real LN34 configurado usando o kit AgPath-ID One-Step RT-PCR (ThermoFisher Scientific AM1005) (Seção 3). A extração de RNA e RT-PCR podem ser realizadas usando outros produtos, mas os kits devem ser validados antes do uso para garantir que o RNA do Lyssavirus seja extraído e amplificado adequadamente.
A Seção 1 descreve a coleta dos tecidos cerebrais adequados a serem usados no ensaio RT-PCR em tempo real LN34. A descrição da necropsia animal, decapitação e remoção do cérebro não estão incluídas. As amostras podem conter agentes infecciosos. Os procedimentos de biossegurança detalhados na Biossegurança em Laboratórios Microbiológicos e Biomédicos 6ª Edição1 devem ser seguidos para mitigar o risco. As amostras devem ser consideradas infecciosas até a conclusão da inativação. A inativação viral e a validação do ensaio devem ser realizadas em cada laboratório de acordo com os padrões dessa instituição. Os laboratórios devem seguir os procedimentos padrão de segurança e qualidade determinados por sua instituição ao implementar um novo teste de diagnóstico.
Com base no que se sabe sobre a disseminação do vírus da raiva durante a infecção, o tronco encefálico e o cerebelo são os melhores tecidos para o diagnóstico da raiva, e esses tecidos são recomendados para testes antirrábicos pela Organização Mundial da Saúde e pela Organização Mundial de Saúde Animal 2,3,4,5. Como a disseminação do vírus pode ser unilateral (Figura 1), especialmente em animais maiores, um corte transversal completo do tronco cerebral e três lobos do cerebelo devem ser examinados para descartar a raiva. Para amostras que não atendem a esses critérios mínimos, o laboratório pode rejeitar a amostra como insuficiente para testes ou optar por testar para fins de vigilância ou regra. Se os tecidos necessários não forem recebidos, mas o laboratório optar por testar a amostra, um resultado de teste negativo deve ser interpretado como inconclusivo para raiva naquele animal, pois a presença de RNA viral em outros tecidos pode ser retardada, de baixa abundância, intermitente ou inexistente. A coleta das amostras necessárias ou testes adicionais é necessária para descartar a raiva nesse caso. No entanto, a identificação do RNA do lyssavirus em qualquer tecido é diagnóstica de infecção por raiva 3,6. Exemplos de amostras que podem ser testadas para RNA do vírus da raiva para infecção por raiva de regra ou vigilância (mas não descartada) são córtex, hipocampo, medula espinhal, amostras degradadas, pele, saliva e córnea. Uma avaliação qualitativa da condição de cada amostra deve ser feita na chegada ao laboratório. A refrigeração pode preservar uma amostra por pelo menos 72 h, mas não deve ser usada a longo prazo. Ciclos repetidos de congelamento e descongelamento podem reduzir a sensibilidade do teste, e mais de cinco ciclos de congelamento e descongelamento devem ser evitados. Se a condição do tecido impedir a identificação segura das estruturas cerebrais, a amostra deve ser identificada como insatisfatória. No caso de uma amostra insatisfatória, o teste ainda pode ser realizado para descartar (mas não descartar) a raiva. Os resultados positivos dos testes são relatados como tal. Resultados negativos ou inconclusivos em tecido insatisfatório devem ser relatados como inconclusivos para evitar interpretações errôneas como um diagnóstico negativo.
Este protocolo foi desenvolvido a partir de procedimentos publicados 7,8,9 e inclui primers atualizados direcionados à região líder do genoma do lyssavirus e à sequência codificadora de nucleoproteínas. A sonda tem como alvo uma sequência curta e altamente conservada e usa nucleotídeos bloqueados para permitir uma ampla detecção. O ensaio detecta RNA de diversos lyssavirus em concentrações variadas8. Este protocolo demonstra os procedimentos laboratoriais para realizar o ensaio de PCR em tempo real LN34, mas a detecção precisa e sensível do RNA do lyssavirus depende de outros elementos que não são amplamente abordados neste protocolo, como armazenamento de amostras, manutenção de registros, treinamento/competência de pessoal, rastreamento de resultados, interpretação de resultados, garantia de qualidade, medidas de segurança laboratorial e solução de problemas. Os ensaios baseados em PCR são propensos à contaminação cruzada devido à sua alta sensibilidade. A contaminação cruzada pode ser evitada aderindo às boas práticas de laboratório, como trocar luvas com frequência, manusear uma amostra de cada vez, desinfetar superfícies de trabalho com agentes descontaminantes eficazes entre as amostras e manter os tubos fechados e as amostras separadas dos reagentes de PCR. Os reagentes e amostras de PCR podem ser facilmente separados empregando um fluxo de trabalho unilateral e separando as áreas de trabalho de pré-amplificação e pós-amplificação. Por exemplo, prepare mastermixes de PCR em um local fisicamente separado de onde as amostras são manuseadas. Troque as luvas com frequência para evitar a contaminação dos reagentes de PCR com amostras, detritos ou RNA de controle positivo. A placa ou tubos de PCR devem ser movidos após a adição do mastermix para um segundo local onde o RNA de amostra e controle possa ser adicionado. É importante ressaltar que os produtos de PCR não devem ser manipulados em áreas onde amostras ou mastermixes são preparados.
Não há substituto para a prática prática e a experiência ao realizar testes de diagnóstico. Todos os novos funcionários devem ser treinados e o pessoal de teste deve ser avaliado quanto à competência pelo menos uma vez por ano, seguindo os requisitos do diretor do laboratório relevante. Quaisquer observações de resultados incomuns ou falha do ensaio devem ser anotadas, investigadas e corrigidas imediatamente. Cada novo lote de reagentes deve ser validado utilizando amostras com valores de Ct conhecidos (tais como um controlo positivo ou uma amostra arquivada). Todos os equipamentos devem passar por manutenção de rotina, conforme sugerido pelo fabricante, e o desempenho do ensaio deve ser verificado após qualquer manutenção ou reparo. Os níveis de temperatura devem ser monitorados no equipamento aplicável para garantir que refrigeradores e freezers permaneçam dentro dos critérios estabelecidos para uma faixa de temperatura aceitável para reagentes usados em testes de diagnóstico.
Modificações processuais podem afetar o desempenho do ensaio e podem levar a resultados falsos positivos, falsos negativos ou não interpretáveis. As recomendações devem ser seguidas rigorosamente para garantir alta sensibilidade e especificidade do ensaio. Um laboratório que deseje incorporar modificações a este protocolo deve validar e confirmar os métodos modificados em consulta com o CDC.
Protocolo
Amostras de tecido cerebral post-mortem foram obtidas por meio de vigilância de rotina ou atividades diagnósticas do Poxvirus and Rabies Branch (CDC; Atlanta, GA, EUA).
1. Coleta de tecido cerebral para diagnóstico post-mortem de raiva em animais pelo ensaio RT-PCR em tempo real do pan-lyssavirus LN34
NOTA: As amostras podem conter agentes infecciosos. Use equipamento de proteção individual (EPI) apropriado (luvas de borracha pesadas ou outras luvas resistentes a cortes, avental de laboratório, avental impermeável, máscara cirúrgica, botas, mangas de proteção e protetor facial) e siga os regulamentos de segurança exigidos para uso, armazenamento e descarte de amostras. Vacinação antirrábica pré-exposição, testes sorológicos regulares e imunizações de reforço (conforme necessário) são necessários para qualquer pessoa antes de trabalhar, testar, produzir ou realizar atividades de pesquisa com lyssavirus ou espécimes conhecidos ou potencialmente infectados 2,3,4,6,10.
- Rotule um tubo de coleta de amostra por amostra com uma etiqueta de acesso. Encha cada sample tubo de coleta com 1 mL de reagente TRIzol ou outro tampão de homogeneização e uma porção de grânulos MagNA Lyser (doravante "grânulos de cerâmica"). Para adicionar contas de cerâmica, despeje cuidadosamente do tubo de contas no tubo de coleta de amostras. Tubos de esferas de cerâmica geralmente contêm contas suficientes para 2-5 amostras, usando pelo menos 20 contas de 1,4 mm de diâmetro por amostra.
CUIDADO: O reagente de TRIzol (doravante "tampão de homogeneização") é um produto químico perigoso; o contato com ácidos ou alvejantes libera gases tóxicos; garantir ventilação adequada; Consulte a ficha de dados de segurança para obter mais informações. Se os usuários substituírem o reagente TRIzol ou o reagente TRI por outro tampão de homogeneização, será necessária uma validação adicional. O TRIzol atua como um tampão de homogeneização/lise de amostras, tampão de inativação de lissavírus e tampão de estabilidade de RNA para este protocolo. O uso de um tampão de homogeneização alternativo exigirá a validação da eficiência de extração, inativação e estabilidade em uma comparação lado a lado controlada. - Limpe e desinfete a superfície de trabalho com desinfetante de compostos de amônio quaternário (QAC) por 2 min e coloque uma almofada absorvente revestida de plástico. Coloque apenas reagentes e suprimentos para a primeira amostra em um gabinete de segurança biológica Classe II (BSC) com recursos para exaurir vapores perigosos fora da sala.
NOTA: Consulte as orientações do fabricante para obter os limites de armazenamento de QAC diluído. Certifique-se de que a almofada revestida de plástico não bloqueie o fluxo de ar do gabinete de segurança biológica. Se o fluxo de ar for interrompido, não use uma almofada. - Colete tecido representando uma seção transversal completa do tronco cerebral e cerebelo usando um bisturi limpo de uso único.
NOTA: A manipulação de tecidos deve ser conduzida de maneira a não aerossolizar líquidos ou produzir partículas transportadas pelo ar. Capelas de exaustão ou cabines de biossegurança não são necessárias, mas cabines de biossegurança ventiladas são recomendadas, pois fornecem proteção adicional contra odores, fumaça, ectoparasitas e fragmentos ósseos.
CUIDADO: O uso de um bisturi com material potencialmente infectado com lyssavirus é perigoso e os usuários devem tomar as devidas precauções de segurança. Recomenda-se o uso de pinças descartáveis.- Para pequenos animais (como morcegos), todo o tronco cerebral e cerebelo podem ser coletados.
- Para animais maiores, colete uma seção transversal completa do tronco cerebral e tecido de cada um dos três lobos do cerebelo.
- OPCIONAL: Se estiver realizando o teste de anticorpo fluorescente direto (DFA), colete impressões cerebrais neste momento. Use o tecido restante após a coleta de impressões cerebrais para DFA para extração de RNA e teste pelo ensaio LN34.
NOTA: Se o TRIzol for adicionado às amostras, as amostras não poderão mais ser usadas para métodos de detecção baseados em antígenos ou isolamento de vírus.
- Preparar amostras para homogeneização e extração de RNA.
NOTA: A eficiência da extração de RNA e da inativação do vírus pode ser afetada pelo uso excessivo de tecido. A quantidade de tecido não deve exceder cerca de 1/10 do volume do tampão de homogeneização utilizado. Se mais tecido for usado, aumente a quantidade de tampão de homogeneização de acordo para garantir uma extração de RNA eficiente e bem-sucedida.- Para animais pequenos, coloque todos os tecidos necessários em um tubo contendo tampão de homogeneização e grânulos para extração. Não exceder 100 mg de amostra em 1 mL de tampão de homogeneização; Para amostras maiores, aumente o volume do tampão de homogeneização ou use vários tubos para refletir uma proporção de 1:10 amostra: tampão.
- Para animais maiores, pique e homogeneize bem o tecido e remova uma porção representativa para um tubo pré-preenchido com tampão de homogeneização e contas. Não exceder 100 mg de amostra em 1 mL de tampão de homogeneização; Para amostras maiores, aumente o volume do tampão de homogeneização ou use vários tubos para refletir uma proporção de 1:10 amostra: tampão.
- Opção 1 (batedor de contas): Homogeneizar o tecido usando um batedor de contas, 1 mL de tampão e esferas de cerâmica. Vários tubos de 2 mL ou tubos maiores podem precisar ser usados.
- Opção 1 (batedor de contas): Limpe e desinfete a estação de trabalho, o equipamento e os tubos de amostra externos com desinfetante QAC (1:256). Deixe repousar por 2 min.
- Opção 1 (Batedor de esferas): Dentro do BSC, carregue um rotor de centrífuga com amostra(s) homogeneizada(s). Centrifugue todas as amostras a 10.000-16.000 × g por 2 min em uma microcentrífuga de mesa. Descarregue o rotor da centrífuga dentro do BSC.
- Opção 1 (batedor de contas): Deixe repousar por 2 min.
- Opção 1 (batedor de esferas): Transfira 120 μL de homogenato para um tubo pré-preenchido com 1 mL de tampão de homogeneização.
CUIDADO: A homogeneização pode produzir aerossóis e deve ser realizada em um BSC. - Opção 2 (bisturi): Pique finamente os tecidos necessários usando um bisturi descartável, esfregue com um cotonete e transfira o cotonete para um tubo pré-preenchido com tampão de homogeneização e contas. Não exceder 100 mg de amostra em 1 mL de tampão de homogeneização; Para amostras maiores, aumente o volume do tampão de homogeneização ou use vários tubos para refletir uma proporção de 1:10 amostra: tampão.
CUIDADO: O uso de um bisturi com material potencialmente infectado com lyssavirus é perigoso e os usuários devem tomar as devidas precauções de segurança.
- Colete todos os tecidos restantes no recipiente original ou em um recipiente novo e vazio rotulado com uma etiqueta de acesso. Armazene este tecido caso seja necessário um novo teste ou caracterização adicional.
- Limpe e desinfete a estação de trabalho, o equipamento e a parte externa dos tubos de amostra com desinfetante QAC 1:256. Deixe repousar por 2 min.
- Repita as etapas 1.2-1.5 para todas as amostras restantes.
- Homogeneizar as amostras com um mini batedor de esferas por pelo menos 60 s. Inspecione visualmente os tubos. Repita o batedor de contas por mais 60 segundos se restarem pedaços grandes de tecido. Esta etapa é opcional se o tecido estiver completamente homogeneizado na etapa 1.4.2.1 acima.
NOTA: É importante garantir que o tecido esteja completamente homogeneizado. A homogeneização incompleta diminuirá o rendimento do RNA. - Deixe descansar por pelo menos 5 min em temperatura ambiente (RT).
- Limpe e desinfete a estação de trabalho, o equipamento e a parte externa dos tubos de amostra com desinfetante QAC (1:256).
NOTA: A amostra é considerada não infecciosa neste momento e pode ser removida do laboratório de raiva. - Processe imediatamente as amostras em tampão de homogeneização para extração de RNA, armazene em RT (20 °C a 25 °C) ou refrigerado (4 °C a 8 °C) por vários dias, ou armazene a -16 °C ou mais frio para armazenamento de longo prazo.
2. Protocolo para extração de RNA usando kit RNA MiniPrep
- Configure o espaço de trabalho no BSC.
- Limpe a superfície de trabalho do BSC usando etanol 70% antes de iniciar o trabalho para remover poeira ou outros contaminantes ambientais. Realize descontaminação adicional da superfície com desinfetante QAC (1:256), RNase AWAY ou RNaseZap (de acordo com as recomendações do fabricante).
- Coloque uma almofada de trabalho absorvente revestida de plástico e coloque reagentes, suprimentos e a amostra no BSC.
NOTA: Certifique-se de que a almofada revestida de plástico não bloqueie o fluxo de ar do BSC. Se o fluxo de ar for interrompido, não use uma almofada. - Disponha todos os tubos de coleta em um rack limpo para tubos de microcentrífuga. Pré-encha um tubo de microcentrífuga de 1,5 mL com 300 μL de etanol 100% para cada amostra de cérebro não morcego. Para amostras com pouco tecido (amostra de cérebro de morcego, amostra sem cérebro ou amostra deteriorada), pré-encha um tubo de microcentrífuga de 1,5 mL com 600 μL de etanol 100% para cada um.
- Preparação da amostra
- Colete todas as amostras preparadas na seção 1 em um suporte para tubos no BSC. Descongele todas as amostras congeladas antes do teste.
- Descongele um controle de extração.
NOTA: Recomenda-se que seja escolhida uma amostra sem RNA de lyssavirus; a amostra deve ser previamente ensaiada com um intervalo de valores de Ct esperado para o ensaio de beta-actina. Por exemplo, células de cultura de tecidos pré-aliquotadas ou um caso de raiva negativo previamente testado (humano ou animal).
- Centrifugue todas as amostras a 10.000-16.000 × g por 2 m em uma microcentrífuga de mesa.
- Transfira o sobrenadante para um novo tubo de microcentrífuga estéril contendo 100% de etanol. Certifique-se de que o sobrenadante esteja claro, sem lipídios óbvios ou tecido sólido. Evite a coleta de lipídios e tecido sólido.
- Para tecido cerebral não morcego: transferir 300 μL de sobrenadante.
- Para amostras com pouco tecido (amostra de cérebro de morcego, amostra sem cérebro ou amostra deteriorada), transfira 600 μL de sobrenadante.
- Armazenar o homogeneizado restante num tubo de microcentrífuga com tampa de rosca a ≤-16 °C.
- Pipete para cima e para baixo 10 vezes para misturar.
- Para cada amostra, transferir 600 μl da mistura etanol-sobrenadante para uma coluna de centrifugação num tubo de recolha.
- Centrifugue até que o líquido passe pela coluna (1 min a 10.000-16.000 × g). Descarte o fluxo.
- Repita se houver mais de 600 μL de mistura tampão de homogeneização de etanol para uma amostra.
- Transfira cada coluna para um novo tubo de coleta.
- Adicione 400 μL de tampão de pré-lavagem de RNA a cada coluna e centrifugue a 10.000-16.000 × g por 30 s.
- Rejeitar o fluxo contínuo e voltar cada coluna para o mesmo tubo de recolha.
- Repita as etapas 2.10-2.11.
- Adicione 700 μL de tampão de lavagem de RNA a cada coluna e centrifugue a 10.000-16.000 × g por 2 min. Certifique-se de que o tampão de lavagem passou completamente por cada coluna.
- Transfira cada coluna cuidadosamente para um tubo livre de RNase.
- Rejeitar o escoamento e o tubo de recolha do ponto 2.13.
- Adicione 50 μL de água livre de DNase / RNase diretamente à matriz da coluna para eluir o RNA.
NOTA: Não toque na matriz da coluna com a ponta da pipeta. - Incubar por 30 s em RT e, em seguida, centrifugar a 10.000-16.000 × g por 1 min.
- Transfira cuidadosamente o RNA para um novo tubo de microcentrífuga rotulado com acesso de fundo plano com tampa de parafuso. Mova o RNA extraído para o gelo para testes imediatos. Armazene a longo prazo a -70 °C ou mais frio.
NOTA: O armazenamento em temperaturas mais quentes ou congelamentos repetidos podem levar à degradação do RNA e afetar os resultados do diagnóstico.
3. Protocolo para ensaio RT-PCR em tempo real do pan-lyssavirus LN34
- Prepare reagentes.
- Controle positivo artificial
- Se um RNA de controle positivo artificial for produzido pelo CDC8, siga as instruções na embalagem para armazenamento, reconstituição e alíquota. Pule esta etapa se alíquotas de uso único de RNA de controle positivo já estiverem disponíveis.
NOTA: O RNA de controle positivo em concentrações de trabalho deve ser manuseado na área de adição do molde e não na mesma área da preparação do mastermix. O RNA de controle positivo deve produzir um valor de limiar de ciclo (Ct) dentro da faixa esperada determinada para um determinado lote. Entre as execuções, o valor de LN34 Ct para o RNA de controle positivo não deve diferir em mais de ±1,5 valores de Ct. - Descongelar uma alíquota descartável de ≤ armazenamento a -70 °C imediatamente antes de usar em gelo ou bloco de gelo. Não congele, descongele e descarte alíquotas mantidas por um longo período de tempo em temperaturas refrigeradas.
NOTA: Um controle positivo deve ser executado em triplicado no ensaio LN34; O controle positivo artificial8 não amplificará no ensaio de beta-actina.
- Se um RNA de controle positivo artificial for produzido pelo CDC8, siga as instruções na embalagem para armazenamento, reconstituição e alíquota. Pule esta etapa se alíquotas de uso único de RNA de controle positivo já estiverem disponíveis.
- Controlo de extracção e amostras: Colocar as amostras recém-extraídas sobre gelo (ou bloco de gelo) ou descongelar amostras de ≤ a -70 °C de armazenamento em gelo (ou bloco de gelo) imediatamente antes da utilização.
NOTA: O RNA deve ser descongelado e processado em uma área designada para adição de amostra ou modelo que seja separada das áreas usadas para preparação de mastermix ou manipulação de produtos de PCR ou grandes quantidades de material viral (por exemplo, geração de controle positivo, propagação viral)
- Controle positivo artificial
- Prepare os reagentes mastermix na área de preparação mastermix.
- Preparação Mastermix do ensaio singleplex LN34 RT-PCR
NOTA: Os usuários podem testar amostras no formato singleplex (etapa 3.2.1) ou multiplex (etapa 3.2.2). Não é necessário executar os números 3.2.1 e 3.3.2. A preparação do mastermix, a alíquota do primer e da sonda e nenhum reagente de controle de modelo devem ser descongelados e manipulados em uma área limpa separada do processamento de amostras, necropsia, PCR e outras áreas onde os materiais virais são manipulados. Isso pode ser alcançado por meio de salas separadas ou um sistema de gabinete com fluxo de amostra unilateral.- Gere misturas de primer e sonda em concentrações de trabalho, conforme indicado na Tabela 1 e na Tabela 2. Saltar esta etapa se as alíquotas de diluição de trabalho dos primers e sondas já estiverem disponíveis.
- Alíquotas de primers e sondas em estoques de armazenamento de 1,5 mL e 50 μL de trabalho e armazene a ≤ -16°C no escuro. Saltar esta etapa se as alíquotas de diluição de trabalho dos primers e sondas já estiverem disponíveis.
NOTA: Recomenda-se executar a etapa 3.2.1.2. - Descongele o tampão RT-PCR de uma etapa, sem controle de modelo, primers e sondas de armazenamento de ≤ -16 °C em gelo ou bloco de gelo na área de preparação do mastermix.
NOTA: Use reagentes até a data de validade ou falha de desempenho, o que ocorrer primeiro. - Rapidamente vórtice e centrifugue todos os tampões, primers e sondas antes de usar.
- Armazene a enzima RT-PCR de uma etapa no gelo ou em um bloco de gelo até o uso.
- Amostras de RNA
NOTA: Use RNA recém-extraído sempre que possível, pois o congelamento e descongelamento pode afetar o desempenho - Armazene amostras de RNA em gelo ou bloco de gelo até o uso.
- Descongele todas as amostras de RNA congeladas no gelo ou em um bloco de gelo.
- Rotular um tubo de microcentrífuga por ensaio (LN34 e βA).
- Determine o número de reações (N) a serem configuradas por ensaio.
- Calcule o número de reações para o ensaio LN34 multiplicando o número de amostras por 3 e adicionando 6 para poços de reação de controle mais 10% de reações extras para contabilizar o volume perdido durante a pipetagem. (por exemplo, para 10 amostras: (10 x 3) + 6 = 36 reações; excesso de reações: (36 x 0,1) + 36 = 3,6 + 36 = 39,6 reações totais, ou 40 reações arredondadas para cima)
NOTA: Para testes clínicos, sugere-se testar todas as amostras em triplicata para LN34. Para fins de vigilância, cada amostra pode ser testada em duplicata. Recomenda-se que o triplicado seja usado durante a integração inicial do ensaio para garantir baixa variabilidade entre as réplicas e boa técnica. - Calcule o número de reações para o ensaio βA adicionando o número de amostras mais 4 poços de reação de controle mais 10% de reações extras para contabilizar o volume perdido durante a pipetagem.
- Determine o volume de cada reagente para os mastermixes LN34 e βA usando a Tabela 2.
- Designar alvéolos para cada amostra a ser ensaiada em triplicado no ensaio LN34 e singlicar para o ensaio βA utilizando um mapa de placas de 96 poços.
- Dispense 23 μL de mastermix de ensaio LN34 em cada poço LN34 designado após um breve vórtice e rotação por 30 s usando uma microcentrífuga de mesa para coletar o líquido no fundo do tubo. Evite introduzir bolhas.
- Dispense reagentes 23 μL de mastermix de ensaio βA em cada poço marcado com βA após um breve vórtice e rotação por 30 s em uma microcentrífuga de mesa para coletar o líquido na parte inferior.
- Configure as reações sem controle de modelo (NTC) pipetando 2 μL de água de grau PCR em cada poço NTC.
- Cubra os poços e transfira a placa para a área de adição do modelo.
- Brevemente vórtice e centrifugue os tubos contendo as amostras de RNA.
- Pipete 2 μL de RNA extraído da primeira amostra em cada poço marcado para essa amostra. Evite introduzir bolhas.
- Certifique-se de que o RNA foi atraído para a pipeta por visualização.
- Pipete na lateral do poço para garantir que a amostra seja adicionada ao poço correto.
- Evite agitar pontas de pipeta contendo RNA sobre poços abertos, tanto quanto possível.
- Repetir o passo 3.2.10 para as restantes amostras e para o ARN de controlo positivo.
- Coloque a tampa adesiva óptica sobre os poços depois de adicionar todas as amostras e controles. Tenha o cuidado de cobrir todos os poços e selar completamente.
- Centrifugue a 500 × g por 1 min em RT em uma centrífuga de mesa ou use um centrifugador de pratos do tipo centrifugador de salada.
- Coloque a placa selada em um instrumento de PCR em tempo real calibrado para corantes repórteres FAM e VIC/HEX e ajuste-o para os parâmetros de ciclagem mostrados na Tabela 3.
- Prepare master mixes para o ensaio LN34 Multiplexed (LN34M).
- Rotule um tubo de microcentrífuga LN34M de acordo com a Tabela 2.
- Determine o número de reações (N) a serem configuradas por ensaio.
- Calcule o número de reações para o ensaio LN34M multiplicando o número de amostras por 3 e adicione 6 para poços de reação de controle mais 10% de reações extras para contabilizar o volume perdido durante a pipetagem. (por exemplo, para 10 amostras: (10 x 3) + 6 = 36 reações; excesso de reações: (36 x 0,1) + 36 = 3,6 + 36 = 39,6 reações totais, ou 40 reações arredondadas para cima)
- Escolha o formato de 25 μL ou 12,5 μL. Determine o volume de cada reagente para o mastermix LN34M usando a Tabela 2.
- Designe poços para cada amostra a ser testada em triplicata no ensaio LN34M usando um mapa de placas de 96 poços.
- Dispense reagentes para o ensaio LN34M nos poços. Brevemente vórtice e rotação para baixo tubos para coletar o líquido no fundo antes de dispensar 23 μL (para reação de 25 μL) ou 10,5 μL (para reação de 12,5 μL) de mistura principal em cada poço designado. Evite introduzir bolhas.
- Configure as reações NTC pipetando 2 μL de água de grau PCR em cada poço NTC.
- Cubra os poços e transfira a placa para a área de adição do modelo.
- Brevemente vórtice e gire para baixo os tubos contendo as amostras de RNA para coletar o líquido no fundo.
- Pipete 2 μL de RNA extraído da primeira amostra em cada poço marcado para essa amostra. Evite introduzir bolhas.
- Certifique-se de que o RNA seja atraído para a visualização da pipeta.
- Pipete na lateral do poço para garantir que a amostra seja adicionada ao poço correto.
- Evite agitar pontas de pipeta contendo RNA sobre poços abertos, tanto quanto possível.
- Repetir o passo 3.3.8 para as restantes amostras e o ARN de controlo positivo.
- Depois de adicionar a última amostra/controle, coloque a tampa adesiva óptica sobre os poços, garantindo que todos os poços estejam cobertos e selados completamente.
- Centrifugue a 500 × g por 1 min em RT em uma centrífuga de mesa ou use um centrifugador de pratos do tipo centrifugador de salada.
- Coloque a placa selada em um instrumento de PCR em tempo real calibrado para corantes repórteres FAM e VIC/HEX e ajuste-a para os parâmetros de ciclagem, conforme mostrado na Tabela 3. Defina o corante de referência passivo para ROX e execute no modo padrão (não execute no modo rápido)
NOTA: Esta configuração é específica para os instrumentos mencionados neste protocolo e requer o uso de um reagente RT-PCR de uma etapa contendo ROX como corante passivo. Instrumentos alternativos requerem abordagens diferentes para determinar as configurações ideais de execução. Garanta a manutenção normal do instrumento de acordo com o fabricante para obter o melhor desempenho.
- Preparação Mastermix do ensaio singleplex LN34 RT-PCR
4. Interpretação dos resultados
- Defina uma linha de base automática e cálculos de limite manual usando um valor de 0,2 para LN34/FAM e 0,05 para βA/HEX/VIC.
NOTA: Esta configuração é específica para os instrumentos mencionados neste protocolo e requer o uso de um reagente RT-PCR de uma etapa contendo ROX como corante passivo. Instrumentos alternativos requerem abordagens diferentes para calcular os valores de linha de base e limiar. - Determine o resultado do diagnóstico usando as orientações da Tabela 4 se todos os controles funcionaram conforme o esperado (Tabela 5).
- Confirme todos os valores de Ct ou Cq visualizando gráficos de amplificação.
- Investigue quaisquer resultados incomuns conforme recomendado.
5. Retenção e armazenamento de amostras
- Armazene todas as amostras congeladas a -16 °C ou menos até que o teste seja concluído e os resultados sejam relatados. Conservar os tecidos originais para confirmar os resultados ou identificar o animal hospedeiro para a espécie em caso de resultados de testes invulgares.
- Use identificadores de amostra exclusivos; Rotule todos os tubos, relatórios e documentos com identificadores de amostra exclusivos completos.
- Reter amostras intermédias (a curto prazo) para o caso de ser necessário repetir o ensaio.
- Reter amostras positivas representativas conforme necessário para uso como controles, tipagem epidemiológica e outros fins.
- Armazene o RNA a ≤-70 °C para armazenamento de longo prazo.
Resultados
Imagens representativas de um ensaio LN34 bem-sucedido executado em um instrumento de PCR em tempo real ABI ViiA7 são mostradas na Figura 2. A visualização dos resultados plotados em uma escala logarítmica permite uma fácil visualização do valor Ct, o ponto em que a curva cruza a linha limite (Figura 2A, C). Quando plotada em uma escala linear, a amplificação bem-sucedida aparecerá como uma curva sigmoidal (ou em forma de "S") (Figura 2B, D), enquanto os resultados negativos devem aparecer como uma linha reta e plana. Recomenda-se a exibição de resultados em exibições lineares e de escala de log para identificar possíveis anomalias ou erros. Resultados positivos e negativos típicos na visualização de gráfico multicomponente podem ser vistos na Figura 2E, F, respectivamente, onde o nível de fluorescência do corante que marca a sonda (FAM para LN34, VIC/HEX para βA) pode ser observado em relação ao corante passivo no tampão de reação (ROX).
Exemplos de resultados anormais são mostrados na Figura 3. Comparações entre os gráficos de execuções bem-sucedidas (Figura 2) e gráficos anormais (Figura 3) podem ser usadas para isolar execuções atípicas e problemas de instrumentos. A Figura 3A mostra um sinal cruzando o limiar, produzindo um valor de Ct para LN34, mas a curva de amplificação é muito atípica, aumentando linearmente. O gráfico multicomponente (Figura 3B) também mostra uma linha ondulada que não é típica de uma amostra positiva. Este exemplo destaca a importância de visualizar os gráficos de amplificação e não simplesmente copiar os valores de Ct. Certifique-se sempre de que as curvas de amplificação pareçam normais para todas as amostras. A visualização do gráfico multicomponente também é recomendada para garantir que não haja irregularidades. Ocasionalmente, sinais de linha de base confusos podem gerar valores de Ct nos casos em que nenhuma amplificação ocorreu. Se os sinais de amplificação parecerem lineares, sugere-se que a linha de base seja ajustada para ver se a curva desaparece. No caso de qualquer sinal incomum, toda a execução deve ser repetida. Recomenda-se limpar e executar uma placa de fundo em seu instrumento de PCR em tempo real se os problemas persistirem. Se disponíveis, os produtos de PCR podem ser executados em um gel de agarose e/ou sequenciados para solucionar problemas de resultados incomuns. Não é recomendado usar os resultados da eletroforese em gel ou sequenciamento para determinar os resultados diagnósticos.
Estudos anteriores mostraram baixa variabilidade entre repetições, ensaio, operador e laboratório para o ensaio LN347. Se for observada uma elevada variabilidade (diferença de >±1,5 Ct) entre os replicados da mesma amostra, esse ARN deve ser novamente testado. A alta variabilidade pode ser causada por problemas com pipetas, práticas de laboratório, pipetagem incorreta ou máquinas de PCR em tempo real. A observação repetida de alta variabilidade em várias amostras ou em ensaios pode indicar problemas sistêmicos. Amostras com baixo RNA, aproximando-se do limiar do ensaio para uma amostra positiva (Ct 35), podem apresentar maior variabilidade nos valores de Ct entre as repetições. A consulta com o CDC e a solução de problemas podem ser necessárias para resolver a causa da variabilidade persistente, resultados inconsistentes ou falha do ensaio.
A alta sensibilidade dos ensaios baseados em PCR os torna inerentemente suscetíveis à contaminação. A adesão estrita às boas práticas de laboratório é a melhor maneira de mitigar a contaminação cruzada. Saber identificar possíveis contaminações é importante. Deve-se suspeitar de contaminação do reagente se nenhum controle de modelo e suspeita de poços de amostra negativos em um ensaio produzirem valores de Ct semelhantes. Repita o teste com novas alíquotas de reagentes de PCR (tampão, água, primers e enzima) e o mesmo RNA. Se todas as amostras e o controlo de extracção produzirem valores de CT semelhantes, mas a NTC for negativa, deve investigar-se a contaminação dos reagentes de extracção e repetir-se a extracção com novos reagentes. É uma boa prática fazer pequenas alíquotas de reagentes para reduzir o risco de contaminação e evitar a possibilidade de descartar grandes volumes de reagentes caros. A contaminação cruzada da amostra é mais difícil de identificar. Se houver suspeita de contaminação da amostra, repita a coleta da amostra a partir dos tecidos originais. Em alguns casos, o sequenciamento do RNA viral pode confirmar a contaminação, especialmente quando o RNA contaminante é muito diferente da variante viral esperada (como um vírus de controle usado em laboratório). O sequenciamento de duas amostras processadas ao mesmo tempo pode determinar se as sequências virais são idênticas, mas pode não ser informativo se as sequências forem muito semelhantes (por exemplo, a mesma variante coletada no mesmo município). Se houver suspeita de contaminação da amostra com o RNA de controle positivo, pode-se executar os amplicons do ensaio LN34 em um gel de agarose para diferenciar o RNA do lyssavirus (165 pb) do RNA de controle positivo (99 pb). A sequência do molde usado para gerar o RNA de controle positivo fornecido pelo CDC8.
Para outros patógenos, os laboratórios podem ser usados para definir o limite manualmente para se livrar do "ruído", como a amplificação fraca mostrada em ciano na Figura 4. Esta prática NÃO é recomendada para o diagnóstico da raiva porque pode levar a resultados falsos negativos com consequências terríveis, uma vez que a raiva é quase 100% fatal. NÃO altere manualmente o limite para produzir resultados negativos para amostras de amplificação fracas ou tardias. Essas amostras devem ser extraídas novamente e/ou testadas novamente para descartar raiva.
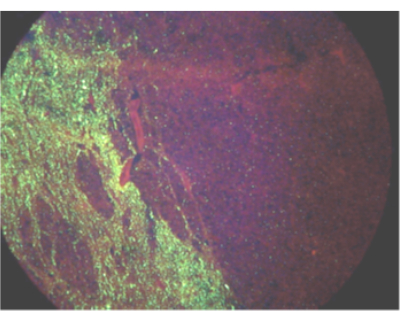
Figura 1: Campo de visão mostrando a disseminação unilateral do antígeno do vírus da raiva em um burro infectado por teste de imunofluorescência direta. Clique aqui para ver uma versão maior desta figura.
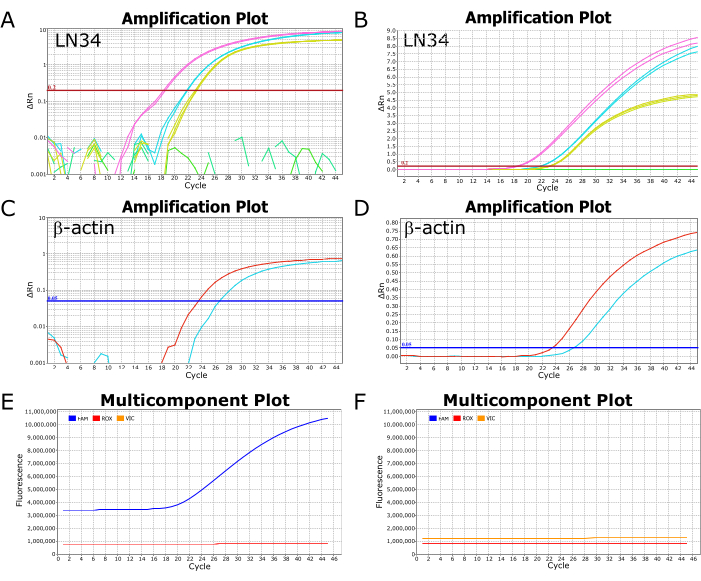
Figura 2: Gráficos de amplificação e multicomponentes de uma execução bem-sucedida do ensaio LN34. (A-D) Os dados dos resultados são plotados em uma escala logarítmica (A,C) e (B,D) linear para o ensaio LN34 e βA. Os painéis A e B representam os resultados do LN34 de duas amostras (em rosa e ciano) em comparação com o controle positivo (em amarelo). No painel B, há uma linha verde plana que representa uma amostra negativa adicional na execução. Em A, a(s) linha(s) verde(s) não mostra(m) nenhuma amplificação e são representadas como segmentos quebrados. O limite para o ensaio LN34 foi definido manualmente para 0,2 e é mostrado pela linha horizontal vermelha. (C, D) Resultados do ensaio βA para duas amostras (vermelho e ciano). O limite para o ensaio βA foi definido manualmente em 0,05. (E, F) Os gráficos multicomponentes representam a fluorescência (RFU) em cada ciclo para FAM (LN34), VIC (βA) e ROX (corante passivo presente no tampão AgPath-ID). Os níveis de ROX devem permanecer estáveis em todos os ciclos. Uma amostra positiva típica é mostrada no painel E; A fluorescência FAM aumenta como uma curva sigmoidal começando no ciclo 18 para esta amostra. Uma amostra negativa típica é mostrada no painel F, onde o nível FAM permanece paralelo ao nível ROX em todos os ciclos. Os dados são de um instrumento de PCR em tempo real ABI ViiA7. Clique aqui para ver uma versão maior desta figura.

Figura 3: Imagens representativas do sinal raro e atípico observado no ensaio LN34 são executadas em um instrumento de PCR em tempo real ViiA7. (A-F) Parcelas de amplificação (A,C,E) e multicomponentes (B,D,F) produzidas devido à contaminação do poço. O aumento linear (A,C) e as flutuações onduladas (B,D) na fluorescência FAM não representam uma amplificação verdadeira com base na forma das curvas e na magnitude da mudança de fluorescência. Os painéis A a D provavelmente representam amostras negativas, embora um valor de Ct tenha sido produzido para a réplica mostrada nos painéis A e B. Os painéis E e F mostram um sinal ondulado estranho que é mais facilmente visto no gráfico multicomponente. Esse tipo de sinal deve ser investigado e pode indicar problemas no instrumento, mesmo que todos os controles tenham funcionado conforme o esperado nesta execução. Clique aqui para ver uma versão maior desta figura.
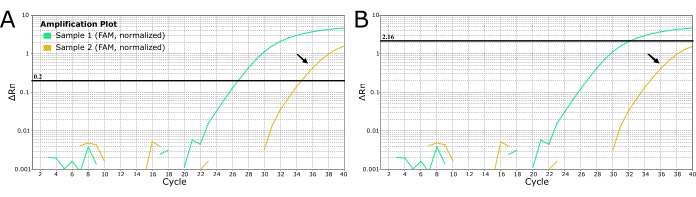
Figura 4: Curvas de RT-PCR em tempo real LN34 de 2 amostras suspeitas de raiva mostrando dois métodos de definição de valores limite. (A) O limite LN34 foi definido como 0,2 (recomendado para todas as execuções). (B) Determinar manualmente um limite diferente para cada corrida para mascarar o sinal determinado como "ruído" (sinal de amplificação tardia). O método usado no painel B NÃO é recomendado para a raiva devido às graves consequências de não ter um resultado positivo verdadeiro. A amplificação tardia pode indicar uma amostra positiva fraca, inibição da reação em cadeia da polimerase ou falha na extração em um caso positivo. Também pode indicar contaminação cruzada. A amostra dourada (indicada por setas pretas) produz um valor de Ct no ponto de corte do ensaio e não deve ser considerada negativa. As amostras com amplificação tardia devem ser extraídas e testadas novamente. Clique aqui para ver uma versão maior desta figura.
Tabela 1: Sequências e concentrações de primer e sonda usadas nos ensaios de RT-PCR em tempo real LN34lys (singleplex LN34), LN34M (LN34 e βA multiplexado). As sondas LN34 são marcadas com o corante fluorescente FAM na extremidade 5' e o supressor de buraco negro (BHQ1) na extremidade 3'. A sonda βA é marcada com o corante HEX fluorescente na extremidade 5' e o supressor de buraco negro (BHQ1) na extremidade 3'. As bases modificadas por nucleotídeos bloqueados são indicadas por um sinal de mais que precede a base na sequência. Clique aqui para baixar esta tabela.
Tabela 2: Configuração do ensaio para os ensaios LN34lys, Actin3 e LN34M. Os nomes, sequências e concentrações de primers e sondas podem ser encontrados na Tabela 1. LN34_F1 corresponde a ACGCTTAACAACCAGATCAAAGAA7. Clique aqui para baixar esta tabela.
Tabela 3: Parâmetros de ciclagem para instrumentos ABI. IMPORTANTE: Certifique-se de executar no modo PADRÃO , não no modo RÁPIDO . ROX deve ser selecionado como o corante de referência passivo. Clique aqui para baixar esta tabela.
Tabela 4: Algoritmo para interpretação dos resultados de RT-PCR em tempo real do LN34 para os formatos singleplex (superior, tabela azul) e multiplex (inferior, tabela vermelha). Um resultado positivo de LN34 deve ser considerado positivo, mesmo que o resultado βA seja negativo ou inconclusivo. Se o amplicon LN34 não for detectado, o βA Ct deve ser ≤ o ponto de corte do valor Ct listado para ser considerado negativo. Os valores de βA Ct indicam a qualidade da amostra que está sendo testada e identificam possíveis inibições. A baixa concentração na amostra clínica original pode afetar as curvas de crescimento βA, levando a nenhuma amplificação discernível. Fatores adicionais que contribuem para a falha na detecção de β-actina incluem extração inadequada de RNA devido à perda de RNA ou transferência de inibidores de PCR, configuração e técnica incorretas do ensaio, tipo ou qualidade de amostra insatisfatórios e mau funcionamento de reagentes ou equipamentos. Clique aqui para baixar esta tabela.
Tabela 5: Ações e interpretações de resultados comuns para controles de ensaio LN34. Todos os três controles (RNA de controle positivo da raiva, controle de extração negativo da raiva e nenhum controle de modelo devem produzir os resultados esperados para que uma corrida seja aprovada. A falha no controle positivo ou nenhum controle de modelo pode indicar falha de pipetagem, reagente ou equipamento. Toda a execução, incluindo todas as amostras de RNA testadas, deve ser repetida. A falha do controle de extração pode indicar um problema durante a extração, como falha do reagente, pipetagem incorreta ou contaminação cruzada. A extracção de todas as amostras deve ser repetida. A falha dos controles deve ser rara para o pessoal experiente do laboratório. Clique aqui para baixar esta tabela.
Discussão
Uma execução bem-sucedida do ensaio LN34 requer um controle positivo, controle de extração e nenhuma reação de controle de modelo funciona conforme o esperado em cada execução de ensaio ou a execução deve ser invalidada e repetida. Todas as três reações de replicação de controle positivo LN34 devem cruzar o limite dentro da faixa especificada ou a execução deve ser repetida. O RNA de controle positivo descrito em publicações anteriores 7,8 não amplificará no ensaio βA. As reações de controle sem modelo não devem exibir curvas de amplificação que cruzem a linha de limiar para o ensaio LN34 ou βA. O controle de extração não deve exibir amplificação para LN34. Se for observada uma amplificação inesperada no NTC ou no controlo de extração, pode indicar contaminação e invalidar o ensaio de execução e repetição para todas as amostras (ver quadro 5). Os usuários podem considerar a adição de controles adicionais, incluindo um controle sem processo ou controle de extração sem amostra, para monitorar a contaminação βA do hospedeiro dos reagentes de extração.
À medida que a letalidade por raiva se aproxima de 100%, recomenda-se que qualquer amplificação fraca ou anormal seja investigada mais a fundo, mesmo que não produza um valor de Ct. As reações negativas ou NTC não devem exibir nenhuma amplificação, e a fluorescência deve aparecer como uma linha plana paralela à fluorescência ROX na visão multicomponente. Observações de curvas, especialmente em múltiplas réplicas, podem indicar contaminação cruzada ou um resultado positivo fraco. Todas as réplicas para uma amostra positiva devem ser amplificadas para um resultado positivo válido. Se apenas um subconjunto de réplicas for amplificado em qualquer um dos ensaios, a amostra deve ser testada novamente. Além disso, qualquer amostra que produza resultados muito variáveis (diferenças no valor de Ct > ±1,5 entre os replicados) deve ser considerada inválida e a amostra deve ser novamente testada. Se o problema persistir, a amostra deve ser extraída novamente.
Espera-se que uma amostra positiva para raiva extraída do tronco cerebral e do tecido cerebelo adequadamente coletado e armazenado tenha um valor de Ct inferior a 35 ciclos para o ensaio LN34. Todas as amostras inconclusivas devem ser testadas novamente por RT-PCR em tempo real LN34. Se a amostra for inconclusiva após a repetição do teste e todos os controles forem realizados conforme o esperado, recomenda-se a reextração do RNA. Amostras com baixo RNA viral (LN34 Ct > 35) podem indicar problemas potenciais, como contaminação, baixa carga viral, inibição de PCR ou falha na extração. Colete pedaços frescos do cérebro do tecido original, faça a extração de RNA e teste novamente a amostra. Da mesma forma, os valores de Ct > 33 (singleplex), 37 (LN34M) ou nenhuma amplificação no ensaio βA podem indicar falha na extração de RNA. Repita a extração para essas amostras e, em seguida, repita o teste para LN34 e actina. Se uma amostra produzir um resultado inconclusivo novamente após testes repetidos, use um método secundário, como o teste DFA (também chamado de FAT), DRIT ou isolamento de vírus. Se forem observados resultados discordantes contínuos ou inconclusivos, consulte um laboratório de referência em raiva para testes confirmatórios.
Se os inibidores estiverem presentes em uma extração de RNA, os ensaios de PCR podem produzir um resultado falso negativo. Se houver suspeita de inibição ou se for observada inibição das reações de controle βA (como valor de Ct > 33 ou valor de Ct > 37) para uma amostra específica, o RNA extraído deve ser testado em 2 ou mais diluições (por exemplo, 1:10 e 1:100 em água livre de nuclease) para diluir quaisquer inibidores de PCR potenciais. Para amostras difíceis, a entrada de RNA pode ser aumentada para 8,5 μL na reação RT-PCR sem adicionar água. Isso pode revelar inibição aumentada (valor de Ct posterior em comparação com 2 μL de RNA de entrada) ou baixo nível de RNA na amostra original (valor de Ct anterior ao usar 8,5 μL em comparação com 2 μL de RNA de entrada).
O ensaio LN34 não diferencia entre lyssavirus nem determina variantes do vírus da raiva. O amplicon do ensaio LN34 pode ser sequenciado para tipagem de variantes do vírus da raiva de baixa resolução ou identificação de espécies de lyssavirus11.
Divulgações
Nenhum a divulgar
Agradecimentos
Reconhecemos os esforços e a colaboração de muitos laboratórios de testes de diagnóstico da raiva que contribuíram para a implementação, validação e otimização do ensaio LN34 por meio de seu compartilhamento de dados abertos e feedback. O uso de nomes comerciais e fontes comerciais é apenas para identificação e não implica endosso dos Centros de Controle e Prevenção de Doenças, do Departamento de Saúde e Serviços Humanos dos EUA ou das instituições afiliadas dos autores. As conclusões, descobertas e opiniões expressas pelos autores não refletem necessariamente a posição oficial do Departamento de Saúde e Serviços Humanos dos EUA, dos Centros de Controle e Prevenção de Doenças ou das instituições afiliadas dos autores.
Materiais
| Name | Company | Catalog Number | Comments |
| 7500 Fast | Applied Biosystems | N/A | Do not substitute without validation |
| 7500 Fast Dx | Applied Biosystems | N/A | Do not substitute without validation |
| ABI ViiA 7 | Applied Biosystems | N/A | Do not substitute without validation |
| AgPath-ID One-Step RT-PCR Kit | ThermoFisher Scientific | AM1005 | Do not substitute without validation |
| Beadbug6 | Benchmark Scientific | D1036 | |
| Direct-zol RNA MiniPrep kit | Zymo Research | R2052 | |
| MagNA Lyser green beads | Roche | 3358941001 | |
| Microcentrifuge | Eppendorf | 5425 R | |
| Optical 96-well Reaction Plates | ThermoFisher Scientific | 4346907 | |
| Optical Adhesive covers | ThermoFisher Scientific | 4311971 | Alternative: caps |
| Polyester fiber-tipped applicator swabs | BD BBL Polyester Fiber Tipped Application Swab | 220690 | |
| QuantStudio 6Flex | Applied Biosystems | 4485691 | Do not substitute without validation |
| Quaternary ammonium disinfectant (1:256) | LYSOL | WBB56939 | Do not substitute without validation |
| RNase AWAY | ThermoFisher Scientific | 7002PK | |
| RNaseZap | ThermoFisher Scientific | AM9780 | |
| Single-use scalpel, a scalpel with a safety mechanism | Integra Miltex | 4-510 | |
| Sterile polyproylene microcentrifuge tubes (1.5 mL), nuclease free | Sarstedt | 72.692.405 | |
| Sterile polyproylene microcentrifuge tubes (2 mL), nuclease free | Sarstedt | 72.694.600 | |
| TRIzol Reagent | ThermoFisher Scientific | 15596026 | Do not substitute without validation |
Referências
- Meechan, P. J., Potts, J. . Biosafety in Microbiological and Biomedical Laboratories. , (2020).
- . Terrestrial Manual 2023 Available from: https://www.woah.org/en/what-we-do/standards/codes-and-manuals (2023)
- . Laboratory Techniques in Rabies Available from: https://iris.who.int/handle/10665/310836 (2018)
- . Laboratory Techniques in Rabies Available from: https://iris.who.int/bitstream/handle/10665/310837/9789241515306-eng.pdf?ua=1 (2019)
- . Protocol for postmortem diagnosis of rabies in animals by direct fluorescent antibody testing: A minimum standard for rabies diagnosis in the United States Available from: https://www.cdc.gov/rabies/pdf/RabiesDFASPv2.pdf (2003)
- World Health Organization. . WHO Expert Consultation on Rabies: Third Report. , (2018).
- Gigante, C. M., et al. Multi-site evaluation of the LN34 pan-lyssavirus real-time RT-PCR assay for postmortem rabies diagnostics. PLoS One. 13 (5), e0197074 (2018).
- Wadhwa, A., et al. A Pan-Lyssavirus Taqman Real-Time RT-PCR assay for the detection of highly variable rabies virus and other lyssaviruses. PLoS Negl Trop Dis. 11 (1), e0005258 (2017).
- Gigante, C. M., Wicker, V., Wilkins, K., Seiders, M., Zhao, H., Patel, P., Orciari, L., Condori, R. E., Dettinger, L., Yager, P., Xia, D., Li, Y., et al. Optimization of pan-lyssavirus LN34 assay for streamlined rabies diagnostics by real-time RT-PCR. Journal Virological Methods. , (2024).
- Rao, A. K., et al. Use of a modified preexposure prophylaxis vaccination schedule to prevent human rabies: recommendations of the advisory committee on immunization practices-United States, 2022. Morbidity and Mortality Weekly Report. 71 (18), 619 (2022).
- Condori, R. E., et al. Using the LN34 Pan-Lyssavirus Real-Time RT-PCR assay for rabies diagnosis and rapid genetic typing from formalin-fixed human brain tissue. Viruses. 12 (1), 120 (2020).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados