Method Article
Postmortale Diagnose von Tollwut bei Tieren mit dem aktualisierten, multiplexierten LN34 Echtzeit-Reverse-Transkriptase-Polymerase-Kettenreaktionsassay
In diesem Artikel
Zusammenfassung
Dieses Protokoll demonstriert den Echtzeit-Assay für die Reverse-Transkriptase-Polymerase-Kettenreaktion (RT-PCR) des Pan-Lyssavirus LN34 von der Gewebeentnahme bis zur Ergebnisinterpretation, einschließlich Aktualisierungen von Primersequenzen und Formulierungen zur Verbesserung der Assay-Leistung für einige Nicht-Tollwut-Lyssaviren und Hasentiere. Wir demonstrieren auch den Assay-Aufbau für ein Single-Well-LN34-Multiplexformat (LN34M).
Zusammenfassung
Tollwut ist eine tödliche Zoonose, die durch das Lyssavirus-Tollwut (RABV) und verwandte Negativstrang-RNA-Viren der Gattung Lyssavirus (Familie Rhabdoviridae) verursacht wird. Der LN34-Assay zielt auf die hochkonservierte Leader-Region und das Nukleoprotein-Gen des Lyssavirus-Genoms ab und verwendet entartete Primer und eine TaqMan-Sonde mit gesperrten Nukleotiden, um RNA in der verschiedenen Lyssavirus-Gattung nachzuweisen. Ein negativer Befund für die Tollwut sollte nur gemacht werden, wenn ein vollständiger Querschnitt des Hirnstamms und drei Hirnlappen untersucht werden; Die Identifizierung von Lyssavirus-RNA in einem Gewebe ist jedoch diagnostisch für eine Tollwutinfektion. Das Gewebe wird gesammelt und in einem TRIzol-Reagenz homogenisiert, das das Virus ebenfalls inaktiviert. Die RNA-Extraktion wird mit einem kommerziellen Extraktionskit auf Basis von Spin-Säulen durchgeführt. Mastermixe werden in einem sauberen Raum hergestellt und in eine 96-Well-Platte aliquotiert, bevor Proben-RNA hinzugefügt wird. Im klinischen Umfeld wird jede Probe mittels real-time RT-PCR auf das Vorhandensein von Lyssavirus-RNA in dreifacher Ausfertigung und einzeln auf Wirt-β--Aktin-mRNA getestet. Positiv- und Negativkontrollen sind in den Extraktions- und Echtzeit-RT-PCR-Schritten des Protokolls enthalten. Die Datenanalyse beinhaltet eine manuelle Anpassung der Schwellenwerte, um die Ct-Werte über Geräteläufe hinweg zu standardisieren. Positive Ergebnisse werden durch das Vorhandensein einer typischen Amplifikation im Pan-Lyssavirus-Assay (Ct ≤ 35) bestimmt. Negative Ergebnisse werden durch das Fehlen einer typischen Amplifikation im Pan-Lyssavirus-Assay und den Nachweis von Wirts-β-Aktin-mRNA (Ct ≤ 33) bestimmt. Die Beobachtung von Werten außerhalb dieser Bereiche oder das Versagen der Assay-Kontrollen kann den Lauf ungültig machen oder zu nicht schlüssigen Ergebnissen für eine Probe führen. Das Protokoll sollte genau befolgt werden, um eine hohe Sensitivität und Spezifität des Assays zu gewährleisten. Verfahrensänderungen können die Assay-Leistung beeinträchtigen und zu falsch positiven, falsch negativen oder nicht interpretierbaren Ergebnissen führen.
Einleitung
Dieses Protokoll beschreibt das Verfahren für die Tollwutdiagnostik mit dem LN34-Pan-Lyssavirus-Echtzeit-Reverse-Transkriptase-Polymerase-Kettenreaktions-Assay (RT-PCR) von der Probenentnahme bis zur Interpretation der Ergebnisse. Das Verfahren wird in drei Abschnitte unterteilt: Entnahme von Gehirnproben, die für den LN34-Assay relevant sind (Abschnitt 1), manuelle säulenbasierte RNA-Extraktion mit dem Direct-zol RNA Miniprep-Kit (Zymo Research R2051) (Abschnitt 2) und LN34-Echtzeit-RT-PCR-Assay, der mit dem AgPath-ID One-Step RT-PCR-Kit (ThermoFisher Scientific AM1005) eingerichtet wurde (Abschnitt 3). RNA-Extraktion und RT-PCR können mit anderen Produkten durchgeführt werden, aber die Kits sollten vor der Verwendung validiert werden, um sicherzustellen, dass die Lyssavirus-RNA angemessen extrahiert und amplifiziert wird.
Abschnitt 1 beschreibt die Entnahme des richtigen Hirngewebes, das für den LN34 real-time RT-PCR-Assay verwendet werden soll. Die Beschreibung der Autopsie von Tieren, der Enthauptung und der Entfernung des Gehirns ist nicht enthalten. Die Proben können Infektionserreger enthalten. Die Biosicherheitsverfahren, die in der 6. Auflage1 der Biosicherheit in mikrobiologischen und biomedizinischen Laboratorien beschrieben sind, sollten befolgt werden, um das Risiko zu mindern. Die Proben sollten bis zum Abschluss der Inaktivierung als infektiös gelten. Die Virusinaktivierung und die Validierung des Assays sollten in jedem Labor gemäß den Standards der jeweiligen Einrichtung durchgeführt werden. Laboratorien sollten bei der Einführung eines neuen diagnostischen Tests die von ihrer Institution festgelegten Standardsicherheits- und Qualitätsverfahren befolgen.
Basierend auf dem, was über die Ausbreitung des Tollwutvirus während einer Infektion bekannt ist, sind der Hirnstamm und das Kleinhirn die besten Gewebe für die Tollwutdiagnose, und diese Gewebe werden von der Weltgesundheitsorganisation und der Weltorganisation für Tiergesundheit für Tollwuttests empfohlen 2,3,4,5. Da die Virusausbreitung vor allem bei größeren Tieren einseitig sein kann (Abbildung 1), sollte ein vollständiger Querschnitt des Hirnstamms und drei Lappen des Kleinhirns auf Tollwutausschluss untersucht werden. Bei Proben, die diese Mindestkriterien nicht erfüllen, kann das Laboratorium die Probe als unzureichend für die Untersuchung ablehnen oder sich für einen Test zu Überwachungs- oder Rule-in-Zwecken entscheiden. Wenn die erforderlichen Gewebe nicht eingegangen sind, das Labor sich jedoch dafür entscheidet, die Probe zu testen, sollte ein negatives Testergebnis als nicht schlüssig für die Tollwut dieses Tieres interpretiert werden, da das Vorhandensein viraler RNA in anderen Geweben verzögert, gering, intermittierend oder nicht vorhanden sein kann. Die Entnahme der erforderlichen Proben oder zusätzliche Tests sind erforderlich, um in diesem Fall eine Tollwut auszuschließen. Die Identifizierung von Lyssavirus-RNA in einem Gewebe ist jedoch diagnostisch für eine Tollwutinfektion 3,6. Beispiele für Proben, die auf Tollwutvirus-RNA getestet werden können, um eine Tollwutinfektion auszuschließen oder zu überwachen (aber nicht auszuschließen), sind Kortex, Hippocampus, Rückenmark, degradierte Proben, Haut, Speichel und Hornhaut. Eine qualitative Beurteilung des Zustands jeder Probe sollte bei der Ankunft im Labor erfolgen. Durch Kühlung kann eine Probe mindestens 72 Stunden aufbewahrt werden, sollte aber nicht langfristig verwendet werden. Wiederholte Frost-Tau-Zyklen können die Testempfindlichkeit verringern, und mehr als fünf Frost-Tau-Zyklen sollten vermieden werden. Wenn der Zustand des Gewebes eine sichere Identifizierung von Hirnstrukturen verhindert, sollte die Probe als unbefriedigend identifiziert werden. Im Falle einer unbefriedigenden Probe kann immer noch getestet werden, um Tollwut auszuschließen (aber nicht auszuschließen). Positive Testergebnisse werden als solche gemeldet. Negative oder nicht schlüssige Ergebnisse bei unbefriedigendem Gewebe sollten als nicht schlüssig gemeldet werden, um eine Fehlinterpretation als negative Diagnose zu vermeiden.
Dieses Protokoll wurde auf der Grundlage der veröffentlichten Verfahren 7,8,9 entwickelt und enthält aktualisierte Primer, die auf die Leader-Region des Lyssavirus-Genoms und die Nukleoprotein-kodierende Sequenz abzielen. Die Sonde zielt auf eine kurze, hochkonservierte Sequenz ab und verwendet gesperrte Nukleotide, um eine breite Detektion zu ermöglichen. Der Assay weist RNA von verschiedenen Lyssaviren in unterschiedlichen Konzentrationennach 8. Dieses Protokoll demonstriert die Laborverfahren zur Durchführung des LN34-Echtzeit-PCR-Assays, aber der genaue und empfindliche Nachweis von Lyssavirus-RNA hängt von anderen Elementen ab, die in diesem Protokoll nicht ausführlich behandelt werden, wie z. B. Probenlagerung, Aufzeichnungen, Personalschulung/-kompetenz, Ergebnisverfolgung, Ergebnisinterpretation, Qualitätssicherung, Laborsicherheitsmaßnahmen und Fehlerbehebung. PCR-basierte Assays sind aufgrund ihrer hohen Sensitivität anfällig für Kreuzkontaminationen. Kreuzkontaminationen können vermieden werden, indem gute Laborpraktiken eingehalten werden, wie z. B. häufiges Wechseln der Handschuhe, Umgang mit einer Probe nach der anderen, Desinfektion von Arbeitsoberflächen mit wirksamen Dekontaminationsmitteln zwischen den Proben und Halten der Röhrchen geschlossen und Proben von PCR-Reagenzien getrennt. PCR-Reagenzien und Proben können leicht getrennt werden, indem ein einseitiger Arbeitsablauf verwendet und Arbeitsbereiche vor und nach der Amplifikation getrennt werden. Bereiten Sie beispielsweise PCR-Mastermixe an einem Ort vor, der physisch von dem Ort getrennt ist, an dem die Proben gehandhabt werden. Wechseln Sie die Handschuhe häufig, um eine Kontamination der PCR-Reagenzien mit Proben, Rückständen oder Positivkontroll-RNA zu vermeiden. Die PCR-Platte oder -Röhrchen sollten nach der Zugabe des Mastermixes an eine zweite Stelle gebracht werden, an der die Probe und die Kontroll-RNA hinzugefügt werden können. Wichtig ist, dass PCR-Produkte nicht in Bereichen manipuliert werden sollten, in denen Proben oder Mastermixe hergestellt werden.
Es gibt keinen Ersatz für praktisches Üben und Erfahrung bei der Durchführung von diagnostischen Tests. Alle neuen Mitarbeiter sollten geschult werden, und das Testpersonal sollte mindestens einmal pro Jahr gemäß den Anforderungen des zuständigen Laborleiters auf seine Kompetenz hin bewertet werden. Jegliche Beobachtungen ungewöhnlicher Ergebnisse oder Untersuchungsfehler sollten sofort notiert, untersucht und korrigiert werden. Jede neue Charge von Reagenzien sollte anhand von Proben mit bekannten Ct-Werten validiert werden (z. B. eine Positivkontrolle oder eine archivierte Probe). Alle Geräte sollten routinemäßig gewartet werden, wie vom Hersteller empfohlen, und die Leistung der Assays sollte nach jeder Wartung oder Reparatur überprüft werden. Die Temperaturniveaus sollten an den entsprechenden Geräten überwacht werden, um sicherzustellen, dass Kühl- und Gefriergeräte innerhalb der Kriterien für einen akzeptablen Temperaturbereich für Reagenzien bleiben, die für diagnostische Tests verwendet werden.
Verfahrensänderungen können die Assay-Leistung beeinträchtigen und zu falsch positiven, falsch negativen oder nicht interpretierbaren Ergebnissen führen. Die Empfehlungen sollten genau befolgt werden, um eine hohe Sensitivität und Spezifität des Assays zu gewährleisten. Ein Laboratorium, das Änderungen an diesem Protokoll vornehmen möchte, sollte die geänderten Methoden in Absprache mit der CDC validieren und bestätigen.
Protokoll
Postmortale Hirngewebeproben wurden im Rahmen routinemäßiger Überwachungs- oder Diagnoseaktivitäten der Pockenvirus- und Tollwutabteilung (CDC; Atlanta, GA, USA).
1. Entnahme von Hirngewebe für die postmortale Diagnose von Tollwut bei Tieren mit dem LN34-Pan-Lyssavirus-Echtzeit-RT-PCR-Assay
HINWEIS: Die Proben können Infektionserreger enthalten. Tragen Sie geeignete persönliche Schutzausrüstung (PSA) (schwere Gummihandschuhe oder andere schnittfeste Handschuhe, Laborkittel, wasserdichte Schürze, chirurgische Maske, Stiefel, Schutzhüllen und Gesichtsschutz) und befolgen Sie die erforderlichen Sicherheitsvorschriften für die Verwendung, Lagerung und Entsorgung von Proben. Eine Präexpositions-Tollwutimpfung, regelmäßige serologische Tests und Auffrischungsimpfungen (falls erforderlich) sind für jedermann erforderlich, der mit Lyssaviren oder bekannten oder potenziell infizierten Proben arbeitet, diese testet, herstellt oder Forschungsaktivitäten durchführt 2,3,4,6,10.
- Beschriften Sie ein Probenentnahmeröhrchen pro Probe mit einem Zugangsetikett. Füllen Sie jedes Probenentnahmeröhrchen mit 1 ml TRIzol-Reagenz oder einem anderen Homogenisierungspuffer und einer Portion MagNA Lyser-Kügelchen (im Folgenden "Keramikkügelchen"). Um Keramikkügelchen hinzuzufügen, gießen Sie vorsichtig aus dem Kügelchenröhrchen in das Probenentnahmeröhrchen. Röhrchen mit Keramikkügelchen enthalten in der Regel genügend Kügelchen für 2-5 Proben, wobei mindestens 20 Kügelchen mit einem Durchmesser von 1,4 mm pro Probe verwendet werden.
ACHTUNG: TRIzol-Reagenz (im Folgenden "Homogenisierungspuffer") ist eine gefährliche Chemikalie; Bei Kontakt mit Säuren oder Bleichmitteln werden giftige Gase freigesetzt; für eine ausreichende Belüftung zu sorgen; Weitere Informationen finden Sie im Sicherheitsdatenblatt. Wenn Benutzer das TRIzol-Reagenz oder das TRI-Reagenz durch einen anderen Homogenisierungspuffer ersetzen, ist eine zusätzliche Validierung erforderlich. TRIzol fungiert als Probenhomogenisierungs-/Lysepuffer, Lyssavirus-Inaktivierungspuffer und RNA-Stabilitätspuffer für dieses Protokoll. Die Verwendung eines alternativen Homogenisierungspuffers erfordert die Validierung der Extraktionseffizienz, Inaktivierung und Stabilität in einem kontrollierten Vergleich nebeneinander. - Reinigen und desinfizieren Sie die Arbeitsfläche 2 Minuten lang mit einem Desinfektionsmittel für quartäre Ammoniumverbindungen (QAC) und legen Sie ein mit Kunststoff ausgekleidetes Saugkissen aus. Legen Sie nur Reagenzien und Verbrauchsmaterialien für die erste Probe in eine biologische Sicherheitswerkbank der Klasse II (BSC) mit Funktionen zur Ableitung gefährlicher Dämpfe außerhalb des Raums.
HINWEIS: Beachten Sie die Anleitung des Herstellers für die Lagergrenzen von verdünntem QAC. Stellen Sie sicher, dass das mit Kunststoff ausgekleidete Pad den Luftstrom der biologischen Sicherheitswerkbank nicht blockiert. Wenn der Luftstrom gestört ist, verwenden Sie kein Pad. - Entnehmen Sie Gewebe, das einen vollständigen Querschnitt des Hirnstamms und des Kleinhirns darstellt, mit einem sauberen Einwegskalpell.
HINWEIS: Die Manipulation von Geweben sollte so durchgeführt werden, dass keine Flüssigkeiten vernebelt oder Partikel in der Luft erzeugt werden. Abzüge oder Biosicherheitswerkbänke sind nicht erforderlich, aber belüftete Biosicherheitswerkbänke werden empfohlen, da sie zusätzlichen Schutz vor Gerüchen, Dämpfen, Ektoparasiten und Knochenfragmenten bieten.
VORSICHT: Die Verwendung eines Skalpells mit Material, das möglicherweise mit dem Lyssavirus infiziert ist, ist gefährlich, und Benutzer sollten angemessene Sicherheitsvorkehrungen treffen. Die Verwendung einer Einmalzange wird empfohlen.- Bei Kleintieren (wie Fledermäusen) kann der gesamte Hirnstamm und das Kleinhirn entnommen werden.
- Bei größeren Tieren entnehmen Sie einen vollständigen Querschnitt des Hirnstamms und des Gewebes von jedem der drei Lappen des Kleinhirns.
- OPTIONAL: Wenn Sie den direkten Fluoreszenz-Antikörper-Test (DFA) durchführen, sammeln Sie an dieser Stelle Abdrücke vom Gehirn. Verwenden Sie das Gewebe, das nach der Entnahme von Hirnabdrücken für DFA übrig bleibt, für die RNA-Extraktion und den Test mit dem LN34-Assay.
HINWEIS: Wenn TRIzol zu Proben hinzugefügt wird, können Proben nicht mehr für antigenbasierte Nachweismethoden oder die Virusisolierung verwendet werden.
- Bereiten Sie Proben für die Homogenisierung und RNA-Extraktion vor.
HINWEIS: Die Effizienz der RNA-Extraktion und der Virusinaktivierung kann durch die Verwendung von zu viel Gewebe beeinträchtigt werden. Die Gewebemenge sollte etwa 1/10 des Volumens des verwendeten Homogenisierungspuffers nicht überschreiten. Wenn mehr Gewebe verwendet wird, erhöhen Sie die Menge des Homogenisierungspuffers entsprechend, um eine effiziente und erfolgreiche RNA-Extraktion zu gewährleisten.- Bei Kleintieren geben Sie alle erforderlichen Gewebe in ein Röhrchen mit Homogenisierungspuffer und Kügelchen für die Extraktion. 100 mg Probe in 1 ml Homogenisierungspuffer dürfen nicht überschritten werden. Erhöhen Sie bei größeren Proben das Volumen des Homogenisierungspuffers oder verwenden Sie mehrere Röhrchen, um eine Probe im Verhältnis 1:10 widerzuspiegeln: Puffer.
- Bei größeren Tieren das Gewebe gründlich zerkleinern und homogenisieren und einen repräsentativen Teil in ein mit Homogenisierungspuffer und Kügelchen vorgefülltes Röhrchen geben. 100 mg Probe in 1 ml Homogenisierungspuffer dürfen nicht überschritten werden. Erhöhen Sie bei größeren Proben das Volumen des Homogenisierungspuffers oder verwenden Sie mehrere Röhrchen, um eine Probe im Verhältnis 1:10 widerzuspiegeln: Puffer.
- Option 1 (Bead Beater): Homogenisieren Sie das Gewebe mit einem Bead Beater, 1 ml Puffer und Keramikkügelchen. Möglicherweise müssen mehrere 2-ml-Röhrchen oder größere Röhrchen verwendet werden.
- Option 1 (Bead Beater): Reinigen und desinfizieren Sie den Arbeitsplatz, die Geräte und die äußeren Probenröhrchen mit QAC-Desinfektionsmittel (1:256). 2 min ziehen lassen.
- Option 1 (Rührbesen): Beladen Sie im Inneren des BSC einen Zentrifugenrotor mit homogenisierten Proben. Zentrifugieren Sie alle Proben(n) bei 10.000-16.000 × g für 2 Minuten in einer Tisch-Mikrozentrifuge. Entladen Sie den Zentrifugenrotor im Inneren des BSC.
- Option 1 (Rührer): 2 Min. ziehen lassen.
- Option 1 (Bead Beater): Übertragen Sie 120 μl Homogenat in ein mit 1 mL Homogenisierungspuffer vorgefülltes Röhrchen.
ACHTUNG: Die Homogenisierung kann Aerosole erzeugen und sollte in einem BSC durchgeführt werden. - Option 2 (Skalpell): Das erforderliche Gewebe mit einem Einwegskalpell fein hacken, mit einem Tupfer abstrichen und den Tupfer in ein mit Homogenisierungspuffer und Kügelchen vorgefülltes Röhrchen geben. 100 mg Probe in 1 ml Homogenisierungspuffer dürfen nicht überschritten werden. Erhöhen Sie bei größeren Proben das Volumen des Homogenisierungspuffers oder verwenden Sie mehrere Röhrchen, um ein Verhältnis von Probe zu Puffer von 1:10 widerzuspiegeln.
VORSICHT: Die Verwendung eines Skalpells mit Material, das möglicherweise mit dem Lyssavirus infiziert ist, ist gefährlich, und Benutzer sollten angemessene Sicherheitsvorkehrungen treffen.
- Sammeln Sie alle verbleibenden Taschentücher in den Originalbehälter oder in einen neuen, leeren Behälter, der mit einem Zugangsetikett gekennzeichnet ist. Bewahren Sie dieses Gewebe für den Fall auf, dass eine erneute Untersuchung oder zusätzliche Charakterisierung erforderlich ist.
- Reinigen und desinfizieren Sie den Arbeitsplatz, die Ausrüstung und die Außenseite der Probenröhrchen mit dem QAC-Desinfektionsmittel 1:256. 2 min ziehen lassen.
- Wiederholen Sie die Schritte 1.2-1.5 für alle verbleibenden Proben.
- Homogenisieren Sie die Proben mit einem Mini-Rührbesen für mindestens 60 s. Überprüfen Sie die Rohre visuell. Wiederholen Sie den Rührer für weitere 60 s, wenn noch große Gewebestücke übrig sind. Dieser Schritt ist fakultativ, wenn das Gewebe in Schritt 1.4.2.1 vollständig homogenisiert wurde.
HINWEIS: Es ist darauf zu achten, dass das Gewebe gründlich homogenisiert wird. Eine unvollständige Homogenisierung verringert die RNA-Ausbeute. - Mindestens 5 Minuten bei Raumtemperatur (RT) ziehen lassen.
- Reinigen und desinfizieren Sie den Arbeitsplatz, die Ausrüstung und die Außenseite der Probenröhrchen mit QAC-Desinfektionsmittel (1:256).
HINWEIS: Die Probe gilt zu diesem Zeitpunkt als nicht infektiös und kann aus dem Tollwutlabor entnommen werden. - Verarbeiten Sie die Proben sofort in Homogenisierungspuffer für die RNA-Extraktion, lagern Sie sie mehrere Tage lang bei RT (20 °C bis 25 °C) oder gekühlt (4 °C bis 8 °C) oder lagern Sie sie bei -16 °C oder kälter.
2. Protokoll für die RNA-Extraktion mit dem RNA MiniPrep Kit
- Richten Sie den Arbeitsbereich unter dem BSC ein.
- Reinigen Sie die BSC-Arbeitsfläche vor Beginn der Arbeiten mit 70 % Ethanol, um Staub oder andere Umweltverunreinigungen zu entfernen. Führen Sie eine zusätzliche Oberflächendekontamination mit QAC-Desinfektionsmittel (1:256), RNase AWAY oder RNaseZap (gemäß den Empfehlungen des Herstellers) durch.
- Legen Sie eine mit Kunststoff ausgekleidete saugfähige Arbeitsunterlage aus und legen Sie Reagenzien, Verbrauchsmaterialien und die Probe in die BSC.
HINWEIS: Stellen Sie sicher, dass das mit Kunststoff ausgekleidete Pad den Luftstrom des BSC nicht blockiert. Wenn der Luftstrom gestört ist, verwenden Sie kein Pad. - Legen Sie alle Entnahmeröhrchen in ein sauberes Gestell für Mikrozentrifugenröhrchen. Füllen Sie ein 1,5-ml-Mikrozentrifugenröhrchen mit 300 μl 100 % Ethanol für jede Gehirnprobe, die nicht von Fledermäusen stammt. Bei Proben mit wenig Gewebe (Fledermaushirnprobe, Nicht-Gehirnprobe oder geschädigte Probe) füllen Sie jeweils ein 1,5-ml-Mikrozentrifugenröhrchen mit 600 μl 100 % Ethanol vor.
- Probenvorbereitung
- Alle in Abschnitt 1 vorbereiteten Proben werden in einem Röhrchengestell im BSC gesammelt. Tauen Sie alle gefrorenen Proben kurz vor dem Test auf.
- Tauen Sie eine Extraktionskontrolle auf.
HINWEIS: Es wird empfohlen, eine Probe ohne Lyssavirus-RNA zu wählen. Die Probe sollte zuvor mit einem erwarteten Ct-Wertbereich für den Beta-Aktin-Assay getestet werden. Zum Beispiel präaliquotierte Gewebekulturzellen oder ein zuvor getesteter negativer Tollwutfall (Mensch oder Tier).
- Zentrifugieren Sie alle Proben bei 10.000-16.000 × g für 2 m in einer Tischmikrozentrifuge.
- Den Überstand in ein neues steriles Mikrozentrifugenröhrchen mit 100 % Ethanol überführen. Stellen Sie sicher, dass der Überstand klar ist, ohne offensichtliche Lipide oder festes Gewebe. Vermeiden Sie die Ansammlung von Lipiden und festem Gewebe.
- Für Hirngewebe, das nicht von Fledermäusen stammt: 300 μl Überstand übertragen.
- Bei Proben mit wenig Gewebe (Fledermaushirnprobe, Nicht-Gehirnprobe oder verschlechterte Probe) werden 600 μl Überstand übertragen.
- Das restliche Homogenat wird in einem Mikrozentrifugenröhrchen mit Schraubverschluss bei ≤-16 °C gelagert.
- Pipettieren Sie zum Mischen 10 Mal auf und ab.
- Für jede Probe werden 600 μl des Ethanol-Überstands-Gemisches in eine Spin-Säule in einem Sammelröhrchen überführt.
- Zentrifugieren, bis die Flüssigkeit die Säule passiert hat (1 min bei 10.000-16.000 × g). Verwerfen Sie den Durchfluss.
- Wiederholen Sie den Vorgang, wenn mehr als 600 μl Ethanol-Homogenisierungspuffer-Gemisch für eine Probe vorhanden sind.
- Übertragen Sie jede Säule in ein neues Entnahmeröhrchen.
- Geben Sie 400 μl RNA-Prewash-Puffer in jede Säule und zentrifugieren Sie 30 s lang bei 10.000-16.000 × g .
- Entsorgen Sie den Durchfluss und geben Sie jede Säule wieder in dasselbe Auffangröhrchen zurück.
- Wiederholen Sie die Schritte 2.10-2.11.
- Geben Sie 700 μl RNA-Waschpuffer in jede Säule und zentrifugieren Sie 2 Minuten lang bei 10.000-16.000 × g . Stellen Sie sicher, dass der Waschpuffer vollständig durch jede Säule gelaufen ist.
- Übertragen Sie jede Säule vorsichtig in ein RNase-freies Röhrchen.
- Die Durchflussdurchführung und das Auffangrohr von Nummer 2.13 entsorgen.
- Geben Sie 50 μl DNase/RNase-freies Wasser direkt in die Säulenmatrix, um RNA zu eluieren.
HINWEIS: Berühren Sie die Säulenmatrize nicht mit der Pipettenspitze. - 30 s bei RT inkubieren, dann 1 min bei 10.000-16.000 × g zentrifugieren.
- Übertragen Sie die RNA vorsichtig in ein neues, mit Schraubendeckel und flachem Boden gekennzeichnetes Mikrozentrifugenröhrchen. Bringen Sie die extrahierte RNA für sofortige Tests auf Eis. Langfristig bei -70 °C oder kälter lagern.
HINWEIS: Die Lagerung bei wärmeren Temperaturen oder wiederholtes Einfrieren und Auftauen kann zum RNA-Abbau führen und die diagnostischen Ergebnisse beeinträchtigen.
3. Protokoll für den LN34-Pan-Lyssavirus-Echtzeit-RT-PCR-Assay
- Bereiten Sie die Reagenzien vor.
- Künstliche Positivkontrolle
- Wenn eine künstliche Positivkontroll-RNA von CDC8 hergestellt wird, befolgen Sie die Anweisungen auf der Verpackung für die Lagerung, Rekonstitution und Aliquotierung. Überspringen Sie diesen Schritt, wenn bereits Einmal-Aliquots der Positivkontroll-RNA zur Hand sind.
HINWEIS: Positivkontroll-RNA in Arbeitskonzentrationen sollte im Bereich der Template-Zugabe und nicht im selben Bereich wie die Mastermix-Zubereitung gehandhabt werden. Positivkontroll-RNA sollte einen Zyklusschwellenwert (Ct) innerhalb des erwarteten Bereichs erzeugen, der für eine bestimmte Charge bestimmt wurde. Zwischen den Durchläufen sollte sich der LN34-Ct-Wert für die Positivkontroll-RNA nicht um mehr als ±1,5 Ct-Werte unterscheiden. - Tauen Sie ein Einmal-Aliquot aus ≤ -70 °C Lagerung kurz vor der Verwendung auf Eis oder Eisblock auf. Aliquote nicht einfrieren und auftauen und über einen längeren Zeitraum bei Kühltemperaturen aufbewahren.
HINWEIS: Eine Positivkontrolle sollte in dreifacher Ausfertigung im LN34-Assay durchgeführt werden. Die künstliche Positivkontrolle8 wird im Beta-Aktin-Assay nicht amplifizieren.
- Wenn eine künstliche Positivkontroll-RNA von CDC8 hergestellt wird, befolgen Sie die Anweisungen auf der Verpackung für die Lagerung, Rekonstitution und Aliquotierung. Überspringen Sie diesen Schritt, wenn bereits Einmal-Aliquots der Positivkontroll-RNA zur Hand sind.
- Extraktionskontrolle und Proben: Legen Sie frisch extrahierte Proben unmittelbar vor der Verwendung auf Eis (oder Eisblock) oder tauen Sie Proben aus ≤ -70 °C Lagerung auf Eis (oder Eisblock) auf.
HINWEIS: RNA sollte aufgetaut und in einem Bereich verarbeitet werden, der für die Proben- oder Template-Zugabe vorgesehen ist und von den Bereichen getrennt ist, die für die Mastermix-Vorbereitung oder die Manipulation von PCR-Produkten oder großen Mengen an viralem Material verwendet werden (z. B. Erzeugung von Positivkontrollen, Virusvermehrung)
- Künstliche Positivkontrolle
- Bereiten Sie die Mastermix-Reagenzien im Mastermix-Vorbereitungsbereich vor.
- Mastermix-Vorbereitung des Singleplex LN34 RT-PCR Assays
HINWEIS: Benutzer können Beispiele im Singleplex- (Schritt 3.2.1) oder Multiplex-Format (Schritt 3.2.2) testen. Es ist nicht erforderlich, sowohl 3.2.1 als auch 3.3.2 auszuführen. Die Mastermix-Vorbereitung, die Aliquotierung von Primern und Sonden sowie keine Reagenzien für die Template-Kontrolle sollten in einem sauberen Bereich aufgetaut und manipuliert werden, der von der Probenverarbeitung, der Autopsie, der PCR und anderen Bereichen, in denen virales Material manipuliert wird, getrennt ist. Dies kann durch getrennte Räume oder ein Schranksystem mit einseitigem Probenfluss erreicht werden.- Erzeugen Sie Primer- und Sondenmischungen in den in Tabelle 1 und Tabelle 2 angegebenen Arbeitskonzentrationen. Überspringen Sie diesen Schritt, wenn die Arbeitsverdünnungsaliquote von Primern und Sonden bereits zur Hand sind.
- Aliquotieren Sie Primer und Sonden in 1,5 mL Lagerbestände und 50 μl Arbeitsmaterialien und lagern Sie sie bei ≤ -16 °C im Dunkeln. Überspringen Sie diesen Schritt, wenn die Arbeitsverdünnungsaliquote von Primern und Sonden bereits zur Hand sind.
HINWEIS: Es wird empfohlen, Schritt 3.2.1.2 auszuführen. - Tauen Sie einstufigen RT-PCR-Puffer, keine Template-Kontrolle, Primer und Sonden aus ≤ -16 °C Lagerung auf Eis oder Eisblock im Mastermix-Vorbereitungsbereich auf.
HINWEIS: Verwenden Sie Reagenzien bis zum Verfallsdatum oder bis zum Leistungsfehler, je nachdem, was zuerst eintritt. - Alle Puffer, Primer und Sonden vor der Verwendung kurz vortexen und zentrifugieren.
- Lagern Sie das einstufige RT-PCR-Enzym bis zur Verwendung auf Eis oder in einem Eisblock.
- RNA-Proben
HINWEIS: Verwenden Sie nach Möglichkeit frisch extrahierte RNA, da Frost-Tauwetter die Leistung beeinträchtigen kann - Lagern Sie RNA-Proben bis zur Verwendung auf Eis oder Eisblock.
- Tauen Sie gefrorene RNA-Proben auf Eis oder einem Eisblock auf.
- Markieren Sie ein Mikrozentrifugenröhrchen pro Assay (LN34 und βA).
- Bestimmen Sie die Anzahl der Reaktionen (N), die pro Assay eingerichtet werden sollen.
- Berechnen Sie die Anzahl der Reaktionen für den LN34-Assay, indem Sie die Anzahl der Proben mit 3 multiplizieren und 6 für Kontrollreaktionsvertiefungen plus 10 % zusätzliche Reaktionen addieren, um den beim Pipettieren verlorenen Volumen zu berücksichtigen. (z.B. Für 10 Proben: (10 x 3) + 6 = 36 Reaktionen; Überreaktionen: (36 x 0,1) + 36 = 3,6 + 36 = 39,6 Reaktionen insgesamt, oder 40 Reaktionen aufgerundet)
HINWEIS: Für klinische Tests wird empfohlen, alle Proben in dreifacher Ausfertigung auf LN34 zu testen. Zu Überwachungszwecken kann jede Probe in zweifacher Ausfertigung untersucht werden. Es wird empfohlen, während des ersten Assay-Onboardings dreifach zu verwenden, um eine geringe Variabilität zwischen den Replikaten und eine gute Technik zu gewährleisten. - Berechnen Sie die Anzahl der Reaktionen für den βA-Assay, indem Sie die Anzahl der Proben plus 4 Kontrollreaktionsvertiefungen plus 10 % zusätzliche Reaktionen addieren, um den beim Pipettieren verlorenen Volumen zu berücksichtigen.
- Bestimmen Sie das Volumen jedes Reagenzes für die LN34- und βA-Mastermixe anhand von Tabelle 2.
- Bestimmen Sie die Vertiefungen für jede zu testende Probe in dreifacher Ausfertigung im LN34-Assay und vereinzeln Sie sie für den βA-Assay unter Verwendung einer 96-Well-Plattenkarte.
- Geben Sie 23 μl LN34-Assay-Mastermix in jedes LN34-Gut, nachdem Sie es kurz vortexen und 30 s lang heruntergeschleudert haben, wobei Sie eine Tischmikrozentrifuge verwendet haben, um Flüssigkeit am Boden des Röhrchens zu sammeln. Vermeiden Sie die Einführung von Blasen.
- Geben Sie 23 μl βA-Assay-Mastermix in jede βA-markierte Welle, nachdem Sie kurz vortext und 30 s lang in einer Tischmikrozentrifuge heruntergeschleudert wurden, um Flüssigkeit am Boden zu sammeln.
- Richten Sie die NTC-Reaktionen (No Template Control) ein, indem Sie 2 μl Wasser in PCR-Qualität in jede NTC-Vertiefung pipettieren.
- Decken Sie die Vertiefungen ab und übertragen Sie die Platte in den Bereich der Schablonenaddition.
- Die Röhrchen mit den RNA-Proben kurz vortexen und zentrifugieren.
- Pipettieren Sie 2 μl extrahierte RNA aus der ersten Probe in jede Vertiefung, die für diese Probe markiert ist. Vermeiden Sie die Einführung von Blasen.
- Stellen Sie sicher, dass die RNA durch Visualisierung in die Pipette gezogen wurde.
- Pipettieren Sie an der Seite der Vertiefung, um sicherzustellen, dass die Probe in die richtige Vertiefung gegeben wird.
- Vermeiden Sie es so weit wie möglich, Pipettenspitzen, die RNA enthalten, über offene Vertiefungen zu schwenken.
- Schritt 3.2.10 für die verbleibenden Proben und die Positivkontroll-RNA wiederholen.
- Legen Sie die optische Klebeabdeckung über die Vertiefungen, nachdem Sie alle Proben und Kontrollen hinzugefügt haben. Achten Sie darauf, alle Vertiefungen abzudecken und vollständig abzudichten.
- Zentrifugieren Sie bei 500 × g für 1 min bei RT in einer Tischzentrifuge oder verwenden Sie eine Salatschleuder.
- Legen Sie die versiegelte Platte in ein real-time PCR-Gerät, das für FAM- und VIC/HEX-Reporterfarbstoffe kalibriert ist, und stellen Sie es auf die in Tabelle 3 gezeigten Zyklusparameter ein.
- Bereiten Sie Mastermixe für den LN34 Multiplexed (LN34M) Assay vor.
- Ein Mikrozentrifugenröhrchen LN34M gemäß Tabelle 2 beschriften.
- Bestimmen Sie die Anzahl der Reaktionen (N), die pro Assay eingerichtet werden sollen.
- Berechnen Sie die Anzahl der Reaktionen für den LN34M-Assay, indem Sie die Anzahl der Proben mit 3 multiplizieren und 6 für Kontrollreaktionsvertiefungen plus 10 % zusätzliche Reaktionen addieren, um den beim Pipettieren verlorenen Volumen zu berücksichtigen. (z. B. Für 10 Proben: (10 x 3) + 6 = 36 Reaktionen; Überreaktionen: (36 x 0,1) + 36 = 3,6 + 36 = 39,6 Reaktionen insgesamt oder 40 Reaktionen aufgerundet)
- Wählen Sie entweder das 25-μl- oder das 12,5-μl-Format. Bestimmen Sie das Volumen jedes Reagenzes für den LN34M-Mastermix anhand von Tabelle 2.
- Bestimmen Sie die Vertiefungen für jede Probe, die im LN34M-Assay in dreifacher Ausfertigung getestet werden soll, unter Verwendung einer 96-Well-Plattenkarte.
- Geben Sie die Reagenzien für den LN34M-Assay in die Wells ab. Röhrchen kurz vortexen und nach unten drehen, um Flüssigkeit am Boden zu sammeln, bevor 23 μl (für eine 25 μl Reaktion) oder 10,5 μl (für eine 12,5 μl Reaktion) Mastermix in jede zugewiesene Vertiefung gegeben werden. Vermeiden Sie die Einführung von Blasen.
- Richten Sie die NTC-Reaktionen ein, indem Sie 2 μl Wasser in PCR-Qualität in jede NTC-Vertiefung pipettieren.
- Decken Sie die Vertiefungen ab und übertragen Sie die Platte in den Bereich der Schablonenaddition.
- Die Röhrchen mit den RNA-Proben kurz durchwirbeln und nach unten drehen, um Flüssigkeit am Boden zu sammeln.
- Pipettieren Sie 2 μl extrahierte RNA aus der ersten Probe in jede Vertiefung, die für diese Probe markiert ist. Vermeiden Sie die Einführung von Blasen.
- Stellen Sie sicher, dass die RNA in die Pipettenvisualisierung gezeichnet wird.
- Pipettieren Sie an der Seite der Vertiefung, um sicherzustellen, dass die Probe in die richtige Vertiefung gegeben wird.
- Vermeiden Sie es so weit wie möglich, Pipettenspitzen, die RNA enthalten, über offene Vertiefungen zu schwenken.
- Schritt 3.3.8 für die verbleibenden Proben und die Positivkontroll-RNA wiederholen.
- Nachdem Sie die letzte Probe/Kontrolle hinzugefügt haben, legen Sie die optische Klebeabdeckung über die Vertiefungen und stellen Sie sicher, dass alle Vertiefungen vollständig abgedeckt und versiegelt sind.
- Zentrifugieren Sie bei 500 × g für 1 min bei RT in einer Tischzentrifuge oder verwenden Sie eine Salatschleuder.
- Legen Sie die versiegelte Platte in ein real-time PCR-Gerät, das für FAM- und VIC/HEX-Reporterfarbstoffe kalibriert ist, und stellen Sie es auf die Zyklusparameter ein, wie in Tabelle 3 gezeigt. Setzen Sie den passiven Referenzfarbstoff auf ROX und führen Sie ihn im Standardmodus aus (nicht im Schnellmodus)
HINWEIS: Diese Einstellung ist spezifisch für die in diesem Protokoll erwähnten Instrumente und erfordert die Verwendung eines einstufigen RT-PCR-Reagenzes, das ROX als passiven Farbstoff enthält. Alternative Instrumente erfordern unterschiedliche Ansätze, um optimale Laufeinstellungen zu bestimmen. Stellen Sie sicher, dass das Gerät gemäß dem Hersteller normal gewartet wird, um eine optimale Leistung zu erzielen.
- Mastermix-Vorbereitung des Singleplex LN34 RT-PCR Assays
4. Interpretation der Ergebnisse
- Legen Sie eine automatische Baseline und manuelle Schwellenwertberechnungen mit einem Wert von 0,2 für LN34/FAM und 0,05 für βA/HEX/VIC fest.
HINWEIS: Diese Einstellung ist spezifisch für die in diesem Protokoll erwähnten Instrumente und erfordert die Verwendung eines einstufigen RT-PCR-Reagenzes, das ROX als passiven Farbstoff enthält. Alternative Instrumente erfordern unterschiedliche Ansätze zur Berechnung von Ausgangs- und Schwellenwerten. - Bestimmen Sie das Diagnoseergebnis anhand der Anleitung in Tabelle 4 , wenn alle Kontrollen wie erwartet funktioniert haben (Tabelle 5).
- Bestätigen Sie alle Ct- oder Cq-Werte, indem Sie Amplifikationsdiagramme anzeigen.
- Untersuchen Sie alle ungewöhnlichen Ergebnisse wie empfohlen.
5. Aufbewahrung und Lagerung von Proben
- Lagern Sie alle Proben eingefroren bei -16 °C oder niedriger, bis die Tests abgeschlossen sind und die Ergebnisse gemeldet werden. Bewahren Sie das ursprüngliche Gewebe auf, um die Ergebnisse zu bestätigen, oder identifizieren Sie das Wirtstier für die Spezies, falls ungewöhnliche Testergebnisse vorliegen.
- Verwenden Sie eindeutige Probenidentifikatoren; Beschriften Sie alle Röhrchen, Berichte und Unterlagen mit vollständigen, eindeutigen Probenidentifikatoren.
- Zwischenproben (kurzfristig) aufbewahren, falls eine Wiederholungsprüfung erforderlich ist.
- Bewahren Sie bei Bedarf repräsentative positive Proben für die Verwendung als Kontrollen, epidemiologische Typisierung und andere Zwecke auf.
- Lagern Sie RNA für die Langzeitlagerung bei ≤-70 °C.
Ergebnisse
Repräsentative Bilder eines erfolgreichen LN34-Assays, der auf einem ABI ViiA7 real-time PCR-Instrument durchgeführt wurde, sind in Abbildung 2 dargestellt. Die Anzeige der Ergebnisse auf einer logarithmischen Skala ermöglicht eine einfache Anzeige des Ct-Wertes, des Punktes, an dem die Kurve die Schwellenwertlinie überschreitet (Abbildung 2A,C). Bei der Darstellung auf einer linearen Skala wird die erfolgreiche Verstärkung als sigmoidale (oder "S"-förmige) Kurve dargestellt (Abbildung 2B, D), während negative Ergebnisse als gerade, flache Linie dargestellt werden sollten. Es wird empfohlen, die Ergebnisse sowohl in linearen als auch in logarithmischen Maßstabsansichten anzuzeigen, um mögliche Anomalien oder Fehler zu identifizieren. Typische positive und negative Ergebnisse in der Mehrkomponenten-Diagrammansicht sind in Abbildung 2E, F zu sehen, wo das Fluoreszenzniveau des Farbstoffs, der die Sonde markiert (FAM für LN34, VIC/HEX für βA), relativ zum passiven Farbstoff im Reaktionspuffer (ROX) beobachtet werden kann.
Beispiele für abnormale Ergebnisse sind in Abbildung 3 dargestellt. Vergleiche zwischen den Diagrammen erfolgreicher Läufe (Abbildung 2) und abnormalen Diagrammen (Abbildung 3) können verwendet werden, um atypische Läufe und Geräteprobleme zu isolieren. Abbildung 3A zeigt ein Signal, das den Schwellenwert überschreitet und einen Ct-Wert für LN34 erzeugt, aber die Verstärkungskurve ist sehr untypisch und steigt linear an. Das Mehrkomponentendiagramm (Abbildung 3B) zeigt auch eine Wellenlinie, die für eine positive Stichprobe nicht typisch ist. Dieses Beispiel zeigt, wie wichtig es ist, die Verstärkungsdiagramme anzuzeigen und nicht einfach Ct-Werte zu kopieren. Stellen Sie immer sicher, dass die Amplifikationskurven für alle Proben normal aussehen. Es wird auch empfohlen, das Diagramm mit mehreren Komponenten zu betrachten, um sicherzustellen, dass keine Unregelmäßigkeiten vorhanden sind. Gelegentlich können unübersichtliche Basissignale Ct-Werte erzeugen, wenn keine Verstärkung stattgefunden hat. Wenn Verstärkungssignale linear erscheinen, wird empfohlen, die Basislinie anzupassen, um zu sehen, ob die Kurve verschwindet. Im Falle eines ungewöhnlichen Signals sollte der gesamte Lauf wiederholt werden. Es wird empfohlen, Ihr real-time PCR-Gerät zu reinigen und eine Hintergrundplatte laufen zu lassen, wenn die Probleme weiterhin bestehen. Falls verfügbar, können PCR-Produkte auf einem Agarose-Gel laufen und/oder sequenziert werden, um ungewöhnliche Ergebnisse zu beheben. Es wird nicht empfohlen, die Ergebnisse der Gelelektrophorese oder Sequenzierung zur Bestimmung der diagnostischen Ergebnisse zu verwenden.
Frühere Studien haben eine geringe Variabilität zwischen Replikaten, Assay-Lauf, Bediener und Labor für den LN34-Assaygezeigt 7. Wenn eine hohe Variabilität (>±1,5 ct Unterschied) zwischen den Replikaten derselben Probe beobachtet wird, sollte diese RNA erneut getestet werden. Eine hohe Variabilität kann durch Probleme mit Pipetten, Laborpraktiken, falschem Pipettieren oder Echtzeit-PCR-Geräten verursacht werden. Die wiederholte Beobachtung einer hohen Variabilität über mehrere Proben oder über Testläufe hinweg kann auf systemische Probleme hinweisen. Proben mit niedriger RNA, die sich der Assay-Schwelle für eine positive Probe (Ct 35) nähern, können eine höhere Variabilität der Ct-Werte zwischen den Replikaten aufweisen. Eine Rücksprache mit der CDC und eine Fehlerbehebung können erforderlich sein, um die Ursache für die anhaltende Variabilität, die inkonsistenten Ergebnisse oder das Versagen des Assays zu beheben.
Die hohe Sensitivität von PCR-basierten Assays macht sie von Natur aus anfällig für Kontaminationen. Die strikte Einhaltung guter Laborpraktiken ist der beste Weg, um Kreuzkontaminationen zu mindern. Es ist wichtig zu wissen, wie man potenzielle Kontaminationen erkennt. Eine Reagenzkontamination sollte vermutet werden, wenn keine Vorlagenkontrolle und vermutete negative Probenvertiefungen in einem Assay-Lauf alle ähnliche Ct-Werte ergeben. Wiederholen Sie den Test mit neuen Aliquoten von PCR-Reagenzien (Puffer, Wasser, Primer und Enzym) und derselben RNA. Wenn alle Proben und die Extraktionskontrolle ähnliche CT-Werte ergeben, der NTC jedoch negativ ist, sollte die Kontamination der Extraktionsreagenzien untersucht und die Extraktion mit neuen Reagenzien wiederholt werden. Es empfiehlt sich, kleine Aliquots von Reagenzien zu erstellen, um das Risiko einer Kontamination zu verringern und die Möglichkeit zu vermeiden, dass große Mengen teurer Reagenzien entsorgt werden. Eine Kreuzkontamination der Probe ist schwieriger zu identifizieren. Bei Verdacht auf eine Probenkontamination ist die Probenentnahme beginnend mit dem ursprünglichen Gewebe zu wiederholen. In einigen Fällen kann die Sequenzierung der viralen RNA eine Kontamination bestätigen, insbesondere wenn sich die kontaminierende RNA stark von der erwarteten Virusvariante unterscheidet (z. B. ein im Labor verwendetes Kontrollvirus). Die Sequenzierung von zwei Proben, die gleichzeitig verarbeitet werden, kann feststellen, ob die Virussequenzen identisch sind, kann aber nicht aussagekräftig sein, wenn erwartet wird, dass die Sequenzen sehr ähnlich sind (z. B. dieselbe Variante, die im selben Landkreis gesammelt wurde). Bei Verdacht auf eine Kontamination der Probe mit der Positivkontroll-RNA kann man die LN34-Assay-Amplikons auf einem Agarose-Gel laufen lassen, um die Lyssavirus-RNA (165 bp) von der Positivkontroll-RNA (99 bp) zu unterscheiden. Die Sequenz des Templates, die zur Erzeugung der von CDC8 bereitgestellten Positivkontroll-RNA verwendet wird.
Bei anderen Krankheitserregern können Laboratorien verwendet werden, um den Schwellenwert manuell einzustellen, um "Rauschen" wie die schwache Verstärkung in Cyan in Abbildung 4 zu beseitigen. Diese Praxis wird für die Tollwutdiagnose NICHT empfohlen, da sie zu falsch negativen Ergebnissen mit schlimmen Folgen führen kann, da Tollwut fast zu 100% tödlich ist. Ändern Sie den Schwellenwert NICHT manuell, um negative Ergebnisse für schwache oder späte Amplifikationsproben zu erhalten. Diese Proben müssen erneut entnommen und/oder erneut getestet werden, um Tollwut auszuschließen.
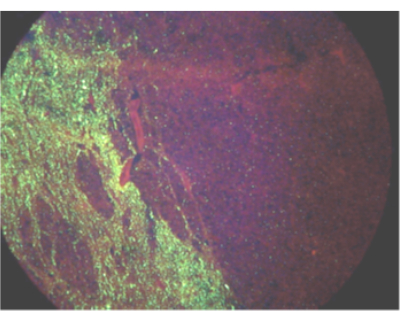
Abbildung 1: Bildfeld, das die einseitige Ausbreitung des Tollwutvirus-Antigens in einem infizierten Esel durch direkten Fluoreszenz-Antikörpertest zeigt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
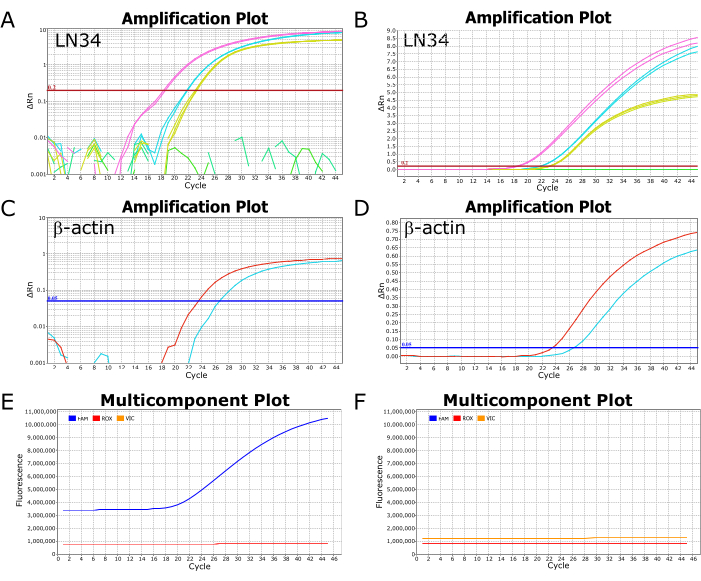
Abbildung 2: Amplifikations- und Mehrkomponenten-Plots aus einem erfolgreichen LN34-Assay-Lauf. (A-D) Die Ergebnisdaten sind auf einer (A,C) logarithmischen Skala und (B,D) auf einer linearen Skala für den LN34- und βA-Assay dargestellt. Die Felder A und B zeigen die LN34-Ergebnisse von zwei Proben (in Rosa und Cyan) im Vergleich zur Positivkontrolle (in Gelb). In Feld B befindet sich eine flache grüne Linie, die eine zusätzliche negative Probe im Lauf darstellt. In A zeigen die grünen Linien keine Verstärkung und werden als unterbrochene Segmente dargestellt. Der Schwellenwert für den LN34-Assay wurde manuell auf 0,2 gesetzt und ist durch die rote horizontale Linie dargestellt. (C,D) Ergebnisse des βA-Assays für zwei Proben (rot und cyan). Der Schwellenwert für den βA-Assay wurde manuell auf 0,05 festgelegt. (E,F) Mehrkomponenten-Plots zeigen die Fluoreszenz (RFU) bei jedem Zyklus für FAM (LN34), VIC (βA) und ROX (passiver Farbstoff im AgPath-ID-Puffer). Die ROX-Werte sollten über alle Zyklen hinweg flach bleiben. Eine typische positive Probe ist in Feld E dargestellt; Die FAM-Fluoreszenz nimmt bei dieser Probe ab Zyklus 18 als sigmoidale Kurve zu. Eine typische negative Stichprobe ist in Panel F dargestellt, bei dem der FAM-Pegel über alle Zyklen hinweg parallel zum ROX-Pegel bleibt. Die Daten stammen von einem ABI ViiA7 Real-Time-PCR-Instrument. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3: Repräsentative Bilder des seltenen, atypischen Signals, das im LN34-Assay auf einem ViiA7 real-time PCR-Instrument beobachtet wurde. (A-F) Amplifikations- (A,C,E) und Mehrkomponenten- (B,D,F) Parzellen, die aufgrund von Bohrlochkontamination erstellt wurden. Die lineare Zunahme (A,C) und die wellenförmigen Fluktuationen (B,D) in der FAM-Fluoreszenz stellen keine echte Verstärkung dar, die auf der Form der Kurven und der Größe der Fluoreszenzänderung basiert. Die Panels A bis D stellen wahrscheinlich negative Stichproben dar, obwohl für das in den Panels A und B gezeigte Replikat ein Ct-Wert erzeugt wurde. Die Felder E und F zeigen ein seltsames wellenförmiges Signal, das im Mehrkomponentendiagramm leichter zu erkennen ist. Diese Art von Signal sollte untersucht werden und kann auf Geräteprobleme hinweisen, obwohl alle Kontrollen in diesem Lauf wie erwartet funktionierten. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
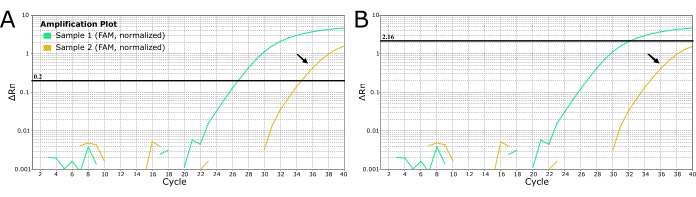
Abbildung 4: LN34 real-time RT-PCR-Kurven von 2 Proben mit Tollwutverdacht zeigen zwei Methoden zur Einstellung von Schwellenwerten. (A) Der LN34-Schwellenwert wurde auf 0,2 gesetzt (empfohlen für alle Durchläufe). (B) Manuelles Bestimmen eines anderen Schwellenwerts für jeden Lauf, um das als "Rauschen" (spätes Verstärkungssignal) ermittelte Signal zu maskieren. Die in Panel B verwendete Methode wird bei Tollwut NICHT empfohlen, da das Fehlen eines wirklich positiven Ergebnisses schwerwiegende Folgen hat. Eine späte Amplifikation kann auf eine schwache positive Probe, eine PCR-Hemmung oder eine fehlgeschlagene Extraktion in einem positiven Fall hinweisen. Es könnte auch auf eine Kreuzkontamination hinweisen. Die goldene Probe (gekennzeichnet durch schwarze Pfeile) ergibt einen Ct-Wert am Cutoff-Ton des Untersuchungsergebnisses und sollte nicht als negativ angesehen werden. Proben mit später Amplifikation sollten erneut extrahiert und erneut getestet werden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Tabelle 1: Primer- und Sondensequenzen und -konzentrationen, die in den Echtzeit-RT-PCR-Assays LN34lys (Singleplex LN34), LN34M (LN34 und βA multiplexed) verwendet wurden. LN34-Sonden werden am 5ʹ-Ende mit dem fluoreszierenden FAM-Farbstoff und am 3ʹ-Ende mit dem Black-Hole-Quencher (BHQ1) markiert. Die βA-Sonde ist am 5ʹ-Ende mit dem fluoreszierenden HEX-Farbstoff und am 3ʹ-Ende mit dem Schwarze-Loch-Quencher (BHQ1) markiert. Gesperrte Nukleotid-modifizierte Basen werden durch ein Plus vor der Base in der Sequenz angezeigt. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Tabelle 2: Assay-Setup für LN34lys-, Actin3- und LN34M-Assays. Namen, Sequenzen und Konzentrationen von Primern und Sonden sind in Tabelle 1 zu finden. LN34_F1 entspricht ACGCTTAACAACCAGATCAAAGAA7. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Tabelle 3: Zyklusparameter für ABI-Instrumente. WICHTIG: Stellen Sie sicher, dass Sie im STANDARD-Modus und nicht im FAST-Modus ausgeführt werden. ROX sollte als passiver Referenzfarbstoff ausgewählt werden. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Tabelle 4: Algorithmus zur Interpretation von LN34 real-time RT-PCR-Ergebnissen für Singleplex- (oben, blaue Tabelle) und Multiplex- (unten, rote Tabelle) Formate. Ein positives LN34-Ergebnis sollte als positiv angesehen werden, auch wenn das βA-Ergebnis negativ oder nicht schlüssig ist. Wenn LN34-Amplikon nicht nachgewiesen wird, muss der βA-Ct-Wert ≤ dem angegebenen Ct-Grenzwert liegen, um als negativ zu gelten. βA Ct-Werte geben Aufschluss über die Qualität der zu untersuchenden Probe und weisen auf eine mögliche Hemmung hin. Eine niedrige Konzentration in der ursprünglichen klinischen Probe kann die βA-Wachstumskurven beeinflussen, was zu keiner erkennbaren Amplifikation führt. Zu den weiteren Faktoren, die zum Scheitern des Nachweises von β-Aktin beitragen, gehören eine schlechte Extraktion von RNA aufgrund von RNA-Verlust oder Verschleppung von PCR-Inhibitoren, falsche Assay-Einrichtung und -Technik, unbefriedigende Probenart oder -qualität sowie Fehlfunktionen von Reagenzien oder Geräten. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Tabelle 5: Maßnahmen und Interpretationen der allgemeinen Ergebnisse für LN34-Assay-Kontrollen. Alle drei Kontrollen (Tollwut-Positivkontroll-RNA, Tollwut-Negativextraktionskontrolle und Kontrolle ohne Schablone) müssen die erwarteten Ergebnisse liefern, damit ein Lauf bestanden wird. Ein Fehlschlagen der Positivkontrolle oder keine Schablonenkontrolle kann auf ein Fehlpipettieren, ein Reagenz oder einen Geräteausfall hinweisen. Der gesamte Lauf, einschließlich aller getesteten RNA-Proben, muss wiederholt werden. Ein Ausfall der Extraktionskontrolle kann auf ein Problem während der Extraktion hinweisen, wie z. B. Reagenzfehler, falsches Pipettieren oder Kreuzkontamination. Die Entnahme aller Proben muss wiederholt werden. Ein Versagen der Kontrollen sollte für erfahrenes Laborpersonal selten sein. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Diskussion
Ein erfolgreicher LN34-Assay-Lauf erfordert eine Positivkontrolle, eine Extraktionskontrolle und keine Template-Kontrollreaktionen, die in jedem Assay-Lauf wie erwartet durchgeführt werden, oder der Lauf muss ungültig gemacht und wiederholt werden. Alle drei LN34-Positivkontroll-Replikatreaktionen sollten den Schwellenwert innerhalb des angegebenen Bereichs überschreiten, oder der Lauf sollte wiederholt werden. Die in früheren Publikationen 7,8 beschriebene Positivkontroll-RNA wird im βA-Assay nicht amplifiziert. Die Kontrollreaktionen ohne Template sollten keine Amplifikationskurven aufweisen, die die Schwellenwertlinie für den LN34- oder den βA-Assay überschreiten. Die Extraktionssteuerung sollte keine Amplifikation für LN34 aufweisen. Wenn in der NTC- oder Extraktionskontrolle eine unerwartete Amplifikation beobachtet wird, kann dies auf eine Kontamination hinweisen und den Lauf und die Wiederholungstests für alle Proben ungültig machen (siehe Tabelle 5). Benutzer können erwägen, zusätzliche Kontrollen hinzuzufügen, einschließlich einer No-Process-Kontrolle oder einer No-Sample-Extraktionskontrolle, um die βA-Kontamination der Extraktionsreagenzien durch den Wirt zu überwachen.
Da sich die Tollwutsterblichkeit 100 % nähert, wird empfohlen, jede schwache oder abnormale Amplifikation weiter zu untersuchen, auch wenn sie keinen Ct-Wert erzeugt. Negative oder NTC-Reaktionen sollten keine Amplifikation aufweisen, und die Fluoreszenz sollte in der Mehrkomponentenansicht als flache Linie parallel zur ROX-Fluoreszenz erscheinen. Beobachtungen von Kurven, insbesondere in mehreren Replikaten, können auf eine Kreuzkontamination oder ein schwaches positives Ergebnis hinweisen. Alle Replikate einer positiven Probe sollten amplifiziert werden, um ein gültiges positives Ergebnis zu erhalten. Wenn nur eine Teilmenge der Replikate in einem der Assays amplifiziert wird, sollte die Probe erneut getestet werden. Darüber hinaus sollte jede Probe, die stark schwankende Ergebnisse liefert (Ct-Wert-Unterschiede > ±1,5 zwischen den Wiederholungen), als ungültig betrachtet und die Probe erneut getestet werden. Wenn das Problem weiterhin besteht, sollte die Probe erneut extrahiert werden.
Es wird erwartet, dass eine tollwutpositive Probe, die aus ordnungsgemäß entnommenem und gelagertem Hirnstamm- und Kleinhirngewebe extrahiert wurde, einen Ct-Wert von weniger als 35 Zyklen für den LN34-Assay aufweist. Alle nicht eindeutigen Proben müssen erneut mittels LN34 real-time RT-PCR getestet werden. Wenn die Probe bei wiederholten Tests nicht schlüssig ist und alle Kontrollen wie erwartet durchgeführt wurden, wird eine erneute Extraktion der RNA empfohlen. Proben mit niedrigem Virus-RNA-Gehalt (LN34 ct > 35) können auf potenzielle Probleme hinweisen, wie z. B. Kontamination, geringe Viruslast, PCR-Hemmung oder fehlgeschlagene Extraktion. Sammeln Sie frische Stücke des Gehirns aus dem ursprünglichen Gewebe, führen Sie eine RNA-Extraktion durch und testen Sie die Probe erneut. Ebenso können Ct-Werte > 33 (Singleplex), 37 (LN34M) oder keine Amplifikation im βA-Assay auf eine fehlgeschlagene RNA-Extraktion hinweisen. Wiederholen Sie die Extraktion für solche Proben und wiederholen Sie dann den Test für LN34 und Aktin. Wenn eine Probe nach wiederholten Tests erneut ein nicht schlüssiges Ergebnis liefert, verwenden Sie eine sekundäre Methode wie den DFA-Test (auch FAT genannt), DRIT oder die Virusisolierung. Wenn weiterhin widersprüchliche oder nicht schlüssige Ergebnisse beobachtet werden, wenden Sie sich bitte an ein Tollwut-Referenzlabor, um einen Bestätigungstest durchzuführen.
Wenn Inhibitoren in einer RNA-Extraktion vorhanden sind, können PCR-Assays ein falsch negatives Ergebnis liefern. Wenn der Verdacht auf eine Hemmung besteht oder eine Hemmung der βA-Kontrollreaktionen (wie Ct-Wert > 33 oder Ct-Wert > 37) für eine bestimmte Probe festgestellt wird, sollte die extrahierte RNA bei 2 oder mehr Verdünnungen (z. B. 1:10 und 1:100 in nukleasefreiem Wasser) getestet werden, um potenzielle PCR-Inhibitoren zu verdünnen. Bei schwierigen Proben kann der RNA-Eintrag in der RT-PCR-Reaktion auf 8,5 μl erhöht werden, indem kein Wasser hinzugefügt wird. Dies kann auf eine erhöhte Hemmung (späterer Ct-Wert im Vergleich zu 2 μL Input-RNA) oder einen niedrigen RNA-Spiegel in der Originalprobe (früherer Ct-Wert bei Verwendung von 8,5 μL im Vergleich zu 2 μL Input-RNA) hindeuten.
Der LN34-Assay unterscheidet nicht zwischen Lyssaviren und bestimmt auch keine Tollwutvirusvarianten. Das LN34-Assay-Amplikon kann für die niedrig aufgelöste Typisierung von Tollwutvirusvarianten oder die Identifizierung von Lyssavirus-Speziessequenziert werden 11.
Offenlegungen
Keine zur Offenlegung
Danksagungen
Wir würdigen die Bemühungen und die Zusammenarbeit vieler Laboratorien für Tollwutdiagnostik, die durch ihren offenen Datenaustausch und ihr Feedback zur Implementierung, Validierung und Optimierung des LN34-Assays beigetragen haben. Die Verwendung von Handelsnamen und kommerziellen Quellen dient nur zur Identifizierung und impliziert keine Billigung durch die Centers for Disease Control and Prevention, das U.S. Department of Health and Human Services oder die mit den Autoren verbundenen Institutionen. Die Schlussfolgerungen, Ergebnisse und Meinungen, die von den Autoren geäußert werden, spiegeln nicht unbedingt die offizielle Position des US-Gesundheitsministeriums, der Centers for Disease Control and Prevention oder der den Autoren angeschlossenen Institutionen wider.
Materialien
| Name | Company | Catalog Number | Comments |
| 7500 Fast | Applied Biosystems | N/A | Do not substitute without validation |
| 7500 Fast Dx | Applied Biosystems | N/A | Do not substitute without validation |
| ABI ViiA 7 | Applied Biosystems | N/A | Do not substitute without validation |
| AgPath-ID One-Step RT-PCR Kit | ThermoFisher Scientific | AM1005 | Do not substitute without validation |
| Beadbug6 | Benchmark Scientific | D1036 | |
| Direct-zol RNA MiniPrep kit | Zymo Research | R2052 | |
| MagNA Lyser green beads | Roche | 3358941001 | |
| Microcentrifuge | Eppendorf | 5425 R | |
| Optical 96-well Reaction Plates | ThermoFisher Scientific | 4346907 | |
| Optical Adhesive covers | ThermoFisher Scientific | 4311971 | Alternative: caps |
| Polyester fiber-tipped applicator swabs | BD BBL Polyester Fiber Tipped Application Swab | 220690 | |
| QuantStudio 6Flex | Applied Biosystems | 4485691 | Do not substitute without validation |
| Quaternary ammonium disinfectant (1:256) | LYSOL | WBB56939 | Do not substitute without validation |
| RNase AWAY | ThermoFisher Scientific | 7002PK | |
| RNaseZap | ThermoFisher Scientific | AM9780 | |
| Single-use scalpel, a scalpel with a safety mechanism | Integra Miltex | 4-510 | |
| Sterile polyproylene microcentrifuge tubes (1.5 mL), nuclease free | Sarstedt | 72.692.405 | |
| Sterile polyproylene microcentrifuge tubes (2 mL), nuclease free | Sarstedt | 72.694.600 | |
| TRIzol Reagent | ThermoFisher Scientific | 15596026 | Do not substitute without validation |
Referenzen
- Meechan, P. J., Potts, J. . Biosafety in Microbiological and Biomedical Laboratories. , (2020).
- . Terrestrial Manual 2023 Available from: https://www.woah.org/en/what-we-do/standards/codes-and-manuals (2023)
- . Laboratory Techniques in Rabies Available from: https://iris.who.int/handle/10665/310836 (2018)
- . Laboratory Techniques in Rabies Available from: https://iris.who.int/bitstream/handle/10665/310837/9789241515306-eng.pdf?ua=1 (2019)
- . Protocol for postmortem diagnosis of rabies in animals by direct fluorescent antibody testing: A minimum standard for rabies diagnosis in the United States Available from: https://www.cdc.gov/rabies/pdf/RabiesDFASPv2.pdf (2003)
- World Health Organization. . WHO Expert Consultation on Rabies: Third Report. , (2018).
- Gigante, C. M., et al. Multi-site evaluation of the LN34 pan-lyssavirus real-time RT-PCR assay for postmortem rabies diagnostics. PLoS One. 13 (5), e0197074 (2018).
- Wadhwa, A., et al. A Pan-Lyssavirus Taqman Real-Time RT-PCR assay for the detection of highly variable rabies virus and other lyssaviruses. PLoS Negl Trop Dis. 11 (1), e0005258 (2017).
- Gigante, C. M., Wicker, V., Wilkins, K., Seiders, M., Zhao, H., Patel, P., Orciari, L., Condori, R. E., Dettinger, L., Yager, P., Xia, D., Li, Y., et al. Optimization of pan-lyssavirus LN34 assay for streamlined rabies diagnostics by real-time RT-PCR. Journal Virological Methods. , (2024).
- Rao, A. K., et al. Use of a modified preexposure prophylaxis vaccination schedule to prevent human rabies: recommendations of the advisory committee on immunization practices-United States, 2022. Morbidity and Mortality Weekly Report. 71 (18), 619 (2022).
- Condori, R. E., et al. Using the LN34 Pan-Lyssavirus Real-Time RT-PCR assay for rabies diagnosis and rapid genetic typing from formalin-fixed human brain tissue. Viruses. 12 (1), 120 (2020).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten