É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Identificação e Caracterização de Espécies imunogênicas de RNA em Alérgenos HDM que modulam inflamação pulmonar eosinofílica
Neste Artigo
Resumo
Alérgenos ambientais, como ácaros domésticos (HDM), geralmente contêm substâncias microbianas que ativam respostas imunes inatas para regular a inflamação alérgica. O protocolo aqui apresentado demonstra a identificação de espécies de dsRNA em alérgenos HDM e caracterização de suas atividades imunogênicas na modulação da inflamação pulmonar eosinofílica.
Resumo
Alérgenos ambientais, como ácaros domésticos (HDM) são frequentemente em formas complexas que contêm tanto proteínas alérgicas que impulsionam respostas aberrantes tipo 2 quanto substâncias microbianas que induzem respostas imunes inatas. Esses componentes microbianos associados ao alérgeno desempenham um papel importante na regulação do desenvolvimento de condições inflamatórias tipo 2, como asma alérgica. No entanto, os mecanismos subjacentes permanecem em grande parte indefinidos. O protocolo aqui apresentado determina as características estruturais e a atividade in vivo do RNA imunoestimulatório associado ao alérgeno. Especificamente, alérgenos comuns são examinados para a presença de espécies de RNA de dupla laminada (dsRNA) que podem estimular as respostas de IFN nos pulmões e conter o desenvolvimento de eosinofilia pulmonar grave em um modelo de camundongos de asma alérgica induzida por HDM. Aqui, incluímos os seguintes três ensaios: Mancha de ponto para mostrar as estruturas dsRNA no total de RNA isolado de alérgenos, incluindo espécies HDM, RT-qPCR para medir as atividades de HDM RNA na expressão de genes estimulantes de interferon (ISGs) em pulmões de camundongos e análise FACS para determinar os efeitos do RNA HDM sobre o número de eosinófilos em BAL e pulmão, respectivamente.
Introdução
Com base na hipótese de higiene originalmente proposta por Strachan1, a exposição infantil a fatores microbianos ambientais, como a endotoxina, pode proteger contra o desenvolvimento de transtornos alérgicos2,,3. Durante infecções microbianas, por exemplo, infecções virais, a detecção imune inata de ácidos nucleicos estranhos (RNA/DNA) desencadeia respostas de defesa do hospedeiro4,,5,,6. No entanto, a existência e prevalência de ácidos nucleicos imunogênicos, como espécies de RNA (dsRNA) de dupla-encalhadas em ácaros domésticos (HDM) ou outros alérgenos de insetos permanecem desconhecidas. Este protocolo foi projetado para determinar se os alérgenos hdm ou insetos e não-insetos contêm espécies de dsRNA longas que podem ativar uma resposta imune protetora para neutralizar o desenvolvimento de inflamação pulmonar eosinofílica grave em um modelo de camundongo de asma alérgica. Aqui, fornecemos três métodos simples e rápidos para avaliar os determinantes estruturais no RNA total HDM que são necessários para regular a inflamação pulmonar eosinofílica induzida por alérgenos.
O sistema imunológico mucosa é o maior órgão imunológico do corpo e serve como primeira linha de defesa hospedeira contra infecções microbianas e insultos alérgicos7,,8. O dsRNA longo, o intermediário de replicação de muitos vírus, é conhecido por funcionar como um padrão molecular associado ao patógeno (PAMP) para estimular potentemente respostas inatas via Toll like receptor 3 (TLR3) para induzir a expressão de genes estimulados por interferon (ISGs)9,,10,,11,12,13,14. Recentemente, mostramos que o RNA total hdm continha estruturas de dsRNA, o que regulava a expressão dos ISGs e reduzia a inflamação pulmonar eosinofílica grave quando administrado através da instilação intratraqueal em um modelo murino de asma alérgica induzida pelos extratos de HDM15. A gravidade das inflamações pulmonares é determinada pela análise dos tipos de células imunes na lavagem broncoalveolar (BAL) e no tecido pulmonar através da citometria de fluxo16,17,,18,,19,20.
Este protocolo inclui três ensaios: 1) detecção rápida de estruturas dsRNA com mancha de ponto RNA usando um anticorpo monoclonal de rato J2 que se liga especificamente ao dsRNA (≥40bp) de forma independente de sequência; 2) avaliação rápida para efeitos in vivo do RNA imunoestimulativo nos pulmões do camundongo medindo a indução de ISGs utilizando RT-qPCR; 3) quantificação precisa de eosinófilos em BAL e pulmão no contexto de inflamação pulmonar induzida pelo HDM utilizando a análise da citometria de fluxo.
Os ensaios acima podem ser usados para estudar não apenas doenças pulmonares alérgicas, mas também infecções bacterianas e virais respiratórias. Por exemplo, o anticorpo J2 específico dsRNA também pode ser usado em outras aplicações como cromatografia imunoaffinity, imunohistoquímica, ensaio imunossorbente ligado à enzima (ELISA) e imunostaining21,,22,23. Além disso, várias aplicações a jusante da coleta de fluidos BAL podem ser utilizadas para quantificar conteúdos solúveis, como citocinas e quimiocinas usando ELISA, e perfil transcricional de células nas vias aéreas (por exemplo, macrófagos alveolares). Embora existam uma variedade de protocolos disponíveis na literatura para avaliar as condições pulmonares, a maioria desses protocolos muitas vezes se concentra na validação do alvo. Os procedimentos aqui descritos podem ser aplicados para identificar componentes em alérgenos ambientais que são importantes para regular o desenvolvimento de doenças alérgicas.
Protocolo
Os procedimentos experimentais descritos aqui foram aprovados pelo Comitê Institucional de Cuidados e Uso de Animais da Universidade do Texas Health San Antonio.
1. Mancha de ponto para mostrar a presença de estruturas dsRNA no RNA total HDM
- Isolamento total do RNA de alérgenos, insetos e alergênicos não-insetos
- Coloque hdm, insetos ou animais não-insetos coletados vivos ou obtidos comercialmente em tubos de 50 mL, e congele rapidamente com líquido-N2. Em seguida, armazene a -70 °C para posterior isolamento total do RNA.
NOTA: Neste experimento, foram selecionados animais HDM, insetos e não-insetos porque são conhecidos por serem fontes comuns de alérgenos. Além disso, uma função imunoestimulatória de suas RNAs permanece incerta. - Transfira uma quantidade adequada (equivalente a 100 μL em volume ou menos) de animais HDM, insetos ou não-insetos armazenados a -70 °C em um tubo de 2 mL contendo esferas cerâmicas (esferas cerâmicas de 1,4 mm), em seguida, congele tubos em um recipiente líquido-N2 por ~10 min.
- Para o isolamento total do RNA, adicione 1 mL de reagente de isolamento de RNA à base de tiocianato de guanidinium24 para cada tubo, em seguida, quebre o inseto e animais pequenos não-insetos com um disruptor celular de alta energia na velocidade máxima para 45 s e frio no gelo. Repita este passo duas vezes.
- Transfira a solução da etapa 1.1.3 para um novo tubo de 1,5 mL e adicione 200 μL de clorofórmio a cada tubo e vórtice. Tubos centrífugas a 14.000 x g para 14 min a 4 °C.
- Uma vez concluída a centrifugação, transfira a fase aquosa superior (200 μL) para um novo tubo de 1,5 mL contendo 500 μL de isopropanol para precipitar a pelota de RNA. Não perturbe a interfase. A razão de volume recomendada da fase superior versus isopropanol é de 1:2.5.
- Misture por vórtice suave, depois tubos de centrífugas a 14.000 x g por 14 min a 4 °C.
- Aspire o supernasce com cautela, depois lave a pelota de RNA com 500 μL de 75% de etanol e centrífuga a 7.500 x g por 10 min a 4 °C. Remova todo o líquido com cuidado, seque a pelota e dissolva a pelota de RNA com 20-50 μL de H2O sem RNase.
- Meça a concentração de RNA com um espectrofotômetro usando os seguintes parâmetros:
- Abra o software associado e selecione o tipo de ácidos nucleicos para medir. Mude o tipo de amostra para RNA.
- Realize a medição em branco com 1-2 μL de H2O sem RNase. Limpe o H2O sem RNase. Agora, o instrumento está pronto para a medição.
- Carregar 1-2 μL da amostra de RNA e medir a concentração de RNA (μg/μL).
NOTA: A razão de absorção em 260 e 280 nm (A260/280) em ~2.0 (1.9-2.2) é geralmente aceita como "pura" para RNA. Se não for processado imediatamente, armazene amostras de RNA a -70 °C e evite os ciclos de congelamento para manter o RNA intacto.
- Coloque hdm, insetos ou animais não-insetos coletados vivos ou obtidos comercialmente em tubos de 50 mL, e congele rapidamente com líquido-N2. Em seguida, armazene a -70 °C para posterior isolamento total do RNA.
- Detecção da estrutura dsRNA no RNA total usando anticorpo J2 específico dsRNA
- Prepare dois 20 μL de amostras de RNA (200 ng/μL). Um com tratamento RNase-III (1 μL para RNA de 1 μg, incubar a 37 °C por 60 min) e o outro sem tratamento RNase-III.
NOTA: O RNase III é usado aqui para degradar especificamente o dsRNA, mas não o RNA25de uma única linha . - Use um lápis para desenhar grades onde as amostras de RNA serão borradas na membrana.
- Ponto 2 μL dos 200 ng/μL da amostra de RNA na membrana de nylon carregada positivamente.
- Transligue as amostras à membrana a 1.200 microjoules x 100 em um crosslinker UV. Repita as etapas 1.2.3 e 1.2.4 mais duas vezes no local da amostra. Isso resultará em um total de 0,8 μg por mancha.
NOTA: Não localmente mais de 2 μL de amostra de RNA na membrana de cada vez. - Bloqueie a ligação não específica com 5% de leite em TBS-T por 1h com agitação à temperatura ambiente. Remova a solução de bloqueio da etapa 1.2.5 e adicione o anticorpo anti-dsRNA J2 na diluição de 1:1.000 em 1% de leite em TBS-T e incubar durante a noite com agitação a 4 °C.
- Lave a membrana com TBS-T por 5 minutos e repita esta etapa por 3 vezes. Adicione o anticorpo secundário (Alcalino fosfattase-conjugado Anti-Mouse IgG diluído em 1% de leite 1:5.000) e incubar por 1h em um shaker à temperatura ambiente. Lave a membrana com TBS-T por 5 minutos e repita esta etapa por 3x.
- Adicione o substrato (BCIP/NBT) e incubar por 5-15 min até que um sinal desejado seja visível.
- Pare a reação enxaguando a membrana com ddH2O.
- Seque a membrana em papéis de tecido e tire uma foto usando um smartphone (um resultado representativo é mostrado na Figura 1).
- Prepare dois 20 μL de amostras de RNA (200 ng/μL). Um com tratamento RNase-III (1 μL para RNA de 1 μg, incubar a 37 °C por 60 min) e o outro sem tratamento RNase-III.
2. RT-qPCR para medir a capacidade do RNA total HDM no estímulo à expressão dos ISGs pulmonares
- Isolamento de RNA de tecidos pulmonares de camundongos
NOTA: Os camundongos (fêmeas, de 8 a 12 semanas, C57BL/6J) foram mantidos em condições específicas sem patógenos.- Anestesia brevemente o animal com isoflurano e administre através da instilação intratraqueal com 5 μg (diluído em 80 μL PBS) de HDM RNAs tratados com ou sem RNase III.
- Após 16-18 h pós tratamento HDM RNA, sacrifique o mouse por inalação de CO2 por alguns minutos. Em seguida, coloque o mouse em uma plataforma e fixe membros com agulhas.
- Desinfete o rato com 70% de etanol e corte a pele do abdômen até o pescoço com uma tesoura esterilizada.
- Conserte a pele com agulhas e corte as costelas para expor os pulmões. Remova os pulmões inteiros e lave-os com PBS frio. Coloque os pulmões em papéis de tecido e extirve um pequeno pedaço de cada lobo-pulmão em um tubo de 2 mL contendo contas (200-300 μL em volume, esferas cerâmicas de 1,4 mm).
NOTA: O objetivo do uso de contas cerâmicas é moer tecidos pulmonares inteiros - Congele as amostras de pulmão colocando tubos em um recipiente líquido-N2 por ~10 min.
- Adicione 500 μL de reagente de isolamento de RNA à base de tiocianato de guanidinium em cada tubo e quebre os tecidos pulmonares com um homogeneizador para 45 s. Esfrie no gelo entre cada passo. Repita este passo duas vezes.
- Siga os passos 1.1.4- 1.1.7 para isolamento do RNA pulmonar.
- Seque a pelota e dissolva a pelota de RNA com a quantidade adequada de H2O sem RNase (~20-30 μL).
- Meça a concentração de RNA conforme descrito na etapa 1.1.8.
- RT-qPCR para determinar a capacidade do RNA HDM em estimular a expressão genética pulmonar.
- Utilizando 100 ng/μL de RNA extraído dos tecidos pulmonares como modelo, realize a síntese de cDNA de acordo com o protocolo referenciado26.
- Configure uma reação RT-qPCR a 10 μL/well para uma placa de 384 poços usando cDNA gerada acima e os pares de primer específicos de genes(Tabela 1 e Tabela 2).
- Sele os poços firmemente com um filme adesivo transparente e vórtice da placa por 30 s. Gire a placa a 1.000 x g por 30 s para coletar amostras na parte inferior dos poços.
- Carregue a placa em uma máquina RT-qPCR e comece a executar a reação RT-qPCR usando o protocolo de cicloviário térmico(Tabela 3).
- Exporte os resultados em um arquivo de planilha ou analise os dados usando o software fornecido pela manufatura após a conclusão do programa (um resultado representativo é mostrado na Figura 2).
3. Análise FACS para determinar os efeitos do RNA HDM na infiltração de eosinófilos em BAL e pulmão
- Coleta de fluidos BAL para análise FACS
- Camundongos eutanizais (fêmeas, 8-12 semanas de idade, C57BL/6J) que foram tratados com extratos de alérgenos HDM (de acordo com o desenho experimental mostrado na Figura 3B) por inalação de CO2.
- Coloque o mouse em uma plataforma e fixe membros com agulhas.
- Desinfete o rato com 70% de etanol. Use uma tesoura para cortar a pele da parte superior do abdômen até o pescoço.
- Suavemente, puxe as glândulas salivares e o músculo esternoide cuidadosamente separado usando os fórceps para expor a traqueia. Coloque uma corda de nylon (~10 cm) sob a traqueia usando fórceps.
- Faça uma incisão na traqueia (~2 mm sob a laringe) apenas o suficiente para inserir uma cânula. Não corte a traqueia. Nó a corda em torno de traqueia e cânula.
- Carregue a seringa com 1 mL de PBS+EDTA e anexe-a à extremidade da cânula. Injete 1 mL de PBS+EDTA no pulmão e aspire completamente a solução. Retire a seringa da cânula cuidadosamente e transfira a solução para um tubo de 15 mL no gelo.
- Recarregue a seringa com o PBS+EDTA fresco e repita esta etapa 2x.
- Centrifugar o tubo contendo o BAL agrupado obtido na etapa 3.1.7 para pelotar as células a 500 x g por 7 min a 4 °C. Regisso o volume do fluido BAL e transfira o supernante para dois tubos de 1,5 mL sem perturbar a pelota.
NOTA: O supernatante de BAL pode ser armazenado a -70 °C para análise futura, por exemplo, ELISA. - No caso de haver RBCs presentes na pelota devido a inflamação pulmonar grave, após a remoção do supernascimento, adicione 500 μL de tampão de lise RBC e misture bem com a resuspensão. Transfira a solução para um novo tubo de 1,5 mL e centrífuga por 7 min a uma velocidade de 500 x g a 4 °C.
- Remova o supernatante e resuspense a pelota em 150 μL de tampão FACS.
- Transfira os 150 μL da amostra resuspended em placa de 96 poços e centrifugar a placa por 7 minutos à velocidade de 500 x g a 4 °C.
- Rapidamente, inverta a placa em papéis de tecido para coletar as células que residem na parte inferior dos poços.
- Manche as células com anticorpos no tampão FACS na presença de anticorpos bloqueadores de 2,4G2 (2,5 μg / 100 μL). Incubar a placa em temperatura ambiente por 30 minutos em um lugar escuro.
- Após a coloração, centrifufique a placa para pelotar as células a 500 x g por 7 min a 4 °C.
- Remova a solução de coloração invertendo a placa em papel de tecido e depois lave resususus com 100 μL de tampão FACS. Em seguida, centrifugar a placa novamente a 500 x g por 7 min a 4 °C e remover o tampão FACS invertendo a placa em papel de tecido.
- Resuspend amostras em 150 μL de tampão FACS e transferir amostras para os tubos FACS contendo 350 μL de tampão FACS. Adicione 25 μL de contas de contagem a cada amostra. As amostras estão prontas para análise de citometria de fluxo.
NOTA: Vários tipos de células em fluido BAL foram rotulados com anticorpos conforme indicado. As contas de contagem foram adicionadas antes da execução do FACS. Os dados de citometria de fluxo foram analisados por meio de um software comercialmente disponível. Consulte a Figura 3 e a Tabela 4 para a estratégia de gating.
- Digestão do tecido pulmonar para a análise FACS
- Siga os passos 3.1.1 - 3.1.3.
- Corte a pele do abdômen até o pescoço com uma tesoura esterilizada. Conserte a pele com agulhas e corte as costelas para expor os pulmões.
- Remova os pulmões inteiros e lave-os com PBS frio. Coloque amostras no tubo de 1,5 mL contendo 50 μL de solução de digestão pulmonar.
- Pique os tecidos pulmonares em pequenos pedaços com uma tesoura curvada. Transfira os tecidos pulmonares para uma placa de 6 poços, depois adicione 8 mL de solução de digestão pulmonar. Coloque a placa em um agitador na incubadora de 37 °C por 45 min.
- Após a incubação, use o tubo superior de 1,5 mL para moer os tecidos pulmonares. Coloque uma placa de 70 μm em uma nova placa de 6 poços e aplique a amostra através de um filtro de 0,22 μm.
- Transfira a solução filtrada para um tubo de 15 mL e, em seguida, centrífugue os tubos a 500 x g por 7 min a 4 °C. Aspire o supernasciente e resuspense a pelota em 1 mL de tampão de lise RBC e deixe-a no gelo por 3 minutos.
- Transfira a amostra para tubo de 1,5 mL e centrífuga a 500 x g por 7 min a 4 °C. Repita 2x
- Lave as células pulmonares 2x com tampão FACS de 1 mL. Aspire o supernasal e resuspenque a pelota em 1 mL de tampão FACS e, em seguida, transfira 100 μL da amostra em placa de 96 poços.
- Centrifugar a placa por 7 min a uma velocidade de 500 x g a 4 °C. Siga os passos descritos na coleta de fluidos BAL para análise facs (3.1.13 a 3.1.16) para manchar as células de amostras de tecido pulmonar digerido.
NOTA: Os eosinófilos nos pulmões foram rotulados com anticorpos como indicado, depois misturados com contas de contagem para posterior análise facs. Os dados de citometria de fluxo foram analisados por meio de software associado. Consulte a Figura 3 para avaliar as respostas imunes induzidas pelo RNA do HDM.
4. Análise estatística
- Realize análises estatísticas usando um software comercialmente disponível.
- Determine os valores p por teste T de estudante de duas caudas não verificados para a comparação de dois grupos.
- Calcule os números absolutos de eosinófilos com base em contas de referência (painel superior) usando a fórmula
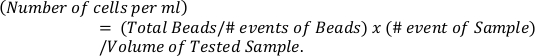
- Determine os valores p por duas vias ANOVA e sidak teste de comparações múltiplas para a comparação de mais de dois grupos.
- Considere um valor p menor que 0,05 como estatisticamente significativo. Os valores p são indicados em parcelas como *p <0,05, **p<0.01, ***p<0.001 e ****p<0.0001.
NOTA: Todas as receitas de buffer são fornecidas na Tabela 5.
Resultados
A presença de estruturas de dsRNA longas em HDM, insetos e animais pequenos não-insetos foi examinada por mancha de ponto usando um anticorpo monoclonal de camundongos específico dsRNA J2 (≥ 40bp). RNase III foi usado para digerir dsRNA em fragmentos de dsRNA de 12 a 15 bp, que eram indetectáveis por J2 (Figura 1).
A capacidade do RNA total hdm de estimular uma resposta imune inata nos pulmões do camundongo de forma dependente de dose foi analisada por RT-q...
Discussão
O protocolo atual descreve como avaliar as propriedades imunoestimulatórias do RNA microbiano associado ao alérgeno e seus impactos no desenvolvimento da inflamação pulmonar eosinofílica em um modelo de camundongo de asma alérgica. Embora os DSRNAs longos sejam conhecidos como intermediários de replicação de muitos vírus que podem potencialmente ativar respostas de interferon em células de mamíferos, suas presenças em alérgenos HDM têm sido desconhecidas até nosso trabalho recente15
Divulgações
Não temos nada para revelar.
Agradecimentos
Agradecemos à Sra. Karla Gorena pela assistência técnica na citometria de fluxo. L.S. é apoiado pelo China Scholarship Council e Hunan Provincial Innovation Foundation for Postgraduate (CX201713068). H.H.A. é apoiado pelo Departamento de Ciências Clínicas do Laboratório, Faculdade de Ciências Médicas Aplicadas, Universidade de Jouf, Sakaka, Arábia Saudita. X.D.L. é apoiado pelo UT Health San Antonio School of Medicine Startup Fund e pelo Max and Minnei Voelcker Fund.
Materiais
| Name | Company | Catalog Number | Comments |
| 0.40 µm Falcon Cell Strainer | Thermo Fisher Scientific | 08-771-1 | |
| 1 mL syringes | Henke Sass Wolf | 5010.200V0 | |
| 15 mL Tube | TH.Geyer | 7696702 | |
| 50 mL Tube | TH.Geyer | 7696705 | |
| 70% ethanol | Decon Labs | 2701 | |
| Absolute Counting Beads | Life Technologies Europe B.V. | C36950 | |
| ACK-RBC lysing buffer | Lonza | 10-548E | |
| Amersham Hybond-N+ Membrane | GE Healthcare | RPN203B | |
| Ant | San Antonio | Note: Locally collected | |
| Antibody dilution buffer | (see Table 5 for recipe) | ||
| Anti-Mouse CD11b V450 Rat (clone M1/70) | BD Bioscience | 560456 | 1 to 200 dilution |
| Anti-Mouse CD11c PE-Cy7 (clone N418) | BioLegend | 117317 | 1 to 200 dilution |
| Anti-Mouse CD19 Alexa Flour 647 (clone 1D3) | eBioscience | 15-0193-81 | 1 to 200 dilution |
| Anti-Mouse CD3e APC (clone 145-2C11) | Invitrogen | 15-0031-81 | 1 to 200 dilution |
| Anti-Mouse CD45 APC-Cy7 (clone: 30-F11) | BioLegend | 103130 | 1 to 200 dilution |
| Anti-Mouse Fixable Viabillity Dye eFluor 506 | Invitrogen | 65-0866-14 | 1 to 200 dilution |
| Anti-Mouse IgG (H+L), AP Conjugate | Promega | S3721 | |
| Anti-Mouse Ly-6G FITC (clone RB6-8C5) | Invitrogen | 11-5931-82 | 1 to 200 dilution |
| Anti-Mouse MHC II APC-eFluor 780 (clone M5/114.15.2) | eBioscience | 47-5321-80 | 1 to 200 dilution |
| Anti-Mouse Siglec-F PE (clone E50-2440) | BD Pharmingen | 552126 | 1 to 200 dilution |
| BCIP/NBT substrate | Thermo Fisher Scientific | PI34042 | |
| Blocking Buffer | (see Table 5 for recipe) | ||
| Cannual, 20G X 1.5” | CADENCE SCIENCE | 9920 | |
| Centrifuge | Thermo Fisher Scientific | 75004030 | |
| CFX384 Touch Real-Time PCR Detection System | Bio-Rad Laboratories | 1855485 | |
| Chloroform | Thermo Fisher Scientific | C298-500 | |
| Cockroach | Greer Laboratories | B26 | |
| Counting beads | Thermo Fisher Scientific | 01-1234-42 | |
| D. farinae | Greer Laboratories | B81 | |
| D. pteronyssinus | Greer Laboratories | B82 | |
| Denville Cell Culture Plates with lid, 96 well cell culture plate | Thomas Scientific | 1156F03 | |
| Digital Dry Bath - Four Blocks | Universal Medical, Inc. | BSH1004 | |
| Earthworm | San Antonio | Note: Locally collected | |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma-Aldrich | E6511 | |
| FACS buffer | (see recipe in Table 5) | ||
| Falcon Round-Bottom Polypropylene Tubes, 5 mL | STEMCELLTM TECHNOLOGIES | 38056 | |
| Flow cytometer (BD FACS Celesta) | BD Biosciences | ||
| Fly | Greer Laboratories | B8 | |
| Forceps | Roboz Surgical Instrument | RS-5135 | |
| Hemocytometer | Hausser Scientific | 3110 | |
| HT-DNA | Sigma | D6898 | |
| In Vivo MAb anti-mouse CD16/CD32 (clone: 2.4G2) | Bio X Cell | BE0307 | |
| iScript cDNA Synthesis Kit | Bio-Rad Laboratories | 1708891 | |
| Isoflurane | Abbott Labs | sc-363629Rx | |
| Isopropanol | Thermo Fisher Scientific | BP2618500 | |
| J2 anti-dsRNA monoclonal antibody | SCICONS | 10010200 | |
| Lung digestion solution | (see recipe in Table 5) | ||
| Lysing Matrix D | MP Biomedicals | 116913050-CF | |
| Lysing Matrix D, 2 mL tube | MP Biomedicals | SKU:116913100 | |
| Mice (female, 8-12 weeks old, C57BL/6J) | Jackson Laboratory | #000664 | |
| Microcentrifuge tube 1.5 mL | Sigma-Aldrich | 30120.094 | |
| Microscope | Olympus | CK30 | |
| Mini-BeadBeater | Homogenizers | SKU:BS:607 | |
| Mini-Beadbeater-16 | Biospec | 607 | |
| Mosquito | Greer Laboratories | B55 | |
| NanoDrop 2000C | Thermo Scientific Spectophotometer Medex Supply | TSCND2000C | |
| Needle, 21 G x 1 1/2 in | BD Biosciences | 305167 | |
| Non-fat milk | Bio-Rad Laboratories | 1706404 | |
| Nylon string | Dynarex | 3243 | |
| Phosphate-buffered Saline (PBS) | Lonza | BE17-516F | |
| RNase III | Thermo Fisher Scientific | AM2290 | |
| RNase T1 | Thermo Fisher Scientific | AM2283 | |
| Scissors | Roboz Surgical Instrument | RS-6802 | |
| Shaker or Small laboratory mixer | Boekel Scientific | 201100 | |
| SPHERO AccuCount Fluorescent | Spherotech | ACFP-70-5 | 1 to 10 dilution |
| Spider | San Antonio | Note: Locally collected | |
| TBS | (see recipe in Table 5) | ||
| TBS-T | (see recipe in Table 5) | ||
| Total cell medium | (see recipe in Table 5) | ||
| TRIzol Reagent | Thermo Fisher Scientific | 15596018 | |
| Tween 20 | Sigma-Aldrich | P9416 | |
| UV Stratalinker 2400 UV | LabX | 20447 | |
| Wasp | San Antonio | Note: Locally collected |
Referências
- Strachan, D. P. Hay fever, hygiene, and household size. BMJ. 299, 1259-1260 (1989).
- Schuijs, M. J., et al. Farm dust and endotoxin protect against allergy through A20 induction in lung epithelial cells. Science. 349, 1106-1110 (2015).
- Stein, M. M., et al. Innate Immunity and Asthma Risk in Amish and Hutterite Farm Children. New England Journal of Medicine. 375, 411-421 (2016).
- Roers, A., Hiller, B., Hornung, V. Recognition of Endogenous Nucleic Acids by the Innate Immune System. Immunity. 44, 739-754 (2016).
- Schlee, M., Hartmann, G. Discriminating self from non-self in nucleic acid sensing. Nature Reviews Immunology. 16, 566-580 (2016).
- Wu, J., Chen, Z. J. Innate immune sensing and signaling of cytosolic nucleic acids. Annual Reviews Immunology. 32, 461-488 (2014).
- O'Hara, A. M., Shanahan, F. The gut flora as a forgotten organ. EMBO Reports. 7 (7), 688-693 (2006).
- . Focused Meeting 2018: Microbes and Mucosal Surfaces Available from: https://microbiologysociety.org/event/society-events-and-meetings/focused-meeting-2018-microbes-and-mucosal-surfaces.html (2018)
- Weber, F., et al. Double-stranded RNA is produced by positive-strand RNA viruses and DNA viruses but not in detectable amounts by negative-strand RNA viruses. Journal of Virology. 80, 5059-5064 (2006).
- Barral, P. M., et al. Functions of the cytoplasmic RNA sensors RIG-I and MDA-5: Key regulators of innate immunity. Pharmacology and Therapeutics. 124, 219-234 (2009).
- Netea, M. G., et al. From the Th1/Th2 paradigm towards a Toll-like receptor/T-helper bias. Antimicrobial Agents and Chemotherapy. 49, 3991-3996 (2005).
- McNally, B., et al. Intranasal administration of dsRNA analog poly(I:C) induces interferon-alpha receptor-dependent accumulation of antigen experienced T cells in the airways. PLoS One. 7, 51351 (2012).
- Seya, T., Takeda, Y., Matsumoto, M. Tumor vaccines with dsRNA adjuvant ARNAX induces antigen-specific tumor shrinkage without cytokinemia. Oncoimmunology. 5, 1043506 (2016).
- Toussi, D. N., Massari, P. Immune Adjuvant Effect of molecularly defined Toll-Like Receptor Ligands. Vaccines (Basel). 2, 323-353 (2014).
- She, L., et al. Immune Sensing of Aeroallergen-Associated Double-Stranded RNA Triggers an IFN Response and Modulates Type 2 Lung Inflammation. Journal of Immunology. 203, 2520-2531 (2019).
- Fujimoto, Y., et al. Pulmonary inflammation and cytokine dynamics of bronchoalveolar lavage fluid from a mouse model of bronchial asthma during A(H1N1)pdm09 influenza infection. Science Reports. 7, 9128 (2017).
- Yao, Y., et al. Induction of Autonomous Memory Alveolar Macrophages Requires T Cell Help and Is Critical to Trained Immunity. Cell. 175, 1634-1650 (2018).
- Dua, K., Shukla, S. D., Hansbro, P. M. Aspiration techniques for bronchoalveolar lavage in translational respiratory research: Paving the way to develop novel therapeutic moieties. Journal of Biological Methods. 4, 73 (2017).
- Van Hoecke, L., et al. Bronchoalveolar Lavage of Murine Lungs to Analyze Inflammatory Cell Infiltration. Journal of Visualized Experiments. (123), e55398 (2017).
- Salahuddin, S., et al. Processing of Bronchoalveolar Lavage Fluid and Matched Blood for Alveolar Macrophage and CD4+ T-cell Immunophenotyping and HIV Reservoir Assessment. Journal of Visualized Experiments. (148), e59427 (2019).
- Son, K. N., Liang, Z., Lipton, H. L. Double-Stranded RNA Is Detected by Immunofluorescence Analysis in RNA and DNA Virus Infections, Including Those by Negative-Stranded RNA Viruses. Journal of Virology. 89, 9383-9392 (2015).
- Monsion, B., et al. Efficient Detection of Long dsRNA in Vitro and in Vivo Using the dsRNA Binding Domain from FHV B2 Protein. Front Plant Sci. 9, 70 (2018).
- Redente, E. F., et al. Age and sex dimorphisms contribute to the severity of bleomycin-induced lung injury and fibrosis. American Journal of Physiology-Lung Cellular and Molecular Physiology. 301, 510-518 (2011).
- Card, J. W., et al. Gender differences in murine airway responsiveness and lipopolysaccharide-induced inflammation. Journal of Immunology. 177, 621-630 (2006).
- Gueders, M. M., et al. Mouse models of asthma: a comparison between C57BL/6 and BALB/c strains regarding bronchial responsiveness, inflammation, and cytokine production. Inflammation Research. 58, 845-854 (2009).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados