É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Imagem e Análise Automatizada para a Quantificação de Macropinoss fluorescentes rotulados
Neste Artigo
Resumo
Ensaios automatizados usando microplatas multi-poços são abordagens vantajosas para identificar reguladores de vias, permitindo a avaliação de uma infinidade de condições em um único experimento. Aqui, adaptamos o bem estabelecido protocolo de imagem e quantificação de macropinoso para um formato de microplacão de 96 poços e fornecemos um contorno abrangente para automação usando um leitor de placas de vários modos.
Resumo
Macropinocistose é uma via de absorção não específica da fase de fluidos que permite que as células internalizem grandes cargas extracelulares, como proteínas, patógenos e detritos celulares, através da endocitose a granel. Esse caminho desempenha um papel essencial em uma variedade de processos celulares, incluindo a regulação de respostas imunes e metabolismo de células cancerosas. Dada essa importância na função biológica, examinar as condições da cultura celular pode fornecer informações valiosas identificando reguladores dessa via e otimizando condições a serem empregadas na descoberta de novas abordagens terapêuticas. O estudo descreve uma técnica automatizada de imagem e análise utilizando equipamentos de laboratório padrão e um leitor de placas multimodestração de imagem celular para a rápida quantificação do índice macropinocítico em células aderentes. O método automatizado baseia-se na absorção de dextran fluorescente de alto peso molecular e pode ser aplicado a microplacões de 96 poços para facilitar avaliações de múltiplas condições em um experimento ou amostras fixas montadas em tampas de vidro. Esta abordagem visa maximizar a reprodutibilidade e reduzir a variação experimental, ao mesmo tempo em que economiza tempo e econômica.
Introdução
A via endocítica não específica da macropinocistose permite que as células internalizem uma variedade de componentes extracelulares, incluindo nutrientes, proteínas, antígenos e patógenos, através da absorção em massa de fluido extracelular e seus constituintes1. Embora importante para a biologia de numerosos tipos celulares, cada vez mais, a via macropinotose é descrita como um papel essencial na biologia tumoral, onde, através da absorção macropinócítica, as células tumorais são capazes de sobreviver e proliferar na presença de um microambiente empobrecido de nutrientes2,3. A absorção de macromoléculas extracelulares, incluindo albumina e matriz extracelular, e detritos de células necróticas, fornece uma fonte alternativa de nutrientes para a produção de biomassa, criando aminoácidos, açúcares, lipídios e nucleotídeos através de macropinoso e catabolismo de carga mediado por fusão4,5,6,7,8.
A indução e regulação da macropinocitose são complexas e podem variar dependendo do contexto celular. Até agora, vários indutores de macropinocistose foram identificados e incluem ligantes, como fator de crescimento epidérmico (EGF), fator de crescimento derivado de plaquetas (PDGF), galectina-3 e Wnt3A9,10,11,12,13. Além disso, condições de cultivo que imitam o microambiente tumoral podem desencadear a ativação da via. Os tumores de adenocarcinoma ductal pancreático (PDAC) são privados de nutrientes, especialmente para a glutamina aminoácido, que faz com que tanto as células cancerígenas quanto os fibroblastos associados ao câncer (CAFs) dependam da macropinocise para sobreviver7,13,14,15. Além disso, as tensões tumorais, como a hipóxia e o estresse oxidativo, podem ativar essa via de limpeza16. Além dos inúmeros influenciadores extrínsecos que podem induzir a macropinocisse, uma variedade de vias intracelulares controlam a formação macropinosa. A transformação oncogênica mediada por Ras é suficiente para iniciar o maquinário macropinocítico, e vários tipos de câncer exibem macropinocistose constitutiva oncogênica baseada em Ras4,5,9,17. Alternativamente, a ativação ras do tipo selvagem e vias independentes de Ras foram identificadas para ativar a macropinocistose em células cancerosas e CAFs10,11,15,18. O uso de vários modelos in vitro em combinação com tratamentos inibidores resultou na identificação de vários moduladores de macropinocise, que incluem trocadores de sódio-hidrogênio, o pequeno GTPase Rac1, fosfoinositida 3-quinase (PI3K), quinase ativada p21 (Pak) e proteína ativada amp(AMPK)4,13,15 . No entanto, dada a multiplicidade de fatores e condições descritos que regulam a macropinocistose, é concebível que muitos mais moduladores e estímulos permaneçam desconhecidos. A identificação de novos moduladores e estímulos pode ser facilitada pela avaliação automatizada de uma infinidade de condições em um único experimento. Essa metodologia pode lançar luz sobre os fatores envolvidos na formação de macropinossomos e pode permitir a descoberta de novas pequenas moléculas ou biológicas que visam esse caminho.
Aqui, adaptamos nosso protocolo previamente estabelecido para determinar a extensão da macropinocistose em células cancerosas in vitro para um formato de microplacão de 96 poços e imagem automatizada e quantificação19,20. Este protocolo é baseado na microscopia fluorescente, que se tornou um padrão no campo para determinar a macropinocise in vitro e in vivo4,5,6,7,9,10,11,12,13,15,16,17,18, 19,20,21,22. Macropinossomos podem ser distinguidos de outras vias endocíticas através de sua capacidade de internalizar grandes macromoléculas, como dextran de alto peso molecular (ou seja, 70 kDa)2,3,4,20,21,22,23. Assim, macropinossomos podem ser definidos através da absorção de fluorforotos extracelularmente rotulados de 70 kDa dextran. Como resultado, as vesículas macropinocísticas se manifestam como aglomerados intracelulares de puncta fluorescente com tamanhos que variam de 0,2-5 μm. Esses punctas podem ser imagens microscopicamente e, posteriormente, quantificadas para determinar a extensão da macropinocise na célula - "o índice macropinocítico".
Neste protocolo, são descritas as etapas essenciais para visualizar macropinossomos em células aderentes in vitro em uma microplacão de 96 poços e são descritas manchas usando equipamentos de laboratório padrão (Figura 1)." Além disso, são fornecidas as instruções para automatizar a aquisição e quantificação da imagem do índice macropinocítico usando um leitor de placas multimodesas de imagem celular. Essa automação reduz o tempo, o custo e o esforço em comparação com nossos protocolos descritos anteriormente19,20. Além disso, evita a aquisição e análise de imagens sem querer tendenciosas e, assim, aumenta a reprodutibilidade e a confiabilidade. Este método pode ser facilmente adaptado a diferentes tipos de células ou leitores de placas ou ser utilizado para determinar características macropinosas alternativas, como tamanho, número e localização. O método aqui descrito é especialmente adequado para a triagem de condições de cultura celular que induzem a macropinocistose, a identificação de novos moduladores ou a otimização de concentrações de medicamentos de inibidores conhecidos.
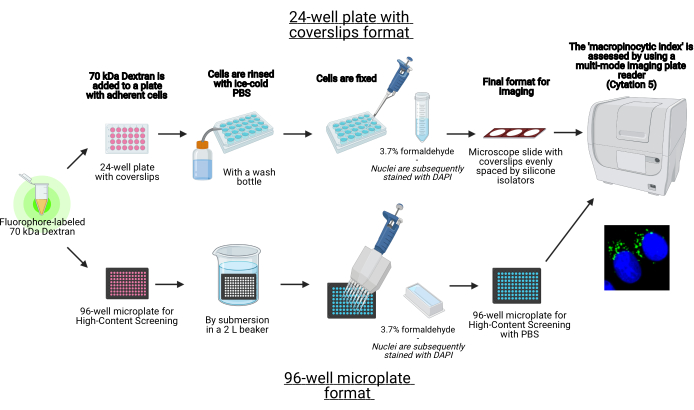
Figura 1: Esquema do ensaio automatizado para determinar o "índice macropinolítico" em células aderentes. Criado usando BioRender. Clique aqui para ver uma versão maior desta figura.
Protocolo
1. Preparação de materiais
- Dissolver 70 kDa dextran rotulado com FITC ou tetrametilrhodamina (TMR) em PBS para obter uma solução de 20 mg/mL. Armazene as alíquotas a -20 °C.
- Dissolver o DAPI no ddH2O para obter uma solução de 1 mg/mL. Armazene as alíquotas a -20 °C.
ATENÇÃO: O DAPI é um potencial cancerígeno e deve ser tratado com cuidado. - No dia da fixação, prepare formaldeído fresco de grau ACS de 3,7% na PBS.
ATENÇÃO: O formaldeído é um carcinógeno fixável e conhecido e é tóxico se inalado. Faça a solução em um capô de fumaça química para evitar a inalação e manuseie com cuidado. - Prepare as tampas lavadas com ácido.
- Use um béquer de 500 mL e banho de água para aquecer 28 g de tampas de vidro borossilicato com um diâmetro de 12 mm e espessura de 1,5 para 24 h a 56 °C em 100 mL de 1 M HCl. Sele o béquer com plástico para evitar evaporação extensiva.
ATENÇÃO: O HCL é um ácido forte altamente corrosivo e deve, portanto, ser manuseado com cuidado e usado em uma capa de fumaça química para evitar a inalação. - Lave as tampas com água destilada. Repita a lavagem 4 vezes. Em seguida, lave as tampas com 95% de etanol. Repita a lavagem 4 vezes.
- Armazene as tampas lavadas com ácido em um prato de cultura celular à temperatura ambiente para uso futuro, mantendo a esterilidade através da submersão em 95% de etanol. Sele o prato com parafilm para evitar evaporação extensiva.
- Use um béquer de 500 mL e banho de água para aquecer 28 g de tampas de vidro borossilicato com um diâmetro de 12 mm e espessura de 1,5 para 24 h a 56 °C em 100 mL de 1 M HCl. Sele o béquer com plástico para evitar evaporação extensiva.
2. Preparação de células
- Utilizando uma placa de cultura tecidual confluente de 10 cm com as células aderentes de interesse, aspire a mídia e enxágue as células com 5 mL de DPBS, pré-armadas a 37 °C.
- Retire as células da placa adicionando 1,5 mL de trippsina pré-armada de 0,25% e incubando a 37 °C.
NOTA: O tempo de incubação de trippsina necessário para desprender as células de interesse deve ser empiricamente determinado e pode ser confirmado observando o desprendimento sob um microscópio de luz convencional. - Colete as células em um tubo de centrífuga de 15 mL e adicione 4,5 mL de mídia completa para saciar a trippsina.
- Pelota as células por centrifugação por 3 min a 200 x g e aspire o supernasce.
- Resuspengem a pelota de célula em um volume adequado de mídia completa pré-armada para obter uma única suspensão celular para semeadura.
- Prossiga para semear as células em uma placa de 24 poços com tampas ou um formato de microplacão de 96 poços (Figura 1).
NOTA: O número de células a serem semeadas deve ser determinado empiricamente para cada linha celular, pois as taxas de proliferação e o tamanho variam entre as linhas celulares. Este protocolo foi otimizado para células cancerígenas aderentes com 80% de confluência celular no dia da rotulagem macropinosa. A confluência celular pode afetar a capacidade macropinocícica, e isso também deve ser determinado empiricamente.- Placa de 24 poços com formato de deslizamento de tampas
- Adicione tampas a uma placa de cultura de tecido de 24 poços, use fórceps para segurar uma única tampa do banho de etanol. Bata na tampa na parede interna da placa para remover o excesso de etanol e coloque o deslizamento de tampas na parte inferior de um poço.
- Deixe o etanol evaporar e lave o deslizamento 2 vezes com DPBS.
- Semear as células em cima do deslizamento de tampa adicionando 500 μL da suspensão celular a cada poço. Coloque as células em uma incubadora celular de 37 °C com 5% de CO2 até que a confluência celular atinja 60%-80% no dia anterior à rotulagem macropinosome.
- Um dia antes da rotulagem macropinosa, aspire a mídia dos poços e adicione 500 μL de mídia sem soro pré-armado a cada poço e coloque as células em uma incubadora de células de 37 °C com 5% de CO2 por 16-24 h.
NOTA: Dependendo das condições a serem estudadas, recomenda-se que a mídia livre de soro reduza os efeitos de fatores de crescimento que podem induzir a macropinocisse e que normalmente estão presentes no soro. No entanto, deve-se considerar que a fome de soro pode afetar outros processos celulares, como a proliferação e a autofagia. Como o soro residual pode afetar a capacidade macropinocítica das células, bem como a atividade inibidora, a remoção do soro pode ser melhorada enxaguando suavemente as células 1 ou 2 vezes com 500 μL de DPBS pré-armados.
- Formato de microplacão de 96 poços
- Transfira a suspensão da célula para um reservatório de reagente de 25 mL. Usando uma pipeta multicanal (8 ou 12 canais), semente 100 μL da suspensão celular para cada poço de uma microplaca de triagem preta de 96-bem-de-alto teor com olefina cíclica opticamente clara ou fundo de vidro.
- Coloque as células em uma incubadora celular de 37 °C com 5% de CO2 até que a confluência celular atinja 60%-80% no dia anterior à rotulagem macropinosome.
- Um dia antes da rotulagem macropinosa, remova e descarte a mídia de cada poço usando uma pipeta multicanal (8 ou 12 canais) ou um adaptador de aspiração multicanal para pontas padrão anexadas a uma bomba de vácuo.
- Usando um reservatório de reagente e pipeta multicanal (8 ou 12 canais), adicione suavemente 100 μL de mídia pré-armada sem soro a cada poço. Coloque as células em uma incubadora celular de 37 °C com 5% de CO2 para 16-24 h.
NOTA: Dependendo das condições a serem estudadas, recomenda-se que a mídia livre de soro reduza os efeitos de fatores de crescimento que podem induzir a macropinocisse e que normalmente estão presentes no soro. No entanto, deve-se considerar que a fome de soro pode afetar outros processos celulares, como a proliferação e a autofagia. Como o soro residual pode afetar a capacidade macropinocítica das células, bem como a atividade inibidora, a remoção do soro pode ser melhorada enxaguando suavemente as células 1 ou 2 vezes com 100 μL de DPBS pré-armados.
- Placa de 24 poços com formato de deslizamento de tampas
3. Rotulagem macropinosa
- Placa de 24 poços com formato de deslizamento de tampas
- Aspire os poços e adicione de volta 200 μL de mídia sem soro com dextran de alto peso molecular (70 kDa) com 1 mg/mL com alto peso molecular (70 kDa). Coloque as células em uma incubadora celular de 37 °C por 30 minutos.
NOTA: Dependendo das condições a serem estudadas, em vez de usar mídia fresca, a reutilização da mídia condicionada para carregamento de dextran pode ser preferida, pois conteria fatores secretados ou suplementados, como EGF ou compostos inibidores, respectivamente, que podem afetar a capacidade macropinocítica das células. - Aspire a mídia e lave suavemente, mas rapidamente, as células 5 vezes com PBS gelado usando uma garrafa de lavagem pré-motor. Agite firmemente a placa à mão durante as lavagens para ajudar na desalojação dos agregados do Dextran que ficam presos às tampas.
- Fixar as células adicionando 350 μL de 3,7% de formaldeído e incubando por 20 min. Em seguida, aspire a solução de fixação e lave as células com PBS duas vezes.
- Manche os núcleos com 350 μL de 2 μg/mL DAPI em PBS. Após 20 min, aspire a solução DAPI e lave as células com PBS três vezes.
- Adere aos isoladores de silicone lado a lado em um slide de microscópio para obter mesmo espaçamento e localização reprodutível das tampas, necessárias para automação de imagem (Figura 2A,B).
NOTA: Todo o slide do microscópio pode ser preenchido com um total de 3 isoladores. - Para cada deslizamento de cobertura, adicione uma gota de mídia de montagem de fluorescência de endurecimento no slide do microscópio dentro do espaço aberto do isolador (Figura 2C). Pegue uma mancha de cobertura usando fórceps e remova o excesso de PBS tocando suavemente o lado das tampas em uma limpeza sem fiapos.
- Coloque o deslizamento de cabeça para baixo na queda da mídia de montagem (Figura 2D). Toque suavemente na tampa usando fórceps fechados para remover bolhas da mídia de montagem (Figura 2E).
- Armazene os slides em um ambiente escuro e permita que a mídia de montagem seque à temperatura ambiente, normalmente levando 16-24 h. Os slides agora podem ser imagedos ou armazenados a -20 °C por até 2 semanas.
- Antes de fotografar, remova os isoladores do slide do microscópio. Deixe os slides equilibrarem-se à temperatura ambiente e limpe as tampas usando um aplicador de ponta de algodão molhado com limpador de vidro sem amônia. Posteriormente, use um aplicador de ponta de algodão limpo molhado com 70% de etanol para limpar e deixar o deslizamento seco.
- Aspire os poços e adicione de volta 200 μL de mídia sem soro com dextran de alto peso molecular (70 kDa) com 1 mg/mL com alto peso molecular (70 kDa). Coloque as células em uma incubadora celular de 37 °C por 30 minutos.
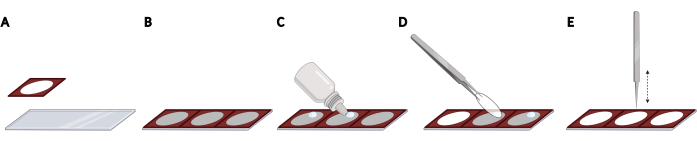
Figura 2: Colocar tampas em um slide de microscópio com isoladores de silicone. (A) Isoladores de silicone são pressionados e aderidos a um slide de microscópio. (B) Todo o slide do microscópio pode ser preenchido com um total de 3 isoladores, resultando em até espaçamento e localização reprodutível das tampas. (C) Para cada deslizamento de cobertura, adicione uma gota de mídia de montagem de fluorescência no slide do microscópio dentro do espaço aberto do isolador. (D) Usando fórceps, pegue uma mancha de cobertura da placa de 24 poços e coloque-a de cabeça para baixo na gota da mídia de montagem. (E) Quando as bolhas estiverem presentes entre o deslizamento de tampas e o deslizamento do microscópio, toque suavemente no deslizamento de tampas usando fórceps fechados para remover bolhas. Criado usando BioRender. Clique aqui para ver uma versão maior desta figura.
- Formato de microplacão de 96 poços
- Aspire os poços usando um adaptador de aspiração multicanal ligado a um vácuo e adicione 40 μL de mídia sem soro com 1 mg/mL de alto peso molecular (70 kDa) dextran de volta aos poços. Incubar as células em uma incubadora de células de 37 °C por 30 minutos.
NOTA: Dependendo das condições a serem estudadas, em vez de usar mídia fresca, a reutilização da mídia condicionada para carregamento de dextran pode ser preferida, pois conteria fatores secretados ou suplementados, como EGF ou compostos inibidores, respectivamente, que podem afetar a capacidade macropinocítica das células. - Descarte a mídia na microplacão, movendo manualmente a placa de cabeça para baixo em um béquer 5 L vazio (Figura 3A).
- Enxágüe as células da microplaca submergindo lentamente a placa verticalmente, em um leve ângulo, em um béquer de 2 L cheio de PBS gelado (Figura 3B) e, posteriormente, elimine o PBS na microplaca, movendo a placa de cabeça para baixo no béquer 5 L (Figura 3C). Repita duas vezes.
NOTA: As células que se prendem fracamente à microplacão de imagem podem se desprender durante este processo. Se necessário, os poços também podem ser aspirados com um adaptador de aspiração multicanal ou lavados suavemente com PBS usando uma pipeta multicanal. O processamento de uma microplacão de 96 poços exigirá aproximadamente 2 L de PBS gelado. Se mais placas forem analisadas, use um béquer maior e adicione 1 L de PBS gelado para cada placa adicional ou refresque o PBS gelado conforme necessário. - Após o descarte do PBS da última lavagem, fixe as células por 20 minutos à temperatura ambiente adicionando 100 μL de formaldeído de 3,7% em PBS a cada poço utilizando um reservatório de reagente de 25 mL e uma pipeta multicanal (Figura 3D).
- Remova a solução de fixação e lave as células com PBS duas vezes usando a técnica de submersão e movimento. Manche os núcleos com 100 μL de 2 μg/mL DAPI em PBS por poço.
- Após 20 min, enxágue as células três vezes com PBS gelado usando a técnica de submersão e movimento descrita acima (passo 3.2.3). Remova qualquer PBS residual tocando a microplacão de cabeça para baixo em uma limpeza sem fiapos e adicione 100 μL de PBS fresco a cada poço usando um reservatório de reagente de 25 mL e pipeta multicanal. Imagem as células agora ou armazená-las cobertas de luz a 4 °C por até uma semana.
NOTA: Alternativamente, uma solução de glicerol em PBS (em vez de PBS) pode ser usada para imagem e armazenamento para estabilizar melhor a fluorescência (Figura 4A). - Antes da imagem, deixe a placa equilibrar-se à temperatura ambiente. Limpe a microplacão com um lenço sem fiapos.
- Aspire os poços usando um adaptador de aspiração multicanal ligado a um vácuo e adicione 40 μL de mídia sem soro com 1 mg/mL de alto peso molecular (70 kDa) dextran de volta aos poços. Incubar as células em uma incubadora de células de 37 °C por 30 minutos.
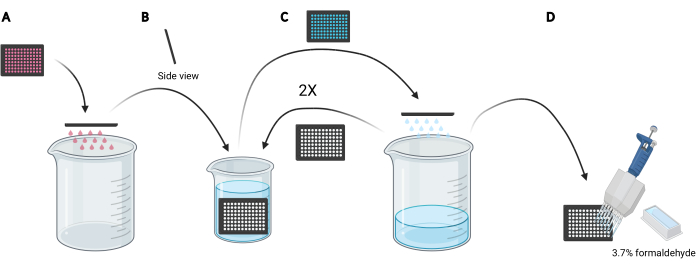
Figura 3: Enxaguar a microplacão de 96 poços para se preparar para fixação. (A) Esvazie a microplacão da mídia em um béquer de 5 L, piscando manualmente. (B) Verticalmente e em um leve ângulo, submergir lentamente a microplaca em um béquer de 2 L cheio de PBS gelado. (C) Esvazie a microplacão de PBS no béquer 5 L, piscando manualmente. Repita os passos de lavagem conforme descrito em B duas vezes. (D) Depois de esvaziar o PBS na microplacão pela última vez, adicione 100 μL 3,7% de formaldeído aos poços, utilizando uma pipeta multicanal. Criado usando BioRender. Clique aqui para ver uma versão maior desta figura.
4. Imagem macropinosa automatizada
Imagens de macropinossomos podem ser capturadas usando um microscópio fluorescente padrão, como descrito anteriormente19,20. No entanto, tal procedimento pode ser aprimorado em termos de eficiência através da automação, especialmente quando se avalia inúmeras condições diferentes de cultura celular. A automação da aquisição de imagens pode ser realizada através de um leitor de placas multimodesto de imagem celular, o que diminui o esforço reduzindo os procedimentos de manuseio e, principalmente, aumenta a reprodutibilidade e a confiabilidade dos dados através da aquisição de imagens de forma imparcial. Vários sistemas de imagem estão disponíveis comercialmente, e as direções diferem entre os instrumentos. Aqui, a aquisição de imagens usando uma Cytation 5 é descrita. No entanto, o protocolo abaixo pode ser adaptado a cada instrumento individual, aderindo às seguintes diretrizes:
- Crie um protocolo de automação para adquirir as imagens com um objetivo de ar de 40x no canal de comprimento de onda do fluoroforeiro de dextran (FITC/TMR) e DAPI.
NOTA: O inibidor de macropinotose comumente usado EIPA exibe autofluorescência no canal FITC, especialmente quando previamente animado no canal DAPI. Outros compostos que estão sendo testados também podem exibir autofluorescência. Para contornar esse problema, definir a aquisição de imagem para ocorrer primeiro no canal com o maior comprimento de onda de excitação (FITC/TMR) e segundo no canal DAPI ajuda a evitar essa ocorrência. - Otimizar as configurações de exposição usando uma amostra prevista para ter o mais alto nível de macropinocistose para evitar a superexposição, o que pode resultar em saturação do sinal e perda de dados de intensidade. Use configurações de foco que localizem a amostra de forma facilmente e consistente para produzir imagens de alta qualidade.
- Adquira várias imagens em cada poço ou se encobre para explicar a variabilidade da amostra e obter uma representação precisa da amostra.
- Uma vez determinadas as configurações de imagem, use as mesmas configurações para cada amostra dentro do experimento.
- Siga estas instruções para aquisição de imagens macropinossa ao usar um software Cytation 5 e Gen5:
- Placa de 24 poços com formato de deslizamento de tampas
- Inicie o leitor de placas e insira os slides do microscópio de cabeça para baixo usando o suporte de slides.
- Abra o leitor de microplaca e software de imagem, crie um novo protocolo clicando em Protocolos e Crie novos. Clique duas vezes em Procedimento e selecione o tipo de placa.
NOTA: Se o tipo de placa não estiver disponível, adicione o tipo de placa ao software clicando em System > Plate Types > Add Plate, e usando as dimensões da placa fornecidas pelo fabricante. Para facilitar o uso, o modelo para dois slides de microscópio com três tampas espaçadas usando os isoladores de silicone é fornecido no Arquivo Suplementar 1. - Para acessar as configurações de imagem, selecione Ações > Imagem > Imagem Invertida e clique em OK. Use o objetivo de fase 40x, PL FL com Wide FOV e Autofocus Binning.
- Para o primeiro canal, selecione o cubo LED correspondente ao rótulo fluorohore (GFP ou RFP) do Dextran. Desciclar a exposição automática e clique no botão ícone do microscópio para otimizar as configurações de exposição. Quando as configurações de exposição apropriadas tiverem sido determinadas, clique em Salvar configurações.
NOTA: Ajuste as configurações de exposição usando uma amostra prevista para ter o mais alto nível de macropinocistose para evitar imagens superexpostas, o que pode resultar em saturação do sinal e perda de dados de intensidade. - Repita a etapa anterior para o segundo canal usando o cubo LED DAPI.
- Defina as configurações de foco automático para cada canal de fluorescência, selecione Opções de foco. Desmarque o método de foco padrão e use Foco automático com Digitalização Opcional e Foco Automático sem Varredura Opcional para o canal dextran-fluorophore e DAPI, respectivamente. Clique em OK para salvar as configurações.
NOTA: A distância de varredura pode ser reduzida para 200 μm e o incremento para 20 μm para aumentar a eficiência do foco automático. A varredura opcional é necessária para foco automático adequado em amostras com baixa fluorescência. Isso ocorre normalmente ao analisar condições com baixa macropinocitose, como quando a macropinocise é inibida ou não está inatamente presente. - Use a opção Definir faróis para automatizar a aquisição de imagens em diferentes regiões do deslizamento de cobertura. Clique no ícone do microscópio e adicione sinalizadores clicando na janela da imagem e movendo o palco para a próxima região. Quando o número apropriado de regiões for selecionado, mova-se para o próximo deslizamento de cobertura e repita o processo. Para finalizar, clique em Salvar configurações.
NOTA: Para obter uma boa representação da macropinocistose em toda a amostra, selecione aproximadamente 20 balizas que estão distribuídas uniformemente através do deslizamento de tampas (Figura 4B). Menos sinalizadores podem ser usados, mas algumas imagens podem ter que ser excluídas da análise de imagem após a aquisição devido a discrepâncias de qualidade, como quando a imagem está fora de foco ou contém bolhas ou manchas fluorescentes e manchas fluorescentes. - Para concluir o ajuste das configurações de imagem, clique em OK. Para imaginar os clipes de cobertura, selecione Criar um novo experimento e ler agora a partir das ferramentas de protocolo. Salve o protocolo e o experimento quando solicitado.
- Placa de 24 poços com formato de deslizamento de tampas
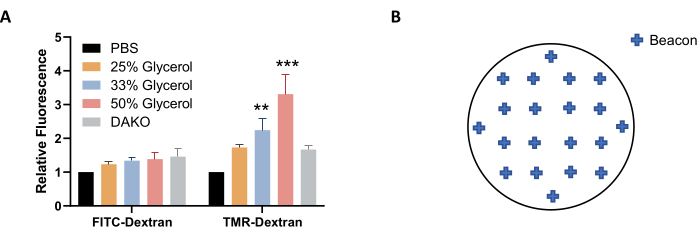
Figura 4: Otimização das condições para aquisição de imagens. (A) O aumento da concentração de glicerol aumenta a fluorescência TMR-dextran, conforme determinado nas células AsPC-1 tratadas com EGF. (B) Exemplos de coordenadas de faróis de imagem para aquisição automática de imagens ao usar a placa de 24 poços com formato de clipes de tampa. O gráfico da barra mostra a fluorescência relativa média com o SEM de 5 experimentos. A significância estatística foi determinada pela ANOVA bidirecional, em relação à PBS. ** p < 0,01; p < 0,001. Clique aqui para ver uma versão maior desta figura.
- Formato de microplacão de 96 poços
- Inicie o leitor de placas e insira a microplacão.
- Abra o leitor de microplaca e software de imagem, crie um novo protocolo clicando em Protocolos e Crie novos. Clique duas vezes em Procedimento e selecione o tipo de placa.
NOTA: Se o tipo de placa não estiver disponível, adicione o tipo de placa ao software clicando em System > Plate Types > Add Plate, e usando as dimensões da placa fornecidas pelo fabricante. Para facilitar o uso, o modelo para o CellCarrier-96 Ultra Microplates da PerkinElmer é fornecido no Arquivo Suplementar 2. - Para acessar as configurações de imagem, selecione Ações > Imagem > Imagem Invertida e clique em OK. Use o objetivo de fase 40x PL FL com Amplo FOV e Autofoco Binning.
- Para o primeiro canal, selecione o cubo LED correspondente ao rótulo fluorohore (GFP ou RFP) do Dextran. Desciclar a exposição automática e clique no botão ícone do microscópio para otimizar as configurações de exposição. Quando as configurações de exposição apropriadas tiverem sido determinadas, clique em Salvar configurações.
NOTA: Durante a otimização das configurações de exposição, pode ocorrer branqueamento significativo da fluorescência. Isso pode resultar em configurações que causam superexposição quando um novo campo é imageado. Portanto, valide as configurações de exposição verificando um campo ainda não exposto e garantindo que nenhuma saturação do sinal ocorra nas configurações selecionadas. Não inclua os poços utilizados para a otimização do ajuste de exposição na quantificação macropinosa, pois a fluorescência diminuiu como resultado do branqueamento durante a otimização. Ajuste as configurações de exposição usando uma amostra prevista para ter o mais alto nível de macropinocitose para evitar imagens superexpostas, o que pode resultar em saturação do sinal e perda de dados de intensidade. - Repita a etapa anterior para o segundo canal usando o cubo LED DAPI.
- Defina as configurações de foco automático para cada canal de fluorescência, selecione Opções de foco. Desmarque o método de foco padrão e use Laser Autofocus. Capture uma varredura de referência após determinar o plano focal para uma visualização ideal de macropinossomos e núcleos. Clique em OK para salvar as configurações.
NOTA: A distância de varredura pode ser reduzida para 400 μm e o incremento para 3 μm para aumentar a eficiência do foco automático. Para que o laser se concentre automaticamente para funcionar corretamente, limpe a parte inferior da placa, seque e limpe a placa com uma limpeza sem fiapos antes da imagem. O foco automático a laser é um método superior de foco, pois requer tempo mínimo para encontrar o plano focal. Outros métodos de foco podem ser usados, mas, como nenhum anti-desbotamento foi adicionado aos poços, esses métodos podem causar branqueamento significativo das amostras que impactarão negativamente a coleta de dados. - Defina o deslocamento horizontal e vertical para zero e em Imagem Única selecione Montagem sem sobreposição e use imagens 3 x 3, dependendo de quantas células são desejadas para serem incluídas na análise.
NOTA: Dependendo do tamanho e densidade das células, mais ou menos imagens podem ser tiradas para obter uma avaliação representativa da macropinocise ao longo da amostra. Avaliar a macropinocise nas células AsPC-1 ou MIA PaCa-2 em condições variadas, não são observadas diferenças na interpretação dos dados entre um quadro fotográfico de 2 x 2 ou 4 x 4, embora a variação entre as amostras de réplica possa aumentar ao tirar menos fotos (Figura 5A,B). Aumentar ou diminuir o tamanho do quadro afetará o tempo necessário para digitalizar a placa. Dependendo do tempo de exposição, uma microplacão completa de 96 poços levará cerca de 1-1,5 h para digitalizar completamente usando um quadro 3 x 3. Um quadro 2 x 2 e 4 x 4 reduzirá pela metade ou dobrará esse tempo, respectivamente. - Para concluir o ajuste das configurações de imagem, clique em OK.
- Para imaginar a placa, selecione Criar um novo experimento e ler agora a partir das ferramentas de protocolo. Salve o protocolo e o experimento quando solicitado.
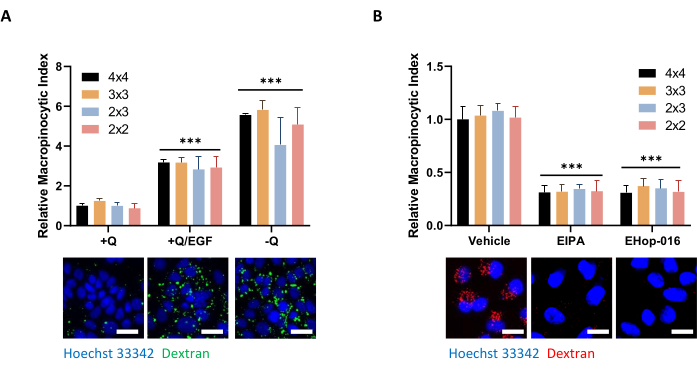
Figura 5: Condições de controle para avaliação da macropinocistose em células PDAC. (A) As células AsPC-1 exibem macropinocistose em resposta à estimulação EGF de 100 ng/mL por 5 min ou privação de glutamina por 24 h. Para aquisição de imagens, foram tiradas molduras de 4 x 4, 3 x 3, 2 x 3 ou 2 x 2 para determinar a influência do número de fotos na qualidade dos dados. (B) As células MIA PaCa-2 apresentam macropinocise constitutiva inibida por tratamento de 30 min com tratamento de 75 μM EIPA ou 2-h com 10 μM EHop-016. Quadros foram tirados como em A. Barra de escala = 25 μm. Gráficos de barras mostram o índice macropinolítico relativo médio com SD de 1 experimento com 4 réplicas. A significância estatística foi determinada por ANOVA bidirecional em relação à condição +Q ou veículo. p < 0.001 Clique aqui para ver uma versão maior desta figura.
5. Determinando o índice macropinocítico
O "índice macropinocítico" é a extensão da macropinocistose celular que é determinada pela quantificação da absorção fluorescente do dextran por célula por meio de imagens microscópicas19. Para isso, as imagens adquiridas são utilizadas para determinar a quantidade de dextran internalizado medindo a intensidade total da fluorescência ou área positiva da fluorescência e o número total de células determinadas pela coloração da DAPI. Essa análise pode ser realizada com softwares de processamento e análise de imagens de código aberto, como Cell Profiler ou FIJI/ImageJ, como descrito anteriormente19,20. No entanto, ao trabalhar com um leitor de placas de vários modos, o software fornecido com o instrumento pode incluir aplicativos de análise incorporados que podem ser usados para a computação do índice macropinóctico. Em alguns casos, o pipeline de análise de software incorporado pode não ser completamente aparente para o usuário. Recomenda-se, portanto, validar o software em um estágio inicial em comparação com um procedimento não automatizado, como Cell Profiler ou FIJI/ImageJ. Este protocolo pode ser adaptado a outras ferramentas de software de processamento e análise de imagens, aderindo às seguintes instruções gerais:
- Para a imagem DAPI e dextran correspondente, subtraia o fundo aplicando a função apropriada, frequentemente chamada de função bola rolando. Ajuste as configurações para que o ruído de fundo seja minimizado e não haja efeito mínimo de subtração no sinal DAPI e dextran.
- Usando um campo com sinal de dextran alto, determine as configurações de sinal de intensidade, frequentemente chamadas de função limiar, para selecionar os núcleos e determinar a configuração de sinal de intensidade mínima necessária para selecionar apenas os macropinoss.
- Para a imagem do Dextran, calcule a fluorescência total dentro da seleção macropinosa criada ou utilize a seleção para determinar a área total positiva para o dextran.
- Para a imagem DAPI, use a seleção para determinar o número de núcleos na imagem para refletir o número de células presentes.
- Para determinar o índice macropinocítico, divida a fluorescência ou área total do dextran pelo número de células determinadas pelo DAPI.
- Repita estas etapas de análise para todas as imagens adquiridas aplicando as mesmas configurações numéricas ao longo de toda parte.
- Siga estas instruções para determinar o índice macropinocítico ao usar o software Gen5:
NOTA: O pipeline de análise incorporado foi validado e não detectou diferenças na computação em relação ao Fiji/ImageJ (Figura 6A).- Após a conclusão da imagem, selecione uma imagem com alto nível de macropinocistose. Remova o sinal de fundo, clique em Processo (Figura Suplementar 1A) e selecione a opção Pré-processamento de imagem.
- Para o canal dextran, desmarque o Auto e utilize um diâmetro de bola rolando de 5 μm, priorize resultados finos e suavize a imagem com 1 ciclo.
- Para o canal DAPI, use pré-processamento automático e 1 ciclo suave. Clique em OK e adicione a etapa de pré-processamento de imagem ao protocolo; clique em ADD STEP. Em seguida, selecione a imagem processada sob o roll-out de imagem (Figura Suplementar 1B) e clique no botão Analisar (Figura Suplementar 1C).
- Em CONFIGURAÇÕES DE ANÁLISE, defina o Tipo de Análise Celular. Selecione o canal DAPI e clique em Opções (Figura Suplementar 2A).
- Para a máscara primária, usando a imagem DAPI processada, crie uma máscara para selecionar núcleos únicos. Use o fundo escuro e a opção Auto . Além disso, determine quais configurações permitem a seleção de máscaras de núcleos únicos e, quando concluída, clique no botão Aplicar para determinar se a máscara é aplicada adequadamente.
NOTA: Ativar as opções Split Touch Objects e Fill Holes in Masks pode funcionar melhor para selecionar núcleos únicos. Os tamanhos mínimos e máximos do objeto podem precisar ser ajustados dependendo da linha celular e são mais comumente definidos na faixa de 5-40 μm. Objetos de borda primária podem ser incluídos e toda a imagem deve ser analisada. O controle deslizante pode ser aplicado para ajustar a seleção da máscara à intensidade do sinal. - Em seguida, aplique uma máscara secundária para otimizar as configurações para selecionar o puncta fluorescente macropinoso. Use a Medida dentro de uma função máscara secundária e expanda a máscara primária em 40 μm, dependendo do tamanho das células.
- Use a função Limiar e o método Limiar na Máscara para selecionar as áreas positivas do dextran. Clique em Solicitar para determinar se as configurações são aplicadas corretamente.
NOTA: Para determinar o valor do limiar, utilize a ferramenta Perfil de Linha de Exibição (Figura Suplementar 2B) e desenhe uma linha sobre uma área dextran positivo (Figura Suplementar 2C). Use a intensidade medida para determinar a melhor configuração para criar uma máscara que seleciona macropinossos e exclui o sinal de fundo (Figura Suplementar 2D). - Depois de criar máscaras apropriadas para selecionar núcleos e macropinossomos, clique na guia Métricas Calculadas e selecione Selecionar ou Criar métricas de interesse em nível de objeto.
- Remova todas as métricas presentes e adicione as métricas Integral e Área para análise da máscara secundária. Clique em OK e selecione Calcular e Mostrar para as novas métricas. Quando terminar, clique em OK e selecione ADD STEP para adicionar a análise e os cálculos ao protocolo.
- Salve o protocolo finalizado para uso futuro, clique em Arquivo e Salvar Protocolo Como.
- Após o término da análise dos dados, selecione as métricas de interesse e exporte os dados para determinar o índice macropinocítico. Determine o índice macropinocítico da seguinte forma:
Fluorescência de dextran por célula = Objeto Int_2[Fluorophore Dextran]
Área do Dextran por célula = Objeto Area_2[Fluorophore Dextran]
NOTA: Para a placa de 24 poços com formato de deslizamentos de tampas, as métricas refletem a média do índice macropinocítico médio por imagem. Alternativamente, o índice macropinocítico pode ser calculado manualmente para toda a amostra dividindo a soma da 'Área' ou 'Integral' para todas as imagens pelo total 'Contagem de Células'. A diferença entre essas abordagens no cálculo do índice macropinocítico é mínima na maioria das configurações. Para o formato de microplacão de 96 poços, o índice macropinocítico é calculado como a média para toda a amostra. - Salve o protocolo para imagens e a análise automatizada subsequente. Reutilize o protocolo para experimentos futuros com os mesmos fluoroforos.
NOTA: Ao usar a função de foco automático a laser, uma nova varredura de referência deve ser feita quando uma linha celular diferente deve ser analisada, uma vez que núcleos e macropinoss são possivelmente localizados para um plano diferente. Cada vez que um novo experimento é realizado usando um protocolo previamente determinado, as configurações de exposição para esse experimento devem ser otimizadas.
6. Adição de tratamentos
Tratamentos celulares (pequenas moléculas, biológicas, fatores de crescimento, metabólitos etc.) podem ser incorporados em qualquer fase do protocolo, e o tempo preciso dependerá dos objetivos e objetivos do estudo.
- Prepare as células como na seção 2.
- Pouco antes de adicionar os tratamentos de interesse, prepare os tratamentos e os controles apropriados em duas concentrações finais em mídia sem soro. Prepare os tratamentos em volume igual ao volume do número de poços de replicação que estão sendo avaliados.
NOTA: Dado o papel que fatores secretados podem desempenhar no controle das funções celulares, pode ser preferível diluir os tratamentos de interesse em mídias condicionadas. Para esses fins, pode ser útil a sementes placas adicionais, como pratos de cultura celular de 6 cm ou 10 cm, ao preparar células conforme descrito na seção 2 para gerar a mídia condicionada para a preparação das soluções de tratamento. - Sem remover a mídia do poço, adicione um bem volume de solução de tratamento a cada poço. Agite a placa para garantir a mistura adequada. Incubar as células pelo tempo desejado.
- Continue com a seção 3.
NOTA: Ao adicionar o dextran, o uso de mídia fresca provoca a remoção dos tratamentos adicionados, o que pode afetar o nível de macropinocistose. Portanto, pode ser preferível adicionar o Dextran diretamente aos poços sem aspirar ou, alternativamente, reassumar os tratamentos ou reutilizar a mídia condicionada para preparar a solução do Dextran.
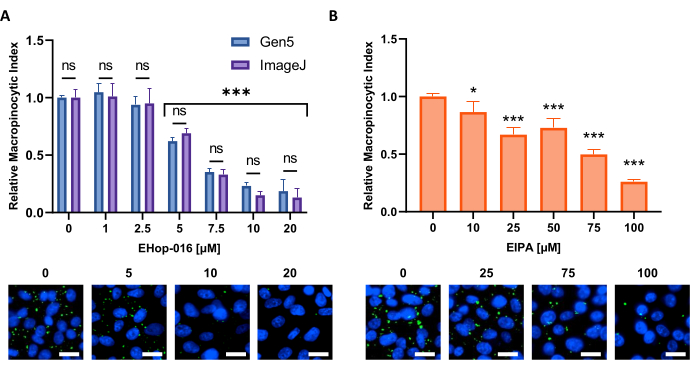
Figura 6: Realizar uma curva dose-resposta para inibidores de macropinocistose. Exemplos de dados obtidos ao testar inibidores conhecidos de macropinocistose em uma nova linha celular. As células PATU8998T foram utilizadas para o formato de microplacão de 96 poços e tratadas por 2h e 30 min com as concentrações indicadas de (A) EHop-16 e (B) EIPA, respectivamente. A comparação dos resultados obtidos através da análise de imagem pelo software Gen5 ou ImageJ não mostra diferenças significativas entre as duas abordagens indicadas por ns in (A). Barra de escala = 25 μm. Gráficos de barras mostram a média e o SD de um único experimento com 4 réplicas. A significância estatística foi determinada por ANOVA de uma ou duas vias, em comparação com as condições não tratadas. * p < 0,05; p < 0,001. Clique aqui para ver uma versão maior desta figura.
Resultados
Quando as etapas e o ajuste do protocolo acima descrito são seguidos em conformidade, os resultados experimentais finais devem fornecer informações sobre se as condições de cultura celular estudadas ou inibidores induzem ou reduzem a macropinocistose na linha celular de interesse. Para fortalecer a validade desses achados, a inclusão das condições de controle permitirá a análise dos resultados para determinar se o experimento foi concluído com sucesso. Os controles de indução da macropinocitose fornecerão i...
Discussão
A qualidade dos experimentos e a aquisição de dados dependem muito da qualidade dos reagentes, da otimização das configurações e da limpeza das tampas e das microplacões. Os resultados finais devem dar uma variação mínima entre as réplicas; no entanto, as variações biológicas ocorrem naturalmente ou podem ser causadas por uma série de fatores. A densidade celular pode fazer com que as células respondam mais ou menos aos indutores ou inibidores da macropinocitose. É, portanto, crucial aderir à confluênc...
Divulgações
C.C. é um inventor de uma patente emitida intitulada ''Diagnóstico de câncer, terapêutica e descoberta de medicamentos associados à macropinocistose'', Patente nº 9.983.194.
Agradecimentos
Este trabalho foi apoiado por subvenções do NIH/NCI (R01CA207189, R21CA243701) a C.C. KMO.G. é um beneficiário do TRDRP Postdoctoral Fellowship Award (T30FT0952). O BioTek Cytation 5 é uma parte do Núcleo de Imagens de Células Prebys de Sanford Burnham, que recebe apoio financeiro do NCI Cancer Center Support Grant (P30 CA030199). As figuras 1-3 foram criadas utilizando-se o BioRender.
Materiais
| Name | Company | Catalog Number | Comments |
| 0.25% Trypsin | Corning | 25053CI | 0.1% EDTA in HBSS w/o Calcium, Magnesium and Sodium Bicarbonate |
| 1.5 mL Microcentrifuge tube | Fisherbrand | 05-408-129 | |
| 10-cm Tissue culture dish | Greiner Bio-One | 664160 | CELLSTAR |
| 15 mL Centrifuge tube | Fisherbrand | 07-200-886 | |
| 2 L Beaker | Fisherbrand | 02-591-33 | |
| 24-well Tissue culture plate | Greiner Bio-One | 662160 | CELLSTAR |
| 25 mL Reagent reservoir | Genesee Scientific Corporation | 28-121 | |
| 500 mL Beaker | Fisherbrand | 02-591-30 | |
| 6-cm Tissue culture dish | Greiner Bio-One | 628160 | CELLSTAR |
| 8-Channel aspiration adapter | Integra Biosciences | 155503 | |
| 8-Channel aspiration adapter for standard tips | Integra Biosciences | 159024 | |
| 95% Ethanol | Decon Laboratories Inc | 4355226 | |
| Ammonia-free glass cleaner | Sparkle | FUN20500CT | |
| Black 96-well high-content screening microplate | PerkinElmer | 6055300 | CellCarrier-96 Ultra |
| Cotton-tipped applicator | Fisherbrand | 23-400-101 | |
| Coverslips | Fisherbrand | 12-545-80 | 12 mm diameter |
| Cytation 5 Cell Imaging Multi-Mode Reader | Biotek | CYT5FW | |
| DAPI | Millipore Sigma | 5.08741 | |
| Dextran 70 kDa - FITC | Life Technologies | D1822 | Lysine-fixable |
| Dextran 70 kDa - TMR | Life Technologies | D1819 | |
| DMSO | Millipore Sigma | D1435 | |
| DPBS | Corning | 21031CV | Without Calcium and Magnesium |
| Forceps | Fine Science Tools | 11251-20 | Dumont #5 |
| Formaldehyde, 37% | Ricca Chemical | RSOF0010-250A | ACS Reagent Grade |
| Glycerol | Fisher BioReagents | BP229-1 | |
| Hardening fluorescence mounting media | Agilent Tech | S302380-2 | DAKO |
| Hoechst 33342 | Millipore Sigma | B2261 | |
| Hydrochloric acid (HCl) | Fisher Chemical | A144-212 | Certified ACS Plus, 36.5%–38.0% |
| Lint-free wipes | Kimberly-Clark | 34155 | Kimwipes |
| Miscroscope slides | Fisherbrand | 12-544-1 | Premium plain glass |
| Multichannel pipette | Gilson | FA10013 | 8 channels, 0.5–10 µL |
| Multichannel pipette | Gilson | FA10012 | 12 channels, 20–200 µL |
| Multichannel pipette | Gilson | FA10011 | 8 channels, 20–200 µL |
| Parafilm M | Pechiney | PM996 | |
| Plastic wrap | Kirkland Signature | 208733 | Stretch-Tite |
| Silicone isolators | Grace Bio Labs Inc | 664107 | 13 mm Diameter X 0.8 mm Depth ID, 25 mm X 25 mm |
| Slide adapter | Biotek | 1220548 | |
| Wash bottle | Fisherbrand | FB0340922C |
Referências
- Lin, X. P., Mintern, J. D., Gleeson, P. A. Macropinocytosis in different cell types: similarities and differences. Membranes. 10 (8), 21 (2020).
- Recouvreux, M. V., Commisso, C. Macropinocytosis: a metabolic adaptation to nutrient stress in cancer. Frontiers in Endocrinology. 8, (2017).
- Zhang, Y. J., Commisso, C. Macropinocytosis in cancer: a complex signaling network. Trends in Cancer. 5 (6), 332-334 (2019).
- Commisso, C., et al. Macropinocytosis of protein is an amino acid supply route in Ras-transformed cells. Nature. 497 (7451), 633-637 (2013).
- Kim, S. M., et al. PTEN deficiency and AMPK activation promote nutrient scavenging and anabolism in prostate cancer cells. Cancer Discovery. 8 (7), 866-883 (2018).
- Jayashankar, V., Edinger, A. L. Macropinocytosis confers resistance to therapies targeting cancer anabolism. Nature Communications. 11 (1), (2020).
- Kamphorst, J. J., et al. Human pancreatic cancer tumors are nutrient poor and tumor cells actively scavenge extracellular protein. Cancer Research. 75 (3), 544-553 (2015).
- Olivares, O., et al. Collagen-derived proline promotes pancreatic ductal adenocarcinoma cell survival under nutrient limited conditions. Nature Communications. 8, (2017).
- Seguin, L., et al. Galectin-3, a druggable vulnerability for KRAS-addicted cancers. Cancer Discovery. 7 (12), 1464-1479 (2017).
- Redelman-Sidi, G., et al. The canonical Wnt pathway drives macropinocytosis in cancer. Cancer Research. 78 (16), 4658-4670 (2018).
- Tejeda-Munoz, N., Albrecht, L. V., Bui, M. H., De Robertis, E. M. Wnt canonical pathway activates macropinocytosis and lysosomal degradation of extracellular proteins. Proceedings of the National Academy of Sciences of the United States of America. 116 (21), 10402-10411 (2019).
- Schmees, C., et al. Macropinocytosis of the PDGF beta-receptor promotes fibroblast transformation by H-RasG12V. Molecular Biology of the Cell. 23 (13), 2571-2582 (2012).
- Lee, S. -. W., et al. EGFR-Pak signaling selectively regulates glutamine deprivation-induced macropinocytosis. Developmental Cell. 50 (3), 381-392 (2019).
- Recouvreux, M. V., et al. Glutamine depletion regulates Slug to promote EMT and metastasis in pancreatic cancer. Journal of Experimental Medicine. 217 (9), (2020).
- Zhang, Y., et al. Macropinocytosis in cancer-associated fibroblasts is dependent on CaMKK2/ARHGEF2 signaling and functions to support tumor and stromal cell fitness. Cancer Discovery. 11 (7), 1808-1825 (2021).
- Su, H., et al. Cancer cells escape autophagy inhibition via NRF2-induced macropinocytosis. Cancer Cell. 39 (5), 678-693 (2021).
- Bar-Sagi, D., Feramisco, J. R. Induction of membrane ruffling and fluid-phase pinocytosis in quiescent fibroblasts by ras proteins. Science. 233 (4768), 1061-1068 (1986).
- Mishra, R., et al. Stromal epigenetic alterations drive metabolic and neuroendocrine prostate cancer reprogramming. Journal of Clinical Investigation. 128 (10), 4472-4484 (2018).
- Commisso, C., Flinn, R. J., Bar-Sagi, D. Determining the macropinocytic index of cells through a quantitative image-based assay. Nature Protocols. 9 (1), 182-192 (2014).
- Galenkamp, K. M. O., Alas, B., Commisso, C. Quantitation of macropinocytosis in cancer cells. Methods in Molecular Biology. 1928, 113-123 (2019).
- Wang, J. T. H., Teasdale, R. D., Liebl, D. Macropinosome quantitation assay. MethodsX. 1, 36-41 (2014).
- Lee, S. -. W., Alas, B., Commisso, C. Detection and quantification of macropinosomes in pancreatic tumors. Methods in Molecular Biology. 1882, 171-181 (2019).
- Williams, T., Kay, R. R. High-throughput measurement of dictyostelium discoideum macropinocytosis by flow cytometry. Journal of Visualized Experiments: JoVE. (139), e58434 (2018).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados