È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Imaging e analisi automatizzati per la quantificazione di macropinosomi marcati fluorescentemente
In questo articolo
Riepilogo
I saggi automatizzati che utilizzano micropiastre multi-pozzo sono approcci vantaggiosi per identificare i regolatori di percorso consentendo la valutazione di una moltitudine di condizioni in un singolo esperimento. Qui, abbiamo adattato il consolidato protocollo di imaging e quantificazione dei macropinosomi a un formato di micropiastre a 96 pozzetti e forniamo uno schema completo per l'automazione utilizzando un lettore di piastre multimodale.
Abstract
La macropinocitosi è una via di assorbimento in fase fluida non specifica che consente alle cellule di internalizzare grandi carichi extracellulari, come proteine, agenti patogeni e detriti cellulari, attraverso l'endocitosi di massa. Questo percorso svolge un ruolo essenziale in una varietà di processi cellulari, tra cui la regolazione delle risposte immunitarie e il metabolismo delle cellule tumorali. Data questa importanza nella funzione biologica, l'esame delle condizioni di coltura cellulare può fornire informazioni preziose identificando i regolatori di questo percorso e ottimizzando le condizioni da impiegare nella scoperta di nuovi approcci terapeutici. Lo studio descrive una tecnica automatizzata di imaging e analisi che utilizza apparecchiature di laboratorio standard e un lettore di piastre multimodale per l'imaging cellulare per la rapida quantificazione dell'indice macropinocitico nelle cellule aderenti. Il metodo automatizzato si basa sull'assorbimento di destrano fluorescente ad alto peso molecolare e può essere applicato a micropiastre a 96 pozzetti per facilitare la valutazione di più condizioni in un esperimento o campioni fissi montati su coperture di vetro. Questo approccio mira a massimizzare la riproducibilità e ridurre la variazione sperimentale, risparmiando tempo ed economicamente vantaggioso.
Introduzione
La via endocitica non specifica della macropinocitosi consente alle cellule di interiorizzare una varietà di componenti extracellulari, tra cui nutrienti, proteine, antigeni e agenti patogeni, attraverso l'assorbimento di massa del fluido extracellulare e dei suoi costituenti1. Sebbene importante per la biologia di numerosi tipi cellulari, sempre più spesso, la via della macropinocitosi è descritta per svolgere un ruolo essenziale nella biologia tumorale, dove, attraverso l'assorbimento macropinocitico, le cellule tumorali sono in grado di sopravvivere e proliferare in presenza di un microambiente impoverito di nutrienti2,3. L'assorbimento di macromolecole extracellulari, tra cui albumina e matrice extracellulare, e detriti cellulari necrotici, fornisce una fonte alternativa di nutrienti per la produzione di biomassa creando aminoacidi, zuccheri, lipidi e nucleotidi attraverso il catabolismo cargo mediato da macropinosomi e lisosomi4,5,6,7,8.
L'induzione e la regolazione della macropinocitosi sono complesse e possono variare a seconda del contesto cellulare. Finora, sono stati identificati diversi induttori della macropinocitosi e includono ligandi, come il fattore di crescita epidermico (EGF), il fattore di crescita derivato dalle piastrine (PDGF), la galectina-3 e Wnt3A9,10,11,12,13. Inoltre, le condizioni di coltura che imitano il microambiente tumorale possono innescare l'attivazione del percorso. I tumori dell'adenocarcinoma duttale pancreatico (PDAC) sono privi di nutrienti, in particolare per l'aminoacido glutammina, che fa sì che sia le cellule tumorali che i fibroblasti associati al cancro (CAS) si affidino alla macropinocitosi per la sopravvivenza7,13,14,15. Inoltre, gli stress tumorali, come l'ipossia e lo stress ossidativo, possono attivare questa via di scavenging16. Oltre ai numerosi influenzatori estrinseci che possono indurre macropinocitosi, una varietà di percorsi intracellulari controlla la formazione di macropinosomi. La trasformazione oncogenica mediata da Ras è sufficiente per avviare il meccanismo macropinocitico e più tipi di cancro presentano macropinocitosi costitutiva oncogenica guidata da Ras4,5,9,17. In alternativa, sono state identificate l'attivazione di Ras wild-type e le vie Ras-indipendenti per attivare la macropinocitosi nelle cellule tumorali e nelle CAF10,11,15,18. L'uso di vari modelli in vitro in combinazione con trattamenti inibitori ha portato all'identificazione di diversi modulatori della macropinocitosi, che includono scambiatori di sodio-idrogeno, la piccola GTPasi Rac1, fosfoinositide 3-chinasi (PI3K), chinasi attivata da p21 (Pak) e proteina chinasi attivata da AMP (AMPK)4,13,15 . Tuttavia, data la moltitudine di fattori e condizioni descritti che regolano la macropinocitosi, è concepibile che molti più modulatori e stimoli rimangano sconosciuti. L'identificazione di nuovi modulatori e stimoli può essere facilitata dalla valutazione automatizzata di una moltitudine di condizioni in un singolo esperimento. Questa metodologia può far luce sui fattori coinvolti nella formazione dei macropinosomi e può consentire la scoperta di nuove piccole molecole o biologici che mirano a questo percorso.
Qui, abbiamo adattato il nostro protocollo precedentemente stabilito per determinare l'estensione della macropinocitosi nelle cellule tumorali in vitro a un formato di micropiastre a 96 pozzetti e l'imaging e la quantificazione automatizzati19,20. Questo protocollo si basa sulla microscopia fluorescente, che è diventata uno standard nel campo per determinare la macropinocitosi in vitro e in vivo4,5,6,7,9,10,11,12,13,15,16,17,18, 19,20,21,22. I macropinosomi possono essere distinti da altre vie endocitiche attraverso la loro capacità di internalizzare macromolecole di grandi dimensioni, come il destrano ad alto peso molecolare (cioè 70 kDa)2,3,4,20,21,22,23. Pertanto, i macropinosomi possono essere definiti attraverso l'assorbimento di 70 kDa destrano 70 kDa somministrato extracellulare. Di conseguenza, le vescicole macropinocitiche si manifestano come cluster intracellulari di puncta fluorescenti con dimensioni comprese tra 0,2 e 5 μm. Questi puncta possono essere ripresi al microscopio e successivamente quantificati per determinare l'estensione della macropinocitosi nella cellula - "l'indice macropinocitico".
In questo protocollo, vengono descritti i passaggi essenziali per visualizzare i macropinosomi nelle cellule aderenti in vitro su una micropiastra a 96 pozzetti e coverslip utilizzando apparecchiature di laboratorio standard (Figura 1). Inoltre, vengono fornite le indicazioni per automatizzare l'acquisizione dell'immagine e la quantificazione dell'indice macropinocitico utilizzando un lettore di lastre multimodale di imaging cellulare. Questa automazione riduce tempi, costi e sforzi rispetto ai nostri protocolli descritti in precedenza19,20. Inoltre, evita l'acquisizione e l'analisi di immagini involontariamente distorte e quindi migliora la riproducibilità e l'affidabilità. Questo metodo può essere facilmente adattato a diversi tipi di celle o lettori di piastre o essere utilizzato per determinare caratteristiche macropinosomiche alternative, come dimensioni, numero e posizione. Il metodo qui descritto è particolarmente adatto per lo screening di condizioni di coltura cellulare che inducono macropinocitosi, l'identificazione di nuovi modulatori o l'ottimizzazione delle concentrazioni di farmaci di inibitori noti.
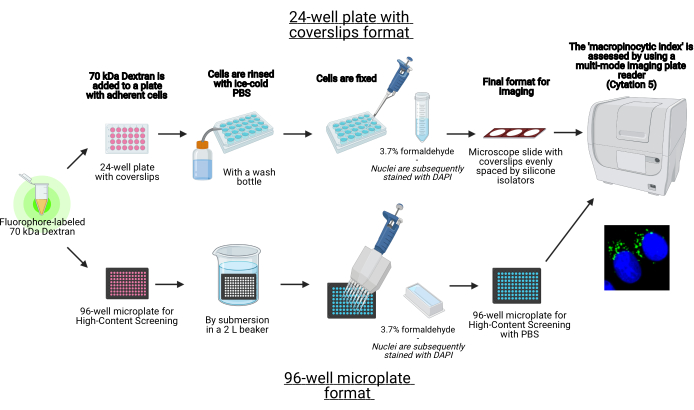
Figura 1: Schema del test automatizzato per determinare l'"indice macropinocitico" nelle cellule aderenti. Creato utilizzando BioRender. Fare clic qui per visualizzare una versione più grande di questa figura.
Protocollo
1. Preparazione dei materiali
- Sciogliere 70 kDa destrano marcato con FITC o tetrametilrodramina (TMR) in PBS per ottenere una soluzione da 20 mg/mL. Conservare le aliquote a -20 °C.
- Sciogliere DAPI in ddH2O per ottenere una soluzione da 1 mg/mL. Conservare le aliquote a -20 °C.
ATTENZIONE: DAPI è un potenziale cancerogeno e deve essere maneggiato con cura. - Il giorno della fissazione, preparare formaldeide fresca al 3,7% di grado ACS in PBS.
ATTENZIONE: La formaldeide è un fissativo, noto cancerogeno ed è tossico se inalato. Preparare la soluzione in una cappa aspirante chimica per evitare l'inalazione e maneggiarla con cura. - Preparare coverslips lavati con acido.
- Utilizzare un becher da 500 mL e un bagno d'acqua per riscaldare 28 g di coperture in vetro borosilicato con un diametro di 12 mm e uno spessore di 1,5 per 24 ore a 56 °C in 100 mL di 1 M HCl. Sigillare il becher con un involucro di plastica per evitare un'evaporazione estesa.
ATTENZIONE: HCl è un acido forte che è altamente corrosivo e deve quindi essere maneggiato con cura e utilizzato in una cappa chimica per evitare l'inalazione. - Lavare i coverslips con acqua distillata. Ripetere il lavaggio 4 volte. Quindi lavare le coperture con etanolo al 95%. Ripetere il lavaggio 4 volte.
- Conservare i coverslips lavati con acido in un piatto di coltura cellulare a temperatura ambiente per un uso futuro mantenendo la sterilità attraverso l'immersione in etanolo al 95%. Sigillare il piatto con parafilm per evitare un'evaporazione prolungata.
- Utilizzare un becher da 500 mL e un bagno d'acqua per riscaldare 28 g di coperture in vetro borosilicato con un diametro di 12 mm e uno spessore di 1,5 per 24 ore a 56 °C in 100 mL di 1 M HCl. Sigillare il becher con un involucro di plastica per evitare un'evaporazione estesa.
2. Preparazione delle cellule
- Utilizzando una piastra di coltura tissutale confluente di 10 cm con le cellule aderenti di interesse, aspirare il mezzo e risciacquare le cellule con 5 ml di DPBS, preriscaldate a 37 °C.
- Staccare le cellule dalla piastra aggiungendo 1,5 ml di tripsina allo 0,25% preriscaldata e incubando a 37 °C.
NOTA: Il tempo di incubazione della tripsina necessario per staccare le cellule di interesse deve essere determinato empiricamente e può essere confermato osservando il distacco al microscopio ottico convenzionale. - Raccogliere le cellule in un tubo di centrifuga da 15 mL e aggiungere 4,5 mL di mezzo completo per estinguere la tripsina.
- Pellet le celle per centrifugazione per 3 min a 200 x g e aspirare il surnatante.
- Risospesso il pellet cellulare in un volume adeguato di mezzi completi preribalizzati per ottenere una sospensione a cella singola per la semina.
- Procedere a seminare le cellule su una piastra a 24 pozzetti con coverslips o un formato micropiastra a 96 pozzetti (Figura 1).
NOTA: Il numero di cellule da seminare deve essere determinato empiricamente per ogni linea cellulare poiché i tassi di proliferazione e le dimensioni variano tra le linee cellulari. Questo protocollo è stato ottimizzato per le cellule tumorali aderenti con confluenza cellulare dell'80% il giorno dell'etichettatura dei macropinosomi. La confluenza cellulare può influenzare la capacità macropinocitica, e questo dovrebbe anche essere determinato empiricamente.- Piastra a 24 pozzetti con formato coverslip
- Aggiungi coverslips a una piastra di coltura tissutale a 24 pozzetti, usa una pinza per afferrare un singolo coverslip dal bagno di etanolo. Toccare il coperchio alla parete interna della piastra per rimuovere l'etanolo in eccesso e posizionare il coperchio piatto sul fondo di un pozzo.
- Lasciare evaporare l'etanolo e lavare il coverslip 2 volte con DPBS.
- Semina le cellule sopra il coperchio aggiungendo 500 μL della sospensione cellulare a ciascun pozzetto. Posizionare le cellule in un incubatore cellulare a 37 °C con il 5% di CO2 fino a quando la confluenza cellulare raggiunge il 60%-80% il giorno prima dell'etichettatura dei macropinosomi.
- Il giorno prima dell'etichettatura dei macropinosomi, aspirare i mezzi dai pozzetti e aggiungere 500 μL di mezzi privi di siero preriscaldati a ciascun pozzetto e posizionare le cellule in un incubatore cellulare a 37 °C con il 5% di CO2 per 16-24 ore.
NOTA: A seconda delle condizioni da studiare, si raccomanda di ridurre gli effetti dei fattori di crescita che possono indurre macropinocitosi e che sono normalmente presenti nel siero. Tuttavia, va considerato che la fame sierica può influenzare altri processi cellulari, come la proliferazione e l'autofagia. Poiché il siero residuo può influenzare la capacità macropinocitica delle cellule, così come l'attività inibitoria, la rimozione del siero può essere migliorata risciacquando delicatamente le cellule 1 o 2 volte con 500 μL di DPBS preriscaldato.
- Formato micropiastra a 96 pozzetti
- Trasferire la sospensione cellulare in un serbatoio di reagenti da 25 ml. Utilizzando una pipetta multicanale (8 o 12 canali), seminare 100 μL della sospensione cellulare in ciascun pozzetto di una micropiastra di screening nera ad alto contenuto a 96 pozzetti con olefina ciclica otticamente trasparente o fondo di vetro.
- Posizionare le cellule in un incubatore cellulare a 37 °C con il 5% di CO2 fino a quando la confluenza cellulare raggiunge il 60%-80% il giorno prima dell'etichettatura dei macropinosomi.
- Il giorno prima dell'etichettatura dei macropinosomi, rimuovere e scartare il supporto da ciascun pozzo utilizzando una pipetta multicanale (8 o 12 canali) o un adattatore di aspirazione multicanale per punte standard collegate a una pompa per vuoto.
- Utilizzando un serbatoio di reagenti e una pipetta multicanale (8 o 12 canali), aggiungere delicatamente 100 μL di supporti privi di siero preriscaldati a ciascun pozzetto. Posizionare le celle in un incubatore cellulare a 37 °C con il 5% di CO2 per 16-24 ore.
NOTA: A seconda delle condizioni da studiare, si raccomanda di ridurre gli effetti dei fattori di crescita che possono indurre macropinocitosi e che sono normalmente presenti nel siero. Tuttavia, va considerato che la fame sierica può influenzare altri processi cellulari, come la proliferazione e l'autofagia. Poiché il siero residuo può influenzare la capacità macropinocitica delle cellule, così come l'attività inibitoria, la rimozione del siero può essere migliorata risciacquando delicatamente le cellule 1 o 2 volte con 100 μL di DPBS preribalta.
- Piastra a 24 pozzetti con formato coverslip
3. Etichettatura dei macropinosomi
- Piastra a 24 pozzetti con formato coverslip
- Aspirare i pozzetti e aggiungere 200 μL di mezzi privi di siero con 1 mg/mL di destrano ad alto peso molecolare (70 kDa) marcato con fluoroforo. Posizionare le celle in un incubatore cellulare a 37 °C per 30 minuti.
NOTA: A seconda delle condizioni da studiare, invece di utilizzare mezzi freschi, può essere preferibile riutilizzare il mezzo condizionato per il carico di destrano in quanto conterrebbe fattori secreti o integrati, come EGF o composti inibitori, rispettivamente, che possono influenzare la capacità macropinocitica delle cellule. - Aspirare il fluido e lavare delicatamente ma rapidamente le celle 5 volte con PBS ghiacciato utilizzando un flacone di lavaggio preraffreddato. Scuotere saldamente la piastra a mano durante i lavaggi per aiutare a rimuovere gli aggregati destrani che si attaccano alle coperture.
- Fissare le cellule aggiungendo 350 μL di formaldeide al 3,7% e incubando per 20 minuti. Quindi, aspirare la soluzione di fissazione e lavare le celle con PBS due volte.
- Colorare i nuclei con 350 μL di 2 μg/mL DAPI in PBS. Dopo 20 minuti, aspirare la soluzione DAPI e lavare le cellule con PBS tre volte.
- Aderire agli isolatori in silicone affiancati su un vetrino per microscopio per ottenere una spaziatura uniforme e una localizzazione riproducibile dei coverslip, necessaria per l'automazione dell'imaging (Figura 2A,B).
NOTA: L'intero vetrino del microscopio può essere popolato con un totale di 3 isolatori. - Per ogni coverslip, aggiungere una goccia di supporto di montaggio a fluorescenza indurente sul vetrino del microscopio all'interno dello spazio aperto dell'isolatore (Figura 2C). Raccogli una copertina usando una pinza e rimuovi il PBS in eccesso picchiettando delicatamente il lato delle coperture su una salvietta priva di lanugine.
- Posizionare il coverslip capovolto sulla goccia del supporto di montaggio (Figura 2D). Toccare delicatamente il coperchio utilizzando una pinza chiusa per rimuovere le bolle dal supporto di montaggio (Figura 2E).
- Conservare le diapositive in un ambiente buio e lasciare asciugare il supporto di montaggio a temperatura ambiente, in genere impiegando 16-24 ore. Le diapositive possono ora essere fotografate o conservate a -20 °C per un massimo di 2 settimane.
- Prima dell'imaging, rimuovere gli isolatori dal vetrino del microscopio. Lasciare che i vetrini si equilibrino a temperatura ambiente e pulire i coperchi utilizzando un applicatore con punta di cotone bagnato con detergente per vetri privo di ammoniaca. Successivamente, utilizzare un applicatore pulito con punta in cotone bagnato con etanolo al 70% per pulire e lasciare asciugare il coperchio.
- Aspirare i pozzetti e aggiungere 200 μL di mezzi privi di siero con 1 mg/mL di destrano ad alto peso molecolare (70 kDa) marcato con fluoroforo. Posizionare le celle in un incubatore cellulare a 37 °C per 30 minuti.
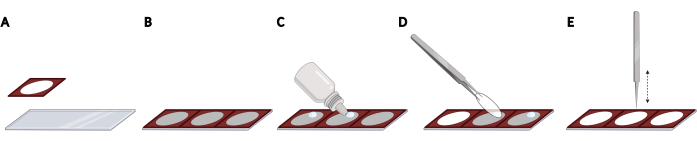
Figura 2: Posizionamento di coverslip su un vetrino per microscopio con isolatori in silicone. (A) Gli isolatori in silicone vengono premuti e aderiti a un vetrino per microscopio. (B) L'intero vetrino del microscopio può essere popolato con un totale di 3 isolatori, con conseguente spaziatura uniforme e localizzazione riproducibile dei coverslip. (C) Per ogni coverslip, aggiungere una goccia di supporto di montaggio a fluorescenza sul vetrino del microscopio all'interno dello spazio aperto dell'isolatore. (D) Usando una pinza, prelevare una slitta dalla piastra a 24 pozzetti e posizionarla a testa in giù sulla goccia del supporto di montaggio. (E) Quando sono presenti bolle tra il coperchio e il vetrino del microscopio, picchiettare delicatamente il coperchio usando una pinza chiusa per rimuovere le bolle. Creato utilizzando BioRender. Fare clic qui per visualizzare una versione più grande di questa figura.
- Formato micropiastra a 96 pozzetti
- Aspirare i pozzetti utilizzando un adattatore di aspirazione multicanale collegato a un vuoto e aggiungere 40 μL di mezzi privi di siero con 1 mg/mL di destrano ad alto peso molecolare (70 kDa) marcati con fluoroforo ai pozzetti. Incubare le cellule in un incubatore cellulare a 37 °C per 30 minuti.
NOTA: A seconda delle condizioni da studiare, invece di utilizzare mezzi freschi, può essere preferibile riutilizzare il mezzo condizionato per il carico di destrano in quanto conterrebbe fattori secreti o integrati, come EGF o composti inibitori, rispettivamente che possono influenzare la capacità macropinocitica delle cellule. - Smaltire il fluido nella micropiastra facendo scorrere manualmente la piastra a testa in giù in un becher vuoto da 5 L (Figura 3A).
- Risciacquare le cellule nella micropiastra immergendo lentamente la piastra verticalmente, con una leggera angolazione, in un becher da 2 L riempito con PBS ghiacciato (Figura 3B) e successivamente smaltire il PBS nella micropiastra facendo scorrere la piastra capovolta nel becher da 5 L (Figura 3C). Ripeti 2 volte.
NOTA: le cellule che si attaccano debolmente alla micropiastra di imaging possono staccarsi durante questo processo. Se necessario, i pozzetti possono anche essere aspirati con un adattatore di aspirazione multicanale o più delicatamente lavati con PBS utilizzando una pipetta multicanale. L'elaborazione di una micropiastra a 96 pozzetti richiederà circa 2 L di PBS ghiacciato. Se devono essere analizzate più piastre, utilizzare un becher più grande e aggiungere 1 L di PBS ghiacciato per ogni piastra aggiuntiva o aggiornare il PBS ghiacciato se necessario. - Dopo lo smaltimento del PBS dall'ultimo risciacquo, fissare le celle per 20 minuti a temperatura ambiente aggiungendo 100 μL di formaldeide al 3,7% in PBS a ciascun pozzo utilizzando un serbatoio di reagenti da 25 ml e una pipetta multicanale (Figura 3D).
- Rimuovere la soluzione di fissaggio e lavare le celle con PBS due volte utilizzando la tecnica di immersione e scorrimento. Macchiare i nuclei con 100 μL di 2 μg/mL DAPI in PBS per pozzetto.
- Dopo 20 minuti, sciacquare le cellule tre volte con PBS ghiacciato utilizzando la tecnica di immersione e scorrimento sopra descritta (fase 3.2.3). Rimuovere eventuali PBS residui picchiettando la micropiastra a testa in giù su una salvietta priva di lanugine e aggiungere 100 μL di PBS fresco a ciascun pozzetto utilizzando un serbatoio di reagenti da 25 ml e una pipetta multicanale. Immaginate le cellule ora o conservatele coperte dalla luce a 4 °C per un massimo di una settimana.
NOTA: In alternativa, una soluzione di glicerolo in PBS (invece di PBS) può essere utilizzata per l'imaging e la conservazione per stabilizzare meglio la fluorescenza (Figura 4A). - Prima dell'imaging, lasciare che la piastra si equilibri a temperatura ambiente. Asciugare la micropiastra con una salvietta priva di lanugine.
- Aspirare i pozzetti utilizzando un adattatore di aspirazione multicanale collegato a un vuoto e aggiungere 40 μL di mezzi privi di siero con 1 mg/mL di destrano ad alto peso molecolare (70 kDa) marcati con fluoroforo ai pozzetti. Incubare le cellule in un incubatore cellulare a 37 °C per 30 minuti.
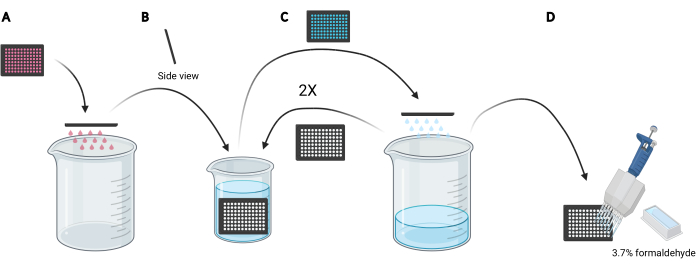
Figura 3: Risciacquo della micropiastra a 96 pozzetti per prepararsi alla fissazione. (A) Svuotare la micropiastra del supporto in un becher da 5 L scorrendo manualmente. (B) Verticalmente e con una leggera angolazione, immergere lentamente la micropiastra in un becher da 2 L riempito con PBS ghiacciato. (C) Svuotare la micropiastra di PBS nel becher da 5 L scorrendo manualmente. Ripetere due volte le fasi di lavaggio descritte al punto B . (D) Dopo aver svuotato per l'ultima volta il PBS nella micropiastra, aggiungere 100 μL di formaldeide al 3,7% ai pozzetti, utilizzando una pipetta multicanale. Creato utilizzando BioRender. Fare clic qui per visualizzare una versione più grande di questa figura.
4. Imaging automatizzato dei macropinosomi
Le immagini dei macropinosomi possono essere catturate utilizzando un microscopio fluorescente standard, come descritto in precedenza19,20. Tuttavia, tale procedura può essere migliorata in termini di efficienza attraverso l'automazione, specialmente quando si valutano numerose condizioni di coltura cellulare diverse. L'automazione dell'acquisizione delle immagini può essere realizzata tramite un lettore di lastre multimodale di imaging cellulare, che riduce lo sforzo riducendo le procedure di gestione e, soprattutto, aumenta la riproducibilità e l'affidabilità dei dati acquisendo immagini in modo imparziale. Sono disponibili in commercio più sistemi di imaging e le direzioni differiranno tra gli strumenti. Qui viene descritta l'acquisizione di immagini utilizzando un Cytation 5. Tuttavia, il protocollo riportato di seguito può essere adattato a ogni singolo strumento aderendo alle seguenti linee guida:
- Creare un protocollo di automazione per acquisire le immagini con un obiettivo d'aria 40x nel canale di lunghezza d'onda del fluoroforo destro (FITC/TMR) e DAPI.
NOTA: L'inibitore della macropinocitosi comunemente usato EIPA presenta autofluorescenza nel canale FITC, specialmente se precedentemente eccitato nel canale DAPI. Altri composti in fase di test potrebbero anche mostrare autofluorescenza. Per aggirare questo problema, l'impostazione dell'acquisizione dell'immagine in modo che avvenga prima nel canale con la lunghezza d'onda di eccitazione più alta (FITC/TMR) e la seconda nel canale DAPI consente di evitare questo evento. - Ottimizzare le impostazioni di esposizione utilizzando un campione che si prevede abbia il più alto livello di macropinocitosi per evitare la sovraesposizione, che può comportare la saturazione del segnale e la perdita di dati di intensità. Utilizzare le impostazioni di messa a fuoco che individuano in modo rapido e coerente il campione per produrre immagini di alta qualità.
- Acquisire più immagini in ogni pozzo o coverslip per tenere conto della variabilità del campione e ottenere una rappresentazione accurata del campione.
- Una volta determinate le impostazioni di imaging, utilizzare le stesse impostazioni per ogni campione all'interno dell'esperimento.
- Seguire queste istruzioni per l'acquisizione di immagini di macropinosomi quando si utilizza un software Cytation 5 e Gen5:
- Piastra a 24 pozzetti con formato coverslip
- Avviare il lettore di lastre e inserire i vetrini del microscopio a testa in giù utilizzando il supporto del vetrino.
- Aprire il lettore di micropiastre e il software di imaging, creare un nuovo protocollo facendo clic su Protocolli e Crea nuovo. Fare doppio clic su Procedura e selezionare il tipo di piastra.
NOTA: se il tipo di piastra non è disponibile, aggiungere il tipo di piastra al software facendo clic su Sistema > tipi di piastre > Aggiungi piastra e utilizzando le dimensioni della piastra fornite dal produttore. Per facilità d'uso, il modello per due vetrini per microscopio con tre coperchi distanziati utilizzando gli isolatori in silicone è fornito nel file supplementare 1. - Per accedere alle impostazioni di imaging, selezionare Azioni > Immagine > Imager invertito e fare clic su OK. Utilizza l'obiettivo di fase PL FL 40x con Wide FOV e Autofocus Binning.
- Per il primo canale, selezionare il cubo LED corrispondente all'etichetta del fluoroforo destro (GFP o RFP). Deselezionare Esposizione automatica e fare clic sul pulsante icona del microscopio per ottimizzare le impostazioni di esposizione. Quando sono state determinate le impostazioni di esposizione appropriate, fare clic su Salva impostazioni.
NOTA: regolare le impostazioni di esposizione utilizzando un campione che si prevede abbia il più alto livello di macropinocitosi per evitare immagini sovraesposte, che possono causare saturazione del segnale e perdita di dati di intensità. - Ripetere il passaggio precedente per il secondo canale utilizzando il cubo LED DAPI.
- Impostare le impostazioni di messa a fuoco automatica per ciascun canale di fluorescenza, selezionare Opzioni di messa a fuoco. Deselezionate il metodo di messa a fuoco predefinito e utilizzate autofocus con scansione opzionale e messa a fuoco automatica senza scansione opzionale rispettivamente per il canale destro-fluoroforo e DAPI. Fare clic su OK per salvare le impostazioni.
NOTA: la distanza di scansione può essere ridotta a 200 μm e l'incremento a 20 μm per aumentare l'efficienza dell'autofocus. La scansione opzionale è necessaria per un'adeguata messa a fuoco automatica su campioni con bassa fluorescenza. Ciò si verifica normalmente quando si analizzano condizioni con bassa macropinocitosi, come quando la macropinocitosi è inibita o non innatamente presente. - Utilizza l'opzione Definisci beacon per automatizzare l'acquisizione di immagini in diverse regioni del coverslip. Fai clic sull'icona del microscopio e aggiungi i beacon facendo clic nella finestra dell'immagine e spostando lo stage nella regione successiva. Quando è stato selezionato il numero appropriato di regioni, passare al coverslip successivo e ripetere il processo. Per finalizzare, fare clic su Salva impostazioni.
NOTA: per ottenere una buona rappresentazione della macropinocitosi nel campione, selezionare circa 20 beacon distribuiti uniformemente nel coverslip (Figura 4B). È possibile utilizzare meno beacon, ma alcune immagini potrebbero dover essere escluse dall'analisi delle immagini dopo l'acquisizione a causa di discrepanze di qualità, ad esempio quando l'immagine è sfocata o contiene bolle o macchie fluorescenti e macchie. - Per completare la regolazione delle impostazioni di imaging, fare clic su OK. Per visualizzare le immagini delle copertine, selezionare Crea un nuovo esperimento e Leggi ora dagli strumenti del protocollo. Salvare il protocollo e l'esperimento quando richiesto.
- Piastra a 24 pozzetti con formato coverslip
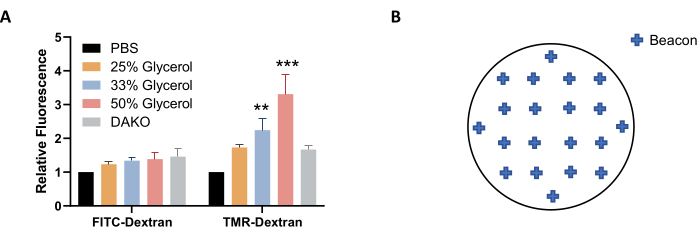
Figura 4: Ottimizzazione delle condizioni per l'acquisizione dell'immagine. (A) L'aumento della concentrazione di glicerolo aumenta la fluorescenza TMR-destrano, come determinato nelle cellule AsPC-1 trattate con EGF. (B) Coordinate di esempio di beacon di imaging per l'acquisizione automatica delle immagini quando si utilizza la piastra a 24 pozzetti con formato coverslips. Il grafico a barre mostra la fluorescenza relativa media con SEM di 5 esperimenti. La significatività statistica è stata determinata da ANOVA bidirezionale, rispetto alla PBS. ** p < 0,01; p < 0,001. Fare clic qui per visualizzare una versione più grande di questa figura.
- Formato micropiastra a 96 pozzetti
- Avviare il lettore di piastre e inserire la micropiastra.
- Aprire il lettore di micropiastre e il software di imaging, creare un nuovo protocollo facendo clic su Protocolli e Crea nuovo. Fare doppio clic su Procedura e selezionare il tipo di piastra.
NOTA: se il tipo di piastra non è disponibile, aggiungere il tipo di piastra al software facendo clic su Sistema > tipi di piastre > Aggiungi piastra e utilizzando le dimensioni della piastra fornite dal produttore. Per facilità d'uso, il modello per le micropiastre CellCarrier-96 Ultra di PerkinElmer è fornito nel file supplementare 2. - Per accedere alle impostazioni di imaging, selezionare Azioni > Immagine > Imager invertito e fare clic su OK. Usa l'obiettivo 40x PL FL Phase con Wide FOV e Autofocus Binning.
- Per il primo canale, selezionare il cubo LED corrispondente all'etichetta del fluoroforo destro (GFP o RFP). Deselezionare Esposizione automatica e fare clic sul pulsante icona del microscopio per ottimizzare le impostazioni di esposizione. Quando sono state determinate le impostazioni di esposizione appropriate, fare clic su Salva impostazioni.
NOTA: Durante l'ottimizzazione delle impostazioni di esposizione, può verificarsi un significativo sbiancamento a fluorescenza. Ciò può comportare impostazioni che causano sovraesposizione quando viene creata un'immagine di un nuovo campo. Pertanto, convalidare le impostazioni di esposizione controllando un campo non ancora esposto e assicurandosi che non si verifichi alcuna saturazione del segnale alle impostazioni selezionate. Non includere i pozzetti utilizzati per l'ottimizzazione dell'impostazione dell'esposizione nella quantificazione del macropinosoma, poiché la fluorescenza è diminuita a seguito dello sbiancamento durante l'ottimizzazione. Regolare le impostazioni di esposizione utilizzando un campione previsto per avere il più alto livello di macropinocitosi per evitare immagini sovraesposte, che possono causare saturazione del segnale e perdita di dati di intensità. - Ripetere il passaggio precedente per il secondo canale utilizzando il cubo LED DAPI.
- Impostare le impostazioni di messa a fuoco automatica per ciascun canale di fluorescenza, selezionare Opzioni di messa a fuoco. Deselezionate il metodo di messa a fuoco predefinito e utilizzate la messa a fuoco automatica laser. Acquisire una scansione di riferimento dopo aver determinato il piano focale per una visualizzazione ottimale di macropinosomi e nuclei. Fare clic su OK per salvare le impostazioni.
NOTA: la distanza di scansione può essere ridotta a 400 μm e l'incremento a 3 μm per aumentare l'efficienza dell'autofocus. Affinché l'opzione di messa a fuoco automatica laser funzioni correttamente, pulire la parte inferiore della piastra, asciugare e pulire la piastra con una salvietta priva di lanugine prima dell'imaging. L'autofocus laser è un metodo superiore per la messa a fuoco in quanto richiede un tempo minimo per trovare il piano focale. Possono essere utilizzati altri metodi di messa a fuoco, ma, poiché non è stato aggiunto alcun anti-dissolvenza ai pozzetti, questi metodi possono causare uno sbiancamento significativo dei campioni che avrà un impatto negativo sulla raccolta dei dati. - Impostare l'offset orizzontale e verticale su zero e in Immagine singola selezionare Montaggio senza sovrapposizione e utilizzare immagini 3 x 3, a seconda del numero di celle che si desidera includere nell'analisi.
NOTA: A seconda delle dimensioni e della densità delle cellule, è possibile acquisire più o meno immagini per ottenere una valutazione rappresentativa della macropinocitosi in tutto il campione. Valutando la macropinocitosi nelle cellule AsPC-1 o MIA PaCa-2 in condizioni variabili, non si osservano differenze nell'interpretazione dei dati tra una cornice per foto 2 x 2 o 4 x 4, sebbene la variazione tra i campioni replicati possa aumentare quando si scattano meno foto (Figura 5A,B). Aumentare o diminuire le dimensioni del telaio influenzerà il tempo necessario per scansionare la piastra. A seconda del tempo di esposizione, una micropiastra completa a 96 pozzetti impiegherà circa 1-1,5 ore per eseguire la scansione completa utilizzando un fotogramma 3 x 3. Un fotogramma 2 x 2 e 4 x 4 dimezzerà o raddoppierà quel tempo, rispettivamente. - Per completare la regolazione delle impostazioni di imaging, fare clic su OK.
- Per creare un'immagine della piastra, selezionate Crea un nuovo esperimento e Leggi ora dagli strumenti del protocollo. Salvare il protocollo e l'esperimento quando richiesto.
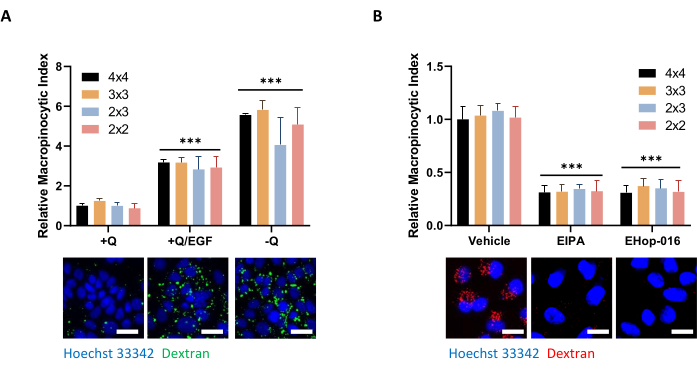
Figura 5: Condizioni di controllo per la valutazione della macropinocitosi nelle cellule PDAC. (A) Le cellule AsPC-1 mostrano macropinocitosi in risposta a 100 ng/mL di stimolazione EGF per 5 minuti o privazione di glutammina per 24 ore. Per l'acquisizione di immagini, sono state scattate cornici di 4 x 4, 3 x 3, 2 x 3 o 2 x 2 per determinare l'influenza del numero di foto sulla qualità dei dati. (B) Le cellule MIA PaCa-2 mostrano una macropinocitosi costitutiva inibita dal trattamento di 30 minuti con EIPA da 75 μM o dal trattamento di 2 ore con EHop-016 da 10 μM. Le cornici sono state scattate come in A. Barra della scala = 25 μm. I grafici a barre mostrano l'indice macropinocitico relativo medio con SD di 1 esperimento con 4 repliche. La significatività statistica è stata determinata da ANOVA bidirezionale rispetto al +Q o alla condizione del veicolo. p < 0,001 Fare clic qui per visualizzare una versione più grande di questa figura.
5. Determinazione dell'indice macropinocitico
L'"indice macropinocitico" è l'estensione della macropinocitosi cellulare che viene determinata quantificando l'assorbimento fluorescente del destrano per cellula utilizzando l'imaging microscopico19. A tal fine, le immagini acquisite vengono utilizzate per determinare la quantità di destrano interiorizzato misurando l'intensità totale della fluorescenza o l'area positiva alla fluorescenza e il numero totale di cellule determinato dalla colorazione DAPI. Questa analisi può essere eseguita con software di elaborazione e analisi delle immagini open source, come Cell Profiler o FIJI / ImageJ, come descritto in precedenza19,20. Tuttavia, quando si lavora con un lettore di piastre multimodale, il software fornito con lo strumento può includere applicazioni di analisi integrate che possono essere utilizzate ai fini del calcolo dell'indice macropinocitico. In alcuni casi, la pipeline di analisi software integrata potrebbe non essere completamente evidente all'utente. Si consiglia pertanto di convalidare il software in una fase iniziale rispetto a una procedura non automatizzata, come Cell Profiler o FIJI/ImageJ. Questo protocollo può essere adattato ad altri strumenti software di elaborazione e analisi delle immagini aderendo alle seguenti istruzioni generali:
- Per il DAPI e l'immagine destrana corrispondente, sottrarre lo sfondo applicando la funzione appropriata, spesso chiamata funzione rolling ball. Regolare le impostazioni in modo che il rumore di fondo sia ridotto al minimo e che vi sia un effetto di sottrazione minimo o nullo sul segnale DAPI e destro.
- Utilizzando un campo con segnale ad alto destro, determinare le impostazioni del segnale di intensità, spesso chiamato funzione di soglia, per selezionare i nuclei e determinare l'impostazione del segnale di intensità minima richiesta per selezionare solo i macropinosomi.
- Per l'immagine del destro, calcolare la fluorescenza totale all'interno della selezione del macropinosoma creata o utilizzare la selezione per determinare l'area totale positiva per il destro.
- Per l'immagine DAPI, utilizzare la selezione per determinare il numero di nuclei nell'immagine per riflettere il numero di celle presenti.
- Per determinare l'indice macropinocitico, dividere la fluorescenza totale del destrano o l'area per il numero di cellule determinato dal DAPI.
- Ripeti questi passaggi di analisi per tutte le immagini acquisite applicando le stesse impostazioni numeriche in tutto.
- Seguire queste istruzioni per determinare l'indice macropinocitico quando si utilizza il software Gen5:
NOTA: la pipeline di analisi integrata è stata convalidata e non ha rilevato differenze nel calcolo rispetto a Fiji/ImageJ (Figura 6A).- Al termine dell'imaging, selezionare un'immagine con un alto livello di macropinocitosi. Rimuovere il segnale di sfondo, fare clic su Elabora (Figura supplementare 1A) e selezionare l'opzione Pre-elaborazione immagine .
- Per il canale destro, deselezionate Auto e utilizzate un diametro della sfera di rotolamento di 5 μm, date la priorità ai risultati finali e uniformate l'immagine con 1 ciclo.
- Per il canale DAPI, utilizzare Pre-elaborazione automatica e 1 ciclo regolare. Fare clic su OK e aggiungere la fase di pre-elaborazione dell'immagine al protocollo; fare clic su AGGIUNGI PASSAGGIO. Quindi, selezionare l'immagine elaborata sotto l'implementazione dell'immagine (Figura supplementare 1B) e fare clic sul pulsante Analizza (Figura supplementare 1C).
- In IMPOSTAZIONI ANALISI impostare Tipo su Analisi cellulare. Selezionare il canale DAPI e fare clic su Opzioni (Figura supplementare 2A).
- Per la maschera primaria, utilizzando l'immagine DAPI elaborata, creare una maschera per selezionare singoli nuclei. Usa lo sfondo scuro e l'opzione Auto . Inoltre, determinare quali impostazioni consentono la selezione della maschera di singoli nuclei e, al termine, fare clic sul pulsante Applica per determinare se la maschera viene applicata in modo appropriato.
NOTA: l'attivazione delle opzioni Dividi oggetti di tocco e Fori di riempimento nelle maschere può funzionare meglio per selezionare singoli nuclei. Potrebbe essere necessario regolare le dimensioni minime e massime degli oggetti in base alla linea cellulare e sono più comunemente impostate nell'intervallo 5-40 μm. È possibile includere oggetti perimetrali primari e analizzare l'intera immagine. Il cursore può essere applicato per regolare la selezione della maschera in base all'intensità del segnale. - Quindi, applicare una maschera secondaria per ottimizzare le impostazioni per la selezione del macropinosoma fluorescente puncta. Utilizzare la funzione Misura all'interno di una maschera secondaria ed espandere la maschera primaria di 40 μm a seconda delle dimensioni delle celle.
- Utilizzare la funzione Soglia e il metodo Soglia in maschera per selezionare le aree di destrano positive. Fare clic su Applica per determinare se le impostazioni sono applicate correttamente.
NOTA: per determinare il valore di soglia, utilizzare lo strumento Visualizza profilo linea (Figura supplementare 2B) e disegnare una linea su un'area destro-positiva (Figura supplementare 2C). Utilizzare l'intensità misurata per determinare l'impostazione migliore per creare una maschera che seleziona i macropinosomi ed esclude il segnale di fondo (Figura supplementare 2D). - Dopo aver creato maschere appropriate per selezionare nuclei e macropinosomi, fare clic sulla scheda Metriche calcolate e selezionare Seleziona o Crea metriche di interesse a livello di oggetto.
- Rimuovi tutte le metriche presenti e aggiungi le metriche Integral e Area per l'analisi della maschera secondaria. Fai clic su OK e seleziona Calcola e mostra per le nuove metriche. Al termine, fare clic su OK e selezionare AGGIUNGI PASSAGGIO per aggiungere l'analisi e i calcoli al protocollo.
- Salvare il protocollo finalizzato per un utilizzo futuro, fare clic su File e Salva protocollo con nome.
- Al termine dell'analisi dei dati, selezionare le metriche di interesse ed esportare i dati per determinare l'indice macropinocitico. Determinare l'indice macropinocitico come segue:
Fluorescenza destrana per cellula = Oggetto Int_2[Fluoroforo destro]
Area destrana per cella = Oggetto Area_2[Fluoroforo destro]
NOTA: per il formato a 24 pozzetti con coverslips, le metriche riflettono la media dell'indice macropinocitico medio per immagine. In alternativa, l'indice macropinocitico può essere calcolato manualmente per l'intero campione dividendo la somma dell'"Area" o "Integrale" per tutte le immagini per il totale "Conteggio delle cellule". La differenza tra questi approcci nel calcolo dell'indice macropinocitico è minima nella maggior parte delle impostazioni. Per il formato micropiastra a 96 pozzetti, l'indice macropinocitico viene calcolato come media per l'intero campione. - Salvare il protocollo per l'imaging e la successiva analisi automatizzata. Riutilizzare il protocollo per esperimenti futuri con gli stessi fluorofori.
NOTA: quando si utilizza la funzione di messa a fuoco automatica laser, è necessario eseguire una nuova scansione di riferimento quando si deve analizzare una linea cellulare diversa poiché nuclei e macropinosomi sono probabilmente localizzati su un piano diverso. Ogni volta che un nuovo esperimento viene eseguito utilizzando un protocollo determinato in precedenza, le impostazioni di esposizione per quell'esperimento devono essere ottimizzate.
6. Aggiunta di trattamenti
I trattamenti cellulari (piccole molecole, biologici, fattori di crescita, metaboliti ecc.) possono essere incorporati in qualsiasi fase del protocollo e la tempistica precisa dipenderà dagli obiettivi e dagli obiettivi dello studio.
- Preparare le cellule come nella sezione 2.
- Poco prima di aggiungere i trattamenti di interesse, preparare i trattamenti e gli opportuni controlli al doppio delle loro concentrazioni finali in mezzi privi di siero. Preparare i trattamenti in un volume pari al volume del numero di pozzetti replicati in fase di valutazione.
NOTA: Dato il ruolo che i fattori secreti possono svolgere nel controllo delle funzioni cellulari, si può preferire diluire i trattamenti di interesse nei mezzi condizionati. Per questi scopi, può essere utile seminare piastre aggiuntive, come piatti di coltura cellulare da 6 cm o 10 cm, durante la preparazione delle cellule come descritto nel paragrafo 2 per generare il mezzo condizionato per la preparazione delle soluzioni di trattamento. - Senza rimuovere il supporto dal pozzo, aggiungere un buon volume di soluzione di trattamento a ciascun pozzetto. Agitare la piastra per garantire una corretta miscelazione. Incubare le cellule per la quantità di tempo desiderata.
- Continuare con la sezione 3.
NOTA: Quando si aggiunge il destrano, l'uso di nuovi mezzi provoca la rimozione dei trattamenti aggiunti, che possono influenzare il livello di macropinocitosi. Pertanto, si può preferire aggiungere il destrano direttamente ai pozzetti senza aspirare o in alternativa riaggiungere i trattamenti o riutilizzare il mezzo condizionato per preparare la soluzione di destrano.
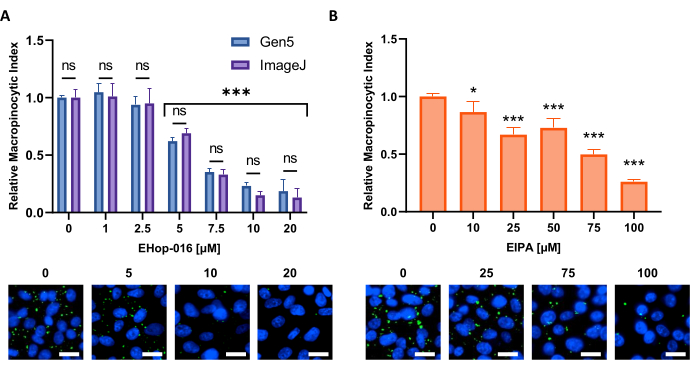
Figura 6: Esecuzione di una curva dose-risposta per gli inibitori della macropinocitosi. Dati di esempio ottenuti durante il test di inibitori noti della macropinocitosi in una nuova linea cellulare. Le cellule PATU8998T sono state utilizzate per il formato micropiastra a 96 pozzetti e trattate per 2 ore e 30 minuti con le concentrazioni indicate di (A) EHop-16 e (B) EIPA, rispettivamente. Il confronto dei risultati ottenuti attraverso l'analisi delle immagini da parte del software Gen5 o ImageJ non mostra differenze significative tra i due approcci come indicato da ns in (A). Barra della scala = 25 μm. I grafici a barre mostrano la media e la SD di un singolo esperimento con 4 repliche. La significatività statistica è stata determinata da ANOVA unidirezionale o bidirezionale, rispetto alle condizioni non trattate. * p < 0,05; p < 0,001. Fare clic qui per visualizzare una versione più grande di questa figura.
Risultati
Quando i passaggi e l'aggiustamento del protocollo sopra descritto vengono seguiti di conseguenza, i risultati sperimentali finali dovrebbero fornire informazioni sul fatto che le condizioni di coltura cellulare studiate o gli inibitori inducano o riducano la macropinocitosi nella linea cellulare di interesse. Per rafforzare la validità di questi risultati, l'inclusione di condizioni di controllo consentirà l'esame dei risultati per determinare se l'esperimento è stato completato con successo. I controlli di induzione...
Discussione
La qualità degli esperimenti e dell'acquisizione dei dati dipende molto dalla qualità dei reagenti, dall'ottimizzazione delle impostazioni e dalla pulizia delle coperture e delle micropiastre. I risultati finali dovrebbero dare una variazione minima tra le repliche; tuttavia, le variazioni biologiche si verificano naturalmente o possono essere altrimenti causate da una serie di fattori. La densità cellulare può indurre le cellule a rispondere più o meno agli induttori o agli inibitori della macropinocitosi. E' quind...
Divulgazioni
C.C. è un inventore di un brevetto rilasciato intitolato "Diagnostica del cancro, terapie e scoperta di farmaci associati alla macropinocitosi", brevetto n.: 9.983.194.
Riconoscimenti
Questo lavoro è stato supportato da sovvenzioni NIH / NCI (R01CA207189, R21CA243701) a C.C. KMO.G. ha ricevuto un TRDRP Postdoctoral Fellowship Award (T30FT0952). BioTek Cytation 5 fa parte del Sanford Burnham Prebys Cell Imaging Core, che riceve sostegno finanziario dal NCI Cancer Center Support Grant (P30 CA030199). Le figure 1-3 sono state create utilizzando BioRender.
Materiali
| Name | Company | Catalog Number | Comments |
| 0.25% Trypsin | Corning | 25053CI | 0.1% EDTA in HBSS w/o Calcium, Magnesium and Sodium Bicarbonate |
| 1.5 mL Microcentrifuge tube | Fisherbrand | 05-408-129 | |
| 10-cm Tissue culture dish | Greiner Bio-One | 664160 | CELLSTAR |
| 15 mL Centrifuge tube | Fisherbrand | 07-200-886 | |
| 2 L Beaker | Fisherbrand | 02-591-33 | |
| 24-well Tissue culture plate | Greiner Bio-One | 662160 | CELLSTAR |
| 25 mL Reagent reservoir | Genesee Scientific Corporation | 28-121 | |
| 500 mL Beaker | Fisherbrand | 02-591-30 | |
| 6-cm Tissue culture dish | Greiner Bio-One | 628160 | CELLSTAR |
| 8-Channel aspiration adapter | Integra Biosciences | 155503 | |
| 8-Channel aspiration adapter for standard tips | Integra Biosciences | 159024 | |
| 95% Ethanol | Decon Laboratories Inc | 4355226 | |
| Ammonia-free glass cleaner | Sparkle | FUN20500CT | |
| Black 96-well high-content screening microplate | PerkinElmer | 6055300 | CellCarrier-96 Ultra |
| Cotton-tipped applicator | Fisherbrand | 23-400-101 | |
| Coverslips | Fisherbrand | 12-545-80 | 12 mm diameter |
| Cytation 5 Cell Imaging Multi-Mode Reader | Biotek | CYT5FW | |
| DAPI | Millipore Sigma | 5.08741 | |
| Dextran 70 kDa - FITC | Life Technologies | D1822 | Lysine-fixable |
| Dextran 70 kDa - TMR | Life Technologies | D1819 | |
| DMSO | Millipore Sigma | D1435 | |
| DPBS | Corning | 21031CV | Without Calcium and Magnesium |
| Forceps | Fine Science Tools | 11251-20 | Dumont #5 |
| Formaldehyde, 37% | Ricca Chemical | RSOF0010-250A | ACS Reagent Grade |
| Glycerol | Fisher BioReagents | BP229-1 | |
| Hardening fluorescence mounting media | Agilent Tech | S302380-2 | DAKO |
| Hoechst 33342 | Millipore Sigma | B2261 | |
| Hydrochloric acid (HCl) | Fisher Chemical | A144-212 | Certified ACS Plus, 36.5%–38.0% |
| Lint-free wipes | Kimberly-Clark | 34155 | Kimwipes |
| Miscroscope slides | Fisherbrand | 12-544-1 | Premium plain glass |
| Multichannel pipette | Gilson | FA10013 | 8 channels, 0.5–10 µL |
| Multichannel pipette | Gilson | FA10012 | 12 channels, 20–200 µL |
| Multichannel pipette | Gilson | FA10011 | 8 channels, 20–200 µL |
| Parafilm M | Pechiney | PM996 | |
| Plastic wrap | Kirkland Signature | 208733 | Stretch-Tite |
| Silicone isolators | Grace Bio Labs Inc | 664107 | 13 mm Diameter X 0.8 mm Depth ID, 25 mm X 25 mm |
| Slide adapter | Biotek | 1220548 | |
| Wash bottle | Fisherbrand | FB0340922C |
Riferimenti
- Lin, X. P., Mintern, J. D., Gleeson, P. A. Macropinocytosis in different cell types: similarities and differences. Membranes. 10 (8), 21 (2020).
- Recouvreux, M. V., Commisso, C. Macropinocytosis: a metabolic adaptation to nutrient stress in cancer. Frontiers in Endocrinology. 8, (2017).
- Zhang, Y. J., Commisso, C. Macropinocytosis in cancer: a complex signaling network. Trends in Cancer. 5 (6), 332-334 (2019).
- Commisso, C., et al. Macropinocytosis of protein is an amino acid supply route in Ras-transformed cells. Nature. 497 (7451), 633-637 (2013).
- Kim, S. M., et al. PTEN deficiency and AMPK activation promote nutrient scavenging and anabolism in prostate cancer cells. Cancer Discovery. 8 (7), 866-883 (2018).
- Jayashankar, V., Edinger, A. L. Macropinocytosis confers resistance to therapies targeting cancer anabolism. Nature Communications. 11 (1), (2020).
- Kamphorst, J. J., et al. Human pancreatic cancer tumors are nutrient poor and tumor cells actively scavenge extracellular protein. Cancer Research. 75 (3), 544-553 (2015).
- Olivares, O., et al. Collagen-derived proline promotes pancreatic ductal adenocarcinoma cell survival under nutrient limited conditions. Nature Communications. 8, (2017).
- Seguin, L., et al. Galectin-3, a druggable vulnerability for KRAS-addicted cancers. Cancer Discovery. 7 (12), 1464-1479 (2017).
- Redelman-Sidi, G., et al. The canonical Wnt pathway drives macropinocytosis in cancer. Cancer Research. 78 (16), 4658-4670 (2018).
- Tejeda-Munoz, N., Albrecht, L. V., Bui, M. H., De Robertis, E. M. Wnt canonical pathway activates macropinocytosis and lysosomal degradation of extracellular proteins. Proceedings of the National Academy of Sciences of the United States of America. 116 (21), 10402-10411 (2019).
- Schmees, C., et al. Macropinocytosis of the PDGF beta-receptor promotes fibroblast transformation by H-RasG12V. Molecular Biology of the Cell. 23 (13), 2571-2582 (2012).
- Lee, S. -. W., et al. EGFR-Pak signaling selectively regulates glutamine deprivation-induced macropinocytosis. Developmental Cell. 50 (3), 381-392 (2019).
- Recouvreux, M. V., et al. Glutamine depletion regulates Slug to promote EMT and metastasis in pancreatic cancer. Journal of Experimental Medicine. 217 (9), (2020).
- Zhang, Y., et al. Macropinocytosis in cancer-associated fibroblasts is dependent on CaMKK2/ARHGEF2 signaling and functions to support tumor and stromal cell fitness. Cancer Discovery. 11 (7), 1808-1825 (2021).
- Su, H., et al. Cancer cells escape autophagy inhibition via NRF2-induced macropinocytosis. Cancer Cell. 39 (5), 678-693 (2021).
- Bar-Sagi, D., Feramisco, J. R. Induction of membrane ruffling and fluid-phase pinocytosis in quiescent fibroblasts by ras proteins. Science. 233 (4768), 1061-1068 (1986).
- Mishra, R., et al. Stromal epigenetic alterations drive metabolic and neuroendocrine prostate cancer reprogramming. Journal of Clinical Investigation. 128 (10), 4472-4484 (2018).
- Commisso, C., Flinn, R. J., Bar-Sagi, D. Determining the macropinocytic index of cells through a quantitative image-based assay. Nature Protocols. 9 (1), 182-192 (2014).
- Galenkamp, K. M. O., Alas, B., Commisso, C. Quantitation of macropinocytosis in cancer cells. Methods in Molecular Biology. 1928, 113-123 (2019).
- Wang, J. T. H., Teasdale, R. D., Liebl, D. Macropinosome quantitation assay. MethodsX. 1, 36-41 (2014).
- Lee, S. -. W., Alas, B., Commisso, C. Detection and quantification of macropinosomes in pancreatic tumors. Methods in Molecular Biology. 1882, 171-181 (2019).
- Williams, T., Kay, R. R. High-throughput measurement of dictyostelium discoideum macropinocytosis by flow cytometry. Journal of Visualized Experiments: JoVE. (139), e58434 (2018).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon