É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Aplicação do Grafeno Monocamada em Grades de Microscopia Crio-Eletrônica para Determinação de Estruturas de Alta Resolução
Neste Artigo
Resumo
A aplicação de camadas de suporte a grades de microscopia eletrônica criogênica (crioEM) pode aumentar a densidade de partículas, limitar as interações com a interface ar-água, reduzir o movimento induzido pelo feixe e melhorar a distribuição das orientações das partículas. Este artigo descreve um protocolo robusto para o revestimento de grades de crioEM com uma monocamada de grafeno para melhorar a preparação de amostras criogênicas.
Resumo
Na microscopia eletrônica criogênica (crioEM), macromoléculas purificadas são aplicadas a uma grade contendo uma folha de carbono holey; As moléculas são então borrifadas para remover o excesso de líquido e rapidamente congeladas em uma camada de gelo vítreo de aproximadamente 20-100 nm de espessura, suspensas em furos de folha de aproximadamente 1 μm de largura. A amostra resultante é imageada usando microscopia eletrônica de transmissão criogênica e, após o processamento da imagem usando software adequado, estruturas de resolução quase atômica podem ser determinadas. Apesar da adoção generalizada do cryoEM, a preparação de amostras continua sendo um gargalo severo nos fluxos de trabalho do cryoEM, com os usuários frequentemente encontrando desafios relacionados ao comportamento inadequado das amostras no gelo vítreo em suspensão. Recentemente, métodos têm sido desenvolvidos para modificar grades crioEM com uma única camada contínua de grafeno, que atua como uma superfície de suporte que frequentemente aumenta a densidade de partículas na área fotografada e pode reduzir as interações entre partículas e a interface ar-água. Aqui, nós fornecemos protocolos detalhados para a aplicação de grafeno em grades crioEM e para avaliar rapidamente a hidrofilicidade relativa das grades resultantes. Adicionalmente, descrevemos um método baseado em ME para confirmar a presença de grafeno visualizando seu padrão de difração característico. Finalmente, demonstramos a utilidade desses suportes de grafeno reconstruindo rapidamente um mapa de densidade de resolução de 2,7 Å de um complexo Cas9 usando uma amostra pura em uma concentração relativamente baixa.
Introdução
A microscopia eletrônica criogênica de partícula única (crioEM) evoluiu para um método amplamente utilizado para visualizar macromoléculas biológicas1. Alimentado pelos avanços na detecção direta de elétrons 2,3,4, aquisição de dados5 e algoritmos de processamento de imagens 6,7,8,9,10, o cryoEM é agora capaz de produzir estruturas 3D de resolução quase atômica de um número crescente de macromoléculas11. Além disso, aproveitando a natureza de molécula única da abordagem, os usuários podem determinar múltiplas estruturas a partir de uma única amostra 12,13,14,15, destacando a promessa de usar os dados gerados para entender conjuntos estruturais heterogêneos 16,17. Apesar desse progresso, persistem gargalos na preparação da grade de crio-espécimes.
Para a caracterização estrutural por crioEM, as amostras biológicas devem ser bem dispersas em solução aquosa e, em seguida, congeladas por meio de um processo denominado vitrificação18,19. O objetivo é capturar partículas em uma camada uniformemente fina de gelo vitrificado, suspensa em buracos regularmente espaçados que normalmente são cortados em uma camada de carbono amorfo. Esta folha de carbono amorfa padronizada é suportada por uma grade TEM com uma malha de barras de suporte de cobre ou ouro. Em fluxos de trabalho padrão, as grades são tornadas hidrofílicas usando um tratamento de plasma de descarga luminosa antes da aplicação da amostra. O excesso de líquido é borrado com papel de filtro, permitindo que a solução proteica forme uma fina película líquida através dos orifícios que pode ser facilmente vitrificada durante o congelamento por imersão. Desafios comuns incluem a localização das partículas para a interface ar-água (AWI) e subsequente desnaturação20,21,22 ou a adoção de orientações preferenciais 23,24,25, a aderência das partículas à folha de carbono em vez de migrar para os orifícios e o agrupamento e agregação das partículas dentro dos orifícios26. A espessura não uniforme do gelo é outra preocupação; gelo espesso pode resultar em níveis mais altos de ruído de fundo nas micrografias devido ao aumento do espalhamento de elétrons, enquanto gelo extremamente fino pode excluir partículas maiores27.
Para enfrentar esses desafios, uma variedade de filmes de suporte finos tem sido usada para revestir superfícies de grade, permitindo que as partículas descansem sobre esses suportes e, idealmente, evitem interações com a interface ar-água. Os suportes de grafeno têm se mostrado muito promissores, em parte devido à sua alta resistência mecânica aliada à sua seção transversal de dispersão mínima, o que reduz o sinal de fundo adicionado pela camada de suporte28. Além de sua mínima contribuição para o ruído de fundo, o grafeno também exibe notável condutividade elétrica e térmica29. Grades revestidas com grafeno e óxido de grafeno mostraram maior densidade de partículas, distribuição de partículas mais uniforme30 e localização reduzida para o AWI22. Além disso, o grafeno fornece uma superfície de suporte que pode ser modificada para: 1) sintonizar as propriedades físico-químicas da superfície da grade através da funcionalização 31,32,33; ou 2) agentes ligantes de casal que facilitam a purificação por afinidade de proteínas de interesse 34,35,36.
Neste artigo, modificamos um procedimento existente para o revestimento de grades crioEM com uma única camada uniforme de grafeno30. As modificações visam minimizar o manuseio da grade ao longo do protocolo, com o objetivo de aumentar o rendimento e a reprodutibilidade. Adicionalmente, discutimos nossa abordagem para avaliar a eficácia de vários tratamentos UV/ozônio em tornar as grades hidrofílicas antes do mergulho. Esta etapa na preparação de amostras de crioEM usando grades revestidas com grafeno é crítica, e achamos que nosso método simples para quantificar a hidrofilicidade relativa das grades resultantes é útil. Usando este protocolo, demonstramos a utilidade de empregar grades revestidas com grafeno para a determinação de estruturas, gerando uma reconstrução 3D de alta resolução de S. pyogenes Cas9 cataliticamente inativo em complexo com RNA guia e DNA-alvo.
Protocolo
1. Preparação do grafeno CVD
- Prepare a solução de corrosão de grafeno conforme descrito abaixo.
- Dissolver 4,6 g de persulfato de amônio (APS) em 20 mL de água de grau molecular em um copo de 50 mL para uma solução de 1 M e cobrir com papel alumínio. Permitir que a SAF se dissolva completamente enquanto prossegue para a etapa 1.2.
- Preparar uma seção de grafeno CVD para revestimento de metacrilato de metila (MMA). Corte cuidadosamente uma seção quadrada de grafeno CVD. Transfira o quadrado para uma lamínula (50 mm x 24 mm) dentro de uma placa de Petri limpa e cubra durante o transporte para o revestidor de spin.
NOTA: Um quadrado de 18 mm x 18 mm deve produzir 25-36 grades revestidas de grafeno.
2. Revestimento de grafeno CVD com MMA
- Ajuste as configurações do revestidor de giro para um giro de alta velocidade de 60 s a 2.500 rpm. Coloque cuidadosamente o grafeno CVD sobre o mandril de tamanho adequado.
NOTA: Idealmente, o grafeno CVD estenderá 1-2 mm sobre a borda do mandril selecionado para evitar a aspiração de MMA no sistema de vácuo. - Pressione o botão Take/Absorb para acoplar a bomba de vácuo e afixar o grafeno CVD no mandril. Aplique o MMA no centro do quadrado de grafeno CVD, feche a tampa e pressione o botão Start/Stop imediatamente. Para um quadrado de 18 mm x 18 mm de grafeno CVD, 40 μL de MMA são suficientes.
- Uma vez que a rotação tenha parado, desengate a bomba de vácuo e recupere cuidadosamente o grafeno CVD revestido de MMA com uma pinça. Inverta o grafeno CVD de tal forma que o lado revestido MMA esteja virado para baixo e coloque-o de volta na tampa de vidro.
3. Corrosão a plasma da parte traseira do grafeno
- Transfira o grafeno CVD na lamínula para o descarregador de brilho e descarga de brilho por 30 s a 25 mA usando pinças de ponta plana.
- Devolva o grafeno CVD na lamínula para a placa de Petri e cubra durante o transporte para a área de corrosão de cobre. O revestimento MMA protegerá a camada de grafeno da parte superior da corrosão a plasma.
4. Corte de quadrados de grafeno CVD revestidos de MMA
- Use dois pares de pinças para apoiar o quadrado de grafeno CVD. Anote a orientação do quadrado de grafeno CVD, mantenha o lado do MMA para cima enquanto estiver preso à pinça (CRÍTICO).
- Corte o grafeno CVD em quadrados de aproximadamente 3 mm x 3 mm. Use dois conjuntos de pinças de ação reversa para esta etapa para segurar o quadrado de grafeno CVD do lado direito para cima e fixá-lo na posição, e também para segurar a borda de um quadrado de 3 mm x 3 mm depois de cortá-lo para longe do resto do quadrado.
5. Dissolução do substrato de cobre do grafeno CVD revestido com MMA
- Flutue cuidadosamente cada quadrado de 3 mm x 3 mm na solução APS. Faça contato com a superfície da solução APS antes de liberar cada quadrado. Inclinar o copo de tal forma que cada quadrado possa ser colocado na solução em um ângulo raso; Isso garante que a praça não afunde.
- Cubra o copo com papel alumínio e incube durante a noite a 25 °C.
6. Remoção de filmes de MMA/grafeno da APS
- Use uma lamínula de vidro com dimensões 12 mm x 50 mm e extraia os quadrados MMA/grafeno da APS mergulhando suavemente a lamínula verticalmente na APS e, em seguida, movendo a lamínula lateralmente de tal forma que ela se encaixe em um quadrado MMA/grafeno flutuante.
- Remova cuidadosamente a lamínula e certifique-se de que o quadrado adere completamente ao lado da lamínula após a remoção.
- Transfira os quadrados de MMA/grafeno para um copo limpo de 50 mL preenchido com água de grau molecular por 20 min. Mergulhe suavemente a lamínula com um quadrado de MMA/grafeno preso à água verticalmente e certifique-se de que o quadrado se desloque da lamínula após a interação com a superfície da água. Repita para todos os quadrados MMA/grafeno.
7. Aderência do grafeno às grades
- Usando pinças de ação negativa, mergulhe suavemente uma grade verticalmente na água com o lado de carbono voltado para um quadrado de MMA/grafeno flutuante. Uma vez em contato com o quadrado, remova cuidadosamente a grade e certifique-se de que o quadrado adere completamente ao lado de carbono da grade após a remoção. Minimizar o movimento lateral da grade quando submersa na água para evitar danificar os quadrados da grade (CRÍTICO).
- Coloque grades em uma tampa limpa MMA/grafeno lado para cima e seque ao ar por 1 a 2 min. Transfira grades revestidas de grafeno/MMA para uma placa quente ajustada a 130 °C usando pinças de ponta plana.
- Cubra com a parte superior de uma placa de Petri de vidro e incube por 20 min. Retire do fogo e deixe esfriar até a temperatura ambiente por 1 a 2 min.
CUIDADO: Tenha cuidado, pois a placa de Petri estará extremamente quente.
8. Dissolver MMA com acetona
- Transfira toda a tampa para uma placa de Petri preenchida com 15 mL de acetona. Incubar por 30 min.
- Usando uma pipeta sorológica de vidro limpa, transfira a acetona, na qual as grades foram incubadas, para um recipiente de resíduos. Adicione cuidadosamente 15 mL de acetona fresca à placa de Petri usando uma pipeta sorológica de vidro limpa. Incubar por 30 min.
- Repita estes passos de lavagem para um total de 3 lavagens de acetona.
9. Remoção da acetona residual com isopropanol
- Após 3 lavagens com acetona, remova a acetona e substitua por 15 mL de isopropanol usando uma pipeta sorológica de vidro limpa. Incubar por 20 min.
- Repita 3x para um total de 4 lavagens com isopropanol. Remova cuidadosamente as grades do isopropanol e seque ao ar em uma tampa limpa usando uma pinça.
NOTA: O isopropanol residual pode dificultar a liberação de grades de pinças. Fazer contato entre a grade e a tampa limpa antes de liberar a grade da pinça pode aliviar esse problema. - Evaporar os orgânicos residuais transferindo a lamínula com grades revestidas de grafeno para uma placa de aquecimento regulada para 100 °C por 10 min. Arrefecer até à temperatura ambiente e conservar sob vácuo até à utilização.
10. Tratamento UV/ozônio de grades revestidas com grafeno
- Usando pinças de ação reversa, coloque delicadamente grades em uma área de iluminação limpa de um limpador UV/ozônio com o lado revestido de grafeno voltado para cima.
- Deslize a área de iluminação fechada e ligue a máquina. Ligue o mostrador de tempo para o tempo de tratamento designado para iniciar o tratamento UV/ozônio nas grades.
NOTA: A duração do tratamento é um parâmetro ajustável, com o excesso de tratamento tendo o potencial de danificar a grade. Han et al.30 recomendam 10 min de tratamento UV/ozônio, e também achamos que 10 min de tratamento são suficientes. Consulte a etapa 12 para obter orientação sobre como ajustar esse tempo de tratamento. - Proceder diretamente ao mergulho de uma amostra de crioEM nas grades tratadas seguindo os passos de imersão descritos em Koh et al.37.
NOTA: Hidrocarbonetos atmosféricos podem se acumular na superfície da grade após tratamento UV/ozônio e aumentar a hidrofobicidade da superfície do grafeno38,39. Para evitar isso, mergulhe as grades tratadas com UV/ozônio imediatamente. Pode ser vantajoso tratar redes UV/ozônio em lotes, por exemplo, UV/ozônio tratando seis grades e imediatamente mergulhando essas seis grades, depois UV/ozônio tratando um segundo lote de grades, seguido de mergulhar o segundo lote.
11. Capturando uma imagem de difração
- Certifique-se de que o microscópio esteja bem ajustado com iluminação paralela estabelecida e ajuste o desfoco final para -0,2 μm.
- Insira a tela fluorescente e o feixe pare totalmente. Entre no modo de difração para visualizar claramente os pontos de difração.
- Adquira uma imagem usando uma câmera CCD e avalie-a usando um software de análise de imagem.
NOTA: Os pontos de difração mais facilmente observados e identificáveis gerados por uma monocamada de grafeno são 6 pontos correspondentes a uma frequência espacial de 2,13 Å. Use uma ferramenta de medição para estimar a distância do centro do feixe difratado a um desses pontos de difração em unidades recíprocas. Notadamente 2,13 Å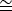 0,47 Å-1.
0,47 Å-1.
12. Avaliação da hidrofilicidade da rede
- Prepare a configuração da geração de imagens. Localize um suporte de telefone, superfície de imagem de mesa, telefone com câmera, tampa de vidro e filme de parafina. As imagens aqui mostradas foram obtidas usando um suporte de telefone e superfície de imagem de mesa que foram impressas em 3D usando os arquivos .stl fornecidos, resultando em medidas de ângulo de contato mais facilmente reprodutíveis e quantificáveis (Figura 1A Suplementar).
- Coloque uma tampa de vidro sobre a superfície plana, depois corte um quadrado de 1 cm por 1 cm de película de parafina e coloque-a sobre a lamínula de vidro. Coloque o telefone no suporte do telefone, orientando o telefone de tal forma que a câmera esteja no plano com a tampa de vidro. Fixe o telefone nesta posição usando elásticos (Figura 1B Suplementar). Tire uma foto de amostra para verificar se a câmera está alinhada com a superfície da imagem.
- Imediatamente após o tratamento UV/ozônio de grades revestidas com grafeno, coloque uma única grade no quadrado de filme de parafina na lamínula de vidro. Verifique se o lado do grafeno está voltado para cima. Adicione uma gota de água de 2 μL no centro da superfície da grade com uma pipeta e tire imediatamente uma foto.
NOTA: Repita esta etapa após: i) intervalos desejados de tratamento UV/ozônio para determinar uma duração suficiente do tratamento; ou ii) intervalos de tempo desejados pós-tratamento UV/ozônio para medir quanto tempo após o tratamento a superfície da grade mantém seu caráter hidrofílico. - Calcule os ângulos de contato das fotos importando-os para o ImageJ43 e usando o plug-in de ângulo de contato.
13. Análise de partículas únicas do conjunto de dados do complexo dCas9
NOTA: Todo o processamento das imagens descritas neste protocolo foi realizado utilizando o cryoSPARC versão 4.2.1.
- Pré-processe filmes usando trabalhos de Correção de movimento de patch e Estimativa de CTF de patch. Execute a coleta de partículas usando o trabalho de seletor de Blobs, usando um blob esférico que varia em diâmetro de 115 Å a 135 Å.
- Extrair partículas usando o trabalho Extract from micrographs, usando coeficiente de correlação normalizado (NCC) e limiares de potência, resultando em aproximadamente 200-300 partículas por micrografia. Observe que os limiares apropriados e a contagem de partículas resultante podem variar, e os usuários devem inspecionar a qualidade da seleção em uma variedade de micrografias para identificar as condições adequadas.
- Realizar reconstruções iniciais multiclasse usando o trabalho de reconstrução Ab-initio, exigindo três classes. Duas das três classes provavelmente conterão partículas não-Cas9, incluindo contaminantes superficiais. Selecione a classe semelhante a dCas9 para processamento posterior.
NOTA: Rodadas adicionais de reconstrução Ab-initio multiclasse ou refinamento heterogêneo podem ser aplicadas para refinar ainda mais a pilha de partículas. - Execute o refinamento 3D usando o trabalho Refinamento não uniforme selecionando os parâmetros padrão. Estimar a resolução da reconstrução usando os trabalhos de Validação (FSC) e ThreeDFSC, empregando os mapas e a máscara do refinamento 3D final.
Resultados
A fabricação bem-sucedida de grades crioEM revestidas com grafeno usando o equipamento (Figura 1) e o protocolo (Figura 2) aqui descritos resultará em uma monocamada de grafeno cobrindo os orifícios da folha, o que pode ser confirmado pelo seu padrão de difração característico. Para promover a adsorção de proteínas na superfície do grafeno, o tratamento UV/ozônio pode ser usado para tornar a superfície hidrofílica através da instalação de grupo...
Discussão
A preparação de amostras CryoEM envolve uma série de desafios técnicos, com a maioria dos fluxos de trabalho exigindo que os pesquisadores manipulem manualmente grades frágeis com extremo cuidado para evitar danificá-las. Além disso, a possibilidade de qualquer amostra ser vitrificação é imprevisível; As partículas frequentemente interagem com a interface ar-água ou com a folha de suporte sólida que sobrepõe as grades, o que pode levar as partículas a adotarem orientações preferenciais ou a não entrare...
Divulgações
Os autores não têm conflitos a revelar.
Agradecimentos
Os espécimes foram preparados e fotografados no CryoEM Facility em MIT.nano em microscópios adquiridos graças à Fundação Arnold e Mabel Beckman. Dispositivos de imagem de ângulo de contato foram impressos no MIT Metropolis Maker Space. Agradecemos aos laboratórios de Nieng Yan e Yimo Han, e à equipe do MIT.nano por seu apoio durante toda a adoção deste método. Em particular, estendemos nossos agradecimentos aos Drs. Guanhui Gao e Sarah Sterling por suas discussões perspicazes e feedback. Este trabalho foi apoiado pelos subsídios do NIH R01-GM144542, 5T32-GM007287 e 2046778 de bolsas NSF-CAREER. A pesquisa no laboratório Davis é apoiada pela Fundação Alfred P. Sloan, pelo James H. Ferry Fund, pelo MIT J-Clinic e pela Família Whitehead.
Materiais
| Name | Company | Catalog Number | Comments |
| 250 mL beaker (3x) | Fisher | 02-555-25B | |
| 50 mL beaker (2x) | Corning | 1000-50 | |
| Acetone | Fisher | A949-4 | |
| Aluminum foil | Fisher | 15-078-292 | |
| Ammonium persulfate | Fisher | (I17874 | |
| Coverslips 50 mm x 24 mm | Mattek | PCS-1.5-5024 | |
| CVD graphene | Graphene Supermarket | CVD-Cu-2x2 | |
| easiGlow discharger | Ted-Pella | 91000S | |
| Ethanol | Millipore-Sigma | 1.11727 | |
| Flat-tip tweezers | Fisher | 50-239-60 | |
| Glass cutter | Grainger | 21UE26 | |
| Glass petri plate and cover | VWR | 75845-544 | |
| Glass serological pipette | Fisher | 13-676-34D | |
| Grid Storage Case | EMS | 71146-02 | |
| Hot plate | Fisher | 07-770-108 | |
| Isopropanol | Sigma | W292907 | |
| Kimwipe | Fisher | 06-666 | |
| Lab scissors | Fisher | 13-806-2 | |
| Methyl-Methacrylate EL-6 | Kayaku | MMA M310006 0500L1GL | |
| Molecular grade water | Corning | 46-000-CM | |
| Negative action tweezers (2x) | Fisher | 50-242-78 | |
| P20 pipette | Rainin | 17014392 | |
| P200 pipette | Rainin | 17008652 | |
| Parafilm | Fisher | 13-374-12 | |
| Pipette tips | Rainin | 30389291 | |
| Quantifoil grids with holey carbon | EMS | Q2100CR1 | |
| Spin coater | SetCas | KW-4A | with chuck SCA-19-23 |
| Straightedge | ULINE | H-6560 | |
| Thermometer | Grainger | 3LRD1 | |
| UV/Ozone cleaner | BioForce | SKU: PC440 | |
| Vacuum desiccator | Thomas Scientific | 1159X11 | |
| Whatman paper | VWR | 28297-216 |
Referências
- Chua, E. Y. D., et al. cheaper: Recent advances in cryo-electron microscopy. Annu Rev Biochem. 91, 1-32 (2022).
- Bai, X. C., Fernandez, I. S., McMullan, G., Scheres, S. H. Ribosome structures to near-atomic resolution from thirty thousand cryo-EM particles. Elife. 2, 00461 (2013).
- Li, X., et al. Electron counting and beam-induced motion correction enable near-atomic-resolution single-particle cryo-EM. Nat Methods. 10 (6), 584-590 (2013).
- Campbell, M. G., et al. Movies of ice-embedded particles enhance resolution in electron cryo-microscopy. Structure. 20 (11), 1823-1828 (2012).
- Cheng, A., et al. Leginon: New features and applications. Protein Sci. 30 (1), 136-150 (2021).
- Scheres, S. H. RELION: implementation of a Bayesian approach to cryo-EM structure determination. J Struct Biol. 180 (3), 519-530 (2012).
- Punjani, A., Rubinstein, J. L., Fleet, D. J., Brubaker, M. A. cryoSPARC: algorithms for rapid unsupervised cryo-EM structure determination. Nat Methods. 14 (3), 290-296 (2017).
- Tegunov, D., Cramer, P. Real-time cryo-electron microscopy data preprocessing with Warp. Nat Methods. 16 (11), 1146-1152 (2019).
- Grant, T., Rohou, A., Grigorieff, N. cisTEM, user-friendly software for single-particle image processing. Elife. 7, 35383 (2018).
- Bell, J. M., Chen, M., Baldwin, P. R., Ludtke, S. J. High resolution single particle refinement in EMAN2.1. Methods. 100, 25-34 (2016).
- Cheng, Y. Single-particle cryo-EM-How did it get here and where will it go. Science. 361 (6405), 876-880 (2018).
- Kinman, L. F., Powell, B., Zhong, E., Berger, B., Davis, J. H. Uncovering structural ensembles from single particle cryo-EM data using cryoDRGN. Nat Protoc. 18 (2), 319-339 (2022).
- Zhong, E. D., Bepler, T., Berger, B., Davis, J. H. CryoDRGN: reconstruction of heterogeneous cryo-EM structures using neural networks. Nat Methods. 18 (2), 176-185 (2021).
- Chen, M., Ludtke, S. J. Deep learning-based mixed-dimensional Gaussian mixture model for characterizing variability in cryo-EM. Nat Methods. 18 (8), 930-936 (2021).
- Punjani, A., Fleet, D. J. 3D variability analysis: Resolving continuous flexibility and discrete heterogeneity from single particle cryo-EM. J Struct Biol. 213 (2), 107702 (2021).
- Dashti, A., et al. Retrieving functional pathways of biomolecules from single-particle snapshots. Nat Commun. 11 (1), 4734 (2020).
- Sun, J., Kinman, L. F., Jahagirdar, D., Ortega, J., Davis, J. H. KsgA facilitates ribosomal small subunit maturation by proofreading a key structural lesion. Nat Struct Mol Biol. , (2023).
- Dubochet, J., Chang, J. J., Freeman, R., Lepault, J., McDowall, A. W. Frozen aqueous suspensions. Ultramicroscopy. 10 (1-2), 55-61 (1982).
- Dubochet, J. Cryo-EM--the first thirty years. J Microsc. 245 (3), 221-224 (2012).
- Glaeser, R. M., et al. Factors that influence the formation and stability of thin, cryo-EM specimens. Biophys J. 110 (4), 749-755 (2016).
- Glaeser, R. M. Proteins, interfaces, and cryo-Em grids. Curr Opin Colloid Interface Sci. 34, 1-8 (2018).
- D'Imprima, E., et al. Protein denaturation at the air-water interface and how to prevent it. Elife. 8, 42747 (2019).
- Tan, Y. Z., et al. Addressing preferred specimen orientation in single-particle cryo-EM through tilting. Nat Methods. 14 (8), 793-796 (2017).
- Chen, J., Noble, A. J., Kang, J. Y., Darst, S. A. Eliminating effects of particle adsorption to the air/water interface in single-particle cryo-electron microscopy: Bacterial RNA polymerase and CHAPSO. J Struct Biol X. 1, 100005 (2019).
- Noble, A. J., et al. Reducing effects of particle adsorption to the air-water interface in cryo-EM. Nat Methods. 15 (10), 793-795 (2018).
- Drulyte, I., et al. Approaches to altering particle distributions in cryo-electron microscopy sample preparation. Acta Crystallogr D Struct Biol. 74, 560-571 (2018).
- Neselu, K., et al. Measuring the effects of ice thickness on resolution in single particle cryo-EM. J Struct Biol X. 7, 100085 (2023).
- Pantelic, R. S., et al. Graphene: Substrate preparation and introduction. J Struct Biol. 174 (1), 234-238 (2011).
- Geim, A. K., Novoselov, K. S. The rise of graphene. Nat Mater. 6 (3), 183-191 (2007).
- Han, Y., et al. High-yield monolayer graphene grids for near-atomic resolution cryoelectron microscopy. Proc Natl Acad Sci U S A. 117 (2), 1009-1014 (2020).
- Fujita, J., et al. Epoxidized graphene grid for highly efficient high-resolution cryoEM structural analysis. Sci Rep. 13 (1), 2279 (2023).
- Lu, Y., et al. Functionalized graphene grids with various charges for single-particle cryo-EM. Nat Commun. 13 (1), 6718 (2022).
- Naydenova, K., Peet, M. J., Russo, C. J. Multifunctional graphene supports for electron cryomicroscopy. Proc Natl Acad Sci U S A. 116 (24), 11718-11724 (2019).
- Liu, N., et al. Bioactive functionalized monolayer graphene for high-resolution cryo-electron microscopy. J Am Chem Soc. 141 (9), 4016-4025 (2019).
- Benjamin, C. J., et al. Selective capture of histidine-tagged proteins from cell lysates using TEM grids modified with NTA-graphene oxide. Sci Rep. 6, 32500 (2016).
- Wang, F., et al. General and robust covalently linked graphene oxide affinity grids for high-resolution cryo-EM. Proc Natl Acad Sci U S A. 117 (39), 24269-24273 (2020).
- Koh, A., et al. Routine Collection of High-Resolution cryo-EM Datasets Using 200 KV Transmission Electron Microscope. J Vis Exp. (181), (2022).
- Schweizer, P., et al. Mechanical cleaning of graphene using in situ electron microscopy. Nat Commun. 11 (1), 1743 (2020).
- Li, Z., et al. Effect of airborne contaminants on the wettability of supported graphene and graphite. Nat Mater. 12 (10), 925-931 (2013).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Zhu, X., et al. Cryo-EM structures reveal coordinated domain motions that govern DNA cleavage by Cas9. Nat Struct Mol Biol. 26 (8), 679-685 (2019).
- Croll, T. I. ISOLDE: a physically realistic environment for model building into low-resolution electron-density maps. Acta Crystallogr D Struct Biol. 74, 519-530 (2018).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ: 25 years of image analysis. Nat Methods. 9 (7), 671-675 (2012).
- Prydatko, A. V., Belyaeva, L. A., Jiang, L., Lima, L. M. C., Schneider, G. F. Contact angle measurement of free-standing square-millimeter single-layer graphene. Nat Commun. 9 (1), 4185 (2018).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados