È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Determinazione del relativo cellulari di superficie e di espressione totale di canali ionici ricombinante in citometria a flusso
In questo articolo
Riepilogo
aritmie cardiache ereditarie sono spesso causate da mutazioni che alterano la consegna di superficie di uno o più canali ionici. Qui, ci adattiamo citometria a flusso saggi di fornire una quantificazione della espressione proteica totale e superficie cellulare relativa dei canali ionici ricombinanti espressi in TSA-201 cellule.
Abstract
Inherited or de novo mutations in cation-selective channels may lead to sudden cardiac death. Alteration in the plasma membrane trafficking of these multi-spanning transmembrane proteins, with or without change in channel gating, is often postulated to contribute significantly in this process. It has thus become critical to develop a method to quantify the change of the relative cell surface expression of cardiac ion channels on a large scale. Herein, a detailed protocol is provided to determine the relative total and cell surface expression of cardiac L-type calcium channels CaV1.2 and membrane-associated subunits in tsA-201 cells using two-color fluorescent cytometry assays. Compared with other microscopy-based or immunoblotting-based qualitative methods, flow cytometry experiments are fast, reproducible, and large-volume assays that deliver quantifiable end-points on large samples of live cells (ranging from 104 to 106 cells) with similar cellular characteristics in a single flow. Constructs were designed to constitutively express mCherry at the intracellular C-terminus (thus allowing a rapid assessment of the total protein expression) and express an extracellular-facing hemagglutinin (HA) epitope to estimate the cell surface expression of membrane proteins using an anti-HA fluorescence conjugated antibody. To avoid false negative, experiments were also conducted in permeabilized cells to confirm the accessibility and proper expression of the HA epitope. The detailed procedure provides: (1) design of tagged DNA (deoxyribonucleic acid) constructs, (2) lipid-mediated transfection of constructs in tsA-201 cells, (3) culture, harvest, and staining of non-permeabilized and permeabilized cells, and (4) acquisition and analysis of fluorescent signals. Additionally, the basic principles of flow cytometry are explained and the experimental design, including the choice of fluorophores, titration of the HA antibody and control experiments, is thoroughly discussed. This specific approach offers objective relative quantification of the total and cell surface expression of ion channels that can be extended to study ion pumps and plasma membrane transporters.
Introduzione
Questo documento fornisce un test affidabile per segnalare il relativo espressione superficie cellulare di proteine di membrana, quali canali ionici espressi in cellule ricombinanti utilizzando la tecnologia di citometria a flusso esistente. I canali ionici sono proteine di membrana che formano pori che sono responsabili del controllo segnali elettrici dal gating il flusso di ioni attraverso la membrana cellulare. Essi sono classificati dal meccanismo di attivazione, la natura e la selettività di specie ioniche in transito attraverso il poro dove sono localizzate. A livello cellulare e dei tessuti, i flussi ionici macroscopici attraverso i canali ionici sono il prodotto di proprietà 1 biofisica (gating e permeazione), biochimici (fosforilazione), e biogenesi (sintesi, glicosilazione, il traffico, e la degradazione). Ciascuno di questi processi è unico per ogni tipo di canali ionici ed è ottimizzato per svolgere il ruolo fisiologico del canale ionico. Di conseguenza, alterazioni in nessuno di questi processi di precisione La attraverso unereditaria o una modificazione genetica, spesso definito come "canalopatia", può essere dannoso per l'omeostasi cellulare. È importante sottolineare che fornire l'importo "diritto" di canali ionici sulla superficie cellulare è fondamentale per l'omeostasi cellulare. Anche piccoli incrementi (guadagno-di-funzione) e lievi diminuzioni (perdita-di-funzione) in attività di canale ionico hanno il potenziale di causare una grave patologia nel corso della vita. Difetti nella consegna superficie cellulare dei canali ionici maturo è un importante determinante in numerosi canalopatie, come la fibrosi cistica (CFTR canale ionico) 2 e aritmie cardiache della forma lunga sindrome del QT (canali del potassio cardiaci) 3.
Canalopatie sono associati con cardiaca improvvisa morte 4. L'attuale diffusione a livello mondiale di tutti i canalopatie cardiache è pensato per essere di almeno 1: 2,000-1: 3.000 per individuo 5 e sono responsabili di circa la metà di improvvisa aritmica ca morte cardiacases 6. Disfunzione cardiaca voltaggio-dipendenti sodio, potassio, calcio-e canali ionici selettivi sono noti per svolgere un ruolo chiave in questo processo. Il 1.2 canali del calcio voltaggio-dipendenti L-tipo Ca V è tenuta ad avviare sincronizzato cuore la contrazione muscolare. Il cardiaca L-tipo Ca V 1.2 canali è un complesso proteico multi-subunità composto principale pore-forming Ca V α1 subunità e Ca V ß e Ca V α2δ1 subunità ausiliari 7-12. Si noti che la serie completa di subunità ausiliari è necessaria per produrre funzionali Ca V 1.2 canali a livello della membrana plasmatica e le interazioni dinamiche tra queste subunità sono essenziali per sostenere la funzione elettrica normale del cuore 13. Ca V ß promuove la superficie espressione cellulare di Ca V 1.2 canali attraverso un non-covalente nanomolari interazione idrofobica 14. Co-espressione del Ca V α2δ1 subunità wi° Ca V ß-bound V α1 Ca stimola l'espressione corrente di picco (da 5 a 10 volte) e promuove attivazione del canale a tensioni più negativi. Guadagno-di-funzione mutazioni della subunità pore-forming Ca V 1.2 sono stati associati con una forma di aritmia ventricolare chiamato la sindrome del QT lungo 15, mentre una serie di mutazioni puntiformi nelle tre principali subunità che formano il L-tipo Ca V 1.2 canali sono stati identificati nei soggetti affetti da aritmie del breve QT modulo sindrome di 16,17. I canali ionici sono proteine di membrana che possono essere studiati da un punto di vista biochimico (chimica delle proteine) o utilizzando gli strumenti di elettrofisiologia (macchine generatori di corrente) e spesso utilizzano questi approcci complementari. Elettrofisiologia, in particolare whole-cell patch-bloccaggio, è un approccio adatto per chiarire la funzione dei canali ionici 15, ma non è in grado di risolvere le modifiche nel traffico di proteine da cambiamenti nella loro biofisicaproprietà. Proteina chimica ha, tuttavia, spesso uso limitato a causa della relativamente bassa espressione di grandi proteine di membrana relativi alle proteine solubili piccoli. Robusti metodi high-throughput utilizzando la fluorescenza di lettura devono essere sviluppate al fine di affrontare specificamente i difetti di biogenesi di proteine che causano cambiamenti nell'espressione superficie cellulare dei canali ionici.
Citometria a flusso è una tecnologia impiegata in biofisica conteggio delle cellule, l'ordinamento, la rilevazione biomarker, e ingegneria proteica 18. Quando una soluzione campione di cellule vive o particelle viene iniettato in un citometro di flusso, le cellule sono ordinate in un unico flusso che può essere controllato di sistema di rilevamento della macchina (figura 1). Il primo citofluorimetro atto prodotto nel 1956 19 rilevato un solo parametro, ma moderni citofluorimetri avere più laser e rilevatori di fluorescenza che consentono il rilevamento di più di 30 parametri fluorescenti 20,21.Filtri e specchi (ottica di emissione) dirigono la diffusione luminosa o luce fluorescente di cellule ad una rete elettronica (fotodiodo e rilevatori) che convertono la luce in proporzione alla sua intensità. I dati digitali vengono analizzati utilizzando software specializzati e l'uscita principale viene visualizzato come un diagramma a punti 21.
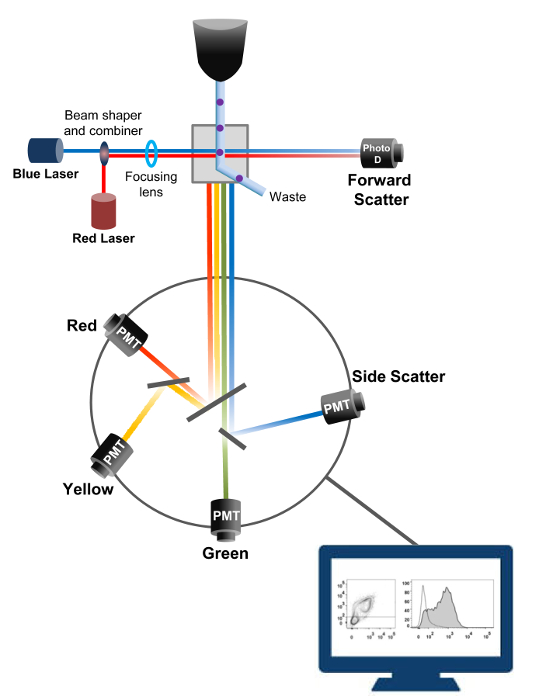
Figura 1:. Principi biofisici di citometria a flusso ordinamento celle singole sono spinti attraverso un ugello ad alta pressione all'interno di un flusso di liquido guaina che li muove attraverso uno o più punti di interrogatorio laser. Il fascio di luce viene deviato da cellule di passaggio e la luce raccolta in direzione avanti (Forward Scatter, FCS) viene inviata ad un fotodiodo che converte la luce in un segnale proporzionale alla dimensione della cella. La luce viene raccolta anche ad un angolo di 90 ° rispetto alla traiettoria laser e inviato ai rivelatori (chiamati anche fotomoltiplicatori (PMT)).Questa luce viene instradato attraverso specchi dicroici che permettono la rilevazione del segnale side scatter (SSC), che riflette la granularità all'interno delle cellule, e le emissioni fluorescenti se fluorocromi eccitati sono presenti nella cellula. Tre rivelatori (verde, giallo e rosso) sono rappresentati con diversi filtri passa-banda di lunghezza d'onda, permettendo la rilevazione simultanea di diversi fluorocromi. I diversi segnali vengono digitalizzati da un computer esterno e convertiti in dati che verranno analizzati per quantificare le caratteristiche delle cellule. Si prega di cliccare qui per vedere una versione più grande di questa figura.
La capacità high-throughput di citometri a flusso è stata sfruttata per quantificare l'espressione di membrana relativo di ricombinante wild-type e la tratta con deficit di voltaggio-dipendenti di tipo L Ca V 1.2 canali e subunità associati in cellule vive. cDNA costruisce coding per le proteine erano doppiamente etichettato avere simultaneamente un epitopo non fluorescente extracellulare che può essere rilevata da un anticorpo coniugato fluorescente impermeabile e un fluoroforo intracellulare che è costitutivamente fluorescente. Sia l'epitopo extracellulare, inserito in un loop extracellulare della proteina, e il fluoroforo intracellulare, inserito dopo il C-terminale, sono tradotti con la proteina. In questa serie di esperimenti, la proteina Ca V α2δ1 è stato progettato per esprimere un emoagglutinina extracellulare (HA) epitopo (YPYDVPDYA) rilevata da un impermeabile FITC (fluoresceina isotiocianato) coniugata anti-HA e mCherry come fluoroforo intracellulare intrinseca. Per determinare il relativo livello di espressione della superficie delle cellule del V α2δ1 mCherry-Ca proteina HA-tag, le cellule ricombinanti che esprimono la proteina di fusione sono state raccolte dopo la transfezione, e colorati con la FITC-coniugato topo monoclonale anti-HA tag epitopo gli anticorpiy (Figura 2). FITC è un composto fluorescente organico che è considerevolmente inferiore rispetto reporter enzimi e quindi improbabile interferire con la funzione biologica. mCherry- Ca V α2δ1-HA sovraespresso in Tsa-201cells, produce un significativo aumento di 3 log in fluorescenza FITC e mCherry fluorescenza appezzamenti bidimensionali 22. Dato che l'epitopo HA si trova nella porzione extracellulare della proteina, l'intensità di fluorescenza per FITC ottenuta in presenza di cellule intatte riflettono l'indice relativa della superficie cellulare espressione di proteine HA-tag. L'accessibilità del epitopo HA nei costrutti è sistematicamente convalidata misurando il segnale FITC dopo permeabilizzazione cellulare. Questa misura serve anche a corroborare l'espressione della proteina totale normalizzato dal momento che le intensità di fluorescenza relative per FITC stimati in cellule permeabilizzate sono qualitativamente paragonabili ai valori di fluorescenza relativi FOR mCherry misurato in condizioni permeabilizzate e non permeabilizzate 22,23. È importante notare che lo spettro di fluorescenza intrinseca è spostato verso valori più elevati dopo permeabilizzazione ma che l'unico valore di essere rilevato è la variazione di intensità di fluorescenza rispetto al costrutto controllo. variazioni relative l'intensità di fluorescenza per i costrutti di prova sono stimate utilizzando la Intensity ΔMean fluorescenza (ΔMFI) i valori per ciascun fluoroforo (mCherry o FITC). Gli esperimenti sono progettati per misurare l'intensità di fluorescenza della prova costrutto relativa all'intensità di fluorescenza del costrutto di controllo espressa nelle stesse condizioni sperimentali per limitare le variazioni nella fluorescenza intrinseca dell'anticorpo fluoroforo-coniugato. Due proteine di membrana sono stati studiati con successo utilizzando questo test: la subunità pore-forming del canale del calcio voltaggio-dipendenti di tipo L Ca V 1.2 14,22 e in una diversa serie diesperimenti, il extracellulare ausiliario Ca V α2δ1 subunità 22,23. Il seguente protocollo è stato utilizzato per determinare l'espressione superficie cellulare del Ca V α2δ1 subunità del cardiaca L-tipo Ca V 1.2 canale in condizioni di controllo e dopo mutazioni colpendo la modifica post-traslazionale del canale ionico. In condizioni sperimentali standardizzate, la fluorescenza superficie cellulare di FITC aumenta quasi linearmente con l'espressione di cDNA codificanti per le proteine α2δ1-HA mCherry-Ca V (Figura 5 dal riferimento 22).
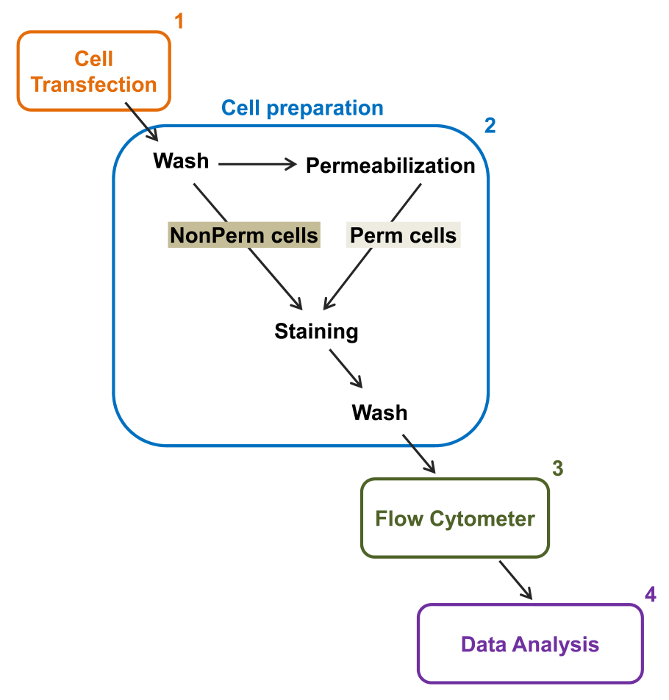
Figura 2:. Rappresentazione schematica di etichettatura totale e membrana in citometria a flusso protocollo sperimentale Lo schema illustra alcuni dei principali passi necessari per quantificare l'espressione relativa totale e superficie cellulare dei canali ionici ricombinanti di flOW citometria. Le cellule sono trasfettate con la costruzione doppia marcatura mCherry-Ca V α2δ1-HA in TSA-201 celle (1) e colorate prima o dopo permeabilizzazione (2). Dati multiparametrica vengono acquisiti in un citofluorimetro (3) per l'analisi multivariata (4). Si prega di cliccare qui per vedere una versione più grande di questa figura.
Protocollo
1. costrutti Doppiamente Tagged DNA
- Inserire l'epitopo HA (YPYDVPDYA) nel linker extracellulare di Ca V α2δ1 tra D676 e R677 per mutagenesi sito-specifica (figura 3B) 20. Utilizzare gccggattatgcgGGAAAACTCCAAACAACC Forward primer e Reverse acatcatacggataTCAATAAATTCATTGAAATTTAAAAGAAATTC primer.
- Subclone la sequenza cDNA della targhetta V α2δ1 HA Ca nel vettore di espressione di mammifero pmCherry-N1 progettato per esprimere la proteina fusa all'N-terminale di mCherry tra i siti SacI e Sali (Figura 3B) 20.
NOTA: La funzione di canale appropriato deve essere testato con il costrutto di controllo utilizzando metodi elettrofisiologici standard di 24.
2. trasfezione transiente liposomi-mediata (30 min, tutti i passaggi vengono eseguite sotto flusso laminare Hood)
- Giorno 1: Piastra mezzo milione di TSA-201 cellule (o HEKT) a35 piatti della cultura mm con 2 ml di Dulbecco di mezzo essenziale minimo di alta glucosio (DMEM-HG) supplementato con 10% siero fetale bovino (FBS) e l'1% di media (PS) cultura di penicillina-streptomicina. Contare le cellule utilizzando un emocitometro standard. Valutare vitalità cellulare da una frazione del campione cella utilizzando Trypan Blue. Piatto abbastanza cellule per raggiungere il 90% di confluenza al momento della transfezione.
- 2 ° giorno: Modifica terreno di coltura con 2 ml di (37 ° C) mezzo fresco pre-riscaldato cultura senza PS.
- Per ogni campione trasfezione, preparare due provette da 1,5 ml. Nel tubo 1, diluire 4 mg di DNA in 250 ml di media siero ridotto. Nel tubo 2, miscelare 10 ml di reagente trasfezione liposomi-mediata con 250 microlitri terreno di coltura siero ridotto. Mescolare delicatamente il reagente di trasfezione prima dell'uso.
- Incubare per 5 minuti a temperatura ambiente.
- Unire il contenuto della provetta 1 e il tubo 2, mescolare delicatamente e incubare almeno 20 minuti a temperatura ambiente.
- Aggiungere i liposomi / DNA complessi alle cellule in coltura e delicatamente roccia piatto cultura per mescolare.
- Incubare a 37 ° C sotto 5% CO 2 ambiente per 24 ore.
3. colorazione delle cellule di citometria a flusso (3 ore)
- Preparazione campioni di cellule
- Giorno 3: Rimuovere media dal piatto cultura accuratamente e lavare le cellule con 400 ml di pre-riscaldato (37 ° C) 0,05% tripsina-EDTA 1x (acido etilendiamminotetraacetico).
- Aggiungere 400 ml di tripsina-EDTA e incubare il piatto a 37 ° C sotto 5% CO 2 ambiente per 5 min per permettere alle cellule di staccarsi dal piatto.
- Fermare la digestione enzimatica con l'aggiunta di 1 ml di terreno di coltura freddo senza PS e lavare tutte le cellule dalla superficie pipettando delicatamente 4-5 volte. Evitare un eccesso di digestione e over-pipettaggio per ridurre la morte cellulare.
- Raccogliere le cellule in provette da 1,5 ml e mettere immediatamente in ghiaccio. Utilizzare soluzioni fredde di ghiaccio e mantenere le cellule a 4 ° C per evitare l'internalizzazionedi superficie antigeni. Ridurre l'illuminazione per limitare photobleaching del segnale fluorescente.
- Tubi centrifugare a 400 xg per 5 minuti a 4 ° C. Con attenzione aspirare e scartare il surnatante.
- Risospendere il pellet in 1 ml di 1x PBS (PBS) per preparare una sospensione singola cella.
- Brevemente vortice i tubi molto delicatamente e ripetere i punti 3.1.5 e 3.1.6 per rimuovere completamente terreno di coltura.
- Risospendere il pellet in 600 ml di PBS 1x e regolare la concentrazione cellulare a un minimo di 3 x 10 6 cellule / ml.
- Dividere le celle in due nuove provette da 1,5 ml per la colorazione extracellulare e intracellulare. Includere controlli appropriati per discriminare colorazione specifica colorazione aspecifica.
NOTA: L'anticorpo controllo isotipico aiuta a valutare il livello di colorazione di fondo e idealmente dovrebbe corrispondere ogni anticorpo primario specie ospite, isotipo e fluoroforo. Utilizzare il controllo isotipo e anticorpo coniugato allo stessoconcentrazione proteica.

Tabella 1: Flow-citometria a campioni di controllo esperimento per non permeabilizzate e permeabilizzate cellule Ogni esperimento deve includere i seguenti controlli negativi:. (1) le cellule Nontransfected (senza anticorpi, con l'isotipo o con l'anticorpo coniugato). (2) Le cellule trasfettate con la proteina di interesse subclonato in un plasmide senza costitutiva fluorocromo intracellulare fluorescente (pCMV- Ca V α2δ1-HA) o con la costruzione doppiamente tag (pmCherry-Ca V α2δ1 e incubate senza anticorpi, con isotipo o con l'anticorpo coniugato). controlli del colore singoli vengono utilizzati per la compensazione delle emissioni di sovrapposizione fluorocromo. Gli stessi controlli vengono eseguiti per non permeabilizzate e condizioni permeabilizzate in ogni serie di esperimenti.
- La colorazione delle cellule della superficie di Intactcellule vive
- Aliquota 1 x 10 6 cellule / ml 100 in provette da 1,5 ml.
- Aggiungere l'anticorpo monoclonale anti-HA FITC-coniugato a 5 ug / ml e, vortex prima incubando le cellule su una piattaforma bilanciere (200 rpm) al buio a 4 ° C per 45 min.
NOTA: La concentrazione ottimale di anticorpi è stata determinata in esperimenti preliminari titolazione (Figura 4). - Rimuovere le cellule dal buio e aggiungere 900 ml di PBS 1x / tubo. Centrifugare a 400 xg per 5 minuti a 4 ° C.
- Aspirare il surnatante e risospendere il pellet in 1 ml di PBS 1x, vortex e centrifugare a 400 xg per 5 minuti a 4 ° C.
- Ripetere il lavaggio (fase 3.2.4) due volte per rimuovere qualsiasi anticorpo non legato. Se viene utilizzato un anticorpo primario non coniugato, incubare con l'anticorpo secondario appropriato.
- Dopo l'ultimo lavaggio, risospendere le cellule in 500 ml di PBS 1x e trasferire la sospensione cellulare singolo in 5 ml citometria a flusso tubi. Tenere il cellulares al buio a 4 ° C fino ad ottenere il campione.
- Eseguire i campioni su un citometro di flusso. Per ottenere risultati ottimali, analizzare le cellule sul citometro appena possibile e non oltre 24 ore dopo.
- Colorazione intracellulare: fissazione, permeabilizzazione, e la colorazione
- Aliquotare 1 x 10 6 cellule / 100 ml in provette da 1,5 ml e centrifugare a 400 xg per 5 minuti a 4 ° C.
- Eliminare le cellule surnatante e risospendere in 100 ml di soluzione di fissaggio-permeabilizzazione direttamente dal magazzino.
- Incubare al buio a 4 ° C per 20 min.
- Aggiungere 100 ml di tampone 1x permeabilizzazione-lavaggio preparati al momento (diluire 10x tampone permeabilizzazione-lavaggio in acqua distillata H 2 O). cellule Vortex e sedimenti utilizzando una centrifuga tavolo a 400 xg per 5 minuti a 4 ° C.
- Aspirare e scartare il surnatante.
- Ripetere i punti 3.3.4 e 3.3.5.
- Aggiungere FITC-coniugato anticorpo monoclonale anti-HA a 5ug / ml in 100 ml di 1x tampone permeabilizzazione lavaggio e, vortex prima incubando le cellule al buio a 4 ° C per 30 min.
NOTA: La colorazione intracellulare viene eseguita seguendo la stessa procedura come quella utilizzata per la colorazione della superficie cellulare. Saponina-mediata permeabilizzazione cella è tuttavia un processo rapido reversibile, quindi è importante sostituire PBS 1x con 1x tampone Perm / Wash per mantenere le cellule in presenza costante di saponina durante la colorazione intracellulare. - Rimuovere le cellule dal buio e aggiungere 100 microlitri di buffer permeabilizzazione-lavaggio. Centrifugare a 400 xg per 5 minuti a 4 ° C.
- Aspirare con attenzione il surnatante e risospendere il pellet in 100 microlitri di buffer permeabilizzazione-lavaggio, vortex e centrifugare a 400 xg per 5 minuti a 4 ° C.
- Ripetere il lavaggio (fase 3.3.9) ancora una volta per rimuovere l'anticorpo non legato.
- Dopo l'ultimo lavaggio, risospendere le cellule in 500 ml di PBS 1x e trasferire il peccatosospensione cellulare GLE a 5 ml flusso citometria a tubi. Mantenere le cellule al buio a 4 ° C fino iniettando il campione nella citofluorimetro.
- Eseguire i campioni su un citometro di flusso. Eseguire i campioni fissi sul citometro nel più breve tempo possibile, e comunque non oltre 1 settimana dopo la colorazione. Eseguire le cellule non permeabilizzate e permeabilizzate nello stesso giorno.
4. Citometria a Flusso
- Citofluorimetro cellulare Sorter giornaliera Setup
- Accendere il software di citometria a flusso. Prima di sperimentare, calibrare e impostare il citofluorimetro cell sorter per garantire prestazioni ottimali dello strumento (ad esempio il laser e ottica sta eseguendo le specifiche, il laser e il flusso delle cellule sono allineati correttamente) utilizzando perline di impostazione dello strumento.
- Utilizzare l'ugello 100 micron con 20 psi pressione guaina.
NOTA: L'ugello non deve essere modificato su una panchina citometro a flusso. - Impostare la portata del citometro secondo il manufacturspecificazione ER. Estremamente alte portate diminuirà sensibilità nella rilevazione di variazioni nella fluorescenza.
- Selezionare blu (488 nm per eccitare fluoresceina Isothiocayanate o FITC) e di colore giallo-verde (561 nm per eccitare mCherry) laser. Raccogliere FITC e mCherry livelli di fluorescenza con un nm 530/30 e 610/20 con un filtro passa-banda nm rispettivamente.
- Acquisire la dispersione in avanti (FCS) rispetto a lato scatter (SSC) diagramma a punti per le cellule non colorate con scala lineare. Regolare l'amplificazione di ogni rivelatore di visualizzare le cellule nel quadrante in basso a sinistra del grafico dot.
- Esempio di lettura di cellule intatte non permeabilizzate
- Impostare la P1gate per cellule non permeabilizzate vivi delineando una forma libera attorno alle cellule da analizzare escluse detriti cellulari e aggregati di cellule, limitando così il segnale di fluorescenza di cellule intatte.
Nota: Live / coloranti di esclusione morti possono essere utilizzati per facilitare il posizionamento cancello a cellule vive. Impostare 10.000 eventi da registrarenel P1 porta arresto. Impostare questo a un maggior numero di eventi se necessario. - Acquisire mCherry contro FITC trama di contorno a due parametri per rilevare autofluorescenza basale delle cellule non colorate. Utilizzare scala bi-logaritmica per mostrare valori negativi e migliorare la risoluzione tra le popolazioni 25. Regolare la tensione di ciascun sensore per impostare le cellule negative non colorate nella porzione inferiore delle prime dieci unità di piazzole intensità log fluorescenza.
- Acquisire tutti i campioni intatti non permeabilizzate utilizzando le impostazioni stabilite in 4.1.5 e 4.1.6 e raccogliere FSC, SSC e segnali nei rivelatori di fluorescenza.
- salvare i file * .fcs Export e da utilizzare per l'analisi mediante citometria a flusso software di analisi.
- Impostare la P1gate per cellule non permeabilizzate vivi delineando una forma libera attorno alle cellule da analizzare escluse detriti cellulari e aggregati di cellule, limitando così il segnale di fluorescenza di cellule intatte.
- Esempio di lettura di celle permeabilizzate
- Spostare il cancello P1 per selezionare cellule vive nei campioni permeabilizzate e regolare FSC e la tensione SSC come indicato in 4.1.5 e 4.1.6.
- Acquisire tutti i campioni permeabilizzate e raccogliere FSC, SSC unasegnali ND nei rivelatori di fluorescenza.
- salvare i file * .fcs Export e da utilizzare per l'analisi mediante citometria a flusso software di analisi.
- Analisi dei dati
- Avviare la citometria a flusso file del software di analisi e di importazione * .fcs salvati in 4.2.4 e 4.3.3.
- Fare clic sul primo campione elencato nella finestra di lavoro. Una nuova finestra prende il nome dal numero di ID tubo si apre automaticamente. Avviare il processo di gating nella trama di SSC contro FSC. Disegnare una porta (P1) utilizzando l'icona Ellisse intorno cellule vive ed eliminare tutti i residui, le cellule morte, o aggregati che hanno diversi scatter in avanti e scatter laterale di cellule vive
- Per disegnare la trama di contorno a due parametri del mCherry (asse y) rispetto FITC (asse x) intensità di fluorescenza delle cellule vive, clicca prima sul ascisse e scegliere la FITC-un canale e poi clicca sul y -axis e scegliere la PE-mCherry-Un canale. Clicca sull'icona "Quad" per posizionare l'indicatore quadrante sul bordo di uncellule utofluorescent in ogni canale di fluorescenza.
NOTA: Il cancello impostare intorno alle cellule positive FITC e mCherry è la porta P2. La popolazione cellulare negativa fluorescenza viene indicato come la porta P3. Vedere la Figura 5 per il metodo di gating rappresentante utilizzato in questo articolo. - Selezionare P2 e P3 porte e cliccare sull'icona "Aggiungi Statistica" nella finestra di lavoro originale. Clicca su "Count" (numero di cellule positive) e cliccare su "media" (Media di Fluorescenza intensità di ogni fluorocromo) o "mediano" (mediana fluorescenza intensità di ogni fluorocromo) statistiche tra l'elenco delle opzioni. Fare clic sull'icona "Aggiungi statistiche" di nuovo. Tutti questi valori vengono trasferiti automaticamente alla finestra di lavoro originale.
NOTA: La "media" è usato solo se l'intensità di fluorescenza segue una distribuzione normale. In ogni altro caso, fare clic sulla scheda "mediana". MFI quindi potrebbe riferirsi a fluorescenza media intesitày o mediana intensità di fluorescenza.
NOTA: Il passo successivo è quello di applicare i parametri e le statistiche dei cancelli 'a tutti i campioni rilevati dal citometro. - Nella finestra di lavoro, utilizzare il mouse per trascinare e rilasciare i parametri cancelli e statistiche sulla linea marcata tutti i campioni.
- Genera un rapporto lotto di trame bidimensionali di contorno (mCherry vs FITC) e istogrammi (conta delle cellule contro intensità di fluorescenza) per le cellule non permeabilizzate e permeabilizzate (figure 6A - B).
- Dalle statistiche generate in fase 4.4.4, calcolare l'intensità di fluorescenza media (MFI) per ogni fluorocromo per le cellule colorate. Da questo valore, sottrarre il valore di MFI ottenuto da cellule non colorate per quantificare la superficie e l'espressione totale della proteina di interesse.
- Riportare i valori ΔMFI per ogni fluoroforo (mCherry e FITC) (Figura 6C - D). Normalizzare il ΔMFI misurato per il Ca V α2δ1 costruire mutanti al valore ΔMFI ottenuto per FITC e mCherry con il costrutto WT.
NOTA: Il valore assoluto dell'intensità della fluorescenza può variare notevolmente a seconda del lotto di anticorpi e le capacità tecniche di ogni laboratorio lavoratore, da qui la necessità di normalizzare l'intensità di fluorescenza del costrutto mutante utilizzando il costrutto WT.
Risultati
Questo articolo descrive un protocollo affidabile per quantificare la superficie totale e delle cellule dei canali ionici ricombinanti espressi in TSA-201cells da un flusso a due colori citometria a test. Come esempio, la superficie di cella relativa e l'espressione di proteine totali è stata quantificata per il V α2δ1subunit Ca. Per eseguire il flusso a due colori citometria assay, Ca V α2δ1 stato doppiamente etichettato esprimere un HA epitopo non fluores...
Discussione
Questo test citometria a base di flusso è stato applicato con successo per la misurazione dei livelli relativi totale e superficie cellulare di subunità fluorescenza marcata formano pori e associati dei canali del calcio voltaggio-dipendenti 14,22,26. Si è utilizzato al meglio quando si indaga l'impatto di mutazioni genetiche e richiede quindi che l'intensità di fluorescenza intrinseca del wild-type costrutto fluorescenza marcata tag essere di almeno 10 a 100 volte più grande per l'intensità...
Divulgazioni
The authors declare that they have no competing financial interests.
Riconoscimenti
We thank Mr. Serge Sénéchal and Dr. Jacques Thibodeau for sharing their expertise and granting us access to their flow cytometry and cell sorting platform. This work was completed with the operating grant 130256 from the Canadian Institutes of Health Research, a grant-in-aid from the Canadian Heart and Stroke Foundation, and support from the "Fondation de l'Institut de Cardiologie de Montréal" to L.P.
Materiali
| Name | Company | Catalog Number | Comments |
| Q5 Site-Directed Mutagenesis Kit | New England Biolabs | E0554S | Can be substitute with QuickChange site-directed mutagenesis Kit (Agilent, #200523). |
| Tubes 1.5 ml | Sarstedt | 72-690-001 | |
| Tubes 15 ml | Sarstedt | 62-554-002 | |
| Disposable graduated Tranfer Pipets | VWR | 160001-192 | |
| 100 mm culture dish | Corning | 430167 | For standard culture of HEKT cells. |
| 35 mm culture dish | Falcon | 353001 | For standard culture of HEKT cells. |
| Serological pipette 1 ml | Sarstedt | 86.1251.001 | |
| Serological pipette 5 ml | Sarstedt | 86.1253.001 | |
| Serological pipette 10 ml | Sarstedt | 86.1254.001 | |
| Serological pipette 25 ml | Sarstedt | 86.1285.001 | |
| Dulbecco's high-glucose medium | Life Technologies | 12100-046 | Warm in 37 °C water bath before use. |
| Fetal Bovine Serum, qualified, heat inactivated, US origin | Life Technologies | 16140-071 | |
| Penicillin-Streptomycin (10,000 U/ml) | Life Technologies | 15140-122 | |
| Lipofectamine 2000 | Life Technologies | 11668-019 | For liposomal transfection. Can be substituted with calcium phosphate transfection. |
| Opti-MEM I Reduced Serum Medium | Life Technologies | 31985-070 | Warm in 37 °C water bath before use. |
| Trypsin-EDTA (1x) 0.05%, phenol red | Life Technologies | 25300-062 | |
| 1.5 ml microtubes | Sarstedt | 72.690.001 | |
| Phosphate Buffered Saline 1x | Fisher | BP661-10 | Can be "home-made". |
| Anti-HA FITC conjugated antibody | Sigma | H7411 | |
| IgG1−FITC Isotype Control antibody | Sigma | F6397 | |
| BD Cytofix/Cytoperm Fixation/Permeabilization Solution Kit | BD Biosciences | 554714 | Fixation/Permeabilization. Permeabilization/Wash solution, store at 4 °C. |
| Hemacytometer | Fisher | 49105161 | |
| Trypan Blue | Fisher | 15250061 | To access cell viability. |
| Refrigerated Microcentrifuge, 5430R | Eppendorf | A14H172200 | |
| Forma Steri-Cycle CO2 Incubator | Fisher | 370 | |
| Laboratory Platform Rocker | Fisher | 545034 | |
| Water Bath | VWR | 89032-216 | |
| BD FACSARIA III | BD Biosciences | 648282 | Flow cytometer. |
| FlowJo Software v10 | FlowJo | FlowJo v10 Dongle | For data analysis. |
Riferimenti
- Delisle, B. P., Anson, B. D., Rajamani, S., January, C. T. Biology of Cardiac Arrhythmias: Ion Channel Protein Trafficking. Circ. Res. 94, 1418-1428 (2004).
- Birault, V., Solari, R., Hanrahan, J., Thomas, D. Y. Correctors of the basic trafficking defect of the mutant F508del-CFTR that causes cystic fibrosis. Curr Opin Chem Biol. 17, 353-360 (2013).
- Balijepalli, S. Y., Anderson, C. L., Lin, E. C., January, C. T. Rescue of Mutated Cardiac Ion Channels in Inherited Arrhythmia Syndromes. J. Cardiovas Pharm. 56, 113-122 (2010).
- Gargus, J. J. Unraveling Monogenic Channelopathies and Their Implications for Complex Polygenic Disease. Am. J. Hum. Genet. 72, 785-803 (2003).
- Abriel, H., Zaklyazminskaya, E. V. Cardiac channelopathies: Genetic and molecular mechanisms. Gene. 517, 1-11 (2013).
- Behr, E. R., et al. Sudden arrhythmic death syndrome: familial evaluation identifies inheritable heart disease in the majority of families. Eur Heart J. 29, 1670-1680 (2008).
- Catterall, W. A. Structure and regulation of voltage-gated Ca2+ channels. Annu. Rev. Cell Dev.Biol. 16, 521-555 (2000).
- Peterson, B. Z., DeMaria, C. D., Adelman, J. P., Yue, D. T. Calmodulin is the Ca2+ sensor for Ca2+ -dependent inactivation of L- type calcium channels. Neuron. 22, 549-558 (1999).
- Dolphin, A. C. Calcium channel diversity: multiple roles of calcium channel subunits. Curr.Opin.Neurobiol. 19, 237-244 (2009).
- Dai, S., Hall, D. D., Hell, J. W. Supramolecular assemblies and localized regulation of voltage-gated ion channels. Physiol Rev. 89, 411-452 (2009).
- Gao, T., et al. Identification and subcellular localization of the subunits of L-type calcium channels and adenylyl cyclase in cardiac myocytes. J. Biol. Chem. 272, 19401-19407 (1997).
- Carl, S. L., et al. Immunolocalization of sarcolemmal dihydropyridine receptor and sarcoplasmic reticular triadin and ryanodine receptor in rabbit ventricle and atrium. J. Cell Biol. 129, 673-682 (1995).
- Abriel, H., Rougier, J. S., Jalife, J. Ion Channel Macromolecular Complexes in Cardiomyocytes: Roles in Sudden Cardiac Death. Circ. Res. 116, 1971-1988 (2015).
- Bourdin, B., et al. Molecular Determinants of the Cavb-induced Plasma Membrane Targeting of the Cav1.2 Channel. J. Biol. Chem. 285, 22853-22863 (2010).
- Raybaud, A., et al. The Role of the GX9GX3G Motif in the Gating of High Voltage-activated Calcium Channels. J. Biol. Chem. 281, 39424-39436 (2006).
- Burashnikov, E., et al. Mutations in the cardiac L-type calcium channel associated with inherited J-wave syndromes and sudden cardiac death. Heart Rhythm. 7, 1872-1882 (2010).
- Hennessey, J. A., et al. A CACNA1C Variant Associated with Reduced Voltage-Dependent Inactivation, Increased Cav1.2 Channel Window Current, and Arrhythmogenesis. PLoS ONE. 9, e106982 (2014).
- Adan, A., Alizada, G., Kiraz, Y., Baran, Y., Nalbant, A. Flow cytometry: basic principles and applications. Crit Rev Biotechnol. , 1-14 (2016).
- Graham, M. D. The Coulter Principle: Foundation of an Industry. J. Lab. Autom. 8, 72-81 (2003).
- Baumgarth, N., Roederer, M. A practical approach to multicolor flow cytometry for immunophenotyping. J. Immunol. Methods. 243, 77-97 (2000).
- Rothe, G., Sack, U., Tarnok, A., Rothe, G. . Cellular Diagnostics. Basics, Methods and Clinical Applications of Flow Cytometry. , 53-88 (2009).
- Bourdin, B., et al. Functional Characterization of Cavalpha2delta Mutations Associated with Sudden Cardiac Death. J. Biol. Chem. 290, 2854-2869 (2015).
- Tetreault, M. P., et al. Identification of glycosylation sites essential for surface expression of the Cavalpha2delta1 subunit and modulation of the cardiac Cav1.2 channel activity. J. Biol. Chem. 291, 4826-4843 (2016).
- Senatore, A., Boone, A. N., Spafford, J. D. Optimized Transfection Strategy for Expression and Electrophysiological Recording of Recombinant Voltage-Gated Ion Channels in HEK-293T Cells. J Vis Exp. (47), (2011).
- Herzenberg, L. A., Tung, J., Moore, W. A., Herzenberg, L. A., Parks, D. R. Interpreting flow cytometry data: a guide for the perplexed. Nat.Immunol. 7, 681-685 (2006).
- Shakeri, B., Bourdin, B., Demers-Giroux, P. O., Sauve, R., Parent, L. A quartet of Leucine residues in the Guanylate Kinase domain of Cavbeta determines the plasma membrane density of the Cav2.3 channel. J Biol Chem. 287, 32835-32847 (2012).
- Morton, R. A., Baptista-Hon, D. T., Hales, T. G., Lovinger, D. M. Agonist- and antagonist-induced up-regulation of surface 5-HT3A receptors. Br. J. Pharmacol. 172, 4066-4077 (2015).
- Hoffmann, C., et al. Fluorescent labeling of tetracysteine-tagged proteins in intact cells. Nat. Protocols. 5, 1666-1677 (2010).
- Cockcroft, C. J., Gamper, N. . Ion Channels: Methods and Protocols. , 233-241 (2013).
- Gonzalez-Gutierrez, G., Miranda-Laferte, E., Neely, A., Hidalgo, P. The Src Homology 3 Domain of the beta-Subunit of Voltage-gated Calcium Channels Promotes Endocytosis via Dynamin Interaction. J. Biol.Chem. 282, 2156-2162 (2007).
- Galizzi, J. P., Borsotto, M., Barhanin, J., Fosset, M., Lazdunski, M. Characterization and photoaffinity labeling of receptor sites for the Calcium channel inhibitors d-cis-diltiazem, (+/-)-bepridil, desmethoxyverapamil, and (+)-PN 200-110 in skeletal muscle transverse tubule membranes. J. Biol.Chem. 261, 1393-1397 (1986).
- Bezanilla, F. The voltage sensor in voltage-dependent ion channels. Physiol.Rev. 80, 555-592 (2000).
- Sigworth, F. J. The variance of sodium current fluctuations at the node of Ranvier. J Physiol. 307, 97-129 (1980).
- Bailey, M. A., Grabe, M., Devor, D. C. Characterization of the PCMBS-dependent modification of KCa3.1 channel gating. J. Gen. Physiol. 136, 367-387 (2010).
- Fletcher, P. A., Scriven, D. R., Schulson, M. N., Moore, E. D. Multi-Image Colocalization and Its Statistical Significance. Biophys. J. 99, 1996-2005 (2010).
- Lizotte, E., Tremblay, A., Allen, B. G., Fiset, C. Isolation and characterization of subcellular protein fractions from mouse heart. Anal. Biochem. 345, 47-54 (2005).
- Mattheyses, A. L., Simon, S. M., Rappoport, J. Z. Imaging with total internal reflection fluorescence microscopy for the cell biologist. J. Cell Sci. 123, 3621-3628 (2010).
- Yamamura, H., Suzuki, Y., Imaizumi, Y. New light on ion channel imaging by total internal reflection fluorescence (TIRF) microscopy. J. Pharmacol. Sci. 128, 1-7 (2015).
- Wible, B. A., et al. HERG-Lite-R: A novel comprehensive high-throughput screen for drug-induced hERG risk. J. Pharmacol. Toxicol. Methods. 52, 136-145 (2005).
- Wilde, A. A. M., Brugada, R. Phenotypical Manifestations of Mutations in the Genes Encoding Subunits of the Cardiac Sodium Channel. Circ. Res. 108, 884-887 (2011).
- Milano, A., et al. Sudden Cardiac Arrest and Rare Genetic Variants in the Community. Circ Cardiovasc Genet. , (2016).
- Schnell, U., Dijk, F., Sjollema, K. A., Giepmans, B. N. G. Immunolabeling artifacts and the need for live-cell imaging. Nat. Meth. 9, 152-158 (2012).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon