Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Monopolar Mitotik İğlerde Dönen Disk Mikroskobu ile Mikrotübül Dinamiğinin Ölçülmesi
Bu Makalede
Özet
Burada canlı hücre dönen disk konfokal mikroskopisi ve MATLAB tabanlı görüntü işleme kullanarak prometafazda senkronize hücrelerde mikrotübül dinamiği analizinin sağlam ve ayrıntılı bir yöntemini sıyoruz.
Özet
Canlı hücrelerdeki mikrotübül dinamiklerini belirlemek için kurulmuş bir yöntemin modifikasyonunu tanımlıyoruz. Protokol, mikrotübüllerin pozitif uçları (tdTomato floresan proteini ile etiketlenmiş EB3) ve dönen disk konfokal mikroskopisi kullanılarak yüksek hızlı, yüksek çözünürlüklü, canlı hücre görüntülemesi için genetik olarak kodlanmış bir belirteç ifadesine dayanmaktadır. Hücre döngüsü senkronizasyonu ve mikrotübüllerin artan yoğunluğu mitotik hücrelerde sentozomal ayırma inhibe ederek elde edilir ve büyüme analizi açık kaynak U-Track yazılımı kullanılarak gerçekleştirilir. Parlak ve kırmızı yayılılmış floresan proteinin kullanımı, düşük lazer gücü ve iplik disk mikroskobu için gerekli azaltılmış maruz kalma süresi ile birlikte fototoksisiteyi ve ışığa bağlı objelerin olasılığını azaltır. Bu standart kültür koşulları altında bir büyüme ortamda hücreleri korurken aynı hazırlık hücrelerin daha fazla sayıda görüntüleme sağlar. Analiz denetlenen otomatik bir şekilde yapıldığından, sonuçlar istatistiksel olarak sağlam ve tekrarlanabilir.
Giriş
Mikrotübüller (MTs) hemen hemen tüm ökaryotik hücrelerde ve bazı bakterilerde bulunan son derece dinamik yapılardır1. Aktin ve ara filamentlerle birlikte sitoiskelet2,3'ün yonttürün. Hücre bölünmesi4, molekül taşıma5, flagellar dayak6, birincil cilium7ile çevrenin hissi , işitme (kinocilium)8,9, embriyogenez10,11,12, invazyon ve metastaz13,14, ve hatta bellek oluşumu15,16,17,18, ve diğer pek çok süreç öncelikle MTs güveniyor. Tüm bu olaylara MTs katılımı hızla büyüme (polimerizasyon) ve büzülme (depolimerizasyon) arasında geçiş olağanüstü yeteneği olmadan imkansız olacaktır. Bu özellik dinamik kararsızlık19olarak tanımlanır. MT dinamikliği birçok patolojik koşullarda değiştirilir20,21,22. Bu nedenle, bu özelliğin doğasını belirleme hastalık mekanizmaları ve daha sonra tedavi anlamak için yardımcı olabilir.
MT dinamiği analizi için uzun bir yöntem listesi geliştirilmiştir ve bunların çoğu görüntüleme tekniklerine dayanmaktadır23. Başlangıçta, geniş alan ışık mikroskoplar in vitro24tubulin polimerlerin oluşumunu gözlemlemek için kullanılmıştır. MT artı uçlarında toplanan son bağlayıcı (EB)-proteinlerin keşfi ve floresan etiket proteinleri için yöntemlerin geliştirilmesi, geniş alan ve konfokal floresan mikroskoplar25,26,27ile canlı hücrelerde doğrudan MT'lerin davranışını gözlemlemek mümkün kılmaktadır. Bir EB-protein ilerler bağlayıcı protein 3 (EB3)28; bir floresan protein erimiş EB3 aşırı ekspresyon ve izleme ile, MT artı uç montaj oranlarıbelirlenebilir 29,30.
Konfokal lazer tarama floresan mikroskopisi (CLSM) sıklıkla MT dinamiklerini takip etmek için kullanılır. Ancak, bu görüntüleme tekniği fototoksisite ve fotobeyazrlama, canlı hücre ve loş örnek görüntüleme için iki istenmeyen süreçler yüksek bir risk teşkil31. Daha iyi bir sinyal-gürültü oranı elde etmek için, lazer gücü ve maruz kalma süresi numunelere zarar vermezken yeterince yüksek olmalıdır ve bu da hız karşılığında çözünürlükten ödün vermelidir. CLSM'ye uygun bir alternatif, dönen disk mikroskobu32'dir. Bu görüntüleme yöntemi bir Nipkow disk33kullanımına dayanmaktadır , hangi iğne delikleri bir dizi taşıyan hareketli bir disk oluşur, ve aynı anda aynı örneği görüntüleme birçok CLS mikroskoplar eşdeğer çalışır34. Bu nedenle, lazerden gelen ışık aynı anda örnekteki çeşitli bölgeleri aydınlatır ancak konfokal tabiatı korur. Nipkow disk, bu nedenle, CLSM benzer ama daha hızlı ve daha az lazer gücü kullanarak görüntüleri elde sağlar. Nipkow disk daha yokogawa Electric tarafından geliştirilmiştir, hangi mikrolensler bir dizi ile ikinci bir disk tanıttı bu ayrı ayrı bir iğne deliği içine ışık doğrudan, daha fazla fototoksisite ve fotobleaching azaltarak35. Böylece, dönen disk lazer tarama mikroskopisi canlı hücre görüntüleme için tercih edilen bir yöntem haline geldi, ve mümkün yüksek hızda yüksek sinyal-gürültü oranı ile görüntü elde etmek için yapar31,36, bu hızlı büyüyen MT biter gibi sinyalleri çözmek için çok önemlidir.
MT dinamiği geçici olarak farklılık gösterir. Örneğin, mitotik MTs interfaz olanlar37,38daha dinamiktir. Benzer şekilde, büyüme hızı ve büzülme farklılıkları aynı hücre döngüsü fazı içinde bile gözlenmiştir, mitoz gibi39,40. Bu nedenle, yanlış veri toplamayı önlemek için, MT dinamiğinin ölçümü hücre döngüsü sırasında dar bir zaman penceresi ile sınırlandırılmalıdır. Örneğin, prometafazda MT dinamiğinin ölçümü, motor kinsin Eg541'i inhibe eden ve bipolar mitotik mil oluşumunu engelleyen bir monastrol analogu olan dimethylenastron (DME) hücreleri tedavi ederek elde edilebilir42. Eg5 inhibitörü DME ve diğer monastrol türevleri ile prometafazda hücrelerin inhibisyonu MT dinamikleri etkilemez43,44,45, Hangi DME sabit ve canlı hücrelerde hem de MT dinamikleri incelemek için yararlı bir araç yapar44.
Burada Ertych ve ark.44 tarafından tanımlanan prometafaz hücrelerinde MT dinamiği analizi yöntemini çift dönen disk görüntüleme ile birleştiriyoruz. Bu yöntem, fotobeyaztma ve minimum fototoksisite olmaksızın, daha yüksek görüntüleme hızına sahip tek bir odak düzleminden toplanan prometafaz hücrelerinde MT dinamiğinin ölçülmesine olanak sağlar. Ayrıca, floresan muhabiri olarak, yeşil floresan protein (EGFP) ile karşılaştırıldığında parlaklık ve fotostabilite geliştirilmiş ve daha düşük enerjiLi ışık ile heyecanlı tandem dimer Domates floresan protein (tdTomato) kullanın46. Bu nedenle, tdTomato uyarma için daha az lazer gücü gerektirir ve daha az fototoksik. Toplamda, fototoksisiteyi azaltarak ve MT dinamikanalizi için gerekli olan çözünürlüğü ve postprocessingi iyileştirerek yöntemi daha da geliştiriyoruz. Ayrıca, diğer senkronizasyon teknikleri ile birleştirerek yöntemin gelecekteki değişiklikler için bir temel oluşturmak.
Protokol
1. HeLa Hücrelerinin Tohumlanması
- Fosfat tamponlu salin (PBS) içinde 2 mL 5 μg/mL fibronectin çözeltisi hazırlayın ve 4 iyi odalı coverslip 'in her kuyuya 450 μL ekleyin (#1.5). 37 °C ve %5 CO2'de15 dakika boyunca kaydırağı kuluçkaya yatırın.
- Dulbecco'nun Fosfat Tamponlu Salini (DPBS) ile eş zamanlı olarak büyüyen HeLa hücrelerini durulayın ve tripsin-EDTA ile inkübün (%0.05: 0.02; w:v) 5 dakika boyunca 37 °C'de. Roswell Park Memorial Institute (RPMI) 1640 orta 3:1 (v:v) eklenen trypsin-EDTA oranı% 10 ısı-inaktive fetal buzağı serum (FCS) ile takviye eklenmesi ile enzimatik reaksiyonu durdurun.
NOT: HeLa hücreleri RPMI 1640'ta 37 °C'de %10 ısı yalıtımlı FCS ve %5 CO2 ile desteklenmiş ve yukarıda açıklandığı gibi %80-90 birleştiğinde rutin olarak geçilmiştir. - Neubauer odasını kullanarak hücre konsantrasyonu belirleyin. Hücre süspansiyonunun 50 μL'lik aliquot'u 1:1 (v:v) oranında trypan mavisi ile karıştırın, yeniden askıya alın ve süspansiyonun 10 μL'sini odaya aktarın. Dört büyük kareiçinde yalnızca trypan mavi-negatif hücreleri sayın (ayrıntılar için Bkz. Phelan ve ark.47). Aşağıdaki formülü kullanarak sayılan hücre sayısından hücre konsantrasyonu elde edin:
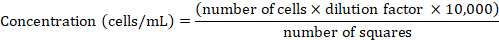
- 1 x 106 hücre/mL elde etmek için hücreleri 300 x g 2 dk. Taze RPMI 1640 ile yeniden askıya alarak santrifüj ile peletleyin.
- Fibronektini odacıklı kapaktan çıkarın, kuyuları DPBS ile iki kez yıkayın ve kuyu başına 50.000 hücre tohumlayın.
- Odacıklı kapak kaymasını hücrelerle birlikte kuvöze geri döndürün ve 37 °C'de 24 saat ve %5 CO2'debüyütün.
2. HeLa Hücrelerinde pEB3-tdTomato ifadesi
- 1,5 mL mikrosantrifüj tüp hazırlayın. Her tüp için 2 μg pEB3-tdTomato48 transfection tamponu (sulu çözeltideki sentetik ürün) ile 396 μL'lik son hacmi seyreltin.
- İlk tüpe 4 μL transfeksiyon reaktifi (polietilenin içeren non-lipidic) ekleyin ve karışımı hemen tam 10 s için girdap.
- Kısaca bir mikrocentrifuge ile tüp aşağı spin ve oda sıcaklığında kuluçka (RT) için 10 dakika.
- HeLa hücrelerini kuvözden çıkarın. Dropwise, transfeksiyon karışımının 100 μL'sini 4 iyi odalı coverslip'in her kuyusuna ekleyin ve hücreleri kuvöze geri döndürün.
- 37 °C'de 4 saat kuluçka ve %5 CO2'densonra, hücreleri 37 °C'de en az 24 saat taze büyüme ortamı ve kuluçka ile tamamlar ve %5 CO2.
NOT: Her hücre tipi için transfeksiyon koşullarını optimize etmek gereklidir. İfade düzeyleri, tek MT büyüyen uçların tanımlanmasına izin verecek kadar düşük olmalıdır. Alternatif olarak, EB3-tdTomato'yı ifade eden bir hücre hattı deneylerde kullanılabilir; bu preparatlar arasında ve aynı hazırlık49hücreler arasında EB3-tdTomato ifade düzeylerinde değişkenlik azaltacaktır.
3. PEB3-tdTomato-ifade eden HeLa Hücrelerinin Senkronizasyonu ve Canlı Hücre Görüntülemesi
- Fenol kırmızısı serbest Dulbecco'nun Modifiye Kartal Ortasında (DMEM) %10 FCS ve 2 mM L-glutamin veya alternatif glutamin kaynağı ile desteklenen 2,5 μM dimethylenastron (DME) çözeltisi hazırlayın.
- Odacıklı kapaktaki büyüme ortamını 2,5 μM DME içeren 500 μL büyüme ortamı ile değiştirin ve hücreleri 37 °C ve %5 CO2'dekuluçkaya yatırın.
- DME ile 3,5 saat kuluçka dan sonra, hücreleri mikroskoba aktarın, 37 °C ve% 5 CO2görüntüleme için koyu paneller ile bir çevre odasına odacıklı kapak monte ve toplam kuluçka süresi 4 saat olana kadar daha fazla kuluçka.
NOT: Sıcaklık 37 °C'de dalgalanma olmadan bakımı deney için çok önemlidir. - 100x 1.49 N.A. yağ daldırma hedefi, çift dönen disk konfokal sistemi ve odak düzleminin sürekli bakımı için güvenilir bir otofokus sistemi ile donatılmış ters bir mikroskopta hızlandırılmış görüntüleme gerçekleştirin. Görüntüleme parametrelerini aşağıdaki gibi tanımlayın.
NOT: Elektron Çarpma Yükü-Birleştirilmiş Cihaz kamerası (EM-CCD) kullanıyoruz.- EB3-tdTomato uyarma için, 200 ms maruz kalma süresine sahip 561 nm lazer hattı kullanın. Yayılan ışığı dörtlü bir bandpass (405, 488, 561, 640 nm) dikroik ayna ve 600/52 nm emisyon filtresi ile toplayın.
NOT: Görüntü doygunluğu önlemek için her görüntülenen hücre için lazer gücü ayarlanabilir. Burada verilen tüm zaman atlamalı filmlerde lazer gücü 5.3 mW olarak belirlenmiştir. - Profaz bir hücre bulun ve z düzleminde monopolar mitotik milin merkezine karşılık gelen odak. Pozlamalar arasında hiçbir binning ve hiçbir aydınlatma ile 1 dakika toplam üzerinden her 0,5 sgörüntüleri elde.
- EB3-tdTomato uyarma için, 200 ms maruz kalma süresine sahip 561 nm lazer hattı kullanın. Yayılan ışığı dörtlü bir bandpass (405, 488, 561, 640 nm) dikroik ayna ve 600/52 nm emisyon filtresi ile toplayın.
4. U-Track v2.2.0 kullanarak MT Dinamiği Analizi
- MT dinamiklerini analiz etmek için sayısal bir bilgi işlem ortamı yazılımı gereklidir (örneğin, MATLAB).
NOT: Yazılımın temel anlayışı analiz için yeterlidir. Kapsamlı yardım materyalleri ve öğreticiler geliştiricinin web sitesinde (https://uk.mathworks.com/products/matlab/getting-started.html) mevcuttur. - Download (https://github.com/DanuserLab/u-track) ve "Readme_u-track.pdf" dosya50,51,52verilen ayrıntılı talimatları aşağıdaki açık kaynak U-Track v2.2.0 yazılımı yükleyin.
- Sayısal analiz yazılımını başlatın ve alt klasörleri içeren U-Track v2.2.0 klasörünü yazılım arama yoluna ekleyin.
- Komut penceresinden "movieSelectorGUI" numaralı telefonu arayın. Bu, mikroskoptaki görüntü edinme yazılımı tarafından oluşturulan ham dosyaların içe aktarılabildiği bir diyalog penceresi açar (Ek Şekil 1, Şekil 2, Şekil 3, Şekil 4).
NOT: U-Track yazılımı diğer görüntü veri biçimleriyle uyumludur. Farklı yaşam bilimi veri formatları53tanır Bio-Formats kullanır. - Her görüntünün boyutu meta verilerden otomatik olarak okunur. Amacın sayısal diyafram açıklığını (bu durumda 1,49) ve görüntüleme için kullanılan zaman aralığını (0,5 s) manuel olarak girin(Ek Şekil 1B). Ayrıca, uyarma dalga boyu hakkında bilgi, florofor, ve maruz kalma süresi de sağlanabilir, ancak daha fazla analiz için kritik değildir.
- Tüm görüntüler yüklendikten sonra, "Film Olarak Kaydet" seçeneğini seçerek girilen hızlandırılmış diziyi film listesi olarak kaydedin. Diyalog penceresinin sağ tarafında "U-Track" seçeneğini seçin ve "Continue" (Ek Şekil 1C)tuşuna basın.
NOT: Değerler HeLa hücreleri için optimize edilebiyi gösterir. Farklı bir hücre satırına geçiyorsanız, değerler yeniden tanımlanmalıdır. Alternatif olarak, yazılım geliştiricileri tarafından önerilen ayarları kullanın. Parametrelerin her birinin ayrıntılı açıklaması ve bunların nasıl tanımlanması gerektiği, yazılımın önceki sürümü plusTipTracker50ile sağlanan teknik raporda bulunabilir. - Açılan pencereden "Microtubule Plus-Ends" seçeneğini seçin ve "Ok" (Ek Şekil 1C)tuşuna basın. Yeni diyalog penceresi, algılama, izleme ve izleme analizi olan analizin üç adımı(Ek Şekil 1D)parametrelerinin belirlenmesine olanak tanır.
- Adım 1'de "Ayarlar"ı seçin ve açılır menüden algılama yöntemi olarak "Kuyruklu yıldız algılama" seçeneğini seçin (Ek Şekil 2B).
- Yeni diyalog penceresinden Gaussians filtre ve havza segmentasyon farkı için parametreleri tanımlamak aşağıdaki gibi (Ek Şekil 2C): Maske işlemi algılama = Yok için kullanılacak; Düşük geçişli Gaussian standart sapma = 1 piksel; Yüksek geçişli Gaussian standart sapma = 3 piksel; Minimum eşik = 3 standart sapma; Eşik adım boyutu = 0,25 standart sapmalar. "Tüm Filmlere Ayarlar Uygula" ve " Uygula " seçeneğinibelirleyin.
- 2. adımda, bağlama, boşluk kapatma, birleştirme ve bölme parametreleri ve Kalman filtre işlevleri pembe, yeşil ve mavi olarak vurgulandığı üzere üç adımda tanımlanır (Ek Şekil 3B). Bu adımlar için sırasıyla "Microtübule Plus-end Dynamics" seçeneğini belirleyin ve "Ayarla" seçeneğini belirleyin, Sırasıyla Ek Şekil 3C-E'debelirtildiği gibi değerleri tanımlayın.
- Boyutsallıkla ilgili sorunlar için açılır menüden "2" seçeneğini belirleyin. Kapatmak için Maksimum Boşluğu Kullanın = 5 kare; İlk Adımdan Parça Segmentlerinin Minimum Uzunluğu = 3 kare. Daha önce olduğu gibi , "Tüm Filmlere Ayarlar Uygula" seçeneğini seçin ve "Uygula" düğmesine tıklayın.
- Analizin 3. Bir parça analiz yöntemi olarak , "Mikrotübül Dinamikleri Sınıflandırması" seçeneğini belirleyin ve parametreleri Ek Şekil 4B,C'debelirtildiği gibi "Ayar" düğmesi ile tanımlayın. Bundan sonra "Tüm Filmlere Ayarlar Uygula" kutusunu seçin ve "Uygula" düğmesine tıklayın.
- Tüm parametreler tanımlandıktan sonra , "Control Panel-U-Track" penceresinden (Ek Şekil 1D)"Tüm Filmlere Kontrol Et/Çeki Çeki Uygula" ve " Tüm FilmleriÇalıştır" kutularını seçin ve "Çalıştır" tuşuna basın. Bu, hızlandırılmış serinin MT analizini başlatacaktır.
- Film işleme tamamlandıktan sonra "Filminiz başarıyla işlendi" mesajı görüntülenir. "Ok" tuşuna basın, sonra "Kaydet".
- Artık sayısal analiz yazılımından çıkmak güvenlidir. Film işleme sonuçları ham dosyaların depolandığı klasörde m-files olarak alt klasör yapılarında saklanır.
5. MT Dinamiklerinin İstatistiksel Analizi
- M-dosyalarını tercih edilen bir istatistik çözümleme programına aktarın.
NOT: Bizim durumumuzda, dosyaları okunabilir hale getirmek için önce standart bir elektronik tabloya aktarıyoruz. M-dosyaları farklı parametreler (örneğin, büyüme hızı, MT dinamikliği) hakkında istatistiksel bilgiler (ortanca, ortalama ve standart sapma) içerir. Parametrelerin ayrıntılı listesi yazılımın önceki sürümü ile sağlanan teknik raporda verilir, plusTipTracker50,52. Oluşturulan m-dosyaları diğer veri işleme yazılımlarına da aktarılabilir. - "Büyüme hızı ortalaması" parametresini seçin ve istatistikler ve görüntüleme için bir tabloya aktarın. Diğer parametrelerle ilgili bilgileri (örneğin, "dinamiklik") yeni bir tabloya veya aynı gruplanmış tablo ve çizimin yeni bir sütununa girin.
Sonuçlar
Şekil 1A'daözetlenen protokolütaki pEB3-tdTomato plazmid, eş zamanlı olarak büyüyen HeLa hücrelerinde geçici olarak ifade edildi. DME tedavisi ile prometafazda transfeksiyon dan sonra hücreler 48 saat senkronize edildi(Şekil 1B). Bu adım, MT dinamiğinin ölçümünün her zaman hücre döngüsünün aynı aşamasında yapılmasını sağlamıştır. Zaman atlamalı filmler daha fazla işlenmiş ve U-Track v2.2.0 ile analiz olarak ek belgelerde açı...
Tartışmalar
Burada, ilk ertych ve ark.44tarafından kurulan bir yöntemin bir değişiklik açıklar. Diğer bazı modifikasyonlarla birlikte, bu MT dinamiği analizi tekniğini çift dönen disk konfokal görüntüleme ile birleştiriyoruz. Çift eğirme diskinin kullanımı fototoksisiteyi azaltırken mt'ler büyüyen çözünürlüğü artırır36. Daha uzun bir dalga boyu floresan muhabirine geçerek hücrelerin fotobeyaztma ve lazer ışık kaynaklı hasarını daha da azaltıyoru...
Açıklamalar
Yazarların açıklayacak bir şeyi yok.
Teşekkürler
Max-Planck Deneysel Tıp Enstitüsü Hafif Mikroskobu Tesisi üyelerine uzman tavsiye ve desteklerinden dolayı teşekkür ederiz.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Dimethylenastron | Merck | 324622 | |
| DMEM w/o phenol red | Gibco | 31053-28 | |
| DPBS | Gibco | 14190-094 | |
| Fetal bovine serum | Biochrom | S0415 | |
| Fibronectin Bovine Plasma | Merck | F4759 | Sterile powder |
| GlutaMAX | Gibco | 35050-038 | Stable glutamine substitutive |
| jetPRIME | Polyplus | 114-15 | |
| EB3-TdTomato | Addgene | plasmid #50708 | |
| RPMI 1640 | Gibco | 61870-010 | |
| Trypan Blue | Merck | T8154-20ML | |
| Trypsin/EDTA solution | Biochrom | L2143 | 0.05% / o.02 % w/o calcium and magnesium |
| µ-slide | Ibidi | 80426 | 4-well slide with #1.5 coverslip |
| Eclipse Ti Inverted microscope | Nikon | NA | |
| Objective | Nikon | MRD01991 | CFI Apo TIRF 100XC Oil |
| ACAL Laser Excahnger | Nikon | Laser box. 405, 458, 488, 514, 561 and 647nm | |
| Spinning disk module | Andor | CSU-W | |
| Camera | Andor | iXon Ultra 888 | |
| Environmental Chamber | Okolab | Dark chamber equipped with CO2 supply, tmeperature control and humidifier | |
| HeLa Cells | DSMZ | ACC-57 | |
| NIS Elements v4 | Nikon | Spinning disk microscope. Acquisition Software | |
| MATLAB | Mathworks | Computing environment | |
| Prism 8 | GraphPad | Statistical analysis and display software |
Referanslar
- Erickson, H. P. Evolution of the cytoskeleton. Bioessays. 29 (7), 668-677 (2007).
- Pollard, T. D., Goldman, R. D. Overview of the Cytoskeleton from an Evolutionary Perspective. Cold Spring Harbor Perspectives in Biology. 10 (7), (2018).
- Wade, R. H. On and around microtubules: an overview. Molecular Biotechnology. 43 (2), 177-191 (2009).
- Forth, S., Kapoor, T. M. The mechanics of microtubule networks in cell division. Journal of Cell Biology. 216 (6), 1525-1531 (2017).
- Franker, M. A., Hoogenraad, C. C. Microtubule-based transport - basic mechanisms, traffic rules and role in neurological pathogenesis. Journal of Cell Science. 126, 2319-2329 (2013).
- Lindemann, C. B., Lesich, K. A. Flagellar and ciliary beating: the proven and the possible. Journal of Cell Science. 123, 519-528 (2010).
- Wheway, G., Nazlamova, L., Hancock, J. T. Signaling through the Primary Cilium. Frontiers in Cell and Developmental Biology. 6, 8 (2018).
- Falk, N., Losl, M., Schroder, N., Giessl, A. Specialized Cilia in Mammalian Sensory Systems. Cells. 4 (3), 500-519 (2015).
- Spoon, C., Grant, W. Biomechanical measurement of kinocilium. Methods in Enzymology. 525, 21-43 (2013).
- Zenker, J., et al. A microtubule-organizing center directing intracellular transport in the early mouse embryo. Science. 357 (6354), 925-928 (2017).
- Goldstein, B. Embryonic polarity: a role for microtubules. Current Biology. 10 (22), 820-822 (2000).
- Uchida, S., Shumyatsky, G. P. Deceivingly dynamic: Learning-dependent changes in stathmin and microtubules. Neurobiology of Learning and Memory. 124, 52-61 (2015).
- Fife, C. M., McCarroll, J. A., Kavallaris, M. Movers and shakers: cell cytoskeleton in cancer metastasis. British Journal of Pharmacology. 171 (24), 5507-5523 (2014).
- Bouchet, B. P., Akhmanova, A. Microtubules in 3D cell motility. Journal of Cell Science. 130 (1), 39-50 (2017).
- Dent, E. W. Of microtubules and memory: implications for microtubule dynamics in dendrites and spines. Molecular Biology of the Cell. 28 (1), 1-8 (2017).
- Craddock, T. J., Tuszynski, J. A., Hameroff, S. Cytoskeletal signaling: is memory encoded in microtubule lattices by CaMKII phosphorylation. PLOS Computational Biology. 8 (3), (2012).
- Smythies, J. Off the beaten track: the molecular structure of long-term memory: three novel hypotheses-electrical, chemical and anatomical (allosteric). Frontiers in Integrative Neuroscience. 9, 4 (2015).
- Kaganovsky, K., Wang, C. Y. How Do Microtubule Dynamics Relate to the Hallmarks of Learning and Memory. Journal of Neuroscience. 36 (22), 5911-5913 (2016).
- Mitchison, T., Kirschner, M. Dynamic instability of microtubule growth. Nature. 312 (5991), 237-242 (1984).
- Dubey, J., Ratnakaran, N., Koushika, S. P. Neurodegeneration and microtubule dynamics: death by a thousand cuts. Frontiers in Cellular Neuroscience. 9, 343 (2015).
- Parker, A. L., Kavallaris, M., McCarroll, J. A. Microtubules and their role in cellular stress in cancer. Frontiers in Oncology. 4, 153 (2014).
- Honore, S., Pasquier, E., Braguer, D. Understanding microtubule dynamics for improved cancer therapy. Cell and Molecular Life Sciences. 62 (24), 3039-3056 (2005).
- Straube, A. . Methods in Molecular Biology. , (2011).
- Budde, P. P., Desai, A., Heald, R. Analysis of microtubule polymerization in vitro and during the cell cycle in Xenopus egg extracts. Methods. 38 (1), 29-34 (2006).
- Gierke, S., Kumar, P., Wittmann, T. Analysis of microtubule polymerization dynamics in live cells. Methods in Cell Biology. 97, 15-33 (2010).
- Matov, A., et al. Analysis of microtubule dynamic instability using a plus-end growth marker. Nature Methods. 7 (9), 761-768 (2010).
- Bailey, M., Conway, L., Gramlich, M. W., Hawkins, T. L., Ross, J. L. Modern methods to interrogate microtubule dynamics. Integrative Biology (Camb). 5 (11), 1324-1333 (2013).
- Galjart, N. Plus-end-tracking proteins and their interactions at microtubule ends. Current Biology. 20 (12), 528-537 (2010).
- Stepanova, T., et al. Visualization of microtubule growth in cultured neurons via the use of EB3-GFP (end-binding protein 3-green fluorescent protein). Journal of Neuroscience. 23 (7), 2655-2664 (2003).
- Zwetsloot, A. J., Tut, G., Straube, A. Measuring microtubule dynamics. Essays in Biochemistry. 62 (6), 725-735 (2018).
- Bayguinov, P. O., et al. Modern Laser Scanning Confocal Microscopy. Current Protocols in Cytometry. 85 (1), 39 (2018).
- Nakano, A. Spinning-disk confocal microscopy -- a cutting-edge tool for imaging of membrane traffic. Cell Structure and Function. 27 (5), 349-355 (2002).
- Nipkow, P. Elektrisches teleskop. Germany patent. , (1884).
- Yin, S., Lu, G., Zhang, J., Yu, F. T., Mait, J. N. Kinoform-based Nipkow disk for a confocal microscope. Applied Optics. 34 (25), 5695-5698 (1995).
- Tanaami, T., Kenta, M. Nipkow disk for confocal optical scanner. European patent application. , (1992).
- Oreopoulos, J., Berman, R., Browne, M. Spinning-disk confocal microscopy: present technology and future trends. Methods in Cell Biology. 123, 153-175 (2014).
- Rusan, N. M., Fagerstrom, C. J., Yvon, A. M., Wadsworth, P. Cell cycle-dependent changes in microtubule dynamics in living cells expressing green fluorescent protein-alpha tubulin. Molecular Biology of the Cell. 12 (4), 971-980 (2001).
- Rusan, N. M., Fagerstrom, C. J., Yvon, A. -. M. C., Wadsworth, P. Cell Cycle-Dependent Changes in Microtubule Dynamics in Living Cells Expressing Green Fluorescent Protein-α Tubulin. Molecular Biology of the Cell. 12 (4), 971-980 (2001).
- Liu, D., Davydenko, O., Lampson, M. A. Polo-like kinase-1 regulates kinetochore-microtubule dynamics and spindle checkpoint silencing. Journal of Cell Biology. 198 (4), 491-499 (2012).
- Maiato, H., Sunkel, C. E. Kinetochore-microtubule interactions during cell division. Chromosome Research. 12 (6), 585-597 (2004).
- Muller, C., et al. Inhibitors of kinesin Eg5: antiproliferative activity of monastrol analogues against human glioblastoma cells. Cancer Chemotherrapy and Pharmacology. 59 (2), 157-164 (2007).
- Mayer, T. U., et al. Small molecule inhibitor of mitotic spindle bipolarity identified in a phenotype-based screen. Science. 286 (5441), 971-974 (1999).
- Kapoor, T. M., Mayer, T. U., Coughlin, M. L., Mitchison, T. J. Probing spindle assembly mechanisms with monastrol, a small molecule inhibitor of the mitotic kinesin Eg5. The Journal of Cell Biology. 150 (5), 975-988 (2000).
- Ertych, N., et al. Increased microtubule assembly rates influence chromosomal instability in colorectal cancer cells. Nature Cell Biology. 16 (8), 779-791 (2014).
- Brito, D. A., Yang, Z., Rieder, C. L. Microtubules do not promote mitotic slippage when the spindle assembly checkpoint cannot be satisfied. The Journal of Cell Biology. 182 (4), 623-629 (2008).
- Shaner, N. C., Patterson, G. H., Davidson, M. W. Advances in fluorescent protein technology. Journal of Cell Science. 120 (24), 4247-4260 (2007).
- Phelan, M. C., Lawler, G. Cell Counting. Current Protocols in Cytometry. 00 (1), 3 (1997).
- Merriam, E. B., et al. Synaptic regulation of microtubule dynamics in dendritic spines by calcium, F-actin, and drebrin. Journal of Neuroscience. 33 (42), 16471-16482 (2013).
- Samora, C. P., et al. MAP4 and CLASP1 operate as a safety mechanism to maintain a stable spindle position in mitosis. Nature Cell Biology. 13 (9), 1040-1050 (2011).
- Applegate, K. T., et al. plusTipTracker: Quantitative image analysis software for the measurement of microtubule dynamics. Journal of Structural Biology. 176 (2), 168-184 (2011).
- Jaqaman, K., et al. Robust single-particle tracking in live-cell time-lapse sequences. Nature Methods. 5 (8), 695-702 (2008).
- Stout, A., D'Amico, S., Enzenbacher, T., Ebbert, P., Lowery, L. A. Using plusTipTracker Software to Measure Microtubule Dynamics in Xenopus laevis Growth Cones. Journal of Visualized Experiments. , e52138 (2014).
- Linkert, M., et al. Metadata matters: access to image data in the real world. The Journal of Cell Biology. 189 (5), 777-782 (2010).
- Brouhard, G. J. Dynamic instability 30 years later: complexities in microtubule growth and catastrophe. Molecular Biology of the Cell. 26 (7), 1207-1210 (2015).
- Burbank, K. S., Mitchison, T. J. Microtubule dynamic instability. Current Biology : CB. 16 (14), 516-517 (2006).
- Caplow, M., Shanks, J., Ruhlen, R. L. Temperature-jump studies of microtubule dynamic instability. Journal of Biological Chemistry. 263 (21), 10344-10352 (1988).
- Prasad, V., Jordan, M. A., Luduena, R. F. Temperature sensitivity of vinblastine-induced tubulin polymerization in the presence of microtubule-associated proteins. Journal of Protein Chemistry. 11 (5), 509-515 (1992).
- Wasteneys, G. O. Microtubules Show their Sensitive Nature. Plant and Cell Physiology. 44 (7), 653-654 (2003).
- Turi, A., Lu, R. C., Lin, P. -. S. Effect of heat on the microtubule disassembly and its relationship to body temperatures. Biochemical and Biophysical Research Communications. 100 (2), 584-590 (1981).
- Safinya, C. R., et al. The effect of multivalent cations and Tau on paclitaxel-stabilized microtubule assembly, disassembly, and structure. Advances in Colloid and Interface Science. 232, 9-16 (2016).
- Sandoval, I. V., Weber, K. Calcium-Induced Inactivation of Microtubule Formation in Brain Extracts. European Journal of Biochemistry. 92 (2), 463-470 (1978).
- Vater, W., Böhm, K. J., Unger, E. Tubulin assembly in the presence of calcium ions and taxol: Microtubule bundling and formation of macrotubule-ring complexes. Cell Motility. 36 (1), 76-83 (1997).
- Yamashita, N., et al. Three-dimensional tracking of plus-tips by lattice light-sheet microscopy permits the quantification of microtubule growth trajectories within the mitotic apparatus. Journal of Biomedical Optics. 20 (10), 1-18 (2015).
- Pamula, M. C., et al. High-resolution imaging reveals how the spindle midzone impacts chromosome movement. Journal of Cell Biology. 218 (8), 2529-2544 (2019).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır