Method Article
فحص بنية الحمض النووي الريبي مع التنميط الطفري لكبريتات ثنائي ميثيل مع التسلسل في المختبر والخلايا
In This Article
Summary
يوفر البروتوكول تعليمات لتعديل الحمض النووي الريبي مع كبريتات ثنائي ميثيل لتجارب التنميط الطفري. ويشمل التحقيق في المختبر وفي الجسم الحي مع طريقتين بديلتين لإعداد المكتبة.
Abstract
أصبح دور بنية الحمض النووي الريبي في أي عملية بيولوجية تقريبا واضحا بشكل متزايد ، خاصة في العقد الماضي. ومع ذلك ، فشلت الأساليب الكلاسيكية لحل بنية الحمض النووي الريبي ، مثل علم بلورات الحمض النووي الريبي أو cryo-EM ، في مواكبة المجال سريع التطور والحاجة إلى حلول عالية الإنتاجية. التنميط الطفري مع التسلسل باستخدام كبريتات ثنائي ميثيل (DMS) MaPseq هو نهج قائم على التسلسل لاستنتاج بنية الحمض النووي الريبي من تفاعل القاعدة مع DMS. يقوم DMS بميثيل النيتروجين N1 في الأدينوزينات و N3 في السيتوزينات في وجه Watson-Crick عندما تكون القاعدة غير متزاوجة. يؤدي النسخ العكسي للحمض النووي الريبي المعدل مع النسخ العكسي للمجموعة الثانية من الإنترون القابل للحرارة (TGIRT-III) إلى دمج القواعد المثيلية كطفرات في cDNA. عند تسلسل cDNA الناتج وتعيينه مرة أخرى إلى نسخة مرجعية ، فإن معدلات الطفرة النسبية لكل قاعدة تشير إلى "حالة" القاعدة على أنها مقترنة أو غير متزاوجة. على الرغم من أن تفاعلات DMS لها نسبة إشارة إلى ضوضاء عالية في كل من المختبر والخلايا ، إلا أن هذه الطريقة حساسة للتحيز في إجراءات المعالجة. للحد من هذا التحيز ، توفر هذه الورقة بروتوكولا لعلاج الحمض النووي الريبي باستخدام DMS في الخلايا ومع الحمض النووي الريبي المنسوخ في المختبر .
Introduction
منذ اكتشاف أن الحمض النووي الريبي له خصائص هيكلية 1,2 و3 تحفيزية ، تم الكشف تدريجيا عن أهمية الحمض النووي الريبي ووظيفته التنظيمية في عدد كبير من العمليات البيولوجية. في الواقع ، اكتسب تأثير بنية الحمض النووي الريبي على تنظيم الجينات اهتماما متزايدا4. مثل البروتينات ، يحتوي الحمض النووي الريبي على هياكل أولية وثانوية وثالثية ، في إشارة إلى تسلسل النيوكليوتيدات ، ورسم الخرائط 2D لتفاعلات إقران القواعد ، والطي ثلاثي الأبعاد لهذه الهياكل المقترنة بالقاعدة ، على التوالي. في حين أن تحديد الهيكل الثلاثي هو المفتاح لفهم الآليات الدقيقة وراء العمليات المعتمدة على الحمض النووي الريبي ، فإن الهيكل الثانوي هو أيضا غني بالمعلومات فيما يتعلق بوظيفة الحمض النووي الريبي وهو الأساس لمزيد من الطي 3D5.
ومع ذلك ، فإن تحديد بنية الحمض النووي الريبي كان يمثل تحديا جوهريا مع الأساليب التقليدية. بينما بالنسبة للبروتينات ، فإن علم البلورات والرنين المغناطيسي النووي (NMR) والمجهر الإلكتروني المبرد (cryo-EM) قد جعل من الممكن تحديد تنوع الزخارف الهيكلية ، مما يسمح بالتنبؤ بالبنية من التسلسل وحده6 ، فإن هذه الأساليب لا تنطبق على نطاق واسع على الحمض النووي الريبي. في الواقع ، الحمض النووي الريبي عبارة عن جزيئات مرنة ذات كتل بناء (نيوكليوتيدات) تتمتع بحرية أكبر في المطابقة والدوران مقارنة بنظيراتها من الأحماض الأمينية. علاوة على ذلك ، فإن التفاعلات من خلال الاقتران القاعدي أكثر ديناميكية وتنوعا من تلك الموجودة في بقايا الأحماض الأمينية. نتيجة لذلك ، كانت الأساليب الكلاسيكية ناجحة فقط للحمض النووي الريبي الصغير نسبيا مع هياكل محددة جيدا ومدمجة للغاية7.
هناك نهج آخر لتحديد بنية الحمض النووي الريبي من خلال الفحص الكيميائي جنبا إلى جنب مع تسلسل الجيل التالي (NGS). تولد هذه الاستراتيجية معلومات حول حالة الربط لكل قاعدة في تسلسل الحمض النووي الريبي (أي هيكلها الثانوي). باختصار ، يتم تعديل القواعد في جزيء الحمض النووي الريبي التي لا تشارك في تزاوج القواعد بشكل تفاضلي بواسطة مركبات كيميائية صغيرة. يدمج النسخ العكسي لهذه الحمض النووي الريبي مع النسخ العكسي المتخصص (RTs) التعديلات في الحمض النووي الريبي منقوص الأكسجين التكميلي (cDNA) كطفرات. ثم يتم تضخيم جزيئات cDNA هذه بواسطة تفاعل البوليميراز المتسلسل (PCR) وتسلسلها. للحصول على معلومات حول "حالتها" على أنها مقيدة أو غير مرتبطة ، يتم حساب ترددات الطفرة في كل قاعدة في الحمض النووي الريبي محل الاهتمام وإدخالها في برنامج التنبؤ بالهيكل كقيود8. استنادا إلى أقرب قواعد الجيران9 والحد الأدنى من حسابات الطاقة الحرة 10 ، يقوم هذا البرنامج بإنشاء نماذج هيكلية تناسب البيانات التجريبية التي تم الحصول عليها11,12.
يستخدم DMS-MaPseq DMS ، الذي يثيل النيتروجين N1 في الأدينوزين والنيتروجين N3 في السيتوزينات في وجه Watson-Crick بطريقة محددة للغاية13. يؤدي استخدام النسخ العكسي للمجموعة الثانية القابلة للحرارة (TGIRT-III) في النسخ العكسي إلى إنشاء ملفات تعريف طفرية بنسب إشارة إلى ضوضاء غير مسبوقة ، حتى السماح بفك التفاف الملامح المتداخلة الناتجة عن اثنين أو أكثر من المطابقات البديلة14,15. علاوة على ذلك ، يمكن لنظام DMS اختراق أغشية الخلايا والأنسجة الكاملة ، مما يجعل التحقيق في السياقات الفسيولوجية ممكنا. ومع ذلك ، فإن توليد بيانات عالية الجودة يمثل تحديا ، حيث يمكن أن تؤثر الاختلافات في إجراءات المعالجة على النتائج. لذلك ، نقدم بروتوكولا مفصلا لكل من DMS-MaPseq في المختبر وداخل الخلية لتقليل التحيز وتوجيه الوافدين الجدد إلى الطريقة من خلال الصعوبات التي قد يواجهونها. خاصة في ضوء جائحة SARS-CoV2 الأخيرة ، تعد البيانات عالية الجودة حول فيروسات الحمض النووي الريبي أداة مهمة لدراسة التعبير الجيني وإيجاد العلاجات الممكنة.
Protocol
ملاحظة: راجع جدول المواد للحصول على التفاصيل المتعلقة بجميع المواد والبرامج والكواشف والأدوات والخلايا المستخدمة في هذا البروتوكول.
1. الجينات الخاصة في المختبر DMS-MaP
- الحمض النووي الريبي في النسخ المختبري
- الحصول على تسلسل الحمض النووي الريبي محل الاهتمام كحمض نووي مزدوج الشريط (ds) (على سبيل المثال ، كشظايا الحمض النووي أو البلازميدات أو تفاعل البوليميراز المتسلسل من الحمض النووي الموجود مسبقا / الجينومي). إذا كان تتابع الحمض النووي (DNA) يحتوي على محفز لإنزيم البلمرة، فانتقل إلى الخطوة 3.
- قم بإجراء تفاعل البوليميراز المتسلسل المتداخل لإرفاق محفز بوليميراز الحمض النووي الريبي في المنبع لجزء الحمض النووي المطلوب (التمهيدي الأمامي لبوليميراز T7: 5 'TAATACGACTCACTATAGG + القواعد الأولى للتسلسل المستهدف 3 ').
- في المختبر نسخ جزء الحمض النووي إلى الحمض النووي الريبي. احتفظ دائما بالحمض النووي الريبي على الجليد.
- هضم الحمض النووي باستخدام DNase.
- عزل الحمض النووي الريبي باستخدام نهج قائم على العمود (الخطوة 2.4) أو عن طريق ترسيب الإيثانول (الخطوة 2.5). Elute في حجم مناسب ، ونتوقع العائد من ~ 50 ميكروغرام.
- ضمان سلامة الحمض النووي الريبي عن طريق تشغيله على هلام الأغاروز ؛ قم بتشويه الحمض النووي الريبي لمدة 2-3 دقائق عند 70 درجة مئوية قبل الجري.
ملاحظة: يمكن أن يحتوي المخزن المؤقت والأغاروز على RNases التي تحط من الحمض النووي الريبي وقد تلوث عينة الحمض النووي الريبي. تم استخدام جل الأغاروز الجاهز سابقا في هذا المختبر. كانت النتائج (خاصة مع الحمض النووي الريبي) غامضة في بعض الأحيان. تم الحصول على أفضل النتائج مع المواد الهلامية agarose أو PAGE. - الاستخدام المباشر لتخزين الحمض النووي الريبي عند -80 درجة مئوية لعدة أشهر ما لم يكن التحلل مرئيا بعد الذوبان.
- في المختبر تعديل DMS (عند 105 mM DMS)
- تحضير كمية كافية من العازلة القابلة لإعادة الطي (0.4 M كاكوديلات الصوديوم ، درجة الحموضة 7.2 ، تحتوي على 6 mM MgCl2).
ملاحظة: لكل تفاعل (الحجم النهائي 100 ميكرولتر) ، أضف 89 ميكرولتر من المخزن المؤقت القابل لإعادة الطي. - لكل تفاعل ، انقل 89 ميكرولتر من المخزن المؤقت القابل لإعادة الطي إلى أنبوب مخصص سعة 1.5 مل ، وقم بتسخينه عند 37 درجة مئوية في جهاز حراري يوضع أسفل غطاء كيميائي.
ملاحظة: DMS شديد السمية ويجب أن يبقى دائما تحت غطاء كيميائي حتى يتم إخماده بواسطة عامل اختزال. - تمييع 1-10 pmol من الحمض النووي الريبي في 10 ميكرولتر من الماء الخالي من النيوكلياز (NF H2O) ؛ نقل إلى أنبوب PCR.
- احتضان في دراجة حرارية عند 95 درجة مئوية لمدة 1 دقيقة لتشويه الحمض النووي الريبي.
- ضعه على كتلة ثلج على الفور لتجنب الطي.
- أضف عينة الحمض النووي الريبي إلى الأنبوب المعين مع إعادة طي المخزن المؤقت عند 37 درجة مئوية ، واخلطها جيدا ، واحتضانها لمدة 10-20 دقيقة لإعادة تشكيل الحمض النووي الريبي.
ملاحظة: سيتم طي معظم الحمض النووي الريبي في حدود ميلي ثانية إلى ثانية ، على الرغم من وجود استثناءات16. - أضف 1 ميكرولتر من DMS بنسبة 100٪ (10.5 م) إلى عينة الحمض النووي الريبي ، واحتضانها لمدة 5 دقائق أثناء الاهتزاز بمعدل 800-1400 دورة في الدقيقة (دورة في الدقيقة).
ملاحظة: يعد الاهتزاز (أو أي وسيلة أخرى للخلط) في هذه الخطوة أمرا بالغ الأهمية لأن DMS كاره للماء وقد لا يذوب تماما في المخزن المؤقت لإعادة الطي. قد تؤثر الانحرافات في أوقات التفاعل على قابلية تكرار تفاعلات DMS. لتقليل خطأ السحب ، يمكن إذابة DMS في الإيثانول بنسبة 100٪ قبل إضافته إلى العينة إذا تم الحفاظ على تركيز نهائي بنسبة 1٪ (105 mM) DMS. بالنسبة للتحكم غير المعالج ، يمكن استبدال DMS بثنائي ميثيل سلفوكسيد (DMSO) أو الماء. - بعد 5 دقائق من وقت رد الفعل ، قم بإخماد 60 ميكرولتر من 100٪ β-mercaptoethanol (BME) ، واخلطه جيدا ، وضع الحمض النووي الريبي على الجليد على الفور.
ملاحظة: يمكن إزالة الحمض النووي الريبي بأمان من الغطاء بعد إخماد التفاعل مع BME لتنظيفه. ومع ذلك ، لا يزال يتعين تجنب التعرض المباشر ل BME للمحيط بسبب رائحته القوية وخصائصه المزعجة. - قم بتنظيف الحمض النووي الريبي عن طريق ترسيب أسيتات الصوديوم والإيثانول (انظر الخطوة 2.5) أو النهج القائم على العمود (انظر الخطوة 2.6) ، وقم بالتخلص منه في 10 ميكرولتر من الماء.
- تحديد كمية الحمض النووي الريبي باستخدام مقياس الطيف.
- الاستخدام المباشر لتخزين الحمض النووي الريبي المعدل عند -80 درجة مئوية.
ملاحظة: يجب تجنب التخزين طويل الأجل ، لأن الحمض النووي الريبي أقل استقرارا بعد علاج DMS.
- تحضير كمية كافية من العازلة القابلة لإعادة الطي (0.4 M كاكوديلات الصوديوم ، درجة الحموضة 7.2 ، تحتوي على 6 mM MgCl2).
- تفاعل البوليميراز المتسلسل RT-PCR الخاص بالجينات للحمض النووي الريبي المعدل
ملاحظة: انظر الشكل 1 لإعداد RT-PCR للشظايا المعالجة بنظام DMS.- قم بتخفيف 100 نانوغرام من الحمض النووي الريبي المعدل في 10 ميكرولتر من H 2 O. الخالي من النيوكلياز (NF) H2O. نقل إلى أنبوب PCR.
- إلى الأنبوب ، أضف 4 ميكرولتر من 5x المخزن المؤقت الأول (FSB) ، 1 ميكرولتر من مزيج dNTP (10 مللي متر لكل منهما) ، 1 ميكرولتر من 0.1 M ثنائي ثيوثريتول (DTT) (تجنب دورات التجميد والذوبان) ، 1 ميكرولتر من مثبط RNase ، 1 ميكرولتر من 10 ميكرومتر التمهيدي العكسي (التمهيدي الفردي أو مجموعة من البادئات) ، و 1 ميكرولتر من TGIRT III.
ملاحظة: بالنسبة لمجموعة من البادئات ، لا تضيف 1 ميكرولتر من 10 ميكرومتر من كل برايمر مباشرة إلى RT ؛ بدلا من ذلك ، امزج البادئات أولا ، وأضف 1 ميكرولتر من المزيج (بتركيز أولي إجمالي 10 ميكرومتر). - احتضان عند 57 درجة مئوية لمدة 30 دقيقة إلى 1.5 ساعة (عادة ، 30 دقيقة كافية لصنع منتج 500 nt) في دراجة حرارية.
- أضف 1 ميكرولتر من 4 M NaOH ، واخلطه عن طريق السحب ، واحتضانه عند 95 درجة مئوية لمدة 3 دقائق لتحلل الحمض النووي الريبي.
ملاحظة: هذه الخطوة حاسمة لأنها تطلق TGIRT من cDNA عن طريق تدهور الحمض النووي الريبي. إذا تم تخطيه ، فقد يتأثر PCR النهائي. - قم بالتنظيف باستخدام نهج قائم على العمود (انظر الخطوة 2.6) يزيل البادئات بشكل كاف ، ويزيل 10 ميكرولتر من NF H2O.
- PCR - تضخيم cDNA باستخدام 1 ميكرولتر من منتج النسخ العكسي لكل 25 ميكرولتر من التفاعل باستخدام مجموعة PCR المصممة لتحقيق التوازن بين العائد والدقة.
ملاحظة: يجب أن يكون للبادئات درجة حرارة انصهار ~ 60 °C. - قم بتشغيل 2 ميكرولتر من منتج PCR على هلام الأغاروز أو هلام الأغاروز مسبق الصب للتحقق من نجاح تفاعل البوليميراز المتسلسل.
- من الناحية المثالية ، يجب أن يظهر نطاق واحد فقط بعد PCR. إذا كان الأمر كذلك ، فقم بتنظيف التفاعل باستخدام نهج قائم على العمود. في حالة وجود نطاقات بديلة ، استخدم تفاعل PCR المتبقي لاستئصال الشريط الصحيح من الجل. تمييع في حجم صغير بما فيه الكفاية (على سبيل المثال ، 10 ميكرولتر).
- تحديد الشظايا المستخرجة باستخدام مقياس الطيف الضوئي.
- فهرسة شظايا dsDNA للتسلسل باستخدام نهج مناسب لمنصة التسلسل المطلوبة.
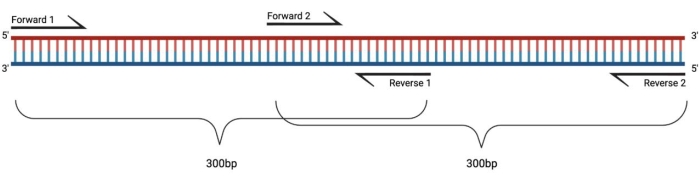
الشكل 1: الإعداد التجريبي لتفاعل البوليميراز المتسلسل RT-PCR للشظايا الكبيرة المعالجة بنظام DMS. عند إجراء النسخ العكسي على الحمض النووي الريبي المعدل ، لن يتم تسجيل التعديلات على التسلسل الذي يلدنه التمهيدي. وبالتالي ، عندما يتجاوز طول الشظايا 400-500 نقطة أساس ، يجب تصميم الأجزاء المتداخلة في مناطق التمهيدي ، كما هو موضح هنا. يعتمد طول الأجزاء على احتياجات التسلسل. عند استخدام تسلسل دورة 150 المزدوجة ، يجب ألا تتجاوز الأجزاء 300 نقطة أساس. الاختصارات: RT-PCR = تفاعل البوليميراز المتسلسل للنسخ العكسي ؛ DMS = ثنائي ميثيل كبريتات. الرجاء الضغط هنا لعرض نسخة أكبر من هذا الشكل.
2. الجينوم الكامل DMS-MaP باستخدام الخلايا المصابة بالفيروس
ملاحظة: في الخلايا ، يمكن أيضا دمج علاج DMS مع نهج التضخيم الخاص بالجينات الموصوف أعلاه. تتطلب مكتبة الجينوم الكاملة عمق تسلسل هائل لتحقيق تغطية كاملة لجين واحد. ومع ذلك ، إذا كان الحمض النووي الريبي الفيروسي يشكل جزءا كبيرا من الحمض النووي الريبي المنضب بعد الاستخراج ، فسيكون تسلسل الجينوم الكامل مناسبا. علاوة على ذلك ، يمكن دمج طرق التخصيب الأخرى مع طريقة توليد مكتبة الجينوم الكامل.
- علاج DMS
- تنمو الخلايا المصابة بالفيروس حتى المرحلة المطلوبة من الإصابة.
- نقل حاوية الخلية إلى غطاء دخان مخصص مناسب للتعامل مع كل من الفيروسات على مستوى السلامة البيولوجية المطلوب والأبخرة الكيميائية الناتجة عن عوامل مثل DMS.
- أضف حجما بنسبة 2.5٪ من DMS إلى وسط الاستزراع ، وأغلق الحاوية (عادة ما تكون لوحة 10 سم) باستخدام البارافيلم.
ملاحظة: من السهل إجراء تعديل ناقص والإفراط في التعديل باستخدام DMS. عند إضافة DMS مباشرة إلى الخلايا ، من المهم جدا الخلط جيدا. بدلا من ذلك ، قم بتسخين الوسط الجديد في أنبوب مخروطي سعة 50 مل عند 37 درجة مئوية ، وأضف DMS مباشرة يهتز بقوة. صب الوسط المستهلك على الخلايا ، وببطء ماصة في الوسط المحتوي على DMS. - انقله إلى حاضنة 37 درجة مئوية لمدة 5 دقائق.
ملاحظة: اعتمادا على مقدار الوقت المستغرق للتعامل مع DMS خارج الحاضنة ، من الممكن أن تؤدي 5 دقائق إلى الإفراط في التعديل. حافظ على الوقت من إضافة DMS إلى الحضانة إلى ≤1 دقيقة. في حالة إجراء التجربة لأول مرة، يوصى بإجراء معايرة DMS وتغيير وقت الحضانة (بين 3 دقائق و10 دقائق) للعثور على معدل التعديل الأمثل والتأكد من أن النتائج قوية عبر نافذة التركيزات. - قم بسحب الوسط المحتوي على DMS بعناية (في النفايات الكيميائية المناسبة) وأضف برفق 10 مل من المخزن المؤقت للإيقاف (PBS مع 30٪ BME [على سبيل المثال ، 3 مل من BME و 7 مل من PBS]).
ملاحظة: يمكن أن تؤدي إضافة DMS و BME إلى رفع الخلايا من اللوحة إذا لم تكن الخلايا ملتصقة بشدة. إذا كانت الخلايا ترفع ، فيمكن معالجتها على أنها خلايا معلقة - بدلا من إزالة الوسط المحتوي على DMS ، أضف المخزن المؤقت للتوقف مباشرة ، وكشط الخلايا باستخدام DMS و BME في أنبوب مخروطي سعة 50 مل. حبيبات الخلايا عن طريق الطرد المركزي لمدة 3 دقائق في 3000 × غرام ؛ تأكد من التخلص من أي DMS المتبقية ، والتي يمكن أن تتساقط تحت الخلايا في قطرات كبيرة. يوصى بخطوة غسيل إضافية في 30٪ BME إذا تعذر إزالة وسيط DMS في البداية. - كشط الخلايا ، ونقلها إلى أنبوب مخروطي 15 مل.
- بيليه بالطرد المركزي عند 3000 × جم لمدة 3 دقائق.
- قم بإزالة المادة الطافية واغسل 2x ب 10 مل من PBS.
- قم بإزالة أكبر قدر ممكن من برنامج تلفزيوني المتبقي بعناية.
- قم بإذابة الحبيبات بكمية مناسبة من كاشف عزل الحمض النووي الريبي (على سبيل المثال ، 3 مل لقارورة ثقافة T75 ، 1 مل للوحة 10 سم).
ملاحظة: قد تؤثر الكميات غير الكافية من الكاشف على عائد الحمض النووي الريبي.
- استخراج الحمض النووي الريبي واستنفاد الحمض النووي الريبوزي الريبوسوم (rRNA)
- إلى 1 مل من الخلايا المتجانسة في كاشف عزل الحمض النووي الريبي ، أضف 200 ميكرولتر من الكلوروفورم ، دوامة لمدة 15-20 ثانية حتى يصبح لونها ورديا ساطعا ، ثم احتضنها لمدة تصل إلى 3 دقائق حتى يصبح فصل الطور مرئيا.
ملاحظة: يجب أن تستقر مرحلة الدهون الوردية في القاع. إذا لم يكن الأمر كذلك ، فمن المحتمل أن يكون وقت الدوامة غير كاف. - تدور بأقصى سرعة (~ 20000 × جم) لمدة 15 دقيقة عند 4 درجات مئوية.
- نقل المرحلة المائية العليا إلى أنبوب جديد.
- قم بتنظيف الحمض النووي الريبي عن طريق ترسيب أسيتات الصوديوم والإيثانول (انظر الخطوة 2.5) أو نهج قائم على العمود (انظر الخطوة 2.6) ، وقم بالتخلص من حجم كاف من NF H2O.
- تحقق من سلامة الحمض النووي الريبي على هلام الأغاروز. ابحث عن شريطين يناظران الوحدتين الفرعيتين الريبوسومية.
- استنفاد rRNAs باستخدام النهج المفضل ، وتذوب في حجم مناسب (عادة 20-50 ميكرولتر) من NF H2O.
ملاحظة: بالنسبة للتطبيقات النهائية ، يقترح ~ 500 نانوغرام من إجمالي الحمض النووي الريبي بحجم 8 ميكرولتر. عادة ما تشكل الحمض النووي الريبي غير الريبوسومي 5٪ -10٪ فقط من إجمالي الحمض النووي الريبي. - تحديد كمي باستخدام مقياس الطيف الضوئي.
- إلى 1 مل من الخلايا المتجانسة في كاشف عزل الحمض النووي الريبي ، أضف 200 ميكرولتر من الكلوروفورم ، دوامة لمدة 15-20 ثانية حتى يصبح لونها ورديا ساطعا ، ثم احتضنها لمدة تصل إلى 3 دقائق حتى يصبح فصل الطور مرئيا.
- جيل المكتبة
- استخدم RT-PCR الخاص بالجينات أو طرق أخرى لإنشاء مكتبات15. في حالة استخدام السداسي العشوائي للتحضير ، أضف خطوة حضانة عند Tm منخفض (37-42 درجة مئوية) للسماح بالتلدين السداسي.
ملاحظة: يمكن أيضا استخدام مجموعات إنشاء المكتبة القياسية عن طريق استبدال إنزيم RT ب TGIRT وتغيير درجة حرارة RT إلى 57 درجة مئوية.
- استخدم RT-PCR الخاص بالجينات أو طرق أخرى لإنشاء مكتبات15. في حالة استخدام السداسي العشوائي للتحضير ، أضف خطوة حضانة عند Tm منخفض (37-42 درجة مئوية) للسماح بالتلدين السداسي.
- تنظيف الحمض النووي الريبي المستند إلى العمود باستخدام أعمدة تنظيف ومكثف الحمض النووي الريبي
ملاحظة: يجب إجراء جميع الخطوات في درجة حرارة الغرفة.- أضف NF H2O إلى أنبوب العينة ليصل حجمه إلى 50 ميكرولتر.
- أضف 100 ميكرولتر من المخزن المؤقت للربط و 150 ميكرولتر من الإيثانول بنسبة 100٪ إلى العينة.
- امزج وانقل إلى عمود الدوران.
- تدور في 10000-16000 × غرام لمدة 30 ثانية ؛ تجاهل التدفق.
- أضف 400 ميكرولتر من المخزن المؤقت لإعداد الحمض النووي الريبي.
- تدور في 10000-16000 × غرام لمدة 30 ثانية ؛ تجاهل التدفق.
- أضف 700 ميكرولتر من محلول غسيل الحمض النووي الريبي.
- تدور في 10000-16000 × غرام لمدة 30 ثانية ؛ تجاهل التدفق.
- أضف 400 ميكرولتر من محلول غسيل الحمض النووي الريبي.
- تدور في 10000-16000 × غرام لمدة 30 ثانية ؛ تجاهل التدفق.
- (اختياري) انقل العمود إلى أنبوب تجميع جديد ، وقم بتدويره بسرعة 10000-16000 × جم لمدة 2 دقيقة.
- انقل العمود إلى أنبوب نظيف خال من الحمض النووي الريبي وأضف كمية مناسبة من NF H2O.
- تدور في 10000-16000 × غرام لمدة 1 دقيقة.
- استخراج الحمض النووي الريبي الفينول كلوروفورم.
- إضافة كمية متساوية من حمض الفينول:الكلوروفورم:كحول إيزو أميل.
- دوامة تماما ، وأجهزة الطرد المركزي في 14000 × غرام لمدة 5 دقائق.
- إذا لم يكن هناك فصل طور ، أضف 20 ميكرولتر من 2 M NaCl ، وكرر الطرد المركزي.
- نقل المرحلة المائية إلى أنبوب جديد.
- أضف 500 ميكرولتر من الأيزوبروبانول و 2 ميكرولتر من المرسب المشترك.
- تخلط وتحتضن في RT لمدة 3 دقائق ؛ ثم احتضن عند -80 درجة مئوية بين عشية وضحاها.
- قم بتكوير الحمض النووي الريبي بالطرد المركزي بأقصى سرعة (~ 20000 × جم) لمدة 30 دقيقة عند 4 درجات مئوية.
- اغسل الحبيبات ب 200 ميكرولتر من الإيثانول المثلج 70٪.
- تدور بأقصى سرعة (~ 20000 × جم) لمدة 5 دقائق ؛ تجاهل التدفق.
- أعد تعليق الحبيبات بالكمية المناسبة من NF H2O.
- تنظيف cDNA المستند إلى العمود باستخدام أعمدة Oligo Clean و Concentrator
ملاحظة: يجب إجراء جميع الخطوات في درجة حرارة الغرفة.- أضف NF H2O إلى أنبوب العينة ليصل حجمه إلى 50 ميكرولتر.
- أضف 100 ميكرولتر من المخزن المؤقت للربط و 400 ميكرولتر من الإيثانول بنسبة 100٪.
- امزج وانقل إلى عمود الدوران.
- تدور في 10000-16000 × غرام لمدة 30 ثانية ؛ تجاهل التدفق.
- أضف 750 ميكرولتر من محلول غسل الحمض النووي.
- تدور في 10000-16000 × غرام لمدة 30 ثانية ؛ تجاهل التدفق.
- (اختياري) انقل العمود إلى أنبوب تجميع جديد وقم بتدويره بسرعة 10000-16000 × جم لمدة 2 دقيقة.
- انقل العمود إلى أنبوب نظيف خال من الحمض النووي الريبي ، وأضف كمية مناسبة من NF H2O.
- تدور في 10000-16000 × غرام لمدة 1 دقيقة.
3. تحليل بيانات التسلسل
ملاحظة: لإنشاء نماذج بنية ثانوية للحمض النووي الريبي من بيانات تسلسل DMS-MaP ، يجب معالجة ملفات .fastq الناتجة بعدة خطوات مختلفة. يمكن تنفيذ هذه الخطوات تلقائيا باستخدام
- قم بقص تسلسلات المحول باستخدام TrimGalore أو Cutadapt.
- قم بتعيين القراءات إلى التسلسلات المرجعية (تنسيق .fasta) باستخدام Bowtie2.
- عد القراءات باستخدام برنامج بنية RNA متخصص (على سبيل المثال ، DREEM14 أو RNA-Framework17 أو ما شابه) ، وقم بإنشاء ملفات تعريف تفاعلية.
- (اختياري) قم بتجميع القراءات للعثور على توافقات RNA بديلة باستخدام DREEM14 أو DRACO 17 أو DANCE-MaP18 أو ما شابه ذلك.
- توقع الحد الأدنى من بنية الطاقة الحرة بناء على ملفات تعريف التفاعل باستخدام RNAStructure12 أو ViennaRNA أو ما شابه ذلك.
- تصور بنية RNA11 باستخدام VARNA (https://varna.lri.fr/) أو ما شابه ذلك.
ملاحظة: من الناحية العملية ، تتضمن برامج مثل DREEM (www.rnadreem.org) و RNA-Framework19 إلى حد كبير الخطوات 1--5 في خطوط الأنابيب الخاصة بها ، مما يبسط عملية التحليل. ومع ذلك ، يجب التعامل مع أي تنبؤ بالهيكل بعناية (على سبيل المثال ، من خلال التحقق من اتفاق الهيكل مع البيانات20.
النتائج
الجينات الخاصة في المختبر DMS-MaP
لدراسة 5'UTR من SARS2 ، تم طلب أول 300 نقطة أساس للفيروس كتسلسل gBlock ، إلى جانب ثلاثة بادئات. وشمل ذلك اثنين من البادئات لنشر الجزء ("FW" و "RV") عبر PCR ، بالإضافة إلى واحد لربط المروج T7 ("FW-T7"). يمكن رؤية هذه التسلسلات في الجدول 1.
| اسم | التسلسل (5'->3') |
| مهاجم | ATTAAAGGTTTATACCTTCCCAGGTAAC |
| آر في | GCAAACTGAGTTGGACGTGT |
| مهاجم-T7 | TAATACGACTCACTATAGG ATTAAAGGTTTATACCTTCCCAGGTAAC |
الجدول 1: تسلسل التمهيدي ل DMS-MaP RT-PCR ل SARS-CoV2 5'UTR. هنا ، هناك حاجة إلى FW-T7 و RV لإنشاء قالب DNA للنسخ في المختبر ، ويتم استخدام RV في النسخ العكسي ويتم استخدام زوج التمهيدي FW-RV في تضخيم PCR اللاحق ل cDNA. تلتصق البادئات ببداية جينوم SARS-CoV2 (FW) والتسلسل في اتجاه مجرى المنطقة محل الاهتمام. الاختصارات: DMS-MaP = التنميط الطفري مع التسلسل باستخدام كبريتات ثنائي ميثيل ؛ RT-PCR = تفاعل البوليميراز المتسلسل للنسخ العكسي ؛ SARS-CoV2 = المتلازمة التنفسية الحادة الوخيمة - فيروس كورونا 2 ؛ UTR = منطقة غير مترجمة; RV = التمهيدي العكسي ؛ FW = التمهيدي إلى الأمام.
لتوليد الحمض النووي الريبي من جزء gBlock ، تم إرفاق تسلسل مروج البوليميراز T7 باستخدام تفاعل البوليميراز المتسلسل المتداخل باستخدام مزيج PCR المسبق وفقا للمخطط الموضح في الشكل 2A. من الجزء الممدود ، تم إنشاء الحمض النووي الريبي باستخدام مجموعة النسخ T7. تم هضم قالب الحمض النووي لاحقا باستخدام DNase و RNA المعزول باستخدام أعمدة RNA Clean و Concentrator.
تم إجراء مراقبة جودة النسخ في المختبر عن طريق تشغيل منتج الحمض النووي الريبي على هلام أغاروز 1٪ جنبا إلى جنب مع سلم ssRNA. نظرا لوجود نطاق واحد فقط مرئي ، تم إجراء فحص DMS في المختبر و RT-PCR (انظر الشكل 2B).
للتحقق من نجاح تفاعل تفاعل البوليميراز المتسلسل ، تم تشغيل العينة على هلام الأغاروز 2٪ باستخدام سلم dsDNA. بعد الفهرسة ، يجب تشغيل النطاق ~ 150 نقطة أساس أعلى على نفس الهلام ، وهو ما يمثل حجم بادئات الفهرسة.
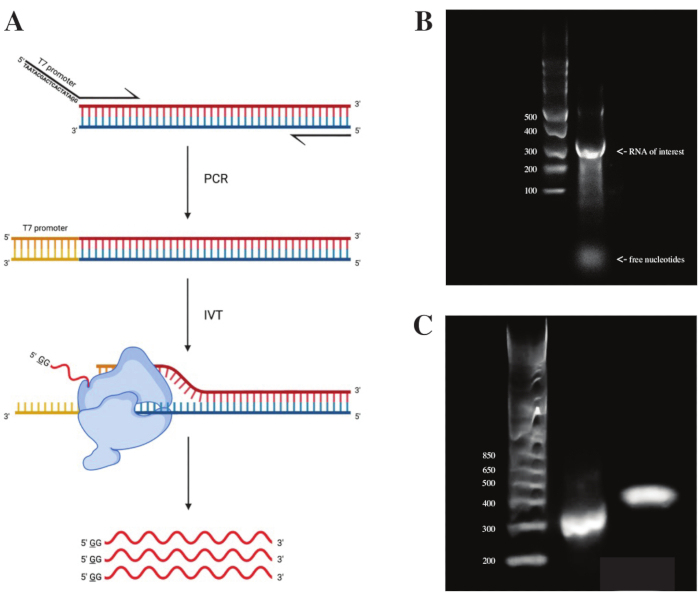
الشكل 2: النسخ المختبري لقالب الحمض النووي (DNA) . (أ) لنسخ قالب الحمض النووي في المختبر الذي لا يحتوي بعد على محفز بوليميراز الحمض النووي الريبي الجوهري ، يجب إرفاق القالب بواسطة تفاعل البوليميراز المتسلسل المتداخل أولا. يتم ذلك باستخدام التمهيدي الأمامي ، والذي يتضمن تسلسل TAATACGACTCACTATAGG (في حالة بوليميراز T7 RNA) المنبع من القواعد الأولى المتداخلة مع الجزء المطلوب. ترمز القاعدة التي تحتها خط هنا إلى موقع بدء النسخ للبوليميراز. بمجرد أن يعلق المروج على جزء dsDNA ، يمكن نسخه بواسطة بوليميراز T7. الأهم من ذلك ، يستخدم البلمرة الشريط المقابل لتسلسل المروج المذكور كقالب (أزرق) ، مما يخلق بشكل فعال الحمض النووي الريبي مطابقا للتسلسل مباشرة في اتجاه مجرى تسلسل المروج المشار إليه (أحمر). (ب) جل أغاروز 1٪ مع سلم ssRNA (الممر 1) ومنتج الحمض النووي الريبي المنسوخ في المختبر عند 300 nt (الممر 2). (ج) جل أغاروز 2٪ مع GeneRuler 1 كيلو بايت بالإضافة إلى سلم (حارة 1) ، ومنتج PCR بعد RT-PCR يعمل بسرعة 300 نقطة أساس (الممر 2) ، والجزء المفهرس بعد إعداد المكتبة يعمل بسرعة 470 نقطة أساس (الممر 3). الاختصارات: RT-PCR = تفاعل البوليميراز المتسلسل للنسخ العكسي ؛ DMS = كبريتات ثنائي ميثيل ؛ nt = النيوكليوتيدات ؛ dsDNA = الحمض النووي المزدوج الذين تقطعت بهم السبل ؛ ssRNA = الحمض النووي الريبي واحد الذين تقطعت بهم السبل. الرجاء الضغط هنا لعرض نسخة أكبر من هذا الشكل.
الجينوم الكامل في الجسم الحي DMS-MaP باستخدام الخلايا المصابة بالفيروس
قبل علاج DMS ، أصيبت خلايا HCT-8 ب OC43. عندما لوحظ تأثير الاعتلال الخلوي (CPE) بعد 4 أيام من الإصابة (dpi) (كما هو موضح في الشكل 3A) ، تمت معالجة هذه الخلايا ، وتم استخراج الحمض النووي الريبي واستنفاده. عند تشغيل إجمالي الحمض النووي الريبي على هلام الأغاروز ، كان شريطان ساطعان مرئيين ، يمثلان الوحدات الفرعية 40S و 60S من الريبوسوم ، والتي تشكل حوالي 95٪ من إجمالي كتلة الحمض النووي الريبي (انظر الشكل 3B). عندما كان استخراج الحمض النووي الريبي غير ناجح أو تدهور (على سبيل المثال ، من خلال دورات التجميد والذوبان المتعددة) ، كانت منتجات تحلل الحمض النووي الريبي مرئية في الجزء السفلي من الجل (انظر الشكل 3C ، الممر الثاني). علاوة على ذلك ، بعد استنفاد rRNA ، اختفى الشريطان الساطعان ، تاركين مسحة في الممر (انظر الشكل 3C ، الممر الثالث). أخيرا ، بعد إعداد المكتبة ، كان للعينات توزيعات متفاوتة الحجم وتم عرضها كمسحة على هلام PAGE النهائي. تم استئصال النطاق بين 200 نيوكليوتيد (nt) و 500 nt ، بالاتفاق مع تسلسل النهاية المزدوجة 150 × 150 المخطط لتحليل هذه المكتبات. الأهم من ذلك ، تم فصل ثنائيات المحول التي تعمل عند ~ 150 nt (انظر الشكل 3D).
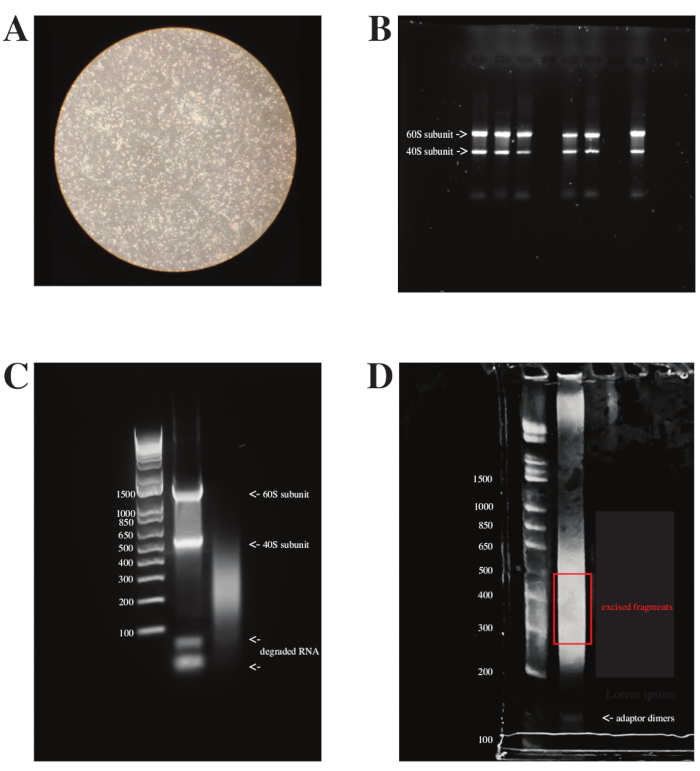
الشكل 3: نقاط التفتيش في الجسم الحي DMS-MaP مع الخلايا المصابة بالفيروس . (أ) صورة مجهرية ضوئية لخلايا HCT-8 المصابة بالفيروس ، 4 أيام نقطة في البوصة. للحصول على أعلى عائد ممكن من الحمض النووي الريبي الفيروسي من إجمالي الحمض النووي الريبي مع تقليل الآثار الضارة بسبب موت الخلايا ، يجب إضافة DMS عند بدء CPE أو حتى قبل ذلك ، كما هو موضح في الصورة. (ب) جل أغاروز 1٪ مع ست عينات من 1 ميكروغرام من الحمض النووي الريبي الكلي. في كل حارة ، يظهر شريطان ساطعان ، يمثلان الوحدات الفرعية 40S و 60S ، حيث يشكل الحمض النووي الريبي الريبوسومي ~ 95٪ من إجمالي الحمض النووي الريبي. ملاحظة: يتسبب علاج DMS داخل الخلية في بعض تجزئة الحمض النووي الريبي وتلطيخه ، ولكن يجب أن يظل نطاقي rRNA مرئيين. يتم التسامح مع التعديل اللاحق للتجزئة الخفيفة لأن المعلومات التي تحتوي على علامة المثيلة يتم إنشاؤها وتقارير عن بنية الحمض النووي الريبي أثناء حضانة DMS بينما لا تزال الخلايا على قيد الحياة. (ج) جل أغاروز 1٪ من GeneRuler 1 كيلو بايت بالإضافة إلى علامة الحمض النووي للسلم (الممر 1) إجمالي الحمض النووي الريبي المخزن مسبقا عند -80 درجة مئوية لمدة 6 أشهر (الممر 2) والحمض النووي الريبي المنضب (الممر 3). عند تخزين الحمض النووي الريبي لفترة طويلة مع عدة دورات تجميد وذوبان الجليد ، يبدأ الحمض النووي الريبي في التدهور وربما لا ينبغي استخدامه في تجارب التحقيق. علاوة على ذلك ، بعد استنفاد إجمالي الحمض النووي الريبي ، يتلاشى النطاقان الساطعان ، اللذان يمثلان الوحدات الفرعية 40S و 60S من الريبوسوم ، وتبدأ مسحة الحمض النووي الريبي المتبقي في الظهور. (د) هلام PAGE من GeneRuler 1 كيلو بايت بالإضافة إلى علامة الحمض النووي للسلم (الممر 1) وعينة مكتبة من الحمض النووي الريبي المحضر بالجينوم الكامل. يجب استئصال الجل بناء على احتياجات التسلسل. للحصول على تسلسل مزدوج النهاية يمتد 150 دورة من كلا الجانبين ، يجب استئصال الجل بين 300 نقطة أساس و 500 نقطة أساس. يجب فصل دايمرات المحول (التي تعمل عند 170 نقطة أساس). الاختصارات: DMS-MaP = التنميط الطفري مع التسلسل باستخدام كبريتات ثنائي ميثيل ؛ نقطة في البوصة = أيام بعد الإصابة ؛ CPE = تأثير الاعتلال الخلوي. الرجاء الضغط هنا لعرض نسخة أكبر من هذا الشكل.
بعد التسلسل ، تم تحليل ملفات .fastq عن طريق إرسال وظيفة إلى خادم الويب DREEM (http://rnadreem.org/) ، جنبا إلى جنب مع ملف مرجعي .fasta. يتضمن الناتج الناتج الذي تم إنشاؤه بواسطة الخادم ملفات مراقبة الجودة التي تم إنشاؤها بواسطة fastqc (https://www.bioinformatics.babraham.ac.uk/projects/fastqc/) و TrimGalore (https://www.bioinformatics.babraham.ac.uk/projects/trim_galore/) ، بالإضافة إلى ملفات الإخراج الأخرى التي تحتوي على ترددات طفرة متوسط السكان. بصرف النظر عن الرسم البياني الذي يوضح ترددات الطفرات بتنسيق .html تفاعلي (انظر الشكل 4 أ) وملف .csv مع عمليات إعادة النشاط الخام لكل قاعدة وملف struct_constraint.txt ، يمكن قراءته بواسطة العديد من برامج التنبؤ ببنية الحمض النووي الريبي ، يتضمن هذا أيضا ملف متجه بت.txt يبلغ عن الطفرات العرضية. من هذه ، تم حساب هياكل متوسط السكان عن طريق إرسال ملفات .fasta و struct_constraint.txt إلى خادم الويب RNAfold (http://rna.tbi.univie.ac.at/cgi-bin/RNAWebSuite/RNAfold.cgi). يستخدم هذا برنامج ViennaRNA لتوليد تنبؤات هيكلية تستند إلى الحد الأدنى من الطاقة المجانية ، والتي يمكن عرضها عبر الإنترنت أو تنزيلها بتنسيق ct أو فيينا. لإنشاء نماذج بنية الحمض النووي الريبي ، تم إرسال هذه الملفات القابلة للتنزيل إلى VARNA (https://varna.lri.fr/ ، انظر الشكل 4 ب). أخيرا ، يمكن استخدام ملفات bitvector.txt بواسطة الإصدار الثابت من DREEM (https://codeocean.com/capsule/6175523/tree/v1) للبحث عن توافقات RNA بديلة. للحصول على نماذج بنية جيدة باستخدام DREEM ، يجب تحقيق تغطية 10000 قراءة لكل قاعدة ؛ للتجميع ، قد تكون هناك حاجة إلى ما يصل إلى 100000 قراءة لكل قاعدة. يمكن العثور على نظرة عامة على سير العمل بأكمله في الشكل 4C.
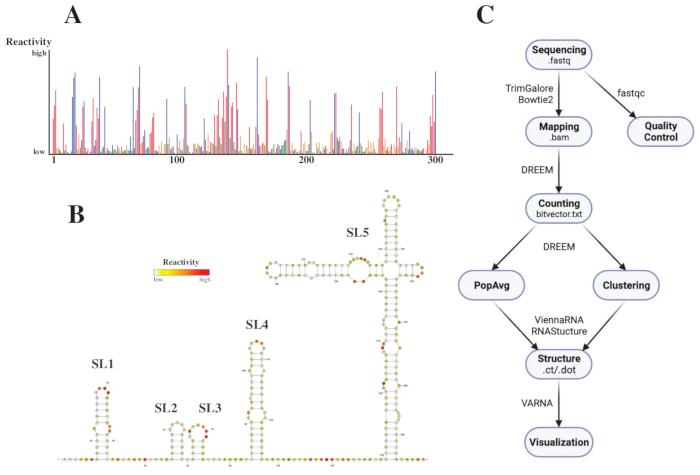
الشكل 4: بيانات نموذجية تم الحصول عليها من تجارب الفحص الكيميائي ل SARS-CoV2 5'UTR. (أ) ملف تعريف تفاعلي لأول 300 قاعدة من جينوم SARS-CoV2 ملونة بالقاعدة (A: أحمر ، C: أزرق ، U: أخضر ، G: أصفر). يتم حساب التفاعلات الخام على أنها تردد الطفرة المطلق مقسوما على التغطية. القواعد ذات التشكل المفتوح لها قيم تفاعلية عالية ؛ القواعد التي تشارك في إقران القواعد لها قيم تفاعلية منخفضة. لا يتم تعديل U و G بواسطة DMS ولهما قيم تفاعلية منخفضة ، ناشئة عن خيانة البلمرة. تم إجراء التنبؤات باستخدام خادم الويب DREEM. (ب) نموذج هيكل SARS-CoV2 5'UTR المتوقع من قيم التفاعل المصنوعة من VARNA. القواعد ذات قيم التفاعل العالية ملونة باللون الأحمر ؛ القواعد ذات قيم التفاعل المنخفضة ملونة باللون الأبيض. (ج) سير عمل تحليل DMS-MaP بدءا من ملفات .fastq التي تم الحصول عليها من التسلسل. يمكن التحكم في الجودة باستخدام fastqc ؛ يتم قطع تسلسلات المحول باستخدام TrimGalore ثم يتم تعيينها مرة أخرى إلى تسلسل مرجعي باستخدام Bowtie2. من ملفات .bam التي تم الحصول عليها ، يقوم DREEM بحساب الطفرات في كل قراءة ، وإنشاء خريطة طفرة أو ملف .bitvector.txt. تقوم هذه التقارير بالإبلاغ عن طفرات كل قراءة بطريقة تعتمد على الموضع ، والتي يمكن بناء عليها إنشاء ملفات تعريف تفاعل متوسط السكان. بدلا من ذلك ، يمكن تجميع متجهات البتات باستخدام DREEM للبحث عن توافقات RNA بديلة. أخيرا ، يتم تصور نماذج الهيكل التي تم الحصول عليها باستخدام البرامج (على سبيل المثال ، VARNA). الاختصارات: DMS-MaP = التنميط الطفري مع التسلسل باستخدام كبريتات ثنائي ميثيل ؛ SARS-CoV2 = المتلازمة التنفسية الحادة الوخيمة - فيروس كورونا 2. الرجاء الضغط هنا لعرض نسخة أكبر من هذا الشكل.
Discussion
يصف البروتوكول هنا كيفية فحص الحمض النووي الريبي في المختبر وفي الخلايا باستخدام تجارب التنميط الطفري DMS. علاوة على ذلك ، فإنه يعطي تعليمات حول كيفية إعداد المكتبات لتسلسل Illumina لإنشاء بيانات خاصة بالجينات وتحليل ملفات .fastq التي تم الحصول عليها. بالإضافة إلى ذلك ، يمكن استخدام أساليب المكتبة على مستوى الجينوم. ومع ذلك ، فإن RT-PCR الخاص بالجينات ينتج أعلى جودة وأقوى البيانات. لذلك ، في حالة المقارنة بين العينات ، من المهم التأكد من إعدادها باستراتيجيات تسلسل متطابقة ، لأن توليد المكتبة يسبب بعض التحيز. يجب دائما قياس قابلية التكرار باستخدام النسخ المتماثلة.
عدة احتياطات
الحمض النووي الريبي هو جزيء غير مستقر حساس للتدهور من خلال درجات الحرارة المرتفعة و RNases. لذلك ، يوصى باتخاذ تدابير خاصة - استخدام معدات الحماية الشخصية (PPE) ، والمواد الخالية من الحمض النووي الريبي ، ومثبطات الحمض النووي الريبي. الأهم من ذلك ، يجب أن تبقى الحمض النووي الريبي على الجليد كلما أمكن ذلك. هذا ينطبق بشكل خاص على الحمض النووي الريبي الميثيلي ، وهو أكثر حساسية لدرجات الحرارة المرتفعة.
من المهم التأكد من أن بنية الحمض النووي الريبي ذات الأهمية ليست حساسة لتركيز DMS والظروف العازلة. تعطي المخازن المؤقتة مثل 100 mM Tris و 100 mM MOPS و 100 mM HEPES عند درجة الحموضة 7-7.5 إشارة عالية ولكنها قد لا تكون كافية للحفاظ على الرقم الهيدروجيني أثناء التفاعل21. نظرا لأن DMS يتحلل بالماء ، مما يقلل من درجة الحموضة ، فإن المخزن المؤقت القوي أمر بالغ الأهمية للحفاظ على درجة حموضة محايدة أثناء تفاعل التعديل. لقد ثبت أن إضافة البيسين تساعد في الحفاظ على الرقم الهيدروجيني كأساسيقليلا 21 ولكنها تؤدي إلى تعديل DMS منخفض على Gs و Us ، والذي يمكن أن يكون مفيدا ولكن يجب تحليله بشكل منفصل بسبب إنتاج إشارة أقل بكثير من As و Cs ولم تتم مناقشته بشكل أكبر في هذا البروتوكول.
في RT-PCR الخاص بالجينات ، يتم نسخ الحمض النووي الريبي المعدل بشكل عكسي إلى الحمض النووي وتضخيمه في أجزاء بواسطة تفاعل البوليميراز المتسلسل. في حين أن حجم الحمض النووي الريبي يمكن أن يكون نظريا غير محدود ، يجب ألا تتجاوز شظايا تفاعل البوليميراز المتسلسل هذه طول 400-500 زوج أساسي (bp) لمنع التحيز أثناء تفاعل النسخ العكسي. من الناحية المثالية ، يجب أن تكون الأجزاء ضمن نطاق تشغيل التسلسل (على سبيل المثال ، إذا تم إجراء التسلسل باستخدام برنامج تسلسل نهاية مزدوجة 150 × 150 دورة ، يجب ألا يتجاوز الجزء الواحد 300 نقطة أساس). عند استخدام برامج التسلسل مع دورات أقل ، يمكن تجزئة منتجات PCR باستخدام dsDNase. علاوة على ذلك ، نظرا لأن التسلسلات داخل تسلسلات التمهيدي لا تحتوي على أي معلومات هيكلية ، يجب أن تتداخل الأجزاء عندما يتكون الحمض النووي الريبي الذي تم فحصه من جزء >1. يمكن أن تحتوي تفاعلات RT على بادئات RT متعددة لأجزاء مختلفة (حتى 10 بادئات RT مختلفة). اعتمادا على التسلسلات ، يمكن أن يؤدي تجميع بادئات RT إلى جعل النسخ العكسي أقل كفاءة ولكنه يعمل بشكل جيد عادة. يجب إجراء كل تفاعل PCR بشكل منفصل.
عند فحص الحمض النووي الريبي باستخدام DMS ، تلعب الظروف التجريبية دورا إضافيا ، حيث أن العديد من الحمض النووي الريبي غير مستقر ديناميكيا حراريا ويغير شكله بناء على عوامل بيئية مثل درجة الحرارة. لتجنب المخالفات ، يجب أن تظل الظروف التجريبية ثابتة قدر الإمكان ، وكذلك فيما يتعلق بأوقات رد الفعل. يبدو أن الظروف العازلة قابلة للتبادل إلى درجة معينة17،20،22،23 عندما يتم الحفاظ على الشروط الأساسية - قدرة التخزين المؤقت ووجود أيونات أحادية التكافؤ (Na) وأيونات ثنائية التكافؤ (Mg) - لضمان الطي السليم للحمض النووي الريبي 24.
فيما يتعلق بإعداد المكتبة للحمض النووي الريبي المعدل ، يجب مراعاة عدة جوانب. أولا ، كما ذكرنا من قبل ، فإن الحمض النووي الريبي المعدل أقل استقرارا من نظيراته غير المعدلة ، مما يعني أنه قد يتطلب تحسين أوقات التجزئة للتوزيع الأمثل لحجم الشظايا. علاوة على ذلك ، تستخدم بعض مجموعات إعداد مكتبة RNA ، بالإضافة إلى العديد من أساليب RNAseq الأخرى ، مواد أولية عشوائية في مجموعة النسخ العكسي. قد يؤدي هذا إلى انخفاض تغطية المرجع ، خاصة في 3 'من الجين ، وفي النهاية ، إلى عمق تغطية غير كاف. إذا كانت تغطية منطقة معينة منخفضة للغاية ، فقد يكون من الضروري إزالة هذه القواعد من التنبؤ بالهيكل. بصرف النظر عن RT-PCR ومجموعات RNAseq للجينوم الكامل ، يمكن استخدام طرق إعداد المكتبة الأخرى. تعتبر البروتوكولات التي تتضمن ربط محولات 3 'و / أو 5' بالحمض النووي الريبي مفيدة عند استخدام أجزاء صغيرة من الحمض النووي الريبي أو عندما يجب تجنب فقدان معلومات الفحص في مناطق التمهيدي.
أخيرا ، يجب دائما تفسير تحليل تجارب الفحص الكيميائي بعناية. حاليا ، لا يوجد برنامج يتنبأ ببنية الحمض النووي الريبي لأي RNA من التسلسل وحده بدقة عالية. على الرغم من أن قيود الفحص الكيميائي تحسن الدقة بشكل كبير ، إلا أن توليد نماذج جيدة للحمض النووي الريبي الطويل (>500 نانومتر) لا يزال يمثل تحديا. يجب اختبار هذه النماذج بشكل أكبر من خلال طرق و / أو طفرات أخرى. يعمل برنامج التنبؤ بالحمض النووي الريبي على تحسين الحد الأقصى لعدد أزواج القواعد ، وبالتالي معاقبة التوافقات المفتوحة بشكل كبير ، والتي قد لا تمثل بدقة طي الحمض النووي الريبي5. وبالتالي ، يجب اختبار نموذج البنية الذي تم الحصول عليه عن طريق تحديد اتفاق التنبؤ الكمي مع بيانات التحقيق الكيميائي الأساسية (على سبيل المثال ، بواسطة AUROC) وبين النسخ المتماثلة (على سبيل المثال ، بواسطة mFMI) ، كما يتضح من Lan et al.20.
من الناحية المثالية ، يجب استخدام العديد من التجارب في أنظمة مختلفة لتحدي نموذج الهيكل الذي تم الحصول عليه لتعزيز فرضية المرء. يمكن أن يشمل ذلك استخدام الأساليب في المختبر وفي الخلية ، والطفرات التعويضية ، وخطوط الخلايا والأنواع المختلفة. علاوة على ذلك ، غالبا ما تكون التفاعلات الخام بنفس القدر أو حتى أكثر إفادة من تنبؤات الهيكل ، لأنها تسجل لقطة "الحقيقة الأرضية" لمجموعة طي الحمض النووي الريبي. على هذا النحو ، فإن التفاعلات الأولية مناسبة جدا وغنية بالمعلومات لمقارنة تغيرات الهيكل بين الظروف المختلفة. الأهم من ذلك ، يجب استخدام أقل هياكل الطاقة الحرة المحسوبة باستخدام قيود التحقيق الكيميائي مع التنبؤ الحسابي فقط كفرضية بداية نحو نموذج بنية كاملة.
Disclosures
ليس لدى المؤلفين أي تضارب في المصالح يعلنونه.
Acknowledgements
اي
Materials
| Name | Company | Catalog Number | Comments |
| 1 Kb Plus DNA Ladder | 10787018 | Thermo | |
| 2-mercaptoethanol | M6250-250ML | Sigma | |
| Acid-Phenol:Chloroform, pH 4.5 | AM9720 | Thermo | |
| Advantage PCR | 639206 | Takara | |
| CloneAmp HiFi PCR Premix | 639298 | Takara | |
| DMS | D186309 | Sigma | |
| dNTPs 10 mM each | U151B | Promega | |
| E-Gel EX Agarose Gels, 2% | G402022 | Thermo | precast agarose gels |
| Ethanol (200 proof) | E7023-4X4L | Sigma | |
| Falcon tubes, 15 mL, 50 mL | |||
| GlycoBlue | co-precipitant | ||
| HCT-8 cells | ATCC #CCL-244 | ||
| Invitrogen MgCl2 (1 M) | AM9530G | fisherscientific | |
| Isopropanol | 278475 | Sigma | |
| Megascript T7 transcription | AM1334 | Thermo | |
| NanoDrop spectrophotometer | |||
| Novex TBE Gels, 8%, 10 well | EC6215BOX | Thermo | |
| OC43 | ATCC #VR-1558 | ||
| RiboRuler Low Range RNA Ladder | SM1831 | Thermo | |
| RNAse H | M0297L | NEB | |
| Sodium Cacodylate, 0.4 M, pH 7.2 | 102090-964 | VWR | |
| Sodium hydroxide solution | S8263-150ML | Sigma | |
| SuperScript II Reverse Transcriptase for FSB and DTT | 18064014 | Thermo | |
| TGIRT-III Enzyme | TGIRT50 | Ingex | |
| The Oligo Clean & Concentrator | D4060 | Genesee | |
| The RNA Clean & Concentrator kits are RNA clean up kits | R1016 | Genesee | |
| TRIzol Reagents | 15596018 | Thermo | RNA isolation reagent |
| Water, (For RNA Work) (DEPC-Treated, DNASE, RNASE free/Mol. Biol.) | BP561-1 | fisherscientific | |
| xGen Broad-range RNA Library Prep 16rxn | 10009865 | IDT | |
| Zymo RNA clean and concentrator columns |
References
- Kim, S. H., et al. Three-dimensional tertiary structure of yeast phenylalanine transfer RNA. Science. 185 (4149), 435-440 (1974).
- Robertus, J. D., et al. Structure of yeast phenylalanine tRNA at 3 Å resolution. Nature. 250 (467), 546-551 (1974).
- Zaug, A. J., Cech, T. R. In vitro splicing of the ribosomal RNA precursor in nuclei of Tetrahymena. Cell. 19 (2), 331-338 (1980).
- Zhao, Y., et al. NONCODE 2016: An informative and valuable data source of long non-coding RNAs. Nucleic Acids Research. 44, D203-D208 (2016).
- Vandivier, L. E., Anderson, S. J., Foley, S. W., Gregory, B. D. The conservation and function of RNA secondary structure in plants. Annual Review of Plant Biology. 67, 463(2016).
- Jumper, J., et al. Highly accurate protein structure prediction with AlphaFold. Nature. 596 (7873), 583-589 (2021).
- Das, R. RNA structure: A renaissance begins. Nature Methods. 18 (5), 439-439 (2021).
- Smola, M. J., Rice, G. M., Busan, S., Siegfried, N. A., Weeks, K. M. Selective 2′-hydroxyl acylation analyzed by primer extension and mutational profiling (SHAPE-MaP) for direct, versatile and accurate RNA structure analysis. Nature Protocols. 10 (11), 1643-1669 (2015).
- Mathews, D. H., et al. Incorporating chemical modification constraints into a dynamic programming algorithm for prediction of RNA secondary structure. Proceedings of the National Academy of Sciences of the United States of America. 101 (19), 7287-7292 (2004).
- Zuker, M., Stiegler, P. Optimal computer folding of large RNA sequences using thermodynamics and auxiliary information. Nucleic Acids Research. 9 (1), 133-148 (1981).
- Lorenz, R., et al. ViennaRNA Package 2.0. Algorithms for Molecular Biology. 6, (2011).
- Reuter, J. S., Mathews, D. H. RNAstructure: Software for RNA secondary structure prediction and analysis. BMC Bioinformatics. 11, (2010).
- Wells, S. E., Hughes, J. M. X., Igel, A. H., Ares, M. Use of dimethyl sulfate to probe RNA structure in vivo. Methods in Enzymology. , 479-493 (2000).
- Tomezsko, P. J., et al. Determination of RNA structural diversity and its role in HIV-1 RNA splicing. Nature. 582 (7812), (2020).
- Zubradt, M., et al. DMS-MaPseq for genome-wide or targeted RNA structure probing in vivo. Nature Methods. 14 (1), (2017).
- Woodson, S. A. Compact intermediates in RNA folding. Annual Reviews in Biophysics. 39, (2010).
- Morandi, E., et al. Genome-scale deconvolution of RNA structure ensembles. Nature Methods. 18 (3), 249-252 (2021).
- Olson, S. W., et al. Discovery of a large-scale, cell-state-responsive allosteric switch in the 7SK RNA using DANCE-MaP. Molecular Cell. 82 (9), 1708-1723 (2022).
- Incarnato, D., Morandi, E., Simon, L. M., Oliviero, S. RNA Framework: An all-in-one toolkit for the analysis of RNA structures and post-transcriptional modifications. Nucleic Acids Research. 46 (16), (2018).
- Lan, T. C. T., et al. Secondary structural ensembles of the SARS-CoV-2 RNA genome in infected cells. Nature Communications. 13 (1), 1128(2022).
- Homan, P. J., et al. Single-molecule correlated chemical probing of RNA. Proceedings of the National Academy of Sciences of the United States of America. 111 (38), 13858-13863 (2014).
- Yang, S. L., et al. Comprehensive mapping of SARS-CoV-2 interactions in vivo reveals functional virus-host interactions. Nature Communications. 12 (1), 5113(2021).
- Manfredonia, I., et al. Genome-wide mapping of SARS-CoV-2 RNA structures identifies therapeutically-relevant elements. Nucleic Acids Research. 48 (22), 12436-12452 (2020).
- Fischer, N. M., Polěto, M. D., Steuer, J., vander Spoel, D. Influence of Na+ and Mg2+ ions on RNA structures studied with molecular dynamics simulations. Nucleic Acids Research. 46 (10), 4872-4882 (2018).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved