A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
Swin-PSAxialNet: تقنية تجزئة متعددة الأعضاء فعالة
In This Article
Summary
يصف البروتوكول الحالي طريقة تجزئة فعالة متعددة الأعضاء تسمى Swin-PSAxialNet ، والتي حققت دقة ممتازة مقارنة بطرق التجزئة السابقة. تتضمن الخطوات الرئيسية لهذا الإجراء جمع مجموعات البيانات ، وتكوين البيئة ، والمعالجة المسبقة للبيانات ، والتدريب على النموذج ومقارنته ، وتجارب الاستئصال.
Abstract
يعد تجزئة أعضاء البطن المتعددة من أهم الموضوعات في مجال تحليل الصور الطبية ، ويلعب دورا مهما في دعم سير العمل السريري مثل تشخيص الأمراض وتخطيط العلاج. في هذه الدراسة ، تم اقتراح طريقة تجزئة فعالة متعددة الأعضاء تسمى Swin-PSAxialNet بناء على بنية nnU-Net. تم تصميمه خصيصا للتجزئة الدقيقة ل 11 عضوا في البطن في صور التصوير المقطعي المحوسب. أجرت الشبكة المقترحة التحسينات التالية مقارنة ب nnU-Net. أولا ، تم إدخال وحدات الفضاء إلى العمق (SPD) وكتل استخراج ميزة الانتباه المحوري المشترك للمعلمات (PSAA) ، مما يعزز قدرة استخراج ميزة الصورة ثلاثية الأبعاد. ثانيا ، تم استخدام نهج دمج الصور متعدد المقاييس لالتقاط المعلومات التفصيلية والميزات المكانية ، مما أدى إلى تحسين القدرة على استخراج الميزات الدقيقة وميزات الحافة. أخيرا ، تم تقديم طريقة مشاركة المعلمات لتقليل التكلفة الحسابية للنموذج وسرعة التدريب. تحقق الشبكة المقترحة متوسط معامل النرد 0.93342 لمهمة التجزئة التي تشمل 11 جهازا. تشير النتائج التجريبية إلى التفوق الملحوظ ل Swin-PSAxialNet على طرق التجزئة السائدة السابقة. تظهر الطريقة دقة ممتازة وتكاليف حسابية منخفضة في تجزئة أعضاء البطن الرئيسية.
Introduction
يعتمد التدخل السريري المعاصر ، بما في ذلك تشخيص الأمراض ، وصياغة خطط العلاج ، وتتبع نتائج العلاج ، على التجزئة الدقيقة للصورالطبية 1. ومع ذلك ، فإن العلاقات الهيكلية المعقدة بين أعضاء البطن2تجعل من الصعب تحقيق تجزئة دقيقة لأعضاء البطنالمتعددة 3. على مدى العقود القليلة الماضية ، قدمت التطورات المزدهرة في التصوير الطبي والرؤية الحاسوبية فرصا وتحديات جديدة في مجال تجزئة الأعضاء متعددة البطن. تمكننا تقنية التصوير بالرنين المغناطيسي المتقدم (MRI)4 والتصوير المقطعي المحوسب (CT)5 من الحصول على صور عالية الدقة للبطن. يحمل التجزئة الدقيقة لأعضاء متعددة من صور التصوير المقطعي المحوسب قيمة سريرية كبيرة لتقييم وعلاج الأعضاء الحيوية مثل الكبد والكلى والطحال والبنكرياس وما إلى ذلك.6،7،8،9،10 ومع ذلك ، فإن التعليق التوضيحي اليدوي لهذه الهياكل التشريحية ، خاصة تلك التي تتطلب تدخلا من أخصائيي الأشعة أو أخصائيي علاج الأورام بالإشعاع ، يستغرق وقتا طويلا وعرضة للتأثيرات الذاتية11. لذلك ، هناك حاجة ملحة لتطوير طرق آلية ودقيقة لتجزئة أعضاء البطن المتعددة.
اعتمدت الأبحاث السابقة حول تجزئة الصور في الغالب على الشبكات العصبية التلافيفية (CNNs) ، والتي تعمل على تحسين كفاءة التجزئة عن طريق تكديس الطبقات وإدخال ResNet12. في عام 2020 ، قدم فريق أبحاث Google نموذج Vision Transformer (VIT)13 ، مما يمثل مثالا رائدا لدمج بنية المحولات في المجال المرئي التقليدي لمجموعة من المهام المرئية14. في حين أن العمليات التلافيفية يمكن أن تفكر فقط في معلومات الميزات المحلية ، فإن آلية الانتباه في المحولات تتيح النظر الشامل في معلومات الميزات العالمية.
بالنظر إلى تفوق البنى القائمة على المحولات على الشبكات التلافيفيةالتقليدية 15 ، أجرت العديد من فرق البحث استكشافا مكثفا في تحسين التآزر بين نقاط القوة في المحولات والشبكات التلافيفية16،17،18،19. قدم Chen et آخرون TransUNet لمهام تجزئة الصور الطبية16 ، والتي تستفيد من المحولات لاستخراج الميزات العالمية من الصور. نظرا لارتفاع تكلفة التدريب على الشبكة والفشل في الاستفادة من مفهوم التسلسل الهرمي لاستخراج الميزات ، لم تتحقق مزايا المحولات بالكامل.
لمعالجة هذه المشكلات ، بدأ العديد من الباحثين في تجربة دمج المحولات كعمود فقري لتدريب شبكات التجزئة. قدم Liu et al.17 محول سوين ، الذي استخدم طريقة بناء هرمية لاستخراج الميزات ذات الطبقات. تم اقتراح مفهوم Windows Multi-Head Self-Attention (W-MSA) ، مما يقلل بشكل كبير من التكلفة الحسابية ، لا سيما في وجود خرائط ميزات ذات مستوى ضحل أكبر. في حين أن هذا النهج قلل من المتطلبات الحسابية ، إلا أنه عزل أيضا نقل المعلومات بين النوافذ المختلفة. لمعالجة هذه المشكلة ، قدم المؤلفون أيضا مفهوم الانتباه الذاتي متعدد الرؤوس (SW-MSA) ، مما يتيح نشر المعلومات بين النوافذ المجاورة. بناء على هذه المنهجية ، صاغ Cao et al. Swin-UNet18 ، واستبدل التلافيفات ثنائية الأبعاد في U-Net بوحدات Swin ودمج W-MSA و SW-MSA في عمليات التشفير وفك التشفير ، مما حقق نتائج تجزئة جديرة بالثناء.
على العكس من ذلك ، Zhou et al. سلط الضوء على أنه لا يمكن تجاهل ميزة تشغيل الإحلال عند معالجة الصور عالية الاستبانة19. يستخدم nnFormer المقترح طريقة حساب الانتباه الذاتي بناء على كتل الصور المحلية ثلاثية الأبعاد ، والتي تشكل نموذج محول يتميز بهيكل متقاطع. أدى استخدام الانتباه القائم على الكتل المحلية ثلاثية الأبعاد إلى تقليل عبء التدريب على الشبكة بشكل كبير.
بالنظر إلى مشاكل الدراسة أعلاه ، تم اقتراح هيكل هرمي هجين فعال لتجزئة الصور الطبية ثلاثية الأبعاد ، يسمى Swin-PSAxialNet. تتضمن هذه الطريقة كتلة تخفيض العينات، الكتلة من الفضاء إلى العمق (SPD)20 ، قادرة على استخراج المعلومات العالمية21. بالإضافة إلى ذلك ، فإنه يضيف وحدة الانتباه المحوري المشترك للمعلمة (PSAA) ، مما يقلل من عدد معلمات التعلم من تربيعي إلى خطي وسيكون له تأثير جيد على دقة تدريب الشبكة وتعقيد نماذجالتدريب 22.
شبكة Swin-PSAxialNet
تتبنى البنية العامة للشبكة الهيكل على شكل حرف U ل nnU-Net23 ، والذي يتكون من هياكل التشفير ووحدة فك التشفير. تشارك هذه الهياكل في استخراج الميزات المحلية وتسلسل الميزات من الصور الكبيرة والصغيرة الحجم ، كما هو موضح في الشكل 1.
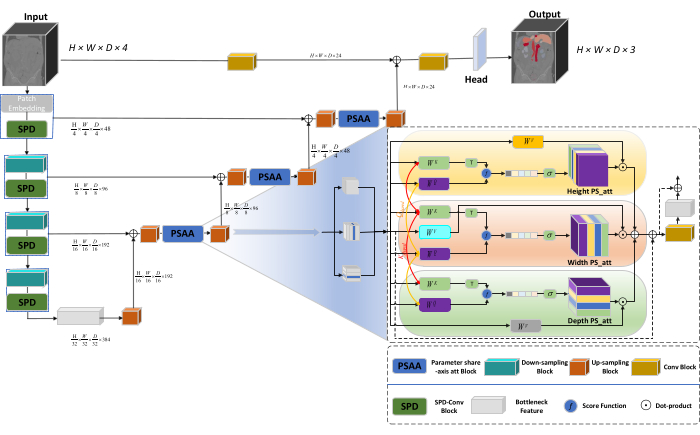
الشكل 1: الرسم التخطيطي Swin-PSAxialNet لبنية الشبكة. الرجاء النقر هنا لعرض نسخة أكبر من هذا الشكل.
في هيكل التشفير ، يتم دمج كتلة Conv التقليدية مع كتلة SPD20 لتشكيل حجم تخفيض العينات. تشتمل الطبقة الأولى من برنامج التشفير على تضمين التصحيح ، وهي وحدة تقسم البيانات ثلاثية الأبعاد إلى تصحيحات 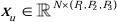 ثلاثية الأبعاد ، (P1 ، P2 ، P3) تمثل تصحيحات غير متداخلة في هذا السياق ،
ثلاثية الأبعاد ، (P1 ، P2 ، P3) تمثل تصحيحات غير متداخلة في هذا السياق ، 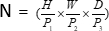 مما يدل على طول تسلسل التصحيحات ثلاثية الأبعاد. بعد طبقة التضمين ، تتضمن الخطوة التالية وحدة تصغير أخذ العينات التلافيفية غير المتداخلة التي تشتمل على كل من كتلة تلافيفية وكتلة SPD. في هذا الإعداد ، تحتوي الكتلة التلافيفية على خطوة محددة على 1 ، ويتم استخدام كتلة SPD لقياس الصورة ، مما يؤدي إلى انخفاض الدقة بمقدار أربعة أضعاف وزيادة مضاعفة في القنوات.
مما يدل على طول تسلسل التصحيحات ثلاثية الأبعاد. بعد طبقة التضمين ، تتضمن الخطوة التالية وحدة تصغير أخذ العينات التلافيفية غير المتداخلة التي تشتمل على كل من كتلة تلافيفية وكتلة SPD. في هذا الإعداد ، تحتوي الكتلة التلافيفية على خطوة محددة على 1 ، ويتم استخدام كتلة SPD لقياس الصورة ، مما يؤدي إلى انخفاض الدقة بمقدار أربعة أضعاف وزيادة مضاعفة في القنوات.
في بنية وحدة فك التشفير، تتكون كل كتلة عينة بعد طبقة ميزة عنق الزجاجة من مجموعة من كتلة أخذ العينات وكتلة PSAA. وتزداد استبانة خريطة المعالم بضعفين، ويخفض عدد القنوات إلى النصف بين كل زوج من مراحل وحدة فك التشفير. لاستعادة المعلومات المكانية وتحسين تمثيل المعالم ، يتم إجراء دمج الميزات بين الصور الكبيرة والصغيرة الحجم بين كتل أخذ العينات. في النهاية ، يتم إدخال نتائج أخذ العينات في طبقة الرأس لاستعادة حجم الصورة الأصلي ، بحجم إخراج (H × W × D × C ، C = 3).
بنية كتلة SPD
في الطرق التقليدية ، يستخدم قسم أخذ العينات خطوة واحدة بحجم خطوة 2. يتضمن ذلك التجميع التلافيفي في المواضع المحلية في الصورة ، مما يحد من المجال الاستقبالي ، ويقصر النموذج على استخراج الميزات من تصحيحات الصور الصغيرة. تستخدم هذه الطريقة كتلة SPD ، التي تقسم الصورة الأصلية بدقة إلى ثلاثة أبعاد. يتم تقسيم الصورة ثلاثية الأبعاد الأصلية بالتساوي على طول المحاور x و y و z ، مما ينتج عنه أربعة أجسام ذات حجم فرعي. (الشكل 2) بعد ذلك ، يتم تسلسل المجلدات الأربعة من خلال عملية "cat" ، وتخضع الصورة الناتجة لتلاف 1 × 1 × 1 للحصول على الصورة المأخوذةمن العينات السفلية 20.
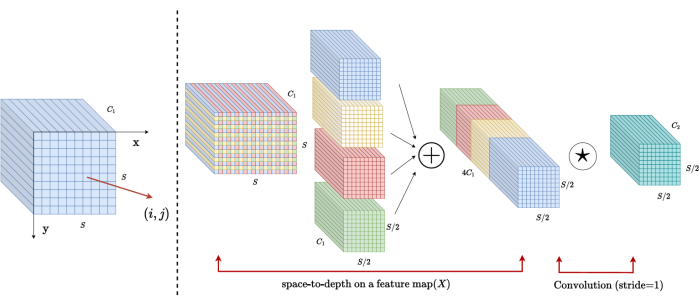
الشكل 2: مخطط كتلة SPD. الرجاء النقر هنا لعرض نسخة أكبر من هذا الشكل.
بنية كتلة PSAA
على عكس شبكات CNN التقليدية ، فإن كتلة PSAA المقترحة أكثر فعالية في إجراء التركيز العالمي على المعلومات وأكثر كفاءة في التعلم والتدريب على الشبكات. يتيح ذلك التقاط صور أكثر ثراء وميزات مكانية. تتضمن كتلة PSAA تعلم الانتباه المحوري بناء على مشاركة المعلمات في ثلاثة أبعاد: الارتفاع والعرض والعمق. بالمقارنة مع آلية الانتباه التقليدية التي تقوم بتعلم الانتباه لكل بكسل في الصورة ، تجري هذه الطريقة بشكل مستقل تعلم الانتباه لكل من الأبعاد الثلاثة ، مما يقلل من تعقيد الانتباه الذاتي من التربيعي إلى الخطي. علاوة على ذلك ، يتم استخدام آلية مشاركة معلمات المفاتيح والاستعلامات القابلة للتعلم ، مما يمكن الشبكة من أداء عمليات آلية الانتباه بالتوازي عبر الأبعاد الثلاثة ، مما يؤدي إلى تمثيل أسرع ومتفوق وأكثر فعالية.
Access restricted. Please log in or start a trial to view this content.
Protocol
تمت الموافقة على هذا البروتوكول من قبل لجنة الأخلاقيات بجامعة نانتونغ. يتضمن التقييم الذكي والبحث للبيانات متعددة الوسائط غير الغازية أو طفيفة التوغل المكتسبة ، بما في ذلك الصور الطبية البشرية وحركات الأطراف وتصوير الأوعية الدموية ، باستخدام تقنية الذكاء الاصطناعي. يوضح الشكل 3 المخطط الانسيابي الكلي للتجزئة متعددة الأعضاء. يتم توفير جميع روابط الويب اللازمة في جدول المواد.
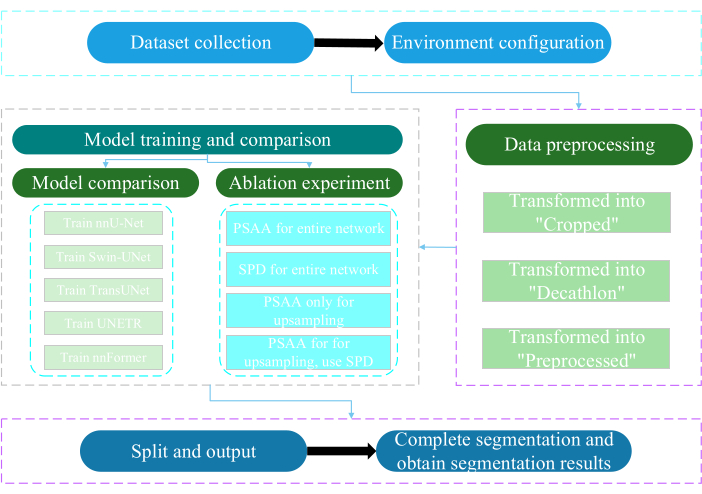
الشكل 3: مخطط انسيابي شامل للتجزئة متعددة الأعضاء. الرجاء النقر هنا لعرض نسخة أكبر من هذا الشكل.
1. جمع مجموعات البيانات
- قم بتنزيل مجموعة البيانات AMOS202224 (انظر جدول المواد).
- حدد 160 صورة كمجموعة بيانات التدريب و 40 صورة كمجموعة بيانات الاختبار.
- تنفيذ mkdir ~/PaddleSeg/contrib/MedicalSeg/data/raw_data.
- افتح مسار raw_data.
- ضع مجموعة البيانات في دليل raw_data ، وتأكد من المراسلات الصحيحة مع أسماء الملفات في دليلي "imagesTr" و "labelsTr".
- قم بإنشاء ملف dataset.json .
- أضف معلومات مجموعة البيانات مثل numTraining وnumTest واسم التسميات في الملف "dataset.json".
ملاحظة: تتكون مجموعة البيانات الخاصة بهذه الدراسة من 200 مجموعة بيانات عالية الجودة للتصوير المقطعي المحوسب تم اختيارها من مجموعة بيانات 500 CT الرسمية AMOS2022 لأغراض التدريب والاختبار. تشمل مجموعة البيانات 11 عضوا ، بما في ذلك الطحال والكلية اليمنى والكلية اليسرى والمرارة والمريء والكبد والمعدة والشريان الأورطي والوريد الأجوف السفلي والبنكرياس والمثانة.
2. تكوين البيئة
- قم بتثبيت بيئة Paddlepaddle (انظر جدول المواد) وإصدار CUDAالمقابل 25.
ملاحظة: يوصى باستخدام الإصدار 2.5.0 من Paddlepaddle والإصدار 11.7 من CUDA. - Git استنساخ مستودع PaddleSeg25.
- تنفيذ القرص المضغوط ~ / PaddleSeg / contrib / MedicalSeg /.
- تنفيذ تثبيت النقطة -r requirements.txt.
- تنفيذ pip install medpy.
3. المعالجة المسبقة للبيانات
- قم بتعديل ملف "yml" ، بما في ذلك معلمات مثل data_root و batch_size و model و train_dataset و val_dataset و Optimizer و lr_scheduler و loss.
- حدد المعلمات المطلوبة للتنفيذ ، بما في ذلك "-config ، --log_iters ، --precision ، --nnunet ، --save_dir ، --save_interval ، إلخ.
ملاحظة: يتم تحويل "raw_data" الأصلي إلى ثلاثة أنواع من ملفات البيانات: "اقتصاص" و "Decathlon" و "معالج مسبقا" ، لتلبية متطلبات التدريب والتقييم المختلفة لتعزيز أداء النموذج وفعالية التدريب.
4. نموذج التدريب والمقارنة
ملاحظة: كخط أساس مستخدم على نطاق واسع في مجال تجزئة الصور ، يعمل nnU-Net23 كنموذج أساسي في الدراسة. عملية مقارنة النموذج المحددة هي كما يلي.
- قطار nnU-Net
- قم بالتكوين بحجم دفعة من 2 و 80,000 تكرار ، وسجل كل 20 تكرارا ، واستخدم دقة fp16.
- قم بتعيين save_dir كمجلد "الإخراج" داخل دليل العمل ، مع تعيين فاصل زمني للحفظ عند 1,000 تكرار.
- أضف المعلمة --use_vdl لتصور معلمة VisualDL.
- اركض train.py.
- قم بإنشاء أربعة ملفات yml ، وحدد الطيات من 1 إلى 4 ، وقم بتدريب النموذج بالتحقق من الصحة المتقاطع بخمسة أضعاف.
- تدريب سوين-أونيت
- الحفاظ على الاتساق مع الظروف التجريبية المحددة مسبقا.
- قم بإزالة المعلمة --nnunet واستخدم --swinunet.
- قم بتشغيل ملف train.py.
- قم بتدريب الطيات من 1 إلى 4 بشكل فردي ثم يخضع لمعالجة المجموعة.
- تدريب TransUNet
- اتبع الإجراءات الموضحة في الخطوة 4.1.
- افتح ملف yml .
- تعديل النوع: nnunet للكتابة: transunet.
- تعديل المعلمة --nnunet إلى --transunet.
- قم بتشغيل ملف train.py.
- قم بتدريب الطيات من 1 إلى 4 بشكل فردي ومعالجة المجموعة.
- تدريب UNETR
- الحفاظ على المتغيرات التجريبية المتسقة.
- كرر الإجراءات الموضحة سابقا مثل Swin-UNet و TransUNet (الخطوة 4.2 والخطوة 4.3).
- اركض train.py.
- قم بتدريب الطيات من 1 إلى 4 بشكل فردي ومعالجة المجموعة.
- تدريب nnالسابق
- قم بتعديل المعلمات مثل Swin-UNet و TransUNet.
- قم بتشغيل train.py طية واحدة.
- تشغيل معالجة المجموعة.
5. تجربة الاستئصال
- كتلة PSAA للشبكة بأكملها
- حافظ على اتساق المتغيرات مع الخطوة 4.1.
- اضبط volume_shape = (64,192,192) و Axial_attention = صواب.
- قم بتشغيل train.py أضعاف واحدة.
- معالجة المجموعة عبر خمس طيات.
- كتلة SPD للشبكة بأكملها
- حافظ على اتساق المتغيرات مع الخطوة 4.1.
- اضبط SPD_enable = صواب و Axial_attention = خطأ.
- قم بتشغيل train.py طية واحدة.
- معالجة المجموعة عبر خمس طيات.
- كتلة PSAA لأخذ العينات
ملاحظة: لا تستخدم كتلة SPD لأخذ العينات.- استخدم كتلة PSAA لأخذ العينات.
- حافظ على اتساق المتغيرات مع الخطوة 4.1.
- اضبط Axial_attention = False في جزء التخفيض و SPD_enable = False.
- قم بتشغيل train.py أضعاف واحدة.
- معالجة مجموعة خمس طيات.
- كتلة PSAA لأخذ العينات
ملاحظة: استخدم كتلة SPD لتقليل العينات.- استخدم كتلة SPD لأخذ العينات المصغرة وكتلة PSAA لأخذ العينات.
- حافظ على اتساق المتغيرات مع الخطوة 4.1.
- اضبط Axial_attention = خطأ في جزء أخذ العينات المصغرة و SPD_enable = صواب.
- قم بتشغيل train.py طية واحدة.
- معالجة مجموعة خمس طيات.
- قم بتعيين عدد التكرارات إلى 2,00,000 بسبب النتائج الفائقة.
Access restricted. Please log in or start a trial to view this content.
النتائج
يستخدم هذا البروتوكول مقياسين لتقييم النموذج: درجة تشابه النرد (DSC) و 95٪ مسافة Hausdorff (HD95). يقيس DSC التداخل بين تنبؤات تجزئة فوكسل والحقيقة الأرضية ، بينما يقوم 95٪ HD بتقييم التداخل بين حدود التنبؤ بتجزئة فوكسل والحقيقة الأرضية ، وتصفية 5٪ من القيم المتطرفة. وفيما يلي تعريف DSC<...
Access restricted. Please log in or start a trial to view this content.
Discussion
تجزئة أعضاء البطن عمل معقد. بالمقارنة مع الهياكل الداخلية الأخرى لجسم الإنسان ، مثل الدماغ أو القلب ، يبدو تجزئة أعضاء البطن أكثر صعوبة بسبب التباين المنخفض والتغيرات الكبيرة في الشكل في صور التصوير المقطعي المحوسب27،28. يقترح Swin-PSAxialNet هنا...
Access restricted. Please log in or start a trial to view this content.
Disclosures
يعلن أصحاب البلاغ عدم وجود تضارب في المصالح.
Acknowledgements
تم دعم هذه الدراسة من قبل المشروع الهندسي "333" لمقاطعة جيانغسو ([2022] 21-003) ، والبرنامج العام للجنة الصحة في Wuxi (M202205) ، وصندوق Wuxi لتطوير العلوم والتكنولوجيا (Y20212002-1) ، والتي كانت مساهماتها لا تقدر بثمن في نجاح هذا العمل ". يشكر المؤلفون جميع مساعدي الباحثين والمشاركين في الدراسة على دعمهم.
Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| AMOS2022 dataset | None | None | Datasets for network training and testing. The weblink is: https://pan.baidu.com/s/1x2ZW5FiZtVap0er55Wk4VQ?pwd=xhpb |
| ASUS mainframe | ASUS | https://www.asusparts.eu/en/asus-13020-01910200 | |
| CUDA version 11.7 | NVIDIA | https://developer.nvidia.com/cuda-11-7-0-download-archive | |
| NVIDIA GeForce RTX 3090 | NVIDIA | https://www.nvidia.com/en-in/geforce/graphics-cards/30-series/rtx-3090-3090ti/ | |
| Paddlepaddle environment | Baidu | None | Environmental preparation for network training. The weblink is: https://www.paddlepaddle.org.cn/ |
| PaddleSeg | Baidu | None | The baseline we use: https://github.com/PaddlePaddle/PaddleSeg |
References
- Liu, X., Song, L., Liu, S., Zhang, Y. A review of deep-learning-based medical image segmentation methods. Sustain. 13 (3), 1224(2021).
- Popilock, R., Sandrasagaren, K., Harris, L., Kaser, K. A. CT artifact recognition for the nuclear technologist. J Nucl Med Technol. 36 (2), 79-81 (2008).
- Rehman, A., Khan, F. G. A deep learning based review on abdominal images. Multimed Tools Appl. 80, 30321-30352 (2021).
- Schenck, J. F. The role of magnetic susceptibility in magnetic resonance imaging: Mri magnetic compatibility of the first and second kinds. Med Phys. 23 (6), 815-850 (1996).
- Palmisano, A., et al. Myocardial late contrast enhancement ct in troponin-positive acute chest pain syndrome. Radiol. 302 (3), 545-553 (2022).
- Chen, E. -L., Chung, P. -C., Chen, C. -L., Tsai, H. -M., Chang, C. -I. An automatic diagnostic system for CT liver image classification. IEEE Trans Biomed Eng. 45 (6), 783-794 (1998).
- Sagel, S. S., Stanley, R. J., Levitt, R. G., Geisse, G. J. R. Computed tomography of the kidney. Radiol. 124 (2), 359-370 (1977).
- Freeman, J. L., Jafri, S., Roberts, J. L., Mezwa, D. G., Shirkhoda, A. CT of congenital and acquired abnormalities of the spleen. Radiographics. 13 (3), 597-610 (1993).
- Almeida, R. R., et al. Advances in pancreatic CT imaging. AJR Am J Roentgenol. 211 (1), 52-66 (2018).
- Eisen, G. M., et al. Guidelines for credentialing and granting privileges for endoscopic ultrasound. Gastrointest Endosc. 54 (6), 811-814 (2001).
- Sykes, J. Reflections on the current status of commercial automated segmentation systems in clinical practice. J Med Radiat Sci. 61 (3), 131-134 (2014).
- He, K., Zhang, X., Ren, S., Sun, J. Deep residual learning for image recognition. 2016 IEEE Conference on Computer Vision and Pattern Recognition (CVPR). , Vegas, NV, USA. 770-778 (2016).
- Dosovitskiy, A., et al. An image is worth 16x16 words: Transformers for image recognition at scale. arXiv. arXiv. , 11929(2020).
- Ramachandran, P., et al. Stand-alone self-attention in vision models. Adv Neural Inf Process Syst. 32, NIPS2019 (2019).
- Vaswani, A., et al. Attention is all you need. Adv Neural Inf Process Syst. 30, NIPS2019 (2017).
- Chen, J., et al. TransUNET: Transformers make strong encoders for medical image segmentation. arXiv. , arXiv:2102 04396(2021).
- Liu, Z., et al. Swin transformer: Hierarchical vision transformer using shifted windows. Proc IEEE Int Conf Comput Vis. , 10012-10022 (2021).
- Cao, H., et al. Swin-UNET: Unet-like pure transformer for medical image segmentation. Comput Vis ECCV. , 205-218 (2022).
- Zhou, H. -Y., et al. nnformer: Interleaved transformer for volumetric segmentation. arXiv. arXiv. , arXiv:2109 03201(2021).
- Sunkara, R., Luo, T. No more strided convolutions or pooling: A new CNN building block for low-resolution images and small objects. Mach Learn Knowl Discov Databases. , 443-459 (2023).
- Çiçek, Ö, Abdulkadir, A., Lienkamp, S. S., Brox, T., Ronneberger, O. 3D U-Net: learning dense volumetric segmentation from sparse annotation. Med Comput Vis Bayesian Graph Models Biomed Imaging. 2016, 424-432 (2016).
- Kim, H., et al. Abdominal multi-organ auto-segmentation using 3D-patch-based deep convolutional neural network. Scientific reports. 10 (1), 6204(2020).
- Isensee, F., et al. nnu-net: Self-adapting framework for u-net-based medical image segmentation. arXiv. , arXiv:1809 10486(2018).
- Ji, Y., et al. A large-scale abdominal multi-organ benchmark for versatile medical image segmentation. Adv Neural Inf Process Syst. 35, 36722-36732 (2022).
- Liu, Y., et al. Paddleseg: A high-efficient development toolkit for image segmentation. arXiv. , arXiv:2101 06175(2021).
- Hatamizadeh, A., et al. UNETR: Transformers for 3D medical image segmentation. Proc IEEE Int Conf Comput Vis. , https://api.semanticscholar.org/CorpusID:232290634 574-584 (2022).
- Zhou, H., Zeng, D., Bian, Z., Ma, J. A semi-supervised network-based tissue-aware contrast enhancement method for ct images. Nan Fang Yi Ke Da Xue Xue Bao. 43 (6), 985-993 (2023).
- Ma, J., et al. Abdomenct-1k: Is abdominal organ segmentation a solved problem. IEEE Trans Pattern Anal Mach Intell. 44 (10), 6695-6714 (2021).
- Galván, E., Mooney, P. Neuroevolution in deep neural networks: Current trends and future challenges. IEEE Trans Artif Intell. 2 (6), 476-493 (2021).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved