需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
Swin-PSAxialNet:一种高效的多器官分割技术
摘要
本协议描述了一种称为 Swin-PSAxialNet 的高效多器官分割方法,与以前的分割方法相比,该方法取得了出色的准确性。此过程的关键步骤包括数据集收集、环境配置、数据预处理、模型训练和比较以及消融实验。
摘要
腹部多器官分割是医学影像分析领域最重要的课题之一,在支持疾病诊断和治疗计划等临床工作流程中发挥着重要作用。在本研究中,提出了一种基于 nnU-Net 架构的高效多器官分割方法,称为 Swin-PSAxialNet。它专为在 CT 图像中精确分割 11 个腹部器官而设计。与 nnU-Net 相比,拟议的网络进行了以下改进。首先,引入空深 (SPD) 模块和参数共享轴向注意力 (PSAA) 特征提取块,增强了 3D 图像特征提取的能力;其次,采用多尺度图像融合方法捕获详细信息和空间特征,提高细微特征和边缘特征的提取能力;最后,引入参数共享方法,降低模型的计算成本和训练速度。对于涉及 11 个器官的分割任务,所提出的网络实现了 0.93342 的平均 Dice 系数。实验结果表明,Swin-PSAxialNet 优于以前的主流分割方法。该方法在分割主要腹部器官方面表现出优异的准确性和较低的计算成本。
引言
现代临床干预,包括疾病诊断、治疗计划的制定和治疗结果的跟踪,都依赖于医学图像的准确分割1。然而,腹部器官2(abdominal organs) 之间复杂的结构关系使得实现多个腹部器官的准确分割成为一项具有挑战性的任务3。在过去的几十年里,医学成像和计算机视觉的蓬勃发展为腹部多器官分割领域带来了新的机遇和挑战。先进的磁共振成像 (MRI) 4 和计算机断层扫描 (CT) 技术5 使我们能够获取高分辨率的腹部图像。从 CT 图像中精确分割多个器官对于评估和治疗肝脏、肾脏、脾脏、胰腺等重要器官具有重要的临床价值6,7,8,9,10 然而,这些解剖结构的手动注释,尤其是那些需要放射科医生或放射肿瘤学家干预的结构,既耗时又容易受到主观影响11. 因此,迫切需要开发自动化和准确的腹部多器官分割方法。
以前关于图像分割的研究主要依赖于卷积神经网络 (CNN),它通过堆叠层和引入 ResNet12 来提高分割效率。2020 年,Google 研究团队推出了 Vision Transformer (VIT) 模型13,标志着将 Transformer 架构整合到传统视觉领域以完成一系列视觉任务的开创性实例14。虽然卷积运算只能考虑局部特征信息,但 Transformers 中的注意力机制可以全面考虑全局特征信息。
考虑到基于 Transformer 的架构优于传统的卷积网络15,许多研究团队在优化 Transformer 和卷积网络之间的协同作用方面进行了广泛的探索 16,17,18,19。Chen 等人介绍了用于医学图像分割任务16 的 TransUNet,它利用 Transformers 从图像中提取全局特征。由于网络训练成本高,且未能利用特征提取层次结构的概念,Transformer 的优势尚未得到充分实现。
为了解决这些问题,许多研究人员已经开始尝试将 Transformers 作为训练分割网络的主干。Liu et al.17 介绍了 Swin Transformer,它采用分层构造方法进行分层特征提取。提出了 Windows 多头自我注意 (W-MSA) 的概念,显著降低了计算成本,尤其是在存在较大的浅层特征图的情况下。虽然这种方法降低了计算要求,但它也隔离了不同窗口之间的信息传输。为了解决这个问题,作者进一步引入了 Shifted Windows Multi-Head Self-Attention (SW-MSA) 的概念,使信息能够在相邻窗口之间传播。基于这种方法,Cao 等人制定了 Swin-UNet18,用 Swin 模块取代了 U-Net 中的 2D 卷积,并将 W-MSA 和 SW-MSA 纳入编码和解码过程,取得了值得称道的分割结果。
相反,周 et al. 强调,在处理高分辨率图像时,conv作的优势不容忽视19。他们提出的 nnFormer 采用了一种基于局部三维图像块的自注意力计算方法,构成了一个以十字形结构为特征的 Transformer 模型。基于局部三维块的注意力的利用显著降低了网络上的训练负载。
鉴于上述研究存在的问题,提出了一种用于 3D 医学图像分割的高效混合分层结构,称为 Swin-PSAxialNet。该方法包含一个下采样模块,即空间到深度 (SPD)20 模块,能够提取全局信息21。此外,它还增加了一个参数共享轴注意力 (PSAA) 模块,将学习参数数量从二次减少到线性,将对网络训练的准确性和训练模型的复杂性产生良好的影响22。
Swin-PSAxialNet 网络
网络整体架构采用 nnU-Net23 的 U 型结构,由编码器和解码器结构组成。这些结构参与局部特征提取和大尺度和小尺度图像的特征串联,如图 1 所示。
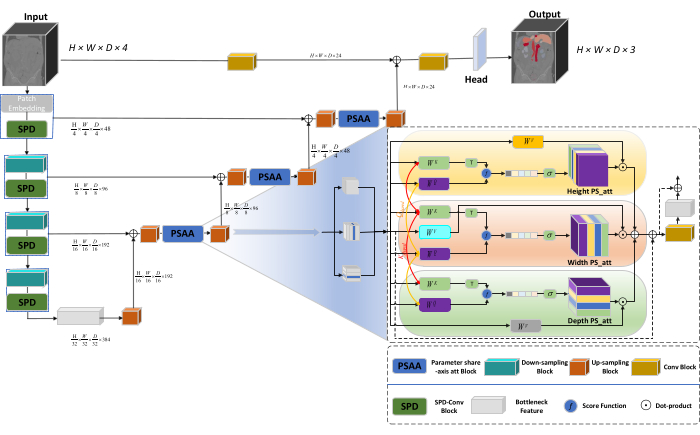
图 1:Swin-PSAxialNet 网络架构示意图。请单击此处查看此图的较大版本。
在编码器结构中,传统的 Conv 模块与 SPD 模块20 组合形成一个下采样体积。编码器的第一层包含 Patch Embedding,这是一个将 3D 数据划分为 3D 补丁 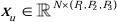 的模块,(P1、 P2、 P3)在此上下文中表示不重叠的补丁,
的模块,(P1、 P2、 P3)在此上下文中表示不重叠的补丁,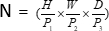 表示 3D 补丁的序列长度。在嵌入层之后,下一步涉及一个非重叠的卷积下采样单元,该单元由一个卷积块和一个 SPD 块组成。在此设置中,卷积块的步幅设置为 1,并且 SPD 块用于图像缩放,导致分辨率降低四倍,通道增加两倍。
表示 3D 补丁的序列长度。在嵌入层之后,下一步涉及一个非重叠的卷积下采样单元,该单元由一个卷积块和一个 SPD 块组成。在此设置中,卷积块的步幅设置为 1,并且 SPD 块用于图像缩放,导致分辨率降低四倍,通道增加两倍。
在解码器结构中,Bottleneck Feature 层之后的每个 upsample 块都由一个 upsampling 块和一个 PSAA 块的组合组成。特征图的分辨率提高了两倍,每对解码器阶段之间的通道数减半。为了恢复空间信息并增强特征表示,在上采样块之间执行大比例和小比例图像之间的特征融合。最终,增加采样结果被馈送到 Head 层以恢复原始图像大小,输出大小为 (H × W × D × C, C = 3)。
SPD 块架构
在传统方法中,缩减采样部分采用步长为 2 的单个步幅。这涉及图像中局部位置的卷积池化,限制感受野,并将模型限制为从小图像块中提取特征。这种方法利用 SPD 块,它将原始图像精细地划分为三个维度。原始 3D 图像沿 x、y 和 z 轴均匀分割,从而产生四个子体积实体。(图 2)随后,通过 “cat”作将四个体积连接起来,得到的图像经过 1 × 1 × 1 卷积,得到下采样图像20。
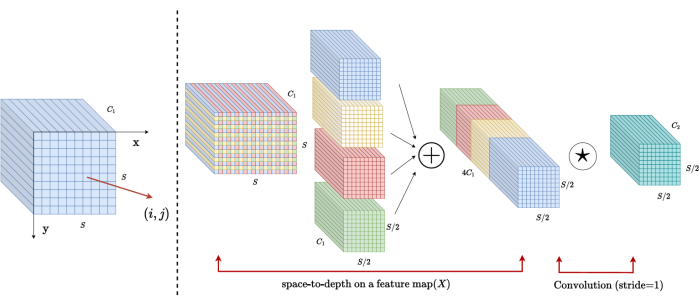
图 2:SPD 框图。请单击此处查看此图的较大版本。
PSAA 块架构
与传统的 CNN 网络相比,所提出的 PSAA 块在进行全球信息聚焦方面更有效,在网络学习和培训方面效率更高。这样可以捕获更丰富的图像和空间特征。PSAA 模块包括基于三个维度共享的参数的轴向注意力学习:高度、宽度和深度。与对图像中的每个像素进行注意力学习的传统注意力机制相比,这种方法独立地对三个维度中的每一个维度进行注意力学习,将自我注意力的复杂性从二次级降低到线性。此外,采用了可学习的 keys-queries 参数共享机制,使网络能够在三个维度上并行执行注意力机制作,从而获得更快、更优越、更有效的特征表示。
研究方案
本方案经南通大学伦理委员会批准。它涉及利用人工智能技术对获取的无创或微创多模态数据进行智能评估和研究,包括人体医学图像、肢体运动和血管成像。 图 3 描述了多器官分割的整体流程图。 材料表中提供了所有必要的网络链接。
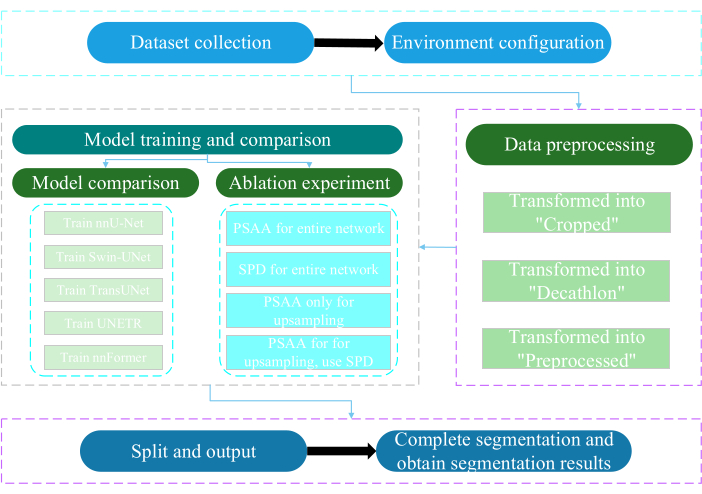
图 3:多器官分割的总体流程图。请单击此处查看此图的较大版本。
1. 数据集集合
- 下载 AMOS2022 数据集24 (参见 材料表)。
- 选择 160 张图像作为训练数据集,选择 40 张图像作为测试数据集。
- 终端执行 mkdir ~/PaddleSeg/contrib/MedicalSeg/data/raw_data。
- 打开 raw_data 路径。
- 将数据集放在 raw_data 目录下,并确保与 imagesTr 和 labelsTr 目录下的文件名正确对应。
- 创建 dataset.json 文件。
- 将 numTraining、 numTest 和 labels name 等数据集信息添加到“dataset.json”文件中。
注:本研究的数据集由从 AMOS2022 官方 500 CT 数据集中选择的 200 个高质量 CT 数据集组成,用于训练和测试目的。该数据集包括 11 个器官,包括脾脏、右肾、左肾、胆囊、食管、肝脏、胃、主动脉、下腔静脉、胰腺和膀胱。
2. 环境配置
- 安装 Paddlepaddle 环境(参见 材料表)和相应的 CUDA 版本25。
注意:建议使用 Paddlepaddle 版本 2.5.0 和 CUDA 版本 11.7。 - Git clone PaddleSeg 仓库25.
- 最终执行 cd ~/PaddleSeg/contrib/MedicalSeg/。
- 执行 pip install -r requirements.txt。
- 执行 pip install medpy。
3. 数据预处理
- 修改 yml 文件,包括 data_root、batch_size、model、train_dataset、val_dataset、optimizer、lr_scheduler 和 loss 等参数。
- 指定执行所需的参数,包括 “-config, --log_iters, --precision, --nnunet, --save_dir, --save_interval 等。
注意:将原来的 “raw_data” 转换为 “Cropped”、“Decathlon” 和 “Preprocessed” 三种类型的数据文件,以满足不同的训练和评估要求,以提高模型性能和训练效果。
4. 模型训练和比较
注意:作为图像分割领域广泛使用的基线,nnU-Net23 在研究中用作基线模型。具体型号对比流程如下。
- 训练 nnU-Net
- 配置批处理大小 2 和 80,000 次迭代,每 20 次迭代记录一次,并利用 fp16 精度。
- 将 save_dir 设置为工作目录中的 “output” 文件夹,保存间隔设置为 1,000 次迭代。
- 添加参数 --use_vdl 用于 VisualDL 参数可视化。
- 运行 train.py。
- 创建 4 个 yml 文件,指定 folds 1-4,并使用 5 fold 交叉验证训练模型。
- 训练 Swin-UNet
- 与先前建立的实验条件保持一致。
- 删除参数 --nnunet 并使用 --swinunet。
- 运行 train.py 文件。
- 火车单独折叠 1-4 次,然后进行集成处理。
- 训练 TransUNet
- 按照步骤 4.1 中概述的过程进行作。
- 打开 yml 文件。
- 将 type: nnunet 修改为 type: transunet。
- 将参数 --nnunet 修改为 --transunet。
- 运行 train.py 文件。
- 单独训练折叠 1-4 并处理集成处理。
- 训练 UNETR
- 保持一致的实验变量。
- 重复前面概述的过程,如 Swin-UNet 和 TransUNet(步骤 4.2 和步骤 4.3)。
- 运行 train.py。
- 单独训练折叠 1-4 并处理集成处理。
- 训练 nnFormer
- 修改 Swin-UNet 和 TransUNet 等参数。
- 运行 single fold train.py。
- 运行 ensemble processing。
5. 消融实验
- 整个网络的 PSAA 块
- 使变量与步骤 4.1 保持一致。
- 设置 volume_shape = (64,192,192) 和 Axial_attention = True。
- 运行 single fold train.py。
- 跨 5 个折叠处理集成处理。
- 整个网络的 SPD 块
- 使变量与步骤 4.1 保持一致。
- 设置 SPD_enable = True 和 Axial_attention = False。
- 运行 single fold train.py。
- 跨 5 个折叠处理集成处理。
- 用于上采样的 PSAA 块
注意:请勿使用 SPD 模块进行下采样。- 使用 PSAA 模块进行上采样。
- 使变量与步骤 4.1 保持一致。
- 在缩减采样部分设置 Axial_attention = False,SPD_enable = False。
- 运行 single fold train.py。
- 处理 5 折 集成 处理。
- 用于上采样的 PSAA 块
注意:使用 SPD 模块进行下采样。- 使用 SPD 模块进行下采样,使用 PSAA 模块进行上采样。
- 使变量与步骤 4.1 保持一致。
- 在缩减采样部分设置 Axial_attention = False,SPD_enable = True。
- 运行 single fold train.py。
- 处理 5 折 集成 处理。
- 将迭代次数设置为 2,00,000,因为结果更好。
结果
该协议采用两个指标来评估模型:骰子相似性分数 (DSC) 和 95% 豪斯多夫距离 (HD95)。DSC 测量体素分割预测与地面实况之间的重叠,而 95% HD 评估体素分割预测边界与地面实况之间的重叠,过滤掉 5% 的异常值。DSC26 的定义如下:
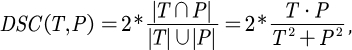 (1)
(1)
其中 T 和 P ...
讨论
腹部器官的分割是一项复杂的工作。与人体的其他内部结构(如大脑或心脏)相比,由于 CT 图像的对比度低且形状变化大,分割腹部器官似乎更具挑战性27,28。这里提出 Swin-PSAxialNet 来解决这个难题。
在数据收集步骤中,本研究从 AMOS2022 官网下载了 200 张图像24.在环境配置步骤中,实验设...
披露声明
作者声明没有利益冲突。
致谢
这项研究得到了江苏省'333'工程项目([2022]21-003)、无锡市卫健委总体计划(M202205)和无锡市科学技术发展基金(Y20212002-1)的支持,他们对这项工作的成功做出了宝贵的贡献。作者感谢所有研究助理和研究参与者的支持。
材料
| Name | Company | Catalog Number | Comments |
| AMOS2022 dataset | None | None | Datasets for network training and testing. The weblink is: https://pan.baidu.com/s/1x2ZW5FiZtVap0er55Wk4VQ?pwd=xhpb |
| ASUS mainframe | ASUS | https://www.asusparts.eu/en/asus-13020-01910200 | |
| CUDA version 11.7 | NVIDIA | https://developer.nvidia.com/cuda-11-7-0-download-archive | |
| NVIDIA GeForce RTX 3090 | NVIDIA | https://www.nvidia.com/en-in/geforce/graphics-cards/30-series/rtx-3090-3090ti/ | |
| Paddlepaddle environment | Baidu | None | Environmental preparation for network training. The weblink is: https://www.paddlepaddle.org.cn/ |
| PaddleSeg | Baidu | None | The baseline we use: https://github.com/PaddlePaddle/PaddleSeg |
参考文献
- Liu, X., Song, L., Liu, S., Zhang, Y. A review of deep-learning-based medical image segmentation methods. Sustain. 13 (3), 1224 (2021).
- Popilock, R., Sandrasagaren, K., Harris, L., Kaser, K. A. CT artifact recognition for the nuclear technologist. J Nucl Med Technol. 36 (2), 79-81 (2008).
- Rehman, A., Khan, F. G. A deep learning based review on abdominal images. Multimed Tools Appl. 80, 30321-30352 (2021).
- Schenck, J. F. The role of magnetic susceptibility in magnetic resonance imaging: Mri magnetic compatibility of the first and second kinds. Med Phys. 23 (6), 815-850 (1996).
- Palmisano, A., et al. Myocardial late contrast enhancement ct in troponin-positive acute chest pain syndrome. Radiol. 302 (3), 545-553 (2022).
- Chen, E. -. L., Chung, P. -. C., Chen, C. -. L., Tsai, H. -. M., Chang, C. -. I. An automatic diagnostic system for CT liver image classification. IEEE Trans Biomed Eng. 45 (6), 783-794 (1998).
- Sagel, S. S., Stanley, R. J., Levitt, R. G., Geisse, G. J. R. Computed tomography of the kidney. Radiol. 124 (2), 359-370 (1977).
- Freeman, J. L., Jafri, S., Roberts, J. L., Mezwa, D. G., Shirkhoda, A. CT of congenital and acquired abnormalities of the spleen. Radiographics. 13 (3), 597-610 (1993).
- Almeida, R. R., et al. Advances in pancreatic CT imaging. AJR Am J Roentgenol. 211 (1), 52-66 (2018).
- Eisen, G. M., et al. Guidelines for credentialing and granting privileges for endoscopic ultrasound. Gastrointest Endosc. 54 (6), 811-814 (2001).
- Sykes, J. Reflections on the current status of commercial automated segmentation systems in clinical practice. J Med Radiat Sci. 61 (3), 131-134 (2014).
- He, K., Zhang, X., Ren, S., Sun, J. Deep residual learning for image recognition. 2016 IEEE Conference on Computer Vision and Pattern Recognition (CVPR). , 770-778 (2016).
- Dosovitskiy, A., et al. An image is worth 16x16 words: Transformers for image recognition at scale. arXiv. arXiv. , 11929 (2020).
- Ramachandran, P., et al. Stand-alone self-attention in vision models. Adv Neural Inf Process Syst. 32, (2019).
- Vaswani, A., et al. Attention is all you need. Adv Neural Inf Process Syst. 30, (2017).
- Chen, J., et al. TransUNET: Transformers make strong encoders for medical image segmentation. arXiv. , 04396 (2021).
- Liu, Z., et al. Swin transformer: Hierarchical vision transformer using shifted windows. Proc IEEE Int Conf Comput Vis. , 10012-10022 (2021).
- Cao, H., et al. Swin-UNET: Unet-like pure transformer for medical image segmentation. Comput Vis ECCV. , 205-218 (2022).
- Zhou, H. -. Y., et al. nnformer: Interleaved transformer for volumetric segmentation. arXiv. arXiv. , 03201 (2021).
- Sunkara, R., Luo, T. No more strided convolutions or pooling: A new CNN building block for low-resolution images and small objects. Mach Learn Knowl Discov Databases. , 443-459 (2023).
- Çiçek, &. #. 2. 1. 4. ;., Abdulkadir, A., Lienkamp, S. S., Brox, T., Ronneberger, O. 3D U-Net: learning dense volumetric segmentation from sparse annotation. Med Comput Vis Bayesian Graph Models Biomed Imaging. 2016, 424-432 (2016).
- Kim, H., et al. Abdominal multi-organ auto-segmentation using 3D-patch-based deep convolutional neural network. Scientific reports. 10 (1), 6204 (2020).
- Isensee, F., et al. nnu-net: Self-adapting framework for u-net-based medical image segmentation. arXiv. , 10486 (2018).
- Ji, Y., et al. A large-scale abdominal multi-organ benchmark for versatile medical image segmentation. Adv Neural Inf Process Syst. 35, 36722-36732 (2022).
- Liu, Y., et al. Paddleseg: A high-efficient development toolkit for image segmentation. arXiv. , 06175 (2021).
- Hatamizadeh, A., et al. UNETR: Transformers for 3D medical image segmentation. Proc IEEE Int Conf Comput Vis. , 574-584 (2022).
- Zhou, H., Zeng, D., Bian, Z., Ma, J. A semi-supervised network-based tissue-aware contrast enhancement method for ct images. Nan Fang Yi Ke Da Xue Xue Bao. 43 (6), 985-993 (2023).
- Ma, J., et al. Abdomenct-1k: Is abdominal organ segmentation a solved problem. IEEE Trans Pattern Anal Mach Intell. 44 (10), 6695-6714 (2021).
- Galván, E., Mooney, P. Neuroevolution in deep neural networks: Current trends and future challenges. IEEE Trans Artif Intell. 2 (6), 476-493 (2021).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。