Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Swin-PSAxialNet: Verimli Bir Çoklu Organ Segmentasyon Tekniği
Bu Makalede
Özet
Mevcut protokol, önceki segmentasyon yöntemlerine kıyasla mükemmel doğruluk elde eden Swin-PSAxialNet adlı verimli bir çoklu organ segmentasyon yöntemini açıklamaktadır. Bu prosedürün temel adımları arasında veri kümesi toplama, ortam yapılandırması, veri ön işleme, model eğitimi ve karşılaştırması ve ablasyon deneyleri yer alır.
Özet
Abdominal çoklu organ segmentasyonu, tıbbi görüntü analizi alanındaki en önemli konulardan biridir ve hastalık teşhisi ve tedavi planlaması gibi klinik iş akışlarını desteklemede önemli bir rol oynar. Bu çalışmada, nnU-Net mimarisine dayalı Swin-PSAxialNet adı verilen verimli bir çoklu organ segmentasyon yöntemi önerilmiştir. BT görüntülerinde 11 abdominal organın hassas segmentasyonu için özel olarak tasarlanmıştır. Önerilen ağ, nnU-Net'e kıyasla aşağıdaki iyileştirmeleri yapmıştır. İlk olarak, Boşluktan derinliğe (SPD) modülleri ve parametre paylaşımlı eksenel dikkat (PSAA) özellik çıkarma blokları tanıtıldı ve 3D görüntü özelliği çıkarma yeteneği geliştirildi. İkinci olarak, ayrıntılı bilgileri ve uzamsal özellikleri yakalamak için çok ölçekli bir görüntü füzyon yaklaşımı kullanıldı ve ince özellikleri ve kenar özelliklerini çıkarma yeteneği geliştirildi. Son olarak, modelin hesaplama maliyetini ve eğitim hızını azaltmak için bir parametre paylaşım yöntemi tanıtıldı. Önerilen ağ, 11 organı içeren segmentasyon görevi için ortalama 0.93342 Zar katsayısına ulaşır. Deneysel sonuçlar, Swin-PSAxialNet'in önceki ana segmentasyon yöntemlerine göre kayda değer üstünlüğünü göstermektedir. Yöntem, ana karın organlarının segmentlere ayrılmasında mükemmel doğruluk ve düşük hesaplama maliyetleri gösterir.
Giriş
Hastalıkların teşhisi, tedavi planlarının formülasyonu ve tedavi sonuçlarının izlenmesi dahil olmak üzere çağdaş klinik müdahale, tıbbi görüntülerin doğru segmentasyonuna dayanır1. Bununla birlikte, abdominal organlar arasındaki karmaşık yapısal ilişkiler2, birden fazla abdominal organın3 doğru segmentasyonunu elde etmeyi zorlu bir görev haline getirir. Son birkaç on yılda, tıbbi görüntüleme ve bilgisayarla görmedeki gelişmeler, abdominal çoklu organ segmentasyonu alanında hem yeni fırsatlar hem de zorluklar sunmuştur. Gelişmiş Manyetik Rezonans Görüntüleme (MRG)4 ve Bilgisayarlı Tomografi (BT)teknolojisi5 yüksek çözünürlüklü karın görüntüleri elde etmemizi sağlar. BT görüntülerinden birden fazla organın kesin segmentasyonu, karaciğer, böbrekler, dalak, pankreas vb. gibi hayati organların değerlendirilmesi ve tedavisi için önemli bir klinik değere sahiptir.6,7,8,9,10 Bununla birlikte, bu anatomik yapıların, özellikle radyologların veya radyasyon onkologlarının müdahalesini gerektirenlerin manuel olarak açıklanması hem zaman alıcıdır hem de öznel etkilere karşı hassastır11. Bu nedenle, abdominal çoklu organ segmentasyonu için otomatik ve doğru yöntemlerin geliştirilmesine acil ihtiyaç vardır.
Görüntü segmentasyonu ile ilgili önceki araştırmalar, ağırlıklı olarak, katmanları istifleyerek ve ResNet12'yi tanıtarak segmentasyon verimliliğini artıran Evrişimli Sinir Ağlarına (CNN'ler) dayanıyordu. 2020'de Google araştırma ekibi, bir dizi görsel görev için Transformer mimarisini geleneksel görsel alana dahil etmenin öncü bir örneği olan Vision Transformer (VIT) model13'ü tanıttı14. Evrişimli işlemler yalnızca yerel özellik bilgilerini düşünebilirken, Transformers'daki dikkat mekanizması, küresel özellik bilgilerinin kapsamlı bir şekilde değerlendirilmesini sağlar.
Transformer tabanlı mimarilerin geleneksel evrişimli ağlara15 göre üstünlüğü göz önüne alındığında, çok sayıda araştırma ekibi, Transformers'ın güçlü yönleri ile evrişimli ağlar arasındaki sinerjiyi optimize etmek için kapsamlı araştırmalar yapmıştır 16,17,18,19. Chen ve arkadaşları, görüntülerden global özellikler çıkarmak için Transformers'tan yararlanan tıbbi görüntü segmentasyon görevleri16 için TransUNet'i tanıttı. Ağ eğitiminin yüksek maliyeti ve özellik çıkarma hiyerarşisi kavramının kullanılamaması nedeniyle, Transformer'ın avantajları tam olarak gerçekleştirilememiştir.
Bu sorunları ele almak için, birçok araştırmacı, segmentasyon ağlarını eğitmek için omurga olarak Transformers'ı dahil etmeyi denemeye başladı. Liu ve ark.17 , katmanlı öznitelik çıkarımı için hiyerarşik bir yapı yöntemi kullanan Swin Transformatörü'nü tanıttı. Windows Çok Kafalı Kendi Kendine Dikkat (W-MSA) kavramı önerildi ve özellikle daha büyük sığ düzey özellik haritalarının varlığında hesaplama maliyetini önemli ölçüde azalttı. Bu yaklaşım hesaplama gereksinimlerini azaltırken, aynı zamanda farklı pencereler arasında bilgi aktarımını da izole etti. Bu sorunu çözmek için yazarlar, bitişik pencereler arasında bilgi yayılımını sağlayan Kaydırılmış Windows Çok Başlı Kendi Kendine Dikkat (SW-MSA) kavramını daha da tanıttılar. Bu metodoloji üzerine inşa edilen Cao ve ark. Swin-UNet18'i formüle etti, U-Net'teki 2D evrişimleri Swin modülleriyle değiştirdi ve W-MSA ve SW-MSA'yı kodlama ve kod çözme süreçlerine dahil ederek övgüye değer segmentasyon sonuçları elde etti.
Tersine, Zhou ve ark. yüksek çözünürlüklü görüntüleri işlerken conv işleminin avantajının göz ardı edilemeyeceğini vurguladı19. Önerdikleri nnFormer, çapraz şekilli bir yapı ile karakterize edilen bir Transformatör modeli oluşturan yerel üç boyutlu görüntü bloklarına dayalı bir öz dikkat hesaplama yöntemi kullanır. Yerel üç boyutlu bloklara dayalı dikkatin kullanılması, ağdaki eğitim yükünü önemli ölçüde azalttı.
Yukarıdaki çalışma ile ilgili sorunlar göz önüne alındığında, Swin-PSAxialNet olarak adlandırılan 3D tıbbi görüntü segmentasyonu için verimli bir hibrit hiyerarşik yapı önerilmiştir. Bu yöntem, küresel bilgileri 20 çıkarabilen bir altörnekleme bloğu olan Boşluktan derinliğe (SPD)21 içerir. Ek olarak, öğrenme parametresi sayısını ikinci dereceden doğrusala düşüren ve ağ eğitiminin doğruluğu ve eğitim modellerinin karmaşıklığı üzerinde iyi bir etkiye sahip olacak bir parametre paylaşılan eksenel dikkat (PSAA) modülü ekler22.
Swin-PSAxialNet ağı
Ağın genel mimarisi, kodlayıcı ve kod çözücü yapılarından oluşan nnU-Net23'ün U şeklindeki yapısını benimser. Bu yapılar, Şekil 1'de gösterildiği gibi, büyük ve küçük ölçekli görüntülerden yerel özellik çıkarma ve özelliklerin birleştirilmesi ile ilgilenir.
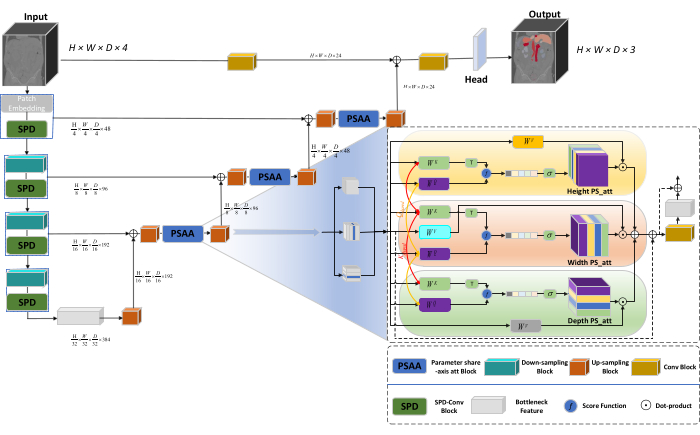
Şekil 1: Ağ mimarisinin Swin-PSAxialNet şematik diyagramı. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Enkoder yapısında, geleneksel Conv bloğu, bir altörnekleme hacmi oluşturmak için SPD bloğu20 ile birleştirilir. Kodlayıcının ilk katmanı, 3B verileri 3B yamalara 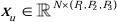 bölen bir modül olan Yama Gömmeyi içerir, (P1, P2, P3) bu bağlamda örtüşmeyen yamaları temsil eder,
bölen bir modül olan Yama Gömmeyi içerir, (P1, P2, P3) bu bağlamda örtüşmeyen yamaları temsil eder, 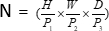 3B yamaların sıra uzunluğunu belirtir. Gömme katmanını takiben, bir sonraki adım, hem evrişimli bir blok hem de bir SPD bloğu içeren, örtüşmeyen bir evrişimli altörnekleme birimini içerir. Bu kurulumda, evrişimli bloğun adımı 1 olarak ayarlanmıştır ve SPD bloğu görüntü ölçekleme için kullanılır, bu da çözünürlükte dört kat azalmaya ve kanallarda iki kat artışa yol açar.
3B yamaların sıra uzunluğunu belirtir. Gömme katmanını takiben, bir sonraki adım, hem evrişimli bir blok hem de bir SPD bloğu içeren, örtüşmeyen bir evrişimli altörnekleme birimini içerir. Bu kurulumda, evrişimli bloğun adımı 1 olarak ayarlanmıştır ve SPD bloğu görüntü ölçekleme için kullanılır, bu da çözünürlükte dört kat azalmaya ve kanallarda iki kat artışa yol açar.
Kod çözücü yapısında, Darboğaz Özelliği katmanından sonraki her yukarı örnekleme bloğu, bir yukarı örnekleme bloğu ve bir PSAA bloğunun bir kombinasyonundan oluşur. Özellik haritasının çözünürlüğü iki kat artırılır ve her bir kod çözücü aşaması çifti arasında kanal sayısı yarıya indirilir. Uzamsal bilgileri geri yüklemek ve özellik temsilini geliştirmek için, yukarı örnekleme blokları arasında büyük ve küçük ölçekli görüntüler arasında özellik füzyonu gerçekleştirilir. Sonuç olarak, yukarı örnekleme sonuçları, orijinal görüntü boyutunu geri yüklemek için (H × W × D × C, C = 3) çıktı boyutuyla Head katmanına beslenir.
SPD blok mimarisi
Geleneksel yöntemlerde, altörnekleme bölümü, adım boyutu 2 olan tek bir adım kullanır. Bu, görüntüdeki yerel konumlarda evrişimli havuzlamayı, alıcı alanı sınırlamayı ve modeli küçük görüntü yamalarından özelliklerin çıkarılmasıyla sınırlamayı içerir. Bu yöntem, orijinal görüntüyü ince bir şekilde üç boyuta bölen SPD bloğunu kullanır. Orijinal 3D görüntü, x, y ve z eksenleri boyunca eşit olarak bölümlere ayrılır ve bu da dört alt hacim gövdesiyle sonuçlanır. (Şekil 2) Daha sonra, dört hacim "kedi" işlemiyle birleştirilir ve elde edilen görüntü, aşağı örneklenmiş görüntüyü20 elde etmek için 1 × 1 × 1 evrişime tabi tutulur.
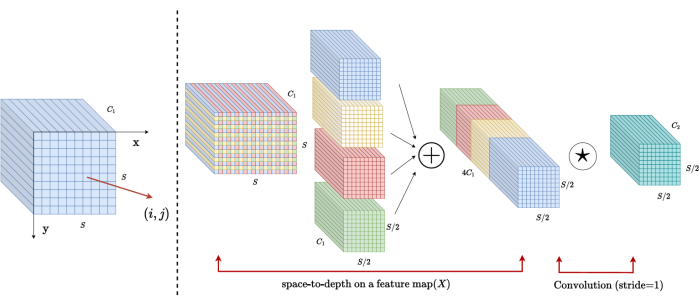
Şekil 2: SPD blok diyagramı. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
PSAA blok mimarisi
Geleneksel CNN ağlarının aksine, önerilen PSAA bloğu, küresel bilgi odağı yürütmede daha etkili ve ağ öğrenimi ve eğitiminde daha verimlidir. Bu, daha zengin görüntülerin ve uzamsal özelliklerin yakalanmasını sağlar. PSAA bloğu, üç boyutta parametre paylaşımına dayalı eksenel dikkat öğrenimini içerir: Yükseklik, Genişlik ve Derinlik. Görüntüdeki her piksel için dikkat öğrenimi gerçekleştiren geleneksel dikkat mekanizmasıyla karşılaştırıldığında, bu yöntem, üç boyutun her biri için bağımsız olarak dikkat öğrenimi yürütür ve kendi kendine dikkatin karmaşıklığını ikinci dereceden doğrusala azaltır. Ayrıca, öğrenilebilir bir anahtar-sorgu parametre paylaşım mekanizması mekanizması kullanılır, bu da ağın dikkat mekanizması işlemlerini üç boyutta paralel olarak gerçekleştirmesini sağlayarak daha hızlı, üstün ve daha etkili özellik gösterimi sağlar.
Protokol
Bu protokol Nantong Üniversitesi Etik Kurulu tarafından onaylanmıştır. Yapay zeka teknolojisini kullanarak insan tıbbi görüntüleri, uzuv hareketleri ve vasküler görüntüleme dahil olmak üzere elde edilen non-invaziv veya minimal invaziv multimodal verilerin akıllı değerlendirmesini ve araştırılmasını içerir. Şekil 3 , çoklu organ segmentasyonunun genel akış şemasını göstermektedir. Gerekli tüm web bağlantıları Malzeme Tablosunda verilmiştir.
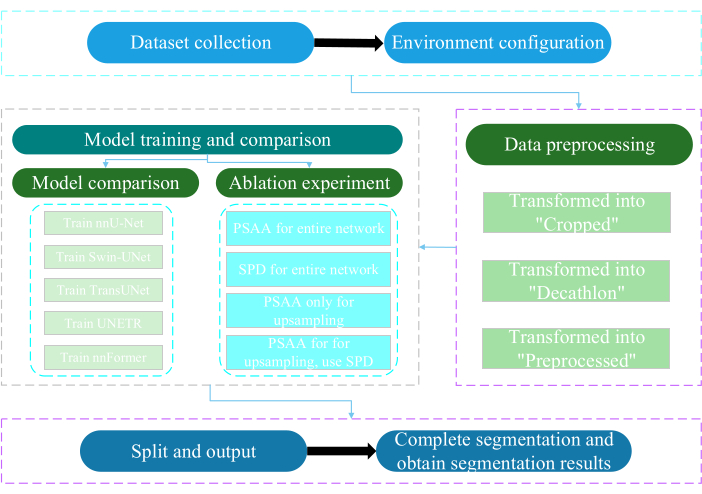
Şekil 3: Çok organlı segmentasyonun genel akış şeması. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
1. Veri kümesi toplama
- AMOS2022 veri setini24 indirin (bkz. Malzeme Tablosu).
- Eğitim veri kümesi olarak 160 görüntü ve test veri kümesi olarak 40 görüntü seçin.
- Terminalde mkdir ~/PaddleSeg/contrib/MedicalSeg/data/raw_data yürütün.
- raw_data yolunu açın.
- Veri setini raw_data dizinine yerleştirin ve "imagesTr" ve "labelsTr" dizinlerindeki dosya adlarıyla doğru yazışmaları sağlayın.
- dataset.json dosyası oluşturun.
- numTraining, numTest ve etiket adı gibi veri kümesi bilgilerini "dataset.json" dosyasına ekleyin.
NOT: Bu çalışmanın veri seti, eğitim ve test amacıyla AMOS2022 resmi 500 CT veri setinden seçilen 200 yüksek kaliteli CT veri setinden oluşmaktadır. Veri seti, dalak, sağ böbrek, sol böbrek, safra kesesi, yemek borusu, karaciğer, mide, aort, inferior vena kava, pankreas ve mesane dahil olmak üzere 11 organı kapsar.
2. Ortam yapılandırması
- Paddlepaddle ortamını ( Malzeme Tablosuna bakın) ve ilgili CUDA sürüm25'i kurun.
NOT: Paddlepaddle sürüm 2.5.0 ve CUDA sürüm 11.7'nin kullanılması önerilir. - Git, PaddleSeg deposunu klonlar25.
- Terminalde cd ~/PaddleSeg/contrib/MedicalSeg/ komutunu çalıştırın.
- pip install -r requirements.txt komutunu çalıştırın.
- Pip install medpy komutunu çalıştırın.
3. Veri ön işleme
- data_root, batch_size, model, train_dataset, val_dataset, optimize edici, lr_scheduler ve kayıp gibi parametreler dahil olmak üzere "yml" dosyasını değiştirin.
- "-config, --log_iters, --precision, --nnunet, --save_dir, --save_interval vb. dahil olmak üzere yürütme için gerekli parametreleri belirtin.
NOT: Orijinal "raw_data", üç tür veri dosyasına dönüştürülür: "Kırpılmış", "Decathlon" ve "Önceden İşlenmiş", model performansını ve eğitim etkinliğini artırmak için farklı eğitim ve değerlendirme gereksinimlerini karşılar.
4. Model eğitimi ve karşılaştırma
NOT: Görüntü segmentasyonu alanında yaygın olarak kullanılan bir temel olan nnU-Net23 , çalışmada bir temel model olarak hizmet vermektedir. Spesifik model karşılaştırma süreci aşağıdaki gibidir.
- nnU-Net'i eğitin
- 2 ve 80.000 yinelemelik toplu iş boyutuyla yapılandırın, her 20 yinelemede bir günlüğe kaydedin ve fp16 hassasiyetinden yararlanın.
- save_dir'yi çalışma dizini içinde "çıktı" klasörü olarak ayarlayın ve kaydetme aralığı 1.000 yineleme olarak ayarlayın.
- VisualDL parametre görselleştirmesi için --use_vdl parametresini ekleyin.
- train.py çalıştırın.
- Dört yml dosyası oluşturun, 1-4 arasındaki katları belirtin ve modeli beş katlı çapraz doğrulama ile eğitin.
- Swin-UNet'i eğitin
- Önceden belirlenmiş deneysel koşullarla tutarlılığı koruyun.
- --nnunet parametresini kaldırın ve --swinunet kullanın.
- train.py dosyasını çalıştırın.
- Tren 1-4'ü ayrı ayrı katlar ve ardından topluluk işlemeye tabi tutar.
- TransUNet'i Eğitin
- Adım 4.1'de belirtilen prosedürleri izleyin.
- Yml dosyasını açın.
- type: nnunet'i şu şekilde değiştirin: transunet.
- --nnunet parametresini --transunet olarak değiştirin.
- train.py dosyasını çalıştırın.
- Tren 1-4'ü ayrı ayrı katlar ve topluluk işlemeyi işler.
- UNETR treni
- Tutarlı deneysel değişkenleri koruyun.
- Swin-UNet ve TransUNet (adım 4.2 ve adım 4.3) gibi daha önce belirtilen prosedürleri tekrarlayın.
- train.py çalıştırın.
- Tren 1-4'ü ayrı ayrı katlar ve topluluk işlemeyi işler.
- nnFormer treni
- Swin-UNet ve TransUNet gibi parametreleri değiştirin.
- Tek kat train.py çalıştırın.
- Topluluk işlemeyi çalıştırın.
5. Ablasyon deneyi
- Tüm ağ için PSAA bloğu
- Değişkenleri adım 4.1 ile tutarlı tutun.
- volume_shape = (64,192,192) ve Axial_attention = Doğru olarak ayarlayın.
- Tek kat train.py çalıştırın.
- Beş kat boyunca topluluk işlemeyi işleyin.
- Tüm ağ için SPD bloğu
- Değişkenleri adım 4.1 ile tutarlı tutun.
- SPD_enable = Doğru ve Axial_attention = Yanlış olarak ayarlayın.
- Tek kat train.py çalıştırın.
- Beş kat boyunca topluluk işlemeyi işleyin.
- Yukarı örnekleme için PSAA bloğu
NOT: Altörnekleme için SPD bloğu kullanmayın.- Yukarı örnekleme için PSAA bloğunu kullanın.
- Değişkenleri adım 4.1 ile tutarlı tutun.
- Altörnekleme kısmında Axial_attention = False olarak ayarlayın ve SPD_enable = False olarak ayarlayın.
- Tek kat train.py çalıştırın.
- Beş kat topluluk işlemeyi işleyin.
- Yukarı örnekleme için PSAA bloğu
NOT: Altörnekleme için SPD bloğunu kullanın.- Altörnekleme için SPD bloğunu ve yukarı örnekleme için PSAA bloğunu kullanın.
- Değişkenleri adım 4.1 ile tutarlı tutun.
- Altörnekleme kısmında Axial_attention = False olarak ayarlayın ve SPD_enable = True olarak ayarlayın.
- Tek kat train.py çalıştırın.
- Beş kat topluluk işlemeyi işleyin.
- Üstün sonuçlar nedeniyle yineleme sayısını 2,00,000 olarak ayarlayın.
Sonuçlar
Bu protokol, modeli değerlendirmek için iki metrik kullanır: Zar Benzerlik Skoru (DSC) ve %95 Hausdorff Mesafesi (HD95). DSC, voksel segmentasyon tahminleri ile temel doğruluk arasındaki örtüşmeyi ölçerken, %95 HD, voksel segmentasyon tahmin sınırları ile temel gerçek arasındaki örtüşmeyi değerlendirir ve aykırı değerlerin %5'ini filtreler. DSC26'nın tanımı aşağıdaki gibidir:
Tartışmalar
Abdominal organların segmentasyonu karmaşık bir iştir. Beyin veya kalp gibi insan vücudunun diğer iç yapılarıyla karşılaştırıldığında, BT görüntülerindeki düşük kontrast ve büyük şekil değişiklikleri nedeniyle karın organlarını segmentlere ayırmak daha zor görünmektedir27,28. Swin-PSAxialNet burada bu zor sorunu çözmek için önerilmiştir.
Veri toplama adımında, b...
Açıklamalar
Yazarlar herhangi bir çıkar çatışması beyan etmemektedir.
Teşekkürler
Bu çalışma, bu çalışmanın başarısında katkıları paha biçilmez olan Jiangsu Eyaleti '333' Mühendislik Projesi ([2022]21-003), Wuxi Sağlık Komisyonu Genel Programı (M202205) ve Wuxi Bilim ve Teknoloji Geliştirme Fonu (Y20212002-1) tarafından desteklenmiştir." Yazarlar, destekleri için tüm araştırma görevlilerine ve çalışma katılımcılarına teşekkür eder.
Malzemeler
| Name | Company | Catalog Number | Comments |
| AMOS2022 dataset | None | None | Datasets for network training and testing. The weblink is: https://pan.baidu.com/s/1x2ZW5FiZtVap0er55Wk4VQ?pwd=xhpb |
| ASUS mainframe | ASUS | https://www.asusparts.eu/en/asus-13020-01910200 | |
| CUDA version 11.7 | NVIDIA | https://developer.nvidia.com/cuda-11-7-0-download-archive | |
| NVIDIA GeForce RTX 3090 | NVIDIA | https://www.nvidia.com/en-in/geforce/graphics-cards/30-series/rtx-3090-3090ti/ | |
| Paddlepaddle environment | Baidu | None | Environmental preparation for network training. The weblink is: https://www.paddlepaddle.org.cn/ |
| PaddleSeg | Baidu | None | The baseline we use: https://github.com/PaddlePaddle/PaddleSeg |
Referanslar
- Liu, X., Song, L., Liu, S., Zhang, Y. A review of deep-learning-based medical image segmentation methods. Sustain. 13 (3), 1224 (2021).
- Popilock, R., Sandrasagaren, K., Harris, L., Kaser, K. A. CT artifact recognition for the nuclear technologist. J Nucl Med Technol. 36 (2), 79-81 (2008).
- Rehman, A., Khan, F. G. A deep learning based review on abdominal images. Multimed Tools Appl. 80, 30321-30352 (2021).
- Schenck, J. F. The role of magnetic susceptibility in magnetic resonance imaging: Mri magnetic compatibility of the first and second kinds. Med Phys. 23 (6), 815-850 (1996).
- Palmisano, A., et al. Myocardial late contrast enhancement ct in troponin-positive acute chest pain syndrome. Radiol. 302 (3), 545-553 (2022).
- Chen, E. -. L., Chung, P. -. C., Chen, C. -. L., Tsai, H. -. M., Chang, C. -. I. An automatic diagnostic system for CT liver image classification. IEEE Trans Biomed Eng. 45 (6), 783-794 (1998).
- Sagel, S. S., Stanley, R. J., Levitt, R. G., Geisse, G. J. R. Computed tomography of the kidney. Radiol. 124 (2), 359-370 (1977).
- Freeman, J. L., Jafri, S., Roberts, J. L., Mezwa, D. G., Shirkhoda, A. CT of congenital and acquired abnormalities of the spleen. Radiographics. 13 (3), 597-610 (1993).
- Almeida, R. R., et al. Advances in pancreatic CT imaging. AJR Am J Roentgenol. 211 (1), 52-66 (2018).
- Eisen, G. M., et al. Guidelines for credentialing and granting privileges for endoscopic ultrasound. Gastrointest Endosc. 54 (6), 811-814 (2001).
- Sykes, J. Reflections on the current status of commercial automated segmentation systems in clinical practice. J Med Radiat Sci. 61 (3), 131-134 (2014).
- He, K., Zhang, X., Ren, S., Sun, J. Deep residual learning for image recognition. 2016 IEEE Conference on Computer Vision and Pattern Recognition (CVPR). , 770-778 (2016).
- Dosovitskiy, A., et al. An image is worth 16x16 words: Transformers for image recognition at scale. arXiv. arXiv. , 11929 (2020).
- Ramachandran, P., et al. Stand-alone self-attention in vision models. Adv Neural Inf Process Syst. 32, (2019).
- Vaswani, A., et al. Attention is all you need. Adv Neural Inf Process Syst. 30, (2017).
- Chen, J., et al. TransUNET: Transformers make strong encoders for medical image segmentation. arXiv. , 04396 (2021).
- Liu, Z., et al. Swin transformer: Hierarchical vision transformer using shifted windows. Proc IEEE Int Conf Comput Vis. , 10012-10022 (2021).
- Cao, H., et al. Swin-UNET: Unet-like pure transformer for medical image segmentation. Comput Vis ECCV. , 205-218 (2022).
- Zhou, H. -. Y., et al. nnformer: Interleaved transformer for volumetric segmentation. arXiv. arXiv. , 03201 (2021).
- Sunkara, R., Luo, T. No more strided convolutions or pooling: A new CNN building block for low-resolution images and small objects. Mach Learn Knowl Discov Databases. , 443-459 (2023).
- Çiçek, &. #. 2. 1. 4. ;., Abdulkadir, A., Lienkamp, S. S., Brox, T., Ronneberger, O. 3D U-Net: learning dense volumetric segmentation from sparse annotation. Med Comput Vis Bayesian Graph Models Biomed Imaging. 2016, 424-432 (2016).
- Kim, H., et al. Abdominal multi-organ auto-segmentation using 3D-patch-based deep convolutional neural network. Scientific reports. 10 (1), 6204 (2020).
- Isensee, F., et al. nnu-net: Self-adapting framework for u-net-based medical image segmentation. arXiv. , 10486 (2018).
- Ji, Y., et al. A large-scale abdominal multi-organ benchmark for versatile medical image segmentation. Adv Neural Inf Process Syst. 35, 36722-36732 (2022).
- Liu, Y., et al. Paddleseg: A high-efficient development toolkit for image segmentation. arXiv. , 06175 (2021).
- Hatamizadeh, A., et al. UNETR: Transformers for 3D medical image segmentation. Proc IEEE Int Conf Comput Vis. , 574-584 (2022).
- Zhou, H., Zeng, D., Bian, Z., Ma, J. A semi-supervised network-based tissue-aware contrast enhancement method for ct images. Nan Fang Yi Ke Da Xue Xue Bao. 43 (6), 985-993 (2023).
- Ma, J., et al. Abdomenct-1k: Is abdominal organ segmentation a solved problem. IEEE Trans Pattern Anal Mach Intell. 44 (10), 6695-6714 (2021).
- Galván, E., Mooney, P. Neuroevolution in deep neural networks: Current trends and future challenges. IEEE Trans Artif Intell. 2 (6), 476-493 (2021).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır