È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Swin-PSAxialNet: un'efficiente tecnica di segmentazione multiorgano
In questo articolo
Riepilogo
Il presente protocollo descrive un efficiente metodo di segmentazione multiorgano chiamato Swin-PSAxialNet, che ha raggiunto un'eccellente accuratezza rispetto ai precedenti metodi di segmentazione. I passaggi chiave di questa procedura includono la raccolta di set di dati, la configurazione dell'ambiente, la pre-elaborazione dei dati, l'addestramento e il confronto dei modelli e gli esperimenti di ablazione.
Abstract
La segmentazione multiorgano addominale è uno degli argomenti più importanti nel campo dell'analisi delle immagini mediche e svolge un ruolo importante nel supportare i flussi di lavoro clinici come la diagnosi della malattia e la pianificazione del trattamento. In questo studio, viene proposto un efficiente metodo di segmentazione multiorgano chiamato Swin-PSAxialNet basato sull'architettura nnU-Net. È stato progettato specificamente per la segmentazione precisa di 11 organi addominali nelle immagini TC. La rete proposta ha apportato i seguenti miglioramenti rispetto a nnU-Net. In primo luogo, sono stati introdotti i moduli SPD (Space-to-depth) e i blocchi di estrazione delle caratteristiche dell'attenzione assiale condivisa con parametri (PSAA), migliorando la capacità di estrazione delle caratteristiche delle immagini 3D. In secondo luogo, è stato impiegato un approccio di fusione di immagini multiscala per acquisire informazioni dettagliate e caratteristiche spaziali, migliorando la capacità di estrarre caratteristiche sottili e caratteristiche di bordo. Infine, è stato introdotto un metodo di condivisione dei parametri per ridurre il costo computazionale del modello e la velocità di addestramento. La rete proposta raggiunge un coefficiente medio di Dice di 0,93342 per il compito di segmentazione che coinvolge 11 organi. I risultati sperimentali indicano la notevole superiorità di Swin-PSAxialNet rispetto ai precedenti metodi di segmentazione tradizionali. Il metodo mostra un'eccellente accuratezza e bassi costi computazionali nella segmentazione dei principali organi addominali.
Introduzione
L'intervento clinico contemporaneo, compresa la diagnosi delle malattie, la formulazione dei piani di trattamento e il monitoraggio dei risultati del trattamento, si basa sull'accurata segmentazione delle immagini mediche1. Tuttavia, le complesse relazioni strutturali tra gli organi addominali2rendono un compito impegnativo ottenere una segmentazione accurata di più organi addominali3. Negli ultimi decenni, i fiorenti sviluppi nell'imaging medico e nella visione artificiale hanno presentato nuove opportunità e sfide nel campo della segmentazione multiorgano addominale. La risonanza magnetica avanzata (MRI)4 e la tomografia computerizzata (TC)5 ci consentono di acquisire immagini addominali ad alta risoluzione. La segmentazione precisa di più organi dalle immagini TC ha un valore clinico significativo per la valutazione e il trattamento di organi vitali come fegato, reni, milza, pancreas, ecc.6,7,8,9,10 Tuttavia, l'annotazione manuale di queste strutture anatomiche, in particolare quelle che richiedono l'intervento di radiologi o radioterapisti, richiede tempo ed è suscettibile di influenze soggettive11. Pertanto, vi è un urgente bisogno di sviluppare metodi automatizzati e accurati per la segmentazione multiorgano addominale.
Le ricerche precedenti sulla segmentazione delle immagini si sono basate prevalentemente sulle reti neurali convoluzionali (CNN), che migliorano l'efficienza della segmentazione impilando i livelli e introducendo ResNet12. Nel 2020, il team di ricerca di Google ha introdotto il modello Vision Transformer (VIT)13, segnando un esempio pionieristico di incorporazione dell'architettura Transformer nel dominio visivo tradizionale per una serie di attività visive14. Mentre le operazioni convoluzionali possono contemplare solo le informazioni sulle caratteristiche locali, il meccanismo di attenzione in Transformers consente la considerazione completa delle informazioni sulle funzionalità globali.
Considerando la superiorità delle architetture basate su Transformer rispetto alle tradizionali reti convoluzionali15, numerosi team di ricerca hanno intrapreso un'ampia esplorazione per ottimizzare la sinergia tra i punti di forza dei Transformers e le reti convoluzionali 16,17,18,19. Chen et al. hanno introdotto il TransUNet per le attività di segmentazione delle immagini mediche16, che sfrutta i trasformatori per estrarre caratteristiche globali dalle immagini. A causa dell'elevato costo della formazione della rete e dell'incapacità di utilizzare il concetto di gerarchia di estrazione delle caratteristiche, i vantaggi di Transformer non sono stati pienamente realizzati.
Per affrontare questi problemi, molti ricercatori hanno iniziato a sperimentare l'incorporazione dei trasformatori come spina dorsale per l'addestramento delle reti di segmentazione. Liu et al.17 hanno introdotto il trasformatore Swin, che impiegava un metodo di costruzione gerarchico per l'estrazione di caratteristiche stratificate. È stato proposto il concetto di Windows Multi-Head Self-Attention (W-MSA), che riduce significativamente i costi computazionali, in particolare in presenza di mappe di funzionalità di livello superficiale più grandi. Se da un lato questo approccio ha ridotto i requisiti computazionali, dall'altro ha isolato la trasmissione delle informazioni tra diverse finestre. Per risolvere questo problema, gli autori hanno ulteriormente introdotto il concetto di Shifted Windows Multi-Head Self-Attention (SW-MSA), che consente la propagazione delle informazioni tra finestre adiacenti. Basandosi su questa metodologia, Cao et al. hanno formulato lo Swin-UNet18, sostituendo le circonvoluzioni 2D in U-Net con i moduli Swin e incorporando W-MSA e SW-MSA nei processi di codifica e decodifica, ottenendo risultati di segmentazione lodevoli.
Al contrario, Zhou et al. hanno evidenziato che il vantaggio del funzionamento conv non può essere ignorato durante l'elaborazione di immagini ad alta risoluzione19. Il loro proposto nnFormer impiega un metodo di calcolo dell'auto-attenzione basato su blocchi di immagini tridimensionali locali, costituendo un modello Transformer caratterizzato da una struttura a forma di croce. L'utilizzo dell'attenzione basata su blocchi tridimensionali locali ha ridotto significativamente il carico di formazione sulla rete.
Dati i problemi con lo studio di cui sopra, viene proposta un'efficiente struttura gerarchica ibrida per la segmentazione di immagini mediche 3D, denominata Swin-PSAxialNet. Questo metodo incorpora un blocco di downsampling, il blocco Space-to-depth (SPD)20 , in grado di estrarre informazioni globali21. Inoltre, aggiunge un modulo PSAA (Parameter Shared Axial Attention), che riduce il numero di parametri di apprendimento da quadratico a lineare e avrà un buon effetto sull'accuratezza dell'addestramento della rete e sulla complessità dei modelli di addestramento22.
Rete Swin-PSAxialNet
L'architettura complessiva della rete adotta la struttura a forma di U di nnU-Net23, costituita da strutture di encoder e decoder. Queste strutture si occupano dell'estrazione di feature locali e della concatenazione di feature da immagini di grandi e piccole dimensioni, come illustrato nella Figura 1.
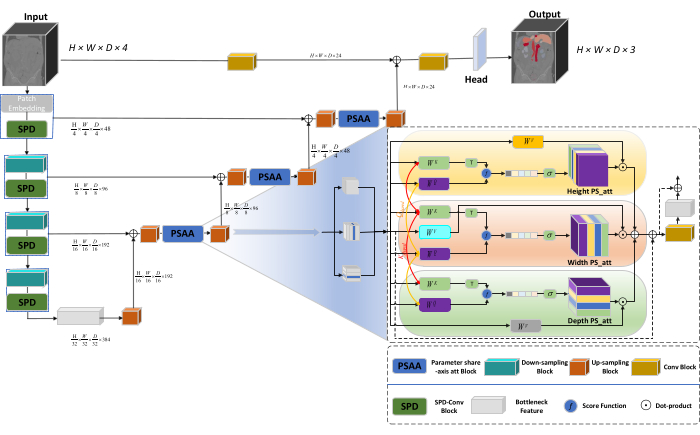
Figura 1: Diagramma schematico dell'architettura di rete di Swin-PSAxialNet. Fare clic qui per visualizzare una versione più grande di questa figura.
Nella struttura dell'encoder, il tradizionale blocco Conv è combinato con il blocco SPD20 per formare un volume di downsampling. Il primo livello dell'encoder incorpora Patch Embedding, un modulo che partiziona i dati 3D in patch 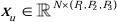 3D, (P1, P2, P3) rappresenta patch non sovrapposte in questo contesto,
3D, (P1, P2, P3) rappresenta patch non sovrapposte in questo contesto, 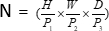 indica la lunghezza della sequenza di patch 3D. Dopo lo strato di inclusione, la fase successiva prevede un'unità di downsampling convoluzionale non sovrapposta che comprende sia un blocco convoluzionale che un blocco SPD. In questa configurazione, il blocco convoluzionale ha uno stride impostato su 1 e il blocco SPD viene impiegato per il ridimensionamento dell'immagine, portando a una riduzione di quattro volte della risoluzione e a un aumento di due volte dei canali.
indica la lunghezza della sequenza di patch 3D. Dopo lo strato di inclusione, la fase successiva prevede un'unità di downsampling convoluzionale non sovrapposta che comprende sia un blocco convoluzionale che un blocco SPD. In questa configurazione, il blocco convoluzionale ha uno stride impostato su 1 e il blocco SPD viene impiegato per il ridimensionamento dell'immagine, portando a una riduzione di quattro volte della risoluzione e a un aumento di due volte dei canali.
Nella struttura del decoder, ogni blocco di upsampling dopo il livello Bottleneck Feature è costituito da una combinazione di un blocco di upsampling e di un blocco PSAA. La risoluzione della mappa delle caratteristiche è raddoppiata e il numero di canali è dimezzato tra ogni coppia di stadi del decoder. Per ripristinare le informazioni spaziali e migliorare la rappresentazione delle caratteristiche, viene eseguita la fusione delle caratteristiche tra immagini su larga e piccola scala tra i blocchi di upsampling. Infine, i risultati dell'upsampling vengono inseriti nel livello Head per ripristinare le dimensioni originali dell'immagine, con una dimensione di output di (H × W × D × C, C = 3).
Architettura a blocchi SPD
Nei metodi tradizionali, la sezione di sottocampionamento impiega un singolo passo con una dimensione del passo di 2. Ciò comporta il pooling convoluzionale in posizioni locali nell'immagine, limitando il campo ricettivo e confinando il modello all'estrazione di caratteristiche da piccole patch dell'immagine. Questo metodo utilizza il blocco SPD, che divide finemente l'immagine originale in tre dimensioni. L'immagine 3D originale viene segmentata uniformemente lungo gli assi x, y e z, ottenendo quattro corpi di sottovolume. (Figura 2) Successivamente, i quattro volumi vengono concatenati attraverso l'operazione "cat" e l'immagine risultante subisce una convoluzione 1 × 1 × 1 per ottenere l'immagine sottocampionata20.
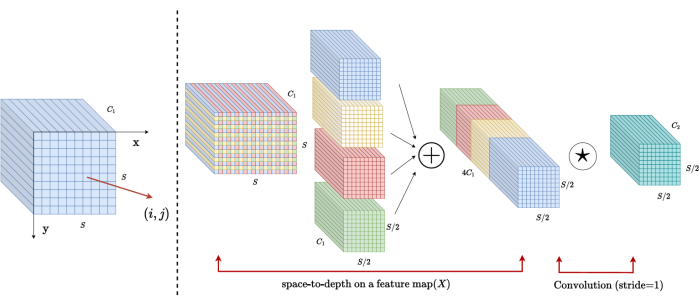
Figura 2: Diagramma a blocchi SPD. Fare clic qui per visualizzare una versione più grande di questa figura.
Architettura a blocchi PSAA
A differenza delle tradizionali reti CNN, il blocco PSAA proposto è più efficace nel condurre un focus sull'informazione globale e più efficiente nell'apprendimento e nella formazione della rete. Ciò consente l'acquisizione di immagini e caratteristiche spaziali più ricche. Il blocco PSAA include l'apprendimento dell'attenzione assiale basato sulla condivisione di parametri in tre dimensioni: altezza, larghezza e profondità. Rispetto al meccanismo di attenzione convenzionale che esegue l'apprendimento dell'attenzione per ogni pixel nell'immagine, questo metodo conduce in modo indipendente l'apprendimento dell'attenzione per ciascuna delle tre dimensioni, riducendo la complessità dell'auto-attenzione da quadratica a lineare. Inoltre, viene impiegato un meccanismo di condivisione dei parametri key-queries apprendibile, che consente alla rete di eseguire operazioni del meccanismo di attenzione in parallelo attraverso le tre dimensioni, ottenendo una rappresentazione delle caratteristiche più veloce, superiore ed efficace.
Protocollo
Il presente protocollo è stato approvato dal Comitato Etico dell'Università di Nantong. Implica la valutazione intelligente e la ricerca di dati multimodali acquisiti non invasivi o minimamente invasivi, tra cui immagini mediche umane, movimenti degli arti e imaging vascolare, utilizzando la tecnologia dell'intelligenza artificiale. La Figura 3 illustra il diagramma di flusso complessivo della segmentazione multiorgano. Tutti i collegamenti web necessari sono forniti nella Tabella dei Materiali.
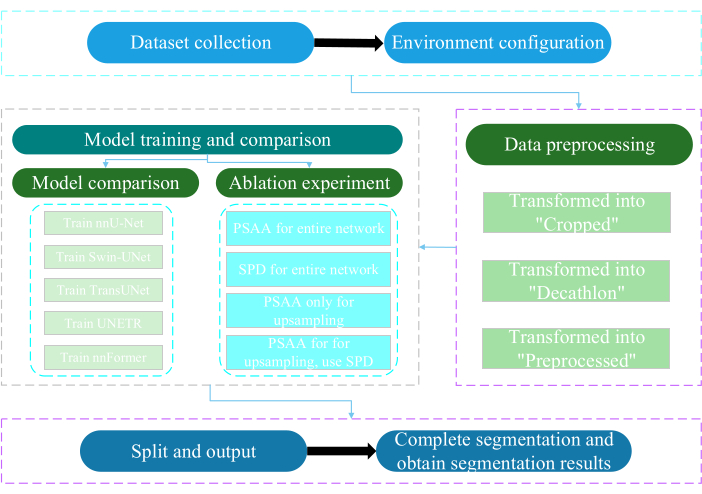
Figura 3: Diagramma di flusso generale della segmentazione multiorgano. Fare clic qui per visualizzare una versione più grande di questa figura.
1. Raccolta di set di dati
- Scarica il set di dati AMOS202224 (vedi Tabella dei materiali).
- Selezionare 160 immagini come set di dati di training e 40 immagini come set di dati di test.
- Esegue terminalmente mkdir ~/PaddleSeg/contrib/MedicalSeg/data/raw_data.
- Apri il percorso di raw_data.
- Posizionare il set di dati nella directory raw_data e assicurarsi che corrisponda correttamente ai nomi dei file nelle directory "imagesTr" e "labelsTr".
- Creare dataset.json file.
- Aggiungere informazioni sul set di dati, ad esempio numTraining, numTest e nome delle etichette , nel file "dataset.json".
NOTA: Il set di dati per questo studio è costituito da 200 set di dati CT di alta qualità selezionati dal set di dati ufficiale AMOS2022 500 CT per scopi di formazione e test. Il set di dati comprende 11 organi, tra cui milza, rene destro, rene sinistro, cistifellea, esofago, fegato, stomaco, aorta, vena cava inferiore, pancreas e vescica.
2. Configurazione dell'ambiente
- Installare l'ambiente Paddlepaddle (vedi Tabella dei materiali) e la corrispondente versioneCUDA 25.
NOTA: Si consiglia di utilizzare Paddlepaddle versione 2.5.0 e CUDA versione 11.7. - Git clona il repository PaddleSeg25.
- Esecuzione terminale cd ~/PaddleSeg/contrib/MedicalSeg/.
- Esegui pip install -r requirements.txt.
- Esegui pip install medpy.
3. Pretrattamento dei dati
- Modifica il file "yml", inclusi parametri come data_root, batch_size, modello, train_dataset, val_dataset, ottimizzatore, lr_scheduler e perdita.
- Specifica i parametri richiesti per l'esecuzione, inclusi "-config, --log_iters, --precision, --nnunet, --save_dir, --save_interval, ecc.
NOTA: Il "raw_data" originale viene trasformato in tre tipi di file di dati: "Ritagliato", "Decathlon" e "Pre-elaborato", che soddisfano diversi requisiti di formazione e valutazione per migliorare le prestazioni del modello e l'efficacia dell'addestramento.
4. Addestramento e confronto dei modelli
NOTA: Come linea di base ampiamente utilizzata nel campo della segmentazione delle immagini, nnU-Net23 funge da modello di base nello studio. Il processo di confronto dei modelli specifico è il seguente.
- Treno nnU-Net
- Configura con una dimensione del batch di 2 e 80.000 iterazioni, registra ogni 20 iterazioni e utilizza la precisione fp16.
- Impostate il save_dir come cartella "output" all'interno della directory di lavoro, con un intervallo di salvataggio impostato a 1.000 iterazioni.
- Aggiungere il parametro --use_vdl per la visualizzazione dei parametri VisualDL.
- Esegui train.py.
- Crea quattro file yml , specifica le pieghe 1-4 e addestra il modello con la convalida incrociata a cinque pieghe.
- Treno Swin-UNet
- Mantenere la coerenza con le condizioni sperimentali precedentemente stabilite.
- Rimuovi il parametro --nnunet e usa --swinunet.
- Esegui il file train.py.
- Il treno piega 1-4 individualmente e poi soggetto a elaborazione d'insieme.
- Treno TransUNet
- Seguire le procedure descritte nel passaggio 4.1.
- Apri il file yml .
- Modificare type: nnunet in type: transunet.
- Modificare il parametro --nnunet in --transunet.
- Eseguire il file train.py.
- Treno piega da 1 a 4 individualmente ed elabora l'elaborazione dell'insieme.
- Treno UNETR
- Mantenere variabili sperimentali coerenti.
- Ripetere le procedure descritte in precedenza come Swin-UNet e TransUNet (passaggio 4.2 e passaggio 4.3).
- Esegui train.py.
- Treno piega da 1 a 4 individualmente ed elabora l'elaborazione dell'insieme.
- Treno nnFormer
- Modifica i parametri come Swin-UNet e TransUNet.
- Esegui una piega singola train.py.
- Eseguire l'elaborazione dell'ensemble.
5. Esperimento di ablazione
- Blocco PSAA per l'intera rete
- Mantieni le variabili coerenti con il passaggio 4.1.
- Imposta volume_shape = (64,192,192) e Axial_attention = Vero.
- Esegui una piega singola train.py.
- Elabora l'elaborazione dell'ensemble su cinque pieghe.
- Blocco SPD per l'intera rete
- Mantieni le variabili coerenti con il passaggio 4.1.
- Imposta SPD_enable = True e Axial_attention = False.
- Esegui una piega singola train.py.
- Elabora l'elaborazione dell'ensemble su cinque pieghe.
- Blocco PSAA per l'upsampling
NOTA: Non utilizzare il blocco SPD per il downsampling.- Utilizzare il blocco PSAA per l'upsampling.
- Mantieni le variabili coerenti con il passaggio 4.1.
- Impostare Axial_attention = False nella parte di downsampling e SPD_enable = False.
- Esegui una piega singola train.py.
- Elabora cinque volte l'elaborazione dell'insieme.
- Blocco PSAA per l'upsampling
NOTA: Utilizzare il blocco SPD per il downsampling.- Utilizzare il blocco SPD per il downsampling e il blocco PSAA per l'upsampling.
- Mantieni le variabili coerenti con il passaggio 4.1.
- Impostare Axial_attention = False nella parte di downsampling e SPD_enable = True.
- Esegui una piega singola train.py.
- Elabora cinque volte l'elaborazione dell'insieme.
- Impostare il numero di iterazioni su 2.00.000 a causa dei risultati superiori.
Risultati
Questo protocollo utilizza due metriche per valutare il modello: il punteggio di somiglianza dei dadi (DSC) e la distanza di Hausdorff del 95% (HD95). Il DSC misura la sovrapposizione tra le previsioni di segmentazione dei voxel e la verità di base, mentre il 95% HD valuta la sovrapposizione tra i limiti di previsione della segmentazione dei voxel e la verità di base, filtrando il 5% dei valori anomali. La definizione di DSC26 è la seguente:
Discussione
La segmentazione degli organi addominali è un lavoro complicato. Rispetto ad altre strutture interne del corpo umano, come il cervello o il cuore, la segmentazione degli organi addominali sembra più impegnativa a causa del basso contrasto e dei grandi cambiamenti di forma nelle immagini TC27,28. Swin-PSAxialNet è proposto qui per risolvere questo difficile problema.
Nella fase di raccolta dei dati, ...
Divulgazioni
Gli autori dichiarano di non avere conflitti di interesse.
Riconoscimenti
Questo studio è stato sostenuto dal progetto di ingegneria '333' della provincia di Jiangsu ([2022]21-003), dal Wuxi Health Commission General Program (M202205) e dal Wuxi Science and Technology Development Fund (Y20212002-1), i cui contributi sono stati inestimabili per il successo di questo lavoro". Gli autori ringraziano tutti gli assistenti di ricerca e i partecipanti allo studio per il loro sostegno.
Materiali
| Name | Company | Catalog Number | Comments |
| AMOS2022 dataset | None | None | Datasets for network training and testing. The weblink is: https://pan.baidu.com/s/1x2ZW5FiZtVap0er55Wk4VQ?pwd=xhpb |
| ASUS mainframe | ASUS | https://www.asusparts.eu/en/asus-13020-01910200 | |
| CUDA version 11.7 | NVIDIA | https://developer.nvidia.com/cuda-11-7-0-download-archive | |
| NVIDIA GeForce RTX 3090 | NVIDIA | https://www.nvidia.com/en-in/geforce/graphics-cards/30-series/rtx-3090-3090ti/ | |
| Paddlepaddle environment | Baidu | None | Environmental preparation for network training. The weblink is: https://www.paddlepaddle.org.cn/ |
| PaddleSeg | Baidu | None | The baseline we use: https://github.com/PaddlePaddle/PaddleSeg |
Riferimenti
- Liu, X., Song, L., Liu, S., Zhang, Y. A review of deep-learning-based medical image segmentation methods. Sustain. 13 (3), 1224 (2021).
- Popilock, R., Sandrasagaren, K., Harris, L., Kaser, K. A. CT artifact recognition for the nuclear technologist. J Nucl Med Technol. 36 (2), 79-81 (2008).
- Rehman, A., Khan, F. G. A deep learning based review on abdominal images. Multimed Tools Appl. 80, 30321-30352 (2021).
- Schenck, J. F. The role of magnetic susceptibility in magnetic resonance imaging: Mri magnetic compatibility of the first and second kinds. Med Phys. 23 (6), 815-850 (1996).
- Palmisano, A., et al. Myocardial late contrast enhancement ct in troponin-positive acute chest pain syndrome. Radiol. 302 (3), 545-553 (2022).
- Chen, E. -. L., Chung, P. -. C., Chen, C. -. L., Tsai, H. -. M., Chang, C. -. I. An automatic diagnostic system for CT liver image classification. IEEE Trans Biomed Eng. 45 (6), 783-794 (1998).
- Sagel, S. S., Stanley, R. J., Levitt, R. G., Geisse, G. J. R. Computed tomography of the kidney. Radiol. 124 (2), 359-370 (1977).
- Freeman, J. L., Jafri, S., Roberts, J. L., Mezwa, D. G., Shirkhoda, A. CT of congenital and acquired abnormalities of the spleen. Radiographics. 13 (3), 597-610 (1993).
- Almeida, R. R., et al. Advances in pancreatic CT imaging. AJR Am J Roentgenol. 211 (1), 52-66 (2018).
- Eisen, G. M., et al. Guidelines for credentialing and granting privileges for endoscopic ultrasound. Gastrointest Endosc. 54 (6), 811-814 (2001).
- Sykes, J. Reflections on the current status of commercial automated segmentation systems in clinical practice. J Med Radiat Sci. 61 (3), 131-134 (2014).
- He, K., Zhang, X., Ren, S., Sun, J. Deep residual learning for image recognition. 2016 IEEE Conference on Computer Vision and Pattern Recognition (CVPR). , 770-778 (2016).
- Dosovitskiy, A., et al. An image is worth 16x16 words: Transformers for image recognition at scale. arXiv. arXiv. , 11929 (2020).
- Ramachandran, P., et al. Stand-alone self-attention in vision models. Adv Neural Inf Process Syst. 32, (2019).
- Vaswani, A., et al. Attention is all you need. Adv Neural Inf Process Syst. 30, (2017).
- Chen, J., et al. TransUNET: Transformers make strong encoders for medical image segmentation. arXiv. , 04396 (2021).
- Liu, Z., et al. Swin transformer: Hierarchical vision transformer using shifted windows. Proc IEEE Int Conf Comput Vis. , 10012-10022 (2021).
- Cao, H., et al. Swin-UNET: Unet-like pure transformer for medical image segmentation. Comput Vis ECCV. , 205-218 (2022).
- Zhou, H. -. Y., et al. nnformer: Interleaved transformer for volumetric segmentation. arXiv. arXiv. , 03201 (2021).
- Sunkara, R., Luo, T. No more strided convolutions or pooling: A new CNN building block for low-resolution images and small objects. Mach Learn Knowl Discov Databases. , 443-459 (2023).
- Çiçek, &. #. 2. 1. 4. ;., Abdulkadir, A., Lienkamp, S. S., Brox, T., Ronneberger, O. 3D U-Net: learning dense volumetric segmentation from sparse annotation. Med Comput Vis Bayesian Graph Models Biomed Imaging. 2016, 424-432 (2016).
- Kim, H., et al. Abdominal multi-organ auto-segmentation using 3D-patch-based deep convolutional neural network. Scientific reports. 10 (1), 6204 (2020).
- Isensee, F., et al. nnu-net: Self-adapting framework for u-net-based medical image segmentation. arXiv. , 10486 (2018).
- Ji, Y., et al. A large-scale abdominal multi-organ benchmark for versatile medical image segmentation. Adv Neural Inf Process Syst. 35, 36722-36732 (2022).
- Liu, Y., et al. Paddleseg: A high-efficient development toolkit for image segmentation. arXiv. , 06175 (2021).
- Hatamizadeh, A., et al. UNETR: Transformers for 3D medical image segmentation. Proc IEEE Int Conf Comput Vis. , 574-584 (2022).
- Zhou, H., Zeng, D., Bian, Z., Ma, J. A semi-supervised network-based tissue-aware contrast enhancement method for ct images. Nan Fang Yi Ke Da Xue Xue Bao. 43 (6), 985-993 (2023).
- Ma, J., et al. Abdomenct-1k: Is abdominal organ segmentation a solved problem. IEEE Trans Pattern Anal Mach Intell. 44 (10), 6695-6714 (2021).
- Galván, E., Mooney, P. Neuroevolution in deep neural networks: Current trends and future challenges. IEEE Trans Artif Intell. 2 (6), 476-493 (2021).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon