Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Swin-PSAxialNet: Una Técnica Eficiente de Segmentación Multi-Orgánica
En este artículo
Resumen
El presente protocolo describe un método eficiente de segmentación multi-órgano llamado Swin-PSAxialNet, que ha logrado una excelente precisión en comparación con los métodos de segmentación anteriores. Los pasos clave de este procedimiento incluyen la recopilación de conjuntos de datos, la configuración del entorno, el preprocesamiento de datos, el entrenamiento y la comparación de modelos y los experimentos de ablación.
Resumen
La segmentación multiorgánica abdominal es uno de los temas más importantes en el campo del análisis de imágenes médicas y desempeña un papel importante en el apoyo a los flujos de trabajo clínicos, como el diagnóstico de enfermedades y la planificación de tratamientos. En este estudio se propone un método eficiente de segmentación multi-órgano denominado Swin-PSAxialNet basado en la arquitectura nnU-Net. Fue diseñado específicamente para la segmentación precisa de 11 órganos abdominales en imágenes de TC. La red propuesta ha realizado las siguientes mejoras en comparación con nnU-Net. En primer lugar, se introdujeron los módulos de espacio-profundidad (SPD) y los bloques de extracción de características de atención axial compartida por parámetros (PSAA), lo que mejoró la capacidad de extracción de características de imágenes 3D. En segundo lugar, se empleó un enfoque de fusión de imágenes a múltiples escalas para capturar información detallada y características espaciales, mejorando la capacidad de extraer características sutiles y características de borde. Por último, se introdujo un método de compartición de parámetros para reducir el coste computacional del modelo y la velocidad de entrenamiento. La red propuesta alcanza un coeficiente de Dice promedio de 0.93342 para la tarea de segmentación que involucra a 11 órganos. Los resultados experimentales indican la notable superioridad de Swin-PSAxialNet sobre los métodos de segmentación convencionales anteriores. El método muestra una excelente precisión y bajos costos computacionales en la segmentación de los principales órganos abdominales.
Introducción
La intervención clínica contemporánea, que incluye el diagnóstico de enfermedades, la formulación de planes de tratamiento y el seguimiento de los resultados del tratamiento, se basa en la segmentación precisa de las imágenes médicas1. Sin embargo, las complejas relaciones estructurales entre los órganos abdominales2hacen que sea una tarea desafiante lograr una segmentación precisa de múltiples órganos abdominales3. En las últimas décadas, los florecientes desarrollos en imágenes médicas y visión por computadora han presentado nuevas oportunidades y desafíos en el campo de la segmentación multiorgánica abdominal. La tecnología avanzada de Resonancia Magnética (RM)4 y Tomografía Computarizada (TC)5 nos permite obtener imágenes abdominales de alta resolución. La segmentación precisa de múltiples órganos a partir de imágenes de TC tiene un valor clínico significativo para la evaluación y el tratamiento de órganos vitales como el hígado, los riñones, el bazo, el páncreas, etc.6,7,8,9,10 Sin embargo, la anotación manual de estas estructuras anatómicas, especialmente aquellas que requieren la intervención de radiólogos u oncólogos radioterápicos, requiere mucho tiempo y es susceptible a influencias subjetivas11. Por lo tanto, existe una necesidad urgente de desarrollar métodos automatizados y precisos para la segmentación multiorgánica abdominal.
Las investigaciones anteriores sobre la segmentación de imágenes se basaron principalmente en las redes neuronales convolucionales (CNN), que mejoran la eficiencia de la segmentación mediante el apilamiento de capas y la introducción de ResNet12. En 2020, el equipo de investigación de Google presentó el modelo Vision Transformer (VIT)13, marcando un ejemplo pionero de incorporación de la arquitectura Transformer en el dominio visual tradicional para una variedad de tareas visuales14. Mientras que las operaciones convolucionales solo pueden contemplar la información de las características locales, el mecanismo de atención de Transformers permite la consideración integral de la información de las características globales.
Teniendo en cuenta la superioridad de las arquitecturas basadas en transformadores sobre las redes convolucionales tradicionales15, numerosos equipos de investigación han emprendido una amplia exploración para optimizar la sinergia entre las fortalezas de los transformadores y las redes convolucionales 16,17,18,19. Chen et al. introdujeron el TransUNet para tareas de segmentación de imágenes médicas16, que aprovecha Transformers para extraer características globales de las imágenes. Debido al alto costo de la capacitación de la red y a la falta de utilización del concepto de jerarquía de extracción de características, las ventajas de Transformer no se han aprovechado por completo.
Para abordar estos problemas, muchos investigadores han comenzado a experimentar con la incorporación de Transformers como columna vertebral para entrenar redes de segmentación. Liu et al.17 introdujeron el transformador Swin, que empleaba un método de construcción jerárquica para la extracción de características en capas. Se propuso el concepto de autoatención multicabezal de Windows (W-MSA), lo que reduce significativamente el costo computacional, particularmente en presencia de mapas de características de nivel superficial más grandes. Si bien este enfoque redujo los requisitos computacionales, también aisló la transmisión de información entre diferentes ventanas. Para abordar este problema, los autores introdujeron el concepto de autoatención multicabezal de ventanas desplazadas (SW-MSA), que permite la propagación de información entre ventanas adyacentes. Sobre la base de esta metodología, Cao et al. formularon el Swin-UNet18, reemplazando las convoluciones 2D en U-Net con módulos Swin e incorporando W-MSA y SW-MSA en los procesos de codificación y decodificación, logrando resultados de segmentación encomiables.
Por el contrario, Zhou et al. destacaron que la ventaja de la operación conv no podía ignorarse cuando se procesaban imágenes de alta resolución19. Su propuesta nnFormer emplea un método de cálculo de autoatención basado en bloques de imágenes tridimensionales locales, constituyendo un modelo Transformer caracterizado por una estructura en forma de cruz. La utilización de la atención basada en bloques tridimensionales locales redujo significativamente la carga de entrenamiento en la red.
Dados los problemas con el estudio anterior, se propone una estructura jerárquica híbrida eficiente para la segmentación de imágenes médicas en 3D, denominada Swin-PSAxialNet. Este método incorpora un bloque de downmuestreo, el bloque Space-to-depth (SPD)20 , capaz de extraer información global21. Además, agrega un módulo de atención axial compartida de parámetros (PSAA), que reduce el recuento de parámetros de aprendizaje de cuadrático a lineal y tendrá un buen efecto en la precisión del entrenamiento de la red y la complejidad de los modelos de entrenamiento22.
Red Swin-PSAxialNet
La arquitectura general de la red adopta la estructura en forma de U de nnU-Net23, que consta de estructuras de codificador y decodificador. Estas estructuras participan en la extracción de características locales y la concatenación de características a partir de imágenes a gran y pequeña escala, como se ilustra en la Figura 1.
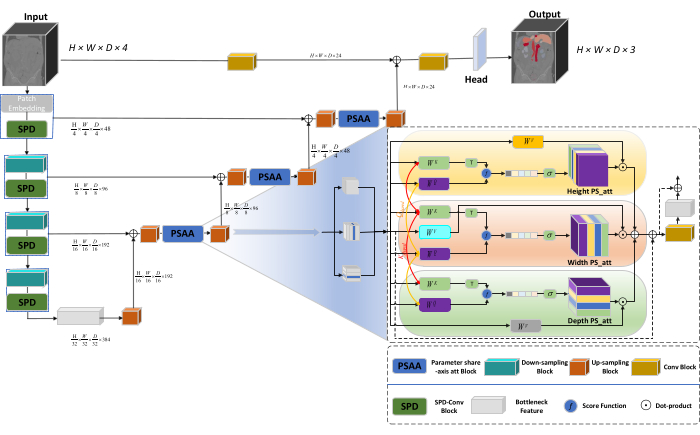
Figura 1: Diagrama esquemático de la arquitectura de red de Swin-PSAxialNet. Haga clic aquí para ver una versión más grande de esta figura.
En la estructura del codificador, el bloque Conv tradicional se combina con el bloque SPD20 para formar un volumen de muestreo descendente. La primera capa del codificador incorpora Patch Embedding, un módulo que divide los datos 3D en parches 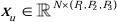 3D, (P1, P2, P3) representa parches no superpuestos en este contexto,
3D, (P1, P2, P3) representa parches no superpuestos en este contexto, 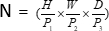 significa la longitud de la secuencia de parches 3D. Después de la capa de incrustación, el siguiente paso implica una unidad de submuestreo convolucional no superpuesta que comprende un bloque convolucional y un bloque SPD. En esta configuración, el bloque convolucional tiene un paso establecido en 1, y el bloque SPD se emplea para el escalado de la imagen, lo que lleva a una reducción de cuatro veces en la resolución y un aumento del doble en los canales.
significa la longitud de la secuencia de parches 3D. Después de la capa de incrustación, el siguiente paso implica una unidad de submuestreo convolucional no superpuesta que comprende un bloque convolucional y un bloque SPD. En esta configuración, el bloque convolucional tiene un paso establecido en 1, y el bloque SPD se emplea para el escalado de la imagen, lo que lleva a una reducción de cuatro veces en la resolución y un aumento del doble en los canales.
En la estructura del descodificador, cada bloque de sobremuestreo después de la capa de entidad de cuello de botella consta de una combinación de un bloque de sobremuestreo y un bloque PSAA. La resolución del mapa de características se duplica y el número de canales se reduce a la mitad entre cada par de etapas del decodificador. Para restaurar la información espacial y mejorar la representación de características, se realiza la fusión de características entre imágenes a gran y pequeña escala entre los bloques de sobremuestreo. En última instancia, los resultados del sobremuestreo se introducen en la capa Head para restaurar el tamaño de la imagen original, con un tamaño de salida de (H × W × D × C, C = 3).
Arquitectura de bloques SPD
En los métodos tradicionales, la sección de reducción de muestreo emplea un solo paso con un tamaño de paso de 2. Esto implica la agrupación convolucional en posiciones locales de la imagen, lo que limita el campo receptivo y limita el modelo a la extracción de características de pequeños parches de imagen. Este método utiliza el bloque SPD, que divide finamente la imagen original en tres dimensiones. La imagen 3D original está segmentada uniformemente a lo largo de los ejes x, y y z, lo que da como resultado cuatro cuerpos de subvolumen. (Figura 2) Posteriormente, los cuatro volúmenes se concatenan mediante la operación "gato", y la imagen resultante se somete a una convolución 1 × 1 × 1 para obtener la imagen submuestreada20.
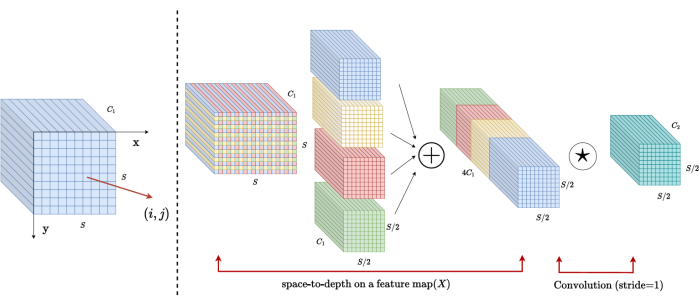
Figura 2: Diagrama de bloques SPD. Haga clic aquí para ver una versión más grande de esta figura.
Arquitectura de bloques PSAA
A diferencia de las redes tradicionales de CNN, el bloque PSAA propuesto es más eficaz para llevar a cabo un enfoque de información global y más eficiente en el aprendizaje y la capacitación de la red. Esto permite la captura de imágenes y características espaciales más ricas. El bloque PSAA incluye el aprendizaje de la atención axial basado en el intercambio de parámetros en tres dimensiones: Altura, Anchura y Profundidad. En comparación con el mecanismo de atención convencional que realiza el aprendizaje de la atención para cada píxel de la imagen, este método lleva a cabo de forma independiente el aprendizaje de la atención para cada una de las tres dimensiones, reduciendo la complejidad de la autoatención de cuadrática a lineal. Además, se emplea un mecanismo de intercambio de parámetros de consultas de claves aprendible, lo que permite a la red realizar operaciones de mecanismo de atención en paralelo en las tres dimensiones, lo que da como resultado una representación de características más rápida, superior y efectiva.
Protocolo
El presente protocolo fue aprobado por el Comité de Ética de la Universidad de Nantong. Implica la evaluación e investigación inteligente de datos multimodales adquiridos no invasivos o mínimamente invasivos, incluidas imágenes médicas humanas, movimientos de extremidades e imágenes vasculares, utilizando tecnología de inteligencia artificial. La Figura 3 muestra el diagrama de flujo general de la segmentación multiorgánica. Todos los enlaces web necesarios se proporcionan en la Tabla de Materiales.
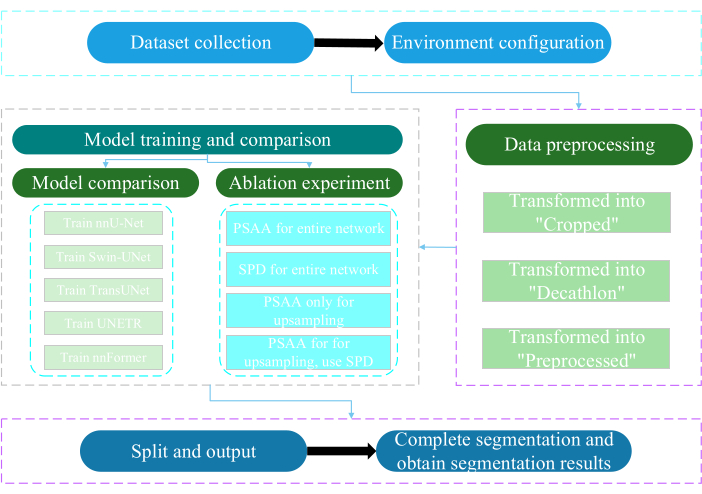
Figura 3: Diagrama de flujo general de la segmentación multiorgánica. Haga clic aquí para ver una versión más grande de esta figura.
1. Recopilación de conjuntos de datos
- Descargue el conjunto de datos AMOS202224 (consulte Tabla de materiales).
- Seleccione 160 imágenes como conjunto de datos de entrenamiento y 40 imágenes como conjunto de datos de prueba.
- Ejecute terminalmente mkdir ~/PaddleSeg/contrib/MedicalSeg/data/raw_data.
- Abre el camino de raw_data.
- Coloque el conjunto de datos en el directorio raw_data y asegúrese de que coincida correctamente con los nombres de archivo de los directorios "imagesTr" y "labelsTr".
- Cree dataset.json archivo.
- Agregue información del conjunto de datos, como numTraining, numTest y el nombre de las etiquetas en el archivo "dataset.json".
NOTA: El conjunto de datos para este estudio consta de 200 conjuntos de datos de TC de alta calidad seleccionados del AMOS2022 conjunto de datos oficial de 500 CT con fines de entrenamiento y pruebas. El conjunto de datos abarca 11 órganos, incluidos el bazo, el riñón derecho, el riñón izquierdo, la vesícula biliar, el esófago, el hígado, el estómago, la aorta, la vena cava inferior, el páncreas y la vejiga.
2. Configuración del entorno
- Instale el entorno Paddlepaddle (ver Tabla de Materiales) y la versión25 de CUDA correspondiente.
NOTA: Se recomienda utilizar la versión 2.5.0 de Paddlepaddle y la versión 11.7 de CUDA. - Git clona el repositorio PaddleSeg25.
- Ejecute terminalmente cd ~/PaddleSeg/contrib/MedicalSeg/.
- Ejecute pip install -r requirements.txt.
- Ejecute pip install medpy.
3. Pretratamiento de datos
- Modifique el archivo "yml", incluidos parámetros como data_root, batch_size, modelo, train_dataset, val_dataset, optimizador, lr_scheduler y pérdida.
- Especifique los parámetros necesarios para la ejecución, incluidos "-config, --log_iters, --precision, --nnunet, --save_dir, --save_interval, etc.
NOTA: El "raw_data" original se transforma en tres tipos de archivos de datos: "Recortado", "Decathlon" y "Preprocesado", que satisfacen diferentes requisitos de capacitación y evaluación para mejorar el rendimiento del modelo y la efectividad de la capacitación.
4. Entrenamiento y comparación de modelos
NOTA: Como línea de base ampliamente utilizada en el campo de la segmentación de imágenes, nnU-Net23 sirve como modelo de referencia en el estudio. El proceso específico de comparación de modelos es el siguiente.
- Tren nnU-Net
- Configure con un tamaño de lote de 2 y 80.000 iteraciones, registre cada 20 iteraciones y utilice la precisión fp16.
- Establezca el save_dir como la carpeta de "salida" dentro del directorio de trabajo, con un intervalo de guardado establecido en 1.000 iteraciones.
- Add parameter --use_vdl para la visualización de parámetros de VisualDL.
- Ejecute train.py.
- Cree cuatro archivos yml , especifique los pliegues 1-4 y entrene el modelo con una validación cruzada quíntuple.
- Tren Swin-UNet
- Mantener la coherencia con las condiciones experimentales previamente establecidas.
- Elimine el parámetro --nnunet y use --swinunet.
- Ejecute el archivo train.py.
- El tren se pliega 1-4 individualmente y luego se somete al procesamiento del conjunto.
- Tren TransUNet
- Siga los procedimientos descritos en el paso 4.1.
- Abra el archivo yml .
- Modifique el tipo: nnunet al tipo: transunet.
- Modifique el parámetro --nnunet a --transunet.
- Ejecute el archivo train.py.
- Entrene los pliegues 1-4 individualmente y procese el procesamiento del conjunto.
- Tren UNETR
- Mantener la consistencia de las variables experimentales.
- Repita los procedimientos descritos anteriormente como Swin-UNet y TransUNet (paso 4.2 y paso 4.3).
- Ejecute train.py.
- Entrene los pliegues 1-4 individualmente y procese el procesamiento del conjunto.
- Tren nnFormer
- Modifique los parámetros como Swin-UNet y TransUNet.
- Ejecute train.py de pliegue único.
- Ejecute el procesamiento de conjuntos.
5. Experimento de ablación
- Bloque PSAA para toda la red
- Mantenga las variables coherentes con el paso 4.1.
- Establezca volume_shape = (64,192,192) y Axial_attention = Verdadero.
- Ejecute train.py de pliegue único.
- Procese el procesamiento de conjuntos en cinco pliegues.
- Bloque SPD para toda la red
- Mantenga las variables coherentes con el paso 4.1.
- Establezca SPD_enable = verdadero y Axial_attention = falso.
- Ejecute train.py de pliegue único.
- Procese el procesamiento de conjuntos en cinco pliegues.
- Bloque PSAA para sobremuestreo
NOTA: No utilice el bloque SPD para reducir el muestreo.- Utilice el bloque PSAA para el muestreo.
- Mantenga las variables coherentes con el paso 4.1.
- Establezca Axial_attention = Falso en la parte de reducción de resolución y SPD_enable = Falso.
- Ejecute train.py de pliegue único.
- Procesamiento de ensamble de cinco pliegues.
- Bloque PSAA para sobremuestreo
NOTA: Utilice el bloque SPD para reducir el muestreo.- Utilice el bloque SPD para reducir el muestreo y el bloque PSAA para el aumento de muestreo.
- Mantenga las variables coherentes con el paso 4.1.
- Establezca Axial_attention = Falso en la parte de reducción de resolución y SPD_enable = Verdadero.
- Ejecute train.py de pliegue único.
- Procesamiento de ensamble de cinco pliegues.
- Establezca el número de iteraciones en 2.00.000 debido a los resultados superiores.
Resultados
Este protocolo emplea dos métricas para evaluar el modelo: Puntuación de similitud de dados (DSC) y Distancia de Hausdorff del 95% (HD95). DSC mide la superposición entre las predicciones de segmentación de vóxeles y la realidad del terreno, mientras que el HD del 95 % evalúa la superposición entre los límites de la predicción de la segmentación de vóxeles y la realidad del terreno, filtrando el 5 % de los valores atípicos. La definición de DSC26 es l...
Discusión
La segmentación de los órganos abdominales es un trabajo complicado. En comparación con otras estructuras internas del cuerpo humano, como el cerebro o el corazón, la segmentación de los órganos abdominales parece más difícil debido al bajo contraste y a los grandes cambios de forma en las imágenes de TC27,28. Swin-PSAxialNet se propone aquí para resolver este difícil problema.
En la etapa d...
Divulgaciones
Los autores declaran no tener conflictos de intereses.
Agradecimientos
Este estudio contó con el apoyo del Proyecto de Ingeniería '333' de la provincia de Jiangsu ([2022]21-003), el Programa General de la Comisión de Salud de Wuxi (M202205) y el Fondo de Desarrollo de Ciencia y Tecnología de Wuxi (Y20212002-1), cuyas contribuciones han sido invaluables para el éxito de este trabajo". Los autores agradecen a todos los asistentes de investigación y participantes en el estudio por su apoyo.
Materiales
| Name | Company | Catalog Number | Comments |
| AMOS2022 dataset | None | None | Datasets for network training and testing. The weblink is: https://pan.baidu.com/s/1x2ZW5FiZtVap0er55Wk4VQ?pwd=xhpb |
| ASUS mainframe | ASUS | https://www.asusparts.eu/en/asus-13020-01910200 | |
| CUDA version 11.7 | NVIDIA | https://developer.nvidia.com/cuda-11-7-0-download-archive | |
| NVIDIA GeForce RTX 3090 | NVIDIA | https://www.nvidia.com/en-in/geforce/graphics-cards/30-series/rtx-3090-3090ti/ | |
| Paddlepaddle environment | Baidu | None | Environmental preparation for network training. The weblink is: https://www.paddlepaddle.org.cn/ |
| PaddleSeg | Baidu | None | The baseline we use: https://github.com/PaddlePaddle/PaddleSeg |
Referencias
- Liu, X., Song, L., Liu, S., Zhang, Y. A review of deep-learning-based medical image segmentation methods. Sustain. 13 (3), 1224 (2021).
- Popilock, R., Sandrasagaren, K., Harris, L., Kaser, K. A. CT artifact recognition for the nuclear technologist. J Nucl Med Technol. 36 (2), 79-81 (2008).
- Rehman, A., Khan, F. G. A deep learning based review on abdominal images. Multimed Tools Appl. 80, 30321-30352 (2021).
- Schenck, J. F. The role of magnetic susceptibility in magnetic resonance imaging: Mri magnetic compatibility of the first and second kinds. Med Phys. 23 (6), 815-850 (1996).
- Palmisano, A., et al. Myocardial late contrast enhancement ct in troponin-positive acute chest pain syndrome. Radiol. 302 (3), 545-553 (2022).
- Chen, E. -. L., Chung, P. -. C., Chen, C. -. L., Tsai, H. -. M., Chang, C. -. I. An automatic diagnostic system for CT liver image classification. IEEE Trans Biomed Eng. 45 (6), 783-794 (1998).
- Sagel, S. S., Stanley, R. J., Levitt, R. G., Geisse, G. J. R. Computed tomography of the kidney. Radiol. 124 (2), 359-370 (1977).
- Freeman, J. L., Jafri, S., Roberts, J. L., Mezwa, D. G., Shirkhoda, A. CT of congenital and acquired abnormalities of the spleen. Radiographics. 13 (3), 597-610 (1993).
- Almeida, R. R., et al. Advances in pancreatic CT imaging. AJR Am J Roentgenol. 211 (1), 52-66 (2018).
- Eisen, G. M., et al. Guidelines for credentialing and granting privileges for endoscopic ultrasound. Gastrointest Endosc. 54 (6), 811-814 (2001).
- Sykes, J. Reflections on the current status of commercial automated segmentation systems in clinical practice. J Med Radiat Sci. 61 (3), 131-134 (2014).
- He, K., Zhang, X., Ren, S., Sun, J. Deep residual learning for image recognition. 2016 IEEE Conference on Computer Vision and Pattern Recognition (CVPR). , 770-778 (2016).
- Dosovitskiy, A., et al. An image is worth 16x16 words: Transformers for image recognition at scale. arXiv. arXiv. , 11929 (2020).
- Ramachandran, P., et al. Stand-alone self-attention in vision models. Adv Neural Inf Process Syst. 32, (2019).
- Vaswani, A., et al. Attention is all you need. Adv Neural Inf Process Syst. 30, (2017).
- Chen, J., et al. TransUNET: Transformers make strong encoders for medical image segmentation. arXiv. , 04396 (2021).
- Liu, Z., et al. Swin transformer: Hierarchical vision transformer using shifted windows. Proc IEEE Int Conf Comput Vis. , 10012-10022 (2021).
- Cao, H., et al. Swin-UNET: Unet-like pure transformer for medical image segmentation. Comput Vis ECCV. , 205-218 (2022).
- Zhou, H. -. Y., et al. nnformer: Interleaved transformer for volumetric segmentation. arXiv. arXiv. , 03201 (2021).
- Sunkara, R., Luo, T. No more strided convolutions or pooling: A new CNN building block for low-resolution images and small objects. Mach Learn Knowl Discov Databases. , 443-459 (2023).
- Çiçek, &. #. 2. 1. 4. ;., Abdulkadir, A., Lienkamp, S. S., Brox, T., Ronneberger, O. 3D U-Net: learning dense volumetric segmentation from sparse annotation. Med Comput Vis Bayesian Graph Models Biomed Imaging. 2016, 424-432 (2016).
- Kim, H., et al. Abdominal multi-organ auto-segmentation using 3D-patch-based deep convolutional neural network. Scientific reports. 10 (1), 6204 (2020).
- Isensee, F., et al. nnu-net: Self-adapting framework for u-net-based medical image segmentation. arXiv. , 10486 (2018).
- Ji, Y., et al. A large-scale abdominal multi-organ benchmark for versatile medical image segmentation. Adv Neural Inf Process Syst. 35, 36722-36732 (2022).
- Liu, Y., et al. Paddleseg: A high-efficient development toolkit for image segmentation. arXiv. , 06175 (2021).
- Hatamizadeh, A., et al. UNETR: Transformers for 3D medical image segmentation. Proc IEEE Int Conf Comput Vis. , 574-584 (2022).
- Zhou, H., Zeng, D., Bian, Z., Ma, J. A semi-supervised network-based tissue-aware contrast enhancement method for ct images. Nan Fang Yi Ke Da Xue Xue Bao. 43 (6), 985-993 (2023).
- Ma, J., et al. Abdomenct-1k: Is abdominal organ segmentation a solved problem. IEEE Trans Pattern Anal Mach Intell. 44 (10), 6695-6714 (2021).
- Galván, E., Mooney, P. Neuroevolution in deep neural networks: Current trends and future challenges. IEEE Trans Artif Intell. 2 (6), 476-493 (2021).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados