Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Swin-PSAxialNet: Eine effiziente Multiorgan-Segmentierungstechnik
In diesem Artikel
Zusammenfassung
Das vorliegende Protokoll beschreibt ein effizientes Multiorgan-Segmentierungsverfahren namens Swin-PSAxialNet, das im Vergleich zu früheren Segmentierungsmethoden eine hervorragende Genauigkeit erreicht hat. Zu den wichtigsten Schritten dieses Verfahrens gehören die Sammlung von Datensätzen, die Umgebungskonfiguration, die Datenvorverarbeitung, das Trainieren und Vergleichen von Modellen sowie Ablationsexperimente.
Zusammenfassung
Die Segmentierung mehrerer Organe im Abdomen ist eines der wichtigsten Themen im Bereich der medizinischen Bildanalyse und spielt eine wichtige Rolle bei der Unterstützung klinischer Arbeitsabläufe wie der Diagnose von Krankheiten und der Behandlungsplanung. In dieser Studie wird eine effiziente Multiorgan-Segmentierungsmethode namens Swin-PSAxialNet vorgeschlagen, die auf der nnU-Net-Architektur basiert. Es wurde speziell für die präzise Segmentierung von 11 Bauchorganen in CT-Bildern entwickelt. Das vorgeschlagene Netzwerk hat im Vergleich zu nnU-Net die folgenden Verbesserungen vorgenommen. Zunächst wurden Space-to-Depth (SPD)-Module und PSAA-Merkmalsextraktionsblöcke (Parameter-Shared Axial Attention) eingeführt, die die Möglichkeiten der 3D-Bildmerkmalsextraktion verbessern. Zweitens wurde ein mehrskaliger Bildfusionsansatz verwendet, um detaillierte Informationen und räumliche Merkmale zu erfassen und die Fähigkeit zur Extraktion subtiler Merkmale und Kantenmerkmale zu verbessern. Schließlich wurde eine Methode zur gemeinsamen Nutzung von Parametern eingeführt, um die Rechenkosten und die Trainingsgeschwindigkeit des Modells zu reduzieren. Das vorgeschlagene Netzwerk erreicht einen durchschnittlichen Würfelkoeffizienten von 0,93342 für die Segmentierungsaufgabe mit 11 Organen. Experimentelle Ergebnisse zeigen die bemerkenswerte Überlegenheit von Swin-PSAxialNet gegenüber früheren gängigen Segmentierungsmethoden. Die Methode zeigt eine hervorragende Genauigkeit und einen geringen Rechenaufwand bei der Segmentierung der wichtigsten Bauchorgane.
Einleitung
Moderne klinische Interventionen, einschließlich der Diagnose von Krankheiten, der Formulierung von Behandlungsplänen und der Verfolgung von Behandlungsergebnissen, beruhen auf der genauen Segmentierung medizinischer Bilder1. Die komplexen strukturellen Beziehungen zwischen den Bauchorganen2machen es jedoch zu einer herausfordernden Aufgabe, eine genaue Segmentierung mehrerer Bauchorganezu erreichen 3. In den letzten Jahrzehnten haben die rasanten Entwicklungen in der medizinischen Bildgebung und im Bereich des maschinellen Sehens sowohl neue Möglichkeiten als auch Herausforderungen auf dem Gebiet der abdominalen Multiorgansegmentierung mit sich gebracht. Fortschrittliche Magnetresonanztomographie (MRT)4 und Computertomographie (CT)-Technologie5 ermöglichen es uns, hochauflösende Bilder des Abdomens zu erfassen. Die präzise Segmentierung mehrerer Organe aus CT-Bildern ist von erheblichem klinischem Wert für die Beurteilung und Behandlung lebenswichtiger Organe wie Leber, Nieren, Milz, Bauchspeicheldrüse usw.6,7,8,9,10 Die manuelle Annotation dieser anatomischen Strukturen, insbesondere solcher, die ein Eingreifen von Radiologen oder Radioonkologen erfordern, ist jedoch sowohl zeitaufwändig als auch anfällig für subjektive Einflüsse11. Daher besteht ein dringender Bedarf, automatisierte und genaue Methoden für die Segmentierung mehrerer abdominaler Organe zu entwickeln.
Frühere Forschungen zur Bildsegmentierung stützten sich hauptsächlich auf Convolutional Neural Networks (CNNs), die die Segmentierungseffizienz durch das Stapeln von Schichten und die Einführung von ResNet12 verbessern. Im Jahr 2020 führte das Google-Forschungsteam das Vision Transformer (VIT)-Modell13 ein und markierte damit ein bahnbrechendes Beispiel für die Integration der Transformer-Architektur in den traditionellen visuellen Bereich für eine Reihe von visuellen Aufgaben14. Während Faltungsoperationen nur lokale Merkmalsinformationen berücksichtigen können, ermöglicht der Aufmerksamkeitsmechanismus in Transformers die umfassende Berücksichtigung globaler Merkmalsinformationen.
In Anbetracht der Überlegenheit von Transformer-basierten Architekturen gegenüber herkömmlichen Faltungsnetzwerken15 haben zahlreiche Forschungsteams umfangreiche Untersuchungen zur Optimierung der Synergie zwischen den Stärken von Transformern und Faltungsnetzwerkendurchgeführt 16,17,18,19. Chen et al. stellten das TransUNet für medizinische Bildsegmentierungsaufgaben16 vor, das Transformer nutzt, um globale Merkmale aus Bildern zu extrahieren. Aufgrund der hohen Kosten für das Netzwerktraining und der fehlenden Nutzung des Konzepts der Merkmalsextraktionshierarchie wurden die Vorteile von Transformer nicht vollständig ausgeschöpft.
Um diese Probleme anzugehen, haben viele Forscher begonnen, mit der Integration von Transformern als Rückgrat für das Training von Segmentierungsnetzwerken zu experimentieren. Liu et al.17 stellten den Swin Transformer vor, der eine hierarchische Konstruktionsmethode für die Extraktion von geschichteten Merkmalen verwendete. Es wurde das Konzept der Windows-Multi-Head-Self-Attention (W-MSA) vorgeschlagen, das den Rechenaufwand erheblich reduziert, insbesondere bei größeren Feature-Maps auf flacher Ebene. Dieser Ansatz reduzierte zwar den Rechenaufwand, isolierte aber auch die Informationsübertragung zwischen verschiedenen Fenstern. Um dieses Problem zu lösen, führten die Autoren das Konzept der Shifted Windows Multi-Head Self-Attention (SW-MSA) ein, das die Informationsweitergabe zwischen benachbarten Fenstern ermöglicht. Aufbauend auf dieser Methodik formulierten Cao et al. das Swin-UNet18, ersetzten die 2D-Faltungen in U-Net durch Swin-Module und integrierten W-MSA und SW-MSA in die Codierungs- und Dekodierungsprozesse, was zu lobenswerten Segmentierungsergebnissen führte.
Umgekehrt betonten Zhou et al., dass der Vorteil des Conv-Betriebs bei der Verarbeitung hochauflösender Bilder nicht ignoriert werden konnte19. Ihr vorgeschlagener nnFormer verwendet eine Selbstaufmerksamkeitsberechnungsmethode, die auf lokalen dreidimensionalen Bildblöcken basiert und ein Transformer-Modell darstellt, das durch eine kreuzförmige Struktur gekennzeichnet ist. Die Nutzung von Aufmerksamkeit auf der Grundlage lokaler dreidimensionaler Blöcke reduzierte die Trainingsbelastung des Netzwerks erheblich.
Angesichts der Probleme mit der obigen Studie wird eine effiziente hybride hierarchische Struktur für die medizinische Bildsegmentierung in 3D, genannt Swin-PSAxialNet, vorgeschlagen. Dieses Verfahren umfasst einen Downsampling-Block, den Space-to-Depth (SPD)20-Block , der in der Lage ist, globale Informationen21 zu extrahieren. Darüber hinaus fügt es ein PSAA-Modul (Parameter Shared Axial Attention) hinzu, das die Anzahl der Lernparameter von quadratisch auf linear reduziert und sich positiv auf die Genauigkeit des Netzwerktrainings und die Komplexität von Trainingsmodellenauswirkt 22.
Swin-PSAxialNet-Netzwerk
Die Gesamtarchitektur des Netzwerks nimmt die U-förmige Struktur von nnU-Net23 an, die aus Encoder- und Decoderstrukturen besteht. Diese Strukturen beschäftigen sich mit der lokalen Merkmalsextraktion und der Verkettung von Merkmalen aus großen und kleinmaßstäblichen Bildern, wie in Abbildung 1 dargestellt.
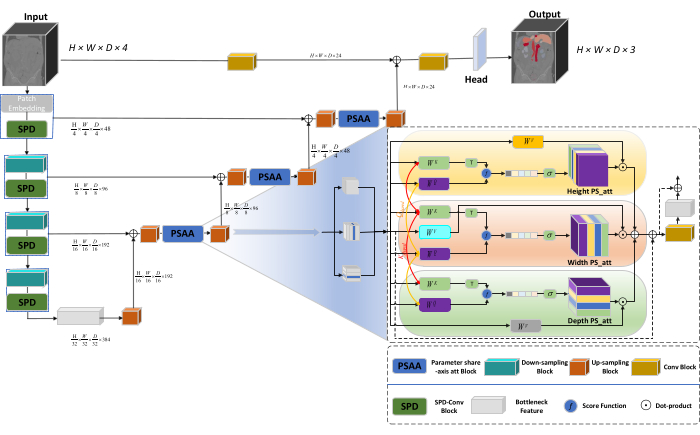
Abbildung 1: Schematische Darstellung der Netzwerkarchitektur von Swin-PSAxialNet. Klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
In der Encoder-Struktur wird der traditionelle Conv-Block mit dem SPD-Block20 kombiniert, um ein Downsampling-Volumen zu bilden. Die erste Schicht des Encoders enthält Patch Embedding, ein Modul, das die 3D-Daten in 3D-Patches 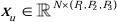 partitioniert, (P1, P2, P3) stellt in diesem Zusammenhang nicht überlappende Patches dar und
partitioniert, (P1, P2, P3) stellt in diesem Zusammenhang nicht überlappende Patches dar und 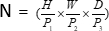 bedeutet die Sequenzlänge von 3D-Patches. Nach der Einbettungsschicht handelt es sich im nächsten Schritt um eine nicht überlappende Faltungs-Downsampling-Einheit, die sowohl aus einem Faltungsblock als auch aus einem SPD-Block besteht. In diesem Setup ist der Schrittwinkel des Faltungsblocks auf 1 gesetzt, und der SPD-Block wird für die Bildskalierung verwendet, was zu einer vierfachen Verringerung der Auflösung und einer zweifachen Erhöhung der Kanäle führt.
bedeutet die Sequenzlänge von 3D-Patches. Nach der Einbettungsschicht handelt es sich im nächsten Schritt um eine nicht überlappende Faltungs-Downsampling-Einheit, die sowohl aus einem Faltungsblock als auch aus einem SPD-Block besteht. In diesem Setup ist der Schrittwinkel des Faltungsblocks auf 1 gesetzt, und der SPD-Block wird für die Bildskalierung verwendet, was zu einer vierfachen Verringerung der Auflösung und einer zweifachen Erhöhung der Kanäle führt.
In der Decoderstruktur besteht jeder Upsampling-Block nach dem Layer Bottleneck Feature aus einer Kombination aus einem Upsampling-Block und einem PSAA-Block. Die Auflösung der Feature-Map wird um das Doppelte erhöht, und die Kanalanzahl wird zwischen den einzelnen Decoder-Paarstufen halbiert. Um räumliche Informationen wiederherzustellen und die Feature-Darstellung zu verbessern, wird die Feature-Fusion zwischen Bildern mit großem und kleinem Maßstab zwischen den Blöcken mit dem Upsampling durchgeführt. Letztendlich werden die Upsampling-Ergebnisse in die Kopfebene eingespeist, um die ursprüngliche Bildgröße mit einer Ausgabegröße von (H × B × T × C, C = 3) wiederherzustellen.
Architektur des SPD-Blocks
Bei herkömmlichen Methoden wird bei der Downsampling-Sektion ein einzelner Schritt mit einer Schrittweite von 2 verwendet. Dies beinhaltet Convolutional Pooling an lokalen Positionen im Bild, die Begrenzung des rezeptiven Feldes und die Beschränkung des Modells auf die Extraktion von Merkmalen aus kleinen Bildflecken. Bei dieser Methode wird der SPD-Block verwendet, der das Originalbild fein in drei Dimensionen unterteilt. Das ursprüngliche 3D-Bild wird gleichmäßig entlang der x-, y- und z-Achse segmentiert, was zu vier Subvolumenkörpern führt. (Abbildung 2) Anschließend werden die vier Volumina durch die "Kat"-Operation verkettet, und das resultierende Bild erfährt eine 1 × 1 × 1 Faltung, um das heruntergesampelte Bild20 zu erhalten.
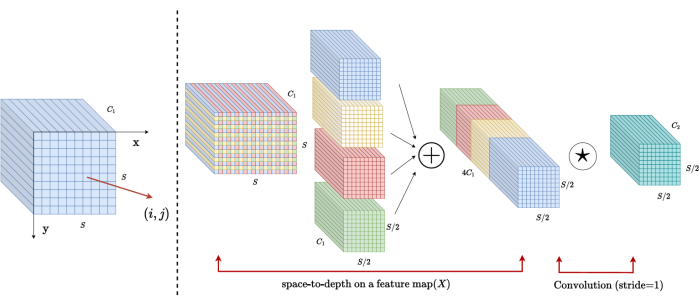
Abbildung 2: SPD-Blockdiagramm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
PSAA-Blockarchitektur
Im Gegensatz zu traditionellen CNN-Netzwerken ist der vorgeschlagene PSAA-Block effektiver bei der globalen Informationsfokussierung und effizienter beim Lernen und Trainieren von Netzwerken. Dies ermöglicht die Erfassung von reichhaltigeren Bildern und räumlichen Merkmalen. Der PSAA-Block umfasst axiales Aufmerksamkeitslernen basierend auf Parametern, die in drei Dimensionen geteilt werden: Höhe, Breite und Tiefe. Im Vergleich zum herkömmlichen Aufmerksamkeitsmechanismus, der Aufmerksamkeitslernen für jedes Pixel im Bild durchführt, führt diese Methode unabhängig voneinander Aufmerksamkeitslernen für jede der drei Dimensionen durch, wodurch die Komplexität der Selbstaufmerksamkeit von quadratisch zu linear reduziert wird. Darüber hinaus wird ein erlernbarer Mechanismus zur gemeinsamen Nutzung von Schlüsselabfragen verwendet, der es dem Netzwerk ermöglicht, Aufmerksamkeitsmechanismusoperationen parallel über die drei Dimensionen durchzuführen, was zu einer schnelleren, überlegeneren und effektiveren Merkmalsdarstellung führt.
Protokoll
Das vorliegende Protokoll wurde von der Ethikkommission der Universität Nantong genehmigt. Es geht um die intelligente Bewertung und Erforschung von erfassten nicht-invasiven oder minimal-invasiven multimodalen Daten, einschließlich medizinischer Bilder des Menschen, Bewegungen von Gliedmaßen und vaskulärer Bildgebung, unter Verwendung von Technologie der künstlichen Intelligenz. Abbildung 3 zeigt das Gesamtflussdiagramm der Multiorgansegmentierung. Alle notwendigen Weblinks finden Sie in der Materialtabelle.
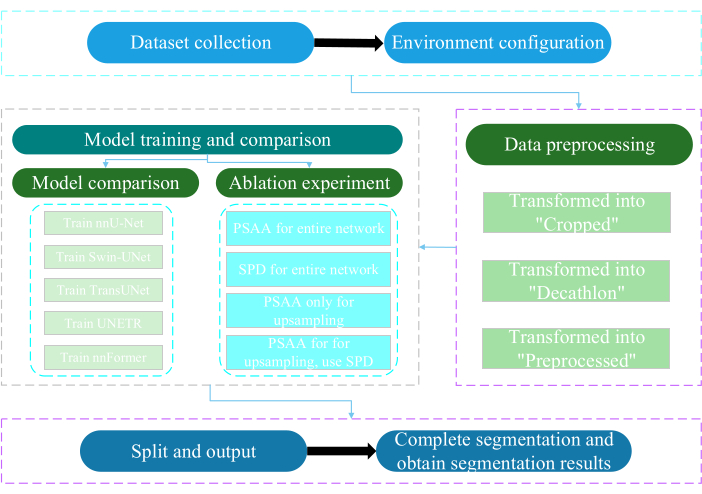
Abbildung 3: Gesamtflussdiagramm der Multiorgansegmentierung. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
1. Sammlung von Datensätzen
- Laden Sie den AMOS2022 Datensatz24 herunter (siehe Materialtabelle).
- Wählen Sie 160 Bilder als Trainingsdataset und 40 Bilder als Testdataset aus.
- Führen Sie mkdir ~/PaddleSeg/contrib/MedicalSeg/data/raw_data aus.
- Öffnen Sie den Pfad der raw_data.
- Legen Sie das Dataset im raw_data Verzeichnis ab und stellen Sie sicher, dass es korrekt mit den Dateinamen in den Verzeichnissen "imagesTr" und "labelsTr" übereinstimmt.
- Erstellen Sie dataset.json Datei.
- Fügen Sie der Datei "dataset.json" Datasetinformationen wie numTraining, numTest und Bezeichnungsname hinzu.
HINWEIS: Der Datensatz für diese Studie besteht aus 200 hochwertigen CT-Datensätzen, die zu Trainings- und Testzwecken aus dem offiziellen 500-CT-Datensatz von AMOS2022 ausgewählt wurden. Der Datensatz umfasst 11 Organe, darunter die Milz, die rechte Niere, die linke Niere, die Gallenblase, die Speiseröhre, die Leber, den Magen, die Aorta, die untere Hohlvene, die Bauchspeicheldrüse und die Blase.
2. Konfiguration der Umgebung
- Installieren Sie die Paddlepaddle-Umgebung (siehe Materialtabelle) und die entsprechende CUDA-Version25.
HINWEIS: Die Verwendung von Paddlepaddle Version 2.5.0 und CUDA Version 11.7 wird empfohlen. - Git-Klonen Sie das PaddleSeg-Repository25.
- Führen Sie cd ~/PaddleSeg/contrib/MedicalSeg/ aus.
- Führen Sie pip install -r requirements.txt aus.
- Führen Sie pip install mampy aus.
3. Datenvorverarbeitung
- Ändern Sie die "yml"-Datei, einschließlich Parametern wie data_root, batch_size, Modell, train_dataset, val_dataset, Optimierer, lr_scheduler und Verlust.
- Geben Sie die erforderlichen Parameter für die Ausführung an, einschließlich "-config, --log_iters, --precision, --nnunet, --save_dir, --save_interval usw.
HINWEIS: Das ursprüngliche "raw_data" wird in drei Arten von Datendateien umgewandelt: "Zugeschnitten", "Zehnkampf" und "Vorverarbeitet", um unterschiedliche Schulungs- und Bewertungsanforderungen zu erfüllen, um die Modellleistung und die Trainingseffektivität zu verbessern.
4. Modelltraining und -vergleich
HINWEIS: Als weit verbreitete Baseline im Bereich der Bildsegmentierung dient nnU-Net23 als Baseline-Modell in der Studie. Der spezifische Modellvergleichsprozess ist wie folgt.
- Zug nnU-Net
- Konfigurieren Sie mit einer Batchgröße von 2 und 80.000 Iterationen, protokollieren Sie alle 20 Iterationen, und nutzen Sie fp16-Präzision.
- Legen Sie den save_dir als "Ausgabe"-Ordner innerhalb des Arbeitsverzeichnisses fest, wobei das Speicherintervall auf 1.000 Iterationen festgelegt ist.
- Fügen Sie den Parameter --use_vdl für die Visualisierung von VisualDL-Parametern hinzu.
- Führen Sie train.py aus.
- Erstellen Sie vier YML-Dateien , geben Sie die Falten 1 bis 4 an, und trainieren Sie das Modell mit fünffacher Kreuzvalidierung.
- Zug Swin-UNet
- Bewahren Sie die Konsistenz mit den zuvor festgelegten Versuchsbedingungen.
- Entfernen Sie den Parameter --nnunet und verwenden Sie --swinunet.
- Führen Sie die train.py Datei aus.
- Der Zug faltet die Faltungen 1-4 einzeln und wird dann einer Ensemble-Verarbeitung unterzogen.
- Zug TransUNet
- Befolgen Sie die in Schritt 4.1 beschriebenen Verfahren.
- Öffnen Sie die yml-Datei .
- Ändern Sie type: nnunet in type: transunet.
- Ändern Sie den Parameter --nnunet in --transunet.
- Führen Sie die train.py Datei aus.
- Der Zug falzt 1-4 einzeln und verarbeitet die Ensembleverarbeitung.
- Zug UNETR
- Konsistente experimentelle Variablen beibehalten.
- Wiederholen Sie die zuvor beschriebenen Verfahren wie Swin-UNet und TransUNet (Schritt 4.2 und Schritt 4.3).
- Führen Sie train.py aus.
- Der Zug falzt 1-4 einzeln und verarbeitet die Ensembleverarbeitung.
- Zug nnFormer
- Ändern Sie die Parameter wie Swin-UNet und TransUNet.
- Führen Sie einfach gefaltet train.py aus.
- Führen Sie die Ensembleverarbeitung aus.
5. Ablationsversuch
- PSAA-Block für das gesamte Netzwerk
- Halten Sie die Variablen konsistent mit Schritt 4.1.
- Legen Sie volume_shape = (64.192.192) und Axial_attention = True fest.
- Führen Sie einfach gefaltet train.py aus.
- Verarbeiten Sie die Ensemble-Verarbeitung über fünf Falten.
- SPD-Block für das gesamte Netzwerk
- Halten Sie die Variablen konsistent mit Schritt 4.1.
- Legen Sie SPD_enable = True und Axial_attention = False fest.
- Führen Sie einfach gefaltet train.py aus.
- Verarbeiten Sie die Ensemble-Verarbeitung über fünf Falten.
- PSAA-Block für Upsampling
HINWEIS: Verwenden Sie den SPD-Block nicht für das Downsampling.- Verwenden Sie den PSAA-Block für das Upsampling.
- Halten Sie die Variablen konsistent mit Schritt 4.1.
- Legen Sie Axial_attention = False im Downsampling-Teil und SPD_enable = False fest.
- Führen Sie einfach gefaltet train.py aus.
- Verarbeiten Sie die fünffache Ensemble-Verarbeitung.
- PSAA-Block für Upsampling
HINWEIS: Verwenden Sie den SPD-Block für das Downsampling.- Verwenden Sie den SPD-Block für das Downsampling und den PSAA-Block für das Upsampling.
- Halten Sie die Variablen konsistent mit Schritt 4.1.
- Legen Sie Axial_attention = False im Downsampling-Teil und SPD_enable = True fest.
- Führen Sie einfach gefaltet train.py aus.
- Verarbeiten Sie die fünffache Ensemble-Verarbeitung.
- Legen Sie die Anzahl der Iterationen aufgrund der besseren Ergebnisse auf 2.00.000 fest.
Ergebnisse
Dieses Protokoll verwendet zwei Metriken, um das Modell zu bewerten: Dice Similarity Score (DSC) und 95% Hausdorff Distance (HD95). DSC misst die Überlappung zwischen Voxelsegmentierungsvorhersagen und Ground Truth, während 95 % HD die Überlappung zwischen den Grenzen der Voxelsegmentierungsvorhersage und der Ground Truth bewertet und 5 % der Ausreißer herausfiltert. Die Definition von DSC26 lautet wie folgt:
Diskussion
Die Segmentierung von Bauchorganen ist eine komplizierte Arbeit. Im Vergleich zu anderen inneren Strukturen des menschlichen Körpers, wie dem Gehirn oder dem Herzen, scheint die Segmentierung von Bauchorganen aufgrund des geringen Kontrasts und der großen Formveränderungen in CT-Bildern schwieriger zu sein27,28. Swin-PSAxialNet wird hier vorgeschlagen, um dieses schwierige Problem zu lösen.
Im Schr...
Offenlegungen
Die Autoren erklären, dass keine Interessenkonflikte bestehen.
Danksagungen
Diese Studie wurde unterstützt durch das '333' Engineering Project der Provinz Jiangsu ([2022]21-003), das Wuxi Health Commission General Program (M202205) und den Wuxi Science and Technology Development Fund (Y20212002-1), deren Beiträge für den Erfolg dieser Arbeit von unschätzbarem Wert waren." Die Autorinnen und Autoren danken allen wissenschaftlichen Mitarbeiterinnen und Mitarbeitern sowie den Studienteilnehmerinnen und -teilnehmern für ihre Unterstützung.
Materialien
| Name | Company | Catalog Number | Comments |
| AMOS2022 dataset | None | None | Datasets for network training and testing. The weblink is: https://pan.baidu.com/s/1x2ZW5FiZtVap0er55Wk4VQ?pwd=xhpb |
| ASUS mainframe | ASUS | https://www.asusparts.eu/en/asus-13020-01910200 | |
| CUDA version 11.7 | NVIDIA | https://developer.nvidia.com/cuda-11-7-0-download-archive | |
| NVIDIA GeForce RTX 3090 | NVIDIA | https://www.nvidia.com/en-in/geforce/graphics-cards/30-series/rtx-3090-3090ti/ | |
| Paddlepaddle environment | Baidu | None | Environmental preparation for network training. The weblink is: https://www.paddlepaddle.org.cn/ |
| PaddleSeg | Baidu | None | The baseline we use: https://github.com/PaddlePaddle/PaddleSeg |
Referenzen
- Liu, X., Song, L., Liu, S., Zhang, Y. A review of deep-learning-based medical image segmentation methods. Sustain. 13 (3), 1224 (2021).
- Popilock, R., Sandrasagaren, K., Harris, L., Kaser, K. A. CT artifact recognition for the nuclear technologist. J Nucl Med Technol. 36 (2), 79-81 (2008).
- Rehman, A., Khan, F. G. A deep learning based review on abdominal images. Multimed Tools Appl. 80, 30321-30352 (2021).
- Schenck, J. F. The role of magnetic susceptibility in magnetic resonance imaging: Mri magnetic compatibility of the first and second kinds. Med Phys. 23 (6), 815-850 (1996).
- Palmisano, A., et al. Myocardial late contrast enhancement ct in troponin-positive acute chest pain syndrome. Radiol. 302 (3), 545-553 (2022).
- Chen, E. -. L., Chung, P. -. C., Chen, C. -. L., Tsai, H. -. M., Chang, C. -. I. An automatic diagnostic system for CT liver image classification. IEEE Trans Biomed Eng. 45 (6), 783-794 (1998).
- Sagel, S. S., Stanley, R. J., Levitt, R. G., Geisse, G. J. R. Computed tomography of the kidney. Radiol. 124 (2), 359-370 (1977).
- Freeman, J. L., Jafri, S., Roberts, J. L., Mezwa, D. G., Shirkhoda, A. CT of congenital and acquired abnormalities of the spleen. Radiographics. 13 (3), 597-610 (1993).
- Almeida, R. R., et al. Advances in pancreatic CT imaging. AJR Am J Roentgenol. 211 (1), 52-66 (2018).
- Eisen, G. M., et al. Guidelines for credentialing and granting privileges for endoscopic ultrasound. Gastrointest Endosc. 54 (6), 811-814 (2001).
- Sykes, J. Reflections on the current status of commercial automated segmentation systems in clinical practice. J Med Radiat Sci. 61 (3), 131-134 (2014).
- He, K., Zhang, X., Ren, S., Sun, J. Deep residual learning for image recognition. 2016 IEEE Conference on Computer Vision and Pattern Recognition (CVPR). , 770-778 (2016).
- Dosovitskiy, A., et al. An image is worth 16x16 words: Transformers for image recognition at scale. arXiv. arXiv. , 11929 (2020).
- Ramachandran, P., et al. Stand-alone self-attention in vision models. Adv Neural Inf Process Syst. 32, (2019).
- Vaswani, A., et al. Attention is all you need. Adv Neural Inf Process Syst. 30, (2017).
- Chen, J., et al. TransUNET: Transformers make strong encoders for medical image segmentation. arXiv. , 04396 (2021).
- Liu, Z., et al. Swin transformer: Hierarchical vision transformer using shifted windows. Proc IEEE Int Conf Comput Vis. , 10012-10022 (2021).
- Cao, H., et al. Swin-UNET: Unet-like pure transformer for medical image segmentation. Comput Vis ECCV. , 205-218 (2022).
- Zhou, H. -. Y., et al. nnformer: Interleaved transformer for volumetric segmentation. arXiv. arXiv. , 03201 (2021).
- Sunkara, R., Luo, T. No more strided convolutions or pooling: A new CNN building block for low-resolution images and small objects. Mach Learn Knowl Discov Databases. , 443-459 (2023).
- Çiçek, &. #. 2. 1. 4. ;., Abdulkadir, A., Lienkamp, S. S., Brox, T., Ronneberger, O. 3D U-Net: learning dense volumetric segmentation from sparse annotation. Med Comput Vis Bayesian Graph Models Biomed Imaging. 2016, 424-432 (2016).
- Kim, H., et al. Abdominal multi-organ auto-segmentation using 3D-patch-based deep convolutional neural network. Scientific reports. 10 (1), 6204 (2020).
- Isensee, F., et al. nnu-net: Self-adapting framework for u-net-based medical image segmentation. arXiv. , 10486 (2018).
- Ji, Y., et al. A large-scale abdominal multi-organ benchmark for versatile medical image segmentation. Adv Neural Inf Process Syst. 35, 36722-36732 (2022).
- Liu, Y., et al. Paddleseg: A high-efficient development toolkit for image segmentation. arXiv. , 06175 (2021).
- Hatamizadeh, A., et al. UNETR: Transformers for 3D medical image segmentation. Proc IEEE Int Conf Comput Vis. , 574-584 (2022).
- Zhou, H., Zeng, D., Bian, Z., Ma, J. A semi-supervised network-based tissue-aware contrast enhancement method for ct images. Nan Fang Yi Ke Da Xue Xue Bao. 43 (6), 985-993 (2023).
- Ma, J., et al. Abdomenct-1k: Is abdominal organ segmentation a solved problem. IEEE Trans Pattern Anal Mach Intell. 44 (10), 6695-6714 (2021).
- Galván, E., Mooney, P. Neuroevolution in deep neural networks: Current trends and future challenges. IEEE Trans Artif Intell. 2 (6), 476-493 (2021).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten