Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Swin-PSAxialNet : une technique de segmentation multi-organes efficace
Dans cet article
Résumé
Le présent protocole décrit une méthode de segmentation multi-organes efficace appelée Swin-PSAxialNet, qui a atteint une excellente précision par rapport aux méthodes de segmentation précédentes. Les étapes clés de cette procédure comprennent la collecte de l’ensemble de données, la configuration de l’environnement, le prétraitement des données, l’entraînement et la comparaison des modèles, ainsi que les expériences d’ablation.
Résumé
La segmentation abdominale multi-organes est l’un des sujets les plus importants dans le domaine de l’analyse d’images médicales, et elle joue un rôle important dans le soutien des flux de travail cliniques tels que le diagnostic des maladies et la planification du traitement. Dans cette étude, une méthode de segmentation multi-organes efficace appelée Swin-PSAxialNet basée sur l’architecture nnU-Net est proposée. Il a été conçu spécifiquement pour la segmentation précise de 11 organes abdominaux dans les images CT. Le réseau proposé a apporté les améliorations suivantes par rapport à nnU-Net. Tout d’abord, des modules d’espace à profondeur (SPD) et des blocs d’extraction de caractéristiques d’attention axiale partagée (PSAA) ont été introduits, améliorant ainsi la capacité d’extraction de caractéristiques d’images 3D. Deuxièmement, une approche de fusion d’images à plusieurs échelles a été utilisée pour capturer des informations détaillées et des caractéristiques spatiales, améliorant ainsi la capacité d’extraction de caractéristiques subtiles et de caractéristiques de bord. Enfin, une méthode de partage de paramètres a été introduite pour réduire le coût de calcul et la vitesse d’entraînement du modèle. Le réseau proposé atteint un coefficient de dés moyen de 0,93342 pour la tâche de segmentation impliquant 11 organes. Les résultats expérimentaux indiquent la supériorité notable de Swin-PSAxialNet sur les méthodes de segmentation traditionnelles précédentes. La méthode montre une excellente précision et de faibles coûts de calcul dans la segmentation des principaux organes abdominaux.
Introduction
L’intervention clinique contemporaine, y compris le diagnostic des maladies, la formulation de plans de traitement et le suivi des résultats du traitement, repose sur la segmentation précise des images médicales1. Cependant, les relations structurelles complexes entre les organes abdominaux2rendent difficile la réalisation d’une segmentation précise de plusieurs organes abdominaux3. Au cours des dernières décennies, les développements florissants de l’imagerie médicale et de la vision par ordinateur ont présenté à la fois de nouvelles opportunités et de nouveaux défis dans le domaine de la segmentation abdominale multi-organes. L’imagerie par résonance magnétique (IRM)4 et la technologie de tomodensitométrie (TDM)5 nous permettent d’acquérir des images abdominales à haute résolution. La segmentation précise de plusieurs organes à partir d’images CT a une valeur clinique significative pour l’évaluation et le traitement d’organes vitaux tels que le foie, les reins, la rate, le pancréas, etc.6,7,8,9,10 Cependant, l’annotation manuelle de ces structures anatomiques, en particulier celles nécessitant l’intervention de radiologues ou de radio-oncologues, prend du temps et est sensible à des influences subjectives 11. Par conséquent, il est urgent de développer des méthodes automatisées et précises pour la segmentation abdominale multi-organes.
Les recherches précédentes sur la segmentation d’images reposaient principalement sur les réseaux neuronaux convolutifs (CNN), qui améliorent l’efficacité de la segmentation en empilant des couches et en introduisant ResNet12. En 2020, l’équipe de recherche de Google a présenté le modèle Vision Transformer (VIT)13, marquant ainsi un exemple pionnier d’intégration de l’architecture Transformer dans le domaine visuel traditionnel pour une gamme de tâches visuelles14. Alors que les opérations convolutives ne peuvent prendre en compte que les informations de caractéristiques locales, le mécanisme d’attention de Transformers permet de prendre en compte de manière exhaustive les informations de caractéristiques globales.
Compte tenu de la supériorité des architectures basées sur les transformateurs sur les réseaux convolutifs traditionnels15, de nombreuses équipes de recherche ont entrepris une exploration approfondie pour optimiser la synergie entre les forces des transformateurs et les réseaux convolutifs 16,17,18,19. Chen et al. ont présenté le TransUNet pour les tâches de segmentation d’images médicales16, qui exploite Transformers pour extraire des caractéristiques globales à partir d’images. En raison du coût élevé de la formation au réseau et de l’incapacité à utiliser le concept de hiérarchie d’extraction de fonctionnalités, les avantages de Transformer n’ont pas été pleinement exploités.
Pour résoudre ces problèmes, de nombreux chercheurs ont commencé à expérimenter l’intégration de Transformers comme épine dorsale pour les réseaux de segmentation de l’entraînement. Liu et al.17 ont introduit le Swin Transformer, qui utilisait une méthode de construction hiérarchique pour l’extraction de caractéristiques en couches. Le concept d’auto-attention multi-têtes Windows (W-MSA) a été proposé, réduisant considérablement les coûts de calcul, en particulier en présence de cartes de fonctionnalités plus grandes et peu profondes. Bien que cette approche réduise les besoins en calcul, elle isole également la transmission d’informations entre différentes fenêtres. Pour résoudre ce problème, les auteurs ont introduit le concept d’auto-attention multi-têtes à fenêtres décalées (SW-MSA), permettant la propagation de l’information entre les fenêtres adjacentes. S’appuyant sur cette méthodologie, Cao et al. ont formulé le Swin-UNet18, remplaçant les circonvolutions 2D dans U-Net par des modules Swin et incorporant W-MSA et SW-MSA dans les processus d’encodage et de décodage, obtenant des résultats de segmentation louables.
À l’inverse, Zhou et al. ont souligné que l’avantage du fonctionnement en conv ne pouvait être ignoré lors du traitement d’images à haute résolution19. Leur proposition nnFormer utilise une méthode de calcul d’auto-attention basée sur des blocs d’images tridimensionnels locaux, constituant un modèle Transformer caractérisé par une structure en forme de croix. L’utilisation de l’attention basée sur des blocs tridimensionnels locaux a considérablement réduit la charge d’entraînement sur le réseau.
Compte tenu des problèmes posés par l’étude ci-dessus, une structure hiérarchique hybride efficace pour la segmentation d’images médicales 3D, appelée Swin-PSAxialNet, est proposée. Cette méthode intègre un bloc de sous-échantillonnage, le bloc Space-to-depth (SPD)20, capable d’extraire des informations globales21. De plus, il ajoute un module d’attention axiale partagée (PSAA), qui réduit le nombre de paramètres d’apprentissage de quadratique à linéaire et aura un effet positif sur la précision de l’entraînement du réseau et la complexité des modèles d’entraînement22.
Réseau Swin-PSAxialNet
L’architecture globale du réseau adopte la structure en forme de U de nnU-Net23, composée de structures d’encodeur et de décodeur. Ces structures s’engagent dans l’extraction de caractéristiques locales et la concaténation de caractéristiques à partir d’images à grande et petite échelle, comme illustré à la figure 1.
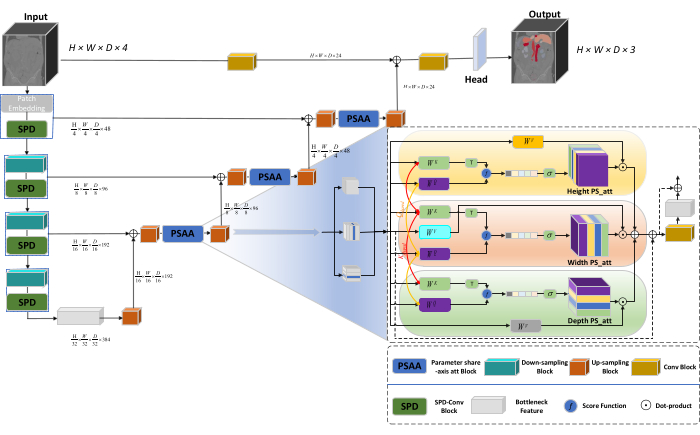
Figure 1 : Schéma de principe de l’architecture réseau de Swin-PSAxialNet. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Dans la structure de l’encodeur, le bloc Conv traditionnel est combiné avec le bloc SPD20 pour former un volume de sous-échantillonnage. La première couche de l’encodeur intègre Patch Embedding, un module qui partitionne les données 3D en patchs 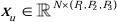 3D, (P1, P2, P3) représente des patchs non chevauchants dans ce contexte,
3D, (P1, P2, P3) représente des patchs non chevauchants dans ce contexte, 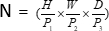 signifie la longueur de la séquence des patchs 3D. Après la couche d’encastrement, l’étape suivante implique une unité de sous-échantillonnage convolutif non chevauchant comprenant à la fois un bloc convolutif et un bloc SPD. Dans cette configuration, le bloc convolutif a une foulée définie sur 1, et le bloc SPD est utilisé pour la mise à l’échelle de l’image, ce qui entraîne une réduction de quatre fois de la résolution et une multiplication par deux du nombre de canaux.
signifie la longueur de la séquence des patchs 3D. Après la couche d’encastrement, l’étape suivante implique une unité de sous-échantillonnage convolutif non chevauchant comprenant à la fois un bloc convolutif et un bloc SPD. Dans cette configuration, le bloc convolutif a une foulée définie sur 1, et le bloc SPD est utilisé pour la mise à l’échelle de l’image, ce qui entraîne une réduction de quatre fois de la résolution et une multiplication par deux du nombre de canaux.
Dans la structure du décodeur, chaque bloc de suréchantillonnage après la couche Fonctionnalité de goulot d’étranglement se compose d’une combinaison d’un bloc de suréchantillonnage et d’un bloc PSAA. La résolution de la carte des fonctions est multipliée par deux et le nombre de canaux est divisé par deux entre chaque paire d’étages de décodeur. Pour restaurer les informations spatiales et améliorer la représentation des entités, la fusion des entités entre les images à grande et à petite échelle est effectuée entre les blocs de suréchantillonnage. En fin de compte, les résultats du suréchantillonnage sont introduits dans la couche Head pour restaurer la taille de l’image d’origine, avec une taille de sortie de (H × W × D × C, C = 3).
Architecture de bloc SPD
Dans les méthodes traditionnelles, la section de sous-échantillonnage utilise une seule foulée avec une taille de pas de 2. Cela implique un regroupement convolutif à des positions locales dans l’image, en limitant le champ récepteur et en confinant le modèle à l’extraction de caractéristiques à partir de petits patchs d’image. Cette méthode utilise le bloc SPD, qui divise finement l’image d’origine en trois dimensions. L’image 3D d’origine est segmentée uniformément le long des axes x, y et z, ce qui donne quatre corps de sous-volume. (Graphique 2) Par la suite, les quatre volumes sont concaténés par une opération « chat », et l’image résultante subit une convolution 1 × 1 × 1 pour obtenir l’image sous-échantillonnée20.
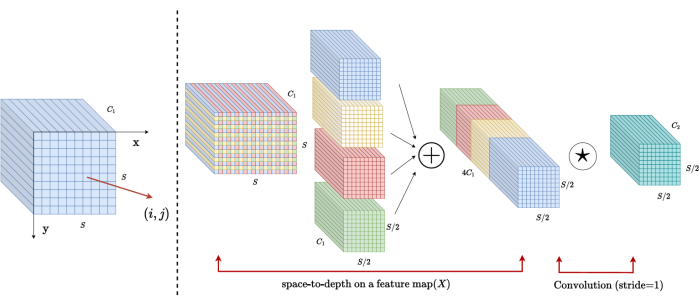
Figure 2 : Schéma fonctionnel du SPD. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Architecture de bloc PSAA
Contrairement aux réseaux CNN traditionnels, le bloc PSAA proposé est plus efficace pour mener une enquête mondiale et plus efficace pour l’apprentissage et la formation en réseau. Cela permet de capturer des images et des caractéristiques spatiales plus riches. Le bloc PSAA comprend l’apprentissage de l’attention axiale basé sur le partage de paramètres en trois dimensions : hauteur, largeur et profondeur. Par rapport au mécanisme d’attention conventionnel qui effectue l’apprentissage de l’attention pour chaque pixel de l’image, cette méthode effectue indépendamment l’apprentissage de l’attention pour chacune des trois dimensions, réduisant ainsi la complexité de l’auto-attention de quadratique à linéaire. De plus, un mécanisme de partage de paramètres keys-queries apprenant est utilisé, permettant au réseau d’effectuer des opérations de mécanisme d’attention en parallèle sur les trois dimensions, ce qui se traduit par une représentation plus rapide, supérieure et plus efficace des caractéristiques.
Access restricted. Please log in or start a trial to view this content.
Protocole
Le présent protocole a été approuvé par le Comité d’éthique de l’Université de Nantong. Il implique l’évaluation et la recherche intelligentes de données multimodales non invasives ou peu invasives, y compris des images médicales humaines, des mouvements de membres et l’imagerie vasculaire, à l’aide de la technologie de l’intelligence artificielle. La figure 3 illustre l’organigramme global de la segmentation multi-organes. Tous les liens Web nécessaires sont fournis dans la table des matériaux.
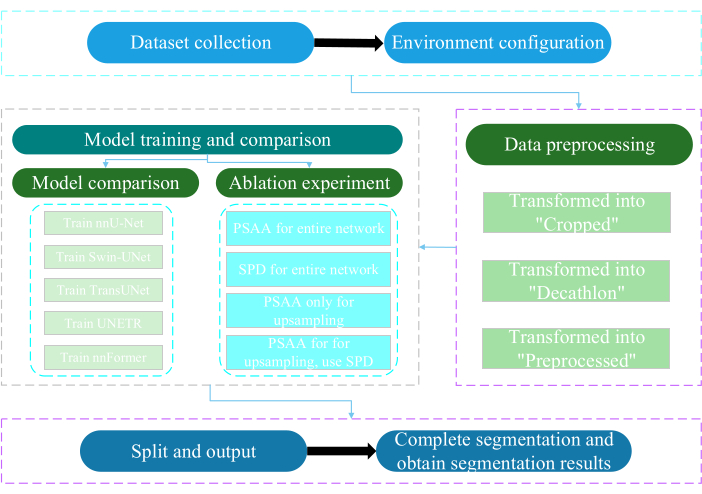
Figure 3 : Organigramme global de la segmentation multi-organes. Veuillez cliquer ici pour voir une version agrandie de cette figure.
1. Collecte de l’ensemble de données
- Téléchargez le jeu de données AMOS202224 (voir Tableau des matériaux).
- Sélectionnez 160 images comme jeu de données d’entraînement et 40 images comme jeu de données de test.
- Exécutez terminal mkdir ~/PaddleSeg/contrib/MedicalSeg/data/raw_data.
- Ouvrez le chemin de raw_data.
- Placez le jeu de données dans le répertoire raw_data et assurez-vous qu’il correspond correctement avec les noms de fichiers dans les répertoires « imagesTr » et « labelsTr ».
- Créez dataset.json fichier.
- Ajoutez des informations de jeu de données telles que numTraining, numTest et le nom des libellés dans le fichier « dataset.json ».
REMARQUE : L’ensemble de données de cette étude se compose de 200 ensembles de données CT de haute qualité sélectionnés à partir de l’ensemble de données officiel AMOS2022 500 CT à des fins de formation et de test. L’ensemble de données comprend 11 organes, dont la rate, le rein droit, le rein gauche, la vésicule biliaire, l’œsophage, le foie, l’estomac, l’aorte, la veine cave inférieure, le pancréas et la vessie.
2. Configuration de l’environnement
- Installez l’environnement Paddlepaddle (voir tableau des matériaux) et la version25 CUDA correspondante.
REMARQUE : Il est recommandé d’utiliser la version 2.5.0 de Paddlepaddle et la version 11.7 de CUDA. - Clonez Git le dépôt PaddleSeg25.
- Exécuter en terminal cd ~/PaddleSeg/contrib/MedicalSeg/.
- Exécutez pip install -r requirements.txt.
- Exécutez pip install medpy.
3. Prétraitement des données
- Modifiez le fichier « yml », y compris les paramètres tels que data_root, batch_size, modèle, train_dataset, val_dataset, optimiseur, lr_scheduler et perte.
- Spécifiez les paramètres requis pour l’exécution, y compris « -config », --log_iters, --precision, --nnunet, --save_dir, --save_interval, etc.
REMARQUE : Le « raw_data » d’origine est transformé en trois types de fichiers de données : « Recadré », « Décathlon » et « Prétraité », répondant à différentes exigences d’entraînement et d’évaluation afin d’améliorer les performances du modèle et l’efficacité de l’entraînement.
4. Formation et comparaison des modèles
REMARQUE : En tant que référence largement utilisée dans le domaine de la segmentation d’images, nnU-Net23 sert de modèle de référence dans l’étude. Le processus de comparaison des modèles est le suivant.
- Train nnU-Net
- Configurez avec une taille de lot de 2 et 80 000 itérations, enregistrez toutes les 20 itérations et utilisez la précision fp16.
- Définissez le save_dir en tant que dossier « output » dans le répertoire de travail, avec un intervalle d’enregistrement défini à 1 000 itérations.
- Ajouter le paramètre --use_vdl pour la visualisation des paramètres VisualDL.
- Exécutez train.py.
- Créez quatre fichiers yml , spécifiez les plis 1 à 4 et entraînez le modèle avec une validation croisée en cinq parties.
- Train Swin-UNet
- Maintenir la cohérence avec les conditions expérimentales précédemment établies.
- Supprimez le paramètre --nnunet et utilisez --swinunet.
- Exécutez le fichier train.py.
- Train plie 1 à 4 individuellement, puis soumis à un traitement d’ensemble.
- Train TransUNet
- Suivez les procédures décrites à l’étape 4.1.
- Ouvrez le fichier yml .
- Modifiez type : nnunet en type : transunet.
- Modifiez le paramètre --nnunet en --transunet.
- Exécutez le fichier train.py.
- Train plie 1 à 4 individuellement et traite l’ensemble du traitement.
- Train UNETR
- Maintenir des variables expérimentales cohérentes.
- Répétez les procédures décrites précédemment, comme Swin-UNet et TransUNet (étapes 4.2 et 4.3).
- Exécutez train.py.
- Train plie 1 à 4 individuellement et traite l’ensemble du traitement.
- Train nnFormer
- Modifiez les paramètres tels que Swin-UNet et TransUNet.
- Exécutez un seul pli train.py.
- Exécutez le traitement d’ensemble.
5. Expérience d’ablation
- Bloc PSAA pour l’ensemble du réseau
- Gardez les variables cohérentes avec l’étape 4.1.
- Définissez volume_shape = (64 192 192) et Axial_attention = Vrai.
- Exécutez un seul pli train.py.
- Traitement d’ensemble du processus sur cinq volets.
- Bloc SPD pour l’ensemble du réseau
- Gardez les variables cohérentes avec l’étape 4.1.
- Définissez SPD_enable = Vrai et Axial_attention = Faux.
- Exécutez un seul pli train.py.
- Traitement d’ensemble du processus sur cinq volets.
- Bloc PSAA pour le suréchantillonnage
REMARQUE : N’utilisez pas le bloc SPD pour le sous-échantillonnage.- Utilisez le bloc PSAA pour le suréchantillonnage.
- Gardez les variables cohérentes avec l’étape 4.1.
- Définissez Axial_attention = False dans la partie sous-échantillonnage et SPD_enable = Faux.
- Exécutez un seul pli train.py.
- Traitement d’ensemble à cinq plis.
- Bloc PSAA pour le suréchantillonnage
REMARQUE : Utilisez le bloc SPD pour le sous-échantillonnage.- Utilisez le bloc SPD pour le sous-échantillonnage et le bloc PSAA pour le suréchantillonnage.
- Gardez les variables cohérentes avec l’étape 4.1.
- Définissez Axial_attention = False dans la partie sous-échantillonnage et SPD_enable = True.
- Exécutez un seul pli train.py.
- Traitement d’ensemble à cinq plis.
- Définissez le nombre d’itérations à 2 00 000 en raison des résultats supérieurs.
Access restricted. Please log in or start a trial to view this content.
Résultats
Ce protocole utilise deux mesures pour évaluer le modèle : le score de similarité des dés (DSC) et la distance de Hausdorff à 95 % (HD95). La DSC mesure le chevauchement entre les prédictions de segmentation des voxels et la vérité terrain, tandis que la HD 95 % évalue le chevauchement entre les limites de prédiction de la segmentation des voxels et la vérité terrain, en filtrant 5 % des valeurs aberrantes. La définition de l’ASR26 est la suivante :...
Access restricted. Please log in or start a trial to view this content.
Discussion
La segmentation des organes abdominaux est un travail compliqué. Par rapport à d’autres structures internes du corps humain, telles que le cerveau ou le cœur, la segmentation des organes abdominaux semble plus difficile en raison du faible contraste et des grands changements de forme des images CT27,28. Swin-PSAxialNet est proposé ici pour résoudre ce problème difficile.
Dans l’étape de coll...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs ne déclarent aucun conflit d’intérêts.
Remerciements
Cette étude a été soutenue par le projet d’ingénierie « 333 » de la province du Jiangsu ([2022]21-003), le programme général de la Commission de la santé de Wuxi (M202205) et le Fonds de développement de la science et de la technologie de Wuxi (Y20212002-1), dont les contributions ont été inestimables pour le succès de ce travail. Les auteurs remercient tous les assistants de recherche et les participants à l’étude pour leur soutien.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| AMOS2022 dataset | None | None | Datasets for network training and testing. The weblink is: https://pan.baidu.com/s/1x2ZW5FiZtVap0er55Wk4VQ?pwd=xhpb |
| ASUS mainframe | ASUS | https://www.asusparts.eu/en/asus-13020-01910200 | |
| CUDA version 11.7 | NVIDIA | https://developer.nvidia.com/cuda-11-7-0-download-archive | |
| NVIDIA GeForce RTX 3090 | NVIDIA | https://www.nvidia.com/en-in/geforce/graphics-cards/30-series/rtx-3090-3090ti/ | |
| Paddlepaddle environment | Baidu | None | Environmental preparation for network training. The weblink is: https://www.paddlepaddle.org.cn/ |
| PaddleSeg | Baidu | None | The baseline we use: https://github.com/PaddlePaddle/PaddleSeg |
Références
- Liu, X., Song, L., Liu, S., Zhang, Y. A review of deep-learning-based medical image segmentation methods. Sustain. 13 (3), 1224(2021).
- Popilock, R., Sandrasagaren, K., Harris, L., Kaser, K. A. CT artifact recognition for the nuclear technologist. J Nucl Med Technol. 36 (2), 79-81 (2008).
- Rehman, A., Khan, F. G. A deep learning based review on abdominal images. Multimed Tools Appl. 80, 30321-30352 (2021).
- Schenck, J. F. The role of magnetic susceptibility in magnetic resonance imaging: Mri magnetic compatibility of the first and second kinds. Med Phys. 23 (6), 815-850 (1996).
- Palmisano, A., et al. Myocardial late contrast enhancement ct in troponin-positive acute chest pain syndrome. Radiol. 302 (3), 545-553 (2022).
- Chen, E. -L., Chung, P. -C., Chen, C. -L., Tsai, H. -M., Chang, C. -I. An automatic diagnostic system for CT liver image classification. IEEE Trans Biomed Eng. 45 (6), 783-794 (1998).
- Sagel, S. S., Stanley, R. J., Levitt, R. G., Geisse, G. J. R. Computed tomography of the kidney. Radiol. 124 (2), 359-370 (1977).
- Freeman, J. L., Jafri, S., Roberts, J. L., Mezwa, D. G., Shirkhoda, A. CT of congenital and acquired abnormalities of the spleen. Radiographics. 13 (3), 597-610 (1993).
- Almeida, R. R., et al. Advances in pancreatic CT imaging. AJR Am J Roentgenol. 211 (1), 52-66 (2018).
- Eisen, G. M., et al. Guidelines for credentialing and granting privileges for endoscopic ultrasound. Gastrointest Endosc. 54 (6), 811-814 (2001).
- Sykes, J. Reflections on the current status of commercial automated segmentation systems in clinical practice. J Med Radiat Sci. 61 (3), 131-134 (2014).
- He, K., Zhang, X., Ren, S., Sun, J. Deep residual learning for image recognition. 2016 IEEE Conference on Computer Vision and Pattern Recognition (CVPR). , Vegas, NV, USA. 770-778 (2016).
- Dosovitskiy, A., et al. An image is worth 16x16 words: Transformers for image recognition at scale. arXiv. arXiv. , 11929(2020).
- Ramachandran, P., et al. Stand-alone self-attention in vision models. Adv Neural Inf Process Syst. 32, NIPS2019 (2019).
- Vaswani, A., et al. Attention is all you need. Adv Neural Inf Process Syst. 30, NIPS2019 (2017).
- Chen, J., et al. TransUNET: Transformers make strong encoders for medical image segmentation. arXiv. , arXiv:2102 04396(2021).
- Liu, Z., et al. Swin transformer: Hierarchical vision transformer using shifted windows. Proc IEEE Int Conf Comput Vis. , 10012-10022 (2021).
- Cao, H., et al. Swin-UNET: Unet-like pure transformer for medical image segmentation. Comput Vis ECCV. , 205-218 (2022).
- Zhou, H. -Y., et al. nnformer: Interleaved transformer for volumetric segmentation. arXiv. arXiv. , arXiv:2109 03201(2021).
- Sunkara, R., Luo, T. No more strided convolutions or pooling: A new CNN building block for low-resolution images and small objects. Mach Learn Knowl Discov Databases. , 443-459 (2023).
- Çiçek, Ö, Abdulkadir, A., Lienkamp, S. S., Brox, T., Ronneberger, O. 3D U-Net: learning dense volumetric segmentation from sparse annotation. Med Comput Vis Bayesian Graph Models Biomed Imaging. 2016, 424-432 (2016).
- Kim, H., et al. Abdominal multi-organ auto-segmentation using 3D-patch-based deep convolutional neural network. Scientific reports. 10 (1), 6204(2020).
- Isensee, F., et al. nnu-net: Self-adapting framework for u-net-based medical image segmentation. arXiv. , arXiv:1809 10486(2018).
- Ji, Y., et al. A large-scale abdominal multi-organ benchmark for versatile medical image segmentation. Adv Neural Inf Process Syst. 35, 36722-36732 (2022).
- Liu, Y., et al. Paddleseg: A high-efficient development toolkit for image segmentation. arXiv. , arXiv:2101 06175(2021).
- Hatamizadeh, A., et al. UNETR: Transformers for 3D medical image segmentation. Proc IEEE Int Conf Comput Vis. , https://api.semanticscholar.org/CorpusID:232290634 574-584 (2022).
- Zhou, H., Zeng, D., Bian, Z., Ma, J. A semi-supervised network-based tissue-aware contrast enhancement method for ct images. Nan Fang Yi Ke Da Xue Xue Bao. 43 (6), 985-993 (2023).
- Ma, J., et al. Abdomenct-1k: Is abdominal organ segmentation a solved problem. IEEE Trans Pattern Anal Mach Intell. 44 (10), 6695-6714 (2021).
- Galván, E., Mooney, P. Neuroevolution in deep neural networks: Current trends and future challenges. IEEE Trans Artif Intell. 2 (6), 476-493 (2021).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon