É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Swin-PSAxialNet: uma técnica eficiente de segmentação de múltiplos órgãos
Neste Artigo
Resumo
O presente protocolo descreve um método eficiente de segmentação de múltiplos órgãos chamado Swin-PSAxialNet, que alcançou excelente precisão em comparação com os métodos de segmentação anteriores. As principais etapas deste procedimento incluem coleta de conjunto de dados, configuração do ambiente, pré-processamento de dados, treinamento e comparação de modelos e experimentos de ablação.
Resumo
A segmentação abdominal de múltiplos órgãos é um dos tópicos mais importantes no campo da análise de imagens médicas e desempenha um papel importante no suporte a fluxos de trabalho clínicos, como diagnóstico de doenças e planejamento de tratamento. Neste estudo, é proposto um método eficiente de segmentação de múltiplos órgãos denominado Swin-PSAxialNet baseado na arquitetura nnU-Net. Ele foi projetado especificamente para a segmentação precisa de 11 órgãos abdominais em imagens de TC. A rede proposta fez as seguintes melhorias em comparação com a nnU-Net. Em primeiro lugar, foram introduzidos módulos Space-to-depth (SPD) e blocos de extração de recursos de atenção axial compartilhada por parâmetros (PSAA), aprimorando a capacidade de extração de recursos de imagem 3D. Em segundo lugar, uma abordagem de fusão de imagens em várias escalas foi empregada para capturar informações detalhadas e características espaciais, melhorando a capacidade de extrair características sutis e características de borda. Por fim, um método de compartilhamento de parâmetros foi introduzido para reduzir o custo computacional e a velocidade de treinamento do modelo. A rede proposta atinge um coeficiente de Dice médio de 0,93342 para a tarefa de segmentação envolvendo 11 órgãos. Os resultados experimentais indicam a notável superioridade do Swin-PSAxialNet sobre os métodos de segmentação convencionais anteriores. O método apresenta excelente precisão e baixos custos computacionais na segmentação dos principais órgãos abdominais.
Introdução
A intervenção clínica contemporânea, incluindo o diagnóstico de doenças, a formulação de planos de tratamento e o rastreamento dos resultados do tratamento, depende da segmentação precisa das imagens médicas1. No entanto, as complexas relações estruturais entre os órgãos abdominais2tornam uma tarefa desafiadora alcançar a segmentação precisa de vários órgãos abdominais3. Nas últimas décadas, os desenvolvimentos florescentes em imagens médicas e visão computacional apresentaram novas oportunidades e desafios no campo da segmentação abdominal de múltiplos órgãos. A tecnologia de Ressonância Magnética (RM)4 e Tomografia Computadorizada (TC)5 nos permite adquirir imagens abdominais de alta resolução. A segmentação precisa de múltiplos órgãos a partir de imagens de TC tem valor clínico significativo para a avaliação e tratamento de órgãos vitais, como fígado, rins, baço, pâncreas, etc.6,7,8,9,10 No entanto, a anotação manual dessas estruturas anatômicas, especialmente aquelas que requerem intervenção de radiologistas ou oncologistas de radiação, é demorada e suscetível a influências subjetivas11. Portanto, há uma necessidade urgente de desenvolver métodos automatizados e precisos para segmentação abdominal de múltiplos órgãos.
Pesquisas anteriores sobre segmentação de imagens dependiam predominantemente de redes neurais convolucionais (CNNs), que melhoram a eficiência da segmentação empilhando camadas e introduzindo o ResNet12. Em 2020, a equipe de pesquisa do Google apresentou o modelo Vision Transformer (VIT)13, marcando um exemplo pioneiro de incorporação da arquitetura Transformer ao domínio visual tradicional para uma variedade de tarefas visuais14. Embora as operações convolucionais possam contemplar apenas informações de recursos locais, o mecanismo de atenção em Transformers permite a consideração abrangente de informações de recursos globais.
Considerando a superioridade das arquiteturas baseadas em Transformers sobre as redes convolucionais tradicionais15, várias equipes de pesquisa realizaram uma extensa exploração na otimização da sinergia entre os pontos fortes dos Transformers e as redes convolucionais 16,17,18,19. Chen et al. introduziram o TransUNet para tarefas de segmentação de imagens médicas16, que aproveita os Transformers para extrair recursos globais de imagens. Devido ao alto custo do treinamento de rede e à falha em utilizar o conceito de hierarquia de extração de recursos, as vantagens do Transformer não foram totalmente realizadas.
Para resolver esses problemas, muitos pesquisadores começaram a experimentar a incorporação de Transformers como a espinha dorsal para treinar redes de segmentação. Liu et al.17 introduziram o Transformador Swin, que empregava um método de construção hierárquica para extração de características em camadas. O conceito de Windows Multi-Head Self-Attention (W-MSA) foi proposto, reduzindo significativamente o custo computacional, particularmente na presença de mapas de recursos de nível superficial maiores. Embora essa abordagem reduzisse os requisitos computacionais, ela também isolava a transmissão de informações entre diferentes janelas. Para resolver esse problema, os autores introduziram ainda o conceito de Shifted Windows Multi-Head Self-Attention (SW-MSA), permitindo a propagação de informações entre janelas adjacentes. Com base nessa metodologia, Cao et al. formularam o Swin-UNet18, substituindo as convoluções 2D em U-Net por módulos Swin e incorporando W-MSA e SW-MSA nos processos de codificação e decodificação, alcançando resultados de segmentação louváveis.
Por outro lado, Zhou et al. destacaram que a vantagem da operação conv não pode ser ignorada ao processar imagens de alta resolução19. Seu nnFormer proposto emprega um método de computação de auto-atenção baseado em blocos de imagem tridimensionais locais, constituindo um modelo Transformer caracterizado por uma estrutura em forma de cruz. A utilização da atenção baseada em blocos tridimensionais locais reduziu significativamente a carga de treinamento na rede.
Dados os problemas com o estudo acima, uma estrutura hierárquica híbrida eficiente para segmentação de imagens médicas 3D, denominada Swin-PSAxialNet, é proposta. Este método incorpora um bloco de redução de amostragem, bloco Space-to-depth (SPD)20 , capaz de extrair informações globais21. Além disso, adiciona um módulo de atenção axial compartilhada por parâmetros (PSAA), que reduz a contagem de parâmetros de aprendizado de quadrático para linear e terá um bom efeito na precisão do treinamento da rede e na complexidade dos modelos de treinamento22.
Rede Swin-PSAxialNet
A arquitetura geral da rede adota a estrutura em forma de U do nnU-Net23, consistindo em estruturas de codificador e decodificador. Essas estruturas se envolvem na extração de características locais e na concatenação de características de imagens de grande e pequena escala, conforme ilustrado na Figura 1.
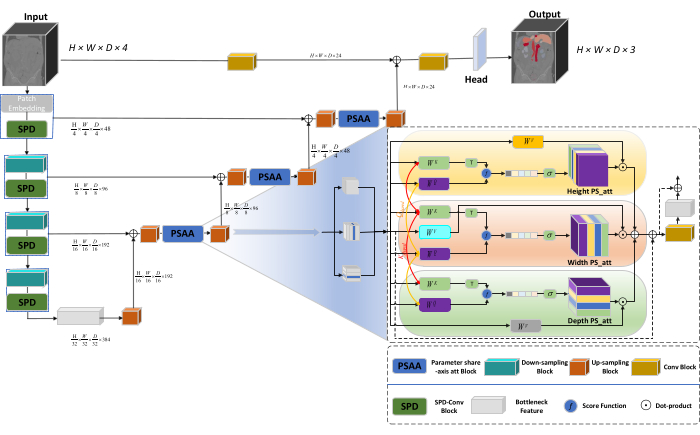
Figura 1: Diagrama esquemático Swin-PSAxialNet da arquitetura de rede. Clique aqui para ver uma versão maior desta figura.
Na estrutura do codificador, o bloco Conv tradicional é combinado com o bloco SPD20 para formar um volume de downsampling. A primeira camada do codificador incorpora Patch Embedding, um módulo que particiona os dados 3D em patches 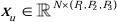 3D, (P1, P2, P3) representa patches não sobrepostos neste contexto,
3D, (P1, P2, P3) representa patches não sobrepostos neste contexto, 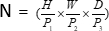 significa o comprimento da sequência de patches 3D. Após a camada de incorporação, a próxima etapa envolve uma unidade de redução de amostragem convolucional não sobreposta, compreendendo um bloco convolucional e um bloco SPD. Nessa configuração, o bloco convolucional tem um passo definido como 1 e o bloco SPD é empregado para dimensionamento de imagem, levando a uma redução de quatro vezes na resolução e um aumento de duas vezes nos canais.
significa o comprimento da sequência de patches 3D. Após a camada de incorporação, a próxima etapa envolve uma unidade de redução de amostragem convolucional não sobreposta, compreendendo um bloco convolucional e um bloco SPD. Nessa configuração, o bloco convolucional tem um passo definido como 1 e o bloco SPD é empregado para dimensionamento de imagem, levando a uma redução de quatro vezes na resolução e um aumento de duas vezes nos canais.
Na estrutura do decodificador, cada bloco de upsample após a camada de Feição de Gargalo consiste em uma combinação de um bloco de upsampling e um bloco PSAA. A resolução do mapa de recursos é aumentada duas vezes e a contagem de canais é reduzida pela metade entre cada par de estágios do decodificador. Para restaurar informações espaciais e melhorar a representação de recursos, a fusão de recursos entre imagens de grande e pequena escala é realizada entre os blocos de aumento de amostragem. Por fim, os resultados do upsampling são alimentados na camada Head para restaurar o tamanho da imagem original, com um tamanho de saída de (H × W × D × C, C = 3).
Arquitetura de bloco SPD
Nos métodos tradicionais, a seção de redução da amostragem emprega uma única passada com um tamanho de passo de 2. Isso envolve o agrupamento convolucional em posições locais na imagem, limitando o campo receptivo e confinando o modelo à extração de recursos de pequenos patches de imagem. Este método utiliza o bloco SPD, que divide finamente a imagem original em três dimensões. A imagem 3D original é segmentada uniformemente ao longo dos eixos x, y e z, resultando em quatro corpos de subvolume. (Figura 2) Posteriormente, os quatro volumes são concatenados por meio da operação "cat", e a imagem resultante sofre uma convolução 1 × 1 × 1 para obter a imagem com amostragemreduzida 20.
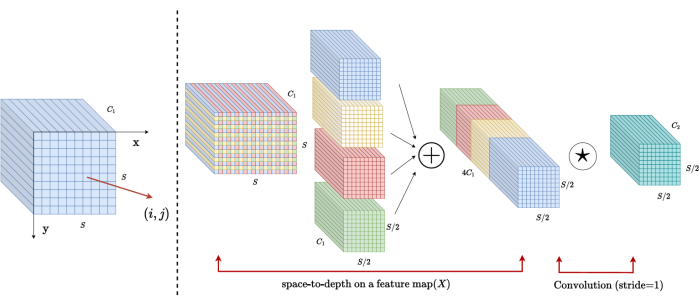
Figura 2: Diagrama de blocos SPD. Clique aqui para ver uma versão maior desta figura.
Arquitetura de blocos PSAA
Em contraste com as redes tradicionais da CNN, o bloco PSAA proposto é mais eficaz na condução do foco global de informações e mais eficiente no aprendizado e treinamento da rede. Isso permite a captura de imagens e recursos espaciais mais ricos. O bloco PSAA inclui aprendizado de atenção axial com base no compartilhamento de parâmetros em três dimensões: Altura, Largura e Profundidade. Em comparação com o mecanismo de atenção convencional que realiza o aprendizado de atenção para cada pixel da imagem, esse método conduz o aprendizado de atenção de forma independente para cada uma das três dimensões, reduzindo a complexidade da autoatenção de quadrática para linear. Além disso, um mecanismo de compartilhamento de parâmetros de consultas de chaves que podem ser aprendidas é empregado, permitindo que a rede execute operações de mecanismo de atenção em paralelo nas três dimensões, resultando em uma representação de recursos mais rápida, superior e eficaz.
Access restricted. Please log in or start a trial to view this content.
Protocolo
O presente protocolo foi aprovado pelo Comitê de Ética da Universidade de Nantong. Envolve a avaliação inteligente e a pesquisa de dados multimodais não invasivos ou minimamente invasivos adquiridos, incluindo imagens médicas humanas, movimentos de membros e imagens vasculares, utilizando tecnologia de inteligência artificial. A Figura 3 mostra o fluxograma geral da segmentação de múltiplos órgãos. Todos os links necessários são fornecidos na Tabela de Materiais.
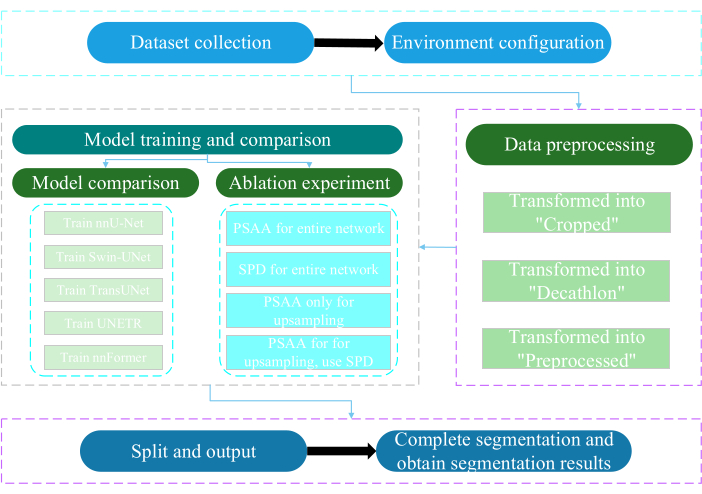
Figura 3: Fluxograma geral da segmentação de múltiplos órgãos. Clique aqui para ver uma versão maior desta figura.
1. Recolha de conjuntos de dados
- Faça o download do conjunto de dados AMOS202224 (consulte a Tabela de Materiais).
- Selecione 160 imagens como o conjunto de dados de treinamento e 40 imagens como o conjunto de dados de teste.
- Execute terminalmente mkdir ~/PaddleSeg/contrib/MedicalSeg/data/raw_data.
- Abra o caminho de raw_data.
- Coloque o conjunto de dados no diretório raw_data e garanta a correspondência correta com os nomes dos arquivos nos diretórios "imagesTr" e "labelsTr".
- Crie dataset.json arquivo.
- Adicione informações do conjunto de dados, como numTraining, numTest e nome de rótulos , ao arquivo "dataset.json".
NOTA: O conjunto de dados para este estudo consiste em 200 conjuntos de dados de TC de alta qualidade selecionados do conjunto de dados oficial de 500 CT da AMOS2022 para fins de treinamento e teste. O conjunto de dados abrange 11 órgãos, incluindo baço, rim direito, rim esquerdo, vesícula biliar, esôfago, fígado, estômago, aorta, veia cava inferior, pâncreas e bexiga.
2. Configuração do ambiente
- Instale o ambiente Paddlepaddle (consulte a Tabela de Materiais) e o CUDA versão25 correspondente.
NOTA: Recomenda-se utilizar o Paddlepaddle versão 2.5.0 e CUDA versão 11.7. - Git clone o repositório PaddleSeg25.
- Execute terminalmente cd ~/PaddleSeg/contrib/MedicalSeg/.
- Execute pip install -r requirements.txt.
- Execute pip install medpy.
3. Pré-processamento de dados
- Modifique o arquivo "yml", incluindo parâmetros como data_root, batch_size, modelo, train_dataset, val_dataset, otimizador, lr_scheduler e perda.
- Especifique os parâmetros necessários para a execução, incluindo "-config, --log_iters, --precision, --nnunet, --save_dir, --save_interval, etc.
NOTA: O "raw_data" original é transformado em três tipos de arquivos de dados: "Cropped", "Decathlon" e "Preprocessed", atendendo a diferentes requisitos de treinamento e avaliação para melhorar o desempenho do modelo e a eficácia do treinamento.
4. Treinamento e comparação de modelos
NOTA: Como uma linha de base amplamente utilizada no campo da segmentação de imagens, o nnU-Net23 serve como um modelo de linha de base no estudo. O processo de comparação de modelos específicos é o seguinte.
- Trem nnU-Net
- Configure com um tamanho de lote de 2 e 80.000 iterações, registre a cada 20 iterações e utilize a precisão fp16.
- Defina o save_dir como a pasta "saída" dentro do diretório de trabalho, com um intervalo de salvamento definido em 1.000 iterações.
- Adicione o parâmetro --use_vdl para visualização do parâmetro VisualDL.
- Execute train.py.
- Crie quatro arquivos yml , especifique as dobras de 1 a 4 e treine o modelo com validação cruzada quíntupla.
- Trem Swin-UNet
- Manter a consistência com as condições experimentais previamente estabelecidas.
- Remova o parâmetro --nnunet e use --swinunet.
- Execute o arquivo train.py.
- Treine dobras de 1 a 4 individualmente e, em seguida, sujeito ao processamento do conjunto.
- Trem TransUNet
- Siga os procedimentos descritos na etapa 4.1.
- Abra o arquivo yml .
- Modifique type: nnunet para type: transunet.
- Modifique o parâmetro --nnunet para --transunet.
- Execute o arquivo train.py.
- Treine as dobras 1-4 individualmente e processe o processamento do conjunto.
- Trem UNETR
- Mantenha variáveis experimentais consistentes.
- Repita os procedimentos descritos anteriormente, como Swin-UNet e TransUNet (etapa 4.2 e etapa 4.3).
- Execute train.py.
- Treine as dobras 1-4 individualmente e processe o processamento do conjunto.
- Formador de Trem
- Modifique os parâmetros como Swin-UNet e TransUNet.
- Execute uma única dobra train.py.
- Execute o processamento de conjunto.
5. Experiência de ablação
- Bloco PSAA para toda a rede
- Mantenha as variáveis consistentes com a etapa 4.1.
- Conjunto volume_shape = (64.192.192) e Axial_attention = Verdadeiro.
- Execute uma única dobra train.py.
- Processe o processamento do conjunto em cinco dobras.
- Bloco SPD para toda a rede
- Mantenha as variáveis consistentes com a etapa 4.1.
- Defina SPD_enable = Verdadeiro e Axial_attention = Falso.
- Execute uma única dobra train.py.
- Processe o processamento do conjunto em cinco dobras.
- Bloco PSAA para aumento da amostragem
NOTA: Não use o bloco SPD para downsampling.- Use o bloco PSAA para upsampling.
- Mantenha as variáveis consistentes com a etapa 4.1.
- Defina Axial_attention = False na parte de redução da resolução e SPD_enable = False.
- Execute uma única dobra train.py.
- Processe o processamento do conjunto de cinco dobras.
- Bloco PSAA para aumento da amostragem
NOTA: Use o bloco SPD para downsampling.- Use o bloco SPD para redução da amostragem e o bloco PSAA para upsampling.
- Mantenha as variáveis consistentes com a etapa 4.1.
- Defina Axial_attention = False na parte de redução da resolução e SPD_enable = True.
- Execute uma única dobra train.py.
- Processe o processamento do conjunto de cinco dobras.
- Defina o número de iterações como 2.00.000 devido aos resultados superiores.
Access restricted. Please log in or start a trial to view this content.
Resultados
Este protocolo emprega duas métricas para avaliar o modelo: Dice Similarity Score (DSC) e 95% Hausdorff Distance (HD95). O DSC mede a sobreposição entre as previsões de segmentação de voxel e a verdade fundamental, enquanto o 95% HD avalia a sobreposição entre os limites de previsão de segmentação de voxel e a verdade fundamental, filtrando 5% dos valores discrepantes. A definição de DSC26 é a seguinte:
Access restricted. Please log in or start a trial to view this content.
Discussão
A segmentação dos órgãos abdominais é um trabalho complicado. Em comparação com outras estruturas internas do corpo humano, como o cérebro ou o coração, a segmentação dos órgãos abdominais parece mais desafiadora devido ao baixo contraste e às grandes mudanças de forma nas imagens de TC27,28. Swin-PSAxialNet é proposto aqui para resolver este difícil problema.
Na etapa de coleta de da...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Os autores declaram não haver conflitos de interesse.
Agradecimentos
Este estudo foi apoiado pelo Projeto de Engenharia '333' da Província de Jiangsu ([2022]21-003), pelo Programa Geral da Comissão de Saúde de Wuxi (M202205) e pelo Fundo de Desenvolvimento de Ciência e Tecnologia de Wuxi (Y20212002-1), cujas contribuições foram inestimáveis para o sucesso deste trabalho." Os autores agradecem a todos os assistentes de pesquisa e participantes do estudo por seu apoio.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| AMOS2022 dataset | None | None | Datasets for network training and testing. The weblink is: https://pan.baidu.com/s/1x2ZW5FiZtVap0er55Wk4VQ?pwd=xhpb |
| ASUS mainframe | ASUS | https://www.asusparts.eu/en/asus-13020-01910200 | |
| CUDA version 11.7 | NVIDIA | https://developer.nvidia.com/cuda-11-7-0-download-archive | |
| NVIDIA GeForce RTX 3090 | NVIDIA | https://www.nvidia.com/en-in/geforce/graphics-cards/30-series/rtx-3090-3090ti/ | |
| Paddlepaddle environment | Baidu | None | Environmental preparation for network training. The weblink is: https://www.paddlepaddle.org.cn/ |
| PaddleSeg | Baidu | None | The baseline we use: https://github.com/PaddlePaddle/PaddleSeg |
Referências
- Liu, X., Song, L., Liu, S., Zhang, Y. A review of deep-learning-based medical image segmentation methods. Sustain. 13 (3), 1224(2021).
- Popilock, R., Sandrasagaren, K., Harris, L., Kaser, K. A. CT artifact recognition for the nuclear technologist. J Nucl Med Technol. 36 (2), 79-81 (2008).
- Rehman, A., Khan, F. G. A deep learning based review on abdominal images. Multimed Tools Appl. 80, 30321-30352 (2021).
- Schenck, J. F. The role of magnetic susceptibility in magnetic resonance imaging: Mri magnetic compatibility of the first and second kinds. Med Phys. 23 (6), 815-850 (1996).
- Palmisano, A., et al. Myocardial late contrast enhancement ct in troponin-positive acute chest pain syndrome. Radiol. 302 (3), 545-553 (2022).
- Chen, E. -L., Chung, P. -C., Chen, C. -L., Tsai, H. -M., Chang, C. -I. An automatic diagnostic system for CT liver image classification. IEEE Trans Biomed Eng. 45 (6), 783-794 (1998).
- Sagel, S. S., Stanley, R. J., Levitt, R. G., Geisse, G. J. R. Computed tomography of the kidney. Radiol. 124 (2), 359-370 (1977).
- Freeman, J. L., Jafri, S., Roberts, J. L., Mezwa, D. G., Shirkhoda, A. CT of congenital and acquired abnormalities of the spleen. Radiographics. 13 (3), 597-610 (1993).
- Almeida, R. R., et al. Advances in pancreatic CT imaging. AJR Am J Roentgenol. 211 (1), 52-66 (2018).
- Eisen, G. M., et al. Guidelines for credentialing and granting privileges for endoscopic ultrasound. Gastrointest Endosc. 54 (6), 811-814 (2001).
- Sykes, J. Reflections on the current status of commercial automated segmentation systems in clinical practice. J Med Radiat Sci. 61 (3), 131-134 (2014).
- He, K., Zhang, X., Ren, S., Sun, J. Deep residual learning for image recognition. 2016 IEEE Conference on Computer Vision and Pattern Recognition (CVPR). , Vegas, NV, USA. 770-778 (2016).
- Dosovitskiy, A., et al. An image is worth 16x16 words: Transformers for image recognition at scale. arXiv. arXiv. , 11929(2020).
- Ramachandran, P., et al. Stand-alone self-attention in vision models. Adv Neural Inf Process Syst. 32, NIPS2019 (2019).
- Vaswani, A., et al. Attention is all you need. Adv Neural Inf Process Syst. 30, NIPS2019 (2017).
- Chen, J., et al. TransUNET: Transformers make strong encoders for medical image segmentation. arXiv. , arXiv:2102 04396(2021).
- Liu, Z., et al. Swin transformer: Hierarchical vision transformer using shifted windows. Proc IEEE Int Conf Comput Vis. , 10012-10022 (2021).
- Cao, H., et al. Swin-UNET: Unet-like pure transformer for medical image segmentation. Comput Vis ECCV. , 205-218 (2022).
- Zhou, H. -Y., et al. nnformer: Interleaved transformer for volumetric segmentation. arXiv. arXiv. , arXiv:2109 03201(2021).
- Sunkara, R., Luo, T. No more strided convolutions or pooling: A new CNN building block for low-resolution images and small objects. Mach Learn Knowl Discov Databases. , 443-459 (2023).
- Çiçek, Ö, Abdulkadir, A., Lienkamp, S. S., Brox, T., Ronneberger, O. 3D U-Net: learning dense volumetric segmentation from sparse annotation. Med Comput Vis Bayesian Graph Models Biomed Imaging. 2016, 424-432 (2016).
- Kim, H., et al. Abdominal multi-organ auto-segmentation using 3D-patch-based deep convolutional neural network. Scientific reports. 10 (1), 6204(2020).
- Isensee, F., et al. nnu-net: Self-adapting framework for u-net-based medical image segmentation. arXiv. , arXiv:1809 10486(2018).
- Ji, Y., et al. A large-scale abdominal multi-organ benchmark for versatile medical image segmentation. Adv Neural Inf Process Syst. 35, 36722-36732 (2022).
- Liu, Y., et al. Paddleseg: A high-efficient development toolkit for image segmentation. arXiv. , arXiv:2101 06175(2021).
- Hatamizadeh, A., et al. UNETR: Transformers for 3D medical image segmentation. Proc IEEE Int Conf Comput Vis. , https://api.semanticscholar.org/CorpusID:232290634 574-584 (2022).
- Zhou, H., Zeng, D., Bian, Z., Ma, J. A semi-supervised network-based tissue-aware contrast enhancement method for ct images. Nan Fang Yi Ke Da Xue Xue Bao. 43 (6), 985-993 (2023).
- Ma, J., et al. Abdomenct-1k: Is abdominal organ segmentation a solved problem. IEEE Trans Pattern Anal Mach Intell. 44 (10), 6695-6714 (2021).
- Galván, E., Mooney, P. Neuroevolution in deep neural networks: Current trends and future challenges. IEEE Trans Artif Intell. 2 (6), 476-493 (2021).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados