このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
Swin-PSAxialNet:効率的な多臓器セグメンテーション技術
要約
本プロトコルは、Swin-PSAxialNetと呼ばれる効率的な多臓器セグメンテーション法を記述しており、これは以前のセグメンテーション方法と比較して優れた精度を達成しています。この手順の主なステップには、データセットの収集、環境設定、データの前処理、モデルのトレーニングと比較、アブレーション実験が含まれます。
要約
腹部多臓器セグメンテーションは、医用画像解析の分野で最も重要なトピックの1つであり、疾患診断や治療計画などの臨床ワークフローを支える重要な役割を果たしています。本研究では、nnU-Netアーキテクチャに基づく効率的な多臓器セグメンテーション法Swin-PSAxialNetを提案します。これは、CT画像内の11の腹部臓器の正確なセグメンテーションのために特別に設計されました。提案ネットワークは、nnU-Netと比較して以下の改善が行われています。まず、SPD(Space-to-Depth)モジュールとPSAA(Parameter-Shared Axial Attention)特徴抽出ブロックが導入され、3D画像特徴抽出の能力が向上しました。次に、詳細な情報と空間的特徴をキャプチャするためにマルチスケール画像融合アプローチが採用され、微妙な特徴とエッジ特徴を抽出する能力が向上しました。最後に、モデルの計算コストとトレーニング速度を削減するために、パラメーター共有方法が導入されました。提案されたネットワークは、11 の臓器が関与するセグメンテーション タスクに対して 0.93342 の平均ダイス係数を達成します。実験結果は、Swin-PSAxialNetが以前の主流のセグメンテーション方法よりも顕著な優位性を示していることを示しています。この方法は、主要な腹部臓器のセグメンテーションにおいて優れた精度と低い計算コストを示しています。
概要
疾患の診断、治療計画の策定、治療結果の追跡など、現代の臨床介入は、医療画像の正確なセグメンテーションに依存しています1。しかし、腹部臓器2間の複雑な構造的関係により、複数の腹部臓器3の正確なセグメンテーションを達成することは困難な課題となっています。過去数十年にわたり、医用画像処理とコンピュータビジョンの盛んな発展は、腹部の多臓器セグメンテーションの分野において新たな機会と課題の両方をもたらしてきました。高度な磁気共鳴画像法(MRI)※4やCT(コンピュータ断層撮影)技術※5により、高解像度の腹部画像を取得することができます。CT画像から複数の臓器を正確にセグメンテーションすることは、肝臓、腎臓、脾臓、膵臓などの重要な臓器の評価と治療に大きな臨床的価値を持っています6,7,8,9,10 しかし、これらの解剖学的構造、特に放射線科医や放射線腫瘍医からの介入が必要な解剖学的構造の手動注釈は、時間がかかり、主観的な影響を受けやすいです11.したがって、腹部の多臓器セグメンテーションのための自動化された正確な方法を開発することが急務です。
画像セグメンテーションに関する以前の研究では、主に畳み込みニューラルネットワーク(CNN)に依存しており、レイヤーを積み重ねてResNet12を導入することでセグメンテーション効率を向上させていました。2020 年、Google の研究チームは Vision Transformer(VIT)モデル13 を導入し、Transformer アーキテクチャを従来の視覚領域に組み込んでさまざまな視覚タスク14 を組み込んだ先駆的な事例となりました。畳み込み演算は局所的な特徴情報しか考慮できませんが、Transformers のアテンション メカニズムにより、グローバルな特徴情報を包括的に考慮できます。
従来の畳み込みネットワーク15に対するTransformerベースのアーキテクチャの優位性を考慮して、多数の研究チームが、Transformerと畳み込みネットワーク16,17,18,19の強みとの間の相乗効果を最適化するための広範な調査を行ってきた。Chenらは、Transformerを活用して画像からグローバルな特徴を抽出する、医用画像セグメンテーションタスク16用のTransUNetを導入しました。ネットワークトレーニングのコストが高く、特徴抽出階層の概念を利用できないため、Transformerの利点は完全には実現されていません。
これらの問題に対処するために、多くの研究者がセグメンテーションネットワークの学習のバックボーンとしてTransformerを組み込む実験を始めています。Liuら17 は、階層化された特徴抽出のための階層的構築方法を採用したSwin Transformerを導入しました。Windows Multi-Head Self-Attention (W-MSA) の概念が提案され、特に大きな浅いレベルの特徴マップが存在する場合に、計算コストが大幅に削減されました。このアプローチにより、計算要件が軽減される一方で、異なるウィンドウ間での情報伝送も分離されました。この問題に対処するために、著者たちはさらに、隣接するウィンドウ間での情報伝達を可能にするShifted Windows Multi-Head Self-Attention(SW-MSA)の概念を導入しました。この方法論に基づいて、CaoらはSwin-UNet18を定式化し、U-Netの2D畳み込みをSwinモジュールに置き換え、W-MSAとSW-MSAをエンコードおよびデコードプロセスに組み込み、称賛に値するセグメンテーション結果を達成しました。
逆に、Zhouらは、高解像度の画像を処理する際にconv演算の利点を無視できないことを強調した19。彼らが提案するnnFormerは、局所的な3次元画像ブロックに基づくセルフアテンション計算法を採用し、十字型構造を特徴とするTransformerモデルを構成する。ローカルの3次元ブロックに基づくアテンションの利用により、ネットワークの学習負荷が大幅に軽減されました。
上記の研究の問題点を踏まえて、3D医用画像セグメンテーションのための効率的なハイブリッド階層構造であるSwin-PSAxialNetが提案されています。この方法には、ダウンサンプリングブロックであるSpace-to-depth(SPD)20 ブロックが組み込まれており、グローバル情報21を抽出できる。さらに、パラメータ共有アキシャルアテンション(PSAA)モジュールを追加し、これにより学習パラメータ数が二次から線形に減少し、ネットワークトレーニングの精度とトレーニングモデル22の複雑さに良い影響を与える。
Swin-PSAxialNetネットワーク
ネットワークの全体的なアーキテクチャは、エンコーダとデコーダの構造からなるnnU-Net23のU字型構造を採用しています。これらの構造は、 図 1 に示すように、ローカルな特徴抽出と、大小の画像からの特徴の連結に関与します。
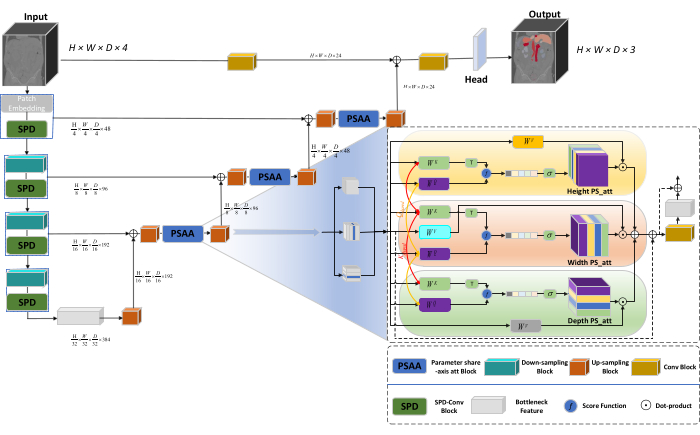
図 1: ネットワーク アーキテクチャの Swin-PSAxialNet 概略図。この図の拡大版を表示するには、ここをクリックしてください。
エンコーダ構造では、従来のConvブロックがSPDブロック20と組み合わされて、ダウンサンプリングボリュームが形成されます。エンコーダの最初の層には、3Dデータを3Dパッチに分割するモジュールであるパッチ埋め込みが組み込まれています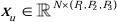 (P1、P2、P 3)は、このコンテキストでは重複しないパッチを表し、
(P1、P2、P 3)は、このコンテキストでは重複しないパッチを表し、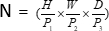 3Dパッチのシーケンス長を示します。埋め込み層に続いて、次のステップには、畳み込みブロックと SPD ブロックの両方で構成されるオーバーラップしない畳み込みダウンサンプリング ユニットが含まれます。この設定では、畳み込みブロックのストライドが 1 に設定され、SPD ブロックがイメージ スケーリングに使用されるため、解像度が 4 倍、チャネルが 2 倍に増加します。
3Dパッチのシーケンス長を示します。埋め込み層に続いて、次のステップには、畳み込みブロックと SPD ブロックの両方で構成されるオーバーラップしない畳み込みダウンサンプリング ユニットが含まれます。この設定では、畳み込みブロックのストライドが 1 に設定され、SPD ブロックがイメージ スケーリングに使用されるため、解像度が 4 倍、チャネルが 2 倍に増加します。
デコーダー構造では、Bottleneck Feature 層の後の各アップサンプル ブロックは、アップサンプリング ブロックと PSAA ブロックの組み合わせで構成されます。特徴マップの解像度は 2 倍に向上し、デコーダ ステージの各ペア間でチャネル数が半分になります。空間情報を復元し、フィーチャ表現を強化するために、アップサンプリング ブロック間で大縮尺イメージと小縮尺イメージ間のフィーチャ フュージョンが実行されます。最終的に、アップサンプリングの結果は Head レイヤーに供給され、出力サイズが (H × W × D × C, C = 3) の元の画像サイズに戻ります。
SPD ブロックのアーキテクチャ
従来の方法では、ダウンサンプリング セクションでは、ステップ サイズが 2 の 1 ストライドが採用されていました。これには、画像内の局所的な位置での畳み込みプーリング、受容野の制限、およびモデルを小さな画像パッチからの特徴の抽出に制限することが含まれます。この方法では、元の画像を3次元に細かく分割するSPDブロックを利用します。元の 3D イメージは、x、y、z 軸に沿って均等にセグメント化され、4 つのサブボリューム ボディになります。(図2)続いて、4つのボリュームを「cat」操作によって連結し、得られた画像を1×1×1畳み込みを受けて、ダウンサンプリングされた画像20を得る。
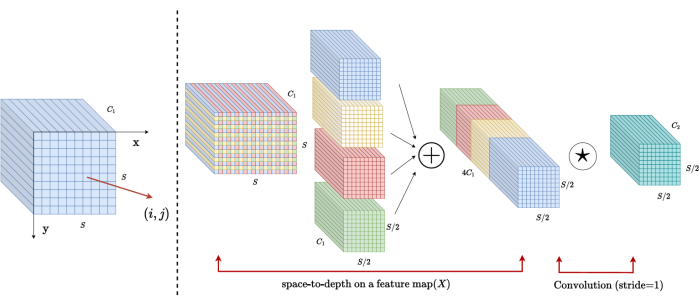
図 2: SPD のブロック図。この図の拡大版を表示するには、ここをクリックしてください。
PSAA ブロックのアーキテクチャ
従来のCNNネットワークとは対照的に、提案されているPSAAブロックは、グローバルな情報に焦点を当てる上でより効果的であり、ネットワークの学習とトレーニングにおいてより効率的です。これにより、より豊富な画像と空間的特徴をキャプチャできます。PSAA ブロックには、高さ、幅、深さの 3 次元で共有するパラメーターに基づく軸方向の注意学習が含まれています。従来のアテンションメカニズムが画像内の各ピクセルに対してアテンション学習を行うのに対し、この手法は3次元ごとに独立してアテンション学習を行うため、セルフアテンションの複雑さを2次から線形に軽減します。さらに、学習可能なキークエリパラメータ共有メカニズムが採用されているため、ネットワークは3次元にわたってアテンションメカニズムの操作を並行して実行できるため、より速く、優れており、より効果的な特徴表現が得られます。
プロトコル
本プロトコルは、南通大学の倫理委員会によって承認されました。これには、人工知能技術を利用して、人間の医療画像、手足の動き、血管イメージングなど、取得した非侵襲的または低侵襲のマルチモーダルデータのインテリジェントな評価と研究が含まれます。 図3 は、多臓器セグメンテーションの全体的なフローチャートを示しています。必要なすべてのウェブリンクは 、資料の表に記載されています。
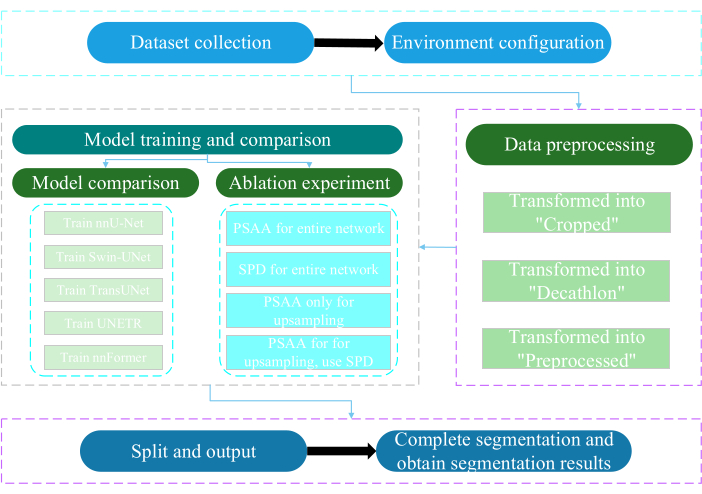
図3:多臓器セグメンテーションの全体的なフローチャート。この図の拡大版を表示するには、ここをクリックしてください。
1. データセットの収集
- AMOS2022データセット24 をダウンロードします( 資料の表を参照)。
- トレーニングデータセットとして 160 枚の画像を選択し、テストデータセットとして 40 枚の画像を選択します。
- mkdir ~/PaddleSeg/contrib/MedicalSeg/data/raw_data を最後に実行します。
- raw_dataのパスを開きます。
- データセットを raw_data ディレクトリに配置し、"imagesTr" ディレクトリと "labelsTr" ディレクトリのファイル名と正しく対応していることを確認します。
- dataset.jsonファイルを作成します。
- numTraining、numTest、ラベル名などのデータセット情報を「dataset.json」ファイルに追加します。
注: この研究のデータセットは、トレーニングおよびテスト目的でAMOS2022公式の 500 CT データセットから選択された 200 の高品質 CT データセットで構成されています。このデータセットには、脾臓、右腎臓、左腎臓、胆嚢、食道、肝臓、胃、大動脈、下大静脈、膵臓、膀胱を含む 11 の臓器が含まれています。
2. 環境設定
- パドルパドル環境( 資料の表を参照)と対応するCUDAバージョン25をインストールします。
注意: Paddlepaddle バージョン 2.5.0 と CUDA バージョン 11.7 の使用をお勧めします。 - GitクローンPaddleSegリポジトリ25。
- cd ~/PaddleSeg/contrib/MedicalSeg/ を末端的に実行します。
- pip install -r requirements.txt を実行します。
- pip install medpy を実行します。
3. データの前処理
- data_root、batch_size、model、train_dataset、val_dataset、optimizer、lr_scheduler、loss などのパラメータを含む "yml" ファイルを変更します。
- 「-config, --log_iters, --precision, --nnunet, --save_dir, --save_interval など、実行に必要なパラメータを指定します。
注: 元の "raw_data" は、"Cropped"、"Decathlon"、"Preprocessed" の 3 種類のデータ ファイルに変換され、モデルのパフォーマンスとトレーニングの有効性を向上させるためのさまざまなトレーニングと評価の要件に対応します。
4. モデルのトレーニングと比較
注:画像セグメンテーションの分野で広く使用されているベースラインとして、nnU-Net23 は本研究のベースラインモデルとして機能します。具体的なモデル比較プロセスは次のとおりです。
- 電車nnU-Net
- バッチ サイズを 2 回と 80,000 回の反復で構成し、20 回の反復ごとにログを記録し、fp16 の精度を利用します。
- save_dirを作業ディレクトリ内の「output」フォルダとして設定し、保存間隔を 1,000 回の反復に設定します。
- VisualDL パラメーターの視覚化にパラメーター --use_vdl を追加します。
- train.py を実行します。
- 4 つの yml ファイルを作成し、フォールド 1 から 4 を指定し、5 フォールド交差検証でモデルをトレーニングします。
- スウィン-UNetの列車
- 以前に確立された実験条件との一貫性を維持します。
- パラメータ --nnunet を削除し、 --swinunet を使用します。
- train.py ファイルを実行します。
- 列車は1〜4を個別に折り畳み、アンサンブル処理を行います。
- 鉄道 TransUNet
- 手順 4.1 で概説されている手順に従います。
- ymlファイルを開きます。
- type: nnunet を type: transunet に変更します。
- パラメータ --nnunet を --transunet に変更します。
- train.py ファイルを実行します。
- 折り畳み1〜4を個別にトレインし、アンサンブル処理を処理します。
- UNETRの列車
- 一貫した実験変数を維持します。
- Swin-UNet や TransUNet など、前に説明した手順を繰り返します (手順 4.2 と手順 4.3)。
- train.py を実行します。
- 折り畳み1〜4を個別にトレインし、アンサンブル処理を処理します。
- トレイン nnFormer
- Swin-UNet や TransUNet などのパラメーターを変更します。
- シングルフォールド train.py を実行します。
- アンサンブル処理を実行します。
5. アブレーション実験
- ネットワーク全体のPSAAブロック
- 変数をステップ 4.1 と一貫性を保ちます。
- volume_shape = (64,192,192) と Axial_attention = True を設定します。
- シングルフォールド train.py を実行します。
- 5つのフォールドにわたるアンサンブル処理を処理します。
- ネットワーク全体のSPDブロック
- 変数をステップ 4.1 と一貫性を保ちます。
- SPD_enable = True と Axial_attention = False を設定します。
- 一折り train.py を実行します。
- 5つのフォールドにわたるアンサンブル処理を処理します。
- アップサンプリング用の PSAA ブロック
注:ダウンサンプリングにSPDブロックを使用しないでください。- アップサンプリングには PSAA ブロックを使用します。
- 変数をステップ 4.1 と一貫性を保ちます。
- ダウンサンプリング部分で Axial_attention = False と SPD_enable = False を設定します。
- シングルフォールド train.py を実行します。
- 5つ折りアンサンブル加工を加工します。
- アップサンプリング用の PSAA ブロック
注:ダウンサンプリングにはSPDブロックを使用します。- ダウンサンプリングには SPD ブロックを使用し、アップサンプリングには PSAA ブロックを使用します。
- 変数をステップ 4.1 と一貫性を保ちます。
- ダウンサンプリング部分で Axial_attention = False と SPD_enable = True を設定します。
- シングルフォールド train.py を実行します。
- 5つ折りアンサンブル加工を加工します。
- 反復回数を 2,00,000 に設定すると、結果が優れているためです。
結果
このプロトコルでは、モデルを評価するために、サイコロ類似性スコア (DSC) と 95% ハウスドルフ距離 (HD95) の 2 つのメトリックを使用します。DSC はボクセル セグメンテーション予測とグラウンド トゥルースの間のオーバーラップを測定し、95% HD はボクセル セグメンテーション予測境界とグラウンド トゥルースの間のオーバーラップを評価し、外れ値の 5% をフィル...
ディスカッション
腹部臓器のセグメンテーションは複雑な作業です。脳や心臓など、人体の他の内部構造と比較して、CT画像のコントラストが低く、形状変化が大きいため、腹部臓器のセグメンテーションはより困難に思えます27,28。ここでは、この難しい問題を解決するためにSwin-PSAxialNetを提案しています。
データ収?...
開示事項
著者は、利益相反を宣言しません。
謝辞
この研究は、江蘇省の「333」エンジニアリングプロジェクト([2022]21-003)、無錫衛生委員会一般プログラム(M202205)、無錫科学技術開発基金(Y20212002-1)の支援を受けており、彼らの貢献はこの研究の成功に非常に貴重です。」著者らは、すべての研究助手と研究参加者の支援に感謝します。
資料
| Name | Company | Catalog Number | Comments |
| AMOS2022 dataset | None | None | Datasets for network training and testing. The weblink is: https://pan.baidu.com/s/1x2ZW5FiZtVap0er55Wk4VQ?pwd=xhpb |
| ASUS mainframe | ASUS | https://www.asusparts.eu/en/asus-13020-01910200 | |
| CUDA version 11.7 | NVIDIA | https://developer.nvidia.com/cuda-11-7-0-download-archive | |
| NVIDIA GeForce RTX 3090 | NVIDIA | https://www.nvidia.com/en-in/geforce/graphics-cards/30-series/rtx-3090-3090ti/ | |
| Paddlepaddle environment | Baidu | None | Environmental preparation for network training. The weblink is: https://www.paddlepaddle.org.cn/ |
| PaddleSeg | Baidu | None | The baseline we use: https://github.com/PaddlePaddle/PaddleSeg |
参考文献
- Liu, X., Song, L., Liu, S., Zhang, Y. A review of deep-learning-based medical image segmentation methods. Sustain. 13 (3), 1224 (2021).
- Popilock, R., Sandrasagaren, K., Harris, L., Kaser, K. A. CT artifact recognition for the nuclear technologist. J Nucl Med Technol. 36 (2), 79-81 (2008).
- Rehman, A., Khan, F. G. A deep learning based review on abdominal images. Multimed Tools Appl. 80, 30321-30352 (2021).
- Schenck, J. F. The role of magnetic susceptibility in magnetic resonance imaging: Mri magnetic compatibility of the first and second kinds. Med Phys. 23 (6), 815-850 (1996).
- Palmisano, A., et al. Myocardial late contrast enhancement ct in troponin-positive acute chest pain syndrome. Radiol. 302 (3), 545-553 (2022).
- Chen, E. -. L., Chung, P. -. C., Chen, C. -. L., Tsai, H. -. M., Chang, C. -. I. An automatic diagnostic system for CT liver image classification. IEEE Trans Biomed Eng. 45 (6), 783-794 (1998).
- Sagel, S. S., Stanley, R. J., Levitt, R. G., Geisse, G. J. R. Computed tomography of the kidney. Radiol. 124 (2), 359-370 (1977).
- Freeman, J. L., Jafri, S., Roberts, J. L., Mezwa, D. G., Shirkhoda, A. CT of congenital and acquired abnormalities of the spleen. Radiographics. 13 (3), 597-610 (1993).
- Almeida, R. R., et al. Advances in pancreatic CT imaging. AJR Am J Roentgenol. 211 (1), 52-66 (2018).
- Eisen, G. M., et al. Guidelines for credentialing and granting privileges for endoscopic ultrasound. Gastrointest Endosc. 54 (6), 811-814 (2001).
- Sykes, J. Reflections on the current status of commercial automated segmentation systems in clinical practice. J Med Radiat Sci. 61 (3), 131-134 (2014).
- He, K., Zhang, X., Ren, S., Sun, J. Deep residual learning for image recognition. 2016 IEEE Conference on Computer Vision and Pattern Recognition (CVPR). , 770-778 (2016).
- Dosovitskiy, A., et al. An image is worth 16x16 words: Transformers for image recognition at scale. arXiv. arXiv. , 11929 (2020).
- Ramachandran, P., et al. Stand-alone self-attention in vision models. Adv Neural Inf Process Syst. 32, (2019).
- Vaswani, A., et al. Attention is all you need. Adv Neural Inf Process Syst. 30, (2017).
- Chen, J., et al. TransUNET: Transformers make strong encoders for medical image segmentation. arXiv. , 04396 (2021).
- Liu, Z., et al. Swin transformer: Hierarchical vision transformer using shifted windows. Proc IEEE Int Conf Comput Vis. , 10012-10022 (2021).
- Cao, H., et al. Swin-UNET: Unet-like pure transformer for medical image segmentation. Comput Vis ECCV. , 205-218 (2022).
- Zhou, H. -. Y., et al. nnformer: Interleaved transformer for volumetric segmentation. arXiv. arXiv. , 03201 (2021).
- Sunkara, R., Luo, T. No more strided convolutions or pooling: A new CNN building block for low-resolution images and small objects. Mach Learn Knowl Discov Databases. , 443-459 (2023).
- Çiçek, &. #. 2. 1. 4. ;., Abdulkadir, A., Lienkamp, S. S., Brox, T., Ronneberger, O. 3D U-Net: learning dense volumetric segmentation from sparse annotation. Med Comput Vis Bayesian Graph Models Biomed Imaging. 2016, 424-432 (2016).
- Kim, H., et al. Abdominal multi-organ auto-segmentation using 3D-patch-based deep convolutional neural network. Scientific reports. 10 (1), 6204 (2020).
- Isensee, F., et al. nnu-net: Self-adapting framework for u-net-based medical image segmentation. arXiv. , 10486 (2018).
- Ji, Y., et al. A large-scale abdominal multi-organ benchmark for versatile medical image segmentation. Adv Neural Inf Process Syst. 35, 36722-36732 (2022).
- Liu, Y., et al. Paddleseg: A high-efficient development toolkit for image segmentation. arXiv. , 06175 (2021).
- Hatamizadeh, A., et al. UNETR: Transformers for 3D medical image segmentation. Proc IEEE Int Conf Comput Vis. , 574-584 (2022).
- Zhou, H., Zeng, D., Bian, Z., Ma, J. A semi-supervised network-based tissue-aware contrast enhancement method for ct images. Nan Fang Yi Ke Da Xue Xue Bao. 43 (6), 985-993 (2023).
- Ma, J., et al. Abdomenct-1k: Is abdominal organ segmentation a solved problem. IEEE Trans Pattern Anal Mach Intell. 44 (10), 6695-6714 (2021).
- Galván, E., Mooney, P. Neuroevolution in deep neural networks: Current trends and future challenges. IEEE Trans Artif Intell. 2 (6), 476-493 (2021).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved