A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
Swin-PSAxialNet: טכניקת פילוח רב-איברים יעילה
In This Article
Summary
הפרוטוקול הנוכחי מתאר שיטת פילוח יעילה מרובת איברים הנקראת Swin-PSAxialNet, שהשיגה דיוק מצוין בהשוואה לשיטות פילוח קודמות. השלבים העיקריים של הליך זה כוללים איסוף מערכי נתונים, תצורת סביבה, עיבוד מקדים של נתונים, אימון והשוואה של מודלים וניסויי אבלציה.
Abstract
פילוח רב איברים בבטן הוא אחד הנושאים החשובים ביותר בתחום ניתוח תמונות רפואיות, והוא ממלא תפקיד חשוב בתמיכה בתהליכי עבודה קליניים כגון אבחון מחלות ותכנון טיפול. במחקר זה מוצעת שיטת פילוח יעילה מרובת איברים הנקראת Swin-PSAxialNet המבוססת על ארכיטקטורת nnU-Net. הוא תוכנן במיוחד לפילוח מדויק של 11 איברי בטן בתמונות CT. הרשת המוצעת ביצעה את השיפורים הבאים בהשוואה ל-nnU-Net. ראשית, הוצגו מודולי מרחב לעומק (SPD) ובלוקים לחילוץ תכונות תשומת לב צירית משותפת לפרמטרים (PSAA), מה ששיפר את היכולת של חילוץ תכונות תמונה תלת מימדית. שנית, נעשה שימוש בגישת היתוך תמונה רב-קנה מידה כדי ללכוד מידע מפורט ותכונות מרחביות, ולשפר את היכולת לחלץ תכונות עדינות ותכונות קצה. לבסוף, הוצגה שיטת שיתוף פרמטרים כדי להפחית את עלות החישוב ומהירות האימון של המודל. הרשת המוצעת משיגה מקדם קוביות ממוצע של 0.93342 עבור משימת הפילוח הכוללת 11 איברים. תוצאות ניסיוניות מצביעות על העליונות הבולטת של Swin-PSAxialNet על פני שיטות פילוח מיינסטרים קודמות. השיטה מציגה דיוק מצוין ועלויות חישוביות נמוכות בפילוח איברי בטן עיקריים.
Introduction
התערבות קלינית עכשווית, כולל אבחון מחלות, גיבוש תוכניות טיפול ומעקב אחר תוצאות הטיפול, מסתמכת על פילוח מדויק של תמונות רפואיות1. עם זאת, היחסים המבניים המורכבים בין איברי הבטן2הופכים את זה למשימה מאתגרת להשיג פילוח מדויק של איברי בטןמרובים 3. במהלך העשורים האחרונים, ההתפתחויות המשגשגות בהדמיה רפואית וראייה ממוחשבת הציגו הזדמנויות ואתגרים חדשים בתחום הסגמנטציה של איברים מרובי איברים בבטן. הדמיית תהודה מגנטית מתקדמת (MRI)4 וטכנולוגיית טומוגרפיה ממוחשבת (CT)5 מאפשרות לנו לרכוש תמונות בטן ברזולוציה גבוהה. לפילוח המדויק של איברים מרובים מתמונות CT יש ערך קליני משמעותי להערכה וטיפול באיברים חיוניים כגון הכבד, הכליות, הטחול, הלבלב וכו'.6,7,8,9,10 עם זאת, ביאור ידני של מבנים אנטומיים אלה, במיוחד אלה הדורשים התערבות של רדיולוגים או אונקולוגים קרינתיים, הוא גם גוזל זמן וגם רגיש להשפעות סובייקטיביות 11. לכן, יש צורך דחוף בפיתוח שיטות אוטומטיות ומדויקות לפילוח רב איברים בבטן.
מחקר קודם על פילוח תמונות הסתמך בעיקר על רשתות עצביות קונבולוציוניות (CNNs), המשפרות את יעילות הפילוח על ידי ערימת שכבות והצגת ResNet12. בשנת 2020, צוות המחקר של גוגל הציג את דגם Vision Transformer (VIT)13, וסימן מופע חלוצי של שילוב ארכיטקטורת שנאי בתחום הוויזואלי המסורתי עבור מגוון משימות חזותיות14. בעוד שפעולות קונבולוציוניות יכולות להרהר רק במידע על תכונות מקומיות, מנגנון הקשב ב-Transformers מאפשר התייחסות מקיפה למידע על תכונות גלובליות.
בהתחשב בעליונותן של ארכיטקטורות מבוססות רובוטריקים על פני רשתות קונבולוציוניות מסורתיות15, צוותי מחקר רבים ביצעו מחקר מקיף באופטימיזציה של הסינרגיה בין נקודות החוזק של רובוטריקים ורשתות קונבולוציוניות 16,17,18,19. צ'ן ועמיתיו הציגו את TransUNet למשימות פילוח תמונות רפואיות16, הממנף רובוטריקים כדי לחלץ תכונות גלובליות מתמונות. בשל העלות הגבוהה של אימון הרשת והכישלון לנצל את הרעיון של היררכיית חילוץ תכונות, היתרונות של Transformer לא מומשו במלואם.
כדי לטפל בבעיות אלה, חוקרים רבים החלו להתנסות בשילוב רובוטריקים כעמוד השדרה של רשתות פילוח הדרכה. ליו ואחרים הציגו את שנאי סווין, שהשתמש בשיטת בנייה היררכית לחילוץ תכונות שכבות. הרעיון של תשומת לב עצמית מרובת ראשים של Windows (W-MSA) הוצע, והפחית משמעותית את עלות החישוב, במיוחד בנוכחות מפות תכונות גדולות יותר ברמה רדודה. בעוד שגישה זו הפחיתה את הדרישות החישוביות, היא גם בודדה העברת מידע בין חלונות שונים. כדי לטפל בבעיה זו, המחברים הציגו עוד את הרעיון של תשומת לב עצמית מרובת ראשים של חלונות מוסטים (SW-MSA), המאפשר הפצת מידע בין חלונות סמוכים. בהתבסס על מתודולוגיה זו, Cao et al. ניסחו את Swin-UNet18, החליפו את הפיתולים הדו-ממדיים ב-U-Net במודולי Swin ושילבו W-MSA ו-SW-MSA בתהליכי הקידוד והפענוח, והשיגו תוצאות פילוח ראויות לשבח.
לעומת זאת, ג'ואו ואחרים הדגישו כי לא ניתן להתעלם מהיתרון של פעולת המרה בעת עיבוד תמונות ברזולוציה גבוהה19. nnFormer המוצע שלהם משתמש בשיטת חישוב תשומת לב עצמית המבוססת על בלוקים מקומיים של תמונה תלת מימדית, המהווים מודל שנאי המאופיין במבנה בצורת צלב. ניצול הקשב המבוסס על בלוקים תלת מימדיים מקומיים הפחית משמעותית את עומס האימון ברשת.
בהתחשב בבעיות במחקר הנ"ל, מוצע מבנה היררכי היברידי יעיל לפילוח תמונות רפואיות תלת מימדיות, המכונה Swin-PSAxialNet. שיטה זו משלבת בלוק דגימה, בלוק מרחב לעומק (SPD)20 , המסוגל לחלץ מידע גלובלי21. בנוסף, הוא מוסיף מודול קשב צירי משותף (PSAA), המפחית את ספירת פרמטרי הלמידה מריבועי לליניארי וישפיע לטובה על הדיוק של אימון הרשת ועל המורכבות של מודלים של אימון22.
רשת Swin-PSAxialNet
הארכיטקטורה הכוללת של הרשת מאמצת את המבנה בצורת U של nnU-Net23, המורכב ממבני מקודד ומפענח. מבנים אלה עוסקים בחילוץ תכונות מקומיות ובשרשור של תכונות מתמונות בקנה מידה גדול וקטן, כפי שמודגם באיור 1.
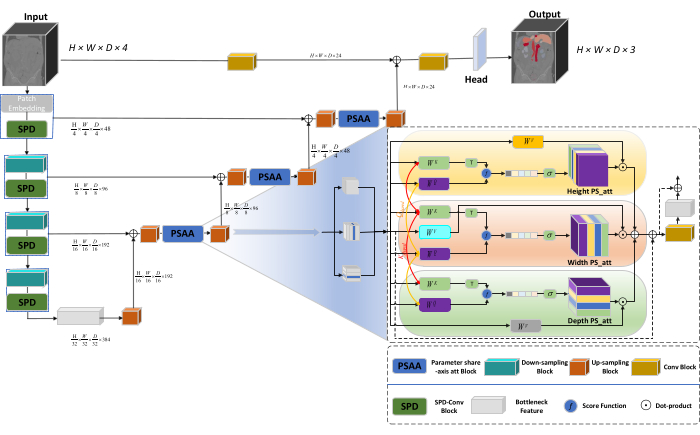
איור 1: דיאגרמה סכמטית של Swin-PSAxialNet של ארכיטקטורת רשת. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
במבנה המקודד, בלוק הקונב המסורתי משולב עם בלוק SPD20 ליצירת נפח דגימה. השכבה הראשונה של המקודד משלבת Patch Embedding, מודול המחלק את נתוני התלת מימד לתיקונים תלת מימדיים 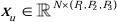 , (P1, P2, P3) מייצג טלאים לא חופפים בהקשר זה,
, (P1, P2, P3) מייצג טלאים לא חופפים בהקשר זה, 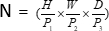 מסמן את אורך הרצף של טלאים תלת מימדיים. לאחר שכבת ההטמעה, השלב הבא כולל יחידת דגימה קונבולוציונית לא חופפת הכוללת גם בלוק קונבולוציוני וגם בלוק SPD. במערך זה, לבלוק הקונבולוציוני יש צעד מוגדר ל-1, ובלוק ה-SPD משמש לשינוי קנה מידה של תמונה, מה שמוביל להפחתה של פי ארבעה ברזולוציה ולעלייה כפולה בערוצים.
מסמן את אורך הרצף של טלאים תלת מימדיים. לאחר שכבת ההטמעה, השלב הבא כולל יחידת דגימה קונבולוציונית לא חופפת הכוללת גם בלוק קונבולוציוני וגם בלוק SPD. במערך זה, לבלוק הקונבולוציוני יש צעד מוגדר ל-1, ובלוק ה-SPD משמש לשינוי קנה מידה של תמונה, מה שמוביל להפחתה של פי ארבעה ברזולוציה ולעלייה כפולה בערוצים.
במבנה המפענח, כל בלוק דגימה לאחר שכבת תכונת צוואר הבקבוק מורכב משילוב של בלוק דגימה ובלוק PSAA. הרזולוציה של מפת התכונות גדלה פי שניים, וספירת הערוצים מצטמצמת בחצי בין כל זוג שלבי מפענח. כדי לשחזר מידע מרחבי ולשפר את ייצוג התכונות, מיזוג תכונות בין תמונות בקנה מידה גדול וקטן מתבצע בין גושי הדגימה. בסופו של דבר, תוצאות ההגדלה מוזנות לשכבת הראש כדי לשחזר את גודל התמונה המקורי, עם גודל פלט של (H × W × D × C, C = 3).
ארכיטקטורת בלוקים SPD
בשיטות מסורתיות, קטע הפחתת הדגימה משתמש בצעד בודד עם גודל צעד של 2. זה כרוך באיגום קונבולוציוני במיקומים מקומיים בתמונה, הגבלת שדה הקליטה והגבלת המודל לחילוץ תכונות מטלאי תמונה קטנים. שיטה זו משתמשת בבלוק SPD, המחלק דק את התמונה המקורית לתלת מימד. התמונה התלת-ממדית המקורית מחולקת באופן שווה לאורך צירי x, y ו-z, והתוצאה היא ארבעה גופים תת-נפחיים. (איור 2) לאחר מכן, ארבעת הכרכים משורשרים באמצעות פעולת "חתול", והתמונה המתקבלת עוברת פיתול של 1 × 1 × 1 כדי לקבל את התמונה שנדגמה למטה20.
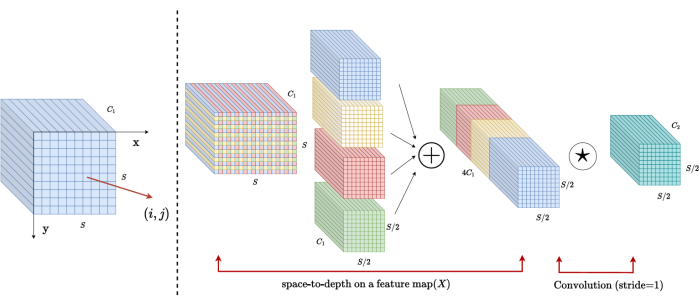
איור 2: דיאגרמת בלוקים SPD. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
ארכיטקטורת בלוק PSAA
בניגוד לרשתות CNN מסורתיות, בלוק ה-PSAA המוצע יעיל יותר בניהול מיקוד מידע גלובלי ויעיל יותר בלמידה והדרכה ברשת. זה מאפשר ללכוד תמונות עשירות יותר ותכונות מרחביות. בלוק ה-PSAA כולל למידת קשב צירית המבוססת על פרמטרים החולקים בשלושה ממדים: גובה, רוחב ועומק. בהשוואה למנגנון הקשב המקובל המבצע למידת קשב עבור כל פיקסל בתמונה, שיטה זו מבצעת באופן עצמאי למידת קשב עבור כל אחד משלושת הממדים, ומפחיתה את מורכבות הקשב העצמי מריבועית לליניארית. יתר על כן, נעשה שימוש במנגנון שיתוף פרמטרים של שאילתות מפתחות הניתן ללמידה, המאפשר לרשת לבצע פעולות מנגנון קשב במקביל על פני שלושת הממדים, וכתוצאה מכך ייצוג תכונות מהיר, מעולה ויעיל יותר.
Protocol
הפרוטוקול הנוכחי אושר על ידי ועדת האתיקה של אוניברסיטת ננטונג. זה כרוך בהערכה ומחקר חכמים של נתונים מולטי-מודאליים לא פולשניים או זעיר פולשניים שנרכשו, כולל תמונות רפואיות אנושיות, תנועות גפיים והדמיית כלי דם, תוך שימוש בטכנולוגיית בינה מלאכותית. איור 3 מתאר את תרשים הזרימה הכולל של פילוח מרובה איברים. כל קישורי האינטרנט הדרושים מסופקים בטבלת החומרים.
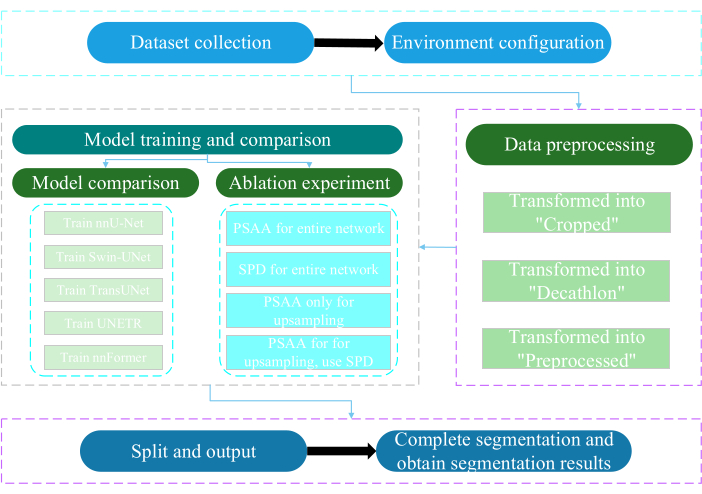
איור 3: תרשים זרימה כולל של פילוח רב-איברים. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
1. איסוף מערכי נתונים
- הורד את מערך הנתונים של AMOS202224 (ראה טבלת חומרים).
- בחר 160 תמונות כמערך נתוני האימון ו-40 תמונות כמערך נתוני הבדיקה.
- בצע באופן סופי mkdir ~/PaddleSeg/contrib/MedicalSeg/data/raw_data.
- פתח את הנתיב של raw_data.
- מקם את מערך הנתונים בספריית raw_data , וודא התאמה נכונה עם שמות הקבצים בספריות "imagesTr" ו-"labelsTr".
- צור קובץ dataset.json .
- הוסף מידע על ערכת נתונים כגון numTraining, numTest ושם תוויות לקובץ "dataset.json".
הערה: מערך הנתונים למחקר זה מורכב מ-200 מערכי נתונים איכותיים של CT שנבחרו ממערך הנתונים הרשמי של AMOS2022 500 CT למטרות אימון ובדיקה. מערך הנתונים מקיף 11 איברים, כולל הטחול, הכליה הימנית, הכליה השמאלית, כיס המרה, הוושט, הכבד, הקיבה, אבי העורקים, הווריד הנבוב התחתון, הלבלב ושלפוחית השתן.
2. תצורת סביבה
- התקן את סביבת ההנעה (ראה טבלת חומרים) ואת ה-CUDA המתאים גרסה25.
הערה: מומלץ להשתמש ב-Paddlepaddle גרסה 2.5.0 וב-CUDA גרסה 11.7. - Git שיבוט את מאגר PaddleSeg25.
- בצע באופן סופי את cd ~/PaddleSeg/contrib/MedicalSeg/.
- בצע התקנת pip -r requirements.txt.
- בצע את pip install medpy.
3. עיבוד מקדים של נתונים
- שנה את קובץ "yml", כולל פרמטרים כגון data_root, batch_size, דגם, train_dataset, val_dataset, אופטימיזציה, lr_scheduler ואובדן.
- ציין את הפרמטרים הנדרשים לביצוע, כולל "-config, --log_iters, --precision, --nnunet, --save_dir, --save_interval וכו'.
הערה: ה-"raw_data" המקורי הופך לשלושה סוגים של קבצי נתונים: "חתוך", "דקטלון" ו"מעובד מראש", הנותן מענה לדרישות הדרכה והערכה שונות כדי לשפר את ביצועי המודל ויעילות האימון.
4. הדרכה והשוואה של מודלים
הערה: כבסיס בשימוש נרחב בתחום פילוח התמונות, nnU-Net23 משמש כמודל בסיס במחקר. תהליך השוואת הדגמים הספציפי הוא כדלקמן.
- רכבת nnU-Net
- הגדר עם גודל אצווה של 2 ו-80,000 איטרציות, רשום כל 20 איטרציות והשתמש בדיוק fp16.
- הגדר את save_dir כתיקיית "פלט" בתוך ספריית העבודה, עם מרווח שמירה מוגדר ל-1,000 איטרציות.
- הוסף פרמטר --use_vdl עבור תצוגה חזותית של פרמטר VisualDL.
- הפעל train.py.
- צור ארבעה קבצי yml , ציין קיפולים 1-4 ואמן את המודל עם אימות צולב פי חמישה.
- רכבת Swin-UNet
- שמור על עקביות עם תנאי הניסוי שנקבעו בעבר.
- הסר את הפרמטר --nnunet והשתמש ב --swinunet.
- הפעל את קובץ train.py.
- הרכבת מתקפלת 1-4 בנפרד ולאחר מכן עוברת עיבוד אנסמבל.
- רכבת TransUNet
- בצע את ההליכים המתוארים בשלב 4.1.
- פתח קובץ yml .
- שינוי סוג: nnunet להקלדה: transunet.
- שנה את הפרמטר --nnunet ל --transunet.
- הפעל את קובץ train.py.
- אימון מתקפל 1-4 בנפרד ועיבוד אנסמבל.
- רכבת UNETR
- שמור על משתני ניסוי עקביים.
- חזור על ההליכים שתוארו קודם לכן כמו Swin-UNet ו- TransUNet (שלב 4.2 ושלב 4.3).
- רוץ train.py.
- אימון מתקפל 1-4 בנפרד ועיבוד אנסמבל.
- Train nnFormer
- שנה את הפרמטרים כמו Swin-UNet ו- TransUNet.
- הפעל קיפול יחיד train.py.
- הפעל עיבוד אנסמבל.
5. ניסוי אבלציה
- בלוק PSAA עבור הרשת כולה
- שמור על עקביות המשתנים עם שלב 4.1.
- הגדר volume_shape = (64,192,192) ו- Axial_attention = True.
- הפעל קיפול יחיד train.py.
- עיבוד אנסמבל תהליכים על פני חמישה קיפולים.
- בלוק SPD לכל הרשת
- שמור על עקביות המשתנים עם שלב 4.1.
- הגדר SPD_enable = True ו- Axial_attention = False.
- הפעל קיפול יחיד train.py.
- עיבוד אנסמבל תהליכים על פני חמישה קיפולים.
- בלוק PSAA לדגימה נוספת
הערה: אל תשתמש בבלוק SPD להפחתת דגימה.- השתמש בבלוק PSAA לדגימה נוספת.
- שמור על עקביות המשתנים עם שלב 4.1.
- הגדר Axial_attention = False בחלק של הפחתת הדגימה ו- SPD_enable = False.
- הפעל קיפול יחיד train.py.
- עיבוד אנסמבל חמישה קיפולים.
- בלוק PSAA לדגימה נוספת
הערה: השתמש בבלוק SPD להפחתת דגימה.- השתמש בבלוק SPD להפחתת דגימה ובבלוק PSAA לדגימה.
- שמור על עקביות המשתנים עם שלב 4.1.
- הגדר Axial_attention = False בחלק של הפחתת הדגימה ו- SPD_enable = True.
- הפעל קיפול יחיד train.py.
- עיבוד אנסמבל חמישה קיפולים.
- הגדר את מספר החזרות ל- 2,00,000 עקב התוצאות המעולות.
תוצאות
פרוטוקול זה משתמש בשני מדדים כדי להעריך את המודל: ציון דמיון בקוביות (DSC) ומרחק האוסדורף 95% (HD95). DSC מודד את החפיפה בין תחזיות פילוח ווקסל לאמת הקרקע, בעוד ש-95% HD מעריך את החפיפה בין גבולות חיזוי סגמנטציה של ווקסל לבין האמת הקרקעית, ומסנן 5% מהחריגים. ההגדרה של DSC26 ?...
Discussion
פילוח איברי הבטן הוא עבודה מורכבת. בהשוואה למבנים פנימיים אחרים של גוף האדם, כגון המוח או הלב, פילוח איברי הבטן נראה מאתגר יותר בגלל הניגודיות הנמוכה ושינויי הצורה הגדולים בתמונות CT27,28. Swin-PSAxialNet מוצע כאן כדי לפתור בעיה קשה זו.
Disclosures
המחברים מצהירים שאין ניגודי אינטרסים.
Acknowledgements
מחקר זה נתמך על ידי פרויקט ההנדסה '333' של מחוז ג'יאנגסו ([2022]21-003), התוכנית הכללית של ועדת הבריאות של וושי (M202205) וקרן פיתוח המדע והטכנולוגיה של וושי (Y20212002-1), שתרומתם לא תסולא בפז להצלחת עבודה זו". המחברים מודים לכל עוזרי המחקר ומשתתפי המחקר על תמיכתם.
Materials
| Name | Company | Catalog Number | Comments |
| AMOS2022 dataset | None | None | Datasets for network training and testing. The weblink is: https://pan.baidu.com/s/1x2ZW5FiZtVap0er55Wk4VQ?pwd=xhpb |
| ASUS mainframe | ASUS | https://www.asusparts.eu/en/asus-13020-01910200 | |
| CUDA version 11.7 | NVIDIA | https://developer.nvidia.com/cuda-11-7-0-download-archive | |
| NVIDIA GeForce RTX 3090 | NVIDIA | https://www.nvidia.com/en-in/geforce/graphics-cards/30-series/rtx-3090-3090ti/ | |
| Paddlepaddle environment | Baidu | None | Environmental preparation for network training. The weblink is: https://www.paddlepaddle.org.cn/ |
| PaddleSeg | Baidu | None | The baseline we use: https://github.com/PaddlePaddle/PaddleSeg |
References
- Liu, X., Song, L., Liu, S., Zhang, Y. A review of deep-learning-based medical image segmentation methods. Sustain. 13 (3), 1224 (2021).
- Popilock, R., Sandrasagaren, K., Harris, L., Kaser, K. A. CT artifact recognition for the nuclear technologist. J Nucl Med Technol. 36 (2), 79-81 (2008).
- Rehman, A., Khan, F. G. A deep learning based review on abdominal images. Multimed Tools Appl. 80, 30321-30352 (2021).
- Schenck, J. F. The role of magnetic susceptibility in magnetic resonance imaging: Mri magnetic compatibility of the first and second kinds. Med Phys. 23 (6), 815-850 (1996).
- Palmisano, A., et al. Myocardial late contrast enhancement ct in troponin-positive acute chest pain syndrome. Radiol. 302 (3), 545-553 (2022).
- Chen, E. -. L., Chung, P. -. C., Chen, C. -. L., Tsai, H. -. M., Chang, C. -. I. An automatic diagnostic system for CT liver image classification. IEEE Trans Biomed Eng. 45 (6), 783-794 (1998).
- Sagel, S. S., Stanley, R. J., Levitt, R. G., Geisse, G. J. R. Computed tomography of the kidney. Radiol. 124 (2), 359-370 (1977).
- Freeman, J. L., Jafri, S., Roberts, J. L., Mezwa, D. G., Shirkhoda, A. CT of congenital and acquired abnormalities of the spleen. Radiographics. 13 (3), 597-610 (1993).
- Almeida, R. R., et al. Advances in pancreatic CT imaging. AJR Am J Roentgenol. 211 (1), 52-66 (2018).
- Eisen, G. M., et al. Guidelines for credentialing and granting privileges for endoscopic ultrasound. Gastrointest Endosc. 54 (6), 811-814 (2001).
- Sykes, J. Reflections on the current status of commercial automated segmentation systems in clinical practice. J Med Radiat Sci. 61 (3), 131-134 (2014).
- He, K., Zhang, X., Ren, S., Sun, J. Deep residual learning for image recognition. 2016 IEEE Conference on Computer Vision and Pattern Recognition (CVPR). , 770-778 (2016).
- Dosovitskiy, A., et al. An image is worth 16x16 words: Transformers for image recognition at scale. arXiv. arXiv. , 11929 (2020).
- Ramachandran, P., et al. Stand-alone self-attention in vision models. Adv Neural Inf Process Syst. 32, (2019).
- Vaswani, A., et al. Attention is all you need. Adv Neural Inf Process Syst. 30, (2017).
- Chen, J., et al. TransUNET: Transformers make strong encoders for medical image segmentation. arXiv. , 04396 (2021).
- Liu, Z., et al. Swin transformer: Hierarchical vision transformer using shifted windows. Proc IEEE Int Conf Comput Vis. , 10012-10022 (2021).
- Cao, H., et al. Swin-UNET: Unet-like pure transformer for medical image segmentation. Comput Vis ECCV. , 205-218 (2022).
- Zhou, H. -. Y., et al. nnformer: Interleaved transformer for volumetric segmentation. arXiv. arXiv. , 03201 (2021).
- Sunkara, R., Luo, T. No more strided convolutions or pooling: A new CNN building block for low-resolution images and small objects. Mach Learn Knowl Discov Databases. , 443-459 (2023).
- Çiçek, &. #. 2. 1. 4. ;., Abdulkadir, A., Lienkamp, S. S., Brox, T., Ronneberger, O. 3D U-Net: learning dense volumetric segmentation from sparse annotation. Med Comput Vis Bayesian Graph Models Biomed Imaging. 2016, 424-432 (2016).
- Kim, H., et al. Abdominal multi-organ auto-segmentation using 3D-patch-based deep convolutional neural network. Scientific reports. 10 (1), 6204 (2020).
- Isensee, F., et al. nnu-net: Self-adapting framework for u-net-based medical image segmentation. arXiv. , 10486 (2018).
- Ji, Y., et al. A large-scale abdominal multi-organ benchmark for versatile medical image segmentation. Adv Neural Inf Process Syst. 35, 36722-36732 (2022).
- Liu, Y., et al. Paddleseg: A high-efficient development toolkit for image segmentation. arXiv. , 06175 (2021).
- Hatamizadeh, A., et al. UNETR: Transformers for 3D medical image segmentation. Proc IEEE Int Conf Comput Vis. , 574-584 (2022).
- Zhou, H., Zeng, D., Bian, Z., Ma, J. A semi-supervised network-based tissue-aware contrast enhancement method for ct images. Nan Fang Yi Ke Da Xue Xue Bao. 43 (6), 985-993 (2023).
- Ma, J., et al. Abdomenct-1k: Is abdominal organ segmentation a solved problem. IEEE Trans Pattern Anal Mach Intell. 44 (10), 6695-6714 (2021).
- Galván, E., Mooney, P. Neuroevolution in deep neural networks: Current trends and future challenges. IEEE Trans Artif Intell. 2 (6), 476-493 (2021).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved