JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
Swin-PSAxialNet: 효율적인 다중 장기 분할 기법
요약
현재 프로토콜은 이전 분할 방법에 비해 뛰어난 정확도를 달성한 Swin-PSAxialNet이라는 효율적인 다기관 분할 방법을 설명합니다. 이 절차의 주요 단계에는 데이터 세트 수집, 환경 구성, 데이터 전처리, 모델 학습 및 비교, 절제 실험이 포함됩니다.
초록
복부 다기관 분할은 의료 영상 분석 분야에서 가장 중요한 주제 중 하나이며, 질병 진단 및 치료 계획과 같은 임상 워크플로우를 지원하는 데 중요한 역할을 합니다. 본 연구에서는 nnU-Net 아키텍처를 기반으로 한 Swin-PSAxialNet이라는 효율적인 다기관 분할 방법을 제안한다. CT 영상에서 11개의 복부 장기를 정밀하게 분할하기 위해 특별히 설계되었습니다. 제안된 네트워크는 nnU-Net에 비해 다음과 같은 개선이 이루어졌습니다. 먼저 SPD(Space-to-Depth) 모듈과 PSAA(Parameter-Shared Axial Attention) 특징 추출 블록이 도입되어 3D 이미지 특징 추출 기능이 향상되었습니다. 둘째, 다중 스케일 이미지 융합 접근 방식을 사용하여 자세한 정보와 공간 기능을 캡처하여 미묘한 기능과 가장자리 기능을 추출하는 기능을 개선했습니다. 마지막으로, 모델의 계산 비용과 학습 속도를 줄이기 위해 매개 변수 공유 방법이 도입되었습니다. 제안된 네트워크는 11개의 장기를 포함하는 분할 작업에 대해 평균 0.93342의 주사위 계수를 달성합니다. 실험 결과는 Swin-PSAxialNet이 이전의 주류 세분화 방법에 비해 현저하게 우수하다는 것을 나타냅니다. 이 방법은 주요 복부 장기를 분할하는 데 뛰어난 정확도와 낮은 계산 비용을 보여줍니다.
서문
질병의 진단, 치료 계획의 수립, 치료 결과의 추적을 포함한 현대의 임상적 개입은 의료 영상의 정확한 세분화에 의존한다1. 그러나 복부 장기 간의 복잡한 구조적 관계2로 인해 여러 복부 장기를 정확하게 세분화하는 것은 어려운 작업입니다3. 지난 수십 년 동안 의료 영상 및 컴퓨터 비전의 번창하는 발전은 복부 다기관 분할 분야에서 새로운 기회와 도전을 동시에 제시했습니다. 첨단 자기공명영상(MRI)4 및 컴퓨터 단층촬영(CT) 기술5을 통해 고해상도 복부 이미지를 얻을 수 있습니다. CT 영상에서 여러 장기를 정밀하게 세분화하는 것은 간, 신장, 비장, 췌장 등과 같은 중요한 장기의 평가 및 치료에 상당한 임상적 가치를 지닙니다.6,7,8,9,10 그러나 이러한 해부학적 구조, 특히 방사선 전문의나 방사선 종양 전문의의 개입이 필요한 구조를 수동으로 주석하는 것은 시간이 많이 걸리고 주관적인 영향에 취약합니다 11. 따라서 복부 다기관 분할을 위한 자동화되고 정확한 방법을 시급히 개발할 필요가 있습니다.
이미지 분할에 대한 이전 연구는 주로 CNN(Convolutional Neural Network)에 의존했는데, CNN은 레이어를 쌓고 ResNet12를 도입하여 분할 효율성을 개선했습니다. 2020년, Google 연구팀은 VIT(Vision Transformer) 모델13을 도입하여 다양한 시각적 작업14을 위해 Transformer 아키텍처를 기존 시각적 영역에 통합하는 선구적인 사례로 기록했습니다. 컨볼루셔널 연산은 로컬 피처 정보만 고려할 수 있지만, 트랜스포머의 어텐션 메커니즘을 사용하면 글로벌 피처 정보를 포괄적으로 고려할 수 있습니다.
기존 컨볼루션 네트워크15에 비해 트랜스포머 기반 아키텍처의 우수성을 고려하여 수많은 연구팀이 트랜스포머와 컨볼루션 네트워크 16,17,18,19의 강점 간의 시너지 효과를 최적화하기 위해 광범위한 연구를 수행했습니다. Chen 등은 트랜스포머를 활용하여 이미지에서 전역 특징을 추출하는 의료 이미지 분할 작업16을 위한 TransUNet을 도입했습니다. 네트워크 교육의 높은 비용과 기능 추출 계층 구조의 개념을 활용하지 못하기 때문에 Transformer의 장점은 완전히 실현되지 않았습니다.
이러한 문제를 해결하기 위해 많은 연구자들이 트랜스포머를 세그멘테이션 네트워크 훈련을 위한 백본으로 통합하는 실험을 시작했습니다. Liu et al.17 은 계층화된 특징 추출을 위해 계층적 구성 방법을 사용하는 Swin Transformer를 도입했습니다. W-MSA(Windows Multi-Head Self-Attention)의 개념이 제안되어 특히 더 큰 얕은 수준의 기능 맵이 있는 경우 계산 비용을 크게 줄였습니다. 이 접근 방식은 계산 요구 사항을 줄였지만 서로 다른 창 간의 정보 전송도 격리했습니다. 이 문제를 해결하기 위해 저자는 SW-MSA(Shifted Windows Multi-Head Self-Attention)의 개념을 추가로 도입하여 인접한 창 간에 정보를 전파할 수 있도록 했습니다. 이 방법론을 기반으로 Cao 등은 Swin-UNet18을 공식화하여 U-Net의 2D 컨볼루션을 Swin 모듈로 대체하고 W-MSA 및 SW-MSA를 인코딩 및 디코딩 프로세스에 통합하여 훌륭한 세분화 결과를 달성했습니다.
반대로, Zhou 등은 고해상도 이미지를 처리할 때 conv 연산의 이점을 무시할 수 없다는 점을 강조했습니다19. 그들이 제안한 nnFormer는 로컬 3차원 이미지 블록을 기반으로 하는 셀프 어텐션 계산 방법을 사용하여 십자형 구조를 특징으로 하는 트랜스포머 모델을 구성합니다. 로컬 3차원 블록을 기반으로 한 어텐션을 활용하면 네트워크의 학습 부하가 크게 줄었습니다.
위 연구의 문제점을 감안하여 Swin-PSAxialNet이라고 하는 3D 의료 이미지 분할을 위한 효율적인 하이브리드 계층 구조를 제안합니다. 이 방법은 다운샘플링 블록, SPD(Space-to-depth)20 블록을 통합하여 글로벌 정보(21)를 추출할 수 있습니다. 또한 PSAA(Parameter Shared Axial Attention) 모듈을 추가하여 학습 매개변수 수를 2차에서 선형으로 줄이고 네트워크 훈련의 정확도와 훈련 모델의 복잡성에 좋은 영향을 미칠 것입니다22.
Swin-PSAxialNet 네트워크
네트워크의 전체 아키텍처는 인코더와 디코더 구조로 구성된 nnU-Net23의 U자형 구조를 채택합니다. 이러한 구조는 그림 1에서 볼 수 있듯이 국소 특징 추출과 큰 크기 및 작은 크기의 이미지에서 특징을 연결하는 데 관여합니다.
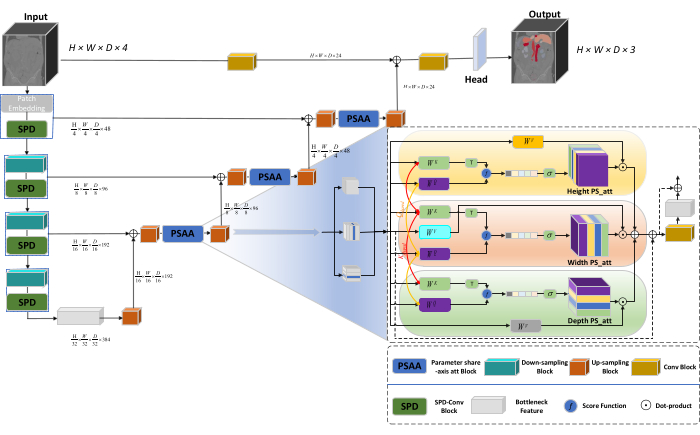
그림 1: 네트워크 아키텍처의 Swin-PSAxialNet 개략도. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
인코더 구조에서 전통적인 Conv 블록은 SPD 블록(20 )과 결합되어 다운샘플링 볼륨을 형성합니다. 엔코더의 첫 번째 계층은 3D 데이터를 3D 패치 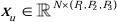 로 분할하는 모듈인 패치 임베딩을 통합하며, (P1, P2, P3)은 이 컨텍스트에서 겹치지 않는 패치를 나타내며
로 분할하는 모듈인 패치 임베딩을 통합하며, (P1, P2, P3)은 이 컨텍스트에서 겹치지 않는 패치를 나타내며 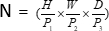 3D 패치의 시퀀스 길이를 의미합니다. 임베딩 레이어에 이어 다음 단계에는 컨볼루션 블록과 SPD 블록을 모두 포함하는 비중첩 컨볼루션 다운샘플링 장치가 포함됩니다. 이 설정에서 컨볼루션 블록은 보폭이 1로 설정되고 SPD 블록은 이미지 스케일링에 사용되어 해상도가 4배 감소하고 채널이 2배 증가합니다.
3D 패치의 시퀀스 길이를 의미합니다. 임베딩 레이어에 이어 다음 단계에는 컨볼루션 블록과 SPD 블록을 모두 포함하는 비중첩 컨볼루션 다운샘플링 장치가 포함됩니다. 이 설정에서 컨볼루션 블록은 보폭이 1로 설정되고 SPD 블록은 이미지 스케일링에 사용되어 해상도가 4배 감소하고 채널이 2배 증가합니다.
디코더 구조에서 병목 현상 기능 레이어 이후의 각 업샘플링 블록은 업샘플링 블록과 PSAA 블록의 조합으로 구성됩니다. 기능 맵의 해상도는 2배 증가하고, 채널 수는 각 디코더 단계 쌍 사이에서 절반으로 줄어듭니다. 공간 정보를 복원하고 특징 표현을 향상시키기 위해 업샘플링 블록 간에 큰 이미지와 작은 크기 이미지 간의 특징 융합이 수행됩니다. 최종적으로 업샘플링 결과는 헤드 레이어에 공급되어 출력 크기가 (H × W × D × C, C = 3)인 원본 이미지 크기를 복원합니다.
SPD 블록 아키텍처
기존 방법에서 다운샘플링 섹션은 단계 크기가 2인 단일 보폭을 사용합니다. 여기에는 이미지의 로컬 위치에서 컨볼루션 풀링(convolutional pooling)이 포함되며, 수용 영역을 제한하고, 모델을 작은 이미지 패치에서 특징을 추출하는 것으로 제한합니다. 이 방법은 원본 이미지를 3차원으로 미세하게 나누는 SPD 블록을 사용합니다. 원본 3D 이미지는 x, y 및 z축을 따라 균등하게 분할되어 4개의 하위 볼륨 바디가 생성됩니다. (그림 2) 이어서, 4개의 볼륨은 "cat" 연산을 통해 연결되고, 결과 이미지는 1 × 1 × 1 컨볼루션을 거쳐 다운 샘플링된 이미지(20)를 얻는다.
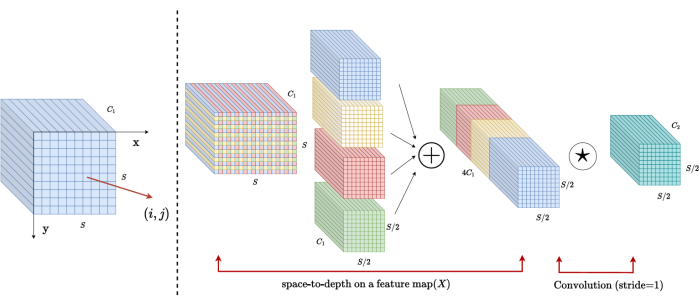
그림 2: SPD 블록 다이어그램. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
PSAA 블록 아키텍처
전통적인 CNN 네트워크와 달리 제안된 PSAA 블록은 글로벌 정보 집중을 수행하는 데 더 효과적이며 네트워크 학습 및 교육에 더 효율적입니다. 이를 통해 보다 풍부한 이미지와 공간 기능을 캡처할 수 있습니다. PSAA 블록에는 높이, 너비, 깊이의 3차원을 공유하는 매개변수를 기반으로 하는 축 주의 학습이 포함되어 있습니다. 이미지 내의 각 픽셀에 대해 어텐션 러닝을 수행하는 종래의 어텐션 메카니즘과 비교해, 이 방법은 3차원 각각에 대해 독립적으로 어텐션 러닝을 수행하여, 셀프 어텐션의 복잡성을 2차에서 선형으로 줄인다. 또한, 학습 가능한 키-쿼리 매개변수 공유 메커니즘이 사용되어 네트워크가 3차원에 걸쳐 병렬로 어텐션 메커니즘 작업을 수행할 수 있도록 하여 더 빠르고 우수하며 효과적인 기능 표현을 가능하게 합니다.
프로토콜
본 의정서는 난퉁대학교 윤리위원회의 승인을 받았다. 여기에는 인공 지능 기술을 활용하여 인간 의료 이미지, 사지 움직임 및 혈관 이미징을 포함하여 획득한 비침습적 또는 최소 침습적 다중 모드 데이터에 대한 지능적인 평가 및 연구가 포함됩니다. 그림 3 은 다기관 분할의 전체 순서도를 보여줍니다. 필요한 모든 웹 링크는 자료 표에 나와 있습니다.
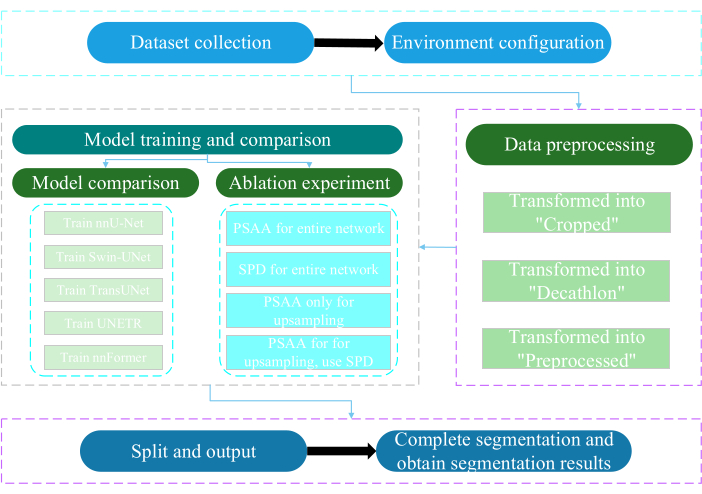
그림 3: 다기관 세분화의 전체 순서도.이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
1. 데이터셋 수집
- AMOS2022 데이터셋24 를 다운로드합니다( 자료표 참조).
- 160개의 이미지를 학습 데이터 세트로 선택하고 40개의 이미지를 테스트 데이터 세트로 선택합니다.
- mkdir ~/PaddleSeg/contrib/MedicalSeg/data/raw_data를 터미널로 실행합니다.
- raw_data의 길을 엽니다.
- 데이터 세트를 raw_data 디렉터리에 배치하고 "imagesTr" 및 "labelsTr" 디렉터리의 파일 이름과 올바른 일치를 확인합니다.
- dataset.json 파일을 만듭니다.
- numTraining, numTest 및 레이블 이름과 같은 데이터셋 정보를 "dataset.json" 파일에 추가합니다.
참고: 이 연구의 데이터 세트는 교육 및 테스트 목적으로 AMOS2022 공식 500 CT 데이터 세트에서 선택한 200개의 고품질 CT 데이터 세트로 구성됩니다. 이 데이터 세트는 비장, 오른쪽 신장, 왼쪽 신장, 담낭, 식도, 간, 위, 대동맥, 하대정맥, 췌장, 방광을 포함한 11개의 기관을 포함합니다.
2. 환경 구성
- Paddlepaddle 환경( Table of Materials) 및 해당 CUDA 버전25를 설치합니다.
참고: Paddlepaddle 버전 2.5.0 및 CUDA 버전 11.7을 사용하는 것이 좋습니다. - Git은 PaddleSeg 저장소를 복제합니다25.
- cd ~/PaddleSeg/contrib/MedicalSeg/를 터미널로 실행합니다.
- pip install -r requirements.txt를 실행합니다.
- pip install medpy를 실행합니다.
3. 데이터 전처리
- data_root, batch_size, model, train_dataset, val_dataset, optimizer, lr_scheduler 및 loss와 같은 매개변수를 포함하여 "yml" 파일을 수정합니다.
- "-config, --log_iters, --precision, --nnunet, --save_dir, --save_interval 등을 포함하여 실행에 필요한 매개 변수를 지정합니다.
참고: 원래 "raw_data"은 "크롭", "데카트론" 및 "전처리"의 세 가지 유형의 데이터 파일로 변환되어 모델 성능과 교육 효율성을 향상시키기 위해 다양한 교육 및 평가 요구 사항을 충족합니다.
4. 모델 학습 및 비교
참고: 이미지 분할 분야에서 널리 사용되는 기준선인 nnU-Net23 은 연구에서 기준선 모델 역할을 합니다. 구체적인 모델 비교 프로세스는 다음과 같습니다.
- nnU-Net 훈련시키기
- 2 및 80,000회 반복의 배치 크기로 구성하고, 20회 반복마다 기록하고, fp16 정밀도를 활용합니다.
- save_dir 작업 디렉토리 내의 "output" 폴더로 설정하고 저장 간격은 1,000회 반복으로 설정합니다.
- VisualDL 매개 변수 시각화를 위해 --use_vdl 매개 변수를 추가합니다.
- train.py 실행합니다.
- 4개의 yml 파일을 만들고, 폴드 1-4를 지정하고, 5겹 교차 검증을 사용하여 모델을 훈련시킵니다.
- 기차 Swin-UNet
- 이전에 설정된 실험 조건과의 일관성을 유지합니다.
- 매개 변수 --nnunet 을 제거하고 --swinunet을 사용합니다.
- train.py 파일을 실행합니다.
- 트레인은 1-4개를 개별적으로 접은 다음 앙상블 처리를 받습니다.
- TransUNet 기차
- 4.1단계에 설명된 절차를 따릅니다.
- yml 파일을 엽니다.
- type: nnunet을 type: transunet으로 수정합니다.
- 매개 변수 --nnunet 을 --transunet으로 수정합니다.
- train.py 파일을 실행합니다.
- 트레인은 1-4를 개별적으로 접고 앙상블 처리를 처리합니다.
- UNETR 훈련
- 실험 변수를 일관되게 유지합니다.
- Swin-UNet 및 TransUNet과 같이 앞에서 설명한 절차를 반복합니다(4.2단계 및 4.3단계).
- train.py 실행합니다.
- 트레인은 1-4를 개별적으로 접고 앙상블 처리를 처리합니다.
- 기차 nnFormer
- Swin-UNet 및 TransUNet과 같은 매개 변수를 수정합니다.
- 한 번 접기 train.py 실행합니다.
- 앙상블 처리를 실행합니다.
5. 절제 실험
- 전체 네트워크에 대한 PSAA 블록
- 변수를 4.1단계와 일관되게 유지합니다.
- volume_shape = (64,192,192) 및 Axial_attention = True로 설정합니다.
- 한 번 접기 train.py 실행합니다.
- 앙상블 가공을 5겹으로 처리합니다.
- 전체 네트워크에 대한 SPD 블록
- 변수를 4.1단계와 일관되게 유지합니다.
- SPD_enable = True 및 Axial_attention = False로 설정합니다.
- 한 번 접기 train.py 실행합니다.
- 앙상블 가공을 5겹으로 처리합니다.
- 업샘플링을 위한 PSAA 블록
참고: 다운샘플링에 SPD 블록을 사용하지 마십시오.- 업샘플링을 위해 PSAA 블록을 사용합니다.
- 변수를 4.1단계와 일관되게 유지합니다.
- 다운샘플링 부분에서 Axial_attention = False 를 설정하고 SPD_enable = False를 설정합니다.
- 한 번 접기 train.py 실행합니다.
- 공정 5중 앙상블 가공.
- 업샘플링을 위한 PSAA 블록
참고: 다운샘플링을 위해 SPD 블록을 사용하십시오.- SPD 블록은 다운샘플링에 사용하고 PSAA 블록은 업샘플링에 사용합니다.
- 변수를 4.1단계와 일관되게 유지합니다.
- 다운샘플링 부분에서 Axial_attention = False 를 설정하고 SPD_enable = True를 설정합니다.
- 한 번 접기 train.py 실행합니다.
- 공정 5중 앙상블 가공.
- 우수한 결과를 위해 반복 횟수를 2,00,000으로 설정합니다.
결과
이 프로토콜은 모델을 평가하기 위해 DSC(Dice Similarity Score)와 HD95(95% Hausdorff Distance)의 두 가지 메트릭을 사용합니다. DSC는 복셀 분할 예측과 실측 자료 간의 중복을 측정하는 반면, 95% HD는 복셀 분할 예측 경계와 실측 자료 간의 중복을 평가하여 이상값의 5%를 필터링합니다. DSC26 의 정의는 다음과 같습니다.
토론
복부 장기의 세분화는 복잡한 작업입니다. 뇌나 심장과 같은 인체의 다른 내부 구조와 비교했을 때, CT 영상에서 대비가 낮고 형태 변화가 크기 때문에 복부 장기를 분할하는 것이 더 어려워 보인다27,28. 이 어려운 문제를 해결하기 위해 Swin-PSAxialNet을 제안합니다.
본 연구는 데이터 수집 단계에서 AMOS2022 공?...
공개
저자는 이해 상충이 없음을 선언합니다.
감사의 말
이 연구는 장쑤성 '333' 엔지니어링 프로젝트([2022]21-003), 우시 보건위원회 일반 프로그램(M202205), 우시 과학기술개발기금(Y20212002-1)의 지원을 받았으며, 이들은 이 작업의 성공에 큰 기여를 했습니다." 저자는 모든 연구 보조원과 연구 참가자의 지원에 감사드립니다.
자료
| Name | Company | Catalog Number | Comments |
| AMOS2022 dataset | None | None | Datasets for network training and testing. The weblink is: https://pan.baidu.com/s/1x2ZW5FiZtVap0er55Wk4VQ?pwd=xhpb |
| ASUS mainframe | ASUS | https://www.asusparts.eu/en/asus-13020-01910200 | |
| CUDA version 11.7 | NVIDIA | https://developer.nvidia.com/cuda-11-7-0-download-archive | |
| NVIDIA GeForce RTX 3090 | NVIDIA | https://www.nvidia.com/en-in/geforce/graphics-cards/30-series/rtx-3090-3090ti/ | |
| Paddlepaddle environment | Baidu | None | Environmental preparation for network training. The weblink is: https://www.paddlepaddle.org.cn/ |
| PaddleSeg | Baidu | None | The baseline we use: https://github.com/PaddlePaddle/PaddleSeg |
참고문헌
- Liu, X., Song, L., Liu, S., Zhang, Y. A review of deep-learning-based medical image segmentation methods. Sustain. 13 (3), 1224 (2021).
- Popilock, R., Sandrasagaren, K., Harris, L., Kaser, K. A. CT artifact recognition for the nuclear technologist. J Nucl Med Technol. 36 (2), 79-81 (2008).
- Rehman, A., Khan, F. G. A deep learning based review on abdominal images. Multimed Tools Appl. 80, 30321-30352 (2021).
- Schenck, J. F. The role of magnetic susceptibility in magnetic resonance imaging: Mri magnetic compatibility of the first and second kinds. Med Phys. 23 (6), 815-850 (1996).
- Palmisano, A., et al. Myocardial late contrast enhancement ct in troponin-positive acute chest pain syndrome. Radiol. 302 (3), 545-553 (2022).
- Chen, E. -. L., Chung, P. -. C., Chen, C. -. L., Tsai, H. -. M., Chang, C. -. I. An automatic diagnostic system for CT liver image classification. IEEE Trans Biomed Eng. 45 (6), 783-794 (1998).
- Sagel, S. S., Stanley, R. J., Levitt, R. G., Geisse, G. J. R. Computed tomography of the kidney. Radiol. 124 (2), 359-370 (1977).
- Freeman, J. L., Jafri, S., Roberts, J. L., Mezwa, D. G., Shirkhoda, A. CT of congenital and acquired abnormalities of the spleen. Radiographics. 13 (3), 597-610 (1993).
- Almeida, R. R., et al. Advances in pancreatic CT imaging. AJR Am J Roentgenol. 211 (1), 52-66 (2018).
- Eisen, G. M., et al. Guidelines for credentialing and granting privileges for endoscopic ultrasound. Gastrointest Endosc. 54 (6), 811-814 (2001).
- Sykes, J. Reflections on the current status of commercial automated segmentation systems in clinical practice. J Med Radiat Sci. 61 (3), 131-134 (2014).
- He, K., Zhang, X., Ren, S., Sun, J. Deep residual learning for image recognition. 2016 IEEE Conference on Computer Vision and Pattern Recognition (CVPR). , 770-778 (2016).
- Dosovitskiy, A., et al. An image is worth 16x16 words: Transformers for image recognition at scale. arXiv. arXiv. , 11929 (2020).
- Ramachandran, P., et al. Stand-alone self-attention in vision models. Adv Neural Inf Process Syst. 32, (2019).
- Vaswani, A., et al. Attention is all you need. Adv Neural Inf Process Syst. 30, (2017).
- Chen, J., et al. TransUNET: Transformers make strong encoders for medical image segmentation. arXiv. , 04396 (2021).
- Liu, Z., et al. Swin transformer: Hierarchical vision transformer using shifted windows. Proc IEEE Int Conf Comput Vis. , 10012-10022 (2021).
- Cao, H., et al. Swin-UNET: Unet-like pure transformer for medical image segmentation. Comput Vis ECCV. , 205-218 (2022).
- Zhou, H. -. Y., et al. nnformer: Interleaved transformer for volumetric segmentation. arXiv. arXiv. , 03201 (2021).
- Sunkara, R., Luo, T. No more strided convolutions or pooling: A new CNN building block for low-resolution images and small objects. Mach Learn Knowl Discov Databases. , 443-459 (2023).
- Çiçek, &. #. 2. 1. 4. ;., Abdulkadir, A., Lienkamp, S. S., Brox, T., Ronneberger, O. 3D U-Net: learning dense volumetric segmentation from sparse annotation. Med Comput Vis Bayesian Graph Models Biomed Imaging. 2016, 424-432 (2016).
- Kim, H., et al. Abdominal multi-organ auto-segmentation using 3D-patch-based deep convolutional neural network. Scientific reports. 10 (1), 6204 (2020).
- Isensee, F., et al. nnu-net: Self-adapting framework for u-net-based medical image segmentation. arXiv. , 10486 (2018).
- Ji, Y., et al. A large-scale abdominal multi-organ benchmark for versatile medical image segmentation. Adv Neural Inf Process Syst. 35, 36722-36732 (2022).
- Liu, Y., et al. Paddleseg: A high-efficient development toolkit for image segmentation. arXiv. , 06175 (2021).
- Hatamizadeh, A., et al. UNETR: Transformers for 3D medical image segmentation. Proc IEEE Int Conf Comput Vis. , 574-584 (2022).
- Zhou, H., Zeng, D., Bian, Z., Ma, J. A semi-supervised network-based tissue-aware contrast enhancement method for ct images. Nan Fang Yi Ke Da Xue Xue Bao. 43 (6), 985-993 (2023).
- Ma, J., et al. Abdomenct-1k: Is abdominal organ segmentation a solved problem. IEEE Trans Pattern Anal Mach Intell. 44 (10), 6695-6714 (2021).
- Galván, E., Mooney, P. Neuroevolution in deep neural networks: Current trends and future challenges. IEEE Trans Artif Intell. 2 (6), 476-493 (2021).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유