Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Swin-PSAxialNet: эффективный метод сегментации нескольких органов
В этой статье
Резюме
Настоящий протокол описывает эффективный метод многоорганной сегментации под названием Swin-PSAxialNet, который достиг превосходной точности по сравнению с предыдущими методами сегментации. Ключевые этапы этой процедуры включают сбор наборов данных, настройку среды, предварительную обработку данных, обучение и сравнение моделей, а также эксперименты по абляции.
Аннотация
Абдоминальная полиорганная сегментация является одной из наиболее важных тем в области анализа медицинских изображений и играет важную роль в поддержке клинических рабочих процессов, таких как диагностика заболеваний и планирование лечения. В данном исследовании предложен эффективный метод многоорганной сегментации под названием Swin-PSAxialNet на основе архитектуры nnU-Net. Он был разработан специально для точной сегментации 11 органов брюшной полости на КТ-изображениях. В предлагаемую сеть были внесены следующие улучшения по сравнению с nnU-Net. Во-первых, были представлены модули Space-to-depth (SPD) и блоки извлечения признаков с разделяемым по параметрам осевого внимания (PSAA), что расширило возможности извлечения признаков 3D-изображений. Во-вторых, для получения подробной информации и пространственных особенностей был использован подход слияния многомасштабных изображений, что улучшило возможности извлечения тонких особенностей и краевых особенностей. Наконец, был введен метод совместного использования параметров, чтобы снизить вычислительные затраты модели и скорость обучения. Предложенная сеть достигает среднего коэффициента Dice 0,93342 для задачи сегментации с участием 11 органов. Экспериментальные результаты указывают на заметное превосходство Swin-PSAxialNet над предыдущими методами сегментации. Метод показывает отличную точность и низкие вычислительные затраты при сегментации основных органов брюшной полости.
Введение
Современное клиническое вмешательство, включая диагностику заболеваний, составление планов лечения и отслеживание результатов лечения, опирается на точную сегментацию медицинских изображений1. Тем не менее, сложные структурные взаимоотношения между органами брюшной полости2делают задачу достижения точной сегментации нескольких органов брюшной полостисложной задачей 3. За последние несколько десятилетий бурное развитие медицинской визуализации и компьютерного зрения открыло как новые возможности, так и вызовы в области сегментации полиорганов брюшной полости. Передовые технологии магнитно-резонансной томографии (МРТ)4 и компьютерной томографии (КТ)5 позволяют получать изображения брюшной полости с высоким разрешением. Точная сегментация нескольких органов по изображениям КТ имеет важное клиническое значение для оценки и лечения жизненно важных органов, таких как печень, почки, селезенка, поджелудочная железа и т. д.6,7,8,9,10 Однако ручное аннотирование этих анатомических структур, особенно тех, которые требуют вмешательства радиологов или онкологов-радиологов, занимает много времени и подвержено субъективному влиянию 11. В связи с этим существует острая необходимость в разработке автоматизированных и точных методов полиорганной сегментации брюшной полости.
Предыдущие исследования сегментации изображений в основном опирались на сверточные нейронные сети (СНС), которые повышают эффективность сегментации за счет наложения слоев и внедрения ResNet12. В 2020 году исследовательская группа Google представила модель Vision Transformer (VIT)13, которая стала первым примером внедрения архитектуры Transformer в традиционную визуальную область для ряда визуальных задач14. В то время как сверточные операции могут рассматривать только информацию о локальных признаках, механизм внимания в «Трансформерах» позволяет всесторонне рассматривать информацию о глобальных признаках.
Учитывая превосходство архитектур на основе трансформаторов над традиционными сверточными сетями15, многочисленные исследовательские группы провели обширные исследования по оптимизации синергии между сильными сторонами трансформаторов и сверточных сетей 16,17,18,19. Чен и др. представили TransUNet для задачсегментации медицинских изображений 16, которые используют трансформеры для извлечения глобальных характеристик из изображений. Из-за высокой стоимости обучения сети и неспособности использовать концепцию иерархии извлечения признаков, преимущества Transformer не были реализованы в полной мере.
Чтобы решить эти проблемы, многие исследователи начали экспериментировать с использованием трансформеров в качестве основы для обучающих сегментационных сетей. Лю и др.17 представили трансформатор Свина, который использовал метод иерархического построения для многоуровневого извлечения признаков. Была предложена концепция Windows Multi-Head Self-Attention (W-MSA), значительно снижающая вычислительные затраты, особенно при наличии более крупных карт функций мелкого уровня. Хотя такой подход снизил вычислительные требования, он также изолировал передачу информации между различными окнами. Чтобы решить эту проблему, авторы ввели концепцию Shifted Windows Multi-Head Self-Attention (SW-MSA), позволяющую распространять информацию между соседними окнами. Основываясь на этой методологии, Цао и др. сформулировали Swin-UNet18, заменив двухмерные свертки в U-Net на модули Swin и включив W-MSA и SW-MSA в процессы кодирования и декодирования, достигнув похвальных результатов сегментации.
И наоборот, Zhou et al. подчеркнули, что преимущество операции conv нельзя игнорировать при обработке изображений с высоким разрешением19. Предложенный ими nnFormer использует метод вычисления собственного внимания, основанный на локальных трехмерных блоках изображений, составляющих модель Трансформера, характеризующуюся крестообразной структурой. Использование внимания на основе локальных трехмерных блоков значительно снижало учебную нагрузку на сеть.
Учитывая проблемы, связанные с вышеупомянутым исследованием, предлагается эффективная гибридная иерархическая структура для сегментации 3D-медицинских изображений, получившая название Swin-PSAxialNet. Этот метод включает в себя блок понижающей дискретизации, блок Space-to-depth (SPD)20 , способный извлекать глобальную информацию21. Кроме того, он добавляет модуль параметра «Разделяемое осевое внимание» (PSAA), который сокращает количество параметров обучения с квадратичного до линейного и окажет хорошее влияние на точность обучения сети и сложность обучающих моделей22.
Сеть Swin-PSAxialNet
Общая архитектура сети использует U-образную структуру nnU-Net23, состоящую из структур энкодера и декодера. Эти структуры участвуют в локальном извлечении признаков и конкатенации признаков из больших и мелких изображений, как показано на рисунке 1.
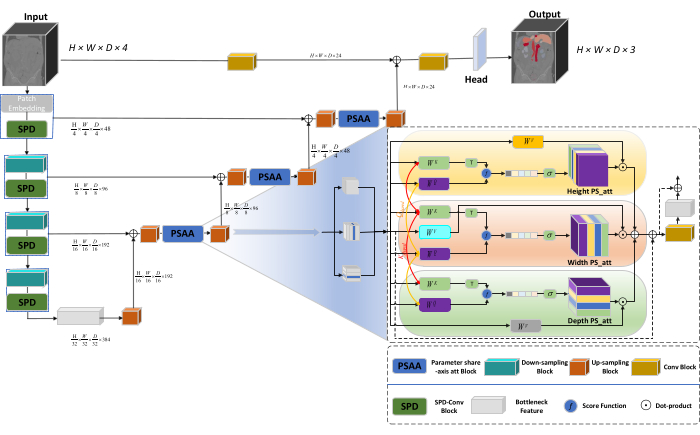
Рисунок 1: Принципиальная схема сетевой архитектуры Swin-PSAxialNet. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
В структуре энкодера традиционный блок Conv объединен с блоком SPD20 для формирования объема понижающей дискретизации. Первый слой кодировщика включает в себя Patch Embedding, модуль, который разделяет 3D-данные на 3D-патчи 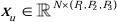 , (P1, P2, P3), представляет неперекрывающиеся патчи в этом контексте,
, (P1, P2, P3), представляет неперекрывающиеся патчи в этом контексте, 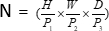 обозначает длину последовательности 3D-патчей. Следующий шаг включает в себя неперекрывающуюся сверточную единицу понижающей дискретизации, содержащую как сверточный блок, так и блок SPD. В этой конфигурации сверточный блок имеет шаг, равный 1, а блок SPD используется для масштабирования изображения, что приводит к четырехкратному снижению разрешения и двукратному увеличению каналов.
обозначает длину последовательности 3D-патчей. Следующий шаг включает в себя неперекрывающуюся сверточную единицу понижающей дискретизации, содержащую как сверточный блок, так и блок SPD. В этой конфигурации сверточный блок имеет шаг, равный 1, а блок SPD используется для масштабирования изображения, что приводит к четырехкратному снижению разрешения и двукратному увеличению каналов.
В структуре декодера каждый блок повышающей дискретизации после уровня Bottleneck Feature состоит из комбинации блока повышающей дискретизации и блока PSAA. Разрешение карты функций увеличивается в два раза, а количество каналов уменьшается вдвое между каждой парой каскадов декодера. Для восстановления пространственной информации и улучшения представления объектов выполняется слияние объектов между большими и мелкомасштабными изображениями между блоками повышающей дискретизации. В конечном счете, результаты повышающей дискретизации подаются в слой Head для восстановления исходного размера изображения с выходным размером (H × W × D × C, C = 3).
Блочная архитектура SPD
В традиционных методах секция понижающей дискретизации выполняется одним шагом с шагом 2. Это включает в себя сверточный пул в локальных положениях изображения, ограничение рецептивного поля и ограничение модели извлечением признаков из небольших участков изображения. В этом методе используется блок SPD, который тонко делит исходное изображение на три измерения. Исходное 3D-изображение равномерно сегментировано по осям x, y и z, в результате чего образуются четыре подобъемных тела. (Рисунок 2) Впоследствии четыре тома объединяются с помощью операции «cat», и полученное изображение подвергается свертке 1 × 1 × 1 для получения изображения20 с пониженной дискретизацией.
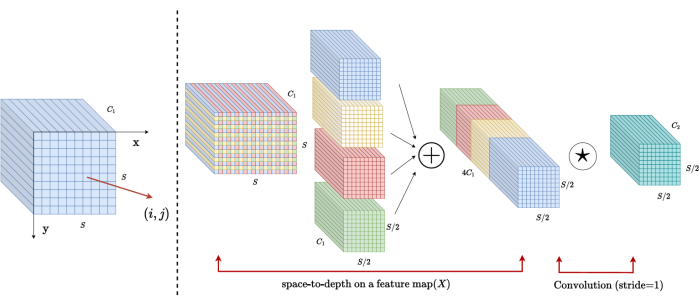
Рисунок 2: Блок-схема SPD. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Блочная архитектура PSAA
В отличие от традиционных сетей CNN, предлагаемый блок PSAA более эффективен в проведении глобального информационного фокуса и более эффективен в обучении и обучении сети. Это позволяет получать более насыщенные изображения и пространственные особенности. Блок PSAA включает в себя осевое обучение внимания на основе совместного использования параметров в трех измерениях: высота, ширина и глубина. По сравнению с обычным механизмом внимания, который выполняет обучение внимания для каждого пикселя на изображении, этот метод независимо проводит обучение внимания для каждого из трех измерений, снижая сложность собственного внимания с квадратичной до линейной. Кроме того, используется обучаемый механизм совместного использования параметров ключей-запросов, позволяющий сети выполнять операции механизма внимания параллельно в трех измерениях, что приводит к более быстрому, превосходному и эффективному представлению признаков.
протокол
Настоящий протокол был одобрен Этическим комитетом Наньтунского университета. Он включает в себя интеллектуальную оценку и исследование полученных неинвазивных или минимально инвазивных мультимодальных данных, включая медицинские изображения человека, движения конечностей и визуализацию сосудов, с использованием технологии искусственного интеллекта. На рисунке 3 показана общая блок-схема многоорганной сегментации. Все необходимые веб-ссылки приведены в Таблице материалов.
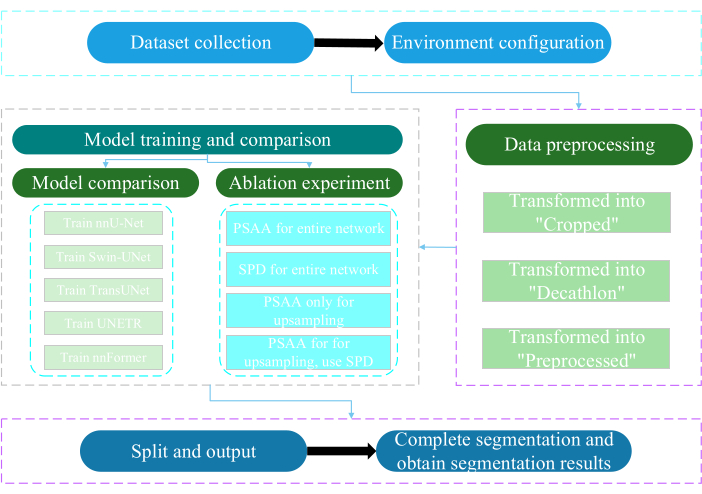
Рисунок 3: Общая блок-схема многоорганной сегментации. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
1. Сбор наборов данных
- Загрузите набор данных AMOS202224 (см. Таблицу материалов).
- Выберите 160 изображений в качестве обучающего набора данных и 40 изображений в качестве тестового набора данных.
- Terminally execute mkdir ~/PaddleSeg/contrib/MedicalSeg/data/raw_data.
- Откройте путь raw_data.
- Поместите набор данных в директорию raw_data и убедитесь в правильном соответствии с именами файлов в директориях "imagesTr" и "labelsTr".
- Создайте файл dataset.json .
- Добавьте сведения о наборе данных, такие как numTraining, numTest и имя меток , в файл dataset.json.
ПРИМЕЧАНИЕ: Набор данных для этого исследования состоит из 200 высококачественных наборов данных КТ, отобранных из AMOS2022 официального набора данных 500 КТ для целей обучения и тестирования. Набор данных охватывает 11 органов, включая селезенку, правую почку, левую почку, желчный пузырь, пищевод, печень, желудок, аорту, нижнюю полую вену, поджелудочную железу и мочевой пузырь.
2. Настройка окружения
- Установите среду Paddlepaddle (см. Таблицу материалов) и соответствующую версию CUDA25.
ПРИМЕЧАНИЕ: Рекомендуется использовать Paddlepaddle версии 2.5.0 и CUDA версии 11.7. - Клонирование репозитория PaddleSeg в Git25.
- Терминально выполните cd ~/PaddleSeg/contrib/MedicalSeg/.
- Выполните pip install -r requirements.txt.
- Выполните pip install medpy.
3. Предварительная обработка данных
- Измените файл "yml", включая такие параметры , как data_root, batch_size, модель, train_dataset, val_dataset, оптимизатор, lr_scheduler и потери.
- Укажите необходимые параметры для выполнения, в том числе "-config, --log_iters, --precision, --nnunet, --save_dir, --save_interval и т.д.
ПРИМЕЧАНИЕ: Исходный «raw_data» преобразуется в три типа файлов данных: «Обрезанные», «Десятиборные» и «Предварительно обработанные», удовлетворяющие различные требования к обучению и оценке для повышения производительности модели и эффективности обучения.
4. Обучение и сравнение моделей
ПРИМЕЧАНИЕ: Являясь широко используемым базовым уровнем в области сегментации изображений, nnU-Net23 служит базовой моделью в исследовании. Процесс сравнения конкретных моделей выглядит следующим образом.
- Поезд nnU-Net
- Конфигурация с размером пакета от 2 до 80 000 итераций, ведение журнала каждые 20 итераций и использование точности fp16.
- Установите save_dir в качестве папки «вывода» в рабочем каталоге с интервалом сохранения в 1 000 итераций.
- Добавьте параметр --use_vdl для визуализации параметров VisualDL.
- Запустите train.py.
- Создайте четыре yml-файла , укажите свертки 1-4 и обучите модель с помощью пятикратной кросс-валидации.
- Поезд Swin-UNet
- Поддерживайте согласованность с ранее установленными условиями эксперимента.
- Удалите параметр --nnunet и используйте --swinunet.
- Запустите файл train.py.
- Шлейф складывается по 1-4 по отдельности и затем подвергается ансамблевой обработке.
- Поезд TransUNet
- Следуйте процедурам, описанным в шаге 4.1.
- Откройте файл yml .
- Измените тип: nnunet на тип: transunet.
- Измените параметр --nnunet на --transunet.
- Запустите файл train.py.
- Тренируйтесь по 1-4 фальца по отдельности и обрабатывайте ансамбль обработки.
- Поезд UNETR
- Поддерживайте согласованность экспериментальных переменных.
- Повторите описанные ранее процедуры, такие как Swin-UNet и TransUNet (шаг 4.2 и шаг 4.3).
- Запустите train.py.
- Тренируйтесь по 1-4 фальца по отдельности и обрабатывайте ансамбль обработки.
- Поезд nnFormer
- Измените параметры, такие как Swin-UNet и TransUNet.
- Запустите одинарный фальцевый train.py.
- Запустите обработку ансамбля.
5. Эксперимент по абляции
- Блок PSAA для всей сети
- Обеспечьте согласованность переменных с шагом 4.1.
- Установите volume_shape = (64,192,192) и Axial_attention = true.
- Запустите одинарный фальцевый train.py.
- Обрабатывайте ансамбль в пять этапов.
- Блок SPD для всей сети
- Обеспечьте согласованность переменных с шагом 4.1.
- Установите SPD_enable = True и Axial_attention = False.
- Запустите одинарный фальцевый train.py.
- Обрабатывайте ансамбль в пять этапов.
- Блок PSAA для повышающей дискретизации
ПРИМЕЧАНИЕ: Не используйте блок SPD для понижающей дискретизации.- Используйте блок PSAA для повышения дискретизации.
- Обеспечьте согласованность переменных с шагом 4.1.
- Установите Axial_attention = False в части понижения дискретизации и SPD_enable = False.
- Запустите одинарный фальцевый train.py.
- Обработайте пятикратным ансамблем обработку.
- Блок PSAA для повышающей дискретизации
ПРИМЕЧАНИЕ: Используйте блок SPD для понижающей дискретизации.- Используйте блок SPD для понижения дискретизации и блок PSAA для повышения дискретизации.
- Обеспечьте согласованность переменных с шагом 4.1.
- Установите Axial_attention = False в части даунсемплинга и SPD_enable = True.
- Запустите одинарный фальцевый train.py.
- Обработайте пятикратным ансамблем обработку.
- Установите количество итераций равным 2 00 000 из-за превосходных результатов.
Результаты
Этот протокол использует две метрики для оценки модели: индекс сходства кубиков (DSC) и 95% расстояние Хаусдорфа (HD95). DSC измеряет перекрытие между прогнозами воксельной сегментации и наземной достоверностью, в то время как 95% HD оценивает перекрытие между границами прогно...
Обсуждение
Сегментация органов брюшной полости – сложная работа. По сравнению с другими внутренними структурами человеческого тела, такими как мозг или сердце, сегментация органов брюшной полости кажется более сложной задачей из-за низкого контраста и больших изменений формы ...
Раскрытие информации
Авторы заявляют об отсутствии конфликта интересов.
Благодарности
Это исследование было поддержано инженерным проектом «333» провинции Цзянсу ([2022]21-003), Общей программой Комиссии по здравоохранению Уси (M202205) и Фондом развития науки и технологий Уси (Y20212002-1), чей вклад был неоценим для успеха этой работы». Авторы благодарят всех научных сотрудников и участников исследования за поддержку.
Материалы
| Name | Company | Catalog Number | Comments |
| AMOS2022 dataset | None | None | Datasets for network training and testing. The weblink is: https://pan.baidu.com/s/1x2ZW5FiZtVap0er55Wk4VQ?pwd=xhpb |
| ASUS mainframe | ASUS | https://www.asusparts.eu/en/asus-13020-01910200 | |
| CUDA version 11.7 | NVIDIA | https://developer.nvidia.com/cuda-11-7-0-download-archive | |
| NVIDIA GeForce RTX 3090 | NVIDIA | https://www.nvidia.com/en-in/geforce/graphics-cards/30-series/rtx-3090-3090ti/ | |
| Paddlepaddle environment | Baidu | None | Environmental preparation for network training. The weblink is: https://www.paddlepaddle.org.cn/ |
| PaddleSeg | Baidu | None | The baseline we use: https://github.com/PaddlePaddle/PaddleSeg |
Ссылки
- Liu, X., Song, L., Liu, S., Zhang, Y. A review of deep-learning-based medical image segmentation methods. Sustain. 13 (3), 1224 (2021).
- Popilock, R., Sandrasagaren, K., Harris, L., Kaser, K. A. CT artifact recognition for the nuclear technologist. J Nucl Med Technol. 36 (2), 79-81 (2008).
- Rehman, A., Khan, F. G. A deep learning based review on abdominal images. Multimed Tools Appl. 80, 30321-30352 (2021).
- Schenck, J. F. The role of magnetic susceptibility in magnetic resonance imaging: Mri magnetic compatibility of the first and second kinds. Med Phys. 23 (6), 815-850 (1996).
- Palmisano, A., et al. Myocardial late contrast enhancement ct in troponin-positive acute chest pain syndrome. Radiol. 302 (3), 545-553 (2022).
- Chen, E. -. L., Chung, P. -. C., Chen, C. -. L., Tsai, H. -. M., Chang, C. -. I. An automatic diagnostic system for CT liver image classification. IEEE Trans Biomed Eng. 45 (6), 783-794 (1998).
- Sagel, S. S., Stanley, R. J., Levitt, R. G., Geisse, G. J. R. Computed tomography of the kidney. Radiol. 124 (2), 359-370 (1977).
- Freeman, J. L., Jafri, S., Roberts, J. L., Mezwa, D. G., Shirkhoda, A. CT of congenital and acquired abnormalities of the spleen. Radiographics. 13 (3), 597-610 (1993).
- Almeida, R. R., et al. Advances in pancreatic CT imaging. AJR Am J Roentgenol. 211 (1), 52-66 (2018).
- Eisen, G. M., et al. Guidelines for credentialing and granting privileges for endoscopic ultrasound. Gastrointest Endosc. 54 (6), 811-814 (2001).
- Sykes, J. Reflections on the current status of commercial automated segmentation systems in clinical practice. J Med Radiat Sci. 61 (3), 131-134 (2014).
- He, K., Zhang, X., Ren, S., Sun, J. Deep residual learning for image recognition. 2016 IEEE Conference on Computer Vision and Pattern Recognition (CVPR). , 770-778 (2016).
- Dosovitskiy, A., et al. An image is worth 16x16 words: Transformers for image recognition at scale. arXiv. arXiv. , 11929 (2020).
- Ramachandran, P., et al. Stand-alone self-attention in vision models. Adv Neural Inf Process Syst. 32, (2019).
- Vaswani, A., et al. Attention is all you need. Adv Neural Inf Process Syst. 30, (2017).
- Chen, J., et al. TransUNET: Transformers make strong encoders for medical image segmentation. arXiv. , 04396 (2021).
- Liu, Z., et al. Swin transformer: Hierarchical vision transformer using shifted windows. Proc IEEE Int Conf Comput Vis. , 10012-10022 (2021).
- Cao, H., et al. Swin-UNET: Unet-like pure transformer for medical image segmentation. Comput Vis ECCV. , 205-218 (2022).
- Zhou, H. -. Y., et al. nnformer: Interleaved transformer for volumetric segmentation. arXiv. arXiv. , 03201 (2021).
- Sunkara, R., Luo, T. No more strided convolutions or pooling: A new CNN building block for low-resolution images and small objects. Mach Learn Knowl Discov Databases. , 443-459 (2023).
- Çiçek, &. #. 2. 1. 4. ;., Abdulkadir, A., Lienkamp, S. S., Brox, T., Ronneberger, O. 3D U-Net: learning dense volumetric segmentation from sparse annotation. Med Comput Vis Bayesian Graph Models Biomed Imaging. 2016, 424-432 (2016).
- Kim, H., et al. Abdominal multi-organ auto-segmentation using 3D-patch-based deep convolutional neural network. Scientific reports. 10 (1), 6204 (2020).
- Isensee, F., et al. nnu-net: Self-adapting framework for u-net-based medical image segmentation. arXiv. , 10486 (2018).
- Ji, Y., et al. A large-scale abdominal multi-organ benchmark for versatile medical image segmentation. Adv Neural Inf Process Syst. 35, 36722-36732 (2022).
- Liu, Y., et al. Paddleseg: A high-efficient development toolkit for image segmentation. arXiv. , 06175 (2021).
- Hatamizadeh, A., et al. UNETR: Transformers for 3D medical image segmentation. Proc IEEE Int Conf Comput Vis. , 574-584 (2022).
- Zhou, H., Zeng, D., Bian, Z., Ma, J. A semi-supervised network-based tissue-aware contrast enhancement method for ct images. Nan Fang Yi Ke Da Xue Xue Bao. 43 (6), 985-993 (2023).
- Ma, J., et al. Abdomenct-1k: Is abdominal organ segmentation a solved problem. IEEE Trans Pattern Anal Mach Intell. 44 (10), 6695-6714 (2021).
- Galván, E., Mooney, P. Neuroevolution in deep neural networks: Current trends and future challenges. IEEE Trans Artif Intell. 2 (6), 476-493 (2021).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены