A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
استراتيجية التعلم الآلي القائمة على الصور الحية لمراقبة تمايز الخلايا الجذعية متعددة القدرات
* These authors contributed equally
In This Article
Summary
يتم حاليا إعاقة أنظمة تمايز الخلايا الجذعية متعددة القدرات (PSC) إلى الخلايا الوظيفية بسبب مشاكل التباين الشديد من خط إلى خط ومن دفعة إلى دفعة. هنا ، باستخدام التمايز القلبي كمثال رئيسي ، نقدم بروتوكولا لمراقبة وتعديل عملية تمايز PSC بذكاء بناء على التعلم الآلي المستند إلى الصور.
Abstract
تم استخدام تقنيات الخلايا الجذعية متعددة القدرات (PSC) على نطاق واسع في اكتشاف الأدوية ونمذجة الأمراض والطب التجديدي. ومع ذلك ، فإن أنظمة التمايز الوظيفية المتاحة من PSC إلى الخلايا الوظيفية تعيقها مشاكل التباين الشديد من خط إلى خط ومن دفعة إلى دفعة. لذلك من المهم التحكم الدقيق في تمايز الخلايا في الوقت الفعلي. في هذا البروتوكول ، نصف استراتيجية ذكية وغير جراحية تتغلب على التباين في تمايز الخلايا باستخدام التعلم الآلي القائم على الصور ذات المجال الساطع. مع أخذ التمايز بين PSC إلى خلايا عضلة القلب كمثال ، توفر هذه المنهجية معلومات مفصلة للتحكم في حالة PSC الأولية ، والتقييم المبكر والتدخل في ظروف التمايز ، والقضاء على تلوث الخلايا غير المتمايز ، معا تحقيق تمايز عالي الجودة باستمرار من PSCs إلى الخلايا الوظيفية. من حيث المبدأ ، يمكن توسيع هذه الاستراتيجية لتشمل أنظمة تمايز الخلايا أو إعادة البرمجة الأخرى بخطوات متعددة لدعم تصنيع الخلايا ، وكذلك لتعزيز فهمنا للآليات أثناء تحويل مصير الخلية.
Introduction
تمتلك الخلايا الجذعية متعددة القدرات (PSCs) قدرة رائعة على التمايز إلى أنواع عديدة من الخلايا في المختبر. يمكن استخدام هذه الخلايا الوظيفية المتمايزة للعلاج الخلوي ونمذجة الأمراض وتطوير الأدوية ، وكلها ذات قيمة للبحث أو التطبيقات السريرية1،2،3. على سبيل المثال ، تم تطوير مجموعة متنوعة من الطرق للتمييز بين PSCs إلى خلايا عضلة القلب (CMs) 4،5،6،7. يمكن تطبيق CMs هذه لاختبار السمية القلبية للأدوية ، ونمذجة أمراض القلب ، وزرع الخلايا8،9،10،11.
ومع ذلك ، فإن التحويل من PSC إلى الخلايا المتمايزة الطرفية هو عملية تدريجية ، وقد تؤدي الاضطرابات المتعددة أثناء عملية التمايز إلى الخلايا إلى مصائر خلايا متباينة. تؤثر الخلفيات الجينية المختلفة والعلامات اللاجينية لخطوط PSC على إمكانية التمايز إلى سلالة معينة12،13،14،15. عدد مقاطع PSC والطفرات الجينية المتراكمة هي أيضا مصادر لعدم تجانس PSC. يمكن أن تؤدي الاختلافات في العمليات التجريبية التي يستخدمها المجربون المختلفون أيضا إلى نتائج تمايز مختلفةتماما 16،17،18،19،20. لذلك ، تتمثل إحدى المشكلات الرئيسية حاليا في إنتاج الخلايا المشتقة من PSC في عدم الاستقرار بين خطوط الخلايا والدفعات21،22،23،24،25. غالبا ما يؤدي عدم الاستقرار في تمايز PSC إلى تجارب متكررة متعددة ، مما يستهلك وقتا كبيرا وموارد عمالة. لمعالجة هذه المشكلة ، من الأهمية بمكان تطوير استراتيجية تقلل من التباين بين خطوط الخلايا والدفعات ، وبالتالي تعزيز استقرار ومتانة التمايز.
في الآونة الأخيرة ، سهلت التطورات في الفحص المجهري عالي الدقة والتعلم الآلي (ML) تطبيق تحليل الصور الكمي المستند إلى التعلم الآلي في بيولوجيا الخلية ، مما يجعل من الممكن استخدام معلومات قيمة في ميزات تصوير الخلايا26،27،28،29،30،31،32،33،34. في عملنا السابق ، اقترحنا استراتيجية ML قائمة على الصور الحية لمراقبة حالة تمايز PSC والتدخل فيها في الوقت الفعلي لتحسين استقرار وكفاءة تمايز PSC (الشكل 1) 35. بأخذ تمايز PSC إلى عضلة القلب كمثال ، قمنا بتقييم حالة PSC الأولية باستخدام نماذج الغابات العشوائية ، وتوقعنا حالة التمايز المثلى باستخدام نماذج الانحدار اللوجستي ، وتعرفنا على الخلايا المتمايزة بنجاح باستخدام Grad-CAM36 القائم على التعلم العميق و pix2pix37. تعلمت نماذج ML تحديد سلالات الخلايا من مجموعة من الميزات المورفولوجية للمجال الساطع ، بما في ذلك ميزات حول المساحة ، والمحيط ، والتحدب ، والصلابة ، والسطوع ، والسرعة المتحركة ، وغيرها من الميزات الضمنية المستخرجة بواسطة الشبكات العصبية التلافيفية العميقة. بناء على الاستدلال من نماذج ML الراسخة هذه ، أدركنا التحكم في حالة PSC الأولية ، والتقييم المبكر والتدخل في ظروف التمايز ، والقضاء على تلوث الخلايا غير المتمايز ، مما يوفر معا تعديلا شاملا ودقيقا لعملية التمايز القلبي. نقدم هنا بروتوكولا خطوة بخطوة لتطوير الاستراتيجية.
Protocol
1. تمايز الخلايا وتوصيفها
- تحضير الكواشف الاستزراعية وألواح الاستزراع
- تحضير وسط استزراع PSC بإضافة 2 مل من المكملات الغذائية و 0.2٪ بنسلين ستربتومايسين إلى 48 مل من الوسط القاعدي. Aliquot وتخزين المكمل في -20 درجة مئوية. قم بتخزين هذه الوسط عند 4 درجات مئوية لمدة تصل إلى 4 أسابيع.
- تحضير وسط تحضير PSC عن طريق إضافة 1 مل من المكملات الغذائية و 0.2٪ بنسلين ستربتومايسين إلى 500 مل من الوسط القاعدي. عند الاستخدام ، سخن الوسط مسبقا للاستخدام لمرة واحدة ، وقم بتخزين الوسط عند 4 درجات مئوية لمدة تصل إلى 3 أسابيع.
- قم بإعداد وسيط تمايز CM عن طريق إضافة 1x B27 مكمل ناقص الأنسولين و 1٪ بنسلين ستربتومايسين إلى RPMI 1640. قم بإعداد وسيط صيانة CM عن طريق إضافة 1x B27 supplement و 1٪ Penicillin-Streptomycin إلى RPMI 1640. قم بتخزين هذين النوعين من الوسائط في درجة حرارة 4 درجات مئوية لمدة أسبوعين.
- اغمر Matrigel في الجليد واحتفظ به عند 4 درجات مئوية طوال الليل. تأكد من إذابة Matrigel تماما ؛ بعد ذلك ، قم بتقطيع 1 مل من الحصص المخزنة عند -80 درجة مئوية لاستخدامها لاحقا ، وأضف 1 مل من Matrigel إلى 49 مل من DMEM / F12 عند 4 درجات مئوية لتحضير محلول عمل Matrigel. أضف 850 ميكرولتر من محلول عمل Matrigel لكل بئر من لوحة استزراع 6 آبار ، واحتضن اللوحة عند 37 درجة مئوية لمدة 30 دقيقة على الأقل. بالنسبة للوحة الاستزراع المكونة من 96 بئرا المستخدمة في تمايز CM ، أضف 35 ميكرولتر من محلول عمل Matrigel لكل بئر.
ملاحظة: حافظ على Matrigel على الجليد في جميع الأوقات. يجب تبريد أطراف وأنابيب الماصة مسبقا عند 4 درجات مئوية. يمكن تخزين محلول عمل Matrigel عند 4 درجات مئوية لمدة أسبوعين. كن حذرا بشأن تجنب الفقاعات عند سحب محلول عمل Matrigel.
- صيانة ومرور PSC
- حافظ على PSCs في ألواح استزراع 6 آبار. تأكد من كثافة الخلية تحت المجهر واستعد للمرور عندما يصل الالتقاء إلى ~ 70٪ (الشكل 2 أ).
- استخدم EDTA لهضم الخلايا للمرور. وسط استزراع PSC الدافئ ، PBS ، و EDTA في حمام مائي 37 درجة مئوية ، وأضف Y27632 في الوسط إلى التركيز النهائي 5 ميكرومتر.
- اغسل PSCs ب 1 مل من PBS ، وأضف 1 مل من EDTA لكل بئر ، واحتضن اللوحة في حاضنة 37 درجة مئوية لمدة 3 دقائق.
- قم بشفط EDTA قبل أن تنفصل الخلايا عن لوحة الثقافة ، ثم استخدم 1 مل من وسط زراعة PSC لشطف قاع البئر برفق 3-4x لإعادة تعليق الخلايا.
ملاحظة: هذا يسمح لنا بتخطي خطوة الطرد المركزي وتقليل تلف الخلايا في عمليات المرور. - قم بشفط DMEM / F12 من بئر مطلي ب Matrigel من صفيحة استزراع مكونة من 6 آبار وأضف 2 مل من وسط استزراع PSC الطازج لكل بئر (تمت إضافة Y27632). أضف تعليق الخلية إلى الآبار بنسبة الانقسام من 1: 6 إلى 1:12. ضع اللوحة في حاضنة 37 درجة مئوية مع 5٪ ثاني أكسيد الكربون2.
- قم بتغيير وسط استزراع PSC (بدون Y27632) بعد 12 إلى 24 ساعة من المرور ؛ بعد ذلك ، قم بتغيير الوسيط وتحقق من حالة الخلية والتقاء كل يوم.
- التمايز القلبي لمركبات PSC
- لتمايز CM ، قم بزرع PSCs في صفيحة استزراع 96 بئرا في وسط تحضير PSC (تمت إضافة Y27632). بالنسبة لتمايز CM ، اتبع نفس الخطوات أعلاه (الخطوات 1.2.2-1.2.6) لزرع PSCs في صفيحة استزراع 96 بئرا في وسط تحضير PSC (تمت إضافة Y27632).
ملاحظة: في هذه المرحلة ، يمكن تمديد مدة علاج EDTA بشكل مناسب (5 دقائق أو أكثر) لفصل PSCs بشكل كاف بحيث يمكن أن تكون المستنسخة أصغر بعد المرور. أظهرت دراستنا أنه في نفس المنطقة ، تساعد المستعمرات الأصغر على التمايز. - المرحلة الأولى: عندما تصل PSCs إلى التقاء 80-90٪ ، قم بتبديل وسط التمايز المتوسط إلى CM مع 2-20 ميكرومتر CHIR99021 (CHIR) (الشكل 2 ب). يشار إلى النقطة الزمنية للتبديل المتوسط باليوم 0. بعد 24-48 ساعة من علاج CHIR ، قم بتغيير وسط تمايز CM المتوسط إلى الجديد.
ملاحظة: CHIR هو مثبط GSK3beta لتحريض الأديم المتوسط. تتقلب جرعة CHIR المثلى لتمايز CM مع خطوط الخلايا والدفعات المختلفة. على سبيل المثال, الجرعة المثلى CHIR هي 8-10 ميكرومتر, 48 ساعة ل iPS-B1. لجمع صور متنوعة للمجال الساطع لتدريب نموذج ML ، قمنا هنا بمعايرة تركيزات CHIR لكل دفعة من التجارب من أجل كفاءات تمايز مختلفة. - المرحلة الثانية: في الساعة 72 (اليوم 3) ، استبدل الوسط بوسط تمايز CM المكمل ب 5 ميكرومولار IWR1 والزراعة لمدة 48 ساعة (اليوم 5). قم بتغيير الوسيط باستخدام وسيط تمايز CM لمدة 1-2 أيام (إلى الأيام 6-7). في هذه المرحلة ، تتمايز PSCs إلى خلايا سلفية للقلب (CPC).
- المرحلة الثالثة: استبدل الوسيط بوسيط صيانة CM واستبدل الوسيط كل 3 أيام. في اليوم 10 أو اليوم 12 ، احصد CM لمزيد من التحليل.
- لتمايز CM ، قم بزرع PSCs في صفيحة استزراع 96 بئرا في وسط تحضير PSC (تمت إضافة Y27632). بالنسبة لتمايز CM ، اتبع نفس الخطوات أعلاه (الخطوات 1.2.2-1.2.6) لزرع PSCs في صفيحة استزراع 96 بئرا في وسط تحضير PSC (تمت إضافة Y27632).
- تلطيخ التألق المناعي ل CM
- في اليوم 10 أو اليوم 12 ، اغسل الخلايا باستخدام PBS وقم بإصلاحها بنسبة 4٪ بارافورمالدهيد في PBS لمدة 15 دقيقة في درجة حرارة الغرفة. اغسل CMs 2x باستخدام PBS وقم بتخزين اللوحة عند 4 درجات مئوية لمدة تصل إلى ~ 1 أسبوع.
ملاحظة: بارافورمالدهيد هو كاشف سام ذو رائحة نفاذة. تعمل في غطاء كيماوي وتحمي العينين واليدين. - في وقت التلوين ، عالج الخلايا بمحلول نفاذية (0.1٪ Triton X-100 في PBS) لمدة 15 دقيقة في درجة حرارة الغرفة ؛ بعد ذلك ، احتضان الخلايا في محلول مانع (0.1٪ Triton X-100 يحتوي على 3٪ مصل حمار في PBS) لمدة 30 دقيقة في درجة حرارة الغرفة.
- احتضان العينة بالجسم المضاد الأساسي للقلب تروبونين تي (cTnT) المخفف في محلول مانع طوال الليل عند 4 درجات مئوية لتحديد CMs.
- اجمع الجسم المضاد الأساسي واغسل الخلايا باستخدام PBS 3x. احتضان العينة بالأجسام المضادة الثانوية في PBS مع ألبومين مصل الأبقار بنسبة 1٪ لمدة ساعة واحدة عند 37 درجة مئوية في بيئة مظلمة.
ملاحظة: يمكن إعادة استخدام الجسم المضاد الأساسي الذي تم جمعه لمدة 2-3x في أسبوع واحد.
- اجمع الجسم المضاد الأساسي واغسل الخلايا باستخدام PBS 3x. احتضان العينة بالأجسام المضادة الثانوية في PBS مع ألبومين مصل الأبقار بنسبة 1٪ لمدة ساعة واحدة عند 37 درجة مئوية في بيئة مظلمة.
- قم بإزالة الجسم المضاد الثانوي من الخلايا واغسل الخلايا 3x باستخدام PBS. استخدم Hoechst 33342 (1: 1,000 في PBS) لتلطيخ النوى لمدة 5 دقائق في درجة حرارة الغرفة. اشطف الخلية 3 مرات باستخدام PBS وأضف 100 ميكرولتر من PBS لكل بئر لتجنب الجفاف. قم بتخزين اللوحة في درجة حرارة 4 درجات مئوية في مكان مظلم واحصل على صور الفلورسنت في غضون أسبوع واحد.
- في اليوم 10 أو اليوم 12 ، اغسل الخلايا باستخدام PBS وقم بإصلاحها بنسبة 4٪ بارافورمالدهيد في PBS لمدة 15 دقيقة في درجة حرارة الغرفة. اغسل CMs 2x باستخدام PBS وقم بتخزين اللوحة عند 4 درجات مئوية لمدة تصل إلى ~ 1 أسبوع.
2. الحصول على دفق الصور طوال عملية التمايز
- إعداد التصميم التجريبي لجمع الصور
- استخدم مجهرا آليا يدعم زراعة الخلايا الحية والتصوير لجمع صور المجال الساطع لمراحل مختلفة من تمايز CM وصور الفلورسنت المناعي cTnT لنتائج التمايز (الشكل 2A-E). استخدم البرنامج الداعم للمجهر لتصميم برنامج التجربة والتحكم في المجهر.
- افتح البرنامج وأنشئ تصميما تجريبيا جديدا. اختر هدفا 5x وعدسة أنبوبية 2x للتصوير. تحقق من خيارات البلاط ومكدس Z.
- أضف قناة TL Brightfield لتصوير المجال الساطع في قائمة القنوات. أضف قنوات AF488 و H3342 للتصوير المناعي الفلوري. قم بتعديل مسار الضوء في قائمة إعداد التصوير للتصوير باستخدام كاميرا CMOS الرقمية الخارجية.
- افتح قائمة وضع الاكتساب ، وقم بتعيين تجميع 2 × 2 لزيادة نسبة الإشارة إلى الضوضاء.
- افتح قائمة Z-stack واختر نموذج المركز. اضبط عدد الشرائح والفترات الزمنية عند المسح الضوئي. اختر 3-5 شرائح بفواصل زمنية تتراوح بين 3 و6 ميكرومتر للصور في التركيز البؤري في التحليل.
- في نافذة التنقل والمربعات، قم بإعداد مناطق التجانب بواسطة الناقل وقم بتعيين 25 بلاطة (5 أعمدة × 5 صفوف) لبئر واحد. في القائمة الإطارات المتجانبة، حدد قالب Multiwell 96 في Sample Carrier، وقم بتعيين تداخل 5٪ -15٪ على المناطق المجاورة.
- إذا كانت هناك حاجة إلى التصوير المستمر، فحدد خيار سلسلة Time واضبط المدة والفاصل الزمني في قائمة السلسلة الزمنية. لجمع دفق الصورة في المرحلة الأولى ، قم بتعيين مدة 12 ساعة دون فترات.
- الحصول على صور المجال الساطع والفلورسنت المناعي
- للحصول على الصور ، ضع أولا لوحة زراعة الخلية في علبة العينات ، وقم بتحميل العينة داخل المجهر. حدد قالب Multiwell 96 لمطابقة التصميم التجريبي. إذا كانت العينة تتكون من خلايا حية ، فافتح نظام التسخين ومضخة ثاني أكسيد الكربون2 للحفاظ على الحالة المناسبة للزراعة (37 درجة مئوية ، 5٪ ثاني أكسيدالكربون 2).
- افتح المشروع التجريبي المحدد مسبقا واحفظه كمشروع جديد. افتح قائمة البلاط وقم بمعايرة موضع اللوحة يدويا. في نافذة التنقل والمربعات ، حدد الآبار المطلوبة وانقر فوق إنشاء لإنشاء مناطق تجانب لهذه الآبار.
- انقر فوق التحقق من مناطق التجانب في قائمة المربعات وقم بتشغيل التركيز البؤري التلقائي للتحقق من كافة الآبار. بعد ذلك ، قم بتصحيح تركيز كل بئر يدويا أسفل الحقل الساطع عن طريق تمرير عجلة الماوس.
- حدد القنوات المطلوبة واضبط أوقات التعرض في قائمة القنوات. أوقات التعريض الضوئي الموصى بها هي 1.625 مللي ثانية للسطوع ، و 100 مللي ثانية ل H3342 ، و 550 مللي ثانية ل AF488. قم بتنفيذ لقطة واحدة للتحقق من صور جميع القنوات.
- أخيرا ، انقر فوق الزر "بدء التجربة" ، وانتظر التصوير التلقائي. عادة ما يستغرق الأمر ~ 1.2 ساعة لإنهاء مسح لوحة ثقافة كاملة مكونة من 96 بئرا (96 بئرا ، ثلاث طبقات ، 25 بلاطة لكل بئر تحت الحقل الساطع). في إطار عمل المعالجة، اختر Image Export، وحدد نوع الملف بتنسيق TIFF غير المضغوط أو تنسيق PNG، ثم قم بتطبيقه. استخدم صورا من 9 مربعات (3 × 3) في وسط البئر لمزيد من التحليل (الشكل 2A-E).
3. وضع استراتيجية التعلم الآلي المستندة إلى الصور في كل مرحلة من مراحل عملية التمايز
- إعداد البرامج
- قم بتثبيت بيئة Python ، مع حزم pytorch (1.9.0) و numpy و scipy و scikit-learn و pandas و visdom و scikit-image و opencv-python و matplotlib.
- قم بتثبيت Jupyter Notebook.
- قم بتثبيت MATLAB.
- قم بتنزيل الكود ومجموعات البيانات النموذجية من https://github.com/zhaoyanglab/ML-for-PSC-differentiation.
ملاحظة: يعتمد أداء التعلم الآلي على جودة مجموعات البيانات واختيار النماذج والمعلمات الفائقة. لا يضمن تنفيذ المثال أن يكون مثاليا لجميع الظروف. يجب على مهندسي ML تخصيص الكود وتخصيص النموذج والمعلمات الفائقة لاحتياجاتهم الخاصة.
- استراتيجية التعلم الآلي في مرحلة PSC: السيطرة على دول مستعمرة PSC الأولية
مجموعة البيانات: أزواج من صور المجال الساطع في 0 ساعة (قبل معالجة CHIR) وكفاءة التمايز النهائية
نموذج: غابة عشوائية- قم بإعداد مجموعة بيانات تتكون من صور المجال الساطع في 0 ساعة (قبل معالجة CHIR) وصور مضان cTnT النهائية. لزيادة تنوع الملامح المورفولوجية لصور المجال الساطع 0 ساعة ، تأكد من أن مجموعة البيانات تتضمن مستعمرات PSC من خطوط خلايا مختلفة ، مع بدء التمايز على مدى فترة زمنية بعد المرور. معالجة الآبار في مجموعة البيانات بظروف CHIR المثلى في المرحلة الأولى.
ملاحظة: لضمان معالجة المستعمرات بظروف CHIR المثلى ، يمكن معالجة الآبار الموجودة في نفس الدفعة بجرعات CHIR الممعايرة. يتم تضمين الآبار ذات ظروف CHIR عالية الكفاءة فقط في مجموعة البيانات. - حدد كفاءة التمايز لكل بئر بواسطة مؤشر كفاءة التمايز المحسوب من صور التألق cTnT ، والتي يتم تحديدها بواسطة
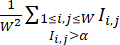 (حيث
(حيث 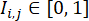 يمثل شدة التألق في موقع البكسل (i ، j) في صورة مضان W × W ، ويتم أخذ المجموع على جميع مواقع البكسل بكثافة أعلى من عتبة α، عادة ما يتم ضبطه على 0.5). داخل كل خط خلية ، احسب الحد الأقصى لمؤشر كفاءة التمايز على جميع الآبار في خط الخلية هذا ، وقم بتطبيع مؤشرات كفاءة التمايز الخاصة بها بالقيمة القصوى.
يمثل شدة التألق في موقع البكسل (i ، j) في صورة مضان W × W ، ويتم أخذ المجموع على جميع مواقع البكسل بكثافة أعلى من عتبة α، عادة ما يتم ضبطه على 0.5). داخل كل خط خلية ، احسب الحد الأقصى لمؤشر كفاءة التمايز على جميع الآبار في خط الخلية هذا ، وقم بتطبيع مؤشرات كفاءة التمايز الخاصة بها بالقيمة القصوى.
ملاحظة: نظرا لأن إمكانات التمايز لكل خط خلوي مختلفة بطبيعتها ، فإن مثل هذا التطبيع سيؤكد أكثر على السمات المورفولوجية للمستعمرة المشتركة بين خطوط الخلايا المختلفة التي تساهم في نتيجة التمايز. التطبيع في الواقع يجعل مستعمرات PSC من خطوط الخلايا المختلفة قابلة للمقارنة ويخفف من التحيز الناجم عن خطوط الخلايا. - حدد الملامح المورفولوجية لصور المجال الساطع 0 ساعة من خلال ميزات عالية الأبعاد تكشف عن خصائص شكل المستعمرة. لتحديد مناطق المستعمرة في صور المجال الساطع ، احسب الانتروبيا المحلية (بنصف قطرها r) ؛ يتم تحديد وحدات البكسل ذات الانتروبيا المحلية الأكبر من σ العتبة بواسطة الخلايا المغطاة بالسكان. استخدم إعداد معلمة r = 10 بكسل و σ = 3. قم بتبديل دليل العمل إلى "./colony_control" ؛ احفظ صور Brightfield والصور الثنائية لمناطق الخلية وصور مضان cTnT في "colony_control/ image_data / CD00-*/[brightfield|cell_region|ctnt]/S *.png"؛ وقم بتشغيل Jupyter Notebook "Compute_features.ipynb" لحساب الميزات المتعلقة بمساحة المستعمرة ، والمحيط ، والصلابة ، والتحدب ، والدائرية ، ومسافات كفاف النقطة الوسطى ، من بين أمور أخرى.
- قسم مجموعة البيانات بشكل عشوائي إلى مجموعة تدريب ومجموعة اختبار ، حيث تضم مجموعة التدريب ما بين 50٪ و 80٪ من البيانات الإجمالية. قم بتشغيل الكتلة النهائية تقسيم مجموعة البيانات في Jupyter Notebook لتقسيم مجموعة البيانات (الشكل 3 أ).
- تدريب نموذج انحدار غابة عشوائي على مجموعة التدريب للتنبؤ بكفاءة التمايز من ميزات صورة المجال الساطع 0 ساعة. قم بتشغيل Jupyter Notebook "Machine_learning.ipynb" لتدريب النموذج واختباره.
- تقييم نموذج الغابة العشوائية المدرب على مجموعة الاختبار. تأكد من أن كفاءة التمايز المتوقعة ترتبط ارتباطا وثيقا بالكفاءة الحقيقية باستخدام معامل ارتباط بيرسون (الشكل 3 ب).
ملاحظة: نظرا لأن مجموعة الاختبار لا تشارك في مرحلة التدريب ، فإن الاتساق العالي بين كفاءة التمايز المتوقعة والحقيقية في مجموعة الاختبار يشير إلى أن النموذج يمكن أن يعمم جيدا على العينات غير المرئية. - احصل على أهمية الميزة من نموذج الغابة العشوائية المدرب وحدد 8 ميزات ذات أهمية أكبر. تصور علاقتها بكفاءة التمايز النهائية عن طريق تقسيم نطاق الميزات إلى صناديق وحساب متوسط كفاءة التمايز في كل صندوق. قم بتشغيل Jupyter Notebook "Feature_importance.ipynb" لإجراء التصور (الشكل 3C).
ملاحظة: قد يساعد التصور في فهم كيف تساهم التغييرات في شكل مستعمرة PSC في التمايز. - تطبيق النموذج المدرب على دفعات جديدة لمراقبة حالات مستعمرة PSC في الوقت الفعلي. التقط صورة المجال الساطع في كل ساعة ، وقم بتمرير الصورة إلى النموذج المدرب ، واحصل على كفاءة التمايز المتوقعة. بمجرد أن تتجاوز الكفاءة المتوقعة عتبة (على سبيل المثال ، 50٪) ، ابدأ عملية التمايز. إذا لم تتمكن الكفاءة المتوقعة من الوصول إلى العتبة ، فتجاهل هذه العينات.
ملاحظة: خلال مرحلة PSC ، سترتفع إمكانات التمايز لمستعمرات PSC أولا وتنخفض لاحقا. تمكن المراقبة في الوقت الفعلي المجربين من تحديد النقطة الزمنية الأكثر ملاءمة لبدء التمايز.
- قم بإعداد مجموعة بيانات تتكون من صور المجال الساطع في 0 ساعة (قبل معالجة CHIR) وصور مضان cTnT النهائية. لزيادة تنوع الملامح المورفولوجية لصور المجال الساطع 0 ساعة ، تأكد من أن مجموعة البيانات تتضمن مستعمرات PSC من خطوط خلايا مختلفة ، مع بدء التمايز على مدى فترة زمنية بعد المرور. معالجة الآبار في مجموعة البيانات بظروف CHIR المثلى في المرحلة الأولى.
- استراتيجية غسل الأموال في المرحلة الأولى: التقييم المبكر لجرعات CHIR وتعديلها
مجموعة البيانات: أزواج من تدفقات صور المجال الساطع في 0-12 ساعة وملصقات تركيز CHIR (منخفضة/مثالية/عالية)
نموذج: الانحدار اللوجستي- قم بإعداد مجموعة بيانات تتكون من تدفقات صور الحقل الساطع بالكامل ، حيث يتم الحصول على الصور كل ساعة بين 0 ساعة و 12 ساعة. سجل جرعات CHIR (أي مجموعات من تركيزات CHIR ومدتها) لكل بئر. تأكد من أن مجموعة البيانات تتضمن دفعات وآبار مختلفة بجرعات CHIR معايرة (على سبيل المثال ، التركيز = 2 و 4 و 6 و 8 و 10 و 12 ميكرومتر ؛ المدة = 24 و 36 و 48 ساعة) لزيادة التنوع. قم بتبديل دليل العمل إلى "./stage_I" ؛ احفظ الصور ك "./data/image/CD01-*/S*/T*.png" ، حيث CD01-* (CD01-1 ، CD01-2 ، CD01-3 ، CD01-4) هو اسم الدفعة ، S * (S1 ، S2 ، ... ، S96) هو مؤشر البئر ، و T * .png (T1.png ، T2.png ، ... ، T10.png) هي صورة المجال الساطع المعالجة مسبقا للبئر في خطوة زمنية مختلفة من دفق الصورة.
- في كل دفعة ، احسب النسبة المئوية لخلايا cTnT + لكل بئر ، والتي يتم حسابها بقسمة عدد خلايا cTnT + على العدد الإجمالي لخلايا Hoechst + في صورة التألق. متوسط النسبة المئوية لخلايا cTnT + على آبار متوازية بنفس حالة جرعة CHIR. احفظ حالة جرعة CHIR والنسبة المئوية لخلايا cTnT + لكل بئر في ملف csv "./data/image/CD01-*/CD01-?_exp_config.csv".
- في كل دفعة ، حدد نطاق تركيز CHIR "المنخفض" و "الأمثل" و "العالي" تحت كل مدة CHIR وفقا للمعايير التالية. يتم تحديد تركيزات CHIR مع متوسط نسبة مئوية من خلايا cTnT + أعلى من العتبة (على سبيل المثال ، 20٪) على أنها "مثالية". يتم تحديد تركيزات CHIR التي تتجاوز النطاق الأمثل على أنها "منخفضة" أو "عالية". تحت كل مدة CHIR ، قم بتسمية الآبار بأنها "منخفضة" و "مثالية" و "عالية" وفقا لتركيزات CHIR المقابلة لها.
ملاحظة: من الناحية التجريبية ، هناك ارتباط سلبي بين تركيزات CHIR ومدة الآبار عالية الكفاءة ضمن دفعة: بالنسبة لفترة CHIR الأعلى ، تنخفض التركيزات المثلى. بمعنى آخر ، يعتمد القدر الأمثل لتركيزات CHIR على مدة CHIR ، وبالتالي ، تختلف ملصقات الآبار بين فترات CHIR المختلفة. - في كل دفعة ، تحت كل مدة CHIR ، احسب تركيز ΔCHIR لكل تركيز c لتحديد انحرافه عن المستوى الأمثل. تشير إلى نطاق تركيز CHIR الأمثل بمقدار [c1 ، c2] ، ثم تركيز ΔCHIR (c) = c - c 1 (إذا كان c < c1) ، 0 (إذا كان c1 ≤ c ≤ c2) ، أو c - c 2 (إذا كان c > c2).
- استخراج ميزات تدفقات الصور في مجموعة البيانات ، مع التأكد من أنها تعكس التغيرات المورفولوجية للخلايا خلال المرحلة الأولى. لحساب ميزات الصورة حول الانتروبيا المحلية والمساحة والمحيطات والسطوع والتدفق البصري والبعد الكسوري، قم بتشغيل
قرص مضغوط ./بيانات
بايثون compute_features.py
قرص مضغوط ..
ملاحظة: لتسليط الضوء على التغييرات المورفولوجية ، يمكن تطبيع قيم بعض الميزات (على سبيل المثال ، المساحة والمحيطات والتدفق البصري) من خلال قيم خط الأساس الأولية في تدفقات الصور. - قسم مجموعة البيانات بشكل عشوائي إلى مجموعة تدريب ومجموعة اختبار. قم بتشغيل Jupyter Notebook "./data/prepare_dataset.ipynb" لتقسيم مجموعة البيانات وتنظيم مجموعات البيانات كإطارات بيانات (الشكل 3D).
- لكل مدة CHIR ، قم بتدريب نموذج انحدار لوجستي للتنبؤ بتسمية تركيز CHIR (منخفضة / مثالية / عالية) من ميزات دفق الصور في مجموعة التدريب. تقييم أداء تصنيف نموذج الانحدار اللوجستي المدرب على مجموعة الاختبار باستخدام الدقة والدقة والاستدعاء ودرجة F1 والمساحة تحت المنحنى (AUC). قم بتشغيل Jupyter Notebook "machine_learning.ipynb" للتدريب والاختبار (الشكل 3E).
- تقييم أداء النموذج في تقييم جرعة CHIR. في مجموعة الاختبار ، قم بدمج الملصقات المتوقعة للآبار المتوازية بنفس تركيز CHIR باستخدام درجات الانحراف (تتراوح من -1 إلى 1) ، المحددة بواسطة
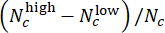 ، حيث يشير Nc إلى عدد الآبار ذات التركيز c والآبار
، حيث يشير Nc إلى عدد الآبار ذات التركيز c والآبار 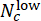
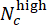 من بينها متوقعة على أنها "منخفضة" و "عالية" ، على التوالي. تأكد من أن درجات الانحراف المتوقعة ترتبط ارتباطا وثيقا بتركيز ΔCHIR الحقيقي لكل جرعة CHIR باستخدام معامل ارتباط بيرسون.
من بينها متوقعة على أنها "منخفضة" و "عالية" ، على التوالي. تأكد من أن درجات الانحراف المتوقعة ترتبط ارتباطا وثيقا بتركيز ΔCHIR الحقيقي لكل جرعة CHIR باستخدام معامل ارتباط بيرسون. - قم بإجراء التحقق من صحة الدفعات لاختبار أداء التعميم للنموذج. في كل جولة ، اترك دفعة واحدة للاختبار أثناء استخدام الآخرين للتدريب. قارن درجات الانحراف المتوقعة وتركيز ΔCHIR الحقيقي لكل جرعة CHIR في كل دفعة اختبار باستخدام معامل ارتباط بيرسون. قم بتشغيل Jupyter Notebook "cross_batch_validation.ipynb" لتنفيذ التحقق من صحة الدفعات المتقاطعة (الشكل 3F).
ملاحظة: بالنسبة لمدة CHIR المختارة h ، سيتم تحويل التنبؤ لجميع الآبار بالتركيز من نموذج الانحدار اللوجستي المدرب تحت المدة h إلى درجة انحراف ، مما يعكس القدر الأمثل لتركيز CHIR c. تشير درجة الانحراف القريبة من 0 إلى أنه من المتوقع أن يكون التركيز c هو الأمثل تحت المدة h ، بينما تشير درجة الانحراف السلبية أو الإيجابية إلى حالة جرعة منخفضة أو جرعة زائدة ، والتي يمكن أن تكون بمثابة إشارة لتعديل جرعات CHIR لاحقا. لاحظ أنه نظرا لأن التنبؤ يتم في وقت مبكر (12 ساعة) ، فلا يزال من الممكن تعديل المدة النهائية ل CHIR وتركيز CHIR خلال بقية المرحلة الأولى. - تطبيق نماذج الانحدار اللوجستي المدربة لتقييم جرعات CHIR على دفعات جديدة. للحصول على تركيز معين من CHIR ، احسب ميزات دفق الصور من 0 إلى 12 ساعة للآبار المتوازية ذات التركيز ، وقم بتمرير الميزات إلى نماذج الانحدار اللوجستي المدربة في ظل فترات مختلفة من CHIR ، وبالتالي ، احصل على الملصقات المتوقعة لكل بئر. بناء على تنبؤ النماذج ، احسب درجات الانحراف لتقييم كل تركيز CHIR تحت فترات CHIR المختلفة.
- من خلال تقييم جرعة CHIR المستند إلى النموذج ، يتم إنقاذ الآبار تحت كل تركيز CHIR دون المستوى الأمثل وفقا لذلك ، عن طريق تعديل مدة أو تركيز CHIR نحو الأمثل قبل 48 ساعة.
- تعديل المدة: وفقا لتوقع النموذج حول المدة المثلى ل CHIR ، اضبط النقطة الزمنية عندما يتم تحويل الوسط المحتوي على CHIR في هذه الآبار إلى وسط تمايز CM. إذا فاتتك النقطة الزمنية المثلى، فتجاهل هذه العينات.
- ضبط التركيز: تحت مدة CHIR المحددة مسبقا ، إذا كان من المتوقع أن يكون التركيز الحالي "منخفضا" ، أضف تركيزا عاليا من CHIR مباشرة إلى الوسط لزيادة تركيز CHIR. قم بتخفيف CHIR في DMSO (50 ملم) باستخدام PBS عند الحاجة ، أضف حجما مناسبا من السائل ، وقم بهز اللوحة برفق. إذا كان التركيز الحالي متوقعا على أنه "مرتفع", استنشاق حجم معين من الوسط المحتوية على CHIR واستكمل بنفس الحجم من وسط التمايز CM.
ملاحظة: في أول 48 ساعة من التمايز ، تفرز الخلايا عوامل لتحسين نمو الخلايا وتمايزها ، لذلك لا تغير الوسط تماما. بالإضافة إلى ذلك ، من الضروري الحفاظ على الحجم المتوسط ثابتا في التمايز. لذلك ، تأكد من أن حجم السائل المضاف صغير قدر الإمكان عند ضبط تركيز CHIR.
- استراتيجية غسل الأموال في المرحلة الثانية: الاعتراف بالتزامات الأولوية للشركات الملتزمة بإدارة الموارد
مجموعة البيانات: أزواج من صور المجال الساطع في اليوم 6 والأقنعة المشروحة يدويا للتكلفة لكل نقرة ملتزمة ب CM
الموديل: ResNeSt38 مع Grad-CAM36 للتعلم الضعيف الإشراف- قم بإعداد مجموعة بيانات تتكون من صور المجال الساطع في اليوم 6. قم بالتعليق التوضيحي يدويا على تكلفة النقرة الملتزمة ب CM في صور الحقل الساطع عن طريق تعقب خلايا cTnT + في تدفقات الصور من اليوم 12 إلى اليوم 6.
- قم بقص صور الحقل الساطع والتعليق التوضيحي اليدوي المقابل (يطلق عليه الأقنعة) لتكلفة النقرة الملتزمة ب CM إلى تصحيحات ، مع حجم كل تصحيح عند 1 / 5.5 من حجم صورة البئر بالكامل. استخرج التصحيحات بطريقة متداخلة حيث تشترك التصحيحات المجاورة في تداخل بنسبة 50٪ في مجموعة التدريب وتداخل بنسبة 75٪ في مجموعة الاختبار. قم بتصنيف التصحيحات التي تحتوي على ≥30٪ من تكلفة النقرة الملتزمة ب CM على أنها "إيجابية". قم بتصنيف التصحيحات التي لا تحتوي على تكلفة نقرة ملتزمة ب CM على أنها "سلبية". قم بتبديل دليل العمل إلى "stage_II". قم بتشغيل "./Crop_and_Reconstruct/mask_crop.m" لاقتصاص الصور. قم بتشغيل "./Crop_and_Reconstruct/classification.m" لتعيين التسميات (0: سلبية؛ 1: موجبة) لتصحيحات الحقل الساطع (الشكل 3G).
- قسم تصحيحات الصور المسماة بشكل عشوائي إلى مجموعة تدريب ومجموعة اختبار.
- تدريب شبكة عصبية تلافيفية عميقة ، ResNeSt38 لتعلم تصنيف هذه التصحيحات. قم بتقييم ResNeSt المدرب على مجموعة الاختبار للتأكد من أن النموذج يوضح دقة تصنيف عالية. قم بتشغيل "train.py" و "test.py" للتدريب والاختبار ، على التوالي (الشكل 3H).
- استخدم Grad-CAM36 لتسليط الضوء على المناطق التي تساهم بشكل أكبر في استنتاج ResNeSt ، ممثلة بخرائط التمثيل اللوني. قم بتجميع خرائط الحرارة ثنائية (باستخدام عتبة ، على سبيل المثال ، 10٪ × كحد أقصى) للحصول على مناطق تكلفة النقرة الملتزمة ب CM المتوقعة ، والتي تسمى مناطق CPC المعترف بها في الصورة (IR-CPC).
- قارن مناطق IR-CPC مع الأقنعة المشروحة يدويا على مجموعة الاختبار باستخدام الدقة ودرجة F1 والدقة والاستدعاء والنوعية والتقاطع فوق الاتحاد (IoU). قارن نسبة مناطق IR-CPC مع مؤشرات كفاءة التمايز (المحسوبة من صور مضان cTnT النهائية) باستخدام معامل ارتباط بيرسون. قم بتشغيل "./Evaluation/index_calculate.m" و "./Evaluation/Pearson_correlation.m" لتقييم الأداء (الشكل 3I).
- تطبيق النموذج المدرب للتعرف على التكلفة الفولرة للدفعات الجديدة في المرحلة الثانية. قم بقص صور الحقول الساطعة بالكامل إلى تصحيحات ، وقم بتمرير التصحيحات إلى ResNeSt المدرب ، واحصل على مناطق IR-CPC على مستوى التصحيح ، والتي يتم دمجها بعد ذلك لإنتاج مناطق IR-CPC الجيدة بالكامل. احسب جزء مناطق الأشعة تحت الحمراء - CPC كمؤشر لكفاءة التمايز النهائية. تنقية IR-CPCs لتقليل تلوث الخلايا (الموضح أدناه).
ملاحظة: نظرا لأن نموذج التعرف على تكلفة المنافسة للبراءات يمكن أن يقوم بتقييم مبكر لكفاءة التمايز النهائي ، فإنه يمكن أن يكون أيضا بمثابة قراءة عالية الإنتاجية لفحص الجزيئات الصغيرة التي تساعد على عملية التمايز ، مما قد يزيد من تحسين بروتوكولالتمايز 35.
- التنقية الانتقائية للمنطقة للتكلفة النقرة المعترف بها بالصور
ملاحظة: تعتمد تجارب التنشيط الضوئي على مجهر مضان مقلوب يتم تسهيله بمرحلة آلية.- وفقا لمناطق IR-CPC التي تنبأ بها النموذج المدرب ، تخلص من الآبار التي لا يمكنها توليد أي IR-CPCs. لتنقية CPCs الملتزمة ب CM ، استخدم مسبارا غير سام للخلايا ، Dual-Activaable Cell Tracker 1 (DACT-1) لتسمية المنطقة غير CPC39 بشكل انتقائي
- قم بإذابة DACT-1 (10 ملم) في DMSO وتخزينه عند -20 درجة مئوية. احتضان خلايا اليوم 6 مع 1 ميكرومتر DACT-1 في وسط التمايز CM لمدة 30 دقيقة عند 37 درجة مئوية.
- افحص الخلايا الحية تحت المجهر وحدد منطقة الاهتمام (ROI) كمضلعات في MATLAB. ارسم خطوطا متوازية بتباعد 20 ميكرومتر داخل المضلع واحسب إحداثيات نقاط التقاطع.
- اضبط المسار المتحرك للمرحلة الآلية وفقا لخطوط التتبع ، ثم تتحرك المرحلة بسرعة 0.12 مم / ثانية بحيث يمكن لخط ليزر 405 نانومتر (قطره 20 ميكرومتر) يتم تجميعه على المسرح مسح عائد الاستثمار عالميا لتنشيط ضوئي غير CPCs بشكل مقيد. بعد التشعيع ، اكتشف الخلايا المسماة DACT-1 باستخدام خط ليزر 561 نانومتر.
- قم بفصل الخلايا باستخدام 0.05٪ Trypsin-EDTA (مخفف بواسطة PBS) عند 37 درجة مئوية لمدة 5-7 دقائق ، وقم بهز الطبق برفق في حاضنة لمدة دقيقتين. قم بتصفية الخلايا من خلال مصفاة خلية 40 ميكرومتر وجهاز طرد مركزي عند 500 × جم لمدة 3 دقائق. أعد تعليق الخلايا في 0.5٪ BSA واحتفظ بالأنبوب على الجليد حتى الفرز. قم أيضا بإعداد عينة غير مشعة كعنصر تحكم سلبي للفرز.
- ابدأ تشغيل نظام فرز قياس التدفق الخلوي وقم بإعداد برنامج الفرز. اختر ليزر الطول الموجي 561 نانومتر واستخدم مرشح ممر النطاق 610/20 لفرز DACT1 +. لكل عينة ، استخدم أداة بوابة متعددة الأضلاع لوضع دائرة حول مجموعة الخلايا في مخطط ثنائي المتغير من التشتت الأمامي (FSC) والمبعثر الجانبي (SSC) لتحديد الخلية الفردية. بعد ذلك ، قم بتشغيل عنصر التحكم السلبي لتأكيد مضان الخلفية ، وقم بتشغيل العينة المشعة بشكل انتقائي في المنطقة ، وارسم البوابة لمجموعة DACT1 + .
- جمع الخلايا المصنفة وإعادة تعليقها في وسط صيانة CM مع 10٪ FBS و 5 ميكرومتر Y27632 ، وزرع الخلايا المصنفة في صفيحة 96 بئرا مطلية ب Matrigel.
- في اليوم التالي ، قم بتغيير وسيط الصيانة المتوسط إلى CM وانتظر حصاد CMs عالية النقاء.
- استراتيجية غسل الأموال في المرحلة الثالثة: الاعتراف بأعضاء هيئة الإدارة
مجموعة البيانات: أزواج من صور المجال الساطع في اليوم 12 وصور التألق النهائي cTnT
الموديل: pix2pix37- قم بإعداد مجموعة بيانات تتكون من صور المجال الساطع ل CMs وصور مضان cTnT المقابلة. لزيادة التنوع ، تأكد من أن مجموعة البيانات تتضمن آبار ذات كفاءات تمايز مختلفة وخطوط خلوية.
- قسم مجموعة البيانات بشكل عشوائي إلى مجموعة تدريب ومجموعة اختبار. قم بتبديل دليل العمل إلى "stage_III". احفظ صور الحقل الساطع وصور التألق cTnT المقابلة في "./pix2pix/datasets/(CM|CM_new_cell_lines)/A/(train|test)/*.png" و ".../B/(train|test)/*.png" ، على التوالي. قم بتشغيل الأمر التالي:
CD Pix2pix / مجموعات البيانات
python combine_A_and_B.py --- fold_A ./CM / A --fold_B ./CM / B --fold_AB ./CM/ --no_multiprocessing
قرص مضغوط .. - قم بتدريب pix2pix موديل37 على مجموعة التدريب. قم بتشغيل الأمر التالي (الشكل 3J):
python train.py --dataroot ./datasets / CM --name brightfield2fluorescence --model pix2pix --input_nc 1 --output_nc 1 --load_size 1536 --crop_size 256 --lr 2e-4 --n_epochs 1000 --n_epochs_decay 0 --instance norm --netD n_layers --n_layers_D 1 --batch_size 16 --direction AtoB --save_epoch_freq 100 --dataset_mode محاذاة --use_resize_conv --seed 1234 - قم بتطبيق نموذج pix2pix المدرب على مجموعة الاختبار. قم بتشغيل الأمر التالي:
python test.py --dataroot ./datasets / CM --name brightfield2fluorescence --model pix2pix --direction AtoB --input_nc 1 --output_nc 1 --load_size 1536 --crop_size 1536 --use_resize_conv --eval --num_test 1000 - قارن الصور الفلورية التي تنبأ بها النموذج مع الصور الحقيقية (أي التي تم الحصول عليها تجريبيا). على مستوى البكسل ، قارن الشدة المتوقعة والشدة الحقيقية باستخدام معامل ارتباط بيرسون. بالإضافة إلى ذلك ، على مستوى الصورة ، قارن بين مؤشرات كفاءة التمايز المتوقعة مع المؤشرات الحقيقية باستخدام معامل ارتباط بيرسون. قم بتشغيل "stage_III/evaluation/pixel_correlation.m" و "image_correlation.ipynb" لهذه المقارنات (الشكل 3K ، L).
- تطبيق النموذج المدرب للتعرف على CMs على دفعات جديدة في المرحلة III. قم بتمرير صور الحقل الساطع الجديدة ل CMs إلى نموذج pix2pix المدرب واحصل على تنبؤ داخل السيليكو للصور الفلورية وكفاءة التمايز.
ملاحظة: لتقليل تلوث الخلايا ، فإن فرز الخلايا المنشطة ضوئيا المستند إلى DACT-1 قابل للتطبيق أيضا لتنقية CMs المعترف بها بواسطة النموذج.
النتائج
استنادا إلى التصوير الساطع والتعلم الآلي ، يمكن مراقبة عملية التمايز الشاملة وتحسينها بذكاء. في مرحلة PSC ، قمنا بتطوير نموذج ML يمكنه التنبؤ بكفاءة التمايز النهائية وفقا للسمات المورفولوجية لمستعمرات PSC الأولية ، لتحديد النقطة الزمنية الأنسب أو الأنسب لبدء التمايز (
Discussion
هنا ، وصفنا بروتوكولا مفصلا للتغلب على إحدى المشكلات الرئيسية في تطبيق PSC الحالي وترجمته - التباين في تمايز الخلايا. من خلال تسخير تصوير المجال الساطع للخلايا الحية والتعلم الآلي ، قمنا بتحسين تمايز PSC بشكل متكرر لتحقيق كفاءة عالية باستمرار عبر خطوط الخلايا والدفعات. ومع...
Disclosures
يقوم يانغ تشاو وجو تشانغ وشياتشون يانغ وياو وانغ ودايتشاو تشن بتقديم براءة اختراع لاستراتيجية التمايز PSC المذكورة في هذه الورقة (202210525166.X).
Acknowledgements
نشكر Qiushi Sun و Yao Wang و Yu Xia و Jinyu Yang و Chang Lin و Zimu Cen و Dongdong Liang و Rong Wei و Ze Xu و Guangyin Xi و Gang Xue و Can Ye و Li-Peng Wang و Peng Zou و Shi-Qiang Wang و Pablo Rivera-Fuentes و Salome Püntener و Zhixing Chen و Yi Liu و Jue Zhang ، على وضع الأساس لهذه الاستراتيجية. تم دعم هذا العمل من قبل البرنامج الوطني للبحث والتطوير الرئيسي في الصين (2018YFA0800504 ، 2019YFA0110000) ومشروع التجارب الطبية الفضائية لبرنامج الفضاء المأهول الصيني (HYZHXM01020) إلى يانغ تشاو. تم إنشاء الشكل 1 باستخدام BioRender.com.
Materials
| Name | Company | Catalog Number | Comments |
| 0.25% Trypsin-EDTA | Gibco | 25200056 | Diluted digests were used for CPC and CM digestion |
| 4% Paraformaldehyde in PBS | KeyGEN BioTECH | KGIHC016 | |
| 6-well Cell Culture Plate | NEST | 703001 | |
| 96-well Cell Culture Plate | NEST | 701001 | |
| B27 Supplement | Gibco | 17504044 | |
| B27 Supplement Minus Insulin | Gibco | A1895601 | |
| Bovine serum albumin (BSA) | GPC BIOTECH | AA904-100G | |
| Celldiscoverer 7 | Zeiss | Instruments used to take bright-field images throughout differentiation and final cTnT images | |
| CHIR99021 | Selleck | S1263 | |
| DMEM/F12 | Gibco | 12634010 | |
| Donkey anti-Mouse IgG (H+L) Highly Cross-Adsorbed Secondary Antibody, Alexa Fluor 488 | Thermo | A-21202 | Secondary Antibody |
| FACSAria III | BD Biosciences | Flow cytometry sorter | |
| Fetal Bovine Serum (FBS) | VISTECH | SE100-B | |
| Hoechst 33342 | YEASEN | 40732ES03 | |
| Human Pluripotent Stem Cell Chemical-defined Medium | Cauliscell Inc | 400105 | Basal medium of PSC preparation medium |
| iPS-18 | TaKaRa | Y00300 | |
| iPS-B1 | Cellapy | CA4025106 | |
| iPS-F | Nuwacell | RC01001-B | |
| iPS-M | Nuwacell | RC01001-A | |
| IWR1-1-endo | Selleck | S7086 | IWR1 |
| Jupyter Notebook | N/A | Version 6.4.0 | https://jupyter.org/ |
| MATLAB | MathWorks | Version R2020a | Software for scientific computation and image annotation |
| Matrigel Matrix | Corning | 354230 | Matrigel |
| Mouse monoclonal IgG1 anti-cTnT | Thermo | MA5-12960 | cTnT primary antibody |
| Normal Donkey Serum | Jackson | 017-000-121 | |
| ORCA-Flash 4.0 V3 digital CMOS camera | Hamamatsu | C13440-20CU | The digital camera assembled on Celldiscoverer7 |
| PBS | NEB | 21-040-CVR | |
| Penicillin-Streptomycin | Gibco | 15140-122 | |
| Pluripotency Growth Mater 1 basal medium | Cellapy | CA1007500-1 | Basal medium of PSC culture medium |
| Pluripotency Growth Mater 1 supplement | Cellapy | CA1007500-2 | Supplement of PSC culture medium |
| Prism | Graphpad | Version 8/9 | Statistical software for statistical analysis and plotting |
| Python | N/A | version 3.6 | Python 3 environment for scientific computation, with packages pytorch (1.9.0), numpy, scipy, pandas, visdom, scikit-learn, scikit-image, opencv-python, and matplotlib software for scientific computation and image annotation. |
| RPMI 1640 | Gibco | 11875176 | |
| Supplement hPSC-CDM (500x) | Cauliscell Inc | 00015 | Supplement of PSC preparation medium |
| TiE | Nikon | An inverted fluorescence microscope (with modification) for region-selevtive purification | |
| Triton X-100 | Amresco | 9002-93-1 | |
| Versene Solution | Thermo | 15040066 | EDTA solution for PSC digestion |
| Y27632 | Selleck | S6390 | |
| Zen | Zeiss | Version 3.1 | A supporting software of Celldiscoverer7 for image acquisition, processing and analysis |
References
- Yoshida, Y., Yamanaka, S. Induced pluripotent stem cells 10 years later: for cardiac applications. Circ Res. 120 (12), 1958-1968 (2017).
- Shi, Y., Inoue, H., Wu, J. C., Yamanaka, S. Induced pluripotent stem cell technology: a decade of progress. Nat Rev Drug Discov. 16 (2), 115-130 (2017).
- Takahashi, K., et al. Induction of pluripotent stem cells from adult human fibroblasts by defined factors. Cell. 131 (5), 861-872 (2007).
- Burridge, P. W., et al. Chemically defined generation of human cardiomyocytes. Nat Methods. 11 (8), 855-860 (2014).
- Mordwinkin, N. M., Burridge, P. W., Wu, J. C. A review of human pluripotent stem cell-derived cardiomyocytes for high-throughput drug discovery, cardiotoxicity screening, and publication standards. J Cardiovasc Transl Res. 6 (1), 22-30 (2013).
- Vanden Berg, C. W., Elliott, D. A., Braam, S. R., Mummery, C. L., Davis, R. P. Differentiation of human pluripotent stem cells to cardiomyocytes under defined conditions. Methods Mol Biol. 1353, 163-180 (2016).
- Burridge, P. W., Holmström, A., Wu, J. C. Chemically defined culture and cardiomyocyte differentiation of human pluripotent stem cells. Curr Protoc Hum Genet. 87, 1-15 (2015).
- Li, J., et al. Human pluripotent stem cell-derived cardiac tissue-like constructs for repairing the infarcted myocardium. Stem Cell Rep. 9 (5), 1546-1559 (2017).
- Smith, A. S. T., Macadangdang, J., Leung, W., Laflamme, M. A., Kim, D. -. H. Human iPSC-derived cardiomyocytes and tissue engineering strategies for disease modeling and drug screening. Biotechnol Adv. 35 (1), 77-94 (2017).
- Sharma, A., et al. Use of human induced pluripotent stem cell-derived cardiomyocytes to assess drug cardiotoxicity. Nat Protoc. 13 (12), 3018-3041 (2018).
- Mehta, A., et al. Identification of a targeted and testable antiarrhythmic therapy for long-QT syndrome type 2 using a patient-specific cellular model. Eur Heart J. 39 (16), 1446-1455 (2018).
- Cahan, P., Daley, G. Q. Origins and implications of pluripotent stem cell variability and heterogeneity. Nat Rev Mol Cell Biol. 14 (6), 357-368 (2013).
- Kim, K., et al. Epigenetic memory in induced pluripotent stem cells. Nature. 467 (7313), 285-290 (2010).
- Rouhani, F., et al. Genetic background drives transcriptional variation in human induced pluripotent stem cells. PLoS Genet. 10 (6), e1004432 (2014).
- Kim, K., et al. Donor cell type can influence the epigenome and differentiation potential of human induced pluripotent stem cells. Nat Biotechnol. 29 (12), 1117-1119 (2011).
- Garitaonandia, I., et al. Increased risk of genetic and epigenetic instability in human embryonic stem cells associated with specific culture conditions. PLoS One. 10 (2), e0118307 (2015).
- Engler, A. J., Sen, S., Sweeney, H. L., Discher, D. E. Matrix elasticity directs stem cell lineage specification. Cell. 126 (4), 677-689 (2006).
- Matoba, N., Yamashita, T., Takayama, K., Sakurai, F., Mizuguchi, H. Optimal human iPS cell culture method for efficient hepatic differentiation. Differentiation. 104, 13-21 (2018).
- Volpato, V., et al. Reproducibility of molecular phenotypes after long-term differentiation to human iPSC-derived neurons: A multi-site omics study. Stem Cell Rep. 11 (4), 897-911 (2018).
- Anderson, N. C., et al. Balancing serendipity and reproducibility: Pluripotent stem cells as experimental systems for intellectual and developmental disorders. Stem Cell Rep. 16 (6), 1446-1457 (2021).
- Sepac, A., et al. Comparison of cardiomyogenic potential among human ESC and iPSC lines. Cell Transplant. 21 (11), 2523-2530 (2012).
- Laco, F., et al. Unraveling the inconsistencies of cardiac differentiation efficiency induced by the GSK3β inhibitor CHIR99021 in human pluripotent stem cells. Stem Cell Rep. 10 (6), 1851-1866 (2018).
- Volpato, V., Webber, C. Addressing variability in iPSC-derived models of human disease: guidelines to promote reproducibility. Dis Model Mech. 13 (1), 042317 (2020).
- Hu, B. -. Y., et al. Neural differentiation of human induced pluripotent stem cells follows developmental principles but with variable potency. Proc Natl Acad Sci USA. 107 (9), 4335-4340 (2010).
- Huo, J., et al. Evaluation of batch variations in induced pluripotent stem cell-derived human cardiomyocytes from 2 major suppliers. Toxicol Sci. 156 (1), 25-38 (2017).
- Rostam, H. M., Reynolds, P. M., Alexander, M. R., Gadegaard, N., Ghaemmaghami, A. M. Image based machine learning for identification of macrophage subsets. Sci Rep. 7 (1), 3521 (2017).
- Christiansen, E. M., et al. In silico labeling: Predicting fluorescent labels in unlabeled images. Cell. 173 (3), 792-803 (2018).
- Buggenthin, F., et al. Prospective identification of hematopoietic lineage choice by deep learning. Nat Methods. 14 (4), 403-406 (2017).
- Smith, Z. D., Nachman, I., Regev, A., Meissner, A. Dynamic single-cell imaging of direct reprogramming reveals an early specifying event. Nat Biotechnol. 28 (5), 521-526 (2010).
- Fan, K., Zhang, S., Zhang, Y., Lu, J., Holcombe, M., Zhang, X. A machine learning assisted, label-free, non-invasive approach for somatic reprogramming in induced pluripotent stem cell colony formation detection and prediction. Sci Rep. 7 (1), 13496 (2017).
- Kusumoto, D., et al. Automated deep learning-based system to identify endothelial cells derived from induced pluripotent stem cells. Stem Cell Rep. 10 (6), 1687-1695 (2018).
- Zhu, Y., et al. Deep learning-based predictive identification of neural stem cell differentiation. Nat Commun. 12 (1), 2614 (2021).
- Qian, T., et al. Label-free imaging for quality control of cardiomyocyte differentiation. Nat Commun. 12 (1), 4580 (2021).
- Ounkomol, C., Seshamani, S., Maleckar, M. M., Collman, F., Johnson, G. R. Label-free prediction of three-dimensional fluorescence images from transmitted-light microscopy. Nat Methods. 15 (11), 917-920 (2018).
- Yang, X., et al. A live-cell image-based machine learning strategy for reducing variability in PSC differentiation systems. Cell Discovery. 9 (1), 53 (2023).
- Selvaraju, R. R., et al. Grad-CAM: Visual explanations from deep networks via gradient-based localization. Proc IEEE Int Conf Computer Vision (ICCV). , 618-626 (2017).
- Isola, P., Zhu, J. -. Y., Zhou, T., Efros, A. A. Image-to-image translation with conditional adversarial networks. Proc IEEE Conf Computer Vision Pattern Recognition (CVPR). , 5967-5976 (2017).
- Zhang, H., et al. ResNeSt: Split-attention networks. Proc IEEE/CVF Conf Computer Vision Pattern Recognition Workshops (CVPRW). , 2735-2745 (2022).
- Halabi, E. A., et al. Dual-activatable cell tracker for controlled and prolonged single-cell labeling). ACS Chem. Biol. 15 (6), 1613-1620 (2020).
- Hannan, N. R. F., Segeritz, C. -. P., Touboul, T., Vallier, L. Production of hepatocyte-like cells from human pluripotent stem cells. Nat Protoc. 8 (2), 430-437 (2013).
- Zhang, H., Shen, M., Wu, J. C. Generation of quiescent cardiac fibroblasts derived from human induced pluripotent stem cells. Methods Mol Biol. 2454, 109-115 (2022).
- Harding, M. J., McGraw, H. F., Nechiporuk, A. The roles and regulation of multicellular rosette structures during morphogenesis. Development. 141 (13), 2549-2558 (2014).
- Najm, F. J., et al. Rapid and robust generation of functional oligodendrocyte progenitor cells from epiblast stem cells. Nat Methods. 8 (11), 957-962 (2011).
- Hogrebe, N. J., Maxwell, K. G., Augsornworawat, P., Millman, J. R. Generation of insulin-producing pancreatic β cells from multiple human stem cell lines. Nat Protoc. 16 (9), 4109-4143 (2021).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved