A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
אסטרטגיית למידת מכונה מבוססת תמונה של תאים חיים לניטור התמיינות תאי גזע פלוריפוטנטית
* These authors contributed equally
In This Article
Summary
מערכות התמיינות תאים פלוריפוטנטיות זמינות (PSC) לתפקוד מעוכבות כיום על ידי בעיות של שונות חמורה מקו לקו ואצווה לאצווה. כאן, תוך שימוש בהתמיינות לבבית כדוגמה העיקרית, אנו מציגים פרוטוקול לניטור חכם ומווסת את תהליך התמיינות PSC המבוסס על למידת מכונה מבוססת תמונה.
Abstract
טכנולוגיות תאי גזע פלוריפוטנטיות (PSC) נמצאות בשימוש נרחב בגילוי תרופות, מידול מחלות ורפואה רגנרטיבית. עם זאת, מערכות התמיינות תאים זמינות PSC לתפקוד מעוכבות על ידי בעיות של שונות חמורה מקו לקו ואצווה לאצווה. לכן חשוב לשלוט במדויק בהתמיינות התאים בזמן אמת. בפרוטוקול זה, אנו מתארים אסטרטגיה לא פולשנית וחכמה המתגברת על השונות בהתמיינות תאים באמצעות למידת מכונה מבוססת תמונה בשדה בהיר. אם ניקח לדוגמה התמיינות PSC לקרדיומיוציטים, מתודולוגיה זו מספקת מידע מפורט לבקרה על מצב PSC הראשוני, הערכה מוקדמת והתערבות בתנאי התמיינות וסילוק זיהום התאים המובחן באופן שגוי, יחד עם מימוש התמיינות באיכות גבוהה באופן עקבי מתאי PSC לתאים פונקציונליים. באופן עקרוני, ניתן להרחיב אסטרטגיה זו למערכות אחרות של התמיינות תאים או תכנות מחדש עם שלבים מרובים לתמיכה בייצור תאים, כמו גם כדי לקדם את הבנתנו את המנגנונים במהלך המרת גורל התא.
Introduction
תאי גזע פלוריפוטנטיים (PSC) הם בעלי יכולת יוצאת דופן להתמיין לסוגים רבים של תאים במבחנה. תאים פונקציונליים ממוינים אלה יכולים לשמש לטיפול תאי, מידול מחלות ופיתוח תרופות, כולם בעלי ערך למחקר או ליישומים קליניים 1,2,3. לדוגמה, מגוון שיטות פותחו כדי להבדיל PSCs לתוך cardiomyocytes (CMs)4,5,6,7. CMs אלה יכולים להיות מיושמים עבור בדיקות cardiotoxicity של תרופות, מודלים של מחלות לב, השתלת תאים 8,9,10,11.
עם זאת, ההמרה מ-PSC לתאים המתמיינים הסופיים היא תהליך הדרגתי, והפרעות מרובות במהלך תהליך ההתמיינות עלולות להוביל תאים לגורלות תאים שונים. רקע גנטי שונה וסימנים אפיגנטיים שונים של קווי PSC משפיעים על פוטנציאל ההתמיינות לשושלת ספציפית 12,13,14,15; מספר מעברי PSC ומוטציות גנטיות מצטברות הם גם מקורות להטרוגניות PSC; הבדלים בפעולות הניסוי המופעלות על ידי נסיינים שונים יכולים גם להוביל לתוצאות התמיינות שונות לחלוטין 16,17,18,19,20. לכן, כיום אחת הבעיות העיקריות בייצור תאים שמקורם ב-PSC היא חוסר היציבות בין קווי תאים ואצוות 21,22,23,24,25. חוסר יציבות בהתמיינות PSC מוביל לעתים קרובות לניסויים חוזרים ונשנים, הגוזלים משאבי זמן ועבודה משמעותיים. כדי להתמודד עם בעיה זו, חיוני לפתח אסטרטגיה שתמזער את השונות בין קווי תאים ואצוות, ובכך תשפר את היציבות והחוסן של ההתמיינות.
לאחרונה, התקדמות במיקרוסקופ ברזולוציה גבוהה ולמידת מכונה (ML) הקלה על היישום של ניתוח תמונה כמותי מבוסס ML בביולוגיה של התא, מה שמאפשר להשתמש במידע רב ערך בתכונות דימות התא 26,27,28,29,30,31,32,33,34. בעבודתנו הקודמת הצענו אסטרטגיית ML מבוססת תמונה של תאים חיים כדי לנטר ולהתערב במצב התמיינות PSC בזמן אמת כדי לשפר את היציבות והיעילות של התמיינות PSC (איור 1)35. אם ניקח לדוגמה התמיינות PSC לקרדיומיוציטים, הערכנו את מצב PSC הראשוני באמצעות מודלים של יער אקראי, חזינו את מצב ההתמיינות האופטימלי באמצעות מודלים של רגרסיה לוגיסטית, וזיהינו תאים ממוינים בהצלחה באמצעות Grad-CAM36 ו-pix2pix37 מבוססי למידה עמוקה. מודלים של ML למדו לזהות שושלות תאים ממגוון תכונות מורפולוגיות של שדה בהיר, כולל תכונות על שטח, היקף, קמור, מוצקות, בהירות, מהירות נעה ותכונות מרומזות אחרות שחולצו על ידי רשתות עצביות קונבולוציוניות עמוקות. בהתבסס על הסקת מסקנות ממודלים מבוססים אלה של ML, הבנו שליטה במצב PSC הראשוני, הערכה מוקדמת והתערבות בתנאי התמיינות וחיסול זיהום התאים הלא ממוין, יחד עם מתן אפנון מקיף ומדויק של תהליך התמיינות הלב. כאן אנו מספקים פרוטוקול שלב אחר שלב לפיתוח האסטרטגיה.
Access restricted. Please log in or start a trial to view this content.
Protocol
1. התמיינות תאים ואפיון
- הכנת ריאגנטים תרבותיים וצלחות תרבות
- הכן מדיום תרבית PSC על ידי הוספת 2 מ"ל של תוספת ו 0.2% פניצילין-סטרפטומיצין ל 48 מ"ל של מדיום בסיסי. Aliquot ולאחסן את התוסף ב -20 °C. יש לאחסן מדיום זה בטמפרטורה של 4°C למשך עד 4 שבועות.
- הכינו מדיום להכנת PSC על ידי הוספת 1 מ"ל תוסף ו-0.2% פניצילין-סטרפטומיצין ל-500 מ"ל מדיום בסיסי. בעת השימוש, חממו מראש את המדיום לשימוש חד פעמי, ואחסנו את המדיום בטמפרטורה של 4°C למשך עד 3 שבועות.
- הכן מדיום התמיינות CM על ידי הוספת 1x B27 תוספת פחות אינסולין ו 1% פניצילין-סטרפטומיצין לתוך RPMI 1640. הכינו מדיום תחזוקת CM על ידי הוספת תוסף B27 1x ו-1% פניצילין-סטרפטומיצין ל-RPMI 1640. אחסנו את שני סוגי המדיום הללו בטמפרטורה של 4°C למשך שבועיים.
- השקיעו את המטריגל בקרח ושמרו אותו על 4 מעלות צלזיוס למשך הלילה. ודא כי Matrigel מופשר לחלוטין; לאחר מכן, aliquot לתוך 1 מ"ל aliquots מאוחסן ב -80 ° C לשימוש מאוחר יותר, ולהוסיף 1 מ"ל של Matrigel לתוך 49 מ"ל של DMEM / F12 ב 4 ° C כדי להכין את פתרון העבודה Matrigel. הוסיפו 850 מיקרוליטר של תמיסת עבודה זו של מטריג'ל לכל באר של צלחת תרבית בעלת 6 בארות, ודגרו על הצלחת בטמפרטורה של 37°C למשך 30 דקות לפחות. עבור צלחת תרבית 96 בארות המשמשת בידול CM, להוסיף 35 μL של פתרון עבודה Matrigel לכל באר.
הערה: שמור את מטריג'ל על קרח בכל עת; קצות פיפטה צינורות צריך להיות precooled ב 4 ° C. פתרון עבודה Matrigel יכול להיות מאוחסן ב 4 ° C במשך 2 שבועות. היזהרו מהימנעות מבועות בעת שימוש בתמיסת העבודה של מטריג'ל.
- תחזוקה ומעבר PSC
- שמרו על PSCs בצלחות תרבית של 6 בארות. אשרו את צפיפות התאים תחת מיקרוסקופ והתכוננו למעבר כאשר המפגש מגיע ל~70% (איור 2A).
- השתמש ב- EDTA כדי לעכל את התאים למעבר. יש לחמם את מדיום PSC, PBS ו-EDTA באמבט מים של 37°C, ולהוסיף Y27632 בתווך לריכוז סופי של 5 מיקרומטר.
- לשטוף את PSCs עם 1 מ"ל של PBS, להוסיף 1 מ"ל של EDTA לכל באר, לדגור את הצלחת באינקובטור 37 מעלות צלזיוס במשך 3 דקות.
- שאפו את ה-EDTA לפני שהתאים יתנתקו מצלחת התרבית, ולאחר מכן השתמשו ב-1 מ"ל של מדיום תרבית PSC כדי לשטוף בעדינות את תחתית הבאר 3-4x להשעיה מחדש של התאים.
הערה: זה מאפשר לנו לדלג על שלב הצנטריפוגה ולהפחית את הנזק לתאים בפעולות מעבר. - שאפו את ה-DMEM/F12 מבאר מצופה מטריג'ל של צלחת תרבית בעלת 6 בארות והוסיפו 2 מ"ל של מדיום תרבית PSC טרי לכל באר (נוסף 27632 יו'). הוסף את תרחיף התא לבארות עם יחס פיצול של 1:6 עד 1:12. מניחים את הצלחת באינקובטור של 37 מעלות צלזיוס עם 5% CO2.
- החליפו את מדיום תרבות PSC (ללא 27632 יו') 12 עד 24 שעות לאחר המעבר; לאחר מכן, לשנות את המדיום ולבדוק את מצב התא ואת מפגש כל יום.
- התמיינות לבבית של PSCs
- לבידול CM, זרעו PSC לצלחת תרבית של 96 בארות במדיום הכנת PSC (נוסף 27632 יו'). לבידול CM, בצע את אותם השלבים לעיל (שלבים 1.2.2-1.2.6) כדי לזרוע PSCs לתוך צלחת תרבית של 96 בארות במדיום הכנת PSC (Y27632 נוסף).
הערה: בשלב זה, משך הטיפול ב- EDTA יכול להיות מוארך כראוי (5 דקות או יותר) כדי לנתק מספיק PSCs כך שהשיבוטים יוכלו להיות קטנים יותר לאחר המעבר. המחקר שלנו הראה כי באותו אזור, מושבות קטנות יותר תורמות להתבדלות. - שלב I: כאשר PSCs מגיעים למפגש של 80-90%, החליפו את מדיום ההתמיינות מדיום ל-CM עם 2-20 מיקרומטר CHIR99021 (CHIR) (איור 2B). נקודת הזמן של מיתוג בינוני מכונה יום 0. לאחר טיפול CHIR של 24-48 שעות, יש לשנות את מדיום הבידול CM הבינוני לרענן.
הערה: CHIR הוא מעכב GSK3beta להשראת מזודרם. מינון ה-CHIR האופטימלי להתמיינות CM משתנה עם קווי תאים ואצוות שונות. לדוגמה, מינון CHIR אופטימלי הוא 8-10 מיקרומטר, 48 שעות עבור iPS-B1. כדי לאסוף תמונות מגוונות של שדות בהירים לאימון מודל ML, כאן ערכנו ריכוזי CHIR עבור כל קבוצה של ניסויים עבור יעילות התמיינות שונה. - שלב II: בשעה 72 שעות (יום 3), החלף את המדיום במדיום התמיינות CM בתוספת 5 מיקרומטר IWR1 ותרבית למשך 48 שעות (יום 5). שנה את המדיום עם מדיום בידול CM למשך 1-2 ימים (לימים 6-7). בשלב זה, PSCs מתמיינים לתאי אב לבביים (CPC).
- שלב III: החלפת המדיום במדיום תחזוקת CM והחלפת המדיום כל 3 ימים. ביום 10 או ביום 12, קצרו את ה-CM לניתוח נוסף.
- לבידול CM, זרעו PSC לצלחת תרבית של 96 בארות במדיום הכנת PSC (נוסף 27632 יו'). לבידול CM, בצע את אותם השלבים לעיל (שלבים 1.2.2-1.2.6) כדי לזרוע PSCs לתוך צלחת תרבית של 96 בארות במדיום הכנת PSC (Y27632 נוסף).
- צביעה אימונופלואורסצנטית של CM
- ביום 10 או ביום 12, שטפו את התאים עם PBS וקבעו אותם עם 4% פרפורמלדהיד ב-PBS למשך 15 דקות בטמפרטורת החדר. שטפו את CMs 2x עם PBS ואחסנו את הצלחת ב 4 °C עד ~ 1 שבוע.
הערה: Paraformaldehyde הוא מגיב רעיל עם ריח חריף. יש לפעול בתוך מכסה מנוע כימי ולהגן על העיניים והידיים. - בזמן הצביעה, יש לטפל בתאים בתמיסה מחדירה (0.1% Triton X-100 ב-PBS) למשך 15 דקות בטמפרטורת החדר; לאחר מכן, יש לדגור על תאים בתמיסת חסימה (0.1% Triton X-100 המכיל 3% סרום חמור ב-PBS) למשך 30 דקות בטמפרטורת החדר.
- יש לדגור על הדגימה עם נוגדן ראשוני מסוג Troponin T (cTnT) (1:300) מדולל בתמיסת חסימה למשך הלילה ב-4°C כדי לזהות את ה-CMs.
- לאסוף את הנוגדן העיקרי ולשטוף את התאים עם PBS 3x. דגרו על הדגימה עם נוגדנים משניים ב-PBS עם אלבומין בסרום בקר 1% למשך שעה אחת ב-37°C בסביבה חשוכה.
הערה: ניתן לעשות שימוש חוזר בנוגדן הראשוני שנאסף במשך 2-3 פעמים בשבוע אחד.
- לאסוף את הנוגדן העיקרי ולשטוף את התאים עם PBS 3x. דגרו על הדגימה עם נוגדנים משניים ב-PBS עם אלבומין בסרום בקר 1% למשך שעה אחת ב-37°C בסביבה חשוכה.
- הסר את הנוגדן המשני מהתאים ושטוף את התאים 3x עם PBS. השתמש Hoechst 33342 (1:1,000 ב PBS) כדי להכתים גרעינים במשך 5 דקות בטמפרטורת החדר. שטפו את התא 3x באמצעות PBS והוסיפו 100 μL של PBS לכל באר כדי למנוע התייבשות. אחסנו את הצלחת בטמפרטורה של 4°C במקום חשוך וקבלו תמונות פלורסנט תוך שבוע.
- ביום 10 או ביום 12, שטפו את התאים עם PBS וקבעו אותם עם 4% פרפורמלדהיד ב-PBS למשך 15 דקות בטמפרטורת החדר. שטפו את CMs 2x עם PBS ואחסנו את הצלחת ב 4 °C עד ~ 1 שבוע.
2. רכישת זרם תמונה לאורך כל תהליך הבידול
- הקמת תכנון ניסיוני לאיסוף תמונות
- השתמשו במיקרוסקופ אוטומטי התומך בתרביות תאים חיים ובהדמיה כדי לאסוף תמונות שדה בהיר של שלבים שונים בהתמיינות CM ובתמונות אימונופלואורסצנטיות cTnT של תוצאות התמיינות (איור 2A-E). השתמש בתוכנה התומכת במיקרוסקופ כדי לתכנן את תוכנית הניסוי ולשלוט במיקרוסקופ.
- פתח את התוכנה וצור עיצוב ניסיוני חדש. בחר עדשת 5x objective ועדשת צינור 2x להדמיה. בדוק את האפשרויות אריחים וערימת Z.
- הוסף ערוץ TL Brightfield להדמיית brightfield בתפריט Channels. הוסף ערוצי AF488 ו- H3342 להדמיית אימונופלואורסצנטיות. שנה את נתיב האור בתפריט הגדרת הדמיה להדמיה באמצעות מצלמת CMOS דיגיטלית חיצונית.
- פתח את התפריט מצב רכישה והגדר איגוד של 2 x 2 כדי להגדיל את יחס האות לרעש.
- פתחו את התפריט Z-stack ובחרו בדגם Center. קבעו את מספר הפרוסות ומרווחי הזמן בשעת הסריקה. בחרו 3-5 פרוסות במרווחים של 3-6 מיקרומטר לתמונות ממוקדות בניתוח.
- בחלון ניווט ואריחים, הגדר את אזורי האריחים לפי ספק והגדר 25 אריחים (5 עמודות x 5 שורות) לבאר אחת. בתפריט 'אריחים', בחרו בתבנית Multiwell 96 ב'ספק דוגמה', וקבעו חפיפה של 5%-15% באזורים סמוכים.
- אם יש צורך בהדמיה רציפה, סמן את האפשרותT IME series והגדר את משך הזמן ואת מרווח הזמן בתפריט סידרת זמן. לאיסוף זרם התמונה בשלב I, קבעו משך של 12 שעות ללא מרווחי זמן.
- רכישת תמונות שדה בהיר ואימונופלואורסצנטי
- לקבלת תמונות, הכניסו תחילה את צלחת תרבית התאים למגש הדגימה, וטענו את הדגימה בתוך המיקרוסקופ. בחר את תבנית Multiwell 96 שתתאים לעיצוב הניסוי. אם הדגימה מורכבת מתאים חיים, פתח את מערכת החימום ואת משאבת CO2 כדי לשמור על המצב המתאים לתרבית (37 ° C, 5% CO2).
- פתח את פרוייקט הניסוי המוגדר מראש ושמור אותו כפרוייקט חדש. פתח את תפריט אריחים וכייל את מיקום הצלחת באופן ידני. בחלון ניווט ואריחים, בחר את הבארות הדרושות ולחץ על צור כדי לבנות אזורי אריחים עבור בארות אלה.
- לחץ על אמת אזורי אריחים בתפריט אריחים והפעל מיקוד אוטומטי כדי לאמת את כל הבארות. לאחר מכן, תקן ידנית את המיקוד של כל באר מתחת לשדה הבהיר על ידי גלילה של גלגל העכבר.
- סמנו את הערוצים הדרושים והגדירו את זמני החשיפה בתפריט 'ערוצים'. זמני החשיפה המומלצים הם 1.625 אלפיות השנייה עבור Bright, 100 אלפיות השנייה עבור H3342 ו- 550 אלפיות השנייה עבור AF488. בצעו הצמדה אחת כדי לבדוק את התמונות של כל הערוצים.
- לבסוף, לחץ על הלחצן התחל ניסוי והמתן להדמיה אוטומטית. בדרך כלל, לוקח ~ 1.2 שעות לסיים את הסריקה של צלחת תרבות שלמה של 96 בארות (96 בארות, שלוש שכבות, 25 אריחים לכל באר מתחת לשדה הבהיר). במסגרת Process, בחר Image Export, בחר בסוג הקובץ של תבנית TIFF או תבנית PNG לא דחוסה והחל. השתמשו בתמונות של 9 אריחים (3X3) במרכז הבאר לניתוח נוסף (איור 2A-E).
3. בניית אסטרטגיית ML מבוססת תמונה בכל שלב בתהליך הבידול
- הכנת תוכנה
- התקן את סביבת Python, עם חבילות pytorch (1.9.0), numpy, scipy, scikit-learn, pandas, visdom, scikit-image, opencv-python ו- matplotlib.
- התקן את Jupyter Notebook.
- התקינו את MATLAB.
- הורד את הקוד ואת ערכות הנתונים לדוגמה מ- https://github.com/zhaoyanglab/ML-for-PSC-differentiation.
הערה: הביצועים של ML תלויים באיכות של ערכות נתונים ובבחירת מודלים והיפר-פרמטרים. יישום הדוגמה אינו מובטח להיות אופטימלי לכל הנסיבות. מהנדסי ML צריכים להתאים אישית את הקוד ולהתאים את המודל ואת היפר-פרמטרים לצרכים הספציפיים שלהם.
- אסטרטגיית ML בשלב PSC: שליטה במדינות מושבת PSC ראשוניות
ערכת נתונים: זוגות של תמונות brightfield ב- 0 שעות (לפני טיפול CHR) ויעילות בידול סופית
דגם: יער אקראי- הכן ערכת נתונים המורכבת מתמונות שדה בהיר בשעה 0 (לפני טיפול CHR) ותמונות פלואורסצנטיות cTnT סופיות. כדי להגדיל את מגוון הפרופילים המורפולוגיים של תמונות 0 שעות בשדה בהיר, ודא שערכת הנתונים כוללת מושבות PSC מקווי תאים שונים, עם התמיינות המופעלת במרווח זמן לאחר המעבר. טפל בבארות במערך הנתונים עם תנאי CHIR אופטימליים בשלב I.
הערה: כדי להבטיח שהמושבות מטופלות בתנאי CHIR אופטימליים, ניתן לטפל בבארות באותה אצווה במנות CHIR עם טיטרציה. רק בארות עם תנאי CHIR בעלי יעילות גבוהה כלולות במערך הנתונים. - כמת את יעילות הבידול של כל באר על ידי מדד יעילות הדיפרנציאציה המחושב מתמונות הפלואורסצנטיות cTnT שלה, המוגדר על ידי
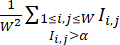 (כאשר
(כאשר 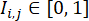 מייצג את עוצמת הפלואורסצנטיות במיקום הפיקסלים (i, j) בתמונה הפלואורסצנטית W × W, והסכום נלקח על כל מיקומי הפיקסלים בעוצמה מעל סף αמוגדר בדרך כלל על 0.5)., בתוך כל קו תא, חשב את מדד יעילות ההתמיינות המרבי על פני כל הבארות בקו תאים זה, ונמל את מדדי יעילות ההתמיינות שלהם בערך המרבי.
מייצג את עוצמת הפלואורסצנטיות במיקום הפיקסלים (i, j) בתמונה הפלואורסצנטית W × W, והסכום נלקח על כל מיקומי הפיקסלים בעוצמה מעל סף αמוגדר בדרך כלל על 0.5)., בתוך כל קו תא, חשב את מדד יעילות ההתמיינות המרבי על פני כל הבארות בקו תאים זה, ונמל את מדדי יעילות ההתמיינות שלהם בערך המרבי.
הערה: מכיוון שפוטנציאל ההתמיינות של כל קו תא שונה מטבעו, נורמליזציה כזו תדגיש יותר את התכונות המורפולוגיות של המושבה המשותפות בין קווי תאים שונים התורמות לתוצאת ההתמיינות. נורמליזציה אכן הופכת מושבות PSC מקווי תאים שונים לדומות וממתנת את ההטיה הנגרמת על ידי קווי תאים. - כמת את הפרופילים המורפולוגיים של תמונות שדה בהיר של 0 שעות באמצעות תכונות ממדיות גבוהות החושפות את תכונות צורת המושבה. למיתאר אזורי מושבה בתמונות שדה בהיר, חשבו את האנטרופיה המקומית (עם רדיוס r); פיקסלים עם אנטרופיה מקומית הגדולה מסף σ מזוהים על ידי תאים מכוסים על ידי תאים. השתמש בהגדרת פרמטר של r = 10 פיקסלים ו- σ = 3. העבר את ספריית העבודה ל- "./colony_control"; שמור את תמונות שדה בהיר, תמונות בינאריות של אזורי תאים ותמונות פלואורסצנטיות cTnT ב- "colony_control/ image_data/CD00-*/[brightfield|cell_region|ctnt]/S*.png"; ולהריץ את מחברת Jupyter "Compute_features.ipynb" כדי לחשב תכונות הקשורות לאזור המושבה, היקף, מוצקות, קמור, מעגליות ומרחקי מתאר צנטרואיד, בין היתר.
- חלקו באופן אקראי את מערך הנתונים לערכת אימונים ולערכת מבחנים, כאשר ערכת האימונים מהווה בין 50% ל-80% מכלל הנתונים. הפעל את הבלוק האחרון פצל את ערכת הנתונים במחברת Jupyter כדי לפצל את ערכת הנתונים (איור 3A).
- אמן מודל רגרסיה אקראית של יער בערכת האימונים כדי לחזות יעילות בידול מתכונות תמונה של 0 שעות בשדה בהיר. הפעל את מחברת Jupyter "Machine_learning.ipynb" כדי לאמן ולבדוק את המודל.
- הערך את מודל היער האקראי המאומן בקבוצת הבדיקה. אשרו שיעילות הבידול החזויה נמצאת בקורלציה גבוהה עם זו האמיתית באמצעות מקדם המתאם של פירסון (איור 3B).
הערה: מאחר שערכת הבדיקות אינה מעורבת בשלב האימון, עקביות גבוהה בין יעילות הבידול החזויה לבין יעילות הבידול האמיתית בקבוצת הבדיקות מצביעה על כך שהמודל יכול להכליל היטב על דגימות בלתי נראות. - קבל חשיבות תכונה ממודל היער האקראי המיומן ובחר 8 תכונות בעלות החשיבות הרבה ביותר. דמיינו את הקשר שלהם עם יעילות הבידול הסופית על ידי חלוקת מגוון התכונות לפחים וחישוב יעילות הבידול הממוצעת בכל פח. הפעל את מחברת Jupyter "Feature_importance.ipynb" כדי לבצע את התצוגה החזותית (איור 3C).
הערה: התצוגה החזותית עשויה לסייע בהבנת האופן שבו שינויים בצורת מושבת PSC תורמים לבידול. - יישם את המודל המאומן באצוות חדשות כדי לנטר מצבי מושבת PSC בזמן אמת. צלם את תמונת השדה הבהיר בכל שעה, העבר את התמונה למודל המיומן והשג את יעילות הבידול החזויה. ברגע שהיעילות החזויה עולה על סף (למשל, 50%), התחל את תהליך הבידול. אם היעילות החזויה לא הצליחה להגיע לסף, השליכו את הדגימות הללו.
הערה: במהלך שלב PSC, פוטנציאל הבידול של מושבות PSC יעלה תחילה ולאחר מכן יקטן. ניטור בזמן אמת מאפשר לנסיינים לזהות את נקודת הזמן המסייעת ביותר ליזום בידול.
- הכן ערכת נתונים המורכבת מתמונות שדה בהיר בשעה 0 (לפני טיפול CHR) ותמונות פלואורסצנטיות cTnT סופיות. כדי להגדיל את מגוון הפרופילים המורפולוגיים של תמונות 0 שעות בשדה בהיר, ודא שערכת הנתונים כוללת מושבות PSC מקווי תאים שונים, עם התמיינות המופעלת במרווח זמן לאחר המעבר. טפל בבארות במערך הנתונים עם תנאי CHIR אופטימליים בשלב I.
- אסטרטגיית ML בשלב I: הערכה מוקדמת והתאמה של מנות CHIR
ערכת נתונים: זוגות של זרמי תמונה בשדה בהיר ב- 0-12 שעות ותוויות ריכוז CHIR (נמוך/אופטימלי/גבוה)
מודל: רגרסיה לוגיסטית- הכן מערך נתונים המורכב מזרמי תמונה בהירים שלמים, שבהם התמונות נרכשות כל שעה בין 0 שעות ל -12 שעות. רשום את מנות ה-CHIR (כלומר, שילובים של ריכוזי CHIR ומשך) של כל באר. ודא שמערך הנתונים כולל אצוות ובארות שונות עם מנות CHIR עם טיטרציה (למשל, ריכוז = 2, 4, 6, 8, 10 ו- 12 מיקרומטר; משך = 24, 36 ו- 48 שעות) כדי להגדיל את המגוון. העבר את ספריית העבודה ל- "./stage_I"; שמור את התמונות כ- "./data/image/CD01-*/S*/T*.png", כאשר CD01-* (CD01-1, CD01-2, CD01-3, CD01-4) הוא שם האצווה, S* (S1, S2, ..., S96) הוא האינדקס של באר, ו- T*.png (T1.png, T2.png, ..., T10.png) הוא תמונת שדה בהיר מעובדת מראש של הבאר בשלב זמן שונה של זרם התמונה.
- בכל אצווה, חשב את אחוז תאי cTnT+ עבור כל באר, אשר מחושב על ידי חלוקת מספר תאי cTnT+ במספר הכולל של תאי Hoechst+ בתמונה הפלואורסצנטית. ממוצע האחוז של תאי cTnT+ על פני בארות מקבילות עם אותו תנאי מינון CHR. שמור את תנאי מינון CHIR ואת אחוז תאי cTnT+ עבור כל באר בקובץ csv "./data/image/CD01-*/CD01-?_exp_config.csv".
- בכל אצווה, קבע את טווח ריכוז ה-CHIR "הנמוך", ה"אופטימלי" וה"גבוה" תחת כל משך CHIR בהתאם לקריטריונים הבאים. ריכוזי CHIR עם אחוז ממוצע של תאי cTnT+ מעל סף (למשל, 20%) מזוהים כ"אופטימליים". ריכוזי CHIR מעבר לטווח האופטימלי מזוהים כ"נמוכים" או "גבוהים". תחת כל משך CHR, תייגו את הבארות כ"נמוכות", "אופטימליות" ו"גבוהות" בהתאם לריכוזי ה-CHIR המתאימים להן.
הערה: מבחינה אמפירית, קיים מתאם שלילי בין ריכוזי CHIR לבין משך הזמן של בארות בעלות יעילות גבוהה בתוך אצווה: עבור משך CHIR גבוה יותר, הריכוזים האופטימליים יורדים. במילים אחרות, האופטימליות של ריכוזי CHIR תלויה במשך CHR, ולכן תוויות הבארות שונות בין משכי CHIR שונים. - בכל אצווה, תחת כל משך CHR, יש לחשב ריכוז ΔCHIR עבור כל ריכוז c כדי לכמת את הסטייה שלו מהאופטימלי. ציין את טווח ריכוז ה- CHIR האופטימלי ב- [c1, c2], ולאחר מכן ריכוז ΔCHIR (c) = c - c1 (אם c < c1), 0 (אם c1 ≤ c ≤ c2), או c - c2 (אם c > c2).
- חלץ תכונות של זרמי התמונה בערכת הנתונים, וודא שהם משקפים את השינויים המורפולוגיים של תאים במהלך שלב I. כדי לחשב תכונות תמונה אודות אנטרופיה מקומית, שטח, היקפים, בהירות, זרימה אופטית וממד פרקטלי, הפעל
תקליטור ./נתונים
Python compute_features.py
תקליטור..
הערה: כדי להדגיש את השינויים המורפולוגיים, ניתן לנרמל את הערכים של תכונות מסוימות (לדוגמה, שטח, היקפים וזרימה אופטית) על-ידי ערכי הבסיס הראשוניים שלהם בזרמי התמונה. - חלק באופן אקראי את ערכת הנתונים לערכת אימונים ולערכת בדיקות. הפעל את מחברת Jupyter "./data/prepare_dataset.ipynb" כדי לפצל את ערכת הנתונים ולארגן את ערכות הנתונים כמסגרות נתונים (איור 3D).
- עבור כל משך זמן של CHR, תרגל מודל רגרסיה לוגיסטית כדי לחזות את תווית ריכוז CHIR (נמוכה/אופטימלית/גבוהה) מתכונות זרם התמונה בערכת האימונים. הערך את ביצועי הסיווג של מודל הרגרסיה הלוגיסטית המאומן בערכת הבדיקה באמצעות דיוק, דיוק, היזכרות, ציון F1 ואזור מתחת לעקומה (AUC). הריצו את מחברת יופיטר "machine_learning.ipynb" לאימון ולבדיקה (איור 3E).
- הערך את ביצועי המודל בהערכת מינון CHR. בקבוצת הבדיקה, יש למזג תוויות חזויות של בארות מקבילות בעלות ריכוז CHIR זהה באמצעות ציוני סטייה (הנעים בין -1 ל-1), המוגדרים על ידי
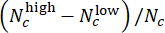 , כאשר Nc מציין את מספר הבארות עם ריכוז c והבארות
, כאשר Nc מציין את מספר הבארות עם ריכוז c והבארות 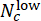
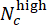 ביניהן נחזות כ"נמוכות" ו"גבוהות", בהתאמה. ודא כי ציוני הסטייה החזויים מתואמים מאוד עם ריכוז ΔCHIR האמיתי עבור כל מנת CHIR באמצעות מקדם המתאם של פירסון.
ביניהן נחזות כ"נמוכות" ו"גבוהות", בהתאמה. ודא כי ציוני הסטייה החזויים מתואמים מאוד עם ריכוז ΔCHIR האמיתי עבור כל מנת CHIR באמצעות מקדם המתאם של פירסון. - בצע אימות חוצה אצווה כדי לבדוק את ביצועי ההכללה של המודל. בכל סבב, השאירו אצווה אחת לבדיקה והשתמשו באחרות לאימונים. השווה את ציוני הסטייה החזויים ואת ריכוז ΔCHIR האמיתי עבור כל מנת CHIR בכל אצוות בדיקה באמצעות מקדם מתאם פירסון. הפעל את מחברת Jupyter "cross_batch_validation.ipynb" כדי ליישם את האימות חוצה אצווה (איור 3F).
הערה: עבור משך CHIR נבחר h, החיזוי עבור כל הבארות עם ריכוז ממודל הרגרסיה הלוגיסטית שאומן תחת משך h יומר לציון סטייה, המשקף את האופטימליות של ריכוז CHIR c. ציון סטייה קרוב ל-0 מצביע על כך שריכוז c צפוי להיות אופטימלי תחת משך h, בעוד שציון סטייה שלילי או חיובי מצביע על מצב של מנת חסר או מנת יתר, מה שיכול לשמש איתות להתאמה מאוחרת יותר של מנות CHR. שים לב שמכיוון שהחיזוי נעשה מוקדם (12 שעות), עדיין ניתן להתאים את משך ה- CHIR הסופי ואת ריכוז ה- CHIR במהלך שאר שלב I. - יישם את מודלי הרגרסיה הלוגיסטית המאומנים כדי להעריך את מנות CHIR באצוות חדשות. עבור ריכוז CHIR נתון, לחשב תכונות זרם תמונה 0-12 שעות עבור בארות מקבילות עם ריכוז , להעביר את התכונות למודלים רגרסיה לוגיסטית מאומנים תחת משכי CHIR שונים, ובהתאם, לקבל את התוויות החזויות עבור כל באר. בהתבסס על חיזוי המודלים, חשב ציוני סטייה כדי להעריך כל ריכוז CHIR תחת משכי CHIR שונים.
- בעזרת הערכת מינון CHIR מבוססת מודל, בארות הצלה תחת כל ריכוז CHIR תת-אופטימלי בהתאם, על ידי התאמת משך או ריכוז ה-CHIR שלהן לכיוון אופטימלי לפני 48 שעות.
- התאמת משך: על פי תחזית המודל לגבי משך CHIR אופטימלי, התאם את נקודת הזמן שבה התווך המכיל CHIR בבארות אלה עובר למדיום התמיינות CM. אם נקודת הזמן האופטימלית מתפספסת, השליכו דגימות אלה.
- התאמת ריכוז: תחת משך CHIR שנקבע מראש, אם הריכוז הנוכחי צפוי כ"נמוך", הוסף ישירות ריכוז גבוה של CHIR למדיום כדי להגדיל את ריכוז CHR. יש לדלל את ה-CHIR ב-DMSO (50 מילימול) עם PBS בעת הצורך, להוסיף נפח מתאים של נוזל ולנער בעדינות את הצלחת. אם הריכוז הנוכחי צפוי להיות "גבוה", לשאוף נפח מסוים של מדיום המכיל CHIR ולהשלים עם אותו נפח של מדיום התמיינות CM.
הערה: ב-48 השעות הראשונות להתמיינות, התאים יפרישו גורמים לשיפור גדילת התאים והתמיינותם, ולכן אל תשנו לחלוטין את התווך. בנוסף, יש צורך לשמור על נפח בינוני קבוע בידול. לכן, יש לוודא שנפח הנוזל שנוסף קטן ככל האפשר בעת התאמת ריכוז ה-CHR.
- אסטרטגיית ML בשלב II: הכרה ב- CPC המחויבים ל- CM
ערכת נתונים: זוגות של תמונות שדה בהיר ביום 6 ומסכות עם הערות ידניות של CPC המחויבים ל-CM
דגם: ResNeSt38 עם Grad-CAM36 ללמידה בפיקוח חלש- הכינו מערך נתונים המורכב מתמונות שדה בהיר ביום השישי. הוסף ביאורים ידניים לקליקים המחויבים ל-CM בתמונות שדה האור על-ידי מעקב אחר תאי cTnT+ בזרמי התמונה מהיום ה-12 ועד היום ה-6.
- חתוך את תמונות שדה הבהיר ואת הביאור הידני המתאים (המכונה מסיכות) של CPC המחויבים ל- CM לטלאים, כאשר גודל כל תיקון הוא 1/5.5 מגודל התמונה המלאה. חלץ טלאים באופן חופף כאשר מדבקות סמוכות חולקות חפיפה של 50% בערכת האימונים וחפיפה של 75% בערכת הבדיקה. תייג טלאים עם ≥30% CPC מחוייבים CM כ"חיובי". תייג טלאים ללא CPC המחויבים ל-CM כ"שליליים". העבר את ספריית העבודה ל- "stage_II". הפעל את "./Crop_and_Reconstruct/mask_crop.m" כדי לחתוך את התמונות. הפעל "./Crop_and_Reconstruct/classification.m" כדי להקצות תוויות (0: שלילי; 1: חיובי) לתיקונים בשדה בהיר (איור 3G).
- חלקו באופן אקראי את טלאי התמונה המסומנים לערכת אימונים ולערכת בדיקות.
- אמן רשת עצבית קונבולוציונית עמוקה, ResNeSt38 כדי ללמוד לסווג את הטלאים האלה. הערך את ResNeSt המיומן בערכת הבדיקה כדי לאשר שהמודל מדגים דיוק סיווג גבוה. הפעל "train.py" ו-"test.py" לאימון ולבדיקה, בהתאמה (איור 3H).
- השתמש ב- Grad-CAM36 כדי להדגיש את האזורים התורמים ביותר להסקת מסקנות של ResNeSt, המיוצגים על-ידי מפות חום. בצע בינאריזציה של מפות החום (באמצעות סף, לדוגמה, 10% × לכל היותר) כדי לקבל את אזורי CPC החזויים המחויבים ל- CM, הנקראים אזורי CPC (IR-CPC) המזוהים עם תמונה.
- השווה בין אזורי IR-CPC עם מסכות עם ביאורים ידניים בערכת הבדיקה באמצעות דיוק, ציון F1, דיוק, היזכרות, ספציפיות והצטלבות-על-איחוד (IoU). השווה את חלקם היחסי של אזורי IR-CPC עם מדדי יעילות בידול (מחושב מתמונות הפלואורסצנטיות הסופיות של cTnT) באמצעות מקדם מתאם פירסון. הפעל את "./Evaluation/index_calculate.m" ו- "./Evaluation/Pearson_correlation.m" להערכת ביצועים (איור 3I).
- יישם את המודל המאומן כדי לזהות CPC עבור אצוות חדשות בשלב II. חתוך תמונות שדה בהיר שלמות לטלאים, העבר את הטלאים ל- ResNeSt המיומן והשג את אזורי IR-CPC ברמת התיקון, אשר לאחר מכן מתמזגים כדי להניב את אזורי IR-CPC של כל הבאר. חשב את החלק של אזורי IR-CPC כאינדיקטור ליעילות הבידול הסופית. לטהר את IR-CPCs כדי להפחית את זיהום התאים (מתואר להלן).
הערה: מכיוון שמודל זיהוי CPC יכול לבצע הערכה מוקדמת של יעילות ההתמיינות הסופית, הוא יכול לשמש גם כקריאת תפוקה גבוהה לסינון מולקולות קטנות התורמות לתהליך ההתמיינות, מה שעשוי לייעל עוד יותר את פרוטוקול ההתמיינות35.
- טיהור סלקטיבי אזורי של CPC המזוהים עם תמונות
הערה: ניסויי פוטואקטיבציה מבוססים על מיקרוסקופ פלואורסצנטי הפוך בעזרת שלב ממונע.- על פי אזורי IR-CPC שנחזו על ידי המודל המאומן, השליכו את הבארות שלא יכלו לייצר שום IR-CPC. כדי לטהר את ה-CPC המחויבים ל-CM, השתמש בבדיקה פוטו-אקוסטית שאינה ציטוטוקסית, Dual-Activatable Cell Tracker 1 (DACT-1) כדי לתייג באופן סלקטיבי אזור שאינו CPC39.
- ממיסים את DACT-1 (10 mM) ב-DMSO ואחסנו אותו בטמפרטורה של -20°C. לדגור יום 6 תאים עם 1 מיקרומטר DACT-1 בתווך התמיינות CM למשך 30 דקות ב 37 ° C.
- בדקו את התאים החיים תחת מיקרוסקופ ובחרו את אזור העניין (ROI) כמצולעים ב-MATLAB. ציירו קווים מקבילים עם מרווח של 20 מיקרומטר בתוך המצולע וחשבו את הקואורדינטות של נקודות החתך.
- הגדר את המסלול הנע של השלב הממונע על פי קווי עקבות, ואז הבמה תנוע במהירות של 0.12 מ"מ לשנייה כך שקו לייזר של 405 ננומטר (קוטר 20 מיקרומטר) שהורכב על הבמה יוכל לסרוק באופן גלובלי את החזר ההשקעה כדי לבצע פוטו-הפעלה של חומרים שאינם CPC באופן מוגבל. לאחר הקרנה, זהה את התאים המסומנים בתווית DACT-1 באמצעות קו לייזר של 561 ננומטר.
- נתקו תאים באמצעות 0.05% טריפסין-EDTA (מדולל על ידי PBS) ב-37°C למשך 5-7 דקות, ונערו בעדינות את הצלחת באינקובטור למשך 2 דקות. סנן את התאים דרך מסננת תאים 40 מיקרומטר וצנטריפוגה ב 500 × גרם במשך 3 דקות. השהה מחדש את התאים ב- 0.5% BSA ושמור את הצינור על קרח עד למיון. הכינו גם דגימה לא מוקרנת כבקרה שלילית למיון.
- הפעל את מערכת מיון ציטומטריית הזרימה והגדר את תוכנת המיון. בחר לייזרים באורך גל של 561 ננומטר והשתמש במסנן פסים של 610/20 למיון DACT1+ . עבור כל דגימה, השתמש בכלי gating מצולע כדי להקיף את אוכלוסיית התא בתרשים דו-משתני של פיזור קדמי (FSC) ופיזור צדדי (SSC) לבחירת התא הבודד. לאחר מכן, הפעל את הבקרה השלילית כדי לאשר את פלואורסצנטיות הרקע, הפעל את הדגימה המוקרנת באופן סלקטיבי באזור, וצייר את השער עבור אוכלוסיית DACT1+ .
- אסוף והשהה מחדש את התאים הממוינים באמצעי תחזוקת CM עם 10% FBS ו- 5 מיקרומטר Y27632, וזרע את התאים הממוינים בצלחת מצופה מטריג'ל של 96 בארות.
- למחרת, החליפו את מדיום התחזוקה בינוני ל-CM והמתינו לקציר CMs בעלי טוהר גבוה.
- אסטרטגיית ML בשלב III: הכרה ב- CMs
ערכת נתונים: זוגות של תמונות brightfield ביום ה-12 ותמונות הפלואורסצנטיות האחרונות של cTnT
דגם: pix2pix37- הכן ערכת נתונים המורכבת מתמונות שדה בהיר של CMs ותמונות פלואורסצנטיות cTnT מתאימות. כדי להגדיל את המגוון, ודא שמערך הנתונים כולל בארות עם יעילות התמיינות וקווי תאים שונים.
- חלק באופן אקראי את ערכת הנתונים לערכת אימונים ולערכת בדיקות. העבר את ספריית העבודה ל- "stage_III". שמור את תמונות שדה הבהירות ואת התמונות הפלואורסצנטיות cTnT המתאימות ב- "./pix2pix/datasets/(CM|CM_new_cell_lines)/A/(train|test)/*.png" ו- ".../B/(train|test)/*.png", בהתאמה. הפעל את הפקודה הבאה:
CD PIX2PIX/ערכות נתונים
פיתון combine_A_and_B.py --fold_A ./CM/A --fold_B ./CM/B --fold_AB ./CM/ --no_multiprocessing
תקליטור.. - אמן את pix2pix דגם37 על סט האימונים. הפעל את הפקודה הבאה (איור 3J):
python train.py --dataroot ./datasets/CM --name brightfield2fluorescence --דגם pix2pix --input_nc 1 --output_nc 1 --load_size 1536 --crop_size 256 --lr 2e-4 --n_epochs 1000 --n_epochs_decay 0 --מופע נורמה --netD n_layers --n_layers_D 1 --batch_size 16 ---כיוון AtoB --save_epoch_freq 100 --dataset_mode מיושר --use_resize_conv --זרע 1234 - החל את מודל pix2pix המאומן על ערכת הבדיקה. הפעל את הפקודה הבאה:
python test.py --dataroot ./datasets/CM --שם brightfield2fluorescence --דגם pix2pix --כיוון AtoB --input_nc 1 --output_nc 1 --load_size 1536 --crop_size 1536 --use_resize_conv --eval --num_test 1000 - השווה את התמונות הפלואורסצנטיות שנחזו על ידי המודל עם התמונות האמיתיות (כלומר, שהתקבלו בניסוי). ברמת הפיקסל, השווה את העוצמות החזויות ואת העוצמות האמיתיות באמצעות מקדם המתאם של פירסון. בנוסף, ברמת התמונה, השווה מדדי יעילות בידול חזויים עם אלה האמיתיים באמצעות מקדם המתאם פירסון. הפעל את "stage_III/evaluation/pixel_correlation.m" ו- "image_correlation.ipynb" עבור השוואות אלה (איור 3K,L).
- יישם את המודל המאומן כדי לזהות CMs באצוות חדשות בשלב III. העבר תמונות חדשות של CMs בשדה בהיר למודל pix2pix המיומן וקבל חיזוי in-silico של תמונות פלואורסצנטיות ויעילות בידול.
הערה: כדי להפחית את זיהום התאים, מיון תאים פוטואקטיביים מבוסס DACT-1 ישים גם לטיהור CMs המוכרים על-ידי המודל.
Access restricted. Please log in or start a trial to view this content.
תוצאות
בהתבסס על הדמיה של שדה בהיר ו-ML, ניתן לנטר ולמטב את תהליך הבידול הכולל בצורה חכמה. בשלב ה-PSC פיתחנו מודל ML שיכול לחזות את יעילות ההתמיינות הסופית בהתאם למאפיינים המורפולוגיים של מושבות PSC ראשוניות, כדי לקבוע את נקודת הזמן המתאימה או המתאימה ביותר ליזום התמיינות (
Access restricted. Please log in or start a trial to view this content.
Discussion
כאן, תיארנו פרוטוקול מפורט כדי להתגבר על אחת הבעיות העיקריות ביישום ובתרגום PSC הנוכחיים – השונות בהתמיינות תאים. על-ידי רתימת דימות שדה בהיר של תאים חיים ו-ML, ביצענו אופטימיזציה איטרטיבית של התמיינות PSC כדי להשיג יעילות גבוהה באופן עקבי בין קווי תאים ואצוות. עם זאת, בתהלי?...
Access restricted. Please log in or start a trial to view this content.
Disclosures
יאנג ג'או, ג'ו ז'אנג, שיאו-צ'ון יאנג, יאו וואנג ודאיצ'או צ'ן מגישים פטנט על אסטרטגיית הבידול של PSC המדווחת במאמר זה (202210525166.X).
Acknowledgements
אנו מודים לצ'יושי סאן, יאו וואנג, יו שיה, ג'יניו יאנג, צ'אנג לין, זימו צ'ן, דונגדונג ליאנג, רונג ווי, זה שו, גואנגיין שי, גאנג שואה, קאן יה, לי-פנג וואנג, פנג זו, שי-קיאנג וואנג, פבלו ריברה-פואנטס, סלומה פונטנר, ג'י-שינג צ'ן, יי ליו וג'ו ז'אנג, על הנחת היסודות לאסטרטגיה זו. עבודה זו נתמכה על ידי תוכנית המו"פ הלאומית של סין (2018YFA0800504, 2019YFA0110000) ופרויקט הניסויים הרפואיים בחלל של תוכנית החלל המאוישת של סין (HYZHXM01020) ליאנג ג'או. איור 1 נוצר באמצעות BioRender.com.
Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| 0.25% Trypsin-EDTA | Gibco | 25200056 | Diluted digests were used for CPC and CM digestion |
| 4% Paraformaldehyde in PBS | KeyGEN BioTECH | KGIHC016 | |
| 6-well Cell Culture Plate | NEST | 703001 | |
| 96-well Cell Culture Plate | NEST | 701001 | |
| B27 Supplement | Gibco | 17504044 | |
| B27 Supplement Minus Insulin | Gibco | A1895601 | |
| Bovine serum albumin (BSA) | GPC BIOTECH | AA904-100G | |
| Celldiscoverer 7 | Zeiss | Instruments used to take bright-field images throughout differentiation and final cTnT images | |
| CHIR99021 | Selleck | S1263 | |
| DMEM/F12 | Gibco | 12634010 | |
| Donkey anti-Mouse IgG (H+L) Highly Cross-Adsorbed Secondary Antibody, Alexa Fluor 488 | Thermo | A-21202 | Secondary Antibody |
| FACSAria III | BD Biosciences | Flow cytometry sorter | |
| Fetal Bovine Serum (FBS) | VISTECH | SE100-B | |
| Hoechst 33342 | YEASEN | 40732ES03 | |
| Human Pluripotent Stem Cell Chemical-defined Medium | Cauliscell Inc | 400105 | Basal medium of PSC preparation medium |
| iPS-18 | TaKaRa | Y00300 | |
| iPS-B1 | Cellapy | CA4025106 | |
| iPS-F | Nuwacell | RC01001-B | |
| iPS-M | Nuwacell | RC01001-A | |
| IWR1-1-endo | Selleck | S7086 | IWR1 |
| Jupyter Notebook | N/A | Version 6.4.0 | https://jupyter.org/ |
| MATLAB | MathWorks | Version R2020a | Software for scientific computation and image annotation |
| Matrigel Matrix | Corning | 354230 | Matrigel |
| Mouse monoclonal IgG1 anti-cTnT | Thermo | MA5-12960 | cTnT primary antibody |
| Normal Donkey Serum | Jackson | 017-000-121 | |
| ORCA-Flash 4.0 V3 digital CMOS camera | Hamamatsu | C13440-20CU | The digital camera assembled on Celldiscoverer7 |
| PBS | NEB | 21-040-CVR | |
| Penicillin-Streptomycin | Gibco | 15140-122 | |
| Pluripotency Growth Mater 1 basal medium | Cellapy | CA1007500-1 | Basal medium of PSC culture medium |
| Pluripotency Growth Mater 1 supplement | Cellapy | CA1007500-2 | Supplement of PSC culture medium |
| Prism | Graphpad | Version 8/9 | Statistical software for statistical analysis and plotting |
| Python | N/A | version 3.6 | Python 3 environment for scientific computation, with packages pytorch (1.9.0), numpy, scipy, pandas, visdom, scikit-learn, scikit-image, opencv-python, and matplotlib software for scientific computation and image annotation. |
| RPMI 1640 | Gibco | 11875176 | |
| Supplement hPSC-CDM (500x) | Cauliscell Inc | 00015 | Supplement of PSC preparation medium |
| TiE | Nikon | An inverted fluorescence microscope (with modification) for region-selevtive purification | |
| Triton X-100 | Amresco | 9002-93-1 | |
| Versene Solution | Thermo | 15040066 | EDTA solution for PSC digestion |
| Y27632 | Selleck | S6390 | |
| Zen | Zeiss | Version 3.1 | A supporting software of Celldiscoverer7 for image acquisition, processing and analysis |
References
- Yoshida, Y., Yamanaka, S. Induced pluripotent stem cells 10 years later: for cardiac applications. Circ Res. 120 (12), 1958-1968 (2017).
- Shi, Y., Inoue, H., Wu, J. C., Yamanaka, S. Induced pluripotent stem cell technology: a decade of progress. Nat Rev Drug Discov. 16 (2), 115-130 (2017).
- Takahashi, K., et al. Induction of pluripotent stem cells from adult human fibroblasts by defined factors. Cell. 131 (5), 861-872 (2007).
- Burridge, P. W., et al. Chemically defined generation of human cardiomyocytes. Nat Methods. 11 (8), 855-860 (2014).
- Mordwinkin, N. M., Burridge, P. W., Wu, J. C. A review of human pluripotent stem cell-derived cardiomyocytes for high-throughput drug discovery, cardiotoxicity screening, and publication standards. J Cardiovasc Transl Res. 6 (1), 22-30 (2013).
- Vanden Berg, C. W., Elliott, D. A., Braam, S. R., Mummery, C. L., Davis, R. P. Differentiation of human pluripotent stem cells to cardiomyocytes under defined conditions. Methods Mol Biol. 1353, 163-180 (2016).
- Burridge, P. W., Holmström, A., Wu, J. C. Chemically defined culture and cardiomyocyte differentiation of human pluripotent stem cells. Curr Protoc Hum Genet. 87, 1-15 (2015).
- Li, J., et al. Human pluripotent stem cell-derived cardiac tissue-like constructs for repairing the infarcted myocardium. Stem Cell Rep. 9 (5), 1546-1559 (2017).
- Smith, A. S. T., Macadangdang, J., Leung, W., Laflamme, M. A., Kim, D. -H. Human iPSC-derived cardiomyocytes and tissue engineering strategies for disease modeling and drug screening. Biotechnol Adv. 35 (1), 77-94 (2017).
- Sharma, A., et al. Use of human induced pluripotent stem cell-derived cardiomyocytes to assess drug cardiotoxicity. Nat Protoc. 13 (12), 3018-3041 (2018).
- Mehta, A., et al. Identification of a targeted and testable antiarrhythmic therapy for long-QT syndrome type 2 using a patient-specific cellular model. Eur Heart J. 39 (16), 1446-1455 (2018).
- Cahan, P., Daley, G. Q. Origins and implications of pluripotent stem cell variability and heterogeneity. Nat Rev Mol Cell Biol. 14 (6), 357-368 (2013).
- Kim, K., et al. Epigenetic memory in induced pluripotent stem cells. Nature. 467 (7313), 285-290 (2010).
- Rouhani, F., et al. Genetic background drives transcriptional variation in human induced pluripotent stem cells. PLoS Genet. 10 (6), e1004432(2014).
- Kim, K., et al. Donor cell type can influence the epigenome and differentiation potential of human induced pluripotent stem cells. Nat Biotechnol. 29 (12), 1117-1119 (2011).
- Garitaonandia, I., et al. Increased risk of genetic and epigenetic instability in human embryonic stem cells associated with specific culture conditions. PLoS One. 10 (2), e0118307(2015).
- Engler, A. J., Sen, S., Sweeney, H. L., Discher, D. E. Matrix elasticity directs stem cell lineage specification. Cell. 126 (4), 677-689 (2006).
- Matoba, N., Yamashita, T., Takayama, K., Sakurai, F., Mizuguchi, H. Optimal human iPS cell culture method for efficient hepatic differentiation. Differentiation. 104, 13-21 (2018).
- Volpato, V., et al. Reproducibility of molecular phenotypes after long-term differentiation to human iPSC-derived neurons: A multi-site omics study. Stem Cell Rep. 11 (4), 897-911 (2018).
- Anderson, N. C., et al. Balancing serendipity and reproducibility: Pluripotent stem cells as experimental systems for intellectual and developmental disorders. Stem Cell Rep. 16 (6), 1446-1457 (2021).
- Sepac, A., et al. Comparison of cardiomyogenic potential among human ESC and iPSC lines. Cell Transplant. 21 (11), 2523-2530 (2012).
- Laco, F., et al. Unraveling the inconsistencies of cardiac differentiation efficiency induced by the GSK3β inhibitor CHIR99021 in human pluripotent stem cells. Stem Cell Rep. 10 (6), 1851-1866 (2018).
- Volpato, V., Webber, C. Addressing variability in iPSC-derived models of human disease: guidelines to promote reproducibility. Dis Model Mech. 13 (1), 042317(2020).
- Hu, B. -Y., et al. Neural differentiation of human induced pluripotent stem cells follows developmental principles but with variable potency. Proc Natl Acad Sci USA. 107 (9), 4335-4340 (2010).
- Huo, J., et al. Evaluation of batch variations in induced pluripotent stem cell-derived human cardiomyocytes from 2 major suppliers. Toxicol Sci. 156 (1), 25-38 (2017).
- Rostam, H. M., Reynolds, P. M., Alexander, M. R., Gadegaard, N., Ghaemmaghami, A. M. Image based machine learning for identification of macrophage subsets. Sci Rep. 7 (1), 3521(2017).
- Christiansen, E. M., et al. In silico labeling: Predicting fluorescent labels in unlabeled images. Cell. 173 (3), 792-803 (2018).
- Buggenthin, F., et al. Prospective identification of hematopoietic lineage choice by deep learning. Nat Methods. 14 (4), 403-406 (2017).
- Smith, Z. D., Nachman, I., Regev, A., Meissner, A. Dynamic single-cell imaging of direct reprogramming reveals an early specifying event. Nat Biotechnol. 28 (5), 521-526 (2010).
- Fan, K., Zhang, S., Zhang, Y., Lu, J., Holcombe, M., Zhang, X. A machine learning assisted, label-free, non-invasive approach for somatic reprogramming in induced pluripotent stem cell colony formation detection and prediction. Sci Rep. 7 (1), 13496(2017).
- Kusumoto, D., et al. Automated deep learning-based system to identify endothelial cells derived from induced pluripotent stem cells. Stem Cell Rep. 10 (6), 1687-1695 (2018).
- Zhu, Y., et al. Deep learning-based predictive identification of neural stem cell differentiation. Nat Commun. 12 (1), 2614(2021).
- Qian, T., et al. Label-free imaging for quality control of cardiomyocyte differentiation. Nat Commun. 12 (1), 4580(2021).
- Ounkomol, C., Seshamani, S., Maleckar, M. M., Collman, F., Johnson, G. R. Label-free prediction of three-dimensional fluorescence images from transmitted-light microscopy. Nat Methods. 15 (11), 917-920 (2018).
- Yang, X., et al. A live-cell image-based machine learning strategy for reducing variability in PSC differentiation systems. Cell Discovery. 9 (1), 53(2023).
- Selvaraju, R. R., et al. Grad-CAM: Visual explanations from deep networks via gradient-based localization. Proc IEEE Int Conf Computer Vision (ICCV). , 618-626 (2017).
- Isola, P., Zhu, J. -Y., Zhou, T., Efros, A. A. Image-to-image translation with conditional adversarial networks. Proc IEEE Conf Computer Vision Pattern Recognition (CVPR). , 5967-5976 (2017).
- Zhang, H., et al. ResNeSt: Split-attention networks. Proc IEEE/CVF Conf Computer Vision Pattern Recognition Workshops (CVPRW). , 2735-2745 (2022).
- Halabi, E. A., et al. Dual-activatable cell tracker for controlled and prolonged single-cell labeling). ACS Chem. Biol. 15 (6), 1613-1620 (2020).
- Hannan, N. R. F., Segeritz, C. -P., Touboul, T., Vallier, L. Production of hepatocyte-like cells from human pluripotent stem cells. Nat Protoc. 8 (2), 430-437 (2013).
- Zhang, H., Shen, M., Wu, J. C. Generation of quiescent cardiac fibroblasts derived from human induced pluripotent stem cells. Methods Mol Biol. 2454, 109-115 (2022).
- Harding, M. J., McGraw, H. F., Nechiporuk, A. The roles and regulation of multicellular rosette structures during morphogenesis. Development. 141 (13), 2549-2558 (2014).
- Najm, F. J., et al. Rapid and robust generation of functional oligodendrocyte progenitor cells from epiblast stem cells. Nat Methods. 8 (11), 957-962 (2011).
- Hogrebe, N. J., Maxwell, K. G., Augsornworawat, P., Millman, J. R. Generation of insulin-producing pancreatic β cells from multiple human stem cell lines. Nat Protoc. 16 (9), 4109-4143 (2021).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved