É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Uma estratégia de aprendizado de máquina baseada em imagem de células vivas para monitorar a diferenciação de células-tronco pluripotentes
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Os sistemas de diferenciação de células-tronco pluripotentes (PSC) para células funcionais disponíveis são atualmente impedidos por problemas de variabilidade severa linha a linha e lote a lote. Aqui, usando a diferenciação cardíaca como exemplo principal, apresentamos um protocolo para monitorar e modular de forma inteligente o processo de diferenciação de CEP com base em aprendizado de máquina baseado em imagem.
Resumo
As tecnologias de células-tronco pluripotentes (PSC) têm sido amplamente utilizadas na descoberta de medicamentos, modelagem de doenças e medicina regenerativa. No entanto, os sistemas de diferenciação PSC para células funcionais disponíveis são impedidos por problemas de variabilidade severa linha a linha e lote a lote. O controle preciso da diferenciação celular em tempo real é, portanto, importante. Neste protocolo, descrevemos uma estratégia não invasiva e inteligente que supera a variabilidade na diferenciação celular usando aprendizado de máquina baseado em imagem de campo claro. Tomando como exemplo a diferenciação de CEP para cardiomiócitos, esta metodologia fornece informações detalhadas para o controle do estado inicial de CEP, avaliação e intervenção precoces em condições de diferenciação e eliminação da contaminação celular indiferenciada, realizando juntos uma diferenciação consistente de alta qualidade de CEPs para células funcionais. Em princípio, essa estratégia pode ser estendida a outros sistemas de diferenciação ou reprogramação celular com várias etapas para apoiar a fabricação de células, bem como para aprofundar nossa compreensão dos mecanismos durante a conversão do destino celular.
Introdução
As células-tronco pluripotentes (PSCs) possuem a notável capacidade de se diferenciar em muitos tipos de células in vitro. Essas células funcionais diferenciadas podem ser usadas para terapia celular, modelagem de doenças e desenvolvimento de medicamentos, todos valiosos para pesquisas ou aplicações clínicas 1,2,3. Por exemplo, uma variedade de métodos foi desenvolvida para diferenciar as CEPs em cardiomiócitos (MCs)4,5,6,7. Esses MCs podem ser aplicados para testes de cardiotoxicidade de drogas, modelagem de doenças cardíacas e transplante de células 8,9,10,11.
No entanto, a conversão de PSC para as células diferenciadas terminais é um processo gradual, e múltiplas perturbações durante o processo de diferenciação podem levar as células a destinos celulares divergentes. Diferentes origens genéticas e marcas epigenéticas das linhagens PSC influenciam o potencial de diferenciação para uma linhagem específica 12,13,14,15; o número de passagens de PSC e mutações genéticas acumuladas também são fontes de heterogeneidade de PSC; Diferenças nas operações experimentais empregadas por diferentes experimentadores também podem levar a resultados de diferenciação completamente diferentes 16,17,18,19,20. Portanto, atualmente um dos principais problemas na produção de células derivadas de PSC é a instabilidade entre linhagens celulares e lotes 21,22,23,24,25. A instabilidade na diferenciação do PSC geralmente leva a vários experimentos repetidos, consumindo tempo e recursos de mão de obra significativos. Para resolver esse problema, é crucial desenvolver uma estratégia que minimize a variabilidade entre linhagens e lotes celulares, aumentando assim a estabilidade e a robustez da diferenciação.
Recentemente, os avanços na microscopia de alta resolução e no aprendizado de máquina (ML) facilitaram a aplicação da análise quantitativa de imagens baseada em ML na biologia celular, tornando possível a utilização de informações valiosas em recursos de imagem celular 26,27,28,29,30,31,32,33,34. Em nosso trabalho anterior, propusemos uma estratégia de ML baseada em imagem de célula viva para monitorar e intervir no status de diferenciação do PSC em tempo real para melhorar a estabilidade e a eficiência da diferenciação do PSC (Figura 1)35. Tomando como exemplo a diferenciação de PSC para cardiomiócitos, avaliamos o estado inicial de PSC usando modelos de floresta aleatória, previmos a condição de diferenciação ideal usando modelos de regressão logística e reconhecemos células diferenciadas com sucesso usando Grad-CAM36 e pix2pix37 baseados em aprendizado profundo. Os modelos de ML aprenderam a identificar linhagens celulares a partir de uma variedade de características morfológicas de campo claro, incluindo características sobre área, circunferência, convexidade, solidez, brilho, velocidade de movimento e outras características implícitas extraídas por redes neurais convolucionais profundas. Com base na inferência desses modelos de ML estabelecidos, realizamos o controle do estado inicial da CEP, avaliação e intervenção precoces em condições de diferenciação e eliminação da contaminação celular indiferenciada, fornecendo uma modulação abrangente e precisa do processo de diferenciação cardíaca. Aqui, fornecemos um protocolo passo a passo para o desenvolvimento da estratégia.
Protocolo
1. Diferenciação e caracterização celular
- Preparação de reagentes de cultura e placas de cultura
- Prepare o meio de cultura PSC adicionando 2 mL de suplemento e 0,2% de penicilina-estreptomicina a 48 mL de meio basal. Aliquotar e conservar o suplemento a -20 °C. Conservar este meio a 4 °C durante um máximo de 4 semanas.
- Prepare o meio de preparação PSC adicionando 1 mL de suplemento e 0,2% de penicilina-estreptomicina a 500 mL de meio basal. Ao usar, pré-aqueça o meio para uso único e armazene-o a 4 ° C por até 3 semanas.
- Prepare o meio de diferenciação do CM adicionando 1x suplemento de B27 menos insulina e 1% de penicilina-estreptomicina no RPMI 1640. Prepare o meio de manutenção do CM adicionando 1x suplemento de B27 e 1% de penicilina-estreptomicina no RPMI 1640. Conservar estes dois tipos de meios a 4 °C durante 2 semanas.
- Mergulhe o Matrigel no gelo e mantenha-o a 4 °C durante a noite. Certifique-se de que o Matrigel esteja completamente descongelado; em seguida, alíquota em alíquotas de 1 mL armazenadas a -80 ° C para uso posterior e adicione 1 mL de Matrigel em 49 mL de DMEM / F12 a 4 ° C para preparar a solução de trabalho Matrigel. Adicione 850 μL desta solução de trabalho Matrigel por poço de uma placa de cultura de 6 poços e incube a placa a 37 °C por pelo menos 30 min. Para a placa de cultura de 96 poços usada na diferenciação de CM, adicione 35 μL de solução de trabalho Matrigel por poço.
NOTA: Mantenha o Matrigel no gelo o tempo todo; as pontas e os tubos da pipeta devem ser pré-resfriados a 4 °C. A solução de trabalho Matrigel pode ser armazenada a 4 °C por 2 semanas. Tenha cuidado para evitar bolhas ao pipetar a solução de trabalho Matrigel.
- Manutenção e passagem do PSC
- Mantenha as PSCs em placas de cultura de 6 poços. Confirme a densidade celular ao microscópio e prepare-se para a passagem quando a confluência atingir ~ 70% (Figura 2A).
- Use EDTA para digerir as células para passagem. Aqueça o meio de cultura PSC, PBS e EDTA em banho-maria a 37 °C e adicione Y27632 no meio até uma concentração final de 5 μM.
- Lave os PSCs com 1 mL de PBS, adicione 1 mL de EDTA por poço e incube a placa em uma incubadora a 37 ° C por 3 min.
- Aspire o EDTA antes que as células se desprendam da placa de cultura e, em seguida, use 1 mL de meio de cultura PSC para enxaguar suavemente o fundo do poço 3-4x para ressuspender as células.
NOTA: Isso nos permite pular a etapa de centrifugação e reduzir os danos às células nas operações de passagem. - Aspire o DMEM/F12 de um poço revestido com Matrigel de uma placa de cultura de 6 poços e adicione 2 mL de meio de cultura PSC fresco por poço (Y27632 adicionado). Adicione a suspensão celular aos poços com a proporção de divisão de 1:6 a 1:12. Coloque a placa na incubadora a 37 °C com 5% de CO2.
- Troque o meio de cultura PSC (sem Y27632) 12 a 24 h após a passagem; Em seguida, mude o meio e verifique o estado da célula e a confluência todos os dias.
- Diferenciação cardíaca de CEPs
- Para diferenciação de CM, semeie PSCs em uma placa de cultura de 96 poços em meio de preparação de PSC (Y27632 adicionado). Para diferenciação de CM, siga as mesmas etapas acima (etapas 1.2.2-1.2.6) para semear PSCs em uma placa de cultura de 96 poços em meio de preparação PSC (Y27632 adicionado).
NOTA: Nesta fase, a duração do tratamento com EDTA pode ser adequadamente estendida (5 min ou mais) para dissociar suficientemente as PSCs para que os clones possam ser menores após a passagem. Nosso estudo demonstrou que, dentro da mesma área, colônias menores são propícias à diferenciação. - Estágio I: Quando as PSCs atingirem 80-90% de confluência, troque o meio para o meio de diferenciação CM com 2-20 μM CHIR99021 (CHIR) (Figura 2B). O ponto de tempo da comutação média é referido como dia 0. Após 24-48 h de tratamento com CHIR, troque o meio por meio de diferenciação de CM fresco.
NOTA: O CHIR é um inibidor de GSK3beta para indução do mesoderma. A dose ideal de CHIR para diferenciação de CM flutua com diferentes linhagens e lotes celulares. Por exemplo, a dose ideal de CHIR é de 8-10 μM, 48 h para iPS-B1. Para coletar diversas imagens de campo claro para treinar o modelo ML, aqui titulamos as concentrações de CHIR para cada lote de experimentos para diferentes eficiências de diferenciação. - Estágio II: Às 72 h (dia 3), substitua o meio pelo meio de diferenciação CM suplementado com 5 μM IWR1 e cultive por 48 h (dia 5). Troque o meio com o meio de diferenciação CM por 1-2 dias (para os dias 6-7). Nesse ponto, as CEPs se diferenciam em células progenitoras cardíacas (CPC).
- Etapa III: Substitua o meio por meio de manutenção CM e substitua o meio a cada 3 dias. No dia 10 ou dia 12, colha o CM para análise posterior.
- Para diferenciação de CM, semeie PSCs em uma placa de cultura de 96 poços em meio de preparação de PSC (Y27632 adicionado). Para diferenciação de CM, siga as mesmas etapas acima (etapas 1.2.2-1.2.6) para semear PSCs em uma placa de cultura de 96 poços em meio de preparação PSC (Y27632 adicionado).
- Coloração por imunofluorescência de CM
- No dia 10 ou dia 12, lave as células com PBS e fixe-as com paraformaldeído a 4% em PBS por 15 min em temperatura ambiente. Lave os CMs 2x com PBS e guarde a placa a 4 °C por até ~1 semana.
NOTA: O paraformaldeído é um reagente tóxico com um odor pungente. Opere com uma capa química e proteja os olhos e as mãos. - No momento da coloração, trate as células com solução permeabilizante (0,1% Triton X-100 em PBS) por 15 min em temperatura ambiente; em seguida, incube as células em solução de bloqueio (0,1% Triton X-100 contendo 3% de soro de burro em PBS) por 30 min em temperatura ambiente.
- Incubar a amostra com anticorpo primário de troponina T cardíaca (cTnT) (1:300) diluído em solução de bloqueio durante a noite a 4 °C para identificar os CMs.
- Colete o anticorpo primário e lave as células com PBS 3x. Incubar a amostra com anticorpos secundários em PBS com albumina sérica bovina a 1% por 1 h a 37 °C em ambiente escuro.
NOTA: O anticorpo primário coletado pode ser reutilizado por 2-3x em 1 semana.
- Colete o anticorpo primário e lave as células com PBS 3x. Incubar a amostra com anticorpos secundários em PBS com albumina sérica bovina a 1% por 1 h a 37 °C em ambiente escuro.
- Remova o anticorpo secundário das células e lave as células 3x com PBS. Use Hoechst 33342 (1:1.000 em PBS) para corar núcleos por 5 min em temperatura ambiente. Enxágue a célula 3x usando PBS e adicione 100 μL de PBS por poço para evitar a secagem. Conservar a placa a 4 °C num local escuro e adquirir imagens fluorescentes no prazo de 1 semana.
- No dia 10 ou dia 12, lave as células com PBS e fixe-as com paraformaldeído a 4% em PBS por 15 min em temperatura ambiente. Lave os CMs 2x com PBS e guarde a placa a 4 °C por até ~1 semana.
2. Aquisição de fluxo de imagens ao longo do processo de diferenciação
- Configuração de projeto experimental para coleta de imagens
- Use um microscópio automatizado que suporte a cultura de células vivas e imagens para coletar imagens de campo claro de diferentes estágios na diferenciação de CM e imagens imunofluorescentes de cTnT do resultado da diferenciação (Figura 2A-E). Use o software de suporte ao microscópio para projetar o programa de experimentos e controlar o microscópio.
- Abra o software e crie um novo experimento experimental. Escolha uma objetiva de 5x e uma lente de tubo de 2x para imagens. Verifique as opções de blocos e pilha Z.
- Adicione o canal TL Brightfield para imagens de campo claro no menu Canais. Adicione canais AF488 e H3342 para imagens de imunofluorescência. Modifique o caminho da luz no menu Configuração de imagem para criar imagens com a câmera CMOS digital externa.
- Abra o menu Modo de aquisição e defina o compartimento 2 x 2 para aumentar a relação sinal-ruído.
- Abra o menu Z-stack e escolha o modelo Center. Defina o número de fatias e intervalos durante a digitalização. Escolha 3-5 fatias com intervalos de 3-6 μm para imagens em foco na análise.
- Na janela Navegação e blocos, configure as regiões de bloco por transportadora e defina 25 blocos (5 colunas x 5 linhas) para um poço. No menu Blocos, selecione o modelo Multiwell 96 em Sample Carrier e defina uma sobreposição de 5% a 15% em regiões adjacentes.
- Se for necessária uma imagem contínua, marque a opção Série temporale defina a duração e o intervalo no menu Série temporal. Para coletar o fluxo de imagens no estágio I, defina uma duração de 12 horas sem intervalos.
- Aquisição de imagens de campo claro e imunofluorescentes
- Para adquirir imagens, primeiro coloque a placa de cultura de células na bandeja de amostra e carregue a amostra dentro do microscópio. Selecione o modelo Multiwell 96 para corresponder ao experimento experimental. Se a amostra for constituída por células vivas, abrir o sistema de aquecimento e a bomba de CO2 para manter o estado adequado para a cultura (37 °C, 5% de CO2).
- Abra o projeto experimental predefinido e salve-o como um novo. Abra o menu Tiles e calibre a posição da placa manualmente. Na janela Navegação e blocos, selecione os poços necessários e clique em Criar para construir regiões de ladrilhos para esses poços.
- Clique em Verificar regiões de bloco no menu Blocos e execute o foco automático para verificar todos os poços. Em seguida, corrija manualmente o foco de cada poço sob o campo claro rolando a roda do mouse.
- Marque os canais necessários e defina os tempos de exposição no menu Canais. Os tempos de exposição recomendados são 1.625 ms para Bright, 100 ms para H3342 e 550 ms para AF488. Execute um único snap para verificar as imagens de todos os canais.
- Por fim, clique no botão Iniciar experimento e aguarde a geração automática de imagens. Normalmente, leva ~ 1,2 h para concluir a varredura de uma placa de cultura inteira de 96 poços (96 poços, três camadas, 25 ladrilhos para cada poço sob o campo claro). Na estrutura de processamento, escolha Exportação de imagem, selecione o tipo de arquivo do formato TIFF descompactado ou do formato PNG e aplique. Use imagens de 9 ladrilhos (3 x 3) no centro do poço para uma análise posterior (Figura 2A-E).
3. Estabelecimento da estratégia de ML baseada em imagem em cada estágio do processo de diferenciação
- Preparação de software
- Instale o ambiente Python, com os pacotes pytorch (1.9.0), numpy, scipy, scikit-learn, pandas, visdom, scikit-image, opencv-python e matplotlib.
- Instale o Jupyter Notebook.
- Instale o MATLAB.
- Baixe o código e os conjuntos de dados de exemplo do https://github.com/zhaoyanglab/ML-for-PSC-differentiation.
NOTA: O desempenho do ML depende da qualidade dos conjuntos de dados e da escolha de modelos e hiperparâmetros. Não há garantia de que a implementação de exemplo seja ideal para todas as circunstâncias. Os engenheiros de ML devem personalizar o código e adaptar o modelo e os hiperparâmetros para suas necessidades específicas.
- Estratégia de ML no estágio PSC: Controle dos estados colônias iniciais do PSC
Conjunto de dados: pares de imagens de campo claro em 0 h (antes do tratamento com CHI) e eficiência de diferenciação final
Modelo: floresta aleatória- Preparar um conjunto de dados constituído por imagens de campo claro a 0 h (antes do tratamento com CHIR) e as imagens finais de fluorescência cTnT. Para aumentar a diversidade de perfis morfológicos de imagens de campo claro de 0 h, certifique-se de que o conjunto de dados inclua colônias de PSC de diferentes linhagens celulares, com diferenciação iniciada em um intervalo de tempo após a passagem. Trate os poços no conjunto de dados com as condições ideais de CHIR no estágio I.
NOTA: Para garantir que as colônias sejam tratadas com as condições ideais de CHI, os poços dentro do mesmo lote podem ser tratados com doses tituladas de CHI. Apenas poços com condições CHIR de alta eficiência são incluídos no conjunto de dados. - Quantifique a eficiência de diferenciação de cada poço pelo Índice de Eficiência de Diferenciação calculado a partir de suas imagens de fluorescência cTnT, que é definido por
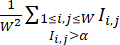 (onde
(onde 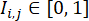 representa a intensidade de fluorescência na localização do pixel (i, j) na imagem de fluorescência W × W, e a soma é tomada em todas as localizações de pixel com intensidade acima de um limite α, normalmente definido em 0,5). Dentro de cada linhagem celular, calcule o Índice de Eficiência de Diferenciação máximo em todos os poços desta linhagem celular e normalize seus Índices de Eficiência de Diferenciação pelo valor máximo.
representa a intensidade de fluorescência na localização do pixel (i, j) na imagem de fluorescência W × W, e a soma é tomada em todas as localizações de pixel com intensidade acima de um limite α, normalmente definido em 0,5). Dentro de cada linhagem celular, calcule o Índice de Eficiência de Diferenciação máximo em todos os poços desta linhagem celular e normalize seus Índices de Eficiência de Diferenciação pelo valor máximo.
NOTA: Como o potencial de diferenciação de cada linhagem celular é inerentemente diferente, essa normalização enfatizará mais as características morfológicas da colônia compartilhadas entre várias linhagens celulares que contribuem para o resultado da diferenciação. A normalização de fato torna as colônias de PSC de diferentes linhagens celulares comparáveis e mitiga o viés induzido pelas linhagens celulares. - Quantifique os perfis morfológicos de imagens de campo claro de 0 h por características de alta dimensão que revelam as propriedades da forma da colônia. Para delinear regiões de colônia em imagens de campo claro, calcule a entropia local (com raio r); pixels com entropia local maior que um limite σ são identificados por células cobertas por células. Use uma configuração de parâmetro de r = 10 pixels e σ = 3. Mude o diretório de trabalho para "./colony_control"; salvar as imagens de campo claro, imagens binárias de regiões celulares e imagens de fluorescência cTnT em "colony_control/ image_data/CD00-*/[campo claro|cell_region|ctnt]/S*.png"; e executar o Jupyter Notebook "Compute_features.ipynb" para calcular recursos relacionados à área da colônia, circunferência, solidez, convexidade, circularidade e distâncias de contorno de centróide, entre outros.
- Divida aleatoriamente o conjunto de dados em um conjunto de treinamento e um conjunto de teste, com o conjunto de treinamento compreendendo entre 50% e 80% dos dados gerais. Execute o bloco final Divida o conjunto de dados no Jupyter Notebook para dividir o conjunto de dados (Figura 3A).
- Treine um modelo de regressão de floresta aleatória no conjunto de treinamento para prever a eficiência de diferenciação de recursos de imagem de campo claro de 0 h. Execute o Jupyter Notebook "Machine_learning.ipynb" para treinar e testar o modelo.
- Avalie o modelo de floresta aleatória treinado no conjunto de teste. Confirme se a eficiência de diferenciação prevista se correlaciona altamente com a verdadeira usando o coeficiente de correlação de Pearson (Figura 3B).
NOTA: Como o conjunto de teste não está envolvido na fase de treinamento, uma alta consistência entre a eficiência de diferenciação prevista e a verdadeira no conjunto de teste indica que o modelo pode generalizar bem em amostras invisíveis. - Obtenha a importância do recurso do modelo de floresta aleatória treinado e selecione 8 recursos com a maior importância. Visualize sua relação com a eficiência de diferenciação final dividindo a gama de recursos em compartimentos e calculando a eficiência média de diferenciação em cada compartimento. Execute o Jupyter Notebook "Feature_importance.ipynb" para executar a visualização (Figura 3C).
NOTA: A visualização pode ajudar a entender como as mudanças na forma da colônia PSC contribuem para a diferenciação. - Aplique o modelo treinado em novos lotes para monitorar os estados da colônia PSC em tempo real. Capture a imagem de campo claro a cada hora, passe a imagem para o modelo treinado e obtenha a eficiência de diferenciação prevista. Quando a eficiência prevista exceder um limite (por exemplo, 50%), inicie o processo de diferenciação. Se a eficiência prevista não puder atingir o limite, descarte essas amostras.
NOTA: Durante o estágio de PSC, o potencial de diferenciação das colônias de PSC aumentará primeiro e depois diminuirá. O monitoramento em tempo real permite que os experimentadores identifiquem o ponto de tempo mais propício para iniciar a diferenciação.
- Preparar um conjunto de dados constituído por imagens de campo claro a 0 h (antes do tratamento com CHIR) e as imagens finais de fluorescência cTnT. Para aumentar a diversidade de perfis morfológicos de imagens de campo claro de 0 h, certifique-se de que o conjunto de dados inclua colônias de PSC de diferentes linhagens celulares, com diferenciação iniciada em um intervalo de tempo após a passagem. Trate os poços no conjunto de dados com as condições ideais de CHIR no estágio I.
- Estratégia de ML no estágio I: avaliação precoce e ajuste das doses de CHIR
Conjunto de dados: pares de fluxos de imagens de campo claro em 0-12 h e rótulos de concentração CHIR (baixo/ótimo/alto)
Modelo: regressão logística- Prepare um conjunto de dados que consista em fluxos de imagens de campo claro de poço inteiro, onde as imagens são adquiridas a cada hora entre 0 h e 12 h. Registre as doses de CHIR (ou seja, combinações de concentrações e duração de CHIR) de cada poço. Certifique-se de que o conjunto de dados inclua diferentes lotes e poços com doses tituladas de CHIR (por exemplo, concentração = 2, 4, 6, 8, 10 e 12 μM; duração = 24, 36 e 48 h) para aumentar a diversidade. Mude o diretório de trabalho para "./stage_I"; salve as imagens como "./data/image/CD01-*/S*/T*.png", onde CD01-* (CD01-1, CD01-2, CD01-3, CD01-4) é o nome do lote, S* (S1, S2, ..., S96) é o índice do poço e T*.png (T1.png, T2.png, ..., T10.png) é a imagem de campo claro pré-processada do poço em diferentes etapas de tempo do fluxo de imagem.
- Em cada lote, calcule a porcentagem de células cTnT+ para cada poço, que é calculada dividindo o número de células cTnT+ pelo número total de células Hoechst+ na imagem de fluorescência. Calcule a média da porcentagem de células cTnT + em poços paralelos com a mesma condição de dose de CHI. Salve a condição de dose CHIR e a porcentagem de células cTnT+ para cada poço em um arquivo csv "./data/image/CD01-*/CD01-?_exp_config.csv".
- Em cada lote, determine a faixa de concentração de CHIR "baixa", "ótima" e "alta" em cada duração de CHIR de acordo com os seguintes critérios. As concentrações de CHIR com uma porcentagem média de células cTnT+ acima de um limite (por exemplo, 20%) são identificadas como "ótimas". As concentrações de CHIR além da faixa ideal são identificadas como "baixas" ou "altas". Em cada duração de CHIR, rotule os poços como "baixo", "ideal" e "alto" de acordo com suas concentrações de CHIR correspondentes.
NOTA: Empiricamente, existe uma correlação negativa entre as concentrações de CHIR e a duração dos poços de alta eficiência dentro de um lote: para maior duração do CHI, as concentrações ideais diminuem. Em outras palavras, a otimização das concentrações de CHIR depende da duração do CHIR e, portanto, os rótulos dos poços diferem entre as diferentes durações de CHIR. - Em cada lote, sob cada duração de CHIR, calcule a concentração de ΔCHIR para cada concentração c para quantificar seu desvio do ótimo. Denote a faixa de concentração ideal de CHIR em [c1, c2] e, em seguida, concentração ΔCHIR (c) = c - c1 (se c < c1), 0 (se c1 ≤ c ≤ c2) ou c - c2 (se c > c2).
- Extraia recursos dos fluxos de imagens no conjunto de dados, garantindo que eles reflitam as mudanças morfológicas das células durante o estágio I. Para calcular os recursos de imagem sobre entropia local, área, circunferências, brilho, fluxo óptico e dimensão fractal, execute
cd ./dados
compute_features.py python
CD..
NOTA: Para destacar as mudanças morfológicas, os valores de algumas características (por exemplo, área, circunferências e fluxo óptico) podem ser normalizados por seus valores iniciais de linha de base nos fluxos de imagem. - Divida aleatoriamente o conjunto de dados em um conjunto de treinamento e um conjunto de teste. Execute o Jupyter Notebook "./data/prepare_dataset.ipynb" para dividir o conjunto de dados e organizá-los como Dataframes (Figura 3D).
- Para cada duração do CHI, treine um modelo de regressão logística para prever o rótulo de concentração de CHIR (baixo/ideal/alto) a partir dos recursos do fluxo de imagens no conjunto de treinamento. Avalie o desempenho de classificação do modelo de regressão logística treinado no conjunto de teste usando exatidão, precisão, recall, pontuação F1 e área sob a curva (AUC). Execute o Jupyter Notebook "machine_learning.ipynb" para treinamento e teste (Figura 3E).
- Avalie o desempenho do modelo na avaliação da dose de CHI. No conjunto de teste, mescle rótulos previstos de poços paralelos com a mesma concentração de CHIR usando pontuações de desvio (variando de -1 a 1), definidas por
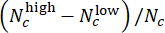 , onde Nc denota o número de poços com concentração c e
, onde Nc denota o número de poços com concentração c e 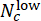 e
e 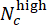 os poços entre eles são previstos como "baixo" e "alto", respectivamente. Confirme se as pontuações de desvio previstas se correlacionam altamente com a verdadeira concentração de ΔCHIR para cada dose de CHIR usando o coeficiente de correlação de Pearson.
os poços entre eles são previstos como "baixo" e "alto", respectivamente. Confirme se as pontuações de desvio previstas se correlacionam altamente com a verdadeira concentração de ΔCHIR para cada dose de CHIR usando o coeficiente de correlação de Pearson. - Execute uma validação entre lotes para testar o desempenho de generalização do modelo. Em cada rodada, deixe um lote para teste enquanto usa outros para treinamento. Compare as pontuações de desvio previstas e a concentração verdadeira de ΔCHIR para cada dose de CHIR em cada lote de teste usando o coeficiente de correlação de Pearson. Execute o Jupyter Notebook "cross_batch_validation.ipynb" para implementar a validação entre lotes (Figura 3F).
NOTA: Para uma duração de CHIR selecionada h, a previsão para todos os poços com concentração do modelo de regressão logística treinado sob a duração h será convertida em uma pontuação de desvio, refletindo a otimização da concentração de CHIR c. Uma pontuação de desvio próxima a 0 sugere que a concentração c é prevista como ótima sob a duração h, enquanto uma pontuação de desvio negativa ou positiva sugere uma condição de subdosagem ou superdosagem, que pode servir como um sinal para ajuste posterior das doses de CHIR. Observe que, como a previsão é feita cedo (12 h), a duração final do CHIR e a concentração do CHIR durante o restante do estágio I ainda podem ser ajustadas. - Aplicar os modelos de regressão logística treinados para avaliar as doses de CHIR em novos lotes. Para uma determinada concentração de CHIR , calcule os recursos de fluxo de imagem de 0 a 12 h para poços paralelos com concentração , passe os recursos para os modelos de regressão logística treinados sob diferentes durações de CHIR e, consequentemente, obtenha os rótulos previstos para cada poço. Com base na previsão dos modelos, calcule as pontuações de desvio para avaliar cada concentração de CHIR em diferentes durações de CHI.
- Com a avaliação da dose de CHIR baseada em modelo, resgate os poços sob cada concentração de CHIR abaixo do ideal de acordo, ajustando sua duração ou concentração de CHIR para o ideal antes de 48 h.
- Ajustando a duração: de acordo com a previsão do modelo sobre a duração ideal do CHIR, ajuste o ponto de tempo em que o meio contendo CHIR nesses poços é trocado pelo meio de diferenciação CM. Se o ponto de tempo ideal for perdido, descarte essas amostras.
- Ajustando a concentração: sob a duração predefinida do CHI, se a concentração atual for prevista como "baixa", adicione diretamente uma alta concentração de CHIR no meio para aumentar a concentração de CHI. Dilua CHIR em DMSO (50 mM) com PBS quando necessário, adicione um volume apropriado de líquido e agite suavemente a placa. Se a concentração atual for prevista como "alta", aspire um certo volume de meio contendo CHIR e suplemente com o mesmo volume de meio de diferenciação de CM.
NOTA: Nas primeiras 48 h de diferenciação, as células secretarão fatores para melhorar o crescimento e a diferenciação celular, portanto, não mude completamente o meio. Além disso, é necessário manter o volume médio constante na diferenciação. Portanto, certifique-se de que o volume de líquido adicionado seja o menor possível ao ajustar a concentração de CHI.
- Estratégia de branqueamento de capitais na fase II: reconhecimento de CPCs comprometidos com o CM
Conjunto de dados: pares de imagens de campo claro no dia 6 e máscaras anotadas manualmente de CPCs confirmados por CM
Modelo: ResNeSt38 com Grad-CAM36 para aprendizado fracamente supervisionado- Prepare um conjunto de dados que consista em imagens de campo claro no dia 6. Anote manualmente os CPCs confirmados por CM nas imagens de campo claro rastreando as células cTnT+ nos fluxos de imagem do dia 12 até o dia 6.
- Corte as imagens de campo claro e a anotação manual correspondente (denominada máscaras) de CPCs confirmados pelo CM em patches, com cada patch dimensionado em 1/5,5 do tamanho da imagem de todo o poço. Extraia patches de maneira sobreposta em que patches adjacentes compartilham uma sobreposição de 50% no conjunto de treinamento e uma sobreposição de 75% no conjunto de teste. Rotule os patches com CPCs comprometidos com CM de ≥30% como "positivos". Rotule os patches sem CPCs confirmados pelo CM como "negativos". Mude o diretório de trabalho para "stage_II". Execute "./Crop_and_Reconstruct/mask_crop.m" para cortar as imagens. Execute "./Crop_and_Reconstruct/classification.m" para atribuir rótulos (0: negativo; 1: positivo) a patches de campo claro (Figura 3G).
- Divida aleatoriamente os patches de imagem rotulados em um conjunto de treinamento e um conjunto de teste.
- Treine uma rede neural convolucional profunda, ResNeSt38 , para aprender a classificar esses patches. Avalie o ResNeSt treinado no conjunto de teste para confirmar se o modelo demonstra uma alta precisão de classificação. Execute "train.py" e "test.py" para treinamento e teste, respectivamente (Figura 3H).
- Use o Grad-CAM36 para destacar as regiões que mais contribuem para a inferência de ResNeSt, representadas por mapas de calor. Binarize os mapas de calor (usando um limite, por exemplo, 10% × máximo) para obter as regiões CPC confirmadas por CM previstas, que são chamadas de regiões CPC reconhecidas por imagem (IR-CPC).
- Compare as regiões IR-CPC com máscaras anotadas manualmente no conjunto de teste usando exatidão, pontuação F1, precisão, recall, especificidade e interseção sobre união (IoU). Compare a proporção de regiões IR-CPC com Índices de Eficiência de Diferenciação (calculados a partir das imagens finais de fluorescência cTnT) usando o coeficiente de correlação de Pearson. Execute "./Evaluation/index_calculate.m" e "./Evaluation/Pearson_correlation.m" para avaliação de desempenho (Figura 3I).
- Aplique o modelo treinado para reconhecer CPCs para novos lotes no estágio II. Corte imagens de campo claro de poço inteiro em patches, passe os patches para o ResNeSt treinado e obtenha as regiões IR-CPC no nível do patch, que são então mescladas para produzir as regiões IR-CPC de todo o poço. Calcule a fração de regiões IR-CPC como um indicador da eficiência de diferenciação final. Purifique os IR-CPCs para reduzir a contaminação celular (descrito abaixo).
NOTA: Como o modelo de reconhecimento CPC pode fazer uma avaliação antecipada da eficiência de diferenciação final, ele também pode servir como uma leitura de alto rendimento para a triagem de pequenas moléculas que são propícias ao processo de diferenciação, o que pode otimizar ainda mais o protocolo de diferenciação35.
- Purificação seletiva por região de CPCs reconhecidos por imagem
NOTA: Os experimentos de fotoativação são baseados em um microscópio de fluorescência invertida facilitado com uma platina motorizada.- De acordo com as regiões IR-CPC previstas pelo modelo treinado, descartar os poços que não puderam gerar nenhum IR-CPCs. Para purificar esses CPCs comprometidos com CM, use uma sonda fotoativável não citotóxica, Dual-Activatable Cell Tracker 1 (DACT-1) para rotular seletivamente não CPC39 por região.
- Dissolver o DACT-1 (10 mM) em DMSO e conservá-lo a -20 °C. Incubar células do dia 6 com 1 μM DACT-1 em meio de diferenciação CM por 30 min a 37 °C.
- Inspecione as células vivas sob um microscópio e selecione a região de interesse (ROI) como polígonos no MATLAB. Desenhe linhas paralelas com espaçamento de 20 μm dentro do polígono e calcule as coordenadas dos pontos de interseção.
- Defina a pista móvel do estágio motorizado de acordo com as linhas de rastreamento, então o estágio se moveria a 0,12 mm / s para que uma linha de laser de 405 nm (20 μm de diâmetro) montada no palco pudesse escanear globalmente o ROI para fotoativar não CPCs de forma restrita. Após a irradiação, detectar as células marcadas com DACT-1 usando uma linha de laser de 561 nm.
- Dissocie as células usando tripsina-EDTA a 0,05% (diluído por PBS) a 37 ° C por 5-7 min e agite suavemente a placa em uma incubadora por 2 min. Filtrar as células através de um filtro de células de 40 μm e centrifugar a 500 × g durante 3 min. Ressuspenda as células em 0,5% de BSA e mantenha o tubo no gelo até a classificação. Preparar também uma amostra não irradiada como controlo negativo para a triagem.
- Inicie o sistema de classificação por citometria de fluxo e configure o software classificador. Escolha os lasers de comprimento de onda de 561 nm e use um filtro passa-banda de 610/20 para classificação DACT1+. Para cada amostra, use uma ferramenta de gating poligonal para circular a população de células em um gráfico bivariado de dispersão direta (FSC) e dispersão lateral (SSC) para selecionar a única célula. Em seguida, execute o controle negativo para confirmar a fluorescência de fundo, execute a amostra irradiada seletivamente por região e desenhe a porta para a população DACT1+ .
- Colete e ressuspenda as células classificadas em meio de manutenção CM com 10% de FBS e 5 μM de Y27632 e semeie as células classificadas em uma placa de 96 poços revestida com Matrigel.
- No dia seguinte, troque o meio por meio de manutenção CM e espere para colher CMs de alta pureza.
- Estratégia de ML na fase III: reconhecimento de CMs
Conjunto de dados: pares de imagens de campo claro no dia 12 e as imagens finais de fluorescência cTnT
Modelo: pix2pix37- Prepare um conjunto de dados que consista em imagens de campo claro de CMs e as imagens de fluorescência cTnT correspondentes. Para aumentar a diversidade, certifique-se de que o conjunto de dados inclua poços com várias eficiências de diferenciação e linhagens celulares.
- Divida aleatoriamente o conjunto de dados em um conjunto de treinamento e um conjunto de teste. Mude o diretório de trabalho para "stage_III". Salve as imagens de campo claro e as imagens de fluorescência cTnT correspondentes em "./pix2pix/datasets/(CM|CM_new_cell_lines)/A/(train|test)/*.png" e ".../B/(train|test)/*.png", respectivamente. Execute o seguinte comando:
CD PIX2PIX/Conjuntos de Dados
python combine_A_and_B.py --fold_A ./CM/A --fold_B ./CM/B --fold_AB ./CM/ --no_multiprocessing
CD.. - Treine o pix2pix modelo37 no conjunto de treinamento. Execute o seguinte comando (Figura 3J):
python train.py --dataroot ./datasets/CM --name brightfield2fluorescence --model pix2pix --input_nc 1 --output_nc 1 --load_size 1536 --crop_size 256 --lr 2e-4 --n_epochs 1000 --n_epochs_decay 0 --norm instance --netD n_layers --n_layers_D 1 --batch_size 16 --direction AtoB --save_epoch_freq 100 --dataset_mode alinhado --use_resize_conv --seed 1234 - Aplique o modelo pix2pix treinado no conjunto de teste. Execute o seguinte comando:
python test.py --dataroot ./datasets/CM --name brightfield2fluorescence --model pix2pix --direction AtoB --input_nc 1 --output_nc 1 --load_size 1536 --crop_size 1536 --use_resize_conv --eval --num_test 1000 - Compare as imagens de fluorescência previstas pelo modelo com as verdadeiras (ou seja, obtidas experimentalmente). No nível do pixel, compare as intensidades previstas e as intensidades reais usando o coeficiente de correlação de Pearson. Além disso, no nível da imagem, compare os Índices de Eficiência de Diferenciação previstos com os verdadeiros usando o coeficiente de correlação de Pearson. Execute "stage_III/evaluation/pixel_correlation.m" e "image_correlation.ipynb" para essas comparações (Figura 3K,L).
- Aplique o modelo treinado para reconhecer CMs em novos lotes no estágio III. Passe novas imagens de campo claro de CMs para o modelo pix2pix treinado e obtenha uma previsão in-silico das imagens de fluorescência e eficiência de diferenciação.
NOTA: Para reduzir a contaminação celular, a classificação de células fotoativadas baseada em DACT-1 também é aplicável para purificar CMs reconhecidos pelo modelo.
Resultados
Com base em imagens de campo claro e ML, o processo geral de diferenciação pode ser monitorado e otimizado de forma inteligente. No estágio PSC, desenvolvemos um modelo ML que poderia prever a eficiência de diferenciação final de acordo com as características morfológicas das colônias iniciais de PSC, para determinar o ponto de tempo mais adequado ou apropriado para iniciar a diferenciação (Figura 4A, B). A eficiência de diferenc...
Discussão
Aqui, descrevemos um protocolo detalhado para superar um dos principais problemas na aplicação e tradução atuais do PSC - a variabilidade na diferenciação celular. Ao aproveitar a imagem de campo claro de células vivas e ML, otimizamos iterativamente a diferenciação de PSC para alcançar uma eficiência consistentemente alta em linhas e lotes celulares. No entanto, no processo de diferenciação acima, várias etapas críticas no protocolo têm uma influência decisiva sobre se ...
Divulgações
Yang Zhao, Jue Zhang, Xiaochun Yang, Yao Wang e Daichao Chen estão registrando uma patente para a estratégia de diferenciação PSC relatada neste artigo (202210525166.X).
Agradecimentos
Agradecemos a Qiushi Sun, Yao Wang, Yu Xia, Jinyu Yang, Chang Lin, Zimu Cen, Dongdong Liang, Rong Wei, Ze Xu, Guangyin Xi, Gang Xue, Can Ye, Li-Peng Wang, Peng Zou, Shi-Qiang Wang, Pablo Rivera-Fuentes, Salome Püntener, Zhixing Chen, Yi Liu e Jue Zhang, por estabelecerem as bases desta estratégia. Este trabalho foi apoiado pelo Programa Nacional de P&D da China (2018YFA0800504, 2019YFA0110000) e pelo Projeto de Experimentos Médicos Espaciais do Programa Espacial Tripulado da China (HYZHXM01020) para Yang Zhao. A Figura 1 foi criada com BioRender.com.
Materiais
| Name | Company | Catalog Number | Comments |
| 0.25% Trypsin-EDTA | Gibco | 25200056 | Diluted digests were used for CPC and CM digestion |
| 4% Paraformaldehyde in PBS | KeyGEN BioTECH | KGIHC016 | |
| 6-well Cell Culture Plate | NEST | 703001 | |
| 96-well Cell Culture Plate | NEST | 701001 | |
| B27 Supplement | Gibco | 17504044 | |
| B27 Supplement Minus Insulin | Gibco | A1895601 | |
| Bovine serum albumin (BSA) | GPC BIOTECH | AA904-100G | |
| Celldiscoverer 7 | Zeiss | Instruments used to take bright-field images throughout differentiation and final cTnT images | |
| CHIR99021 | Selleck | S1263 | |
| DMEM/F12 | Gibco | 12634010 | |
| Donkey anti-Mouse IgG (H+L) Highly Cross-Adsorbed Secondary Antibody, Alexa Fluor 488 | Thermo | A-21202 | Secondary Antibody |
| FACSAria III | BD Biosciences | Flow cytometry sorter | |
| Fetal Bovine Serum (FBS) | VISTECH | SE100-B | |
| Hoechst 33342 | YEASEN | 40732ES03 | |
| Human Pluripotent Stem Cell Chemical-defined Medium | Cauliscell Inc | 400105 | Basal medium of PSC preparation medium |
| iPS-18 | TaKaRa | Y00300 | |
| iPS-B1 | Cellapy | CA4025106 | |
| iPS-F | Nuwacell | RC01001-B | |
| iPS-M | Nuwacell | RC01001-A | |
| IWR1-1-endo | Selleck | S7086 | IWR1 |
| Jupyter Notebook | N/A | Version 6.4.0 | https://jupyter.org/ |
| MATLAB | MathWorks | Version R2020a | Software for scientific computation and image annotation |
| Matrigel Matrix | Corning | 354230 | Matrigel |
| Mouse monoclonal IgG1 anti-cTnT | Thermo | MA5-12960 | cTnT primary antibody |
| Normal Donkey Serum | Jackson | 017-000-121 | |
| ORCA-Flash 4.0 V3 digital CMOS camera | Hamamatsu | C13440-20CU | The digital camera assembled on Celldiscoverer7 |
| PBS | NEB | 21-040-CVR | |
| Penicillin-Streptomycin | Gibco | 15140-122 | |
| Pluripotency Growth Mater 1 basal medium | Cellapy | CA1007500-1 | Basal medium of PSC culture medium |
| Pluripotency Growth Mater 1 supplement | Cellapy | CA1007500-2 | Supplement of PSC culture medium |
| Prism | Graphpad | Version 8/9 | Statistical software for statistical analysis and plotting |
| Python | N/A | version 3.6 | Python 3 environment for scientific computation, with packages pytorch (1.9.0), numpy, scipy, pandas, visdom, scikit-learn, scikit-image, opencv-python, and matplotlib software for scientific computation and image annotation. |
| RPMI 1640 | Gibco | 11875176 | |
| Supplement hPSC-CDM (500x) | Cauliscell Inc | 00015 | Supplement of PSC preparation medium |
| TiE | Nikon | An inverted fluorescence microscope (with modification) for region-selevtive purification | |
| Triton X-100 | Amresco | 9002-93-1 | |
| Versene Solution | Thermo | 15040066 | EDTA solution for PSC digestion |
| Y27632 | Selleck | S6390 | |
| Zen | Zeiss | Version 3.1 | A supporting software of Celldiscoverer7 for image acquisition, processing and analysis |
Referências
- Yoshida, Y., Yamanaka, S. Induced pluripotent stem cells 10 years later: for cardiac applications. Circ Res. 120 (12), 1958-1968 (2017).
- Shi, Y., Inoue, H., Wu, J. C., Yamanaka, S. Induced pluripotent stem cell technology: a decade of progress. Nat Rev Drug Discov. 16 (2), 115-130 (2017).
- Takahashi, K., et al. Induction of pluripotent stem cells from adult human fibroblasts by defined factors. Cell. 131 (5), 861-872 (2007).
- Burridge, P. W., et al. Chemically defined generation of human cardiomyocytes. Nat Methods. 11 (8), 855-860 (2014).
- Mordwinkin, N. M., Burridge, P. W., Wu, J. C. A review of human pluripotent stem cell-derived cardiomyocytes for high-throughput drug discovery, cardiotoxicity screening, and publication standards. J Cardiovasc Transl Res. 6 (1), 22-30 (2013).
- Vanden Berg, C. W., Elliott, D. A., Braam, S. R., Mummery, C. L., Davis, R. P. Differentiation of human pluripotent stem cells to cardiomyocytes under defined conditions. Methods Mol Biol. 1353, 163-180 (2016).
- Burridge, P. W., Holmström, A., Wu, J. C. Chemically defined culture and cardiomyocyte differentiation of human pluripotent stem cells. Curr Protoc Hum Genet. 87, 1-15 (2015).
- Li, J., et al. Human pluripotent stem cell-derived cardiac tissue-like constructs for repairing the infarcted myocardium. Stem Cell Rep. 9 (5), 1546-1559 (2017).
- Smith, A. S. T., Macadangdang, J., Leung, W., Laflamme, M. A., Kim, D. -. H. Human iPSC-derived cardiomyocytes and tissue engineering strategies for disease modeling and drug screening. Biotechnol Adv. 35 (1), 77-94 (2017).
- Sharma, A., et al. Use of human induced pluripotent stem cell-derived cardiomyocytes to assess drug cardiotoxicity. Nat Protoc. 13 (12), 3018-3041 (2018).
- Mehta, A., et al. Identification of a targeted and testable antiarrhythmic therapy for long-QT syndrome type 2 using a patient-specific cellular model. Eur Heart J. 39 (16), 1446-1455 (2018).
- Cahan, P., Daley, G. Q. Origins and implications of pluripotent stem cell variability and heterogeneity. Nat Rev Mol Cell Biol. 14 (6), 357-368 (2013).
- Kim, K., et al. Epigenetic memory in induced pluripotent stem cells. Nature. 467 (7313), 285-290 (2010).
- Rouhani, F., et al. Genetic background drives transcriptional variation in human induced pluripotent stem cells. PLoS Genet. 10 (6), e1004432 (2014).
- Kim, K., et al. Donor cell type can influence the epigenome and differentiation potential of human induced pluripotent stem cells. Nat Biotechnol. 29 (12), 1117-1119 (2011).
- Garitaonandia, I., et al. Increased risk of genetic and epigenetic instability in human embryonic stem cells associated with specific culture conditions. PLoS One. 10 (2), e0118307 (2015).
- Engler, A. J., Sen, S., Sweeney, H. L., Discher, D. E. Matrix elasticity directs stem cell lineage specification. Cell. 126 (4), 677-689 (2006).
- Matoba, N., Yamashita, T., Takayama, K., Sakurai, F., Mizuguchi, H. Optimal human iPS cell culture method for efficient hepatic differentiation. Differentiation. 104, 13-21 (2018).
- Volpato, V., et al. Reproducibility of molecular phenotypes after long-term differentiation to human iPSC-derived neurons: A multi-site omics study. Stem Cell Rep. 11 (4), 897-911 (2018).
- Anderson, N. C., et al. Balancing serendipity and reproducibility: Pluripotent stem cells as experimental systems for intellectual and developmental disorders. Stem Cell Rep. 16 (6), 1446-1457 (2021).
- Sepac, A., et al. Comparison of cardiomyogenic potential among human ESC and iPSC lines. Cell Transplant. 21 (11), 2523-2530 (2012).
- Laco, F., et al. Unraveling the inconsistencies of cardiac differentiation efficiency induced by the GSK3β inhibitor CHIR99021 in human pluripotent stem cells. Stem Cell Rep. 10 (6), 1851-1866 (2018).
- Volpato, V., Webber, C. Addressing variability in iPSC-derived models of human disease: guidelines to promote reproducibility. Dis Model Mech. 13 (1), 042317 (2020).
- Hu, B. -. Y., et al. Neural differentiation of human induced pluripotent stem cells follows developmental principles but with variable potency. Proc Natl Acad Sci USA. 107 (9), 4335-4340 (2010).
- Huo, J., et al. Evaluation of batch variations in induced pluripotent stem cell-derived human cardiomyocytes from 2 major suppliers. Toxicol Sci. 156 (1), 25-38 (2017).
- Rostam, H. M., Reynolds, P. M., Alexander, M. R., Gadegaard, N., Ghaemmaghami, A. M. Image based machine learning for identification of macrophage subsets. Sci Rep. 7 (1), 3521 (2017).
- Christiansen, E. M., et al. In silico labeling: Predicting fluorescent labels in unlabeled images. Cell. 173 (3), 792-803 (2018).
- Buggenthin, F., et al. Prospective identification of hematopoietic lineage choice by deep learning. Nat Methods. 14 (4), 403-406 (2017).
- Smith, Z. D., Nachman, I., Regev, A., Meissner, A. Dynamic single-cell imaging of direct reprogramming reveals an early specifying event. Nat Biotechnol. 28 (5), 521-526 (2010).
- Fan, K., Zhang, S., Zhang, Y., Lu, J., Holcombe, M., Zhang, X. A machine learning assisted, label-free, non-invasive approach for somatic reprogramming in induced pluripotent stem cell colony formation detection and prediction. Sci Rep. 7 (1), 13496 (2017).
- Kusumoto, D., et al. Automated deep learning-based system to identify endothelial cells derived from induced pluripotent stem cells. Stem Cell Rep. 10 (6), 1687-1695 (2018).
- Zhu, Y., et al. Deep learning-based predictive identification of neural stem cell differentiation. Nat Commun. 12 (1), 2614 (2021).
- Qian, T., et al. Label-free imaging for quality control of cardiomyocyte differentiation. Nat Commun. 12 (1), 4580 (2021).
- Ounkomol, C., Seshamani, S., Maleckar, M. M., Collman, F., Johnson, G. R. Label-free prediction of three-dimensional fluorescence images from transmitted-light microscopy. Nat Methods. 15 (11), 917-920 (2018).
- Yang, X., et al. A live-cell image-based machine learning strategy for reducing variability in PSC differentiation systems. Cell Discovery. 9 (1), 53 (2023).
- Selvaraju, R. R., et al. Grad-CAM: Visual explanations from deep networks via gradient-based localization. Proc IEEE Int Conf Computer Vision (ICCV). , 618-626 (2017).
- Isola, P., Zhu, J. -. Y., Zhou, T., Efros, A. A. Image-to-image translation with conditional adversarial networks. Proc IEEE Conf Computer Vision Pattern Recognition (CVPR). , 5967-5976 (2017).
- Zhang, H., et al. ResNeSt: Split-attention networks. Proc IEEE/CVF Conf Computer Vision Pattern Recognition Workshops (CVPRW). , 2735-2745 (2022).
- Halabi, E. A., et al. Dual-activatable cell tracker for controlled and prolonged single-cell labeling). ACS Chem. Biol. 15 (6), 1613-1620 (2020).
- Hannan, N. R. F., Segeritz, C. -. P., Touboul, T., Vallier, L. Production of hepatocyte-like cells from human pluripotent stem cells. Nat Protoc. 8 (2), 430-437 (2013).
- Zhang, H., Shen, M., Wu, J. C. Generation of quiescent cardiac fibroblasts derived from human induced pluripotent stem cells. Methods Mol Biol. 2454, 109-115 (2022).
- Harding, M. J., McGraw, H. F., Nechiporuk, A. The roles and regulation of multicellular rosette structures during morphogenesis. Development. 141 (13), 2549-2558 (2014).
- Najm, F. J., et al. Rapid and robust generation of functional oligodendrocyte progenitor cells from epiblast stem cells. Nat Methods. 8 (11), 957-962 (2011).
- Hogrebe, N. J., Maxwell, K. G., Augsornworawat, P., Millman, J. R. Generation of insulin-producing pancreatic β cells from multiple human stem cell lines. Nat Protoc. 16 (9), 4109-4143 (2021).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados