Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Стратегия машинного обучения на основе изображений живых клеток для мониторинга дифференцировки плюрипотентных стволовых клеток
* Эти авторы внесли равный вклад
В этой статье
Резюме
Доступные системы дифференцировки плюрипотентных стволовых клеток (ПСК) в функциональные клетки в настоящее время затруднены из-за проблем серьезной вариабельности от линии к линии и от партии к партии. Здесь, используя кардиальную дифференцировку в качестве основного примера, мы представляем протокол для интеллектуального мониторинга и модуляции процесса дифференцировки ПСК на основе машинного обучения на основе изображений.
Аннотация
Технологии плюрипотентных стволовых клеток (ПСК) широко используются в разработке лекарств, моделировании заболеваний и регенеративной медицине. Тем не менее, существующие системы дифференцировки ПСХ в функциональные клетки затруднены проблемами серьезной вариабельности от линии к линии и от партии к партии. Поэтому важен точный контроль дифференцировки клеток в режиме реального времени. В этом протоколе мы описываем неинвазивную и интеллектуальную стратегию, которая преодолевает вариабельность дифференцировки клеток с помощью машинного обучения на основе изображений в ярком поле. Взяв в качестве примера дифференцировку ПСХ в кардиомиоциты, данная методология предоставляет подробную информацию для контроля исходного состояния ПСК, ранней оценки и вмешательства в условиях дифференцировки, а также устранения недифференцированной клеточной контаминации, что в совокупности обеспечивает стабильно высокое качество дифференцировки от ПСК к функциональным клеткам. В принципе, эта стратегия может быть распространена на другие системы дифференцировки или перепрограммирования клеток с несколькими ступенями для поддержки производства клеток, а также для углубления нашего понимания механизмов трансформации клеточной судьбы.
Введение
Плюрипотентные стволовые клетки (ПСК) обладают замечательной способностью дифференцироваться во многие типы клеток in vitro. Эти дифференцированные функциональные клетки могут быть использованы для клеточной терапии, моделирования заболеваний и разработки лекарств, и все они представляют ценность для исследований или клинического применения 1,2,3. Например, были разработаны различные методы дифференцировки ПСК в кардиомиоциты (КМ)4,5,6,7. Эти КМ могут быть применены для тестирования кардиотоксичности лекарственных препаратов, моделирования сердечных заболеваний и трансплантации клеток 8,9,10,11.
Тем не менее, превращение ПСК в терминальные дифференцированные клетки является ступенчатым процессом, и множественные возмущения в процессе дифференцировки могут привести клетки к дивергентной клеточной судьбе. Различные генетические предпосылки и эпигенетические признаки линий ПСК влияют на потенциал дифференцировки в конкретную линию 12,13,14,15; количество пассажей ПСК и накопленные генные мутации также являются источниками гетерогенности ПСХ; Различия в экспериментальных операциях, применяемых разными экспериментаторами, также могут привести к совершенно разным результатам дифференцировки 16,17,18,19,20. Таким образом, в настоящее время одной из основных проблем в производстве клеток, полученных из ПСХ, является нестабильность между клеточными линиями и партиями 21,22,23,24,25. Нестабильность дифференцировки ПСК часто приводит к многократным повторным экспериментам, отнимающим значительные временные и трудовые ресурсы. Для решения этой проблемы крайне важно разработать стратегию, которая сводит к минимуму изменчивость между клеточными линиями и партиями, тем самым повышая стабильность и надежность дифференцировки.
В последнее время достижения в области микроскопии высокого разрешения и машинного обучения (ML) облегчили применение количественного анализа изображений на основе ML в клеточной биологии, что сделало возможным использование ценной информации в функциях визуализации клеток 26,27,28,29,30,31,32,33,34. В нашей предыдущей работе мы предложили стратегию машинного обучения на основе изображений живых клеток для мониторинга и вмешательства в состояние дифференцировки ПСК в режиме реального времени для повышения стабильности и эффективности дифференцировки ПСХ (Рисунок 1)35. Взяв в качестве примера дифференцировку ПСХ в кардиомиоциты, мы оценили исходное состояние ПСХ с помощью моделей случайного леса, предсказали оптимальное условие дифференцировки с помощью моделей логистической регрессии и распознали успешно дифференцированные клетки с помощью Grad-CAM36 и pix2pix37 на основе глубокого обучения. Модели машинного обучения научились идентифицировать клеточные линии по целому ряду морфологических признаков яркого поля, включая особенности площади, окружности, выпуклости, плотности, яркости, скорости движения и другие неявные особенности, извлеченные глубокими сверточными нейронными сетями. Основываясь на выводах из этих установленных моделей машинного обучения, мы реализовали контроль исходного состояния ПСХ, раннюю оценку и вмешательство в условиях дифференцировки, а также устранение неправильно дифференцированной клеточной контаминации, в совокупности обеспечив всестороннюю и точную модуляцию процесса дифференцировки сердца. Здесь мы приводим пошаговый протокол разработки стратегии.
протокол
1. Дифференцировка и характеристика клеток
- Приготовление культуральных реактивов и культуральных планшетов
- Питательную среду ПСК приготовить, добавив 2 мл добавки и 0,2% пенициллин-стрептомицина к 48 мл базальной среды. Аликвотируйте и храните добавку при температуре -20 °C. Храните эту среду при температуре 4 °C до 4 недель.
- Приготовьте среду для приготовления ПСК путем добавления 1 мл добавки и 0,2% пенициллин-стрептомицина на 500 мл базальной среды. При использовании подогрейте среду для одноразового использования и храните среду при температуре 4 °C до 3 недель.
- Приготовьте среду для дифференцировки КМ, добавив 1x добавку B27 за вычетом инсулина и 1% пенициллин-стрептомицина в RPMI 1640. Приготовьте поддерживающую среду CM, добавив 1x добавку B27 и 1% пенициллин-стрептомицин в RPMI 1640. Храните эти два вида среды при температуре 4 °C в течение 2 недель.
- Погрузите Matrigel в лед и держите его при температуре 4 °C в течение ночи. Следите за тем, чтобы Матригель полностью разморозился; затем аликвоту в 1 мл аликвот, хранящуюся при -80 °C для последующего использования, и добавьте 1 мл Matrigel в 49 мл DMEM/F12 при 4 °C для приготовления рабочего раствора Matrigel. Добавьте 850 мкл этого рабочего раствора Matrigel в лунку 6-луночного культурального планшета и инкубируйте планшет при 37 °C в течение не менее 30 минут. Для 96-луночного планшета, используемого для дифференцировки CM, добавьте 35 мкл рабочего раствора Matrigel на лунку.
ПРИМЕЧАНИЕ: Всегда держите Матригель на льду; наконечники и трубки для пипеток должны быть предварительно охлаждены до 4 °C. Рабочий раствор Matrigel можно хранить при температуре 4 °C в течение 2 недель. Будьте осторожны и избегайте образования пузырей при пипетировании рабочего раствора Матригель.
- Техническое обслуживание и проход ЧОП
- Поддерживайте ПСК в 6-луночных культуральных планшетах. Подтвердите плотность клеток под микроскопом и подготовьтесь к прохождению, когда слияние достигнет ~70% (рисунок 2A).
- Используйте ЭДТА для переваривания клеток для пассирования. Нагрейте питательную среду PSC, PBS и EDTA на водяной бане при температуре 37 °C и добавьте Y27632 в среду до конечной концентрации 5 мкМ.
- Промойте ПСК 1 мл PBS, добавьте 1 мл ЭДТА на лунку и инкубируйте планшет в инкубаторе при температуре 37 °C в течение 3 минут.
- Отсадите ЭДТА до того, как клетки отделятся от культуральной пластины, затем используйте 1 мл культуральной среды PSC, чтобы осторожно промыть дно лунки 3-4 раза для ресуспендирования клеток.
ПРИМЕЧАНИЕ: Это позволяет нам пропустить этап центрифугирования и уменьшить повреждение клеток при операциях пассирования. - Аспирируйте DMEM/F12 из лунки с покрытием Matrigel из 6-луночного культурального планшета и добавьте 2 мл свежей питательной среды PSC в каждую лунку (добавлен Y27632). Добавьте суспензию ячеек в лунки с соотношением разъемов от 1:6 до 1:12. Поместите планшет в инкубатор с температурой 37 °C и 5%CO2.
- Смените питательную среду PSC (без Y27632) через 12–24 ч после пассирования; Затем смените среду и проверяйте состояние клеток и слияние каждый день.
- Кардиальная дифференцировка ПСК
- Для дифференцировки CM засейте PSC в 96-луночный культуральный планшет в среду для подготовки PSC (добавлен Y27632). Для дифференциации КМ выполните те же действия, описанные выше (шаги 1.2.2-1.2.6), чтобы засеять ПСК в 96-луночный культуральный планшет в среду для приготовления ПСК (добавлен Y27632).
Примечание: На данном этапе продолжительность лечения ЭДТА может быть соответствующим образом увеличена (5 минут или более) для достаточной диссоциации ПСК таким образом, чтобы клоны могли быть меньше после пассажа. Наше исследование показало, что в пределах одной и той же территории небольшие колонии способствуют дифференциации. - Стадия I: Когда ПСК достигают 80-90% конфлюенции, переключите среду на среду дифференцировки СМ с 2-20 мкМ CHIR99021 (CHIR) (Рисунок 2B). Временная точка переключения среды называется днем 0. После 24-48 ч обработки CHIR смените среду на свежую среду для дифференцировки CM.
ПРИМЕЧАНИЕ: CHIR является ингибитором GSK3beta для индукции мезодермы. Оптимальная доза CHIR для дифференцировки КМ колеблется в зависимости от различных клеточных линий и партий. Например, оптимальная доза CHIR составляет 8-10 мкМ, 48 ч для iPS-B1. Чтобы собрать различные изображения в светлом поле для обучения ML-модели, мы титровали концентрации CHIR для каждой партии экспериментов для различной эффективности дифференцировки. - Этап II: Через 72 ч (день 3) замените среду средой для дифференцировки CM с добавлением 5 мкМ IWR1 и культурой на 48 ч (день 5). Смену среды с дифференцировочной средой СМ проводят в течение 1-2 дней (до 6-7 дней). На этом этапе ПСК дифференцируются в сердечные клетки-предшественники (КПК).
- Этап III: Замените среду на среду для технического обслуживания CM и заменяйте среду каждые 3 дня. На 10-й или 12-й день соберите КМ для дальнейшего анализа.
- Для дифференцировки CM засейте PSC в 96-луночный культуральный планшет в среду для подготовки PSC (добавлен Y27632). Для дифференциации КМ выполните те же действия, описанные выше (шаги 1.2.2-1.2.6), чтобы засеять ПСК в 96-луночный культуральный планшет в среду для приготовления ПСК (добавлен Y27632).
- Иммунофлуоресцентное окрашивание КМ
- На 10-й или 12-й день промойте клетки PBS и зафиксируйте их 4% параформальдегидом в PBS на 15 минут при комнатной температуре. Промойте CM 2x с PBS и храните пластину при температуре 4 °C до ~1 недели.
ПРИМЕЧАНИЕ: Параформальдегид является токсичным реагентом с резким запахом. Работайте в капюшоне с химическими веществами и защищайте глаза и руки. - Во время окрашивания клетки обработать проникающим раствором (0,1% Triton X-100 в PBS) в течение 15 мин при комнатной температуре; затем инкубировать клетки в блокирующем растворе (0,1% Triton X-100, содержащем 3% сыворотку Donkey Serum в PBS) в течение 30 минут при комнатной температуре.
- Инкубируйте образец с первичным антителом к сердечному тропонину Т (cTnT) (1:300), разведенным в блокирующем растворе в течение ночи при 4 °C, чтобы идентифицировать КМ.
- Соберите первичное антитело и промойте клетки PBS 3x. Инкубируют образец со вторичными антителами в PBS с 1% бычьим сывороточным альбумином в течение 1 ч при 37 °C в темноте.
ПРИМЕЧАНИЕ: Собранное первичное антитело можно повторно использовать 2-3 раза в течение 1 недели.
- Соберите первичное антитело и промойте клетки PBS 3x. Инкубируют образец со вторичными антителами в PBS с 1% бычьим сывороточным альбумином в течение 1 ч при 37 °C в темноте.
- Удалите вторичное антитело из клеток и промойте клетки 3 раза PBS. Используйте Hoechst 33342 (1:1,000 в PBS) для окрашивания ядер в течение 5 минут при комнатной температуре. Промойте ячейку 3 раза с помощью PBS и добавьте 100 μL PBS на лунку, чтобы избежать высыхания. Храните пластину при температуре 4 °C в темном месте и получайте флуоресцентные изображения в течение 1 недели.
- На 10-й или 12-й день промойте клетки PBS и зафиксируйте их 4% параформальдегидом в PBS на 15 минут при комнатной температуре. Промойте CM 2x с PBS и храните пластину при температуре 4 °C до ~1 недели.
2. Получение потока изображений на протяжении всего процесса дифференциации
- Настройка экспериментального дизайна для сбора изображений
- Используйте автоматизированный микроскоп, поддерживающий культуру живых клеток и визуализацию, для получения изображений в светлом поле различных стадий дифференцировки КМ и иммунофлуоресцентных изображений результата дифференцировки cTnT (рис. 2A-E). Используйте программное обеспечение, поддерживающее микроскоп, для разработки программы эксперимента и управления микроскопом.
- Откройте программу и создайте новый экспериментальный дизайн. Выберите объектив с 5-кратным увеличением и объектив с 2-кратным увеличением для визуализации. Отметьте опции Тайлы и Z-стек.
- Добавьте TL Brightfield channel для получения изображений светлого поля в меню «Каналы». Добавьте каналы AF488 и H3342 для иммунофлуоресцентной визуализации. Измените траекторию света в меню «Настройка изображения» для получения изображений с помощью внешней цифровой КМОП-камеры.
- Откройте меню «Режим захвата» и установите группирование 2 x 2 для увеличения отношения сигнал/шум.
- Откройте меню Z-стек и выберите модель «Центр». Задайте количество срезов и интервалов при сканировании. Выберите 3–5 срезов с интервалом 3–6 мкм для получения изображений в фокусе при анализе.
- В окне «Навигация и плитки» настройте области плитки по Carrier и установите 25 плиток (5 столбцов x 5 строк) для одной ячейки. В меню «Мозаика» выберите шаблон Multiwell 96 в Sample Carrier и установите перекрытие 5%-15% на смежных областях.
- Если требуется непрерывное изображение, установите флажок Time series и установите продолжительность и интервал в меню Time series. Для сбора потока изображений на этапе I установите продолжительность 12 часов без интервалов.
- Получение светлопольных и иммунофлуоресцентных изображений
- Для получения изображений сначала поместите планшет для культивирования клеток в лоток для образцов и загрузите образец внутрь микроскопа. Выберите шаблон Multiwell 96 в соответствии с планом эксперимента. Если образец состоит из живых клеток, откройте систему нагрева и насосCO2 , чтобы поддерживать соответствующие условия для культивирования (37 °C, 5%CO2).
- Откройте предустановленный экспериментальный проект и сохраните его как новый. Откройте меню «Плитки» и откалибруйте положение пластины вручную. В окне «Навигация и мозаика» выберите нужные скважины и нажмите кнопку «Создать», чтобы создать области мозаики для этих скважин.
- Щелкните Проверить области мозаики в меню Мозаики и запустите автофокусировку, чтобы проверить все скважины. Затем вручную корректируйте фокусировку каждой лунки под светлым полем, прокручивая колесико мыши.
- Отметьте галочками нужные каналы и установите время экспонирования в меню «Каналы». Рекомендуемое время экспозиции составляет 1,625 мс для яркого освещения, 100 мс для H3342 и 550 мс для AF488. Выполните один снимок, чтобы проверить изображения всех каналов.
- Наконец, нажмите кнопку «Начать эксперимент» и дождитесь автоматического создания образа. Как правило, требуется ~1,2 часа, чтобы закончить сканирование целой 96-луночной культуральной пластины (96 лунок, три слоя, 25 плиток для каждой лунки под светлым полем). В среде обработки выберите «Экспорт изображений», выберите тип файла в формате TIFF или PNG в несжатом формате и примените файл. Для дальнейшего анализа используйте изображения 9 плиток (3 x 3) в центре колодца (Рисунок 2A-E).
3. Создание ML-стратегии на основе изображений на каждом этапе процесса дифференциации
- Подготовка программного обеспечения
- Установите среду Python с пакетами pytorch (1.9.0), numpy, scipy, scikit-learn, pandas, visdom, scikit-image, opencv-python и matplotlib.
- Установите Jupyter Notebook.
- Установите MATLAB.
- Скачайте код и примеры наборов данных из https://github.com/zhaoyanglab/ML-for-PSC-differentiation.
ПРИМЕЧАНИЕ: Производительность машинного обучения зависит от качества наборов данных и выбора моделей и гиперпараметров. Не гарантируется, что пример реализации будет оптимальным для всех обстоятельств. Инженеры машинного обучения должны настроить код и адаптировать модель и гиперпараметры под свои конкретные нужды.
- ML-стратегия на стадии PSC: контроль исходных состояний колоний PSC
Набор данных: пары светлопольных изображений через 0 ч (до обработки CHIR) и итоговая эффективность дифференцировки
Модель: случайный лес- Подготовьте набор данных, состоящий из светлопольных изображений через 0 ч (до обработки CHIR) и окончательных флуоресцентных изображений cTnT. Чтобы увеличить разнообразие морфологических профилей светлых изображений с точностью до 0 часов, убедитесь, что набор данных включает колонии ПСХ из разных клеточных линий, при этом дифференцировка начинается в течение временного интервала после пассажа. Обработка скважин в датасете с оптимальными условиями CHIR на I этапе.
ПРИМЕЧАНИЕ: Чтобы обеспечить обработку колоний в оптимальных условиях CHIR, лунки в пределах одной партии могут быть обработаны титруемыми дозами CHIR. В набор данных включаются только скважины с высокоэффективными условиями CHIR. - Количественно оцените эффективность дифференцировки каждой лунки с помощью индекса эффективности дифференцировки, вычисленного на основе флуоресцентных изображений cTnT, который определяется как
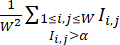 (где
(где 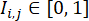 представляет интенсивность флуоресценции в месте пикселя (i, j) на флуоресцентном изображении W × W, и сумма берется по всем местам расположения пикселей с интенсивностью выше порогового α, обычно устанавливается на 0,5). В каждой клеточной линии вычислите максимальный индекс эффективности дифференцировки по всем лункам в этой клеточной линии и нормализуйте их индексы эффективности дифференцировки на максимальное значение.
представляет интенсивность флуоресценции в месте пикселя (i, j) на флуоресцентном изображении W × W, и сумма берется по всем местам расположения пикселей с интенсивностью выше порогового α, обычно устанавливается на 0,5). В каждой клеточной линии вычислите максимальный индекс эффективности дифференцировки по всем лункам в этой клеточной линии и нормализуйте их индексы эффективности дифференцировки на максимальное значение.
Примечание: Поскольку потенциал дифференцировки каждой клеточной линии по своей природе различен, такая нормализация будет больше акцентировать внимание на морфологических особенностях колоний, общих для различных клеточных линий, которые способствуют результату дифференцировки. Нормализация действительно делает колонии ПСК из разных клеточных линий сопоставимыми и смягчает смещение, вызванное клеточными линиями. - Количественно оцените морфологические профили изображений светлого поля 0 ч с помощью многомерных признаков, которые раскрывают свойства формы колонии. Чтобы очертить области колоний на изображениях в светлом поле, вычислите локальную энтропию (с радиусом r); Пиксели с локальной энтропией, превышающей пороговое σ, идентифицируются по ячейкам, покрытым ячейками. Используйте параметр r = 10 пикселей и σ = 3. Переключите рабочий каталог на "./colony_control"; сохраняйте изображения светлого поля, двоичные изображения областей клеток и флуоресцентные изображения cTnT в "colony_control/ image_data/CD00-*/[brightfield|cell_region|ctnt]/S*.png"; и запустите Jupyter Notebook "Compute_features.ipynb" для вычисления особенностей, связанных с площадью колонии, окружностью, плотностью, выпуклостью, круговостью и расстояниями до центроидных контуров, среди прочего.
- Случайным образом разделите набор данных на обучающий и тестовый наборы, при этом обучающий набор должен составлять от 50 % до 80 % от общего объема данных. Запустите последний блок: Разделите набор данных в Jupyter Notebook, чтобы разделить набор данных (рис. 3A).
- Обучите модель регрессии случайного леса на обучающем наборе для прогнозирования эффективности дифференцировки по объектам светлого поля 0 часов. Запустите записную книжку Jupyter Notebook "Machine_learning.ipynb" для обучения и тестирования модели.
- Оцените обученную модель случайного леса на тестовом наборе. Подтвердите, что прогнозируемая эффективность дифференцировки сильно коррелирует с истинной, используя коэффициент корреляции Пирсона (рис. 3B).
ПРИМЕЧАНИЕ: Поскольку тестовый набор не участвует в фазе обучения, высокая согласованность между прогнозируемой и истинной эффективностью дифференцировки на тестовом наборе указывает на то, что модель может хорошо обобщать на невидимых выборках. - Получите важность признаков из обученной модели случайного леса и выберите 8 признаков с наибольшей важностью. Визуализируйте их связь с итоговой эффективностью дифференцирования, разделив диапазон признаков на ячейки и вычислив среднюю эффективность дифференцировки в каждой ячейке. Запустите записную книжку Jupyter Notebook "Feature_importance.ipynb" для выполнения визуализации (рис. 3C).
ПРИМЕЧАНИЕ: Визуализация может помочь понять, как изменения в форме колонии ПСК способствуют дифференцировке. - Применяйте обученную модель в новых пакетах для мониторинга состояний колонии PSC в режиме реального времени. Захватывайте светлопольное изображение каждый час, передавайте изображение обученной модели и получайте прогнозируемую эффективность дифференцировки. Как только прогнозируемая эффективность превысит пороговое значение (например, 50%), начните процесс дифференциации. Если прогнозируемая эффективность не смогла достичь порогового значения, отбраковывайте эти образцы.
ПРИМЕЧАНИЕ: На стадии ПСК дифференцировочный потенциал колоний ПСК сначала повышается, а затем снижается. Мониторинг в режиме реального времени позволяет экспериментаторам определить наиболее благоприятный момент времени для начала дифференцировки.
- Подготовьте набор данных, состоящий из светлопольных изображений через 0 ч (до обработки CHIR) и окончательных флуоресцентных изображений cTnT. Чтобы увеличить разнообразие морфологических профилей светлых изображений с точностью до 0 часов, убедитесь, что набор данных включает колонии ПСХ из разных клеточных линий, при этом дифференцировка начинается в течение временного интервала после пассажа. Обработка скважин в датасете с оптимальными условиями CHIR на I этапе.
- Стратегия ML на I этапе: ранняя оценка и корректировка доз CHIR
Набор данных: пары потоков светлопольных изображений через 0-12 часов и метки концентрации CHIR (низкая/оптимальная/высокая)
Модель: логистическая регрессия- Подготовьте набор данных, состоящий из полнолуночных потоков изображений в светлом поле, где изображения собираются каждый час в период от 0 до 12 часов. Регистрируйте дозы CHIR (т.е. комбинации концентраций и продолжительности CHIR) в каждой скважине. Убедитесь, что набор данных включает различные партии и лунки с титрованными дозами CHIR (например, концентрация = 2, 4, 6, 8, 10 и 12 мкМ; продолжительность = 24, 36 и 48 ч) для увеличения разнообразия. Переключите рабочий каталог на "./stage_I"; сохранить изображения как "./data/image/CD01-*/S*/T*.png", где CD01-* (CD01-1, CD01-2, CD01-3, CD01-4) - название партии, S* (S1, S2, ..., S96) - индекс скважины, а T*.png (T1.png, T2.png, ..., T10.png) - предварительно обработанное светлопольное изображение скважины на разных временных шагах потока изображений.
- В каждой партии вычисляется процент элементов cTnT+ для каждой лунки, который рассчитывается путем деления количества элементов cTnT+ на общее количество элементов Hoechst+ на флуоресцентном изображении. Усредняют процентное содержание клеток cTnT+ в параллельных лунках с одинаковым условием дозы CHIR. Сохраните условие дозы CHIR и процентное содержание клеток cTnT+ для каждой лунки в файле csv "./data/image/CD01-*/CD01-?_exp_config.csv".
- В каждой партии определяйте «низкий», «оптимальный» и «высокий» диапазон концентраций CHIR при каждой продолжительности CHIR в соответствии со следующими критериями. Концентрации CHIR со средним процентом клеток cTnT+ выше порогового значения (например, 20%) определяются как «оптимальные». Концентрации CHIR за пределами оптимального диапазона определяются как «низкие» или «высокие». Под каждой продолжительностью CHIR пометьте скважины как «низкие», «оптимальные» и «высокие» в соответствии с их соответствующими концентрациями CHIR.
ПРИМЕЧАНИЕ: Эмпирически существует отрицательная корреляция между концентрациями CHIR и продолжительностью скважин с высокой эффективностью в пределах партии: при большей продолжительности CHIR оптимальные концентрации снижаются. Другими словами, оптимальность концентраций CHIR зависит от длительности CHIR, и, таким образом, маркировки скважин различаются в зависимости от длительности CHIR. - В каждой партии, при каждой длительности CHIR, вычислите концентрацию ΔCHIR для каждой концентрации c , чтобы количественно оценить ее отклонение от оптимального. Обозначьте оптимальный диапазон концентраций CHIR через [c1, c2], и тогда концентрация ΔCHIR (c) = c - c1 (если c < c1), 0 (если c1 ≤ c ≤ c2) или c - c2 (если c > c2).
- Извлекайте особенности потоков изображений в наборе данных, гарантируя, что они отражают морфологические изменения клеток на стадии I. Чтобы вычислить особенности изображения о локальной энтропии, площади, окружности, яркости, оптическом потоке и фрактальной размерности, выполните команду
CD ./данные
Python compute_features.py
компакт-диск..
ПРИМЕЧАНИЕ: Чтобы выделить морфологические изменения, значения некоторых объектов (например, площади, окружностей и оптического потока) могут быть нормализованы по их начальным базовым значениям в потоках изображений. - Случайным образом разделите набор данных на обучающий и тестовый наборы. Запустите записную книжку Jupyter Notebook "./data/prepare_dataset.ipynb", чтобы разделить набор данных и организовать наборы данных в виде кадров данных (рис. 3D).
- Для каждой длительности CHIR обучите модель логистической регрессии для прогнозирования метки концентрации CHIR (низкая/оптимальная/высокая) на основе объектов потока изображений в обучающем наборе. Оцените производительность классификации обученной модели логистической регрессии на тестовом наборе с помощью точности, точности, полноты, оценки F1 и площади под кривой (AUC). Запустите записную книжку Jupyter Notebook "machine_learning.ipynb" для обучения и тестирования (рис. 3E).
- Оцените эффективность модели при оценке дозы CHIR. В тестовом наборе объедините прогнозируемые метки параллельных скважин с одинаковой концентрацией CHIR с помощью оценки отклонения (в диапазоне от -1 до 1), определяемой как
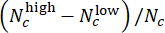 , где Nc обозначает количество скважин с концентрацией c , а
, где Nc обозначает количество скважин с концентрацией c , а 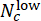
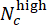 скважины среди них прогнозируются как «низкие» и «высокие» соответственно. Подтвердите, что прогнозируемые оценки отклонения сильно коррелируют с истинной концентрацией ΔCHIR для каждой дозы CHIR, используя коэффициент корреляции Пирсона.
скважины среди них прогнозируются как «низкие» и «высокие» соответственно. Подтвердите, что прогнозируемые оценки отклонения сильно коррелируют с истинной концентрацией ΔCHIR для каждой дозы CHIR, используя коэффициент корреляции Пирсона. - Выполните межпакетную проверку для проверки производительности генерализации модели. На каждом раунде оставляйте одну партию для тестирования, а другие используйте для обучения. Сравните прогнозируемые оценки отклонения и истинную концентрацию ΔCHIR для каждой дозы CHIR в каждой испытуемой партии, используя коэффициент корреляции Пирсона. Запустите записную книжку Jupyter Notebook "cross_batch_validation.ipynb", чтобы реализовать межпакетную проверку (рис. 3F).
ПРИМЕЧАНИЕ: Для выбранной длительности CHIR h прогноз для всех скважин с концентрацией из модели логистической регрессии, обученной при длительности h , будет преобразован в оценку отклонения, отражающую оптимальность концентрации CHIR c. Оценка отклонения, близкая к 0, предполагает, что концентрация c прогнозируется оптимальной при длительности h, в то время как отрицательная или положительная оценка отклонения предполагает состояние недостаточной дозы или передозировки, что может служить сигналом для последующей коррекции доз CHIR. Обратите внимание, что поскольку прогноз сделан на ранней стадии (12 ч), окончательная продолжительность CHIR и концентрация CHIR в течение оставшейся части этапа I все еще могут быть скорректированы. - Применяйте обученные модели логистической регрессии для оценки доз CHIR в новых партиях. Для заданной концентрации CHIR вычислите признаки потока изображений за 0-12 часов для параллельных скважин с концентрацией, передайте эти признаки в модели логистической регрессии, обученные при разной длительности CHIR, и, соответственно, получите прогнозируемые метки для каждой скважины. На основе прогноза моделей вычислите оценки отклонения для оценки каждой концентрации CHIR при разной длительности CHIR.
- С помощью оценки дозы CHIR на основе модели спасательные скважины при каждой субоптимальной концентрации CHIR соответственно, регулируя их продолжительность или концентрацию CHIR в сторону оптимальной до 48 часов.
- Корректировка длительности: в соответствии с прогнозом модели об оптимальной продолжительности CHIR скорректировать момент времени, когда CHIR-содержащая среда в этих скважинах переключается на среду дифференцировки CM. Если оптимальная временная точка упущена, выбросьте эти образцы.
- Корректировка концентрации: при заданной продолжительности CHIR, если текущая концентрация прогнозируется как «низкая», напрямую добавьте высокую концентрацию CHIR в среду для увеличения концентрации CHIR. При необходимости разбавьте CHIR в ДМСО (50 мМ) с PBS, добавьте соответствующий объем жидкости и осторожно встряхните пластину. Если текущая концентрация прогнозируется как «высокая», аспирируйте определенный объем среды, содержащей CHIR, и дополните таким же объемом среду для дифференцировки КМ.
Примечание: В течение первых 48 часов дифференцировки клетки будут секретировать факторы, улучшающие рост и дифференцировку клеток, поэтому не следует полностью менять среду. Кроме того, необходимо поддерживать постоянный объем среды в дифференцировке. Поэтому при регулировке концентрации CHIR убедитесь, что объем добавляемой жидкости как можно меньше.
- Стратегия машинного обучения на втором этапе: признание CPC, зафиксированных CM
Набор данных: пары изображений в светлом поле на 6-й день и вручную аннотированные маски зафиксированных CPC
Модель: ResNeSt38 с Grad-CAM36 для обучения со слабым учителем- Подготовьте набор данных, состоящий из светлопольных изображений на 6-й день. Вручную аннотируйте зафиксированные CM-фиксированные CPC на светлопольных изображениях, отслеживая клетки cTnT+ в потоках изображений с 12-го по 6-й день.
- Обрежьте изображения светлого поля и соответствующие ручные аннотации (называемые масками) зафиксированных CM-фиксированных CPC в патчи, размер каждого из которых должен составлять 1/5,5 от размера изображения всей лунки. Извлекайте патчи перекрывающимся образом, где соседние патчи перекрываются на 50 % в обучающем наборе и на 75 % в тестовом наборе. Пометьте патчи с ≥30% зафиксированных CM-зафиксированных CPC как «положительные». Помечайте патчи без CM-зафиксированных CPC как "отрицательные". Переключите рабочий каталог в положение "stage_II". Запустите "./Crop_and_Reconstruct/mask_crop.m", чтобы обрезать изображения. Запустите "./Crop_and_Reconstruct/classification.m", чтобы присвоить метки (0: отрицательные; 1: положительные) участкам светлого поля (Рисунок 3G).
- Случайным образом разделите помеченные участки изображений на обучающий набор и тестовый набор.
- Обучите глубокую сверточную нейронную сеть ResNeSt38 , чтобы научиться классифицировать эти участки. Оцените обученный ResNeSt на тестовом наборе, чтобы убедиться, что модель демонстрирует высокую точность классификации. Запустите "train.py" и "test.py" для обучения и тестирования соответственно (рис. 3H).
- Используйте Grad-CAM36 , чтобы выделить регионы, которые вносят наибольший вклад в вывод ResNeSt, представленный тепловыми картами. Бинаризуйте тепловые карты (используя пороговое значение, например, максимум 10 % × пределе), чтобы получить прогнозируемые регионы CPC с фиксированным CM, которые называются областями CPC С РАСПОЗНАВАНИЕМ ИЗОБРАЖЕНИЙ (IR-CPC).
- Сравните области IR-CPC с аннотированными вручную масками на тестовом наборе, используя точность, оценку F1, прецизионность, полноту памяти, специфичность и пересечение через объединение (IoU). Сравните долю областей IR-CPC с индексами эффективности дифференцировки (вычисленными на основе окончательных флуоресцентных изображений cTnT) с использованием коэффициента корреляции Пирсона. Выполните команды "./Evaluation/index_calculate.m" и "./Evaluation/Pearson_correlation.m" для оценки производительности (рис. 3I).
- Применение обученной модели для распознавания CPC для новых партий на этапе II. Обрезайте полнолуночные светлые поля в патчи, передайте патчи обученному ResNeSt и получите области IR-CPC на уровне патчей, которые затем объединяются для получения целых областей IR-CPC. Вычислите долю областей IR-CPC в качестве показателя итоговой эффективности дифференцировки. Очистите IR-CPC, чтобы уменьшить загрязнение клеток (описано ниже).
Примечание: Поскольку модель распознавания CPC может проводить раннюю оценку эффективности окончательной дифференцировки, она также может служить в качестве высокопроизводительного считывания для скрининга малых молекул, способствующих процессу дифференцировки, что может еще больше оптимизировать протокол дифференцировки35.
- Регионарно-селективная очистка распознаваемых изображений ЦПК
ПРИМЕЧАНИЕ: Эксперименты по фотоактивации основаны на использовании инвертированного флуоресцентного микроскопа с моторизованным предметным столиком.- В соответствии с областями IR-CPC, предсказанными обученной моделью, отбраковываются скважины, которые не смогли генерировать IR-CPC. Чтобы очистить эти СРК, зафиксированные КМ, используйте нецитотоксический фотоактивируемый зонд Dual-Activatable Cell Tracker 1 (DACT-1) для селективного маркирования не-СРК39.
- Растворите DACT-1 (10 мМ) в ДМСО и храните при температуре -20 °C. Инкубировать клетки 6-го дня с 1 мкМ DACT-1 в среде для дифференцировки CM в течение 30 мин при 37 °C.
- Осмотрите живые клетки под микроскопом и выберите область интереса (ROI) в виде полигонов в MATLAB. Проведите параллельные линии с шагом 20 мкм внутри многоугольника и вычислите координаты точек пересечения.
- Установите движущуюся дорожку моторизованного столика в соответствии с линиями трассировки, затем столик будет двигаться со скоростью 0,12 мм/с, чтобы лазерная линия с длиной волны 405 нм (диаметром 20 мкм), собранная на столике, могла глобально сканировать ROI для фотоактивации не-CPC. После облучения обнаруживайте клетки, меченные DACT-1, с помощью лазерной линии с длиной волны 561 нм.
- Диссоциируйте клетки с помощью 0,05% трипсина-ЭДТА (разбавленного PBS) при 37 °C в течение 5-7 минут и осторожно встряхните планшет в инкубаторе в течение 2 минут. Профильтруйте клетки через сетчатое фильтр 40 μм и центрифугируйте при 500 × г в течение 3 минут. Ресуспендируйте клетки в 0,5% BSA и держите трубку на льду до сортировки. Также подготовьте необлученную пробу в качестве отрицательного контроля для сортировки.
- Запустите систему сортировки проточной цитометрии и настройте программное обеспечение сортировщика. Выберите лазеры с длиной волны 561 нм и используйте полосовой фильтр 610/20 для сортировки DACT1+. Для каждой выборки используйте инструмент полигонального стробирования, чтобы обвести популяцию клеток на двумерном графике прямого рассеяния (FSC) и бокового рассеяния (SSC) для выбора отдельной ячейки. Затем запустите отрицательный контроль, чтобы подтвердить фоновую флуоресценцию, запустите выборочно облученный образец и нарисуйте затвор для популяции DACT1+ .
- Соберите и ресуспендируйте отсортированные клетки в поддерживающей среде CM с 10% FBS и 5 μM Y27632, а также засейте отсортированные клетки в 96-луночный планшет, покрытый Матригелем.
- На следующий день смените среду на поддерживающую среду CM и дождитесь сбора CM высокой чистоты.
- ML-стратегия на III этапе: признание CM
Набор данных: пары светлопольных изображений на 12-й день и окончательные флуоресцентные изображения cTnT
Модель: pix2pix37- Подготовьте набор данных, состоящий из светлопольных изображений CM и соответствующих флуоресцентных изображений cTnT. Чтобы увеличить разнообразие, убедитесь, что набор данных включает лунки с различной эффективностью дифференцировки и клеточные линии.
- Случайным образом разделите набор данных на обучающий и тестовый наборы. Переключите рабочий каталог в положение "stage_III". Сохраните светлопольные изображения и соответствующие флуоресцентные изображения cTnT в папках "./pix2pix/datasets/(CM|CM_new_cell_lines)/A/(train|test)/*.png" и ".../B/(train|test)/*.png" соответственно. Выполните следующую команду:
CD pix2pix/наборы данных
python combine_A_and_B.py --fold_A ./CM/A --fold_B ./CM/B --fold_AB ./CM/ --no_multiprocessing
компакт-диск.. - Обучите pix2pix model37 на обучающем наборе. Выполните следующую команду (рисунок 3J):
python train.py --dataroot ./datasets/CM --name brightfield2fluorescence --model pix2pix --input_nc 1 --output_nc 1 --load_size 1536 --crop_size 256 --lr 2e-4 --n_epochs 1000 --n_epochs_decay 0 --norm instance --netD n_layers --n_layers_D 1 --batch_size 16 --direction AtoB --save_epoch_freq 100 --dataset_mode aligned --use_resize_conv --seed 1234 - Примените обученную модель pix2pix к тестовому набору. Выполните следующую команду:
python test.py --dataroot ./datasets/CM --name brightfield2fluorescence --model pix2pix --direction AtoB --input_nc 1 --output_nc 1 --load_size 1536 --crop_size 1536 --use_resize_conv --eval --num_test 1000 - Сравните предсказанные моделью флуоресцентные изображения с истинными (т.е. экспериментально полученными). На уровне пикселей сравните прогнозируемую интенсивность и истинную интенсивность, используя коэффициент корреляции Пирсона. Кроме того, на уровне изображений сравните прогнозируемые индексы эффективности дифференцировки с истинными, используя коэффициент корреляции Пирсона. Запустите "stage_III/evaluation/pixel_correlation.m" и "image_correlation.ipynb" для этих сравнений (рис. 3K,L).
- Примените обученную модель для распознавания CM в новых пакетах на этапе III. Передайте новые светлопольные изображения КМ обученной модели pix2pix и получите in-silico прогнозирование флуоресцентных изображений и эффективности дифференцировки.
ПРИМЕЧАНИЕ: Для снижения контаминации клеток фотоактивированная сортировка клеток на основе DACT-1 также применима для очистки CM, распознаваемых моделью.
Результаты
На основе визуализации в светлом поле и машинного обучения можно интеллектуально контролировать и оптимизировать общий процесс дифференциации. На этапе ПСК мы разработали ML-модель, которая могла бы предсказать окончательную эффективность дифференцировки в соответ...
Обсуждение
В этой статье мы подробно описали протокол для преодоления одной из основных проблем в современном применении и трансляции ПСК — вариабельности дифференцировки клеток. Используя визуализацию в светлом поле живых клеток и машинное обучение, мы итеративно оптимизиро...
Раскрытие информации
Ян Чжао, Цзюэ Чжан, Сяочунь Ян, Яо Ван и Дайчао Чен подали заявку на патент на стратегию дифференциации PSC, описанную в этом документе (202210525166.X).
Благодарности
Мы благодарим Цюши Суня, Яо Вана, Юй Ся, Цзиньью Яна, Чан Линя, Цзыму Цэня, Дундуна Ляна, Жун Вэя, Цзэ Сюя, Гуанъинь Си, Ган Сюэ, Цань Е, Ли-Пэн Вана, Пэн Цзоу, Ши-Цян Вана, Пабло Ривера-Фуэнтеса, Саломе Пюнтенер, Чжисин Чэнь, И Лю и Цзюэ Чжана за то, что они заложили основу этой стратегии. Эта работа была поддержана Национальной ключевой программой исследований и разработок Китая (2018YFA0800504, 2019YFA0110000) и Проектом космического медицинского эксперимента Китайской пилотируемой космической программы (HYZHXM01020) Ян Чжао. Рисунок 1 был создан с помощью BioRender.com.
Материалы
| Name | Company | Catalog Number | Comments |
| 0.25% Trypsin-EDTA | Gibco | 25200056 | Diluted digests were used for CPC and CM digestion |
| 4% Paraformaldehyde in PBS | KeyGEN BioTECH | KGIHC016 | |
| 6-well Cell Culture Plate | NEST | 703001 | |
| 96-well Cell Culture Plate | NEST | 701001 | |
| B27 Supplement | Gibco | 17504044 | |
| B27 Supplement Minus Insulin | Gibco | A1895601 | |
| Bovine serum albumin (BSA) | GPC BIOTECH | AA904-100G | |
| Celldiscoverer 7 | Zeiss | Instruments used to take bright-field images throughout differentiation and final cTnT images | |
| CHIR99021 | Selleck | S1263 | |
| DMEM/F12 | Gibco | 12634010 | |
| Donkey anti-Mouse IgG (H+L) Highly Cross-Adsorbed Secondary Antibody, Alexa Fluor 488 | Thermo | A-21202 | Secondary Antibody |
| FACSAria III | BD Biosciences | Flow cytometry sorter | |
| Fetal Bovine Serum (FBS) | VISTECH | SE100-B | |
| Hoechst 33342 | YEASEN | 40732ES03 | |
| Human Pluripotent Stem Cell Chemical-defined Medium | Cauliscell Inc | 400105 | Basal medium of PSC preparation medium |
| iPS-18 | TaKaRa | Y00300 | |
| iPS-B1 | Cellapy | CA4025106 | |
| iPS-F | Nuwacell | RC01001-B | |
| iPS-M | Nuwacell | RC01001-A | |
| IWR1-1-endo | Selleck | S7086 | IWR1 |
| Jupyter Notebook | N/A | Version 6.4.0 | https://jupyter.org/ |
| MATLAB | MathWorks | Version R2020a | Software for scientific computation and image annotation |
| Matrigel Matrix | Corning | 354230 | Matrigel |
| Mouse monoclonal IgG1 anti-cTnT | Thermo | MA5-12960 | cTnT primary antibody |
| Normal Donkey Serum | Jackson | 017-000-121 | |
| ORCA-Flash 4.0 V3 digital CMOS camera | Hamamatsu | C13440-20CU | The digital camera assembled on Celldiscoverer7 |
| PBS | NEB | 21-040-CVR | |
| Penicillin-Streptomycin | Gibco | 15140-122 | |
| Pluripotency Growth Mater 1 basal medium | Cellapy | CA1007500-1 | Basal medium of PSC culture medium |
| Pluripotency Growth Mater 1 supplement | Cellapy | CA1007500-2 | Supplement of PSC culture medium |
| Prism | Graphpad | Version 8/9 | Statistical software for statistical analysis and plotting |
| Python | N/A | version 3.6 | Python 3 environment for scientific computation, with packages pytorch (1.9.0), numpy, scipy, pandas, visdom, scikit-learn, scikit-image, opencv-python, and matplotlib software for scientific computation and image annotation. |
| RPMI 1640 | Gibco | 11875176 | |
| Supplement hPSC-CDM (500x) | Cauliscell Inc | 00015 | Supplement of PSC preparation medium |
| TiE | Nikon | An inverted fluorescence microscope (with modification) for region-selevtive purification | |
| Triton X-100 | Amresco | 9002-93-1 | |
| Versene Solution | Thermo | 15040066 | EDTA solution for PSC digestion |
| Y27632 | Selleck | S6390 | |
| Zen | Zeiss | Version 3.1 | A supporting software of Celldiscoverer7 for image acquisition, processing and analysis |
Ссылки
- Yoshida, Y., Yamanaka, S. Induced pluripotent stem cells 10 years later: for cardiac applications. Circ Res. 120 (12), 1958-1968 (2017).
- Shi, Y., Inoue, H., Wu, J. C., Yamanaka, S. Induced pluripotent stem cell technology: a decade of progress. Nat Rev Drug Discov. 16 (2), 115-130 (2017).
- Takahashi, K., et al. Induction of pluripotent stem cells from adult human fibroblasts by defined factors. Cell. 131 (5), 861-872 (2007).
- Burridge, P. W., et al. Chemically defined generation of human cardiomyocytes. Nat Methods. 11 (8), 855-860 (2014).
- Mordwinkin, N. M., Burridge, P. W., Wu, J. C. A review of human pluripotent stem cell-derived cardiomyocytes for high-throughput drug discovery, cardiotoxicity screening, and publication standards. J Cardiovasc Transl Res. 6 (1), 22-30 (2013).
- Vanden Berg, C. W., Elliott, D. A., Braam, S. R., Mummery, C. L., Davis, R. P. Differentiation of human pluripotent stem cells to cardiomyocytes under defined conditions. Methods Mol Biol. 1353, 163-180 (2016).
- Burridge, P. W., Holmström, A., Wu, J. C. Chemically defined culture and cardiomyocyte differentiation of human pluripotent stem cells. Curr Protoc Hum Genet. 87, 1-15 (2015).
- Li, J., et al. Human pluripotent stem cell-derived cardiac tissue-like constructs for repairing the infarcted myocardium. Stem Cell Rep. 9 (5), 1546-1559 (2017).
- Smith, A. S. T., Macadangdang, J., Leung, W., Laflamme, M. A., Kim, D. -. H. Human iPSC-derived cardiomyocytes and tissue engineering strategies for disease modeling and drug screening. Biotechnol Adv. 35 (1), 77-94 (2017).
- Sharma, A., et al. Use of human induced pluripotent stem cell-derived cardiomyocytes to assess drug cardiotoxicity. Nat Protoc. 13 (12), 3018-3041 (2018).
- Mehta, A., et al. Identification of a targeted and testable antiarrhythmic therapy for long-QT syndrome type 2 using a patient-specific cellular model. Eur Heart J. 39 (16), 1446-1455 (2018).
- Cahan, P., Daley, G. Q. Origins and implications of pluripotent stem cell variability and heterogeneity. Nat Rev Mol Cell Biol. 14 (6), 357-368 (2013).
- Kim, K., et al. Epigenetic memory in induced pluripotent stem cells. Nature. 467 (7313), 285-290 (2010).
- Rouhani, F., et al. Genetic background drives transcriptional variation in human induced pluripotent stem cells. PLoS Genet. 10 (6), e1004432 (2014).
- Kim, K., et al. Donor cell type can influence the epigenome and differentiation potential of human induced pluripotent stem cells. Nat Biotechnol. 29 (12), 1117-1119 (2011).
- Garitaonandia, I., et al. Increased risk of genetic and epigenetic instability in human embryonic stem cells associated with specific culture conditions. PLoS One. 10 (2), e0118307 (2015).
- Engler, A. J., Sen, S., Sweeney, H. L., Discher, D. E. Matrix elasticity directs stem cell lineage specification. Cell. 126 (4), 677-689 (2006).
- Matoba, N., Yamashita, T., Takayama, K., Sakurai, F., Mizuguchi, H. Optimal human iPS cell culture method for efficient hepatic differentiation. Differentiation. 104, 13-21 (2018).
- Volpato, V., et al. Reproducibility of molecular phenotypes after long-term differentiation to human iPSC-derived neurons: A multi-site omics study. Stem Cell Rep. 11 (4), 897-911 (2018).
- Anderson, N. C., et al. Balancing serendipity and reproducibility: Pluripotent stem cells as experimental systems for intellectual and developmental disorders. Stem Cell Rep. 16 (6), 1446-1457 (2021).
- Sepac, A., et al. Comparison of cardiomyogenic potential among human ESC and iPSC lines. Cell Transplant. 21 (11), 2523-2530 (2012).
- Laco, F., et al. Unraveling the inconsistencies of cardiac differentiation efficiency induced by the GSK3β inhibitor CHIR99021 in human pluripotent stem cells. Stem Cell Rep. 10 (6), 1851-1866 (2018).
- Volpato, V., Webber, C. Addressing variability in iPSC-derived models of human disease: guidelines to promote reproducibility. Dis Model Mech. 13 (1), 042317 (2020).
- Hu, B. -. Y., et al. Neural differentiation of human induced pluripotent stem cells follows developmental principles but with variable potency. Proc Natl Acad Sci USA. 107 (9), 4335-4340 (2010).
- Huo, J., et al. Evaluation of batch variations in induced pluripotent stem cell-derived human cardiomyocytes from 2 major suppliers. Toxicol Sci. 156 (1), 25-38 (2017).
- Rostam, H. M., Reynolds, P. M., Alexander, M. R., Gadegaard, N., Ghaemmaghami, A. M. Image based machine learning for identification of macrophage subsets. Sci Rep. 7 (1), 3521 (2017).
- Christiansen, E. M., et al. In silico labeling: Predicting fluorescent labels in unlabeled images. Cell. 173 (3), 792-803 (2018).
- Buggenthin, F., et al. Prospective identification of hematopoietic lineage choice by deep learning. Nat Methods. 14 (4), 403-406 (2017).
- Smith, Z. D., Nachman, I., Regev, A., Meissner, A. Dynamic single-cell imaging of direct reprogramming reveals an early specifying event. Nat Biotechnol. 28 (5), 521-526 (2010).
- Fan, K., Zhang, S., Zhang, Y., Lu, J., Holcombe, M., Zhang, X. A machine learning assisted, label-free, non-invasive approach for somatic reprogramming in induced pluripotent stem cell colony formation detection and prediction. Sci Rep. 7 (1), 13496 (2017).
- Kusumoto, D., et al. Automated deep learning-based system to identify endothelial cells derived from induced pluripotent stem cells. Stem Cell Rep. 10 (6), 1687-1695 (2018).
- Zhu, Y., et al. Deep learning-based predictive identification of neural stem cell differentiation. Nat Commun. 12 (1), 2614 (2021).
- Qian, T., et al. Label-free imaging for quality control of cardiomyocyte differentiation. Nat Commun. 12 (1), 4580 (2021).
- Ounkomol, C., Seshamani, S., Maleckar, M. M., Collman, F., Johnson, G. R. Label-free prediction of three-dimensional fluorescence images from transmitted-light microscopy. Nat Methods. 15 (11), 917-920 (2018).
- Yang, X., et al. A live-cell image-based machine learning strategy for reducing variability in PSC differentiation systems. Cell Discovery. 9 (1), 53 (2023).
- Selvaraju, R. R., et al. Grad-CAM: Visual explanations from deep networks via gradient-based localization. Proc IEEE Int Conf Computer Vision (ICCV). , 618-626 (2017).
- Isola, P., Zhu, J. -. Y., Zhou, T., Efros, A. A. Image-to-image translation with conditional adversarial networks. Proc IEEE Conf Computer Vision Pattern Recognition (CVPR). , 5967-5976 (2017).
- Zhang, H., et al. ResNeSt: Split-attention networks. Proc IEEE/CVF Conf Computer Vision Pattern Recognition Workshops (CVPRW). , 2735-2745 (2022).
- Halabi, E. A., et al. Dual-activatable cell tracker for controlled and prolonged single-cell labeling). ACS Chem. Biol. 15 (6), 1613-1620 (2020).
- Hannan, N. R. F., Segeritz, C. -. P., Touboul, T., Vallier, L. Production of hepatocyte-like cells from human pluripotent stem cells. Nat Protoc. 8 (2), 430-437 (2013).
- Zhang, H., Shen, M., Wu, J. C. Generation of quiescent cardiac fibroblasts derived from human induced pluripotent stem cells. Methods Mol Biol. 2454, 109-115 (2022).
- Harding, M. J., McGraw, H. F., Nechiporuk, A. The roles and regulation of multicellular rosette structures during morphogenesis. Development. 141 (13), 2549-2558 (2014).
- Najm, F. J., et al. Rapid and robust generation of functional oligodendrocyte progenitor cells from epiblast stem cells. Nat Methods. 8 (11), 957-962 (2011).
- Hogrebe, N. J., Maxwell, K. G., Augsornworawat, P., Millman, J. R. Generation of insulin-producing pancreatic β cells from multiple human stem cell lines. Nat Protoc. 16 (9), 4109-4143 (2021).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены