Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Une stratégie d’apprentissage automatique basée sur l’image de cellules vivantes pour surveiller la différenciation des cellules souches pluripotentes
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Les systèmes de différenciation des cellules souches pluripotentes (CSP) disponibles en cellules fonctionnelles sont actuellement entravés par des problèmes de variabilité sévère d’une ligne à l’autre et d’un lot à l’autre. Ici, en utilisant la différenciation cardiaque comme exemple principal, nous présentons un protocole pour surveiller et moduler intelligemment le processus de différenciation des CSP basé sur l’apprentissage automatique basé sur l’image.
Résumé
Les technologies de cellules souches pluripotentes (CSP) ont été largement utilisées dans la découverte de médicaments, la modélisation de maladies et la médecine régénérative. Cependant, les systèmes de différenciation cellulaire fonctionnelle PSC disponibles sont entravés par des problèmes de variabilité sévère d’une ligne à l’autre et d’un lot à l’autre. Il est donc important de contrôler précisément la différenciation cellulaire en temps réel. Dans ce protocole, nous décrivons une stratégie non invasive et intelligente qui surmonte la variabilité de la différenciation cellulaire en utilisant l’apprentissage automatique basé sur l’image en champ clair. Si l’on prend l’exemple de la différenciation des CSP en cardiomyocytes, cette méthodologie fournit des informations détaillées pour le contrôle de l’état initial des CSP, l’évaluation et l’intervention précoces dans les conditions de différenciation, et l’élimination de la contamination cellulaire indifférenciée, tout en réalisant une différenciation de haute qualité et constante des CSP en cellules fonctionnelles. En principe, cette stratégie peut être étendue à d’autres systèmes de différenciation ou de reprogrammation cellulaire avec plusieurs étapes pour soutenir la fabrication des cellules, ainsi que pour approfondir notre compréhension des mécanismes lors de la conversion du destin cellulaire.
Introduction
Les cellules souches pluripotentes (CSP) possèdent la remarquable capacité de se différencier en de nombreux types de cellules in vitro. Ces cellules fonctionnelles différenciées pourraient être utilisées pour la thérapie cellulaire, la modélisation de maladies et le développement de médicaments, toutes précieuses pour la recherche ou les applications cliniques 1,2,3. Par exemple, diverses méthodes ont été développées pour différencier les CSP en cardiomyocytes (CM)4,5,6,7. Ces MC peuvent être utilisés pour les tests de cardiotoxicité des médicaments, la modélisation des maladies cardiaques et la transplantation cellulaire 8,9,10,11.
Cependant, la conversion de la CSP en cellules différenciées terminales est un processus par étapes, et de multiples perturbations au cours du processus de différenciation peuvent conduire les cellules à des destins cellulaires divergents. Les différents antécédents génétiques et marques épigénétiques des lignées PSC influencent le potentiel de différenciation vers une lignée spécifique 12,13,14,15 ; le nombre de passages de CSP et les mutations génétiques accumulées sont également des sources d’hétérogénéité des CSP ; Les différences dans les opérations expérimentales employées par différents expérimentateurs peuvent également conduire à des résultats de différenciation complètement différents 16,17,18,19,20. Par conséquent, à l’heure actuelle, l’un des principaux problèmes de production de cellules dérivées de PSC est l’instabilité entre les lignées cellulaires et les lots 21,22,23,24,25. L’instabilité de la différenciation des CSP conduit souvent à de multiples expériences répétées, ce qui consomme beaucoup de temps et de ressources en main-d’œuvre. Pour résoudre ce problème, il est crucial de développer une stratégie qui minimise la variabilité entre les lignées cellulaires et les lots, améliorant ainsi la stabilité et la robustesse de la différenciation.
Récemment, les progrès de la microscopie à haute résolution et de l’apprentissage automatique (ML) ont facilité l’application de l’analyse quantitative d’images basée sur l’apprentissage automatique en biologie cellulaire, ce qui a permis d’utiliser des informations précieuses dans les caractéristiques d’imagerie cellulaire 26,27,28,29,30,31,32,33,34. Dans nos travaux précédents, nous avons proposé une stratégie de ML basée sur l’image de cellules vivantes pour surveiller et intervenir dans l’état de différenciation de la PSC en temps réel afin d’améliorer la stabilité et l’efficacité de la différenciation de la PSC (Figure 1)35. En prenant l’exemple de la différenciation PSC en cardiomyocytes, nous avons évalué l’état initial de la PSC à l’aide de modèles de forêt aléatoires, prédit la condition de différenciation optimale à l’aide de modèles de régression logistique et reconnu les cellules différenciées avec succès à l’aide de Grad-CAM36 et pix2pix37 basés sur l’apprentissage profond. Les modèles d’apprentissage automatique ont appris à identifier les lignées cellulaires à partir d’une gamme de caractéristiques morphologiques en fond clair, y compris des caractéristiques relatives à la surface, à la circonférence, à la convexité, à la solidité, à la luminosité, à la vitesse de déplacement et à d’autres caractéristiques implicites extraites par des réseaux neuronaux convolutifs profonds. Sur la base de l’inférence de ces modèles ML établis, nous avons réalisé le contrôle de l’état initial de la CSP, l’évaluation et l’intervention précoces dans des conditions de différenciation, et l’élimination de la contamination cellulaire indifférenciée, fournissant ensemble une modulation complète et précise du processus de différenciation cardiaque. Nous vous présentons ici un protocole étape par étape pour l’élaboration de la stratégie.
Protocole
1. Différenciation et caractérisation cellulaires
- Préparation des réactifs de culture et des plaques de culture
- Préparez le milieu de culture PSC en ajoutant 2 mL de supplément et 0,2 % de pénicilline-streptomycine à 48 mL de milieu basal. Aliquote et conservez le supplément à -20 °C. Conservez ce milieu à 4 °C jusqu’à 4 semaines.
- Préparez le milieu de préparation PSC en ajoutant 1 mL de supplément et 0,2 % de pénicilline-streptomycine à 500 mL de milieu basal. Lors de l’utilisation, préchauffez le milieu pour une utilisation unique et conservez-le à 4 °C jusqu’à 3 semaines.
- Préparez le milieu de différenciation de la MC en ajoutant 1 supplément de B27 moins l’insuline et 1 % de pénicilline-streptomycine dans le RPMI 1640. Préparez le milieu d’entretien de la MC en ajoutant 1 supplément de B27 et 1 % de pénicilline-streptomycine dans le RPMI 1640. Conservez ces deux types de milieu à 4 °C pendant 2 semaines.
- Immergez le Matrigel dans de la glace et maintenez-le à 4 °C pendant la nuit. Assurez-vous que le Matrigel est complètement décongelé ; ensuite, aliquote dans 1 mL d’aliquotes stockées à -80 °C pour une utilisation ultérieure, et ajouter 1 mL de Matrigel dans 49 mL de DMEM/F12 à 4 °C pour préparer la solution de travail de Matrigel. Ajouter 850 μL de cette solution de travail Matrigel par puits d’une plaque de culture à 6 puits et incuber la plaque à 37 °C pendant au moins 30 min. Pour la plaque de culture à 96 puits utilisée dans la différenciation CM, ajouter 35 μL de solution de travail Matrigel par puits.
REMARQUE : Gardez Matrigel sur la glace en tout temps ; Les pointes et les tubes de la pipette doivent être pré-refroidis à 4 °C. La solution de travail Matrigel peut être conservée à 4 °C pendant 2 semaines. Veillez à ne pas laisser de bulles lors du pipetage de la solution de travail Matrigel.
- Maintenance et passage des PSC
- Maintenir les CSP dans des plaques de culture à 6 puits. Confirmez la densité cellulaire au microscope et préparez-vous au passage lorsque la confluence atteint ~70 % (Figure 2A).
- Utilisez l’EDTA pour digérer les cellules pour le passage. Réchauffez le milieu de culture PSC, le PBS et l’EDTA dans un bain-marie à 37 °C, et ajoutez du Y27632 dans le milieu jusqu’à une concentration finale de 5 μM.
- Lavez les CSP avec 1 ml de PBS, ajoutez 1 ml d’EDTA par puits et incubez la plaque dans un incubateur à 37 °C pendant 3 min.
- Aspirez l’EDTA avant que les cellules ne se détachent de la plaque de culture, puis utilisez 1 ml de milieu de culture PSC pour rincer doucement le fond du puits 3 à 4 fois pour remettre les cellules en suspension.
REMARQUE : Cela nous permet de sauter l’étape de centrifugation et de réduire les dommages aux cellules lors des opérations de passage. - Aspirer le DMEM/F12 d’un puits recouvert de Matrigel d’une plaque de culture à 6 puits et ajouter 2 mL de milieu de culture PSC frais par puits (Y27632 ajouté). Ajoutez la suspension cellulaire dans les puits avec un rapport de division de 1:6 à 1:12. Placez la plaque dans l’incubateur à 37 °C avec 5% de CO2.
- Changer le milieu de culture PSC (sans Y27632) 12 à 24 h après le passage ; Ensuite, changez de milieu et vérifiez l’état et la confluence des cellules tous les jours.
- Différenciation cardiaque des CSP
- Pour la différenciation des CM, ensemencer les CSP dans une plaque de culture à 96 puits dans un milieu de préparation pour CSP (Y27632 ajouté). Pour la différenciation des MC, suivez les mêmes étapes ci-dessus (étapes 1.2.2-1.2.6) pour ensemencer les CSP dans une plaque de culture à 96 puits dans un milieu de préparation des CSP (Y27632 ajouté).
REMARQUE : À ce stade, la durée du traitement par EDTA pourrait être prolongée de manière appropriée (5 min ou plus) pour dissocier suffisamment les CSP afin que les clones puissent être plus petits après le passage. Notre étude a démontré qu’au sein d’une même zone, des colonies plus petites sont propices à la différenciation. - Étape I : Lorsque les CSP atteignent une confluence de 80 à 90 %, passer du milieu au milieu de différenciation CM avec une CHIR99021 de 2 à 20 μM (CHIR) (Figure 2B). Le point temporel de la commutation de milieu est appelé jour 0. Après un traitement CHIR de 24 à 48 h, remplacez le milieu par un milieu de différenciation CM frais.
REMARQUE : CHIR est un inhibiteur de GSK3beta pour l’induction du mésoderme. La dose optimale de CHIR pour la différenciation des CM fluctue selon les lignées cellulaires et les lots. Par exemple, la dose optimale de CHIR est de 8 à 10 μM, 48 h pour l’iPS-B1. Pour collecter diverses images en fond clair afin d’entraîner le modèle ML, nous avons titré les concentrations CHIR pour chaque lot d’expériences afin d’obtenir différentes efficacités de différenciation. - Stade II : À 72 h (jour 3), remplacer le milieu par le milieu de différenciation CM complété par 5 μM IWR1 et cultiver pendant 48 h (jour 5). Changez le milieu avec le milieu de différenciation CM pendant 1 à 2 jours (jusqu’aux jours 6 et 7). À ce stade, les CSP se différencient en cellules progénitrices cardiaques (CPC).
- Étape III : Remplacer le milieu par un milieu d’entretien CM et remplacer le milieu tous les 3 jours. Le jour 10 ou le jour 12, récoltez le MC pour une analyse plus approfondie.
- Pour la différenciation des CM, ensemencer les CSP dans une plaque de culture à 96 puits dans un milieu de préparation pour CSP (Y27632 ajouté). Pour la différenciation des MC, suivez les mêmes étapes ci-dessus (étapes 1.2.2-1.2.6) pour ensemencer les CSP dans une plaque de culture à 96 puits dans un milieu de préparation des CSP (Y27632 ajouté).
- Coloration par immunofluorescence de la CM
- Au jour 10 ou au jour 12, lavez les cellules avec du PBS et fixez-les avec du paraformaldéhyde à 4% dans du PBS pendant 15 min à température ambiante. Lavez les CM 2 fois avec du PBS et conservez la plaque à 4 °C jusqu’à ~1 semaine.
REMARQUE : Le paraformaldéhyde est un réactif toxique avec une odeur piquante. Utiliser avec une cagoule chimique et protéger les yeux et les mains. - Au moment de la coloration, traiter les cellules avec une solution de perméabilisation (0,1% de Triton X-100 dans du PBS) pendant 15 min à température ambiante ; ensuite, incubez des cellules dans une solution bloquante (0,1 % de Triton X-100 contenant 3 % de sérum d’âne dans du PBS) pendant 30 min à température ambiante.
- Incuber l’échantillon avec l’anticorps primaire de la troponine cardiaque T (cTnT) (1:300) dilué dans une solution bloquante pendant la nuit à 4 °C pour identifier les CM.
- Prélevez l’anticorps primaire et lavez les cellules avec du PBS 3x. Incuber l’échantillon avec des anticorps secondaires dans du PBS avec 1 % d’albumine sérique bovine pendant 1 h à 37 °C dans un environnement sombre.
REMARQUE : L’anticorps primaire collecté peut être réutilisé 2 à 3 fois en 1 semaine.
- Prélevez l’anticorps primaire et lavez les cellules avec du PBS 3x. Incuber l’échantillon avec des anticorps secondaires dans du PBS avec 1 % d’albumine sérique bovine pendant 1 h à 37 °C dans un environnement sombre.
- Retirez l’anticorps secondaire des cellules et lavez les cellules 3 fois avec du PBS. Utilisez Hoechst 33342 (1:1 000 en PBS) pour colorer les noyaux pendant 5 minutes à température ambiante. Rincez la cellule 3 fois avec du PBS et ajoutez 100 μL de PBS par puits pour éviter le dessèchement. Conservez la plaque à 4 °C dans un endroit sombre et acquérez des images fluorescentes dans un délai de 1 semaine.
- Au jour 10 ou au jour 12, lavez les cellules avec du PBS et fixez-les avec du paraformaldéhyde à 4% dans du PBS pendant 15 min à température ambiante. Lavez les CM 2 fois avec du PBS et conservez la plaque à 4 °C jusqu’à ~1 semaine.
2. Acquisition du flux d’images tout au long du processus de différenciation
- Mise en place d’un plan expérimental pour la collecte d’images
- Utiliser un microscope automatisé prenant en charge la culture et l’imagerie de cellules vivantes pour collecter des images en fond clair des différents stades de la différenciation de la CM et des images immunofluorescentes cTnT du résultat de la différenciation (Figure 2A-E). Utilisez le logiciel de support de microscope pour concevoir le programme d’expérience et contrôler le microscope.
- Ouvrez le logiciel et créez un nouveau modèle d’expérience. Choisissez un objectif 5x et une lentille tubulaire 2x pour l’imagerie. Vérifiez les options Tuiles et Z-stack.
- Ajoutez le canal TL Fond clair pour l’imagerie en fond clair dans le menu Canaux. Ajoutez les canaux AF488 et H3342 pour l’imagerie par immunofluorescence. Modifiez le trajet de la lumière dans le menu Configuration de l’imagerie pour l’imagerie avec la caméra CMOS numérique externe.
- Ouvrez le menu Mode d’acquisition et définissez le binning 2 x 2 pour augmenter le rapport signal/bruit.
- Ouvrez le menu Z-stack et choisissez le modèle Centre. Définissez le nombre de tranches et d’intervalles lors de la numérisation. Choisissez 3 à 5 coupes avec des intervalles de 3 à 6 μm pour des images nettes lors de l’analyse.
- Dans la fenêtre Navigation et vignettes, configurez les régions de vignette par transporteur et définissez 25 vignettes (5 colonnes x 5 lignes) pour un puits. Dans le menu Tuiles, sélectionnez le modèle Multiwell 96 dans Sample Carrier et définissez un chevauchement de 5 % à 15 % sur les régions adjacentes.
- Si vous avez besoin d’une imagerie continue, cochez l’option Série Time et définissez la durée et l’intervalle dans le menu Série chronologique. Pour collecter le flux d’images à l’étape I, définissez une durée de 12 h sans intervalles.
- Acquisition d’images en fond clair et immunofluorescentes
- Pour acquérir des images, placez d’abord la plaque de culture cellulaire dans le plateau d’échantillon et chargez l’échantillon à l’intérieur du microscope. Sélectionnez le modèle Multiwell 96 pour correspondre au plan expérimental. Si l’échantillon est constitué de cellules vivantes, ouvrez le système de chauffage et la pompe à CO2 pour maintenir les conditions appropriées pour la culture (37 °C, 5 % de CO2).
- Ouvrez le projet expérimental prédéfini et enregistrez-le en tant que nouveau. Ouvrez le menu Tuiles et calibrez manuellement la position de la plaque. Dans la fenêtre Navigation et mosaïques, sélectionnez les puits nécessaires et cliquez sur Créer pour construire des régions de tuiles pour ces puits.
- Cliquez sur Vérifier les régions de tuile dans le menu Tuiles et exécutez le focus automatique pour vérifier tous les puits. Ensuite, corrigez manuellement la mise au point de chaque puits sous le fond clair en faisant défiler la molette de la souris.
- Cochez les canaux nécessaires et réglez les temps d’exposition dans le menu Canaux. Les temps d’exposition recommandés sont de 1,625 ms pour Bright, 100 ms pour H3342 et 550 ms pour AF488. Exécutez un seul cliché pour vérifier les images de toutes les chaînes.
- Enfin, cliquez sur le bouton Démarrer l’expérience et attendez l’imagerie automatique. En règle générale, il faut ~1,2 h pour terminer le balayage d’une plaque de culture entière de 96 puits (96 puits, trois couches, 25 tuiles pour chaque puits sous le fond clair). Dans l’infrastructure de traitement, choisissez Exportation d’image, sélectionnez le type de fichier au format TIFF ou PNG non compressé, puis appliquez-le. Utilisez des images de 9 tuiles (3 x 3) au centre du puits pour une analyse plus approfondie (Figure 2A-E).
3. Etablissement de la stratégie de ML basée sur l’image à chaque étape du processus de différenciation
- Préparation du logiciel
- Installez l’environnement Python, avec les paquets pytorch (1.9.0), numpy, scipy, scikit-learn, pandas, visdom, scikit-image, opencv-python et matplotlib.
- Installez Jupyter Notebook.
- Installez MATLAB.
- Téléchargez le code et les exemples de jeux de données à partir de https://github.com/zhaoyanglab/ML-for-PSC-differentiation.
REMARQUE : Les performances du ML dépendent de la qualité des jeux de données et du choix des modèles et des hyper-paramètres. Il n’est pas garanti que l’implémentation de l’exemple soit optimale en toutes circonstances. Les ingénieurs ML doivent personnaliser le code et adapter le modèle et les hyperparamètres à leurs besoins spécifiques.
- Stratégie de ML au stade de la PSC : contrôle des états initiaux de la colonie PSC
Jeu de données : paires d’images en fond clair à 0 h (avant traitement CHIR) et efficacité de différenciation finale
Modèle : forêt aléatoire- Préparez un ensemble de données composé d’images en fond clair à 0 h (avant le traitement CHIR) et des images finales de fluorescence cTnT. Pour augmenter la diversité des profils morphologiques des images en fond clair 0 h, assurez-vous que l’ensemble de données comprend des colonies PSC de différentes lignées cellulaires, avec une différenciation initiée sur un intervalle de temps après le passage. Traitez les puits de l’ensemble de données avec les conditions CHIR optimales au stade I.
REMARQUE : Pour s’assurer que les colonies sont traitées dans des conditions CHIR optimales, les puits d’un même lot peuvent être traités avec des doses titrées de CHIR. Seuls les puits présentant des conditions CHIR à haut rendement sont inclus dans l’ensemble de données. - Quantifiez l’efficacité de différenciation de chaque puits à l’aide de l’indice d’efficacité de différenciation calculé à partir de ses images de fluorescence cTnT, qui est défini par
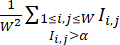 (où
(où 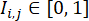 représente l’intensité de fluorescence à l’emplacement des pixels (i, j) dans l’image de fluorescence W × W, et la somme est prise sur tous les emplacements de pixels dont l’intensité est supérieure à un seuil α, généralement fixé à 0,5). Au sein de chaque lignée cellulaire, calculez l’indice d’efficacité de différenciation maximal sur tous les puits de cette lignée cellulaire et normalisez leurs indices d’efficacité de différenciation en fonction de la valeur maximale.
représente l’intensité de fluorescence à l’emplacement des pixels (i, j) dans l’image de fluorescence W × W, et la somme est prise sur tous les emplacements de pixels dont l’intensité est supérieure à un seuil α, généralement fixé à 0,5). Au sein de chaque lignée cellulaire, calculez l’indice d’efficacité de différenciation maximal sur tous les puits de cette lignée cellulaire et normalisez leurs indices d’efficacité de différenciation en fonction de la valeur maximale.
REMARQUE : Étant donné que le potentiel de différenciation de chaque lignée cellulaire est intrinsèquement différent, une telle normalisation mettra davantage l’accent sur les caractéristiques morphologiques de la colonie partagées entre les différentes lignées cellulaires qui contribuent au résultat de la différenciation. La normalisation rend en effet comparables les colonies de CSP de différentes lignées cellulaires et atténue le biais induit par les lignées cellulaires. - Quantifier les profils morphologiques des images en fond clair 0 h par des caractéristiques de grande dimension qui révèlent les propriétés de la forme de la colonie. Pour délimiter les régions de colonie dans les images en fond clair, calculez l’entropie locale (avec le rayon r) ; Les pixels dont l’entropie locale est supérieure à un seuil σ sont identifiés par des cellules recouvertes par . Utilisez un paramètre de r = 10 pixels et σ = 3. Basculez le répertoire de travail sur « ./colony_control » ; enregistrer les images en fond clair, les images binaires des régions cellulaires et les images de fluorescence cTnT à « colony_control/ image_data/CD00-*/[fond clair|cell_region|ctnt]/S*.png » ; et exécuter le Jupyter Notebook « Compute_features.ipynb » pour calculer les caractéristiques liées à la surface de la colonie, à la circonférence, à la solidité, à la convexité, à la circularité et aux distances centoïdes-contours, entre autres.
- Divisez de manière aléatoire l’ensemble de données en un ensemble d’entraînement et un ensemble de test, l’ensemble d’entraînement représentant entre 50 % et 80 % des données globales. Exécuter le dernier bloc Fractionnez le jeu de données dans le bloc-notes Jupyter pour diviser le jeu de données (Figure 3A).
- Entraînez un modèle de régression de forêt aléatoire sur le jeu d’apprentissage pour prédire l’efficacité de la différenciation à partir de caractéristiques d’image en fond clair 0 h. Exécutez le Jupyter Notebook « Machine_learning.ipynb » pour entraîner et tester le modèle.
- Évaluez le modèle de forêt aléatoire formé sur le jeu de test. Confirmez que l’efficacité de différenciation prédite est fortement corrélée à la véritable à l’aide du coefficient de corrélation de Pearson (Figure 3B).
REMARQUE : Étant donné que l’ensemble de test n’est pas impliqué dans la phase d’entraînement, une grande cohérence entre l’efficacité de différenciation prédite et l’efficacité réelle sur l’ensemble de test indique que le modèle peut bien généraliser sur des échantillons invisibles. - Obtenez l’importance des caractéristiques à partir du modèle de forêt aléatoire formé et sélectionnez les 8 entités les plus importantes. Visualisez leur relation avec l’efficacité de différenciation finale en divisant la plage de caractéristiques en groupes et en calculant l’efficacité moyenne de différenciation dans chaque groupe. Exécutez le bloc-notes Jupyter « Feature_importance.ipynb » pour effectuer la visualisation (Figure 3C).
REMARQUE : La visualisation peut aider à comprendre comment les changements dans la forme de la colonie de CSP contribuent à la différenciation. - Appliquez le modèle entraîné dans de nouveaux lots pour surveiller les états des colonies PSC en temps réel. Capturez l’image en fond clair toutes les heures, transmettez l’image au modèle entraîné et obtenez l’efficacité de différenciation prévue. Une fois que l’efficacité prévue dépasse un seuil (par exemple, 50 %), lancez le processus de différenciation. Si l’efficacité prévue n’a pas pu atteindre le seuil, jetez ces échantillons.
REMARQUE : Au cours de la phase PSC, le potentiel de différenciation des colonies PSC augmentera d’abord, puis diminuera. La surveillance en temps réel permet aux expérimentateurs d’identifier le moment le plus propice pour initier la différenciation.
- Préparez un ensemble de données composé d’images en fond clair à 0 h (avant le traitement CHIR) et des images finales de fluorescence cTnT. Pour augmenter la diversité des profils morphologiques des images en fond clair 0 h, assurez-vous que l’ensemble de données comprend des colonies PSC de différentes lignées cellulaires, avec une différenciation initiée sur un intervalle de temps après le passage. Traitez les puits de l’ensemble de données avec les conditions CHIR optimales au stade I.
- Stratégie d’apprentissage automatique au stade I : évaluation précoce et ajustement des doses de CHIR
Ensemble de données : paires de flux d’images en fond clair à 0-12 h et étiquettes de concentration CHIR (faible/optimal/élevé)
Modèle : régression logistique- Préparez un ensemble de données composé de flux d’images en fond clair bien compris, où les images sont acquises toutes les heures entre 0 h et 12 h. Consigner les doses de CHIR (c.-à-d. les combinaisons de concentrations de CHIR et de durée) de chaque puits. Assurez-vous que l’ensemble de données comprend différents lots et puits avec des doses CHIR titrées (p. ex., concentration = 2, 4, 6, 8, 10 et 12 μM ; durée = 24, 36 et 48 h) pour augmenter la diversité. Basculez le répertoire de travail sur « ./stage_I » ; enregistrez les images sous le nom « ./data/image/CD01-*/S*/T*.png », où CD01-* (CD01-1, CD01-2, CD01-3, CD01-4) est le nom du lot, S* (S1, S2, ..., S96) est l’indice du puits et T*.png (T1.png, T2.png, ..., T10.png) est l’image en fond clair prétraitée du puits à différents pas de temps du flux d’images.
- Dans chaque lot, calculez le pourcentage de cellules cTnT+ pour chaque puits, qui est calculé en divisant le nombre de cellules cTnT+ par le nombre total de cellules Hoechst+ dans l’image de fluorescence. Faites la moyenne du pourcentage de cellules cTnT+ sur des puits parallèles avec la même condition de dose CHIR. Enregistrez la condition de dose CHIR et le pourcentage de cellules cTnT+ pour chaque puits dans un fichier csv « ./data/image/CD01-*/CD01- ?_exp_config.csv ».
- Dans chaque lot, déterminer la plage de concentration CHIR « faible », « optimale » et « élevée » pour chaque durée CHIR selon les critères suivants. Les concentrations CHIR avec un pourcentage moyen de cellules cTnT+ au-dessus d’un seuil (p. ex., 20 %) sont identifiées comme « optimales ». Les concentrations CHIR au-delà de la plage optimale sont identifiées comme « faibles » ou « élevées ». Pour chaque durée CHIN, étiquetez les puits comme étant « faible », « optimal » et « élevé » en fonction de leurs concentrations CHIR correspondantes.
REMARQUE : Empiriquement, il existe une corrélation négative entre les concentrations CHIR et la durée des puits à haut rendement au sein d’un lot : pour une durée CHIR plus élevée, les concentrations optimales diminuent. En d’autres termes, l’optimalité des concentrations de CHIR dépend de la durée du CHIR et, par conséquent, les étiquettes des puits diffèrent entre les différentes durées de CHIR. - Dans chaque lot, pour chaque durée CHIR, calculez la concentration ΔCHIR pour chaque concentration c afin de quantifier son écart par rapport à l’optimum. Indiquer la plage de concentration optimale du CHIR par [c1, c2], puis la concentration ΔCHIR (c) = c - c1 (si c < c1), 0 (si c1 ≤ c ≤ c2) ou c - c 2 (si c > c2).
- Extraire les caractéristiques des flux d’images dans l’ensemble de données, en s’assurant qu’ils reflètent les changements morphologiques des cellules au cours de l’étape I. Pour calculer les caractéristiques de l’image concernant l’entropie locale, la surface, les circonférences, la luminosité, le flux optique et la dimension fractale, exécutez
cd ./données
python compute_features.py
CD..
REMARQUE : Pour mettre en évidence les changements morphologiques, les valeurs de certaines caractéristiques (par exemple, la surface, les circonférences et le flux optique) peuvent être normalisées par leurs valeurs de base initiales dans les flux d’images. - Divisez de manière aléatoire l’ensemble de données en un ensemble d’apprentissage et un ensemble de test. Exécutez le bloc-notes Jupyter « ./data/prepare_dataset.ipynb » pour fractionner le jeu de données et organiser les jeux de données sous forme de trames de données (Figure 3D).
- Pour chaque durée CHIR, entraînez un modèle de régression logistique pour prédire l’étiquette de concentration CHIR (faible/optimale/élevée) à partir des caractéristiques du flux d’images sur le jeu d’apprentissage. Évaluez les performances de classification du modèle de régression logistique entraîné sur l’ensemble de test à l’aide de l’exactitude, de la précision, du rappel, du score F1 et de l’aire sous la courbe (AUC). Exécutez le Jupyter Notebook « machine_learning.ipynb » pour l’entraînement et les tests (Figure 3E).
- Évaluez les performances du modèle dans l’évaluation de la dose CHIR. Dans l’ensemble de test, fusionnez les étiquettes prédites de puits parallèles avec la même concentration CHIR à l’aide de scores d’écart (allant de -1 à 1), définis par
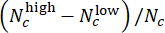 , où Nc désigne le nombre de puits avec une concentration c et
, où Nc désigne le nombre de puits avec une concentration c et 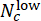
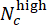 et les puits parmi eux sont prédits comme « faible » et « élevé », respectivement. Confirmez que les scores d’écart prédits sont fortement corrélés avec la concentration réelle de ΔCHIR pour chaque dose CHIR à l’aide du coefficient de corrélation de Pearson.
et les puits parmi eux sont prédits comme « faible » et « élevé », respectivement. Confirmez que les scores d’écart prédits sont fortement corrélés avec la concentration réelle de ΔCHIR pour chaque dose CHIR à l’aide du coefficient de corrélation de Pearson. - Effectuez une validation inter-lots pour tester les performances de généralisation du modèle. À chaque tour, laissez un lot pour le test tout en utilisant les autres pour l’entraînement. Comparez les scores d’écart prédits et la concentration réelle de ΔCHIR pour chaque dose de CHIR dans chaque lot d’essai à l’aide du coefficient de corrélation de Pearson. Exécutez le bloc-notes Jupyter « cross_batch_validation.ipynb » pour implémenter la validation inter-lots (Figure 3F).
REMARQUE : Pour une durée CHIR h sélectionnée, la prédiction de tous les puits avec concentration à partir du modèle de régression logistique entraîné sous la durée h sera convertie en un score d’écart, reflétant l’optimalité de la concentration CHIR c. Un score d’écart proche de 0 suggère que la concentration c est prédite optimale sous la durée h, tandis qu’un score d’écart négatif ou positif suggère une condition de sous-dosage ou de surdosage, qui peut servir de signal pour un ajustement ultérieur des doses CHIR. Notez que puisque la prédiction est faite tôt (12 h), la durée finale du CHIR et la concentration du CHIR pendant le reste du stade I peuvent encore être ajustées. - Appliquez les modèles de régression logistique entraînés pour évaluer les doses CHIR dans les nouveaux lots. Pour une concentration CHIR donnée, calculez les caractéristiques de flux d’images de 0 à 12 h pour les puits parallèles avec concentration, transmettez les caractéristiques aux modèles de régression logistique entraînés sous différentes durées CHIR et, en conséquence, obtenez les étiquettes prédites pour chaque puits. Sur la base de la prédiction des modèles, calculez les scores d’écart pour évaluer chaque concentration CHIR sous différentes durées CHIR.
- Grâce à l’évaluation de la dose CHIR basée sur un modèle, les puits de secours sous chaque concentration CHIR sous-optimale sont en conséquence, en ajustant leur durée CHIR ou leur concentration vers l’optimum avant 48 h.
- Ajustement de la durée : en fonction de la prédiction du modèle sur la durée optimale du CHIR, ajustez le moment où le milieu contenant le CHIR dans ces puits est basculé sur le milieu de différenciation CM. Si le point temporel optimal n’est pas atteint, jetez ces échantillons.
- Réglage de la concentration : sous la durée CHIR prédéfinie, si la concentration actuelle est prédite comme « faible », ajoutez directement une concentration élevée de CHIR dans le milieu pour augmenter la concentration CHIR. Diluez le CHIR dans du DMSO (50 mM) avec du PBS si nécessaire, ajoutez un volume approprié de liquide et secouez doucement la plaque. Si la concentration actuelle est prédite comme étant « élevée », aspirer un certain volume de milieu contenant du CHIR et compléter avec le même volume de milieu de différenciation CM.
REMARQUE : Dans les 48 premières heures de différenciation, les cellules sécrèteront des facteurs pour améliorer la croissance et la différenciation cellulaires, donc ne changez pas complètement le milieu. De plus, il est nécessaire de maintenir le volume moyen constant dans la différenciation. Par conséquent, assurez-vous que le volume de liquide ajouté est aussi petit que possible lors de l’ajustement de la concentration CHIR.
- Stratégie ML à l’étape II : reconnaissance des CPC engagés dans le CM
Jeu de données : paires d’images en fond clair au jour 6 et masques annotés manuellement de CPC validés par CM
Modèle : ResNeSt38 avec Grad-CAM36 pour l’apprentissage faiblement supervisé- Préparez un ensemble de données composé d’images en fond clair le jour 6. Annotez manuellement les CPC validés par CM dans les images en fond clair en suivant les cellules cTnT+ dans les flux d’images du jour 12 au jour 6.
- Recadrez les images en fond clair et l’annotation manuelle correspondante (appelées masques) des CPC validés par CM en patchs, chaque patch ayant une taille de 1/5,5 de la taille de l’image de l’ensemble du puits. Extrayez les correctifs de manière à ce qu’ils se chevauchent lorsque les correctifs adjacents partagent un chevauchement de 50 % dans l’ensemble d’apprentissage et un chevauchement de 75 % dans l’ensemble de test. Étiquetez les patchs avec ≥30 % de CPC engagés par CM comme « positifs ». Étiquetez les correctifs sans CPC validés par CM comme « négatifs ». Basculez le répertoire de travail sur « stage_II ». Exécutez « ./Crop_and_Reconstruct/mask_crop.m » pour recadrer les images. Exécutez « ./Crop_and_Reconstruct/classification.m » pour attribuer des étiquettes (0 : négatif ; 1 : positif) aux taches en fond clair (Figure 3G).
- Divisez de manière aléatoire les correctifs d’image étiquetés en un ensemble d’apprentissage et un ensemble de test.
- Entraînez un réseau de neurones convolutifs profonds, ResNeSt38 , pour apprendre à classifier ces patchs. Évaluez le ResNeSt entraîné sur le banc d’essai pour confirmer que le modèle démontre une grande précision de classification. Exécutez « train.py » et « test.py » pour l’entraînement et le test, respectivement (Figure 3H).
- Utilisez Grad-CAM36 pour mettre en évidence les régions qui contribuent le plus à l’inférence de ResNeSt, représentées par des cartes thermiques. Binarisez les cartes thermiques (à l’aide d’un seuil, par exemple, 10 % × maximum) pour obtenir les régions CPC prédites validées par CM, qui sont appelées régions CPC IMAGÉES-RECONNUES (IR-CPC).
- Comparez les régions IR-CPC avec des masques annotés manuellement sur l’ensemble de test à l’aide de la précision, du score F1, de la précision, du rappel, de la spécificité et de l’intersection sur l’union (IoU). Comparez la proportion de régions IR-CPC avec des indices d’efficacité de différenciation (calculés à partir des images finales de fluorescence cTnT) à l’aide du coefficient de corrélation de Pearson. Exécutez « ./Evaluation/index_calculate.m » et « ./Evaluation/Pearson_correlation.m » pour l’évaluation des performances (Figure 3I).
- Appliquez le modèle entraîné pour reconnaître les CPC pour les nouveaux lots à l’étape II. Recadrez les images en fond clair de puits entiers en patchs, transmettez les patchs au ResNeSt entraîné et obtenez les régions IR-CPC au niveau du patch, qui sont ensuite fusionnées pour produire les régions IR-CPC de puits entiers. Calculez la fraction de régions IR-CPC comme indicateur de l’efficacité de la différenciation finale. Purifier les CPC-IR pour réduire la contamination cellulaire (décrit ci-dessous).
REMARQUE : Étant donné que le modèle de reconnaissance CPC peut effectuer une évaluation précoce de l’efficacité de la différenciation finale, il peut également servir de lecture à haut débit pour le criblage de petites molécules propices au processus de différenciation, ce qui peut optimiser davantage le protocole de différenciation35.
- Purification sélective par région des CPC reconnus par l’imagerie
REMARQUE : Les expériences de photoactivation sont basées sur un microscope à fluorescence inversée facilité par une platine motorisée.- Selon les régions IR-CPC prédites par le modèle entraîné, jetez les puits qui n’ont pas pu générer de CPC-IR. Pour purifier ces CPC engagés dans le CM, utilisez une sonde photoactivable non cytotoxique, Dual-Activatable Cell Tracker 1 (DACT-1) pour marquer sélectivement les non-CPC39 de manière régionale.
- Dissoudre le DACT-1 (10 mM) dans du DMSO et le conserver à -20 °C. Incuber 6 cellules avec 1 μM DACT-1 dans un milieu de différenciation CM pendant 30 min à 37 °C.
- Inspectez les cellules vivantes au microscope et sélectionnez la région d’intérêt (ROI) sous forme de polygones dans MATLAB. Tracez des lignes parallèles espacées de 20 μm à l’intérieur du polygone et calculez les coordonnées des points d’intersection.
- Réglez la piste mobile de la platine motorisée en fonction des lignes de trace, puis la platine se déplacerait à 0,12 mm/s de sorte qu’une ligne laser de 405 nm (20 μm de diamètre) assemblée sur la platine puisse balayer globalement le retour d’intérêt pour photoactiver les non-CPC de manière restreinte. Après l’irradiation, détectez les cellules marquées par DACT-1 à l’aide d’une raie laser de 561 nm.
- Dissociez les cellules à l’aide de trypsine-EDTA à 0,05 % (diluée par du PBS) à 37 °C pendant 5 à 7 minutes, et secouez doucement la plaque dans un incubateur pendant 2 minutes. Filtrer les cellules à travers une crépine de 40 μm et centrifuger à 500 × g pendant 3 min. Remettre les cellules en suspension dans du BSA à 0,5 % et maintenir le tube sur de la glace jusqu’au tri. Préparez également un échantillon non irradié comme témoin négatif pour le tri.
- Démarrez le système de tri par cytométrie en flux et configurez le logiciel de tri. Choisissez les lasers de longueur d’onde de 561 nm et utilisez un filtre passe-bande de 610/20 pour le tri DACT1+. Pour chaque échantillon, utilisez un outil de contrôle polygonal pour entourer la population de cellules dans un graphique bivarié de diffusion directe (FSC) et de diffusion latérale (SSC) pour sélectionner la cellule unique. Ensuite, exécutez le contrôle négatif pour confirmer la fluorescence de fond, analysez l’échantillon irradié de manière sélective par région et tracez la porte pour la population DACT1+ .
- Collecter et remettre en suspension les cellules triées dans un milieu de maintenance CM avec 10 % de FBS et 5 μM Y27632, et ensemencer les cellules triées dans une plaque à 96 puits recouverte de Matrigel.
- Le lendemain, changez le milieu pour un milieu d’entretien CM et attendez de récolter des CM de haute pureté.
- Stratégie ML à l’étape III : reconnaissance des MC
Jeu de données : paires d’images en fond clair du jour 12 et des images finales de fluorescence cTnT
Modèle : pix2pix37- Préparez un ensemble de données composé d’images en fond clair de MC et des images de fluorescence cTnT correspondantes. Pour augmenter la diversité, assurez-vous que l’ensemble de données comprend des puits avec différentes efficacités de différenciation et lignées cellulaires.
- Divisez de manière aléatoire l’ensemble de données en un ensemble d’apprentissage et un ensemble de test. Basculez le répertoire de travail sur « stage_III ». Enregistrez les images en fond clair et les images de fluorescence cTnT correspondantes dans « ./pix2pix/datasets/(CM|CM_new_cell_lines)/A/(train|test)/*.png » et « .../B/(train|test)/*.png », respectivement. Exécutez la commande suivante :
cd pix2pix/datasets
python combine_A_and_B.py --fold_A ./CM/A --fold_B ./CM/B --fold_AB ./CM/ --no_multiprocessing
CD.. - Entraînez le pix2pix model37 sur le kit d’entraînement. Exécutez la commande suivante (Figure 3J) :
python train.py --dataroot ./datasets/CM --name brightfield2fluorescence --model pix2pix --input_nc 1 --output_nc 1 --load_size 1536 --crop_size 256 --lr 2e-4 --n_epochs 1000 --n_epochs_decay 0 --norm instance --netD n_layers --n_layers_D 1 --batch_size 16 --direction AtoB --save_epoch_freq 100 --dataset_mode aligné --use_resize_conv --seed 1234 - Appliquez le modèle pix2pix entraîné sur le kit de test. Exécutez la commande suivante :
python test.py --dataroot ./datasets/CM --name brightfield2fluorescence --model pix2pix --direction AtoB --input_nc 1 --output_nc 1 --load_size 1536 --crop_size 1536 --use_resize_conv --eval --num_test 1000 - Comparez les images de fluorescence prédites par le modèle avec les vraies images (c’est-à-dire obtenues expérimentalement). Au niveau du pixel, comparez les intensités prédites et les intensités réelles à l’aide du coefficient de corrélation de Pearson. De plus, au niveau de l’image, comparez les indices d’efficacité de différenciation prédits avec les vrais à l’aide du coefficient de corrélation de Pearson. Exécutez « stage_III/evaluation/pixel_correlation.m » et « image_correlation.ipynb » pour ces comparaisons (Figure 3K,L).
- Appliquez le modèle entraîné pour reconnaître les CM dans les nouveaux lots à l’étape III. Transmettez de nouvelles images en fond clair de CM au modèle pix2pix entraîné et obtenez une prédiction in silico des images de fluorescence et de l’efficacité de différenciation.
REMARQUE : Pour réduire la contamination cellulaire, le tri cellulaire photoactivé basé sur DACT-1 est également applicable pour purifier les MC reconnus par le modèle.
Résultats
Sur la base de l’imagerie en fond clair et du ML, le processus global de différenciation peut être surveillé et optimisé de manière intelligente. Au stade de la CSP, nous avons développé un modèle ML capable de prédire l’efficacité finale de la différenciation en fonction des caractéristiques morphologiques des colonies initiales de CSP, afin de déterminer le moment le plus approprié pour initier la différenciation (Figure 4A,B
Discussion
Ici, nous avons décrit un protocole détaillé pour surmonter l’un des principaux problèmes de l’application et de la traduction actuelles de la CSP : la variabilité de la différenciation cellulaire. En exploitant l’imagerie en fond clair de cellules vivantes et le ML, nous avons optimisé de manière itérative la différenciation des CSP pour obtenir une efficacité élevée et constante entre les lignées cellulaires et les lots. Cependant, dans le processus de différenciat...
Déclarations de divulgation
Yang Zhao, Jue Zhang, Xiaochun Yang, Yao Wang et Daichao Chen déposent un brevet pour la stratégie de différenciation PSC rapportée dans cet article (202210525166.X).
Remerciements
Nous remercions Qiushi Sun, Yao Wang, Yu Xia, Jinyu Yang, Chang Lin, Zimu Cen, Dongdong Liang, Rong Wei, Ze Xu, Guangyin Xi, Gang Xue, Can Ye, Li-Peng Wang, Peng Zou, Shi-Qiang Wang, Pablo Rivera-Fuentes, Salome Püntener, Zhixing Chen, Yi Liu et Jue Zhang, d’avoir jeté les bases de cette stratégie. Ce travail a été soutenu par le Programme national de R&D clé de la Chine (2018YFA0800504, 2019YFA0110000) et le Projet d’expérience médicale spatiale du Programme spatial habité de Chine (HYZHXM01020) à Yang Zhao. La figure 1 a été créée avec BioRender.com.
matériels
| Name | Company | Catalog Number | Comments |
| 0.25% Trypsin-EDTA | Gibco | 25200056 | Diluted digests were used for CPC and CM digestion |
| 4% Paraformaldehyde in PBS | KeyGEN BioTECH | KGIHC016 | |
| 6-well Cell Culture Plate | NEST | 703001 | |
| 96-well Cell Culture Plate | NEST | 701001 | |
| B27 Supplement | Gibco | 17504044 | |
| B27 Supplement Minus Insulin | Gibco | A1895601 | |
| Bovine serum albumin (BSA) | GPC BIOTECH | AA904-100G | |
| Celldiscoverer 7 | Zeiss | Instruments used to take bright-field images throughout differentiation and final cTnT images | |
| CHIR99021 | Selleck | S1263 | |
| DMEM/F12 | Gibco | 12634010 | |
| Donkey anti-Mouse IgG (H+L) Highly Cross-Adsorbed Secondary Antibody, Alexa Fluor 488 | Thermo | A-21202 | Secondary Antibody |
| FACSAria III | BD Biosciences | Flow cytometry sorter | |
| Fetal Bovine Serum (FBS) | VISTECH | SE100-B | |
| Hoechst 33342 | YEASEN | 40732ES03 | |
| Human Pluripotent Stem Cell Chemical-defined Medium | Cauliscell Inc | 400105 | Basal medium of PSC preparation medium |
| iPS-18 | TaKaRa | Y00300 | |
| iPS-B1 | Cellapy | CA4025106 | |
| iPS-F | Nuwacell | RC01001-B | |
| iPS-M | Nuwacell | RC01001-A | |
| IWR1-1-endo | Selleck | S7086 | IWR1 |
| Jupyter Notebook | N/A | Version 6.4.0 | https://jupyter.org/ |
| MATLAB | MathWorks | Version R2020a | Software for scientific computation and image annotation |
| Matrigel Matrix | Corning | 354230 | Matrigel |
| Mouse monoclonal IgG1 anti-cTnT | Thermo | MA5-12960 | cTnT primary antibody |
| Normal Donkey Serum | Jackson | 017-000-121 | |
| ORCA-Flash 4.0 V3 digital CMOS camera | Hamamatsu | C13440-20CU | The digital camera assembled on Celldiscoverer7 |
| PBS | NEB | 21-040-CVR | |
| Penicillin-Streptomycin | Gibco | 15140-122 | |
| Pluripotency Growth Mater 1 basal medium | Cellapy | CA1007500-1 | Basal medium of PSC culture medium |
| Pluripotency Growth Mater 1 supplement | Cellapy | CA1007500-2 | Supplement of PSC culture medium |
| Prism | Graphpad | Version 8/9 | Statistical software for statistical analysis and plotting |
| Python | N/A | version 3.6 | Python 3 environment for scientific computation, with packages pytorch (1.9.0), numpy, scipy, pandas, visdom, scikit-learn, scikit-image, opencv-python, and matplotlib software for scientific computation and image annotation. |
| RPMI 1640 | Gibco | 11875176 | |
| Supplement hPSC-CDM (500x) | Cauliscell Inc | 00015 | Supplement of PSC preparation medium |
| TiE | Nikon | An inverted fluorescence microscope (with modification) for region-selevtive purification | |
| Triton X-100 | Amresco | 9002-93-1 | |
| Versene Solution | Thermo | 15040066 | EDTA solution for PSC digestion |
| Y27632 | Selleck | S6390 | |
| Zen | Zeiss | Version 3.1 | A supporting software of Celldiscoverer7 for image acquisition, processing and analysis |
Références
- Yoshida, Y., Yamanaka, S. Induced pluripotent stem cells 10 years later: for cardiac applications. Circ Res. 120 (12), 1958-1968 (2017).
- Shi, Y., Inoue, H., Wu, J. C., Yamanaka, S. Induced pluripotent stem cell technology: a decade of progress. Nat Rev Drug Discov. 16 (2), 115-130 (2017).
- Takahashi, K., et al. Induction of pluripotent stem cells from adult human fibroblasts by defined factors. Cell. 131 (5), 861-872 (2007).
- Burridge, P. W., et al. Chemically defined generation of human cardiomyocytes. Nat Methods. 11 (8), 855-860 (2014).
- Mordwinkin, N. M., Burridge, P. W., Wu, J. C. A review of human pluripotent stem cell-derived cardiomyocytes for high-throughput drug discovery, cardiotoxicity screening, and publication standards. J Cardiovasc Transl Res. 6 (1), 22-30 (2013).
- Vanden Berg, C. W., Elliott, D. A., Braam, S. R., Mummery, C. L., Davis, R. P. Differentiation of human pluripotent stem cells to cardiomyocytes under defined conditions. Methods Mol Biol. 1353, 163-180 (2016).
- Burridge, P. W., Holmström, A., Wu, J. C. Chemically defined culture and cardiomyocyte differentiation of human pluripotent stem cells. Curr Protoc Hum Genet. 87, 1-15 (2015).
- Li, J., et al. Human pluripotent stem cell-derived cardiac tissue-like constructs for repairing the infarcted myocardium. Stem Cell Rep. 9 (5), 1546-1559 (2017).
- Smith, A. S. T., Macadangdang, J., Leung, W., Laflamme, M. A., Kim, D. -. H. Human iPSC-derived cardiomyocytes and tissue engineering strategies for disease modeling and drug screening. Biotechnol Adv. 35 (1), 77-94 (2017).
- Sharma, A., et al. Use of human induced pluripotent stem cell-derived cardiomyocytes to assess drug cardiotoxicity. Nat Protoc. 13 (12), 3018-3041 (2018).
- Mehta, A., et al. Identification of a targeted and testable antiarrhythmic therapy for long-QT syndrome type 2 using a patient-specific cellular model. Eur Heart J. 39 (16), 1446-1455 (2018).
- Cahan, P., Daley, G. Q. Origins and implications of pluripotent stem cell variability and heterogeneity. Nat Rev Mol Cell Biol. 14 (6), 357-368 (2013).
- Kim, K., et al. Epigenetic memory in induced pluripotent stem cells. Nature. 467 (7313), 285-290 (2010).
- Rouhani, F., et al. Genetic background drives transcriptional variation in human induced pluripotent stem cells. PLoS Genet. 10 (6), e1004432 (2014).
- Kim, K., et al. Donor cell type can influence the epigenome and differentiation potential of human induced pluripotent stem cells. Nat Biotechnol. 29 (12), 1117-1119 (2011).
- Garitaonandia, I., et al. Increased risk of genetic and epigenetic instability in human embryonic stem cells associated with specific culture conditions. PLoS One. 10 (2), e0118307 (2015).
- Engler, A. J., Sen, S., Sweeney, H. L., Discher, D. E. Matrix elasticity directs stem cell lineage specification. Cell. 126 (4), 677-689 (2006).
- Matoba, N., Yamashita, T., Takayama, K., Sakurai, F., Mizuguchi, H. Optimal human iPS cell culture method for efficient hepatic differentiation. Differentiation. 104, 13-21 (2018).
- Volpato, V., et al. Reproducibility of molecular phenotypes after long-term differentiation to human iPSC-derived neurons: A multi-site omics study. Stem Cell Rep. 11 (4), 897-911 (2018).
- Anderson, N. C., et al. Balancing serendipity and reproducibility: Pluripotent stem cells as experimental systems for intellectual and developmental disorders. Stem Cell Rep. 16 (6), 1446-1457 (2021).
- Sepac, A., et al. Comparison of cardiomyogenic potential among human ESC and iPSC lines. Cell Transplant. 21 (11), 2523-2530 (2012).
- Laco, F., et al. Unraveling the inconsistencies of cardiac differentiation efficiency induced by the GSK3β inhibitor CHIR99021 in human pluripotent stem cells. Stem Cell Rep. 10 (6), 1851-1866 (2018).
- Volpato, V., Webber, C. Addressing variability in iPSC-derived models of human disease: guidelines to promote reproducibility. Dis Model Mech. 13 (1), 042317 (2020).
- Hu, B. -. Y., et al. Neural differentiation of human induced pluripotent stem cells follows developmental principles but with variable potency. Proc Natl Acad Sci USA. 107 (9), 4335-4340 (2010).
- Huo, J., et al. Evaluation of batch variations in induced pluripotent stem cell-derived human cardiomyocytes from 2 major suppliers. Toxicol Sci. 156 (1), 25-38 (2017).
- Rostam, H. M., Reynolds, P. M., Alexander, M. R., Gadegaard, N., Ghaemmaghami, A. M. Image based machine learning for identification of macrophage subsets. Sci Rep. 7 (1), 3521 (2017).
- Christiansen, E. M., et al. In silico labeling: Predicting fluorescent labels in unlabeled images. Cell. 173 (3), 792-803 (2018).
- Buggenthin, F., et al. Prospective identification of hematopoietic lineage choice by deep learning. Nat Methods. 14 (4), 403-406 (2017).
- Smith, Z. D., Nachman, I., Regev, A., Meissner, A. Dynamic single-cell imaging of direct reprogramming reveals an early specifying event. Nat Biotechnol. 28 (5), 521-526 (2010).
- Fan, K., Zhang, S., Zhang, Y., Lu, J., Holcombe, M., Zhang, X. A machine learning assisted, label-free, non-invasive approach for somatic reprogramming in induced pluripotent stem cell colony formation detection and prediction. Sci Rep. 7 (1), 13496 (2017).
- Kusumoto, D., et al. Automated deep learning-based system to identify endothelial cells derived from induced pluripotent stem cells. Stem Cell Rep. 10 (6), 1687-1695 (2018).
- Zhu, Y., et al. Deep learning-based predictive identification of neural stem cell differentiation. Nat Commun. 12 (1), 2614 (2021).
- Qian, T., et al. Label-free imaging for quality control of cardiomyocyte differentiation. Nat Commun. 12 (1), 4580 (2021).
- Ounkomol, C., Seshamani, S., Maleckar, M. M., Collman, F., Johnson, G. R. Label-free prediction of three-dimensional fluorescence images from transmitted-light microscopy. Nat Methods. 15 (11), 917-920 (2018).
- Yang, X., et al. A live-cell image-based machine learning strategy for reducing variability in PSC differentiation systems. Cell Discovery. 9 (1), 53 (2023).
- Selvaraju, R. R., et al. Grad-CAM: Visual explanations from deep networks via gradient-based localization. Proc IEEE Int Conf Computer Vision (ICCV). , 618-626 (2017).
- Isola, P., Zhu, J. -. Y., Zhou, T., Efros, A. A. Image-to-image translation with conditional adversarial networks. Proc IEEE Conf Computer Vision Pattern Recognition (CVPR). , 5967-5976 (2017).
- Zhang, H., et al. ResNeSt: Split-attention networks. Proc IEEE/CVF Conf Computer Vision Pattern Recognition Workshops (CVPRW). , 2735-2745 (2022).
- Halabi, E. A., et al. Dual-activatable cell tracker for controlled and prolonged single-cell labeling). ACS Chem. Biol. 15 (6), 1613-1620 (2020).
- Hannan, N. R. F., Segeritz, C. -. P., Touboul, T., Vallier, L. Production of hepatocyte-like cells from human pluripotent stem cells. Nat Protoc. 8 (2), 430-437 (2013).
- Zhang, H., Shen, M., Wu, J. C. Generation of quiescent cardiac fibroblasts derived from human induced pluripotent stem cells. Methods Mol Biol. 2454, 109-115 (2022).
- Harding, M. J., McGraw, H. F., Nechiporuk, A. The roles and regulation of multicellular rosette structures during morphogenesis. Development. 141 (13), 2549-2558 (2014).
- Najm, F. J., et al. Rapid and robust generation of functional oligodendrocyte progenitor cells from epiblast stem cells. Nat Methods. 8 (11), 957-962 (2011).
- Hogrebe, N. J., Maxwell, K. G., Augsornworawat, P., Millman, J. R. Generation of insulin-producing pancreatic β cells from multiple human stem cell lines. Nat Protoc. 16 (9), 4109-4143 (2021).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon