È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Una strategia di apprendimento automatico basata su immagini di cellule vive per monitorare la differenziazione delle cellule staminali pluripotenti
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
I sistemi di differenziazione da cellule staminali pluripotenti (PSC) a cellule funzionali sono attualmente ostacolati da problemi di grave variabilità da linea a linea e da lotto a lotto. Qui, utilizzando la differenziazione cardiaca come esempio principale, presentiamo un protocollo per monitorare e modulare in modo intelligente il processo di differenziazione della PSC basato sull'apprendimento automatico basato su immagini.
Abstract
Le tecnologie delle cellule staminali pluripotenti (PSC) sono state ampiamente utilizzate nella scoperta di farmaci, nella modellazione delle malattie e nella medicina rigenerativa. Tuttavia, i sistemi di differenziazione PSC-cellula funzionale disponibili sono ostacolati da problemi di grave variabilità da linea a linea e da lotto a lotto. È quindi importante un controllo preciso della differenziazione cellulare in tempo reale. In questo protocollo, descriviamo una strategia non invasiva e intelligente che supera la variabilità nella differenziazione cellulare utilizzando l'apprendimento automatico basato su immagini in campo chiaro. Prendendo come esempio la differenziazione da PSC a cardiomiociti, questa metodologia fornisce informazioni dettagliate per il controllo dello stato iniziale della PSC, la valutazione precoce e l'intervento in condizioni di differenziazione e l'eliminazione della contaminazione cellulare disdifferenziata, realizzando insieme una differenziazione costantemente di alta qualità dalle PSC alle cellule funzionali. In linea di principio, questa strategia può essere estesa ad altri sistemi di differenziazione o riprogrammazione cellulare con più passaggi per supportare la produzione cellulare, nonché per approfondire la nostra comprensione dei meccanismi durante la conversione del destino cellulare.
Introduzione
Le cellule staminali pluripotenti (PSC) possiedono la notevole capacità di differenziarsi in molti tipi di cellule in vitro. Queste cellule funzionali differenziate potrebbero essere utilizzate per la terapia cellulare, la modellazione di malattie e lo sviluppo di farmaci, tutti preziosi per la ricerca o le applicazioni cliniche 1,2,3. Ad esempio, è stata sviluppata una varietà di metodi per differenziare le PSC in cardiomiociti (CM)4,5,6,7. Questi CM possono essere applicati per test di cardiotossicità di farmaci, modellazione di malattie cardiache e trapianto di cellule 8,9,10,11.
Tuttavia, la conversione dalla PSC alle cellule differenziate terminali è un processo graduale e molteplici perturbazioni durante il processo di differenziazione possono portare le cellule a destini cellulari divergenti. Diversi background genetici e segni epigenetici delle linee PSC influenzano il potenziale di differenziamento in un lignaggio specifico 12,13,14,15; anche il numero di passaggi della PSC e le mutazioni geniche accumulate sono fonti di eterogeneità della PSC; Le differenze nelle operazioni sperimentali impiegate da diversi sperimentatori possono anche portare a risultati di differenziazione completamente diversi 16,17,18,19,20. Pertanto, attualmente uno dei problemi principali nella produzione di cellule derivate da PSC è l'instabilità tra le linee cellulari e i lotti 21,22,23,24,25. L'instabilità nella differenziazione della PSC spesso porta a più esperimenti ripetuti, consumando tempo e risorse lavorative significative. Per affrontare questo problema, è fondamentale sviluppare una strategia che minimizzi la variabilità tra linee cellulari e lotti, migliorando così la stabilità e la robustezza del differenziamento.
Recentemente, i progressi nella microscopia ad alta risoluzione e nell'apprendimento automatico (ML) hanno facilitato l'applicazione dell'analisi quantitativa delle immagini basata su ML nella biologia cellulare, rendendo possibile l'utilizzo di informazioni preziose nelle funzioni di imaging cellulare 26,27,28,29,30,31,32,33,34. Nel nostro lavoro precedente, abbiamo proposto una strategia di ML basata su immagini di cellule vive per monitorare e intervenire sullo stato di differenziazione della PSC in tempo reale per migliorare la stabilità e l'efficienza della differenziazione della PSC (Figura 1)35. Prendendo come esempio la differenziazione da PSC a cardiomiociti, abbiamo valutato lo stato iniziale della PSC utilizzando modelli forestali casuali, previsto la condizione di differenziazione ottimale utilizzando modelli di regressione logistica e riconosciuto cellule differenziate con successo utilizzando Grad-CAM36 e pix2pix37 basati sul deep learning. I modelli ML hanno imparato a identificare le linee cellulari da una serie di caratteristiche morfologiche del campo chiaro, comprese le caratteristiche relative all'area, alla circonferenza, alla convessità, alla solidità, alla luminosità, alla velocità di movimento e ad altre caratteristiche implicite estratte da reti neurali convoluzionali profonde. Sulla base dell'inferenza di questi modelli di ML consolidati, abbiamo realizzato il controllo dello stato iniziale della PSC, la valutazione precoce e l'intervento in condizioni di differenziazione e l'eliminazione della contaminazione cellulare disdifferenziata, fornendo insieme una modulazione completa e accurata del processo di differenziazione cardiaca. Qui forniamo un protocollo passo dopo passo per lo sviluppo della strategia.
Access restricted. Please log in or start a trial to view this content.
Protocollo
1. Differenziamento e caratterizzazione cellulare
- Preparazione dei reagenti di coltura e delle piastre di coltura
- Preparare il terreno di coltura della PSC aggiungendo 2 mL di integratore e lo 0,2% di Penicillina-Streptomicina a 48 mL di terreno basale. Aliquotare e conservare l'integratore a -20 °C. Conservare questo terreno a 4 °C per un massimo di 4 settimane.
- Preparare il terreno di preparazione della PSC aggiungendo 1 ml di integratore e lo 0,2% di penicillina-streptomicina a 500 ml di terreno basale. Durante l'uso, preriscaldare il terreno per un utilizzo una tantum e conservare il terreno a 4 °C per un massimo di 3 settimane.
- Preparare il terreno di differenziazione CM aggiungendo 1 integratore di B27 meno insulina e l'1% di penicillina-streptomicina in RPMI 1640. Preparare il terreno di mantenimento CM aggiungendo 1 integratore di B27 e l'1% di penicillina-streptomicina in RPMI 1640. Conservare questi due tipi di terreno a 4 °C per 2 settimane.
- Immergere il Matrigel nel ghiaccio e mantenerlo a 4 °C per una notte. Assicurarsi che Matrigel sia completamente scongelato; quindi, aliquotare in aliquote da 1 mL conservate a -80 °C per un uso successivo e aggiungere 1 mL di Matrigel in 49 mL di DMEM/F12 a 4 °C per preparare la soluzione di lavoro Matrigel. Aggiungere 850 μl di questa soluzione di lavoro Matrigel per pozzetto di una piastra di coltura a 6 pozzetti e incubare la piastra a 37 °C per almeno 30 minuti. Per la piastra di coltura a 96 pozzetti utilizzata nella differenziazione CM, aggiungere 35 μl di soluzione di lavoro Matrigel per pozzetto.
NOTA: Tenere Matrigel sempre sul ghiaccio; i puntali e le provette delle pipette devono essere preraffreddati a 4 °C. La soluzione di lavoro Matrigel può essere conservata a 4 °C per 2 settimane. Prestare attenzione a evitare bolle durante il pipettaggio della soluzione di lavoro Matrigel.
- Manutenzione e passaggio PSC
- Mantenere le PSC in piastre di coltura a 6 pozzetti. Confermare la densità cellulare al microscopio e prepararsi per il passaggio quando la confluenza raggiunge ~70% (Figura 2A).
- Utilizzare l'EDTA per digerire le cellule per il passaggio. Scaldare il terreno di coltura PSC, PBS e EDTA in un bagno d'acqua a 37 °C e aggiungere Y27632 nel terreno fino a una concentrazione finale di 5 μM.
- Lavare le PSC con 1 mL di PBS, aggiungere 1 mL di EDTA per pozzetto e incubare la piastra in un'incubatrice a 37 °C per 3 minuti.
- Aspirare l'EDTA prima che le cellule si stacchino dalla piastra di coltura, quindi utilizzare 1 mL di terreno di coltura PSC per sciacquare delicatamente il fondo del pozzetto 3-4 volte per risospendere le cellule.
NOTA: Questo ci permette di saltare la fase di centrifugazione e ridurre i danni alle celle nelle operazioni di passaggio. - Aspirare il DMEM/F12 da un pozzetto rivestito di Matrigel di una piastra di coltura a 6 pozzetti e aggiungere 2 mL di terreno di coltura PSC fresco per pozzetto (aggiunto Y27632). Aggiungere la sospensione cellulare ai pozzetti con il rapporto di divisione da 1:6 a 1:12. Posizionare la piastra nell'incubatore a 37 °C con il 5% di CO2.
- Cambiare il terreno di coltura PSC (senza Y27632) da 12 a 24 ore dopo il passaggio; Quindi, cambia il mezzo e controlla lo stato e la confluenza della cella ogni giorno.
- Differenziazione cardiaca delle PSC
- Per la differenziazione CM, seminare le PSC in una piastra di coltura a 96 pozzetti nel terreno di preparazione PSC (aggiunto Y27632). Per la differenziazione CM, seguire gli stessi passaggi precedenti (passaggi 1.2.2-1.2.6) per seminare le PSC in una piastra di coltura a 96 pozzetti nel terreno di preparazione PSC (aggiunto Y27632).
NOTA: In questa fase, la durata del trattamento con EDTA potrebbe essere opportunamente estesa (5 minuti o più) per dissociare sufficientemente le PSC in modo che i cloni possano essere più piccoli dopo il passaggio. Il nostro studio ha dimostrato che all'interno della stessa area, le colonie più piccole favoriscono la differenziazione. - Stadio I: quando le PSC raggiungono l'80-90% di confluenza, passare dal mezzo al mezzo di differenziazione CM con 2-20 μM CHIR99021 (CHIR) (Figura 2B). Il punto temporale della commutazione del mezzo è indicato come giorno 0. Dopo 24-48 ore di trattamento CHIR, cambiare il mezzo di differenziazione CM con quello fresco.
NOTA: CHIR è un inibitore di GSK3beta per l'induzione del mesoderma. La dose ottimale di CHIR per la differenziazione di CM varia a seconda delle diverse linee cellulari e dei lotti. Ad esempio, la dose ottimale di CHIR è di 8-10 μM, 48 ore per iPS-B1. Per raccogliere diverse immagini in campo chiaro per l'addestramento del modello ML, qui abbiamo titolato le concentrazioni di CHIR per ogni lotto di esperimenti per diverse efficienze di differenziazione. - Fase II: A 72 ore (giorno 3), sostituire il terreno con il terreno di differenziazione CM integrato con 5 μM IWR1 e coltura per 48 ore (giorno 5). Cambiare il mezzo con il mezzo di differenziazione CM per 1-2 giorni (fino ai giorni 6-7). A questo punto, le PSC si differenziano in cellule progenitrici cardiache (CPC).
- Fase III: Sostituire il mezzo con un mezzo di manutenzione CM e sostituire il mezzo ogni 3 giorni. Il giorno 10 o il giorno 12, raccogli il CM per ulteriori analisi.
- Per la differenziazione CM, seminare le PSC in una piastra di coltura a 96 pozzetti nel terreno di preparazione PSC (aggiunto Y27632). Per la differenziazione CM, seguire gli stessi passaggi precedenti (passaggi 1.2.2-1.2.6) per seminare le PSC in una piastra di coltura a 96 pozzetti nel terreno di preparazione PSC (aggiunto Y27632).
- Colorazione in immunofluorescenza di CM
- Al giorno 10 o al giorno 12, lavare le cellule con PBS e fissarle con paraformaldeide al 4% in PBS per 15 minuti a temperatura ambiente. Lavare i CM 2 volte con PBS e conservare la piastra a 4 °C per un massimo di ~1 settimana.
NOTA: La paraformaldeide è un reagente tossico con un odore pungente. Operare in una cappa chimica e proteggere occhi e mani. - Al momento della colorazione, trattare le cellule con soluzione permeabilizzante (0,1% Triton X-100 in PBS) per 15 min a temperatura ambiente; quindi, incubare le cellule in soluzione bloccante (Triton X-100 allo 0,1% contenente il 3% di siero d'asino in PBS) per 30 minuti a temperatura ambiente.
- Incubare il campione con l'anticorpo primario della troponina T cardiaca (cTnT) (1:300) diluito in soluzione bloccante per una notte a 4 °C per identificare i CM.
- Raccogliere l'anticorpo primario e lavare le cellule con PBS 3x. Incubare il campione con anticorpi secondari in PBS con albumina sierica bovina all'1% per 1 ora a 37 °C in un ambiente buio.
NOTA: L'anticorpo primario raccolto potrebbe essere riutilizzato per 2-3 volte in 1 settimana.
- Raccogliere l'anticorpo primario e lavare le cellule con PBS 3x. Incubare il campione con anticorpi secondari in PBS con albumina sierica bovina all'1% per 1 ora a 37 °C in un ambiente buio.
- Rimuovere l'anticorpo secondario dalle cellule e lavare le cellule 3 volte con PBS. Utilizzare Hoechst 33342 (1:1.000 in PBS) per colorare i nuclei per 5 minuti a temperatura ambiente. Sciacquare la cella 3 volte con PBS e aggiungere 100 μl di PBS per pozzetto per evitare che si secchi. Conservare la lastra a 4 °C in un luogo buio e acquisire immagini fluorescenti entro 1 settimana.
- Al giorno 10 o al giorno 12, lavare le cellule con PBS e fissarle con paraformaldeide al 4% in PBS per 15 minuti a temperatura ambiente. Lavare i CM 2 volte con PBS e conservare la piastra a 4 °C per un massimo di ~1 settimana.
2. Acquisizione del flusso di immagini durante il processo di differenziazione
- Impostazione del disegno sperimentale per la raccolta di immagini
- Utilizzare un microscopio automatizzato a supporto della coltura e dell'imaging di cellule vive per raccogliere immagini in campo chiaro di diversi stadi della differenziazione CM e immagini in immunofluorescenza cTnT dell'esito della differenziazione (Figura 2A-E). Utilizzare il software di supporto del microscopio per progettare il programma dell'esperimento e controllare il microscopio.
- Apri il software e crea un nuovo progetto sperimentale. Scegli un obiettivo 5x e una lente tubolare 2x per l'imaging. Controlla le opzioni Tiles e Z-stack.
- Aggiungere il canale TL Brightfield per l'imaging in campo chiaro nel menu Canali. Aggiungere i canali AF488 e H3342 per l'imaging in immunofluorescenza. Modificare il percorso della luce nel menu Imaging Setup per l'imaging con la fotocamera CMOS digitale esterna.
- Aprire il menu Modalità di acquisizione e impostare il binning 2 x 2 per aumentare il rapporto segnale/rumore.
- Apri il menu Z-stack e scegli il modello Center. Impostare il numero di sezioni e gli intervalli durante la scansione. Scegli 3-5 fette con intervalli di 3-6 μm per immagini a fuoco in analisi.
- Nella finestra Navigazione e riquadri, impostare le aree dei riquadri in base al vettore e impostare 25 riquadri (5 colonne x 5 righe) per un pozzetto. Nel menu Riquadri, selezionare il modello Multiwell 96 in Sample Carrier e impostare una sovrapposizione del 5%-15% sulle regioni adiacenti.
- Se è necessaria l'imaging continuo, selezionare l'opzione Seriedi immagini e impostare la durata e l'intervallo nel menu Serie temporali. Per raccogliere il flusso di immagini nella fase I, impostare una durata di 12 ore senza intervalli.
- Acquisizione di immagini in campo chiaro e in immunofluorescenza
- Per acquisire le immagini, inserire prima la piastra di coltura cellulare nel vassoio del campione e caricare il campione all'interno del microscopio. Selezionare il modello Multiwell 96 in modo che corrisponda al progetto sperimentale. Se il campione è costituito da cellule vive, aprire il sistema di riscaldamento e la pompa di CO2 per mantenere le condizioni appropriate per la coltura (37 °C, 5% CO2).
- Apri il progetto sperimentale preimpostato e salvalo come nuovo. Apri il menu Piastrelle e calibra manualmente la posizione della piastra. Nella finestra Navigazione e piastrelle, selezionare i pozzetti necessari e fare clic su Crea per costruire le regioni di tessere per questi pozzetti.
- Fare clic su Verifica regioni riquadro nel menu Pannelli ed eseguire la messa a fuoco automatica per verificare tutti i pozzetti. Quindi, correggi manualmente la messa a fuoco di ciascun pozzetto sotto il campo chiaro scorrendo la rotellina del mouse.
- Spuntare i canali necessari e impostare i tempi di esposizione nel menu Canali. I tempi di esposizione consigliati sono 1,625 ms per Bright, 100 ms per H3342 e 550 ms per AF488. Esegui un singolo scatto per controllare le immagini di tutti i canali.
- Infine, fai clic sul pulsante Avvia esperimento e attendi l'imaging automatico. In genere, sono necessarie ~1,2 ore per completare la scansione di un'intera piastra di coltura da 96 pozzetti (96 pozzetti, tre strati, 25 tessere per ogni pozzetto sotto il campo chiaro). Nel framework di elaborazione, scegliete Esportazione immagine, selezionate il tipo di file in formato TIFF o PNG non compresso e applicate. Utilizzare le immagini di 9 tessere (3 x 3) al centro del pozzo per ulteriori analisi (Figura 2A-E).
3. Definizione della strategia di ML basata su immagini in ogni fase del processo di differenziazione
- Preparazione del software
- Installa l'ambiente Python, con i pacchetti pytorch (1.9.0), numpy, scipy, scikit-learn, pandas, visdom, scikit-image, opencv-python e matplotlib.
- Installare Jupyter Notebook.
- Installare MATLAB.
- Scarica il codice e i set di dati di esempio da https://github.com/zhaoyanglab/ML-for-PSC-differentiation.
NOTA: Le prestazioni del ML dipendono dalla qualità dei set di dati e dalla scelta dei modelli e degli iperparametri. Non è garantito che l'implementazione di esempio sia ottimale per tutte le circostanze. Gli ingegneri ML devono personalizzare il codice e adattare il modello e gli iperparametri alle loro esigenze specifiche.
- Strategia ML allo stadio PSC: controllo degli stati iniziali delle colonie PSC
Set di dati: coppie di immagini in campo chiaro a 0 h (prima del trattamento CHIR) ed efficienza di differenziazione finale
Modello: foresta casuale- Preparare un set di dati composto da immagini in campo chiaro a 0 h (prima del trattamento CHIR) e le immagini finali di fluorescenza cTnT. Per aumentare la diversità dei profili morfologici delle immagini in campo chiaro a 0 ore, assicurarsi che il set di dati includa colonie di PSC provenienti da diverse linee cellulari, con la differenziazione avviata in un intervallo di tempo dopo il passaggio. Trattare i pozzetti nel set di dati con le condizioni CHIR ottimali allo stadio I.
NOTA: Per garantire che le colonie siano trattate con le condizioni CHIR ottimali, i pozzetti all'interno dello stesso lotto possono essere trattati con dosi di CHIR titolate. Solo i pozzi con condizioni CHIR ad alta efficienza sono inclusi nel set di dati. - Quantificare l'efficienza di differenziazione di ciascun pozzetto in base all'indice di efficienza di differenziazione calcolato dalle sue immagini di fluorescenza cTnT, che è definito da
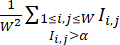 (dove
(dove 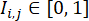 rappresenta l'intensità di fluorescenza nella posizione dei pixel (i, j) nell'immagine di fluorescenza W × W e la somma viene presa su tutte le posizioni dei pixel con intensità superiore a una soglia α, tipicamente impostato a 0,5). All'interno di ciascuna linea cellulare, calcolare l'indice di efficienza di differenziazione massimo su tutti i pozzetti di questa linea cellulare e normalizzare i loro indici di efficienza di differenziazione per il valore massimo.
rappresenta l'intensità di fluorescenza nella posizione dei pixel (i, j) nell'immagine di fluorescenza W × W e la somma viene presa su tutte le posizioni dei pixel con intensità superiore a una soglia α, tipicamente impostato a 0,5). All'interno di ciascuna linea cellulare, calcolare l'indice di efficienza di differenziazione massimo su tutti i pozzetti di questa linea cellulare e normalizzare i loro indici di efficienza di differenziazione per il valore massimo.
NOTA: Poiché il potenziale di differenziazione di ciascuna linea cellulare è intrinsecamente diverso, tale normalizzazione enfatizzerà maggiormente le caratteristiche morfologiche delle colonie condivise tra le varie linee cellulari che contribuiscono al risultato della differenziazione. La normalizzazione infatti rende comparabili le colonie di PSC provenienti da diverse linee cellulari e mitiga il bias indotto dalle linee cellulari. - Quantifica i profili morfologici delle immagini in campo chiaro 0 h in base a caratteristiche ad alta dimensione che rivelano le proprietà della forma della colonia. Per delineare le regioni delle colonie in immagini in campo chiaro, calcolare l'entropia locale (con raggio r); I pixel con entropia locale superiore a una soglia σ sono identificati da coperti da celle. Utilizzare un'impostazione dei parametri di r = 10 pixel e σ = 3. Cambia la directory di lavoro su "./colony_control"; salvare le immagini in campo chiaro, le immagini binarie delle regioni cellulari e le immagini a fluorescenza cTnT a "colony_control/ image_data/CD00-*/[campo chiaro|cell_region|ctnt]/S*.png"; ed eseguire il Jupyter Notebook "Compute_features.ipynb" per calcolare le caratteristiche relative all'area della colonia, alla circonferenza, alla solidità, alla convessità, alla circolarità e alle distanze del contorno del centroide, tra le altre.
- Dividi in modo casuale il set di dati in un set di addestramento e un set di test, con il set di addestramento che comprende tra il 50% e l'80% dei dati complessivi. Eseguire il blocco finale Dividi il set di dati nel Jupyter Notebook per dividere il set di dati (Figura 3A).
- Eseguire il training di un modello di regressione della foresta casuale sul set di addestramento per prevedere l'efficienza di differenziazione dalle funzionalità dell'immagine in campo chiaro 0 h. Eseguire il notebook Jupyter "Machine_learning.ipynb" per eseguire il training e testare il modello.
- Valutare il modello di foresta casuale sottoposto a training nel set di test. Confermare che l'efficienza di differenziazione prevista è altamente correlata con quella vera utilizzando il coefficiente di correlazione di Pearson (Figura 3B).
NOTA: Poiché il set di test non è coinvolto nella fase di addestramento, un'elevata coerenza tra l'efficienza di differenziazione prevista e quella reale sul set di test indica che il modello può generalizzare bene su campioni non visti. - Ottenere l'importanza delle funzionalità dal modello di foresta casuale sottoposto a training e selezionare le 8 funzionalità con l'importanza massima. Visualizza la loro relazione con l'efficienza di differenziazione finale dividendo la gamma di funzioni in contenitori e calcolando l'efficienza di differenziazione media in ciascun contenitore. Eseguire il Jupyter Notebook "Feature_importance.ipynb" per eseguire la visualizzazione (Figura 3C).
NOTA: La visualizzazione può aiutare a capire come i cambiamenti nella forma della colonia di PSC contribuiscano alla differenziazione. - Applicare il modello addestrato in nuovi batch per monitorare gli stati delle colonie di PSC in tempo reale. Acquisisci l'immagine in campo chiaro ogni ora, passa l'immagine al modello addestrato e ottieni l'efficienza di differenziazione prevista. Una volta che l'efficienza prevista supera una soglia (ad esempio, il 50%), avviare il processo di differenziazione. Se l'efficienza prevista non è in grado di raggiungere la soglia, scartare questi campioni.
NOTA: Durante la fase di PSC, il potenziale di differenziazione delle colonie di PSC aumenterà prima e poi diminuirà. Il monitoraggio in tempo reale consente agli sperimentatori di identificare il punto temporale più favorevole per avviare la differenziazione.
- Preparare un set di dati composto da immagini in campo chiaro a 0 h (prima del trattamento CHIR) e le immagini finali di fluorescenza cTnT. Per aumentare la diversità dei profili morfologici delle immagini in campo chiaro a 0 ore, assicurarsi che il set di dati includa colonie di PSC provenienti da diverse linee cellulari, con la differenziazione avviata in un intervallo di tempo dopo il passaggio. Trattare i pozzetti nel set di dati con le condizioni CHIR ottimali allo stadio I.
- Strategia di ML nella fase I: valutazione precoce e aggiustamento delle dosi di CHIR
Set di dati: coppie di flussi di immagini in campo chiaro a 0-12 ore ed etichette di concentrazione CHIR (bassa/ottimale/alta)
Modello: regressione logistica- Preparare un set di dati costituito da flussi di immagini in campo chiaro a pozzetto intero, in cui le immagini vengono acquisite ogni ora tra 0 h e 12 h. Registrare le dosi di CHIR (cioè le combinazioni di concentrazioni e durata di CHIR) di ciascun pozzetto. Assicurarsi che il set di dati includa diversi lotti e pozzetti con dosi di CHIR titolate (ad esempio, concentrazione = 2, 4, 6, 8, 10 e 12 μM; durata = 24, 36 e 48 ore) per aumentare la diversità. Cambia la directory di lavoro su "./stage_I"; salva le immagini come "./data/image/CD01-*/S*/T*.png", dove CD01-* (CD01-1, CD01-2, CD01-3, CD01-4) è il nome del lotto, S* (S1, S2, ..., S96) è l'indice del pozzo e T*.png (T1.png, T2.png, ..., T10.png) è l'immagine in campo chiaro pre-elaborata del pozzo in un diverso passo temporale del flusso di immagini.
- In ogni batch, calcolare la percentuale di cellule cTnT+ per ciascun pozzetto, che viene calcolata dividendo il numero di cellule cTnT+ per il numero totale di cellule Hoechst+ nell'immagine a fluorescenza. Fare la media della percentuale di cellule cTnT+ su pozzetti paralleli con la stessa condizione di dose CHIR. Salvare la condizione di dose CHIR e la percentuale di cellule cTnT+ per ciascun pozzetto in un file csv "./data/image/CD01-*/CD01-?_exp_config.csv".
- In ogni lotto, determinare l'intervallo di concentrazione CHIR "basso", "ottimale" e "alto" in ciascuna durata CHIR in base ai seguenti criteri. Le concentrazioni di CHIR con una percentuale media di cellule cTnT+ al di sopra di una soglia (ad esempio, il 20%) sono identificate come "ottimali". Le concentrazioni di CHIR oltre l'intervallo ottimale sono identificate come "basse" o "alte". Sotto ogni durata CHIR, etichettare i pozzetti come "basso", "ottimale" e "alto" in base alle corrispondenti concentrazioni di CHIR.
NOTA: Empiricamente, esiste una correlazione negativa tra le concentrazioni di CHIR e la durata dei pozzi ad alta efficienza all'interno di un lotto: per una maggiore durata del CHIR, le concentrazioni ottimali diminuiscono. In altre parole, l'ottimalità delle concentrazioni di CHIR dipende dalla durata del CHIR e, quindi, le etichette dei pozzi differiscono tra le diverse durate del CHIR. - In ogni lotto, sotto ogni durata CHIR, calcolare la concentrazione di ΔCHIR per ogni concentrazione c per quantificarne la deviazione dall'ottimale. Denotare l'intervallo di concentrazione CHIR ottimale con [c1, c2], quindi la concentrazione di ΔCHIR (c) = c - c1 (se c < c1), 0 (se c1 ≤ c ≤ c2) o c - c 2 (se c > c2).
- Estrarre le caratteristiche dei flussi di immagini nel set di dati, assicurandosi che riflettano i cambiamenti morfologici delle cellule durante la fase I. Per calcolare le caratteristiche dell'immagine relative all'entropia locale, all'area, alle circonferenze, alla luminosità, al flusso ottico e alla dimensione frattale, eseguire
cd ./dati
Pitone compute_features.py
CD..
NOTA: Per evidenziare i cambiamenti morfologici, i valori di alcune caratteristiche (ad esempio, area, circonferenze e flusso ottico) possono essere normalizzati dai loro valori di base iniziali nei flussi di immagini. - Dividi in modo casuale il set di dati in un set di addestramento e un set di test. Eseguire Jupyter Notebook "./data/prepare_dataset.ipynb" per dividere il set di dati e organizzare i set di dati come frame di dati (Figura 3D).
- Per ogni durata CHIR, eseguire il training di un modello di regressione logistica per stimare l'etichetta di concentrazione CHIR (bassa/ottimale/alta) dalle funzionalità del flusso di immagini nel set di training. Valutare le prestazioni di classificazione del modello di regressione logistica sottoposto a training nel set di test utilizzando accuratezza, precisione, richiamo, punteggio F1 e area sotto la curva (AUC). Eseguire il notebook Jupyter "machine_learning.ipynb" per il training e il test (Figura 3E).
- Valutare le prestazioni del modello nella valutazione della dose CHIR. Nel set di test, unire le etichette previste di pozzi paralleli con la stessa concentrazione CHIR utilizzando i punteggi di deviazione (compresi tra -1 e 1), definiti da
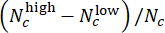 , dove Nc denota il numero di pozzi con concentrazione c e
, dove Nc denota il numero di pozzi con concentrazione c e 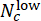 e
e 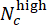 i pozzi tra di loro sono previsti rispettivamente come "basso" e "alto". Confermare che i punteggi di deviazione previsti siano altamente correlati con la vera concentrazione di ΔCHIR per ciascuna dose di CHIR utilizzando il coefficiente di correlazione di Pearson.
i pozzi tra di loro sono previsti rispettivamente come "basso" e "alto". Confermare che i punteggi di deviazione previsti siano altamente correlati con la vera concentrazione di ΔCHIR per ciascuna dose di CHIR utilizzando il coefficiente di correlazione di Pearson. - Eseguire una convalida cross-batch per testare le prestazioni di generalizzazione del modello. Ad ogni round, lascia un lotto per il test mentre usi gli altri per l'allenamento. Confronta i punteggi di deviazione previsti e la vera concentrazione di ΔCHIR per ciascuna dose di CHIR in ogni lotto di test utilizzando il coefficiente di correlazione di Pearson. Eseguire il Jupyter Notebook "cross_batch_validation.ipynb" per implementare la convalida cross-batch (Figura 3F).
NOTA: Per una durata CHIR selezionata h, la previsione per tutti i pozzi con concentrazione dal modello di regressione logistica addestrato sotto la durata h sarà convertita in un punteggio di deviazione, che riflette l'ottimalità della concentrazione CHIR c. Un punteggio di deviazione vicino a 0 suggerisce che si prevede che la concentrazione c sia ottimale sotto la durata h, mentre un punteggio di deviazione negativo o positivo suggerisce una condizione di sottodosaggio o sovradosaggio, che può fungere da segnale per un successivo aggiustamento delle dosi di CHIR. Si noti che, poiché la previsione viene effettuata in anticipo (12 ore), la durata finale del CHIR e la concentrazione del CHIR durante il resto della fase I possono ancora essere regolate. - Applicare i modelli di regressione logistica addestrati per valutare le dosi di CHIR in nuovi batch. Per una data concentrazione di CHIR, calcola le caratteristiche del flusso di immagini 0-12 h per pozzi paralleli con concentrazione, passa le funzionalità ai modelli di regressione logistica addestrati con diverse durate CHIR e, di conseguenza, ottieni le etichette previste per ciascun pozzo. Sulla base della previsione dei modelli, calcola i punteggi di deviazione per valutare ciascuna concentrazione di CHIR con diverse durate di CHIR.
- Con la valutazione della dose di CHIR basata su modelli, i pozzetti di salvataggio al di sotto di ogni concentrazione di CHIR non ottimale si trovano di conseguenza, regolando la durata o la concentrazione del CHIR verso l'ottimale prima di 48 ore.
- Regolazione della durata: in base alla previsione del modello sulla durata ottimale del CHIR, regolare il punto temporale in cui il mezzo contenente CHIR in questi pozzetti viene commutato nel mezzo di differenziazione CM. Se il punto temporale ottimale non viene raggiunto, scartare questi campioni.
- Regolazione della concentrazione: sotto la durata CHIR preimpostata, se la concentrazione attuale è prevista come "bassa", aggiungere direttamente un'alta concentrazione di CHIR nel mezzo per aumentare la concentrazione di CHIR. Diluire CHIR in DMSO (50 mM) con PBS quando necessario, aggiungere un volume appropriato di liquido e agitare delicatamente la piastra. Se la concentrazione attuale è prevista come "alta", aspirare un certo volume di terreno contenente CHR e integrare con lo stesso volume di terreno di differenziazione CM.
NOTA: Nelle prime 48 ore di differenziazione, le cellule secerneranno fattori per migliorare la crescita e la differenziazione cellulare, quindi non cambiano completamente il mezzo. Inoltre, è necessario mantenere costante il volume del mezzo nella differenziazione. Pertanto, assicurarsi che il volume di liquido aggiunto sia il più piccolo possibile quando si regola la concentrazione di CHIR.
- Strategia di ML nella fase II: riconoscimento dei CPC CM-committed
Set di dati: coppie di immagini in campo chiaro il giorno 6 e maschere annotate manualmente di CPC impegnati in CM
Modello: ResNeSt38 con Grad-CAM36 per un apprendimento debolmente supervisionato- Prepara un set di dati composto da immagini in campo chiaro il giorno 6. Annotare manualmente i CPC impegnati in CM nelle immagini in campo chiaro tracciando le cellule cTnT+ nei flussi di immagini dal giorno 12 al giorno 6.
- Ritaglia le immagini in campo chiaro e la corrispondente annotazione manuale (denominate maschere) dei CPC impegnati in CM in patch, con ogni patch dimensionato a 1/5,5 della dimensione dell'immagine a pozzetto intero. Estrarre le patch in modo sovrapposto in cui le patch adiacenti condividono una sovrapposizione del 50% nel set di addestramento e una sovrapposizione del 75% nel set di test. Etichetta le patch con il ≥30% di CPC impegnati in CM come "positive". Etichettare le patch senza CPC impegnati in CM come "negative". Impostare la directory di lavoro su "stage_II". Esegui "./Crop_and_Reconstruct/mask_crop.m" per ritagliare le immagini. Eseguire "./Crop_and_Reconstruct/classification.m" per assegnare etichette (0: negativo; 1: positivo) alle patch in campo chiaro (Figura 3G).
- Dividi in modo casuale le patch di immagine etichettate in un set di training e un set di test.
- Addestra una rete neurale convoluzionale profonda, ResNeSt38 per imparare a classificare queste patch. Valutare il ResNeSt addestrato sul set di test per confermare che il modello dimostra un'elevata precisione di classificazione. Eseguire "train.py" e "test.py" rispettivamente per l'addestramento e il test (Figura 3H).
- Utilizzare Grad-CAM36 per evidenziare le regioni che contribuiscono maggiormente all'inferenza di ResNeSt, rappresentate dalle mappe di calore. Binarizzare le mappe termiche (utilizzando una soglia, ad esempio il 10% × massimo) per ottenere le regioni CPC previste con commit CM, che sono denominate regioni CPC riconosciute con immagini (IR-CPC).
- Confronta le regioni IR-CPC con maschere annotate manualmente sul set di test utilizzando accuratezza, punteggio F1, precisione, richiamo, specificità e intersezione su unione (IoU). Confrontare la proporzione di regioni IR-CPC con gli indici di efficienza di differenziazione (calcolati dalle immagini finali di fluorescenza cTnT) utilizzando il coefficiente di correlazione di Pearson. Eseguire "./Evaluation/index_calculate.m" e "./Evaluation/Pearson_correlation.m" per la valutazione delle prestazioni (Figura 3I).
- Applicare il modello addestrato per riconoscere i CPC per i nuovi batch nella fase II. Ritaglia le immagini in campo chiaro a pozzetto intero in patch, passa le patch a ResNeSt addestrato e ottieni le regioni IR-CPC a livello di patch, che vengono poi unite per ottenere le regioni IR-CPC a pozzetto intero. Calcola la frazione di regioni IR-CPC come indicatore dell'efficienza di differenziazione finale. Purificare gli IR-CPC per ridurre la contaminazione cellulare (descritto di seguito).
NOTA: Poiché il modello di riconoscimento CPC può effettuare una valutazione precoce dell'efficienza di differenziazione finale, può anche fungere da lettura ad alto rendimento per lo screening di piccole molecole che favoriscono il processo di differenziazione, che può ottimizzare ulteriormente il protocollo di differenziazione35.
- Purificazione selettiva per regione di CPC riconosciuti con immagini
NOTA: Gli esperimenti di fotoattivazione si basano su un microscopio a fluorescenza invertita facilitato da un tavolino motorizzato.- Secondo le regioni IR-CPC previste dal modello addestrato, scartare i pozzi che non sono stati in grado di generare alcun IR-CPC. Per purificare queste CPC commesse con CM, utilizzare una sonda fotoattivabile non citotossica, Dual-Activatable Cell Tracker 1 (DACT-1) per marcare selettivamente la non-CPC39.
- Sciogliere il DACT-1 (10 mM) in DMSO e conservarlo a -20 °C. Incubare le cellule del giorno 6 con 1 μM di DACT-1 in terreno di differenziazione CM per 30 minuti a 37 °C.
- Ispeziona le cellule vive al microscopio e seleziona la regione di interesse (ROI) come poligoni in MATLAB. Disegna linee parallele con spaziatura di 20 μm all'interno del poligono e calcola le coordinate dei punti di intersezione.
- Impostare la traccia mobile del tavolino motorizzato in base alle linee di traccia, quindi il tavolino si muoverebbe a 0,12 mm/s in modo che una linea laser da 405 nm (20 μm di diametro) assemblata sul tavolino possa scansionare globalmente il ROI per fotoattivare i non-CPC in modo limitato. Dopo l'irradiazione, rilevare le cellule marcate con DACT-1 utilizzando una linea laser a 561 nm.
- Dissociare le cellule utilizzando tripsina-EDTA allo 0,05% (diluito da PBS) a 37 °C per 5-7 minuti e agitare delicatamente la piastra in un incubatore per 2 minuti. Filtrare le celle attraverso un colino da 40 μm e centrifugare a 500 × g per 3 minuti. Risospendere le cellule in BSA allo 0,5% e mantenere la provetta in ghiaccio fino alla cernita. Preparare anche un campione non irradiato come controllo negativo per la cernita.
- Avviare il sistema di smistamento per citometria a flusso e configurare il software di smistamento. Scegli i laser con lunghezza d'onda di 561 nm e utilizza un filtro passa-banda di 610/20 per l'ordinamento DACT1+. Per ogni campione, utilizzare uno strumento di gating poligonale per circondare la popolazione cellulare in un grafico bivariato di dispersione diretta (FSC) e dispersione laterale (SSC) per selezionare la singola cella. Quindi, eseguire il controllo negativo per confermare la fluorescenza di fondo, eseguire il campione irradiato selettivamente nella regione e disegnare il cancello per la popolazione DACT1+ .
- Raccogliere e risospendere le cellule selezionate in terreno di mantenimento CM con FBS al 10% e Y27632 da 5 μM e seminare le cellule selezionate in una piastra a 96 pozzetti rivestita da Matrigel.
- Il giorno successivo, cambiate il terreno con il mezzo di mantenimento CM e attendete di raccogliere i CM ad alta purezza.
- Strategia di ML nella fase III: riconoscimento dei CM
Set di dati: coppie di immagini in campo chiaro il giorno 12 e le immagini finali a fluorescenza cTnT
Modello: pix2pix37- Preparare un set di dati costituito da immagini in campo chiaro di CM e le corrispondenti immagini a fluorescenza cTnT. Per aumentare la diversità, assicurarsi che il set di dati includa pozzetti con varie efficienze di differenziazione e linee cellulari.
- Dividi in modo casuale il set di dati in un set di addestramento e un set di test. Impostare la directory di lavoro su "stage_III". Salva le immagini in campo chiaro e le corrispondenti immagini a fluorescenza cTnT rispettivamente in "./pix2pix/datasets/(CM|CM_new_cell_lines)/A/(train|test)/*.png" e ".../B/(train|test)/*.png". Eseguire il comando seguente:
cd pix2pix/datasets
python combine_A_and_B.py --fold_A ./CM/A --fold_B ./CM/B --fold_AB ./CM/ --no_multiprocessing
CD.. - Addestra il modello pix2pix37 sul set di allenamento. Eseguire il seguente comando (Figura 3J):
python train.py --dataroot ./datasets/CM --name brightfield2fluorescence --model pix2pix --input_nc 1 --output_nc 1 --load_size 1536 --crop_size 256 --lr 2e-4 --n_epochs 1000 --n_epochs_decay 0 --norm instance --netD n_layers --n_layers_D 1 --batch_size 16 --direction AtoB --save_epoch_freq 100 --dataset_mode allineato --use_resize_conv --seed 1234 - Applicare il modello pix2pix addestrato sul set di test. Eseguire il comando seguente:
python test.py --dataroot ./datasets/CM --name brightfield2fluorescence --model pix2pix --direction AtoB --input_nc 1 --output_nc 1 --load_size 1536 --crop_size 1536 --use_resize_conv --eval --num_test 1000 - Confrontare le immagini di fluorescenza previste dal modello con quelle reali (cioè ottenute sperimentalmente). A livello di pixel, confronta le intensità previste e le intensità reali utilizzando il coefficiente di correlazione di Pearson. Inoltre, a livello di immagine, confrontare gli indici di efficienza di differenziazione previsti con quelli reali utilizzando il coefficiente di correlazione di Pearson. Eseguire "stage_III/valutazione/pixel_correlation.m" e "image_correlation.ipynb" per questi confronti (Figura 3K,L).
- Applicare il modello sottoposto a training per riconoscere i CM nei nuovi batch nella fase III. Passa nuove immagini in campo chiaro di CM al modello addestrato pix2pix e ottieni una previsione in silico delle immagini di fluorescenza e dell'efficienza di differenziazione.
NOTA: Per ridurre la contaminazione cellulare, la selezione delle cellule fotoattivate basata su DACT-1 è applicabile anche per la purificazione dei CM riconosciuti dal modello.
Access restricted. Please log in or start a trial to view this content.
Risultati
Sulla base dell'imaging in campo chiaro e del ML, il processo di differenziazione complessivo può essere monitorato e ottimizzato in modo intelligente. Nella fase di PSC, abbiamo sviluppato un modello ML in grado di prevedere l'efficienza di differenziazione finale in base alle caratteristiche morfologiche delle colonie iniziali di PSC, per determinare il punto temporale più adatto o appropriato per iniziare la differenziazione (Figura 4A, B
Access restricted. Please log in or start a trial to view this content.
Discussione
Qui, abbiamo descritto un protocollo dettagliato per superare uno dei principali problemi nell'attuale applicazione e traduzione della PSC: la variabilità nella differenziazione cellulare. Sfruttando l'imaging in campo chiaro su cellule vive e il ML, abbiamo ottimizzato iterativamente la differenziazione PSC per ottenere un'efficienza costantemente elevata su linee cellulari e lotti. Tuttavia, nel processo di differenziazione di cui sopra, diversi passaggi critici del protocollo hanno u...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Yang Zhao, Jue Zhang, Xiaochun Yang, Yao Wang e Daichao Chen stanno depositando un brevetto per la strategia di differenziazione PSC riportata in questo articolo (202210525166.X).
Riconoscimenti
Ringraziamo Qiushi Sun, Yao Wang, Yu Xia, Jinyu Yang, Chang Lin, Zimu Cen, Dongdong Liang, Rong Wei, Ze Xu, Guangyin Xi, Gang Xue, Can Ye, Li-Peng Wang, Peng Zou, Shi-Qiang Wang, Pablo Rivera-Fuentes, Salome Püntener, Zhixing Chen, Yi Liu e Jue Zhang, per aver gettato le basi di questa strategia. Questo lavoro è stato supportato dal National Key R&D Program of China (2018YFA0800504, 2019YFA0110000) e dallo Space Medical Experiment Project del China Manned Space Program (HYZHXM01020) a Yang Zhao. La Figura 1 è stata creata con BioRender.com.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| 0.25% Trypsin-EDTA | Gibco | 25200056 | Diluted digests were used for CPC and CM digestion |
| 4% Paraformaldehyde in PBS | KeyGEN BioTECH | KGIHC016 | |
| 6-well Cell Culture Plate | NEST | 703001 | |
| 96-well Cell Culture Plate | NEST | 701001 | |
| B27 Supplement | Gibco | 17504044 | |
| B27 Supplement Minus Insulin | Gibco | A1895601 | |
| Bovine serum albumin (BSA) | GPC BIOTECH | AA904-100G | |
| Celldiscoverer 7 | Zeiss | Instruments used to take bright-field images throughout differentiation and final cTnT images | |
| CHIR99021 | Selleck | S1263 | |
| DMEM/F12 | Gibco | 12634010 | |
| Donkey anti-Mouse IgG (H+L) Highly Cross-Adsorbed Secondary Antibody, Alexa Fluor 488 | Thermo | A-21202 | Secondary Antibody |
| FACSAria III | BD Biosciences | Flow cytometry sorter | |
| Fetal Bovine Serum (FBS) | VISTECH | SE100-B | |
| Hoechst 33342 | YEASEN | 40732ES03 | |
| Human Pluripotent Stem Cell Chemical-defined Medium | Cauliscell Inc | 400105 | Basal medium of PSC preparation medium |
| iPS-18 | TaKaRa | Y00300 | |
| iPS-B1 | Cellapy | CA4025106 | |
| iPS-F | Nuwacell | RC01001-B | |
| iPS-M | Nuwacell | RC01001-A | |
| IWR1-1-endo | Selleck | S7086 | IWR1 |
| Jupyter Notebook | N/A | Version 6.4.0 | https://jupyter.org/ |
| MATLAB | MathWorks | Version R2020a | Software for scientific computation and image annotation |
| Matrigel Matrix | Corning | 354230 | Matrigel |
| Mouse monoclonal IgG1 anti-cTnT | Thermo | MA5-12960 | cTnT primary antibody |
| Normal Donkey Serum | Jackson | 017-000-121 | |
| ORCA-Flash 4.0 V3 digital CMOS camera | Hamamatsu | C13440-20CU | The digital camera assembled on Celldiscoverer7 |
| PBS | NEB | 21-040-CVR | |
| Penicillin-Streptomycin | Gibco | 15140-122 | |
| Pluripotency Growth Mater 1 basal medium | Cellapy | CA1007500-1 | Basal medium of PSC culture medium |
| Pluripotency Growth Mater 1 supplement | Cellapy | CA1007500-2 | Supplement of PSC culture medium |
| Prism | Graphpad | Version 8/9 | Statistical software for statistical analysis and plotting |
| Python | N/A | version 3.6 | Python 3 environment for scientific computation, with packages pytorch (1.9.0), numpy, scipy, pandas, visdom, scikit-learn, scikit-image, opencv-python, and matplotlib software for scientific computation and image annotation. |
| RPMI 1640 | Gibco | 11875176 | |
| Supplement hPSC-CDM (500x) | Cauliscell Inc | 00015 | Supplement of PSC preparation medium |
| TiE | Nikon | An inverted fluorescence microscope (with modification) for region-selevtive purification | |
| Triton X-100 | Amresco | 9002-93-1 | |
| Versene Solution | Thermo | 15040066 | EDTA solution for PSC digestion |
| Y27632 | Selleck | S6390 | |
| Zen | Zeiss | Version 3.1 | A supporting software of Celldiscoverer7 for image acquisition, processing and analysis |
Riferimenti
- Yoshida, Y., Yamanaka, S. Induced pluripotent stem cells 10 years later: for cardiac applications. Circ Res. 120 (12), 1958-1968 (2017).
- Shi, Y., Inoue, H., Wu, J. C., Yamanaka, S. Induced pluripotent stem cell technology: a decade of progress. Nat Rev Drug Discov. 16 (2), 115-130 (2017).
- Takahashi, K., et al. Induction of pluripotent stem cells from adult human fibroblasts by defined factors. Cell. 131 (5), 861-872 (2007).
- Burridge, P. W., et al. Chemically defined generation of human cardiomyocytes. Nat Methods. 11 (8), 855-860 (2014).
- Mordwinkin, N. M., Burridge, P. W., Wu, J. C. A review of human pluripotent stem cell-derived cardiomyocytes for high-throughput drug discovery, cardiotoxicity screening, and publication standards. J Cardiovasc Transl Res. 6 (1), 22-30 (2013).
- Vanden Berg, C. W., Elliott, D. A., Braam, S. R., Mummery, C. L., Davis, R. P. Differentiation of human pluripotent stem cells to cardiomyocytes under defined conditions. Methods Mol Biol. 1353, 163-180 (2016).
- Burridge, P. W., Holmström, A., Wu, J. C. Chemically defined culture and cardiomyocyte differentiation of human pluripotent stem cells. Curr Protoc Hum Genet. 87, 1-15 (2015).
- Li, J., et al. Human pluripotent stem cell-derived cardiac tissue-like constructs for repairing the infarcted myocardium. Stem Cell Rep. 9 (5), 1546-1559 (2017).
- Smith, A. S. T., Macadangdang, J., Leung, W., Laflamme, M. A., Kim, D. -H. Human iPSC-derived cardiomyocytes and tissue engineering strategies for disease modeling and drug screening. Biotechnol Adv. 35 (1), 77-94 (2017).
- Sharma, A., et al. Use of human induced pluripotent stem cell-derived cardiomyocytes to assess drug cardiotoxicity. Nat Protoc. 13 (12), 3018-3041 (2018).
- Mehta, A., et al. Identification of a targeted and testable antiarrhythmic therapy for long-QT syndrome type 2 using a patient-specific cellular model. Eur Heart J. 39 (16), 1446-1455 (2018).
- Cahan, P., Daley, G. Q. Origins and implications of pluripotent stem cell variability and heterogeneity. Nat Rev Mol Cell Biol. 14 (6), 357-368 (2013).
- Kim, K., et al. Epigenetic memory in induced pluripotent stem cells. Nature. 467 (7313), 285-290 (2010).
- Rouhani, F., et al. Genetic background drives transcriptional variation in human induced pluripotent stem cells. PLoS Genet. 10 (6), e1004432(2014).
- Kim, K., et al. Donor cell type can influence the epigenome and differentiation potential of human induced pluripotent stem cells. Nat Biotechnol. 29 (12), 1117-1119 (2011).
- Garitaonandia, I., et al. Increased risk of genetic and epigenetic instability in human embryonic stem cells associated with specific culture conditions. PLoS One. 10 (2), e0118307(2015).
- Engler, A. J., Sen, S., Sweeney, H. L., Discher, D. E. Matrix elasticity directs stem cell lineage specification. Cell. 126 (4), 677-689 (2006).
- Matoba, N., Yamashita, T., Takayama, K., Sakurai, F., Mizuguchi, H. Optimal human iPS cell culture method for efficient hepatic differentiation. Differentiation. 104, 13-21 (2018).
- Volpato, V., et al. Reproducibility of molecular phenotypes after long-term differentiation to human iPSC-derived neurons: A multi-site omics study. Stem Cell Rep. 11 (4), 897-911 (2018).
- Anderson, N. C., et al. Balancing serendipity and reproducibility: Pluripotent stem cells as experimental systems for intellectual and developmental disorders. Stem Cell Rep. 16 (6), 1446-1457 (2021).
- Sepac, A., et al. Comparison of cardiomyogenic potential among human ESC and iPSC lines. Cell Transplant. 21 (11), 2523-2530 (2012).
- Laco, F., et al. Unraveling the inconsistencies of cardiac differentiation efficiency induced by the GSK3β inhibitor CHIR99021 in human pluripotent stem cells. Stem Cell Rep. 10 (6), 1851-1866 (2018).
- Volpato, V., Webber, C. Addressing variability in iPSC-derived models of human disease: guidelines to promote reproducibility. Dis Model Mech. 13 (1), 042317(2020).
- Hu, B. -Y., et al. Neural differentiation of human induced pluripotent stem cells follows developmental principles but with variable potency. Proc Natl Acad Sci USA. 107 (9), 4335-4340 (2010).
- Huo, J., et al. Evaluation of batch variations in induced pluripotent stem cell-derived human cardiomyocytes from 2 major suppliers. Toxicol Sci. 156 (1), 25-38 (2017).
- Rostam, H. M., Reynolds, P. M., Alexander, M. R., Gadegaard, N., Ghaemmaghami, A. M. Image based machine learning for identification of macrophage subsets. Sci Rep. 7 (1), 3521(2017).
- Christiansen, E. M., et al. In silico labeling: Predicting fluorescent labels in unlabeled images. Cell. 173 (3), 792-803 (2018).
- Buggenthin, F., et al. Prospective identification of hematopoietic lineage choice by deep learning. Nat Methods. 14 (4), 403-406 (2017).
- Smith, Z. D., Nachman, I., Regev, A., Meissner, A. Dynamic single-cell imaging of direct reprogramming reveals an early specifying event. Nat Biotechnol. 28 (5), 521-526 (2010).
- Fan, K., Zhang, S., Zhang, Y., Lu, J., Holcombe, M., Zhang, X. A machine learning assisted, label-free, non-invasive approach for somatic reprogramming in induced pluripotent stem cell colony formation detection and prediction. Sci Rep. 7 (1), 13496(2017).
- Kusumoto, D., et al. Automated deep learning-based system to identify endothelial cells derived from induced pluripotent stem cells. Stem Cell Rep. 10 (6), 1687-1695 (2018).
- Zhu, Y., et al. Deep learning-based predictive identification of neural stem cell differentiation. Nat Commun. 12 (1), 2614(2021).
- Qian, T., et al. Label-free imaging for quality control of cardiomyocyte differentiation. Nat Commun. 12 (1), 4580(2021).
- Ounkomol, C., Seshamani, S., Maleckar, M. M., Collman, F., Johnson, G. R. Label-free prediction of three-dimensional fluorescence images from transmitted-light microscopy. Nat Methods. 15 (11), 917-920 (2018).
- Yang, X., et al. A live-cell image-based machine learning strategy for reducing variability in PSC differentiation systems. Cell Discovery. 9 (1), 53(2023).
- Selvaraju, R. R., et al. Grad-CAM: Visual explanations from deep networks via gradient-based localization. Proc IEEE Int Conf Computer Vision (ICCV). , 618-626 (2017).
- Isola, P., Zhu, J. -Y., Zhou, T., Efros, A. A. Image-to-image translation with conditional adversarial networks. Proc IEEE Conf Computer Vision Pattern Recognition (CVPR). , 5967-5976 (2017).
- Zhang, H., et al. ResNeSt: Split-attention networks. Proc IEEE/CVF Conf Computer Vision Pattern Recognition Workshops (CVPRW). , 2735-2745 (2022).
- Halabi, E. A., et al. Dual-activatable cell tracker for controlled and prolonged single-cell labeling). ACS Chem. Biol. 15 (6), 1613-1620 (2020).
- Hannan, N. R. F., Segeritz, C. -P., Touboul, T., Vallier, L. Production of hepatocyte-like cells from human pluripotent stem cells. Nat Protoc. 8 (2), 430-437 (2013).
- Zhang, H., Shen, M., Wu, J. C. Generation of quiescent cardiac fibroblasts derived from human induced pluripotent stem cells. Methods Mol Biol. 2454, 109-115 (2022).
- Harding, M. J., McGraw, H. F., Nechiporuk, A. The roles and regulation of multicellular rosette structures during morphogenesis. Development. 141 (13), 2549-2558 (2014).
- Najm, F. J., et al. Rapid and robust generation of functional oligodendrocyte progenitor cells from epiblast stem cells. Nat Methods. 8 (11), 957-962 (2011).
- Hogrebe, N. J., Maxwell, K. G., Augsornworawat, P., Millman, J. R. Generation of insulin-producing pancreatic β cells from multiple human stem cell lines. Nat Protoc. 16 (9), 4109-4143 (2021).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon