このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
多能性幹細胞の分化を監視するための生細胞画像ベース機械学習戦略
* これらの著者は同等に貢献しました
要約
利用可能な多能性幹細胞(PSC)から機能細胞への分化システムは、現在、深刻なライン間およびバッチ間のばらつきの問題によって妨げられています。ここでは、心臓の分化を主な例として、画像ベースの機械学習に基づいてPSCの分化プロセスをインテリジェントに監視および調整するためのプロトコルを紹介します。
要約
多能性幹細胞(PSC)技術は、創薬、疾患モデリング、再生医療などで広く利用されています。しかし、利用可能なPSCから機能細胞への分化システムは、ライン間およびバッチ間の深刻な変動の問題によって妨げられています。そのため、細胞分化をリアルタイムで正確に制御することが重要です。このプロトコルでは、明視野画像ベースの機械学習を使用して細胞分化の変動を克服する非侵襲的でインテリジェントな戦略について説明します。PSCから心筋細胞への分化を例にとると、PSCの初期状態の制御、分化条件での早期評価と介入、分化ミスのある細胞汚染の排除など、詳細な情報を提供するとともに、PSCから機能細胞への高品質な分化を一貫して実現します。原則として、この戦略は、細胞製造をサポートするだけでなく、細胞運命変換中のメカニズムの理解を深めるために、複数のステップを持つ他の細胞分化またはリプログラミングシステムに拡張できます。
概要
多能性幹細胞(PSC)は、in vitroで多くの種類の細胞に分化する優れた能力を持っています。これらの分化した機能細胞は、細胞治療、疾患モデリング、および医薬品開発に利用できる可能性があり、これらはすべて研究や臨床応用に有用です1,2,3。例えば、PSCを心筋細胞(CM)4,5,6,7に区別するための様々な方法が開発されてきた。これらのCMは、薬物の心毒性試験、心臓病のモデリング、および細胞移植8,9,10,11に適用できます。
しかし、PSCから末端分化細胞への変換は段階的なプロセスであり、分化過程での複数の摂動により、細胞は分岐した細胞運命につながる可能性があります。PSC系統のさまざまな遺伝的背景とエピジェネティックなマークは、特定の系統への分化の可能性に影響を与えます12,13,14,15;PSC継代の数と蓄積された遺伝子変異も、PSCの不均一性の原因です。異なる実験者が用いる実験操作の違いもまた、全く異なる分化結果をもたらすことがある16,17,18,19,20。したがって、現在、PSC由来の細胞産生における主要な問題の1つは、細胞株およびバッチ21,22,23,24,25の間の不安定性である。PSCの分化が不安定な場合、実験が何度も繰り返されることが多く、多大な時間と労力を浪費します。この問題に対処するには、細胞株やバッチ間のばらつきを最小限に抑え、分化の安定性と頑健性を高める戦略を開発することが重要です。
近年、高解像度顕微鏡法と機械学習(ML)の進歩により、MLベースの定量的画像解析の細胞生物学への応用が容易になり、細胞イメージングの特徴26,27,28,29,30,31,32,33,34に貴重な情報を活用することが可能になった.以前の研究では、PSC分化の安定性と効率を向上させるために、PSCの分化状態をリアルタイムで監視および介入する生細胞画像ベースのML戦略を提案しました(図1)35。PSCから心筋細胞への分化を例にとると、ランダムフォレストモデルを用いてPSCの初期状態を評価し、ロジスティック回帰モデルを用いて最適な分化条件を予測し、深層学習に基づくGrad-CAM36とpix2pix37を用いて成功した分化細胞を認識しました。MLモデルは、面積、円周、凸性、堅牢性、輝度、移動速度、および深層畳み込みニューラルネットワークによって抽出されたその他の暗黙的な特徴を含む、さまざまな明視野形態学的特徴から細胞系統を特定することを学習しました。これらの確立されたMLモデルからの推論に基づき、PSCの初期状態の制御、分化条件の早期評価と介入、誤分化細胞汚染の排除を実現し、心臓の分化プロセスを包括的かつ正確に調節することを実現しました。ここでは、戦略を策定するためのステップバイステップのプロトコルを提供します。
Access restricted. Please log in or start a trial to view this content.
プロトコル
1. 細胞の分化と特性評価
- 培養試薬および培養プレートの調製
- 48 mLの基礎培地に2 mLのサプリメントと0.2%ペニシリン-ストレプトマイシンを加えて、PSC培地を調製します。サプリメントを分注し、-20°Cで保存します。 この培地は4°Cで最大4週間保存してください。
- 500 mLの基礎培地に1 mLのサプリメントと0.2%ペニシリン-ストレプトマイシンを加えて、PSC調製培地を調製します。使用する場合は、培地を1回だけ使用するために予熱し、培地を4°Cで最大3週間保存してください。
- 1x B27サプリメントからインスリンを差し引いたものと1%ペニシリン-ストレプトマイシンをRPMI 1640に追加して、CM分化培地を調製します。1x B27サプリメントと1%ペニシリン-ストレプトマイシンをRPMI 1640に追加して、CM維持培地を準備します。これら2種類の培地は、4°Cで2週間保存してください。
- マトリゲルを氷に沈め、4°Cで一晩保管します。マトリゲルが完全に解凍されていることを確認してください。次に、後で使用するために-80°Cで保存した1 mLのアリコートに分注し、4°Cで49 mLのDMEM/F12に1 mLのマトリゲルを加えて、マトリゲルの作業溶液を調製します。このマトリゲル作業溶液を6ウェル培養プレートのウェルあたり850 μL加え、プレートを37°Cで少なくとも30分間インキュベートします。CM分化に使用する96ウェル培養プレートの場合、ウェルあたり35 μLのマトリゲル作業溶液を添加します。
注:マトリゲルは常に氷の上に置いてください。ピペットの先端とチューブは4°Cで予冷する必要があります。 マトリゲル作業溶液は、4°Cで2週間保存できます。Matregelの作業溶液をピペッティングする際には、気泡を避けるように注意してください。
- PSCのメンテナンスと通過
- PSCを6ウェル培養プレートに保持します。顕微鏡で細胞密度を確認し、コンフルエント度が~70%に達したら継代の準備をします(図2A)。
- EDTAを使用して細胞を消化し、継代します。PSC培地、PBS、およびEDTAを37°Cのウォーターバスで温め、培地にY27632を添加して最終濃度5 μMにします。
- PSCを1 mLのPBSで洗浄し、1ウェルあたり1 mLのEDTAを添加し、プレートを37°Cのインキュベーターで3分間インキュベートします。
- 細胞が培養プレートから剥離する前にEDTAを吸引し、1mLのPSC培地を使用してウェルの底を3〜4倍穏やかにすすぎ、細胞を再懸濁します。
注:これにより、遠心分離のステップをスキップし、継代操作における細胞損傷を減らすことができます。 - 6ウェル培養プレートのマトリゲルコーティングウェルからDMEM/F12を吸引し、ウェルあたり2 mLの新鮮なPSC培地を添加します(Y27632を添加)。細胞懸濁液を1:6から1:12の分割比でウェルに加えます。プレートを5% CO2を入れた37°Cインキュベーターに入れます。
- PSC培地(Y27632なし)を継代後12〜24時間で交換します。その後、培地を交換し、細胞の状態とコンフルエンスを毎日チェックします。
- PSCの心臓分化
- CM分化のためには、PSC調製培地(Y27632を添加)中の96ウェル培養プレートにPSCを播種します。CMの分化には、上記と同じ手順(手順1.2.2-1.2.6)に従って、PSC調製培地(Y27632を追加)の96ウェル培養プレートにPSCを播種します。
注:この段階では、EDTA治療期間を適切に延長(5分以上)してPSCを十分に解離させ、継代後にクローンを小さくすることができます。私たちの研究は、同じエリア内で、より小さなコロニーが分化を助長することを示しています。 - ステージI:PSCが80〜90%のコンフルエントに達したら、培地を2〜20μMのCHIR99021(CHIR)を含むCM分化培地に切り替えます(図2B)。中程度のスイッチングの時点は、デイ 0 と呼ばれます。24-48時間のCHIR処理後、培地を新鮮なCM分化培地に変更します。
注:CHIRは、中胚葉誘導用のGSK3β阻害剤です。CM分化に最適なCHIR用量は、細胞株やバッチによって異なります。例えば、最適なCHIR用量は、iPS-B1の場合、8-10μM、48時間です。MLモデルのトレーニングのために多様な明視野画像を収集するために、ここでは、さまざまな分化効率について、実験の各バッチのCHIR濃度を滴定しました。 - ステージII:72時間(3日目)に、培地を5μMのIWR1を添加したCM分化培地と交換し、48時間(5日目)培養します。CM分化培地で培地を1〜2日間(6〜7日目)交換します。この時点で、PSCは心前駆細胞(CPC)に分化します。
- ステージIII:メディアをCMメンテナンスメディアと交換し、3日ごとにメディアを交換します。10日目または12日目に、CMを収穫してさらに分析します。
- CM分化のためには、PSC調製培地(Y27632を添加)中の96ウェル培養プレートにPSCを播種します。CMの分化には、上記と同じ手順(手順1.2.2-1.2.6)に従って、PSC調製培地(Y27632を追加)の96ウェル培養プレートにPSCを播種します。
- CMの免疫蛍光染色
- 10日目または12日目に、細胞をPBSで洗浄し、室温で15分間PBS中の4%パラホルムアルデヒドで固定します。CMをPBSで2回洗浄し、プレートを4°Cで最大~1週間保存します。
注:パラホルムアルデヒドは、刺激臭のある有毒な試薬です。ケミカルフードで操作し、目と手を保護します。 - 染色時には、細胞を透過化溶液(PBS中の0.1% Triton X-100)で室温で15分間処理します。次に、ブロッキング溶液(PBS中に3%Donkey Serumを含む0.1% Triton X-100)で細胞を室温で30分間インキュベートします。
- サンプルを心筋トロポニンT(cTnT)一次抗体(1:300)とインキュベートし、ブロッキング溶液で4°Cで一晩希釈し、CMを同定します。
- 一次抗体を採取し、PBS 3xで細胞を洗浄します。暗所で、サンプルをPBS中の二次抗体と1%ウシ血清アルブミン、37°Cで1時間インキュベートします。
注:採取した一次抗体は、1週間で2〜3回再利用できます。
- 一次抗体を採取し、PBS 3xで細胞を洗浄します。暗所で、サンプルをPBS中の二次抗体と1%ウシ血清アルブミン、37°Cで1時間インキュベートします。
- 細胞から二次抗体を取り出し、PBSで細胞を3回洗浄します。Hoechst 33342(PBSで1:1,000)を使用して、室温で5分間核を染色します。PBSを使用してセルを3回すすぎ、乾燥を避けるためにウェルあたり100μLのPBSを加えます。プレートを4°Cの暗所で保存し、1週間以内に蛍光画像を取得してください。
- 10日目または12日目に、細胞をPBSで洗浄し、室温で15分間PBS中の4%パラホルムアルデヒドで固定します。CMをPBSで2回洗浄し、プレートを4°Cで最大~1週間保存します。
2. 分化プロセス全体にわたる画像ストリーム取得
- 画像収集のための実験計画法の設定
- 生細胞の培養とイメージングをサポートする自動顕微鏡を使用して、CM分化のさまざまな段階の明視野画像と分化結果のcTnT免疫蛍光画像を収集します(図2A-E)。顕微鏡支援ソフトウェアを使用して、実験プログラムを設計し、顕微鏡を制御します。
- ソフトウェアを開き、新しい実験計画を作成します。イメージングには、5倍対物レンズと2倍チューブレンズを選択してください。[タイル] と [Z-スタック] のオプションを確認します。
- Channels(チャンネル)メニューに明視野イメージング用のTL Brightfieldチャンネルを追加します。免疫蛍光イメージング用のAF488およびH3342チャンネルを追加します。[イメージング設定]メニューで光路を変更し、外部デジタルCMOSカメラでイメージングします。
- [Acquisition Mode] メニューを開き、2 x 2 ビニングを設定して S/N 比を上げます。
- Zスタックメニューを開き、センターモデルを選択します。スキャン時のスライス数と間隔を設定します。分析でピントの合った画像を得るために、3〜6μm間隔で3〜5スライスを選択します。
- 「Navigation & Tiles」ウィンドウで、Carrier ごとにタイル領域を設定し、1 つのウェルに対して 25 個のタイル (5 列 x 5 行) を設定します。[Tiles]メニューで、[Sample Carrier]の[Multiwell 96]テンプレートを選択し、隣接する領域に5%〜15% のオーバーラップを設定します。
- 連続イメージングが必要な場合は、 Time seriesオプションをチェックし、Time seriesメニューで期間と間隔を設定します。ステージ I でイメージ・ストリームを収集するには、インターバルなしで 12 時間のデュレーションを設定します。
- 明視野および免疫蛍光画像の取得
- 画像を取得するためには、まず細胞培養プレートをサンプルトレイに入れ、サンプルを顕微鏡内にロードします。実験デザインに一致するMultiwell 96テンプレートを選択します。サンプルが生細胞で構成されている場合は、加熱システムとCO2 ポンプを開いて、培養に適した条件(37°C、5%CO2)を維持します。
- プリセットの実験プロジェクトを開き、新しいプロジェクトとして保存します。[タイル] メニューを開き、プレートの位置を手動で調整します。[ナビゲーションとタイル] ウィンドウで、必要な井戸を選択し、[作成] をクリックして、これらの井戸のタイル領域を構築します。
- [Tiles] メニューの [Verify Tile Regions] をクリックし、オートフォーカスを実行してすべてのウェルを確認します。次に、マウスホイールをスクロールして、明視野の下の各ウェルの焦点を手動で修正します。
- 必要なチャンネルにチェックを入れ、チャンネルメニューで露出時間を設定します。推奨される露光時間は、ブライトで 1.625 ミリ秒、H3342 で 100 ミリ秒、AF488 で 550 ミリ秒です。1回のスナップで全チャンネルの画像を確認します。
- 最後に、[実験の開始] ボタンをクリックし、自動イメージングを待ちます。通常、96ウェル培養プレート全体(96ウェル、3層、明視野下の各ウェルに25枚のタイル)のスキャンを完了するには、~1.2時間かかります。プロセッシングフレームワークで、「イメージのエクスポート」を選択し、非圧縮のTIFF形式またはPNG形式のファイルタイプを選択して適用します。ウェルの中央にある9つのタイル(3 x 3)の画像を使用して、さらに分析します(図2A-E)。
3. 差別化プロセスの各ステージにおける画像ベースML戦略の確立
- ソフトウェアの準備
- パッケージ pytorch (1.9.0)、numpy、scipy、scikit-learn、pandas、visdom、scikit-image、opencv-python、matplotlib を含む Python 環境をインストールします。
- Jupyter Notebook をインストールします。
- MATLAB をインストールします。
- https://github.com/zhaoyanglab/ML-for-PSC-differentiation からコードとサンプル データセットをダウンロードします。
注: ML のパフォーマンスは、データセットの品質と、モデルとハイパーパラメーターの選択に依存します。実装例は、すべての状況に最適であることを保証するものではありません。ML エンジニアは、コードをカスタマイズし、モデルとハイパーパラメーターを特定のニーズに合わせて調整する必要があります。
- PSC 段階の ML 戦略: PSC コロニーの初期状態の制御
データセット:0時間(CHIR処理前)の明視野画像のペアと最終分化効率
モデル:ランダムフォレスト- 0時間(CHIR処理前)の明視野画像と最終的なcTnT蛍光画像からなるデータセットを準備します。0時間明視野画像の形態学的プロファイルの多様性を高めるには、データセットに異なる細胞株のPSCコロニーが含まれていることを確認し、継代後の時間間隔で分化を開始します。データセット内の井戸をステージIで最適なCHIR条件で処理します。
注:コロニーが最適なCHIR条件で処理されるように、同じバッチ内のウェルを滴定CHIR用量で処理できます。データセットには、高効率の CHIR 条件を持つ井戸のみが含まれます。 - 各ウェルの分化効率は、
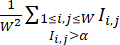 (
(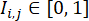 は W 蛍光画像× W 蛍光画像のピクセル位置 (i, j) での蛍光強度を表します) で定義される cTnT 蛍光画像から計算された分化効率指数によって定量化し、その合計はしきい値を超える強度を持つすべてのピクセル位置で取得されますαで、通常は 0.5 に設定されます)。各細胞株内で、この細胞株のすべてのウェルの最大分化効率指数を計算し、それらの分化効率指数を最大値で正規化します。
は W 蛍光画像× W 蛍光画像のピクセル位置 (i, j) での蛍光強度を表します) で定義される cTnT 蛍光画像から計算された分化効率指数によって定量化し、その合計はしきい値を超える強度を持つすべてのピクセル位置で取得されますαで、通常は 0.5 に設定されます)。各細胞株内で、この細胞株のすべてのウェルの最大分化効率指数を計算し、それらの分化効率指数を最大値で正規化します。
注:各細胞株の分化電位は本質的に異なるため、このような標準化は、分化結果に寄与するさまざまな細胞株間で共有されるコロニーの形態学的特徴により強調されます。標準化により、異なる細胞株由来のPSCコロニーが同等になり、細胞株によって誘発されるバイアスが軽減されます。 - 0 時間明視野画像の形態学的プロファイルを、コロニー形状の特性を明らかにする高次元の特徴によって定量化します。明視野画像でコロニー領域の輪郭を描くには、局所エントロピーを計算します(半径 rを使用)。閾値より大きい局所エントロピーを持つピクセルは 、σ Covered by Cells によって識別されます。 r = 10 ピクセル、 σ = 3 のパラメータ設定を使用します。作業ディレクトリを "./colony_control" に切り替えます。明視野画像、細胞領域のバイナリ画像、およびcTnT蛍光画像を「colony_control/ image_data/CD00-*/[明視野|cell_region|ctnt]/S*.png」に保存します。また、Jupyter Notebook「Compute_features.ipynb」を実行して、コロニーエリア、円周、ソリッドリティ、凸面、真円度、重心等高線距離などに関連するフィーチャを計算します。
- データセットをトレーニング セットとテスト セットにランダムに分割し、トレーニング セットはデータ全体の 50% から 80% を占めます。Jupyter Notebook で最後のブロック [データセットの分割] を実行して、データセットを分割します (図 3A)。
- 学習セットでランダム フォレスト回帰モデルに学習させ、0 時間の明視野画像の特徴から微分効率を予測します。Jupyter Notebook "Machine_learning.ipynb" を実行して、モデルをトレーニングおよびテストします。
- テスト セットで学習済みのランダム フォレスト モデルを評価します。ピアソン相関係数を使用して、予測された分化効率が真の効率と高い相関性があることを確認します(図3B)。
注: テスト セットはトレーニング フェーズに関与していないため、テスト セットの予測された微分効率と実際の微分効率の間の一貫性が高い場合は、モデルが未知のサンプルで適切に一般化できることを示します。 - 学習済みのランダム フォレスト モデルから特徴の重要度を取得し、最も重要な 8 つの特徴を選択します。フィーチャの範囲をビンに分割し、各ビンの平均微分効率を計算することで、最終的な微分効率との関係を視覚化します。Jupyter Notebook "Feature_importance.ipynb" を実行して、視覚化を実行します (図 3C)。
注:この視覚化は、PSCコロニー形状の変化が分化にどのように寄与するかを理解するのに役立つ場合があります。 - 学習済みのモデルを新しいバッチで適用して、PSC コロニーの状態をリアルタイムで監視します。明視野画像を毎時キャプチャし、その画像を学習済みモデルに渡し、予測された微分効率を取得します。予測された効率がしきい値(50%など)を超えたら、差別化プロセスを開始します。予測された効率がしきい値に達しなかった場合は、これらのサンプルを破棄します。
注:PSCの段階では、PSCコロニーの分化ポテンシャルは最初に上昇し、後に減少します。リアルタイムモニタリングにより、実験者は分化を開始するのに最も適した時点を特定できます。
- 0時間(CHIR処理前)の明視野画像と最終的なcTnT蛍光画像からなるデータセットを準備します。0時間明視野画像の形態学的プロファイルの多様性を高めるには、データセットに異なる細胞株のPSCコロニーが含まれていることを確認し、継代後の時間間隔で分化を開始します。データセット内の井戸をステージIで最適なCHIR条件で処理します。
- ステージIでのML戦略:CHIR用量の早期評価と調整
データセット:0〜12時間の明視野画像ストリームとCHIR濃度ラベル(低/最適/高)のペア
モデル: ロジスティック回帰- 0 時間から 12 時間の間に 1 時間ごとに画像が取得される、全ウェル明視野画像ストリームで構成されるデータセットを準備します。各ウェルのCHIR用量(すなわち、CHIR濃度と期間の組み合わせ)を記録します。多様性を高めるために、データセットに滴定されたCHIR用量の異なるバッチとウェルが含まれていることを確認してください(例:濃度= 2、4、6、8、10、12 μM、持続時間= 24、36、48時間)。作業ディレクトリを "./stage_I" に切り替えます。.png CD01-* (CD01-1, CD01-2, CD01-3, CD01-3, CD01-4) はバッチ名、S* (S1, S2, ..., S96) はウェルのインデックス、T*.png (T1.png, T2.png, ..., T10.png) はイメージ ストリームの異なるタイム ステップにおけるウェルの前処理された明視野イメージです。
- 各バッチで、各ウェルのcTnT+ 細胞の割合を計算します。これは、cTnT+ 細胞の数を蛍光画像中のHoechst+ 細胞の総数で割ることによって計算されます。同じCHIR線量条件の平行ウェル上のcTnT+ 細胞の割合を平均化します。各ウェルのCHIR用量条件とcTnT+ 細胞の割合をcsvファイル「./data/image/CD01-*/CD01-?_exp_config.csv」に保存します。
- 各バッチで、次の基準に従って、各 CHIR 期間の「低」、「最適」、および「高」の CHIR 濃度範囲を決定します。cTnT+細胞の平均割合が閾値(例:20%)を超えるCHIR濃度は、「最適」と識別されます。最適な範囲を超えるCHIR濃度は、「低」または「高」として識別されます。各CHIR期間で、対応するCHIR濃度に従って、ウェルに「低」、「最適」、および「高」のラベルを付けます。
注:経験的には、CHIR濃度とバッチ内の高効率ウェルの持続時間との間には負の相関関係があり、CHIRの持続時間が長いほど、最適な濃度は減少します。言い換えれば、CHIR濃度の最適性はCHIRの持続時間に依存するため、ウェルのラベルはCHIRの持続時間によって異なります。 - 各バッチで、各 CHIR 期間で、各濃度 c の ΔCHIR 濃度を計算して、最適値からの偏差を定量化します。最適な CHIR 濃度範囲を [c1, c2] で表し、次に ΔCHIR 濃度 (c) = c - c1 ( c < c1 の場合)、0 ( c1 ≤ c ≤ c2 の場合)、または c - c2 ( c > c2 の場合) で表します。
- データセット内の画像ストリームの特徴を抽出し、ステージIの細胞の形態学的変化を反映していることを確認します。ローカル エントロピー、面積、円周、明るさ、オプティカル フロー、フラクタル次元に関する画像の特徴を計算するには、次のコマンドを実行します。
cd ./データ
Pythonのcompute_features.py
cd ..
注:形態学的変化を強調するために、一部の特徴(例えば、面積、円周、およびオプティカルフロー)の値は、画像ストリーム内のそれらの初期ベースライン値によって正規化することができる。 - データセットをトレーニング セットとテスト セットにランダムに分割します。Jupyter Notebook "./data/prepare_dataset.ipynb" を実行して、データセットを分割し、データセットをデータフレームとして整理します (図 3D)。
- CHIR 期間ごとに、ロジスティック回帰モデルをトレーニングして、トレーニング セットの画像ストリームの特徴から CHIR 濃度ラベル (低/最適/高) を予測します。テストセットで学習済みのロジスティック回帰モデルの分類パフォーマンスを、精度、精度、再現率、F1スコア、および曲線下面積(AUC)を使用して評価します。Jupyter Notebook 「machine_learning.ipynb」をトレーニングとテストのために実行します (図 3E)。
- CHIR線量評価でモデル性能を評価します。テストセットでは、
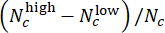 偏差スコア(-1 から 1 の範囲)を使用して、同じ CHIR 濃度の平行ウェルの予測ラベルをマージします。ここで、 Nc は濃度 c と
偏差スコア(-1 から 1 の範囲)を使用して、同じ CHIR 濃度の平行ウェルの予測ラベルをマージします。ここで、 Nc は濃度 c と 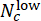 のウェルの数を示し、そのうちの
のウェルの数を示し、そのうちの 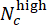 ウェルはそれぞれ「低」と「高」と予測されます。ピアソン相関係数を使用して、予測された偏差スコアが各CHIR用量の真のΔCHIR濃度と高い相関があることを確認します。
ウェルはそれぞれ「低」と「高」と予測されます。ピアソン相関係数を使用して、予測された偏差スコアが各CHIR用量の真のΔCHIR濃度と高い相関があることを確認します。 - クロスバッチ検証を実行して、モデルの汎化パフォーマンスをテストします。各ラウンドで、1つのバッチをテスト用に残し、他のバッチをトレーニングに使用します。ピアソン相関係数を使用して、各テストバッチの各CHIR用量の予測偏差スコアと真のΔCHIR濃度を比較します。Jupyter Notebook 「cross_batch_validation.ipynb」を実行して、クロスバッチ検証を実装します (図 3F)。
注:選択したCHIR期間 hの場合、期間 h でトレーニングされたロジスティック回帰モデルからの濃度を持つすべてのウェルの予測は、CHIR濃度 cの最適性を反映して偏差スコアに変換されます。偏差スコアが0に近い場合は、濃度 c が期間 hで最適であると予測されることを示し、負または正の偏差スコアは、低用量または過剰摂取の状態を示唆しており、これは後でCHIR用量を調整するためのシグナルとして機能する可能性があります。予測は早期(12時間)に行われるため、ステージIの残りの期間の最終的なCHIR期間とCHIR濃度はまだ調整できることに注意してください。 - 学習済みのロジスティック回帰モデルを適用して、新しいバッチの CHIR 線量を評価します。特定の CHIR 濃度について、濃度のある平行ウェルの 0-12 h 画像ストリーム特徴を計算し、異なる CHIR 持続時間で訓練されたロジスティック回帰モデルに特徴を渡し、それに応じて各ウェルの予測ラベルを取得します。モデルの予測に基づいて、偏差スコアを計算して、異なる CHIR 期間で各 CHIR 濃度を評価します。
- モデルベースのCHIR線量評価では、48時間前にCHIRの持続時間または濃度を最適に調整することにより、それに応じて最適でない各CHIR濃度でウェルをレスキューします。
- 期間の調整:最適なCHIR期間に関するモデル予測に従って、これらのウェル内のCHIR含有培地がCM分化培地に切り替わる時点を調整します。最適な時点を逃した場合は、これらのサンプルを廃棄します。
- 濃度の調整:プリセットのCHIR期間で、現在の濃度が「低」と予測される場合は、高濃度のCHIRを培地に直接追加してCHIR濃度を上げます。必要に応じて、CHIRをDMSO(50 mM)でPBSで希釈し、適量の液体を加えて、プレートを静かに振とうします。現在の濃度が「高い」と予測される場合は、一定量のCHIR含有培地を吸引し、同量のCM分化培地を補充します。
注:分化の最初の48時間で、細胞は細胞の成長と分化を改善するための因子を分泌しますので、培地を完全に交換しないでください。また、差別化のために中量を一定に保つ必要があります。したがって、CHIR濃度を調整するときは、添加する液体の量ができるだけ少ないことを確認してください。
- ステージ II の ML 戦略: CM コミット CPC の認識
データセット:6日目の明視野画像のペアと、CMコミットCPCの手動注釈付きマスク
モデル:ResNeSt38 とGrad-CAM36 (弱教師あり学習用)- 6日目に明視野画像からなるデータセットを準備します。12日目から6日目までの画像ストリーム内のcTnT+ 細胞を追跡することにより、明視野画像内のCMコミットCPCに手動で注釈を付けます。
- 明視野画像と、CM コミット CPC の対応する手動アノテーション (マスクと呼ばれる) をパッチにトリミングし、各パッチのサイズをウェル全体の画像サイズの 1/5.5 にします。隣接するパッチがトレーニング セットで 50% のオーバーラップを共有し、テスト セットで 75% のオーバーラップを共有するように、オーバーラップ方式でパッチを抽出します。CMコミットCPCが≥30%のパッチを「陽性」とラベル付けします。CM コミットされた CPC のないパッチには「否定性」のラベルを付けます。作業ディレクトリを "stage_II" に切り替えます。「./Crop_and_Reconstruct/mask_crop.m」を実行して画像をトリミングします。"./Crop_and_Reconstruct/classification.m" を実行して、明視野パッチにラベル (0: ネガティブ、1: ポジティブ) を割り当てます (図 3G)。
- ラベル付けされた画像パッチを学習セットとテストセットにランダムに分割します。
- 深層畳み込みニューラル ネットワーク ResNeSt38 に学習させて、これらのパッチの分類を学習します。テストセットで学習済みのResNeStを評価して、モデルが高い分類精度を示すことを確認します。トレーニングとテストのためにそれぞれ「train.py」と「test.py」を実行します(図3H)。
- Grad-CAM36 を使用して、ヒートマップで表される ResNeSt の推論に最も寄与する領域を強調表示します。ヒートマップを二値化して (しきい値 (最大 10×% など) を使用)、予測された CM コミット CPC 領域 (画像認識 CPC (IR-CPC) 領域) を取得します。
- 精度、F1 スコア、精度、再現率、特異性、および結合交差 (IoU) を使用して、IR-CPC 領域をテスト セット上の手動で注釈付けされたマスクと比較します。ピアソン相関係数を使用して、IR-CPC 領域の割合を Differentiation Efficiency Indexes (最終的な cTnT 蛍光画像から計算) と比較します。パフォーマンス評価のために、"./Evaluation/index_calculate.m" と "./Evaluation/Pearson_correlation.m" を実行します (図 3I)。
- 学習済みモデルを適用して、ステージ II の新しいバッチの CPC を認識します。全ウェル明視野画像をパッチにクロップし、そのパッチを学習済みのResNeStに渡し、パッチレベルでIR-CPC領域を取得し、それらをマージして全ウェルIR-CPC領域を生成します。最終的な分化効率の指標として、IR-CPC 領域の割合を計算します。IR-CPCを精製して細胞汚染を減らします(後述)。
注:CPC認識モデルは、最終的な分化効率を早期に評価することができるので、分化プロセスを助長する小分子をスクリーニングするためのハイスループットな読み出しとしても機能し、これにより分化プロトコル35をさらに最適化することができる。
- 画像認識されたCPCの領域選択的精製
注:光活性化実験は、電動ステージを備えた倒立型蛍光顕微鏡に基づいています。- 学習済みモデルによって予測された IR-CPC 領域に従って、IR-CPC を生成できなかったウェルを破棄します。これらのCMコミットCPCを精製するには、非細胞毒性光活性化プローブであるDual-Activatable Cell Tracker 1(DACT-1)を使用して、非CPC39を領域選択的に標識します。
- DACT-1(10 mM)をDMSOに溶解し、-20°Cで保存します。 6日目の細胞を1 μM DACT-1とCM分化培地中で30分間、37°Cでインキュベートします。
- 顕微鏡で生細胞を検査し、MATLAB で関心領域 (ROI) を多角形として選択します。ポリゴンの内側に20μm間隔で平行線を引き、交点の座標を計算します。
- 電動ステージの移動軌跡をトレースラインに従って設定すると、ステージは0.12 mm / sで移動し、ステージ上に組み立てられた405nmレーザーライン(直径20μm)がROIをグローバルにスキャンして、非CPCを制限的に光活性化できます。照射後、561 nmのレーザーラインを使用してDACT-1標識細胞を検出します。
- 0.05%トリプシン-EDTA(PBSで希釈)を37°Cで5〜7分間使用して細胞を解離し、インキュベーター内でプレートを2分間静かに振とうします。40 μmのセルストレーナーで細胞をろ過し、500 × g で3分間遠心分離します。細胞を0.5% BSAに再懸濁し、選別するまでチューブを氷上に保ちます。また、ソーティング用のネガティブコントロールとして未照射のサンプルを準備します。
- フローサイトメトリーソーティングシステムを起動し、ソーターソフトウェアをセットアップします。波長561nmのレーザーを選択し、DACT1+ソーティングには610/20のバンドパスフィルターを使用します。各サンプルについて、多角形ゲートツールを使用して、単一セルを選択するための前方散乱 (FSC) と側方散乱 (SSC) の二変量プロットで細胞集団を円で囲みます。次に、ネガティブコントロールを実行してバックグラウンド蛍光を確認し、領域選択的に照射したサンプルを実行し、DACT1+ 集団のゲートを描画します。
- 選別した細胞を10% FBSと5 μM Y27632を含むCM維持培地に回収して再懸濁し、選別した細胞をマトリゲルでコーティングした96ウェルプレートに播種します。
- 翌日、培地をCMメンテナンス培地に変更し、高純度のCMの収穫を待ちます。
- ステージIIIのML戦略:CMの認識
データセット:12日目の明視野画像のペアと最終的なcTnT蛍光画像のペア
モデル: pix2pix37- CMの明視野画像と対応するcTnT蛍光画像からなるデータセットを準備します。多様性を高めるには、データセットにさまざまな分化効率を持つウェルと細胞株が含まれていることを確認してください。
- データセットをトレーニング セットとテスト セットにランダムに分割します。作業ディレクトリを "stage_III" に切り替えます。明視野画像と対応するcTnT蛍光画像をそれぞれ「./pix2pix/datasets/(CM|CM_new_cell_lines)/A/(train|test)/*.png」と「.../B/(train|test)/*.png」に保存します。次のコマンドを実行します。
CDのpix2pix/datasets
python combine_A_and_B.py --fold_A ./CM/A --fold_B ./CM/B --fold_AB ./CM/ --no_multiprocessing
cd .. - トレーニング セットで pix2pix モデル37 をトレーニングします。次のコマンドを実行します(図3J)。
python train.py --dataroot ./datasets/CM --name brightfield2fluorescence --model pix2pix --input_nc 1 --output_nc 1 --load_size 1536 --crop_size 256 --lr 2e-4 --n_epochs 1000 --n_epochs_decay 0 --norm instance --netD のn_layers --n_layers_D 1 --batch_size 16 --方向 AtoB --save_epoch_freq 100 --dataset_mode整列 --use_resize_conv --seed 1234 - トレーニング済みのpix2pixモデルをテストセットに適用します。次のコマンドを実行します。
python test.py --dataroot ./datasets/CM --name brightfield2fluorescence --model pix2pix --direction AtoB --input_nc 1 --output_nc 1 --load_size 1536 --crop_size 1536 --use_resize_conv --eval --num_test 1000 - モデルによって予測された蛍光画像を、真の(つまり、実験的に得られた)画像と比較します。ピクセル レベルで、ピアソン相関係数を使用して、予測された強度と真の強度を比較します。さらに、画像レベルでは、ピアソン相関係数を使用して、予測された差別化効率指数と実際の指数を比較します。これらの比較のために、「stage_III/evaluation/pixel_correlation.m」と「image_correlation.ipynb」を実行します(図3K、L)。
- トレーニング済みのモデルを適用して、ステージ III の新しいバッチの CM を認識します。CMの新しい明視野画像を学習済みのpix2pixモデルに渡し、蛍光画像と分化効率の in-silico 予測を取得します。
注:細胞汚染を低減するために、DACT-1ベースの光活性化細胞ソーティングは、モデルによって認識されたCMの精製にも適用できます。
Access restricted. Please log in or start a trial to view this content.
結果
明視野イメージングとMLに基づいて、全体的な差別化プロセスをインテリジェントに監視し、最適化することができます。PSC段階では、初期のPSCコロニーの形態学的特徴に従って最終的な分化効率を予測できるMLモデルを開発し、分化を開始するのに最も適した、または適切な時点を決定しました(図4A、B)。ランダムフォレストモデ?...
Access restricted. Please log in or start a trial to view this content.
ディスカッション
ここでは、現在のPSCのアプリケーションと翻訳における主要な問題の1つである細胞分化のばらつきを克服するための詳細なプロトコルについて説明しました。生細胞明視野イメージングとMLを活用することで、PSCの分化を繰り返し最適化し、細胞株やバッチ間で一貫して高い効率を達成しました。しかし、上記の分化プロセスでは、プロトコールのいくつかの重要な...
Access restricted. Please log in or start a trial to view this content.
開示事項
Yang Zhao、Jue Zhang、Xiaochun Yang、Yao Wang、およびDaichao Chenは、この論文(202210525166.X)で報告されたPSC差別化戦略の特許を申請しています。
謝辞
この戦略の基礎を築いてくれたQiushi Sun氏、Yao Wang氏、Yu Xia氏、Jinyu Yang氏、Chang Lin氏、Zimu Cen氏、Dongdong Liang氏、Rong Wei氏、Ze Xu氏、Guangyin Xi氏、Gang Xue氏、Can Ye氏、Li-Peng Wang氏、Peng Zou氏、Shi-Qiang Wang氏、Pablo Rivera-Fuentes氏、Salome Püntener氏、Zhixing Chen氏、Yi Liu氏、Jue Zhang氏に感謝します。この作業は、中国の国家重点研究開発プログラム(2018YFA0800504、2019YFA0110000)および中国の有人宇宙計画の宇宙医学実験プロジェクト(HYZHXM01020)の支援を受けて、Yang Zhaoに行われました。 図 1 は BioRender.com を使用して作成されました。
Access restricted. Please log in or start a trial to view this content.
資料
| Name | Company | Catalog Number | Comments |
| 0.25% Trypsin-EDTA | Gibco | 25200056 | Diluted digests were used for CPC and CM digestion |
| 4% Paraformaldehyde in PBS | KeyGEN BioTECH | KGIHC016 | |
| 6-well Cell Culture Plate | NEST | 703001 | |
| 96-well Cell Culture Plate | NEST | 701001 | |
| B27 Supplement | Gibco | 17504044 | |
| B27 Supplement Minus Insulin | Gibco | A1895601 | |
| Bovine serum albumin (BSA) | GPC BIOTECH | AA904-100G | |
| Celldiscoverer 7 | Zeiss | Instruments used to take bright-field images throughout differentiation and final cTnT images | |
| CHIR99021 | Selleck | S1263 | |
| DMEM/F12 | Gibco | 12634010 | |
| Donkey anti-Mouse IgG (H+L) Highly Cross-Adsorbed Secondary Antibody, Alexa Fluor 488 | Thermo | A-21202 | Secondary Antibody |
| FACSAria III | BD Biosciences | Flow cytometry sorter | |
| Fetal Bovine Serum (FBS) | VISTECH | SE100-B | |
| Hoechst 33342 | YEASEN | 40732ES03 | |
| Human Pluripotent Stem Cell Chemical-defined Medium | Cauliscell Inc | 400105 | Basal medium of PSC preparation medium |
| iPS-18 | TaKaRa | Y00300 | |
| iPS-B1 | Cellapy | CA4025106 | |
| iPS-F | Nuwacell | RC01001-B | |
| iPS-M | Nuwacell | RC01001-A | |
| IWR1-1-endo | Selleck | S7086 | IWR1 |
| Jupyter Notebook | N/A | Version 6.4.0 | https://jupyter.org/ |
| MATLAB | MathWorks | Version R2020a | Software for scientific computation and image annotation |
| Matrigel Matrix | Corning | 354230 | Matrigel |
| Mouse monoclonal IgG1 anti-cTnT | Thermo | MA5-12960 | cTnT primary antibody |
| Normal Donkey Serum | Jackson | 017-000-121 | |
| ORCA-Flash 4.0 V3 digital CMOS camera | Hamamatsu | C13440-20CU | The digital camera assembled on Celldiscoverer7 |
| PBS | NEB | 21-040-CVR | |
| Penicillin-Streptomycin | Gibco | 15140-122 | |
| Pluripotency Growth Mater 1 basal medium | Cellapy | CA1007500-1 | Basal medium of PSC culture medium |
| Pluripotency Growth Mater 1 supplement | Cellapy | CA1007500-2 | Supplement of PSC culture medium |
| Prism | Graphpad | Version 8/9 | Statistical software for statistical analysis and plotting |
| Python | N/A | version 3.6 | Python 3 environment for scientific computation, with packages pytorch (1.9.0), numpy, scipy, pandas, visdom, scikit-learn, scikit-image, opencv-python, and matplotlib software for scientific computation and image annotation. |
| RPMI 1640 | Gibco | 11875176 | |
| Supplement hPSC-CDM (500x) | Cauliscell Inc | 00015 | Supplement of PSC preparation medium |
| TiE | Nikon | An inverted fluorescence microscope (with modification) for region-selevtive purification | |
| Triton X-100 | Amresco | 9002-93-1 | |
| Versene Solution | Thermo | 15040066 | EDTA solution for PSC digestion |
| Y27632 | Selleck | S6390 | |
| Zen | Zeiss | Version 3.1 | A supporting software of Celldiscoverer7 for image acquisition, processing and analysis |
参考文献
- Yoshida, Y., Yamanaka, S. Induced pluripotent stem cells 10 years later: for cardiac applications. Circ Res. 120 (12), 1958-1968 (2017).
- Shi, Y., Inoue, H., Wu, J. C., Yamanaka, S. Induced pluripotent stem cell technology: a decade of progress. Nat Rev Drug Discov. 16 (2), 115-130 (2017).
- Takahashi, K., et al. Induction of pluripotent stem cells from adult human fibroblasts by defined factors. Cell. 131 (5), 861-872 (2007).
- Burridge, P. W., et al. Chemically defined generation of human cardiomyocytes. Nat Methods. 11 (8), 855-860 (2014).
- Mordwinkin, N. M., Burridge, P. W., Wu, J. C. A review of human pluripotent stem cell-derived cardiomyocytes for high-throughput drug discovery, cardiotoxicity screening, and publication standards. J Cardiovasc Transl Res. 6 (1), 22-30 (2013).
- Vanden Berg, C. W., Elliott, D. A., Braam, S. R., Mummery, C. L., Davis, R. P. Differentiation of human pluripotent stem cells to cardiomyocytes under defined conditions. Methods Mol Biol. 1353, 163-180 (2016).
- Burridge, P. W., Holmström, A., Wu, J. C. Chemically defined culture and cardiomyocyte differentiation of human pluripotent stem cells. Curr Protoc Hum Genet. 87, 1-15 (2015).
- Li, J., et al. Human pluripotent stem cell-derived cardiac tissue-like constructs for repairing the infarcted myocardium. Stem Cell Rep. 9 (5), 1546-1559 (2017).
- Smith, A. S. T., Macadangdang, J., Leung, W., Laflamme, M. A., Kim, D. -H. Human iPSC-derived cardiomyocytes and tissue engineering strategies for disease modeling and drug screening. Biotechnol Adv. 35 (1), 77-94 (2017).
- Sharma, A., et al. Use of human induced pluripotent stem cell-derived cardiomyocytes to assess drug cardiotoxicity. Nat Protoc. 13 (12), 3018-3041 (2018).
- Mehta, A., et al. Identification of a targeted and testable antiarrhythmic therapy for long-QT syndrome type 2 using a patient-specific cellular model. Eur Heart J. 39 (16), 1446-1455 (2018).
- Cahan, P., Daley, G. Q. Origins and implications of pluripotent stem cell variability and heterogeneity. Nat Rev Mol Cell Biol. 14 (6), 357-368 (2013).
- Kim, K., et al. Epigenetic memory in induced pluripotent stem cells. Nature. 467 (7313), 285-290 (2010).
- Rouhani, F., et al. Genetic background drives transcriptional variation in human induced pluripotent stem cells. PLoS Genet. 10 (6), e1004432(2014).
- Kim, K., et al. Donor cell type can influence the epigenome and differentiation potential of human induced pluripotent stem cells. Nat Biotechnol. 29 (12), 1117-1119 (2011).
- Garitaonandia, I., et al. Increased risk of genetic and epigenetic instability in human embryonic stem cells associated with specific culture conditions. PLoS One. 10 (2), e0118307(2015).
- Engler, A. J., Sen, S., Sweeney, H. L., Discher, D. E. Matrix elasticity directs stem cell lineage specification. Cell. 126 (4), 677-689 (2006).
- Matoba, N., Yamashita, T., Takayama, K., Sakurai, F., Mizuguchi, H. Optimal human iPS cell culture method for efficient hepatic differentiation. Differentiation. 104, 13-21 (2018).
- Volpato, V., et al. Reproducibility of molecular phenotypes after long-term differentiation to human iPSC-derived neurons: A multi-site omics study. Stem Cell Rep. 11 (4), 897-911 (2018).
- Anderson, N. C., et al. Balancing serendipity and reproducibility: Pluripotent stem cells as experimental systems for intellectual and developmental disorders. Stem Cell Rep. 16 (6), 1446-1457 (2021).
- Sepac, A., et al. Comparison of cardiomyogenic potential among human ESC and iPSC lines. Cell Transplant. 21 (11), 2523-2530 (2012).
- Laco, F., et al. Unraveling the inconsistencies of cardiac differentiation efficiency induced by the GSK3β inhibitor CHIR99021 in human pluripotent stem cells. Stem Cell Rep. 10 (6), 1851-1866 (2018).
- Volpato, V., Webber, C. Addressing variability in iPSC-derived models of human disease: guidelines to promote reproducibility. Dis Model Mech. 13 (1), 042317(2020).
- Hu, B. -Y., et al. Neural differentiation of human induced pluripotent stem cells follows developmental principles but with variable potency. Proc Natl Acad Sci USA. 107 (9), 4335-4340 (2010).
- Huo, J., et al. Evaluation of batch variations in induced pluripotent stem cell-derived human cardiomyocytes from 2 major suppliers. Toxicol Sci. 156 (1), 25-38 (2017).
- Rostam, H. M., Reynolds, P. M., Alexander, M. R., Gadegaard, N., Ghaemmaghami, A. M. Image based machine learning for identification of macrophage subsets. Sci Rep. 7 (1), 3521(2017).
- Christiansen, E. M., et al. In silico labeling: Predicting fluorescent labels in unlabeled images. Cell. 173 (3), 792-803 (2018).
- Buggenthin, F., et al. Prospective identification of hematopoietic lineage choice by deep learning. Nat Methods. 14 (4), 403-406 (2017).
- Smith, Z. D., Nachman, I., Regev, A., Meissner, A. Dynamic single-cell imaging of direct reprogramming reveals an early specifying event. Nat Biotechnol. 28 (5), 521-526 (2010).
- Fan, K., Zhang, S., Zhang, Y., Lu, J., Holcombe, M., Zhang, X. A machine learning assisted, label-free, non-invasive approach for somatic reprogramming in induced pluripotent stem cell colony formation detection and prediction. Sci Rep. 7 (1), 13496(2017).
- Kusumoto, D., et al. Automated deep learning-based system to identify endothelial cells derived from induced pluripotent stem cells. Stem Cell Rep. 10 (6), 1687-1695 (2018).
- Zhu, Y., et al. Deep learning-based predictive identification of neural stem cell differentiation. Nat Commun. 12 (1), 2614(2021).
- Qian, T., et al. Label-free imaging for quality control of cardiomyocyte differentiation. Nat Commun. 12 (1), 4580(2021).
- Ounkomol, C., Seshamani, S., Maleckar, M. M., Collman, F., Johnson, G. R. Label-free prediction of three-dimensional fluorescence images from transmitted-light microscopy. Nat Methods. 15 (11), 917-920 (2018).
- Yang, X., et al. A live-cell image-based machine learning strategy for reducing variability in PSC differentiation systems. Cell Discovery. 9 (1), 53(2023).
- Selvaraju, R. R., et al. Grad-CAM: Visual explanations from deep networks via gradient-based localization. Proc IEEE Int Conf Computer Vision (ICCV). , 618-626 (2017).
- Isola, P., Zhu, J. -Y., Zhou, T., Efros, A. A. Image-to-image translation with conditional adversarial networks. Proc IEEE Conf Computer Vision Pattern Recognition (CVPR). , 5967-5976 (2017).
- Zhang, H., et al. ResNeSt: Split-attention networks. Proc IEEE/CVF Conf Computer Vision Pattern Recognition Workshops (CVPRW). , 2735-2745 (2022).
- Halabi, E. A., et al. Dual-activatable cell tracker for controlled and prolonged single-cell labeling). ACS Chem. Biol. 15 (6), 1613-1620 (2020).
- Hannan, N. R. F., Segeritz, C. -P., Touboul, T., Vallier, L. Production of hepatocyte-like cells from human pluripotent stem cells. Nat Protoc. 8 (2), 430-437 (2013).
- Zhang, H., Shen, M., Wu, J. C. Generation of quiescent cardiac fibroblasts derived from human induced pluripotent stem cells. Methods Mol Biol. 2454, 109-115 (2022).
- Harding, M. J., McGraw, H. F., Nechiporuk, A. The roles and regulation of multicellular rosette structures during morphogenesis. Development. 141 (13), 2549-2558 (2014).
- Najm, F. J., et al. Rapid and robust generation of functional oligodendrocyte progenitor cells from epiblast stem cells. Nat Methods. 8 (11), 957-962 (2011).
- Hogrebe, N. J., Maxwell, K. G., Augsornworawat, P., Millman, J. R. Generation of insulin-producing pancreatic β cells from multiple human stem cell lines. Nat Protoc. 16 (9), 4109-4143 (2021).
Access restricted. Please log in or start a trial to view this content.
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved