JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
만능줄기세포 분화를 모니터링하기 위한 살아있는 세포 이미지 기반 기계 학습 전략
* 이 저자들은 동등하게 기여했습니다
요약
사용 가능한 만능줄기세포(PSC)-기능성 세포 분화 시스템은 현재 심각한 line-to-line 및 batch-to-batch 변동성 문제로 인해 어려움을 겪고 있습니다. 여기에서는 심장 분화를 주요 예로 사용하여 이미지 기반 기계 학습을 기반으로 PSC 분화 과정을 지능적으로 모니터링하고 조절하는 프로토콜을 제시합니다.
초록
만능줄기세포(PSC) 기술은 신약 개발, 질병 모델링 및 재생 의학에 널리 사용되어 왔습니다. 그러나 사용 가능한 PSC-to-functional cell differentiation 시스템은 심각한 line-to-line 및 batch-to-batch 변동성 문제로 인해 어려움을 겪고 있습니다. 따라서 실시간으로 세포 분화를 정밀하게 제어하는 것이 중요합니다. 이 프로토콜에서는 명시야 이미지 기반 기계 학습을 사용하여 세포 분화의 가변성을 극복하는 비침습적이고 지능적인 전략을 설명합니다. PSC-to-cardiomyocyte 분화를 예로 들면, 이 방법론은 초기 PSC 상태의 제어, 분화 조건에서의 조기 평가 및 개입, 잘못 분화된 세포 오염 제거에 대한 자세한 정보를 제공하며, PSC에서 기능성 세포로의 일관된 고품질 분화를 실현합니다. 원칙적으로 이 전략은 세포 제조를 지원하기 위해 여러 단계를 통해 다른 세포 분화 또는 재프로그래밍 시스템으로 확장될 수 있을 뿐만 아니라 세포 운명 변환 중 메커니즘에 대한 이해를 높일 수 있습니다.
서문
만능줄기세포(PSC)는 체외에서 다양한 유형의 세포로 분화할 수 있는 놀라운 능력을 가지고 있습니다. 이러한 분화된 기능 세포는 세포 치료, 질병 모델링 및 약물 개발에 사용될 수 있으며, 모두 연구 또는 임상 응용 분야에 유용합니다 1,2,3. 예를 들어, PSC를 심근세포(CM)4,5,6,7로 분화하기 위한 다양한 방법이 개발되었습니다. 이 CM은 약물의 심장 독성 검사, 심장 질환 모델링 및 세포 이식에 적용 할 수 있습니다 8,9,10,11.
그러나 PSC에서 말단 분화 세포로의 전환은 단계적 과정이며, 분화 과정 중 여러 번의 섭동으로 인해 세포의 운명이 발산할 수 있습니다. PSC 계통의 상이한 유전적 배경과 후성유전학적 특징은 특정 계통으로의 분화 가능성에 영향을 미친다 12,13,14,15; PSC 통로의 수와 축적된 유전자 돌연변이도 PSC 이질성의 원인입니다. 다른 실험자에 의해 채택된 실험 연산의 차이는 또한 완전히 다른 분화 결과로 이어질 수있다 16,17,18,19,20. 따라서 현재 PSC 유래 세포 생산의 주요 문제 중 하나는 세포주 및 배치 21,22,23,24,25 간의 불안정성입니다. PSC 분화의 불안정성으로 인해 여러 번의 반복적인 실험이 발생하여 상당한 시간과 노동력이 소모되는 경우가 많습니다. 이 문제를 해결하려면 세포주와 배치 간의 변동성을 최소화하여 분화의 안정성과 견고성을 향상시키는 전략을 개발하는 것이 중요합니다.
최근 고해상도 현미경 및 머신 러닝(ML)의 발전으로 세포 생물학에서 ML 기반 정량 이미지 분석의 적용이 용이해짐에 따라 세포 이미징 기능 26,27,28,29,30,31,32,33,34에서 귀중한 정보를 활용할 수 있게 되었습니다. 이전 연구에서는 PSC 분화의 안정성과 효율성을 개선하기 위해 실시간으로 PSC 분화 상태를 모니터링하고 개입하는 라이브 셀 이미지 기반 ML 전략을 제안했습니다(그림 1)35. PSC-to-cardiomyocyte 분화를 예로 들어 랜덤 포레스트 모델을 사용하여 초기 PSC 상태를 평가하고, 로지스틱 회귀 모델을 사용하여 최적의 분화 조건을 예측하고, 딥러닝 기반 Grad-CAM36 및 pix2pix37을 사용하여 성공적으로 분화된 세포를 인식했습니다. ML 모델은 면적, 둘레, 볼록성, 견고성, 밝기, 이동 속도 및 심층 컨볼루션 신경망에서 추출한 기타 암시적 특징을 포함한 다양한 명시야 형태학적 특징에서 세포 계통을 식별하는 방법을 학습했습니다. 이러한 확립된 ML 모델의 추론을 기반으로 초기 PSC 상태의 제어, 분화 조건에 대한 조기 평가 및 개입, 오분화된 세포 오염 제거를 실현하여 심장 분화 과정에 대한 포괄적이고 정확한 조절을 제공했습니다. 여기에서는 전략을 개발하기 위한 단계별 프로토콜을 제공합니다.
Access restricted. Please log in or start a trial to view this content.
프로토콜
1. 세포 분화 및 특성화
- 배양 시약 및 배양 플레이트 준비
- 48mL의 기초 배지에 보충제 2mL와 페니실린-스트렙토마이신 0.2%를 첨가하여 PSC 배양 배지를 제조합니다. 보충제를 분취하여 -20 °C에서 보관하십시오. 이 매체를 4°C에서 최대 4주 동안 보관하십시오.
- 기저 배지 500mL에 보충제 1mL와 페니실린-스트렙토마이신 0.2%를 첨가하여 PSC 제조 배지를 준비합니다. 사용할 때 한 번 사용하기 위해 매체를 예열하고 매체를 최대 4주 동안 3°C에서 보관하십시오.
- RPMI 1640에 인슐린을 뺀 1x B27 보충제와 1% 페니실린-스트렙토마이신을 첨가하여 CM 분화 배지를 준비합니다. RPMI 1640에 1x B27 보충제와 1% 페니실린-스트렙토마이신을 추가하여 CM 유지 매체를 준비합니다. 이 두 종류의 매체를 4°C에서 2주 동안 보관합니다.
- Matrigel을 얼음에 담그고 4°C에서 밤새 보관합니다. Matrigel이 완전히 해동되었는지 확인하십시오. 그런 다음 나중에 사용하기 위해 -80 °C에서 보관된 1 mL 부분 표본에 분취하고, 4 °C에서 49 mL의 DMEM/F12에 1 mL의 Matrigel 을 첨가하여 Matrigel 작업 용액을 준비합니다. 6-웰 배양 플레이트의 웰당 850 μL의 이 Matrigel 작업 용액을 추가하고 플레이트를 37°C에서 최소 30분 동안 배양합니다. CM 분화에 사용되는 96-웰 배양 플레이트의 경우 웰당 35μL의 Matrigel 작업 용액을 추가합니다.
알림: Matrigel을 항상 얼음 위에 두십시오. 피펫 팁과 튜브는 4°C에서 사전 냉각해야 합니다. Matrigel 작업 용액은 4°C에서 2주 동안 보관할 수 있습니다. Matrigel 작업 용액을 피펫팅할 때 기포가 생기지 않도록 주의하십시오.
- PSC 유지 보수 및 통과
- 6-well 배양 플레이트에서 PSC를 유지합니다. 현미경으로 세포 밀도를 확인하고 밀도가 ~70%에 도달하면 통과를 준비합니다(그림 2A).
- EDTA를 사용하여 passaging을 위한 세포를 분해합니다. 37°C 수조에서 PSC 배양 배지, PBS 및 EDTA를 데우고 배지에 Y27632를 첨가하여 최종 농도 5μM까지 합니다.
- PBS 1mL로 PSC를 세척하고, 웰당 1mL의 EDTA를 추가하고, 플레이트를 37°C 인큐베이터에서 3분 동안 배양합니다.
- 세포가 배양 플레이트에서 분리되기 전에 EDTA를 흡인한 다음 1mL의 PSC 배양 배지를 사용하여 웰 바닥을 3-4배 부드럽게 헹구어 세포를 재현탁시킵니다.
알림: 이를 통해 원심분리 단계를 건너뛰고 패세이징 작업에서 세포 손상을 줄일 수 있습니다. - 6-웰 배양 플레이트의 Matrigel 코팅 웰에서 DMEM/F12를 흡입하고 웰당 2mL의 새로운 PSC 배양 배지를 추가합니다(Y27632 추가). 셀 현탁액을 1:6에서 1:12의 분할 비율로 웰에 추가합니다. 플레이트를 37% CO5가 있는 2°C 인큐베이터에 놓습니다.
- 통과 후 PSC 배양 배지(Y27632 제외)를 12시간에서 24시간으로 변경합니다. 그런 다음 배지를 교체하고 매일 세포 상태와 합류점을 확인합니다.
- PSC의 심장 분화
- CM 분화를 위해 PSC 준비 배지(Y27632 추가)의 96웰 배양 플레이트에 PSC를 시드합니다. CM 분화를 위해 위와 동일한 단계(단계 1.2.2-1.2.6)에 따라 PSC 준비 배지(Y27632 추가)의 96웰 배양 플레이트에 PSC를 시드합니다.
참고: 이 단계에서, EDTA 처리 기간을 적절하게 연장(5분 이상)하여 PSC를 충분히 해리시켜 클론이 통과 후 더 작아질 수 있습니다. 우리의 연구는 동일한 지역 내에서 더 작은 군체가 분화에 도움이 된다는 것을 보여주었습니다. - I단계: PSC가 80-90% 밀도에 도달하면 배지를 2-20μM CHIR99021(CHIR)의 CM 분화 배지로 전환합니다(그림 2B). 매체 전환의 시점을 day 0이라고 합니다. 24-48 시간 CHIR 처리 후 매체를 새로운 CM 분화 매체로 변경하십시오.
참고: CHIR은 중배엽 유도를 위한 GSK3beta 억제제입니다. CM 분화를 위한 최적의 CHIR 투여량은 세포주와 배치에 따라 변동합니다. 예를 들어, 최적의 CHIR 선량은 8-10 μM, iPS-B1의 경우 48 시간입니다. ML 모델 훈련을 위한 다양한 명시야 이미지를 수집하기 위해 여기서는 다양한 차별화 효율성에 대해 각 실험 배치에 대한 CHIR 농도를 적정했습니다. - 2단계: 72시간(3일)에 배지를 5μM IWR1이 보충된 CM 분화 배지로 교체하고 48시간(5일) 동안 배양합니다. CM 분화 배지로 매체를 1-2 일 동안 변경하십시오 (6-7 일까지). 이 시점에서 PSC는 심장 전구 세포(CPC)로 분화합니다.
- 3 단계 : 매체를 CM 유지 매체로 교체하고 3 일마다 매체를 교체합니다. 10일 또는 12일째에 추가 분석을 위해 CM을 수확합니다.
- CM 분화를 위해 PSC 준비 배지(Y27632 추가)의 96웰 배양 플레이트에 PSC를 시드합니다. CM 분화를 위해 위와 동일한 단계(단계 1.2.2-1.2.6)에 따라 PSC 준비 배지(Y27632 추가)의 96웰 배양 플레이트에 PSC를 시드합니다.
- CM의 면역형광 염색
- 10일 또는 12일째에 PBS로 세포를 세척하고 실온에서 15분 동안 PBS에 4% 파라포름알데히드로 고정합니다. PBS로 CM을 2배 세척하고 플레이트를 4°C에서 최대 ~1주일 동안 보관합니다.
참고: 파라포름알데히드는 톡 쏘는 냄새가 나는 독성 시약입니다. 화학 후드를 착용하고 작동하고 눈과 손을 보호하십시오. - 염색 시 세포를 투과성 용액(PBS의 0.1% Triton X-100)으로 실온에서 15분 동안 처리합니다. 그런 다음 차단 용액(PBS에 3% 당나귀 혈청을 함유한 0.1% Triton X-100)에 실온에서 30분 동안 세포를 배양합니다.
- CM을 확인하기 위해 4°C에서 차단 용액에 밤새 희석한 Cardiac Troponin T(cTnT) 1차 항체(1:300)로 검체를 배양합니다.
- 1차 항체를 채취하고 PBS 3x로 세포를 세척합니다. 어두운 환경에서 37°C에서 1시간 동안 1% 소 혈청 알부민이 있는 PBS의 2차 항체와 함께 샘플을 배양합니다.
참고: 수집된 1차 항체는 1주일에 2-3회 재사용할 수 있습니다.
- 1차 항체를 채취하고 PBS 3x로 세포를 세척합니다. 어두운 환경에서 37°C에서 1시간 동안 1% 소 혈청 알부민이 있는 PBS의 2차 항체와 함께 샘플을 배양합니다.
- 세포에서 2차 항체를 제거하고 PBS로 세포를 3회 세척합니다. Hoechst 33342(PBS의 1:1,000)를 사용하여 실온에서 5분 동안 핵을 염색합니다. PBS를 사용하여 셀을 3번 헹구고 건조를 방지하기 위해 웰당 100μL의 PBS를 추가합니다. 플레이트를 4°C의 어두운 곳에 보관하고 1주일 이내에 형광 이미지를 얻을 수 있습니다.
- 10일 또는 12일째에 PBS로 세포를 세척하고 실온에서 15분 동안 PBS에 4% 파라포름알데히드로 고정합니다. PBS로 CM을 2배 세척하고 플레이트를 4°C에서 최대 ~1주일 동안 보관합니다.
2. 차별화 프로세스 전반에 걸친 이미지 스트림 획득
- 이미지 수집을 위한 실험 설계 설정
- 살아있는 세포 배양 및 이미징을 지원하는 자동 현미경을 사용하여 CM 분화의 여러 단계에 대한 명시야 이미지와 분화 결과의 cTnT 면역형광 이미지를 수집합니다(그림 2A-E). 현미경 지원 소프트웨어를 사용하여 실험 프로그램을 설계하고 현미경을 제어합니다.
- 소프트웨어를 열고 새 실험 설계를 만듭니다. 이미징을 위해 5x objective와 2x tube lens를 선택하십시오. 타일 및 Z 스택 옵션을 선택합니다.
- 채널 메뉴에서 명시야 이미징을 위한 TL Brightfield 채널을 추가합니다. 면역형광 이미징을 위해 AF488 및 H3342 채널을 추가합니다. 외부 디지털 CMOS 카메라로 이미징하기 위해 이미징 설정 메뉴에서 광 경로를 수정합니다.
- Acquisition Mode 메뉴를 열고 2 x 2 binning을 설정하여 신호 대 노이즈 비율을 높입니다.
- Z-stack 메뉴를 열고 중심 모델을 선택합니다. 스캔할 때 슬라이스 수와 간격을 설정합니다. 분석에서 초점이 맞는 이미지를 위해 3-6μm 간격으로 3-5개의 슬라이스를 선택합니다.
- Navigation & Tiles(탐색 및 타일) 창에서 Carrier(캐리어)별로 타일 영역을 설정하고 하나의 웰에 대해 25개의 타일(5개 열 x 5개 행)을 설정합니다. Tiles 메뉴의 Sample Carrier에서 Multiwell 96 템플릿을 선택하고 인접 영역에서 5%-15% 의 중첩을 설정합니다.
- 연속 이미징이 필요한 경우 Time series 옵션을 선택하고 Time series 메뉴에서 지속 시간과 간격을 설정합니다. 스테이지 I에서 이미지 스트림을 수집하려면 간격 없이 12시간 지속 시간을 설정합니다.
- bright-field 및 immunofluorescent 이미지 획득
- 이미지를 획득하려면 먼저 세포 배양 플레이트를 샘플 트레이에 넣고 샘플을 현미경 내부에 로드합니다. 실험 설계와 일치하는 Multiwell 96 템플릿을 선택합니다. 샘플이 살아있는 세포로 구성된 경우 가열 시스템과 CO2 펌프를 열어 배양에 적합한 조건(37°C, 5% CO2)을 유지합니다.
- 사전 설정된 실험 프로젝트를 열고 새 프로젝트로 저장합니다. Tiles 메뉴를 열고 플레이트의 위치를 수동으로 보정합니다. Navigation & Tiles 창에서 필요한 웰을 선택하고 Create를 클릭하여 이러한 웰에 대한 타일 영역을 구성합니다.
- Tiles 메뉴에서 Verify Tile Regions를 클릭하고 Autofocus를 실행하여 모든 웰을 확인합니다. 그런 다음 마우스 휠을 스크롤하여 명시야 아래에 있는 각 웰의 초점을 수동으로 수정합니다.
- 필요한 채널을 선택하고 채널 메뉴에서 노출 시간을 설정합니다. 권장 노출 시간은 Bright의 경우 1.625ms, H3342의 경우 100ms, AF488의 경우 550ms입니다. 스냅 한 번으로 모든 채널의 이미지를 확인할 수 있습니다.
- 마지막으로 실험 시작 버튼을 클릭하고 자동 이미징을 기다립니다. 일반적으로 전체 96웰 배양 플레이트(96웰, 3개 레이어, 명시야 아래 각 웰에 대해 25타일)의 스캔을 완료하는 데 ~1.2시간이 걸립니다. [Processing framework]에서 [Image Export]를 선택하고 압축되지 않은 TIFF 형식 또는 PNG 형식의 파일 형식을 선택한 다음 적용합니다. 추가 분석을 위해 우물 중앙에 있는 9개의 타일(3 x 3) 이미지를 사용합니다(그림 2A-E).
3. 차별화 프로세스의 각 단계에서 이미지 기반 ML 전략 수립
- 소프트웨어 준비
- pytorch(1.9.0), numpy, scipy, scikit-learn, pandas, visdom, scikit-image, opencv-python 및 matplotlib 패키지를 사용하여 Python 환경을 설치합니다.
- Jupyter Notebook을 설치합니다.
- MATLAB을 설치합니다.
- https://github.com/zhaoyanglab/ML-for-PSC-differentiation 에서 코드와 예제 데이터 세트를 다운로드합니다.
참고: ML의 성능은 데이터 세트의 품질과 모델 및 하이퍼 파라미터의 선택에 따라 달라집니다. 예제 구현이 모든 상황에서 최적이라고 보장되는 것은 아닙니다. ML 엔지니어는 코드를 사용자 지정하고 특정 요구 사항에 맞게 모델 및 하이퍼 매개 변수를 조정해야 합니다.
- PSC 단계에서의 ML 전략: 초기 PSC 콜로니 상태 제어
데이터 세트: 0시간(CHIR 처리 전)의 명시야 이미지 쌍 및 최종 차별화 효율
모델: 랜덤 포레스트- CHIR 처리 전인 0h의 명시야 이미지와 최종 cTnT 형광 이미지로 구성된 데이터 세트를 준비합니다. 0h 명시야 이미지의 형태학적 프로파일의 다양성을 높이려면 데이터 세트에 서로 다른 세포주의 PSC 콜로니가 포함되어 있고 통과 후 시간 간격에 걸쳐 분화가 시작되는지 확인하십시오. 스테이지 I에서 최적의 CHIR 조건으로 데이터 세트의 웰을 처리합니다.
참고: 콜로니가 최적의 CHIR 조건으로 처리되도록 하기 위해 동일한 배치 내의 웰을 적정된 CHIR 용량으로 처리할 수 있습니다. 고효율 CHIR 조건을 가진 우물만 데이터 세트에 포함됩니다. - cTnT 형광 이미지에서 계산된 분화 효율 지수(Differentiation Efficiency Index)로 각 웰의 분화 효율을 정량화합니다(
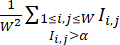 여기서
여기서 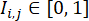 는 W × W 형광 이미지의 픽셀 위치(i, j)에서의 형광 강도를 나타내고, 합계는 임계값 이상의 강도를 가진 모든 픽셀 위치에 대해 취해집니다α, 일반적으로 0.5로 설정됨). 각 세포주 내에서 이 세포주의 모든 웰에 대한 최대 분화 효율 지수를 계산하고 해당 분화 효율 지수를 최대값으로 정규화합니다.
는 W × W 형광 이미지의 픽셀 위치(i, j)에서의 형광 강도를 나타내고, 합계는 임계값 이상의 강도를 가진 모든 픽셀 위치에 대해 취해집니다α, 일반적으로 0.5로 설정됨). 각 세포주 내에서 이 세포주의 모든 웰에 대한 최대 분화 효율 지수를 계산하고 해당 분화 효율 지수를 최대값으로 정규화합니다.
참고: 각 세포주의 분화 잠재력은 본질적으로 다르기 때문에 이러한 정규화는 분화 결과에 기여하는 다양한 세포주 간에 공유되는 colony 형태학적 특징에 더 중점을 둡니다. 정규화는 실제로 서로 다른 세포주의 PSC 콜로니를 비교할 수 있게 하고 세포주에 의해 유도되는 편향을 완화합니다. - 0h 명시야 이미지의 형태학적 프로파일을 콜로니 모양의 특성을 나타내는 고차원 특징으로 정량화합니다. 명시야 영상에서 콜로니 영역의 윤곽을 그리려면 국소 엔트로피(반지름 r)를 계산하십시오. 임계값 σ보다 큰 로컬 엔트로피를 가진 픽셀은 cells로 덮여 식별됩니다. r = 10픽셀 및 σ = 3의 매개 변수 설정을 사용합니다. 작업 디렉토리를 "./colony_control"로 전환하십시오. 명시야 이미지, 세포 영역의 이진 이미지 및 cTnT 형광 이미지를 "colony_control/ image_data/CD00-*/[brightfield|cell_region|ctnt]/S*.png"에 저장합니다. Jupyter Notebook "Compute_features.ipynb"를 실행하여 식민지 면적, 원주, 솔리디션, 볼록성, 원형도 및 중심 윤곽 거리와 관련된 기능을 계산합니다.
- 데이터 세트를 학습 세트와 테스트 세트로 무작위로 나누며, 학습 세트는 전체 데이터의 50%에서 80% 사이로 구성됩니다. 마지막 블록인 Jupyter Notebook에서 데이터 세트 분할을 실행하여 데이터 세트를 분할합니다(그림 3A).
- 훈련 세트에서 랜덤 포레스트 회귀 모델을 훈련시켜 0시간 명시야 이미지 특징과의 차별화 효율성을 예측합니다. Jupyter Notebook "Machine_learning.ipynb"를 실행하여 모델을 학습시키고 테스트합니다.
- 테스트 세트에서 훈련된 랜덤 포레스트 모델을 평가합니다. Pearson 상관 계수를 사용하여 예측된 차별화 효율이 실제 차별화 효율과 높은 상관 관계가 있는지 확인합니다(그림 3B).
참고: 테스트 세트는 학습 단계에 포함되지 않기 때문에 테스트 세트에서 예측된 차별화 효율성과 실제 차별화 효율성 간의 일관성이 높다는 것은 모델이 보이지 않는 샘플에서 잘 일반화될 수 있음을 나타냅니다. - 훈련된 랜덤 포레스트 모델에서 기능 중요도를 얻고 중요도가 가장 높은 8개의 기능을 선택합니다. 특징 범위를 Bin으로 나누고 각 Bin의 평균 미분 효율을 계산하여 최종 미분 효율과의 관계를 시각화합니다. Jupyter Notebook "Feature_importance.ipynb"를 실행하여 시각화를 수행합니다(그림 3C).
참고: 시각화는 PSC 콜로니 모양의 변화가 분화에 어떻게 기여하는지 이해하는 데 도움이 될 수 있습니다. - 훈련된 모델을 새로운 배치에 적용하여 PSC 콜로니 상태를 실시간으로 모니터링할 수 있습니다. 매 시간마다 명시야 이미지를 캡처하고, 이미지를 훈련된 모델에 전달하고, 예측된 차별화 효율성을 얻습니다. 예측된 효율이 임계값(예: 50%)을 초과하면 미분 프로세스를 시작합니다. 예측된 효율이 임계값에 도달할 수 없는 경우 이러한 샘플을 폐기합니다.
참고: PSC 단계에서는 PSC 콜로니의 분화 잠재력이 먼저 증가하고 나중에 감소합니다. 실시간 모니터링을 통해 실험자는 차별화를 시작하는 데 가장 도움이 되는 시점을 식별할 수 있습니다.
- CHIR 처리 전인 0h의 명시야 이미지와 최종 cTnT 형광 이미지로 구성된 데이터 세트를 준비합니다. 0h 명시야 이미지의 형태학적 프로파일의 다양성을 높이려면 데이터 세트에 서로 다른 세포주의 PSC 콜로니가 포함되어 있고 통과 후 시간 간격에 걸쳐 분화가 시작되는지 확인하십시오. 스테이지 I에서 최적의 CHIR 조건으로 데이터 세트의 웰을 처리합니다.
- 단계 I의 ML 전략: CHIR 용량의 조기 평가 및 조정
데이터 세트: 0-12시간의 명시야 이미지 스트림 쌍 및 CHIR 농도 레이블(낮음/최적/높음)
모형: 로지스틱 회귀 분석- 0시간에서 12시간 사이에 매시간 이미지를 획득하는 전체 웰 명시야 이미지 스트림으로 구성된 데이터 세트를 준비합니다. 각 웰의 CHIR 투여량(즉, CHIR 농도와 지속 시간의 조합)을 기록합니다. 다양성을 높이기 위해 데이터 세트에 적정된 CHIR 용량(예: 농도 = 2, 4, 6, 8, 10 및 12μM, 기간 = 24, 36 및 48시간)이 있는 다양한 배치 및 웰이 포함되어 있는지 확인하십시오. 작업 디렉토리를 "./stage_I"로 전환하십시오. 이미지를 "./data/image/CD01-*/S*/T*.png"로 저장하며, 여기서 CD01-* (CD01-1, CD01-2, CD01-3, CD01-4)는 배치 이름, S* (S1, S2, ..., S96)은 웰의 인덱스, T*.png(T1.png, T2.png, ..., T10.png)은 이미지 스트림의 다른 시간 단계에서 웰의 전처리된 명시야 이미지입니다.
- 각 배치에서 각 웰에 대한 cTnT+ 셀의 백분율을 계산하며, 이는 cTnT+ 셀의 수를 형광 이미지의 총 Hoechst+ 셀 수로 나누어 계산합니다. 동일한 CHIR 투여 조건을 가진 평행 웰에 대한 cTnT+ 세포의 백분율을 평균화합니다. CHIR 투여 조건과 각 웰에 대한 cTnT+ 세포의 백분율을 csv 파일 "./data/image/CD01-*/CD01-?_exp_config.csv"에 저장합니다.
- 각 배치에서 다음 기준에 따라 각 CHIR 지속 시간 동안 "낮음", "최적" 및 "높음" CHIR 농도 범위를 결정합니다. 임계값(예: 20%)을 초과하는 cTnT+ 세포의 평균 백분율을 가진 CHIR 농도는 "최적"으로 식별됩니다. 최적 범위를 벗어난 CHIR 농도는 "낮음" 또는 "높음"으로 식별됩니다. 각 CHIR 지속 시간에서 해당 CHIR 농도에 따라 우물을 "낮음", "최적" 및 "높음"으로 레이블을 지정합니다.
참고: 경험적으로, CHIR 농도와 배치 내 고효율 웰의 지속 시간 사이에는 음의 상관 관계가 있습니다: CHIR 지속 시간이 높을수록 최적의 농도가 감소합니다. 즉, CHIR 농도의 최적성은 CHIR 지속 시간에 따라 달라지므로 웰의 레이블은 CHIR 지속 시간에 따라 다릅니다. - 각 배치의 각 CHIR 지속 시간에서 각 농도 c에 대한 ΔCHIR 농도를 계산하여 최적과의 편차를 정량화합니다. 최적의 CHIR 농도 범위를 [c1, c2]로 표시한 다음 ΔCHIR 농도(c) = c - c1(c < c1인 경우), 0(c1 ≤ c ≤ c2) 또는 c - c 2(c > c2인 경우)로 표시합니다.
- 데이터 세트에서 이미지 스트림의 특징을 추출하여 단계 I 동안 세포의 형태학적 변화를 반영하도록 합니다. 국소 엔트로피, 면적, 둘레, 밝기, 광학 흐름, 프랙탈 차원에 대한 영상 특징을 계산하려면 다음을 실행하십시오
cd ./데이터
파이썬 compute_features.py
시디..
참고: 형태학적 변화를 강조하기 위해 일부 특징(예: 면적, 둘레 및 광학 흐름)의 값은 이미지 스트림의 초기 기준선 값으로 정규화될 수 있습니다. - 데이터 세트를 학습 세트와 테스트 세트로 무작위로 나눕니다. Jupyter Notebook "./data/prepare_dataset.ipynb"를 실행하여 데이터 세트를 분할하고 데이터 프레임으로 구성합니다(그림 3D).
- 각 CHIR 기간에 대해 로지스틱 회귀 모델을 훈련시켜 훈련 세트의 이미지 스트림 기능에서 CHIR 농도 레이블(낮음/최적/높음)을 예측합니다. 정확도, 정밀도, 재현율, F1 점수 및 AUC(Area Under the Curve)를 사용하여 테스트 세트에서 훈련된 로지스틱 회귀 모델의 분류 성능을 평가합니다. 학습 및 테스트를 위해 Jupyter Notebook "machine_learning.ipynb"를 실행합니다(그림 3E).
- CHIR 용량 평가에서 모델 성능을 평가합니다. 테스트 세트에서 로 정의
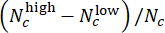 되는 편차 점수(-1에서 1까지의 범위)를 사용하여 CHIR 농도가 동일한 평행 웰의 예측 레이블을 병합합니다. 여기서 Nc는 농도가 c인 웰의 수를 나타내고
되는 편차 점수(-1에서 1까지의 범위)를 사용하여 CHIR 농도가 동일한 평행 웰의 예측 레이블을 병합합니다. 여기서 Nc는 농도가 c인 웰의 수를 나타내고 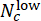 그
그 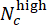 중 웰은 각각 "낮음" 및 "높음"으로 예측됩니다. Pearson 상관 계수를 사용하여 예측된 편차 점수가 각 CHIR 용량에 대한 실제 ΔCHIR 농도와 높은 상관 관계가 있는지 확인합니다.
중 웰은 각각 "낮음" 및 "높음"으로 예측됩니다. Pearson 상관 계수를 사용하여 예측된 편차 점수가 각 CHIR 용량에 대한 실제 ΔCHIR 농도와 높은 상관 관계가 있는지 확인합니다. - 교차 배치 검증을 수행하여 모델의 일반화 성능을 테스트합니다. 각 라운드에서 한 배치는 테스트용으로 남겨두고 다른 배치는 훈련용으로 사용합니다. Pearson 상관 계수를 사용하여 모든 테스트 배치에서 각 CHIR 용량에 대한 예측된 편차 점수와 실제 ΔCHIR 농도를 비교합니다. Jupyter Notebook "cross_batch_validation.ipynb"를 실행하여 일괄 처리 간 유효성 검사를 구현합니다(그림 3F).
참고: 선택한 CHIR 지속 시간 h의 경우, 지속 시간 h 에서 훈련된 로지스틱 회귀 모델의 농도가 있는 모든 웰에 대한 예측은 CHIR 농도 c의 최적성을 반영하는 편차 점수로 변환됩니다. 편차 점수가 0에 가까우면 농도 c 가 지속 시간 h 하에서 최적으로 예측됨을 시사하는 반면, 음수 또는 양수 편차 점수는 과소 투여 또는 과량 투여 상태를 암시하며, 이는 나중에 CHIR 투여량을 조정하기 위한 신호로 작용할 수 있습니다. 예측이 일찍(12시간) 이루어지기 때문에 스테이지 I의 나머지 부분 동안의 최종 CHIR 지속 시간과 CHIR 농도는 여전히 조정될 수 있습니다. - 훈련된 로지스틱 회귀 모델을 적용하여 새로운 배치에서 CHIR 투여량을 평가합니다. 주어진 CHIR 농도에 대해 농도가 있는 평행 웰에 대한 0-12시간 이미지 스트림 기능을 계산하고, 기능을 서로 다른 CHIR 지속 시간 하에서 훈련된 로지스틱 회귀 모델에 전달하고, 그에 따라 각 웰에 대한 예측 레이블을 얻습니다. 모델의 예측에 따라 편차 점수를 계산하여 서로 다른 CHIR 지속 시간에서 각 CHIR 농도를 평가합니다.
- 모델 기반 CHIR 선량 평가를 통해 CHIR 지속 시간 또는 농도를 48시간 전에 최적으로 조정하여 최적이 아닌 각 CHIR 농도 하에서 우물을 구출할 수 있습니다.
- 지속 시간 조정: 최적의 CHIR 지속 시간에 대한 모델 예측에 따라 이러한 웰의 CHIR 함유 매체가 CM 분화 매체로 전환되는 시점을 조정합니다. 최적의 시점을 놓친 경우 이러한 샘플을 폐기하십시오.
- 농도 조정: 사전 설정된 CHIR 지속 시간 내에서 현재 농도가 "낮음"으로 예측되면 매체에 고농도의 CHIR을 직접 추가하여 CHIR 농도를 높입니다. 필요한 경우 PBS로 DMSO(50mM)에 CHIR을 희석하고 적절한 양의 액체를 추가한 다음 플레이트를 부드럽게 흔듭니다. 현재 농도가 "높음"으로 예측되면 일정량의 CHIR 함유 매체를 흡입하고 동일한 부피의 CM 분화 매체를 보충합니다.
참고: 분화의 처음 48시간 동안 세포는 세포 성장과 분화를 개선하기 위해 인자를 분비하므로 배지를 완전히 변경하지 마십시오. 또한 차별화 시 매체 볼륨을 일정하게 유지해야 합니다. 따라서 CHIR 농도를 조정할 때 첨가되는 액체의 부피가 가능한 한 작은지 확인하십시오.
- 2단계 ML 전략: CM 커밋 CPC 인식
데이터 세트: 6일차의 명시야 이미지 쌍과 CM 커밋 CPC의 수동 주석 마스크
모델: ResNeSt38 (약한 지도 학습을 위한 Grad-CAM36 포함)- 6일차에 명시야 이미지로 구성된 데이터 세트를 준비합니다. 12일째부터 6일째까지 이미지 스트림의 cTnT+ 세포를 추적하여 명시야 이미지에서 CM 커밋 CPC에 수동으로 주석을 달 수 있습니다.
- CM-커밋 CPC의 명시야 이미지와 해당 수동 주석(마스크라고 함)을 패치로 자르고 각 패치 크기는 전체 웰 이미지 크기의 1/5.5로 합니다. 인접한 패치가 훈련 세트에서 50% 겹치고 테스트 세트에서 75% 겹치는 중복 방식으로 패치를 추출합니다. ≥30% CM 커밋 CPC가 있는 패치를 '긍정적'으로 라벨링합니다. CM 커밋 CPC가 없는 패치를 '부정적'으로 라벨링합니다. 작업 디렉토리를 "stage_II"로 전환합니다. "./Crop_and_Reconstruct/mask_crop.m"을 실행하여 이미지를 자릅니다. "./Crop_and_Reconstruct/classification.m"을 실행하여 명시야 패치에 레이블(0: 음수, 1: 양수)을 할당합니다(그림 3G).
- 레이블이 지정된 영상 패치를 무작위로 훈련 세트와 테스트 세트로 나눕니다.
- 심층 컨벌루션 신경망 ResNeSt38 을 훈련시켜 이러한 패치를 분류하는 방법을 학습합니다. 테스트 세트에서 훈련된 ResNeSt를 평가하여 모델이 높은 분류 정확도를 보여주는지 확인합니다. 훈련 및 테스트를 위해 각각 "train.py"와 "test.py"를 실행합니다(그림 3H).
- Grad-CAM36 을 사용하여 히트맵으로 표시되는 ResNeSt의 추론에 가장 많이 기여하는 영역을 강조 표시합니다. 히트맵을 이진화하여(임계값, 예: 최대 10% × 사용) 예측된 CM 커밋 CPC 영역(IR-CPC(imaged-recognized CPC) 영역)을 가져옵니다.
- 정확도, F1 점수, 정밀도, 재현율, 특이성 및 IoU(Intersection-over-Union)를 사용하여 테스트 세트에서 IR-CPC 영역을 수동으로 주석이 달린 마스크와 비교합니다. Pearson 상관 계수를 사용하여 IR-CPC 영역의 비율을 Differentiation Efficiency Indexes(최종 cTnT 형광 이미지에서 계산)와 비교합니다. 성능 평가를 위해 "./Evaluation/index_calculate.m" 및 "./Evaluation/Pearson_correlation.m"을 실행합니다(그림 3I).
- 훈련된 모델을 적용하여 스테이지 II에서 새 배치에 대한 CPC를 인식합니다. 전체-웰 명시야 이미지를 패치로 자르고, 패치를 훈련된 ResNeSt에 전달하고, 패치 수준에서 IR-CPC 영역을 얻은 다음, 이를 병합하여 전체-웰 IR-CPC 영역을 생성합니다. 최종 미분 효율의 지표로 IR-CPC 영역의 분율을 계산합니다. IR-CPC를 정제하여 세포 오염을 줄입니다(아래 설명 참조).
참고: CPC 인식 모델은 최종 분화 효율의 조기 평가를 할 수 있기 때문에, 분화 과정에 도움이 되는 소분자를 스크리닝하기 위한 고처리량 판독 역할도 할 수 있으며, 이는 분화 프로토콜(35)을 더욱 최적화할 수 있습니다.
- 이미징 인식된 CPC의 지역 선택적 정제
참고: 광활성화 실험은 전동 스테이지가 있는 도립 형광 현미경을 기반으로 합니다.- 훈련된 모델에 의해 예측된 IR-CPC 영역에 따라 IR-CPC를 생성할 수 없는 웰을 버립니다. 이러한 CM 투입 CPC를 정제하려면 비세포독성 광활성화 프로브인 DACT-1(Dual-Activatable Cell Tracker 1)을 사용하여 비CPC39를 지역 선택적으로 라벨링합니다.
- DACT-1(10mM)을 DMSO에 용해시키고 -20°C에서 보관합니다. 37°C에서 30분 동안 CM 분화 배지에 1μM DACT-1로 6일차 세포를 배양합니다.
- 현미경으로 살아있는 세포를 관찰하고 MATLAB에서 관심 영역(ROI)을 다각형으로 선택합니다. 다각형 내부에 20μm 간격의 평행선을 그리고 교차점의 좌표를 계산합니다.
- 트레이스 라인에 따라 전동 스테이지의 이동 트랙을 설정하면 스테이지가 0.12mm/s로 이동하여 스테이지에 조립된 405nm 레이저 라인(직경 20μm)이 ROI를 전역적으로 스캔하여 비 CPC를 제한적으로 광활성화할 수 있습니다. 방사선 조사 후 561nm 레이저 라인을 사용하여 DACT-1 표지된 세포를 검출합니다.
- 37°C에서 5-7분 동안 0.05% Trypsin-EDTA(PBS로 희석)를 사용하여 세포를 해리하고 인큐베이터에서 2분 동안 플레이트를 부드럽게 흔듭니다. 40μm 셀 스트레이너와 500× g 의 원심분리기를 통해 3분 동안 세포를 여과합니다. 0.5% BSA에 세포를 재현탁시키고 분류할 때까지 튜브를 얼음 위에 두십시오. 또한 분류를 위한 음성 대조군으로 방사선 조사되지 않은 샘플을 준비합니다.
- 유세포 분석 분류 시스템을 시작하고 분류기 소프트웨어를 설정합니다. 561nm 파장 레이저를 선택하고 DACT1+ 분류를 위해 610/20의 대역 통과 필터를 사용하십시오. 각 샘플에 대해 다각형 게이팅 도구를 사용하여 단일 세포를 선택하기 위한 전방 산란(FSC) 및 측면 산란(SSC)의 이변량 플롯에서 세포 집단에 원을 그립니다. 그런 다음 negative control을 실행하여 배경 형광을 확인하고, region-selectively irradiated sample을 실행하고, DACT1+ population에 대한 게이트를 그립니다.
- 10% FBS 및 5μM Y27632가 있는 CM 유지 매체에서 분류된 세포를 수집 및 재현탁하고 Matrigel 코팅된 96웰 플레이트에서 분류된 세포를 시드합니다.
- 다음날 배지를 CM 유지 매체로 변경하고 고순도 CM을 수확할 때까지 기다립니다.
- 3단계 ML 전략: CM 인식
데이터 세트: 12일째의 명시야 이미지 쌍과 최종 cTnT 형광 이미지
모델: pix2pix37- CM의 명시야 이미지와 해당 cTnT 형광 이미지로 구성된 데이터 세트를 준비합니다. 다양성을 높이려면 데이터 세트에 다양한 분화 효율과 세포주를 가진 웰이 포함되어 있는지 확인하십시오.
- 데이터 세트를 학습 세트와 테스트 세트로 무작위로 나눕니다. 작업 디렉토리를 "stage_III"로 전환합니다. 명시야 이미지와 해당 cTnT 형광 이미지를 각각 "./pix2pix/datasets/(CM|CM_new_cell_lines)/A/(train|test)/*.png" 및 ".../B/(train|test)/*.png"에 저장합니다. 다음 명령을 실행합니다.
cd pix2pix/데이터셋
파이썬 combine_A_and_B.py --fold_A ./CM/A --fold_B ./CM/B --fold_AB ./CM/ --no_multiprocessing
시디.. - 훈련 세트에서 pix2pix 모델37 을 훈련시킵니다. 다음 명령을 실행합니다(그림 3J).
파이썬 train.py --dataroot ./datasets/CM --name brightfield2fluorescence --model pix2pix --input_nc 1 --output_nc 1 --load_size 1536 --crop_size 256 --lr 2e-4 --n_epochs 1000 --n_epochs_decay 0 --norm 인스턴스 --netD n_layers --n_layers_D 1 --batch_size 16 --방향 AtoB --save_epoch_freq 100 --dataset_mode 정렬 --use_resize_conv --seed 1234 - 훈련된 pix2pix 모델을 테스트 세트에 적용합니다. 다음 명령을 실행합니다.
파이썬 test.py --dataroot ./datasets/CM --name brightfield2fluorescence --model pix2pix --방향 AtoB --input_nc 1 --output_nc 1 --load_size 1536 --crop_size 1536 --use_resize_conv --eval --num_test 1000 - 모델에 의해 예측된 형광 이미지를 실제로(즉, 실험적으로 얻어진) 형광 이미지와 비교합니다. 픽셀 수준에서는 Pearson 상관 계수를 사용하여 예측된 농도와 실제 농도를 비교합니다. 또한 이미지 수준에서 Pearson 상관 계수를 사용하여 예측된 차별화 효율 지수를 실제 지수와 비교합니다. 이러한 비교를 위해 "stage_III/evaluation/pixel_correlation.m" 및 "image_correlation.ipynb"를 실행합니다(그림 3K,L).
- 훈련된 모델을 적용하여 III단계에서 새로운 배치의 CM을 인식합니다. CM의 새로운 명시야 이미지를 훈련된 pix2pix 모델에 전달하고 형광 이미지 및 분화 효율성에 대한 인실리코(in-silico ) 예측을 얻을 수 있습니다.
참고: 세포 오염을 줄이기 위해 DACT-1 기반 광활성화 세포 분류는 모델에서 인식한 CM을 정제하는 데에도 적용할 수 있습니다.
Access restricted. Please log in or start a trial to view this content.
결과
명시야 이미징 및 ML을 기반으로 전체 차별화 프로세스를 지능적으로 모니터링하고 최적화할 수 있습니다. PSC 단계에서는 초기 PSC 콜로니의 형태학적 특징에 따라 최종 분화 효율을 예측할 수 있는 ML 모델을 개발하여 분화를 시작하기에 가장 적합하거나 적절한 시점을 결정했습니다(그림 4A, B). 랜덤 포레스트 모델에 의해 예측된 분화 ?...
Access restricted. Please log in or start a trial to view this content.
토론
여기에서는 현재 PSC 응용 및 변환의 주요 문제 중 하나인 세포 분화의 가변성을 극복하기 위한 자세한 프로토콜에 대해 설명했습니다. 살아있는 세포 명시야 이미징 및 ML을 활용하여 PSC 분화를 반복적으로 최적화하여 세포주와 배치 전반에 걸쳐 일관되게 높은 효율성을 달성했습니다. 그러나 위의 차별화 과정에서 프로토콜의 몇 가지 중요한 단계는 차별화의 성공 여부?...
Access restricted. Please log in or start a trial to view this content.
공개
Yang Zhao, Jue Zhang, Xiaochun Yang, Yao Wang 및 Daichao Chen은 본 논문(202210525166.X)에 보고된 PSC 차별화 전략에 대한 특허를 출원하고 있습니다.
감사의 말
이 전략의 토대를 마련해 주신 Qiushi Sun, Yao Wang, Yu Xia, Jinyu Yang, Chang Lin, Zimu Cen, Dongdong Liang, Rong Wei, Ze Xu, Guangyin Xi, Gang Xue, Can Ye, Li-Peng Wang, Peng Zou, Shi-Qiang Wang, Pablo Rivera-Fuentes, Salome Püntener, Zhixing Chen, Yi Liu, Jue Zhang에게 감사드립니다. 이 연구는 중국 국가 핵심 R&D 프로그램(2018YFA0800504, 2019YFA0110000)과 중국 유인 우주 프로그램(HYZHXM01020)의 우주 의료 실험 프로젝트의 지원을 받았습니다. 그림 1 은 BioRender.com 로 작성되었습니다.
Access restricted. Please log in or start a trial to view this content.
자료
| Name | Company | Catalog Number | Comments |
| 0.25% Trypsin-EDTA | Gibco | 25200056 | Diluted digests were used for CPC and CM digestion |
| 4% Paraformaldehyde in PBS | KeyGEN BioTECH | KGIHC016 | |
| 6-well Cell Culture Plate | NEST | 703001 | |
| 96-well Cell Culture Plate | NEST | 701001 | |
| B27 Supplement | Gibco | 17504044 | |
| B27 Supplement Minus Insulin | Gibco | A1895601 | |
| Bovine serum albumin (BSA) | GPC BIOTECH | AA904-100G | |
| Celldiscoverer 7 | Zeiss | Instruments used to take bright-field images throughout differentiation and final cTnT images | |
| CHIR99021 | Selleck | S1263 | |
| DMEM/F12 | Gibco | 12634010 | |
| Donkey anti-Mouse IgG (H+L) Highly Cross-Adsorbed Secondary Antibody, Alexa Fluor 488 | Thermo | A-21202 | Secondary Antibody |
| FACSAria III | BD Biosciences | Flow cytometry sorter | |
| Fetal Bovine Serum (FBS) | VISTECH | SE100-B | |
| Hoechst 33342 | YEASEN | 40732ES03 | |
| Human Pluripotent Stem Cell Chemical-defined Medium | Cauliscell Inc | 400105 | Basal medium of PSC preparation medium |
| iPS-18 | TaKaRa | Y00300 | |
| iPS-B1 | Cellapy | CA4025106 | |
| iPS-F | Nuwacell | RC01001-B | |
| iPS-M | Nuwacell | RC01001-A | |
| IWR1-1-endo | Selleck | S7086 | IWR1 |
| Jupyter Notebook | N/A | Version 6.4.0 | https://jupyter.org/ |
| MATLAB | MathWorks | Version R2020a | Software for scientific computation and image annotation |
| Matrigel Matrix | Corning | 354230 | Matrigel |
| Mouse monoclonal IgG1 anti-cTnT | Thermo | MA5-12960 | cTnT primary antibody |
| Normal Donkey Serum | Jackson | 017-000-121 | |
| ORCA-Flash 4.0 V3 digital CMOS camera | Hamamatsu | C13440-20CU | The digital camera assembled on Celldiscoverer7 |
| PBS | NEB | 21-040-CVR | |
| Penicillin-Streptomycin | Gibco | 15140-122 | |
| Pluripotency Growth Mater 1 basal medium | Cellapy | CA1007500-1 | Basal medium of PSC culture medium |
| Pluripotency Growth Mater 1 supplement | Cellapy | CA1007500-2 | Supplement of PSC culture medium |
| Prism | Graphpad | Version 8/9 | Statistical software for statistical analysis and plotting |
| Python | N/A | version 3.6 | Python 3 environment for scientific computation, with packages pytorch (1.9.0), numpy, scipy, pandas, visdom, scikit-learn, scikit-image, opencv-python, and matplotlib software for scientific computation and image annotation. |
| RPMI 1640 | Gibco | 11875176 | |
| Supplement hPSC-CDM (500x) | Cauliscell Inc | 00015 | Supplement of PSC preparation medium |
| TiE | Nikon | An inverted fluorescence microscope (with modification) for region-selevtive purification | |
| Triton X-100 | Amresco | 9002-93-1 | |
| Versene Solution | Thermo | 15040066 | EDTA solution for PSC digestion |
| Y27632 | Selleck | S6390 | |
| Zen | Zeiss | Version 3.1 | A supporting software of Celldiscoverer7 for image acquisition, processing and analysis |
참고문헌
- Yoshida, Y., Yamanaka, S. Induced pluripotent stem cells 10 years later: for cardiac applications. Circ Res. 120 (12), 1958-1968 (2017).
- Shi, Y., Inoue, H., Wu, J. C., Yamanaka, S. Induced pluripotent stem cell technology: a decade of progress. Nat Rev Drug Discov. 16 (2), 115-130 (2017).
- Takahashi, K., et al. Induction of pluripotent stem cells from adult human fibroblasts by defined factors. Cell. 131 (5), 861-872 (2007).
- Burridge, P. W., et al. Chemically defined generation of human cardiomyocytes. Nat Methods. 11 (8), 855-860 (2014).
- Mordwinkin, N. M., Burridge, P. W., Wu, J. C. A review of human pluripotent stem cell-derived cardiomyocytes for high-throughput drug discovery, cardiotoxicity screening, and publication standards. J Cardiovasc Transl Res. 6 (1), 22-30 (2013).
- Vanden Berg, C. W., Elliott, D. A., Braam, S. R., Mummery, C. L., Davis, R. P. Differentiation of human pluripotent stem cells to cardiomyocytes under defined conditions. Methods Mol Biol. 1353, 163-180 (2016).
- Burridge, P. W., Holmström, A., Wu, J. C. Chemically defined culture and cardiomyocyte differentiation of human pluripotent stem cells. Curr Protoc Hum Genet. 87, 1-15 (2015).
- Li, J., et al. Human pluripotent stem cell-derived cardiac tissue-like constructs for repairing the infarcted myocardium. Stem Cell Rep. 9 (5), 1546-1559 (2017).
- Smith, A. S. T., Macadangdang, J., Leung, W., Laflamme, M. A., Kim, D. -H. Human iPSC-derived cardiomyocytes and tissue engineering strategies for disease modeling and drug screening. Biotechnol Adv. 35 (1), 77-94 (2017).
- Sharma, A., et al. Use of human induced pluripotent stem cell-derived cardiomyocytes to assess drug cardiotoxicity. Nat Protoc. 13 (12), 3018-3041 (2018).
- Mehta, A., et al. Identification of a targeted and testable antiarrhythmic therapy for long-QT syndrome type 2 using a patient-specific cellular model. Eur Heart J. 39 (16), 1446-1455 (2018).
- Cahan, P., Daley, G. Q. Origins and implications of pluripotent stem cell variability and heterogeneity. Nat Rev Mol Cell Biol. 14 (6), 357-368 (2013).
- Kim, K., et al. Epigenetic memory in induced pluripotent stem cells. Nature. 467 (7313), 285-290 (2010).
- Rouhani, F., et al. Genetic background drives transcriptional variation in human induced pluripotent stem cells. PLoS Genet. 10 (6), e1004432(2014).
- Kim, K., et al. Donor cell type can influence the epigenome and differentiation potential of human induced pluripotent stem cells. Nat Biotechnol. 29 (12), 1117-1119 (2011).
- Garitaonandia, I., et al. Increased risk of genetic and epigenetic instability in human embryonic stem cells associated with specific culture conditions. PLoS One. 10 (2), e0118307(2015).
- Engler, A. J., Sen, S., Sweeney, H. L., Discher, D. E. Matrix elasticity directs stem cell lineage specification. Cell. 126 (4), 677-689 (2006).
- Matoba, N., Yamashita, T., Takayama, K., Sakurai, F., Mizuguchi, H. Optimal human iPS cell culture method for efficient hepatic differentiation. Differentiation. 104, 13-21 (2018).
- Volpato, V., et al. Reproducibility of molecular phenotypes after long-term differentiation to human iPSC-derived neurons: A multi-site omics study. Stem Cell Rep. 11 (4), 897-911 (2018).
- Anderson, N. C., et al. Balancing serendipity and reproducibility: Pluripotent stem cells as experimental systems for intellectual and developmental disorders. Stem Cell Rep. 16 (6), 1446-1457 (2021).
- Sepac, A., et al. Comparison of cardiomyogenic potential among human ESC and iPSC lines. Cell Transplant. 21 (11), 2523-2530 (2012).
- Laco, F., et al. Unraveling the inconsistencies of cardiac differentiation efficiency induced by the GSK3β inhibitor CHIR99021 in human pluripotent stem cells. Stem Cell Rep. 10 (6), 1851-1866 (2018).
- Volpato, V., Webber, C. Addressing variability in iPSC-derived models of human disease: guidelines to promote reproducibility. Dis Model Mech. 13 (1), 042317(2020).
- Hu, B. -Y., et al. Neural differentiation of human induced pluripotent stem cells follows developmental principles but with variable potency. Proc Natl Acad Sci USA. 107 (9), 4335-4340 (2010).
- Huo, J., et al. Evaluation of batch variations in induced pluripotent stem cell-derived human cardiomyocytes from 2 major suppliers. Toxicol Sci. 156 (1), 25-38 (2017).
- Rostam, H. M., Reynolds, P. M., Alexander, M. R., Gadegaard, N., Ghaemmaghami, A. M. Image based machine learning for identification of macrophage subsets. Sci Rep. 7 (1), 3521(2017).
- Christiansen, E. M., et al. In silico labeling: Predicting fluorescent labels in unlabeled images. Cell. 173 (3), 792-803 (2018).
- Buggenthin, F., et al. Prospective identification of hematopoietic lineage choice by deep learning. Nat Methods. 14 (4), 403-406 (2017).
- Smith, Z. D., Nachman, I., Regev, A., Meissner, A. Dynamic single-cell imaging of direct reprogramming reveals an early specifying event. Nat Biotechnol. 28 (5), 521-526 (2010).
- Fan, K., Zhang, S., Zhang, Y., Lu, J., Holcombe, M., Zhang, X. A machine learning assisted, label-free, non-invasive approach for somatic reprogramming in induced pluripotent stem cell colony formation detection and prediction. Sci Rep. 7 (1), 13496(2017).
- Kusumoto, D., et al. Automated deep learning-based system to identify endothelial cells derived from induced pluripotent stem cells. Stem Cell Rep. 10 (6), 1687-1695 (2018).
- Zhu, Y., et al. Deep learning-based predictive identification of neural stem cell differentiation. Nat Commun. 12 (1), 2614(2021).
- Qian, T., et al. Label-free imaging for quality control of cardiomyocyte differentiation. Nat Commun. 12 (1), 4580(2021).
- Ounkomol, C., Seshamani, S., Maleckar, M. M., Collman, F., Johnson, G. R. Label-free prediction of three-dimensional fluorescence images from transmitted-light microscopy. Nat Methods. 15 (11), 917-920 (2018).
- Yang, X., et al. A live-cell image-based machine learning strategy for reducing variability in PSC differentiation systems. Cell Discovery. 9 (1), 53(2023).
- Selvaraju, R. R., et al. Grad-CAM: Visual explanations from deep networks via gradient-based localization. Proc IEEE Int Conf Computer Vision (ICCV). , 618-626 (2017).
- Isola, P., Zhu, J. -Y., Zhou, T., Efros, A. A. Image-to-image translation with conditional adversarial networks. Proc IEEE Conf Computer Vision Pattern Recognition (CVPR). , 5967-5976 (2017).
- Zhang, H., et al. ResNeSt: Split-attention networks. Proc IEEE/CVF Conf Computer Vision Pattern Recognition Workshops (CVPRW). , 2735-2745 (2022).
- Halabi, E. A., et al. Dual-activatable cell tracker for controlled and prolonged single-cell labeling). ACS Chem. Biol. 15 (6), 1613-1620 (2020).
- Hannan, N. R. F., Segeritz, C. -P., Touboul, T., Vallier, L. Production of hepatocyte-like cells from human pluripotent stem cells. Nat Protoc. 8 (2), 430-437 (2013).
- Zhang, H., Shen, M., Wu, J. C. Generation of quiescent cardiac fibroblasts derived from human induced pluripotent stem cells. Methods Mol Biol. 2454, 109-115 (2022).
- Harding, M. J., McGraw, H. F., Nechiporuk, A. The roles and regulation of multicellular rosette structures during morphogenesis. Development. 141 (13), 2549-2558 (2014).
- Najm, F. J., et al. Rapid and robust generation of functional oligodendrocyte progenitor cells from epiblast stem cells. Nat Methods. 8 (11), 957-962 (2011).
- Hogrebe, N. J., Maxwell, K. G., Augsornworawat, P., Millman, J. R. Generation of insulin-producing pancreatic β cells from multiple human stem cell lines. Nat Protoc. 16 (9), 4109-4143 (2021).
Access restricted. Please log in or start a trial to view this content.
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유