需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
一种基于活细胞图像的机器学习策略,用于监测多能干细胞分化
* 这些作者具有相同的贡献
摘要
目前的多能干细胞 (PSC) 到功能细胞分化系统目前受到严重的线间和批次间差异问题的阻碍。在这里,以心脏分化为主要示例,我们提出了一种基于基于图像的机器学习智能监测和调节 PSC 分化过程的协议。
摘要
多能干细胞 (PSC) 技术已广泛用于药物发现、疾病建模和再生医学。然而,可用的 PSC 到功能细胞分化系统受到严重的线间和批次间差异问题的阻碍。因此,实时精确控制细胞分化非常重要。在该协议中,我们描述了一种非侵入性和智能策略,它通过使用基于明场图像的机器学习来克服细胞分化的可变性。以 PSC 到心肌细胞的分化为例,该方法为控制初始 PSC 状态、分化条件下的早期评估和干预以及消除错误分化的细胞污染提供了详细信息,共同实现了从 PSC 到功能细胞的始终如一的高质量分化。原则上,该策略可以扩展到其他具有多个步骤的细胞分化或重编程系统,以支持细胞制造,以及进一步了解细胞命运转换过程中的机制。
引言
多能干细胞 (PSC) 具有在体外分化成多种类型细胞的显着能力。这些分化的功能细胞可用于细胞治疗、疾病建模和药物开发,所有这些都对研究或临床应用很有价值 1,2,3。例如,已经开发了多种方法将 PSC 区分为心肌细胞 (CM)4,5,6,7。这些 CM 可应用于药物的心脏毒性测试、心脏病建模和细胞移植 8,9,10,11。
然而,从 PSC 到终末分化细胞的转化是一个逐步的过程,分化过程中的多次扰动可能导致细胞命运不同。PSC 系的不同遗传背景和表观遗传标记会影响分化为特定谱系的潜力 12,13,14,15;PSC 传代次数和累积的基因突变也是 PSC 异质性的来源;不同实验者采用的实验作的差异也会导致完全不同的分化结果 16,17,18,19,20。因此,目前 PSC 衍生细胞生产的主要问题之一是细胞系和批次21、22、23、24、25 之间的不稳定性。PSC 分化的不稳定性通常会导致多次重复实验,消耗大量时间和人力资源。为了解决这个问题,开发一种策略以最大限度地减少细胞系和批次之间的差异,从而提高分化的稳定性和稳健性至关重要。
最近,高分辨率显微镜和机器学习 (ML) 的进步促进了基于 ML 的定量图像分析在细胞生物学中的应用,从而可以在细胞成像特征中利用有价值的信息 26,27,28,29,30,31,32,33,34.在我们之前的工作中,我们提出了一种基于活细胞图像的 ML 策略,以实时监测和干预 PSC 分化状态,以提高 PSC 分化的稳定性和效率(图 1)35。以 PSC 到心肌细胞分化为例,我们使用随机森林模型评估初始 PSC 状态,使用 logistic 回归模型预测最佳分化条件,并使用基于深度学习的 Grad-CAM36 和 pix2pix37 识别成功分化的细胞。ML 模型学会了从一系列明场形态特征中识别细胞谱系,包括面积、周长、凸度、固体、亮度、移动速度等特征,以及深度卷积神经网络提取的其他隐含特征。基于这些已建立的 ML 模型的推断,我们实现了对初始 PSC 状态的控制、分化条件的早期评估和干预,以及消除错误分化的细胞污染,共同提供了对心脏分化过程的全面和准确的调节。在这里,我们提供了制定策略的分步协议。
Access restricted. Please log in or start a trial to view this content.
研究方案
1. 细胞分化和表征
- 培养试剂和培养板的制备
- 向 48 mL 基础培养基中加入 2 mL 补充剂和 0.2% 青霉素-链霉素,制备 PSC 培养基。将补充剂分装并储存在 -20 °C。 将此培养基在 4 °C 下储存长达 4 周。
- 向 500 mL 基础培养基中加入 1 mL 补充剂和 0.2% 青霉素-链霉素,制备 PSC 制备培养基。使用时,预热培养基以供一次性使用,并将培养基在 4 °C 下储存长达 3 周。
- 通过在 RPMI 1640 中加入 1x B27 补充剂(不含胰岛素)和 1% 青霉素-链霉素来制备 CM 分化培养基。通过将 1x B27 补充剂和 1% 青霉素-链霉素添加到 RPMI 1640 中来制备 CM 维持培养基。将这两种培养基在 4 °C 下储存 2 周。
- 将 Matrigel 浸入冰中,并在 4 °C 下保持过夜。确保 Matrigel 完全解冻;然后,分装成 1 mL 等分试样,储存在 -80 °C 以备后用,并在 4 °C 下将 1 mL Matrigel 加入 49 mL DMEM/F12 中,以制备 Matrigel 工作溶液。在 6 孔培养板的每孔中加入 850 μL 这种 Matrigel 工作溶液,并将板在 37 °C 下孵育至少 30 分钟。对于用于 CM 分化的 96 孔培养板,每孔添加 35 μL Matrigel 工作溶液。
注意:始终将 Matrigel 置于冰上;移液器吸头和试管应在 4 °C 下预冷。 Matrigel 工作溶液可在 4 °C 下储存 2 周。移液 Matrigel 工作溶液时要小心避免气泡。
- PSC 维持和传代
- 将 PSC 维持在 6 孔培养板中。在显微镜下确认细胞密度,并准备在汇合度达到 ~70% 时传代(图 2A)。
- 使用 EDTA 消化细胞以进行传代。在 37 °C 水浴中加热 PSC 培养基、PBS 和 EDTA,并在培养基中加入 Y27632 至终浓度为 5 μM。
- 用 1 mL PBS 洗涤 PSC,每孔加入 1 mL EDTA,并将板在 37 °C 培养箱中孵育 3 分钟。
- 在细胞从培养板上分离之前吸出 EDTA,然后使用 1 mL PSC 培养基轻轻冲洗孔底部 3-4 次以重悬细胞。
注:这使我们能够跳过离心步骤并减少传代作中的细胞损伤。 - 从 6 孔培养板的基质胶涂层孔中吸出 DMEM/F12,每孔加入 2 mL 新鲜 PSC 培养基(添加 Y27632)。将细胞悬液以 1:6 至 1:12 的分流比添加到孔中。将板放入 37 °C 和 5% CO2 的培养箱中。
- 传代后 12 至 24 小时更换 PSC 培养基(不含 Y27632);然后,每天更换培养基并检查细胞状态和汇合度。
- PSC 的心脏分化
- 对于 CM 分化,将 PSC 接种到 PSC 制备培养基(添加 Y27632)中的 96 孔培养板中。对于 CM 分化,按照上述相同步骤(步骤 1.2.2-1.2.6)将 PSC 接种到 PSC 制备培养基(添加 Y27632)中的 96 孔培养板中。
注意:在此阶段,可以适当延长 EDTA 处理持续时间(5 分钟或更长时间)以充分解离 PSC,以便克隆在传代后可以更小。我们的研究表明,在同一区域内,较小的菌落有利于分化。 - 第一阶段:当 PSC 达到 80-90% 汇合时,将培养基切换到具有 2-20 μM CHIR99021 (CHIR) 的 CM 分化培养基(图 2B)。介质切换的时间点称为第 0 天。CHIR 处理 24-48 小时后,将培养基更换为新鲜的 CM 分化培养基。
注意:CHIR 是一种用于中胚层诱导的 GSK3β 抑制剂。CM 分化的最佳 CHIR 剂量随不同的细胞系和批次而波动。例如,iPS-B1 的最佳 CHIR 剂量为 8-10 μM,48 小时。为了收集不同的明场图像来训练 ML 模型,我们在这里滴定了每批实验的 CHIR 浓度,以获得不同的差异化效率。 - 第二阶段:在 72 小时(第 3 天),用补充有 5 μM IWR1 的 CM 分化培养基替换培养基并培养 48 小时(第 5 天)。用 CM 分化培养基更换培养基 1-2 天(至第 6-7 天)。此时,PSC 分化为心脏祖细胞 (CPC)。
- III 期:用 CM 维持培养基更换培养基,每 3 天更换一次培养基。在第 10 天或第 12 天,收获 CM 以进行进一步分析。
- 对于 CM 分化,将 PSC 接种到 PSC 制备培养基(添加 Y27632)中的 96 孔培养板中。对于 CM 分化,按照上述相同步骤(步骤 1.2.2-1.2.6)将 PSC 接种到 PSC 制备培养基(添加 Y27632)中的 96 孔培养板中。
- CM 的免疫荧光染色
- 在第 10 天或第 12 天,用 PBS 洗涤细胞,并在室温下用 4% 多聚甲醛的 PBS 溶液固定 15 分钟。用 PBS 洗涤 CMs 2 次,并将板在 4 °C 下储存长达 ~1 周。
注意:多聚甲醛是一种有毒试剂,具有刺激性气味。在化学罩中作,保护眼睛和手部。 - 染色时,在室温下用透化溶液(PBS 中的 0.1% Triton X-100)处理细胞 15 分钟;然后,在室温下将细胞在封闭液(0.1% Triton X-100 含 3% 驴血清的 PBS 溶液)中孵育 30 分钟。
- 将样品与用封闭溶液稀释的心肌肌钙蛋白 T (cTnT) 一抗 (1:300) 在 4 °C 下孵育过夜,以鉴定 CM。
- 收集一抗并用 PBS 3x 洗涤细胞。将样品与含 1% 牛血清白蛋白的 PBS 中的二抗在 37 °C 下在黑暗环境中孵育 1 小时。
注:收集的一抗可在 1 周内重复使用 2-3 次。
- 收集一抗并用 PBS 3x 洗涤细胞。将样品与含 1% 牛血清白蛋白的 PBS 中的二抗在 37 °C 下在黑暗环境中孵育 1 小时。
- 从细胞中取出二抗,并用 PBS 洗涤细胞 3 次。使用 Hoechst 33342(PBS 中 1:1,000)在室温下对细胞核染色 5 分钟。使用 PBS 冲洗细胞 3 次,每孔加入 100 μL PBS 以避免干燥。将板在 4 °C 下储存在黑暗处,并在 1 周内获得荧光图像。
- 在第 10 天或第 12 天,用 PBS 洗涤细胞,并在室温下用 4% 多聚甲醛的 PBS 溶液固定 15 分钟。用 PBS 洗涤 CMs 2 次,并将板在 4 °C 下储存长达 ~1 周。
2. 整个分化过程中的图像流采集
- 用于收集图像的实验设计设置
- 使用支持活细胞培养和成像的自动显微镜收集 CM 分化不同阶段的明场图像和分化结果的 cTnT 免疫荧光图像(图 2A-E)。使用显微镜支持软件设计实验程序并控制显微镜。
- 打开软件并创建新的实验设计。选择一个 5 倍物镜和一个 2 倍镜筒透镜进行成像。检查 Tiles 和 Z-stack 选项。
- 在 Channels 菜单中添加用于明场成像的 TL Brightfield 通道。添加 AF488 和 H3342 通道用于免疫荧光成像。修改成像设置菜单中的光路,以便使用外部数码 CMOS 相机进行成像。
- 打开 Acquisition Mode 菜单,并设置 2 x 2 像素合并以提高信噪比。
- 打开 Z-stack 菜单,然后选择 Center 模型。设置扫描时的切片数和间隔。选择 3-5 个切片,间隔为 3-6 μm,用于分析中的聚焦图像。
- 在导航和磁贴窗口中,按载体设置磁贴区域,并为一个井设置25个磁贴(5列x 5行)。在 Tiles 菜单中,选择 Sample Carrier 中的 Multiwell 96 模板,并在相邻区域上设置 5%-15% 的重叠。
- 如果需要连续成像,请选中 Time series 选项,并在 Time series 菜单中设置持续时间和间隔。要在阶段 I 收集图像流,请设置 12 小时的持续时间,不带间隔。
- 获取明场和免疫荧光图像
- 要获取图像,首先将细胞培养板放入样品盘中,然后将样品装入显微镜内。选择 Multiwell 96 模板以匹配实验设计。如果样品由活细胞组成,请打开加热系统和 CO2 泵以保持适当的培养条件(37 °C,5% CO2)。
- 打开预设的实验项目并将其另存为新项目。打开 Tiles 菜单并手动校准板的位置。在导航和瓦片窗口中,选择所需的井,然后点击创建以构建这些井的瓦片区域。
- 单击 Tiles 菜单中的 Verify Tile Regions,然后运行 Autofocus 以验证所有孔。然后,通过滚动鼠标滚轮手动校正明场下每个井的聚焦。
- 勾选所需的频道并在 Channels 菜单中设置曝光时间。建议的曝光时间为 Bright 的 1.625 毫秒、H3342 的 100 毫秒和 AF488 的 550 毫秒。执行一次捕捉以检查所有通道的图像。
- 最后,单击 Start experiment 按钮,然后等待自动成像。通常,完成整个 96 孔培养板的扫描需要 ~1.2 小时(明场下 96 孔,三层,每个孔 25 个平铺)。在 Processing (处理) 框架中,选择 Image Export (图像导出),选择未压缩的 TIFF 格式或 PNG 格式的文件类型,然后应用。在孔中心使用 9 个瓦片 (3 x 3) 的图像进行进一步分析(图 2A-E)。
3. 在差异化过程的每个阶段建立基于图像的 ML 策略
- 软件准备
- 安装 Python 环境,其中包含 pytorch (1.9.0)、numpy、scipy、scikit-learn、pandas、visdom、scikit-image、opencv-python 和 matplotlib 软件包。
- 安装 Jupyter Notebook。
- 安装 MATLAB。
- 从 https://github.com/zhaoyanglab/ML-for-PSC-differentiation 下载代码和示例数据集。
注意:ML 的性能取决于数据集的质量以及模型和超参数的选择。不保证示例实现在所有情况下都是最佳的。ML 工程师应自定义代码,并根据其特定需求定制模型和超参数。
- PSC 阶段的 ML 策略:控制初始 PSC 集落状态
数据集:0 小时(CHIR 处理前)的明场图像对和最终区分效率
型号: random forest- 准备一个数据集,其中包含 0 小时(CHIR 处理前)的明场图像和最终的 cTnT 荧光图像。为了增加 0 h 明场图像形态学特征的多样性,请确保数据集包括来自不同细胞系的 PSC 集落,并在传代后的时间间隔内开始分化。在第一阶段用最佳 CHIR 条件处理数据集中的孔。
注:为确保用最佳 CHIR 条件处理菌落,同一批次内的孔可以用滴定的 CHIR 剂量处理。数据集中仅包含具有高效 CHIR 条件的井。 - 通过从其 cTnT 荧光图像计算的分化效率指数量化每个孔的分化效率,该指数由(
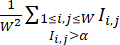 其中
其中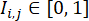 (表示 W × W 荧光图像中像素位置 (i, j) 的荧光强度,并且总和取强度高于阈值 α,通常设置为 0.5)。在每个细胞系中,计算该细胞系中所有孔的最大分化效率指数,并通过最大值对其分化效率指数进行归一化。
(表示 W × W 荧光图像中像素位置 (i, j) 的荧光强度,并且总和取强度高于阈值 α,通常设置为 0.5)。在每个细胞系中,计算该细胞系中所有孔的最大分化效率指数,并通过最大值对其分化效率指数进行归一化。
注意:由于每种细胞系的分化潜力本质上是不同的,因此这种标准化将更加强调各种细胞系之间共享的菌落形态特征,这些特征有助于分化结果。归一化确实使来自不同细胞系的 PSC 集落具有可比性,并减轻了细胞系诱导的偏差。 - 通过揭示菌落形状特性的高维特征量化 0 h 明场图像的形态学特征。要在明场图像中勾勒出菌落区域,请计算局部熵(半径 为 r);局部熵大于阈值的像素 σ由 Covered by Cells 标识。使用 r = 10 像素和 σ = 3 的参数设置。将工作目录切换到 "./colony_control";将明场图像、细胞区域的二元图像和 cTnT 荧光图像保存在"colony_control/ image_data/CD00-*/[brightfield|cell_region|ctnt]/S*.png";并运行 Jupyter Notebook "Compute_features.ipynb" 来计算与菌落面积、周长、坚固性、凸度、圆度和质心-等值线距离等相关的特征。
- 将数据集随机划分为训练集和测试集,训练集占总数据的 50% 到 80%。运行最后一个块 Split the dataset in the Jupyter Notebook 以拆分数据集(图 3A)。
- 在训练集上训练随机森林回归模型,以预测 0 h 明场图像特征的区分效率。运行 Jupyter Notebook "Machine_learning.ipynb" 来训练和测试模型。
- 在测试集上评估经过训练的随机森林模型。使用 Pearson 相关系数确认预测的分化效率与真实分化效率高度相关(图 3B)。
注意:由于测试集不参与训练阶段,因此测试集上的预测效率和真实区分效率之间的高度一致性表明该模型可以很好地泛化到看不见的样本上。 - 从训练好的随机森林模型中获取特征重要性,并选择最重要的 8 个特征。通过将特征范围划分为 bin 并计算每个 bin 中的平均区分效率,可视化它们与最终区分效率的关系。运行 Jupyter Notebook "Feature_importance.ipynb" 以执行可视化(图 3C)。
注意:可视化可能有助于了解 PSC 集落形状的变化如何促进分化。 - 将训练后的模型应用于新批次,以实时监测 PSC 集落状态。每小时捕获一次明场图像,将图像传递给训练好的模型,并获得预测的区分效率。一旦预测的效率超过阈值(例如 50%),就开始差异化过程。如果预测的效率无法达到阈值,则丢弃这些样本。
注:在 PSC 阶段,PSC 集落的分化潜力将先上升,然后下降。实时监测使实验人员能够确定最有利的时间点来启动分化。
- 准备一个数据集,其中包含 0 小时(CHIR 处理前)的明场图像和最终的 cTnT 荧光图像。为了增加 0 h 明场图像形态学特征的多样性,请确保数据集包括来自不同细胞系的 PSC 集落,并在传代后的时间间隔内开始分化。在第一阶段用最佳 CHIR 条件处理数据集中的孔。
- 第一阶段的 ML 策略:早期评估和调整 CHIR 剂量
数据集:0-12 h 的明场图像流对和 CHIR 浓度标签(低/最佳/高)
模型:logistic regression- 准备一个由全阱明场图像流组成的数据集,其中在 0 小时到 12 小时之间每小时采集一次图像。记录每个孔的 CHIR 剂量(即 CHIR 浓度和持续时间的组合)。确保数据集包括具有不同批次和孔的滴定 CHIR 剂量(例如,浓度 = 2、4、6、8、10 和 12 μM;持续时间 = 24、36 和 48 小时)以增加多样性。将工作目录切换到 "./stage_I";将图像保存为 "./data/image/CD01-*/S*/T*.png",其中 CD01-* (CD01-1, CD01-2, CD01-3, CD01-4) 是批次名称,S* (S1, S2, ..., S96) 是井的索引,T*.png (T1.png, T2.png, ..., T10.png) 是图像流不同时间步长井的预处理明场图像。
- 在每个批次中,计算每个孔的 cTnT + 细胞的百分比,其计算方法是将 cTnT + 细胞的数量除以荧光图像中 Hoechst + 细胞的总数。在相同 CHIR 剂量条件下,平行孔中 cTnT + 细胞的平均百分比。将每个孔的 CHIR 剂量条件和 cTnT + 细胞百分比保存在 csv 文件"./data/image/CD01-*/CD01-?_exp_config.csv"中。
- 在每批中,根据以下标准确定每个 CHIR 持续时间下的"低"、"最佳"和"高"CHIR 浓度范围。cTnT + 细胞的平均百分比高于阈值(例如 20%)的 CHIR 浓度被确定为"最佳"。超出最佳范围的 CHIR 浓度被标识为"低"或"高"。在每个 CHIR 持续时间下,根据相应的 CHIR 浓度将孔标记为"低"、"最佳"和"高"。
注意:根据经验,CHIR 浓度与批次内高效井的持续时间之间存在负相关:CHIR 持续时间越高,最佳浓度越低。换句话说,CHIR 浓度的最优性取决于 CHIR 持续时间,因此,孔的标签在不同的 CHIR 持续时间之间有所不同。 - 在每个批次中,在每个 CHIR 持续时间下,计算每个浓度 c 的 ΔCHIR 浓度,以量化其与最佳值的偏差。用 [c1, c2] 表示最佳 CHIR 浓度范围,然后 ΔCHIR 浓度 (c) = c - c1 (如果 c < c1)、0(如果 c1 ≤ c ≤ c2)或 c - c2(如果 c > c2)。
- 提取数据集中图像流的特征,确保它们反映第一阶段细胞的形态变化。要计算有关局部熵、面积、周长、亮度、光流和分形维数的图像特征,请运行
cd ./数据
Python compute_features.py
CD ..
注意:为了突出形态变化,某些特征的值(例如,面积、周长和光流)可以通过它们在图像流中的初始基线值进行归一化。 - 将数据集随机划分为训练集和测试集。运行 Jupyter Notebook "./data/prepare_dataset.ipynb" 以拆分数据集并将数据集组织为数据帧(图 3D)。
- 对于每个 CHIR 持续时间,训练一个逻辑回归模型,以根据训练集上的图像流特征预测 CHIR 浓度标签(低/最佳/高)。使用准确度、精度、召回率、F1 分数和曲线下面积 (AUC) 评估经过训练的 Logistic 回归模型在测试集上的分类性能。运行 Jupyter Notebook "machine_learning.ipynb" 进行训练和测试(图 3E)。
- 评估 CHIR 剂量评估中的模型性能。在测试集中,使用偏差分数(范围从 -1 到 1)合并具有相同 CHIR 浓度的平行孔的预测标签,其中
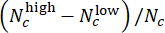 NC 表示浓度为 c 的孔数,
NC 表示浓度为 c 的孔数,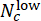
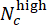 其中孔分别预测为"低"和"高"。使用 Pearson 相关系数确认预测的偏差分数与每个 CHIR 剂量的真实 ΔCHIR 浓度高度相关。
其中孔分别预测为"低"和"高"。使用 Pearson 相关系数确认预测的偏差分数与每个 CHIR 剂量的真实 ΔCHIR 浓度高度相关。 - 执行跨批次验证以测试模型的泛化性能。在每一轮中,留下一个批次进行测试,而使用其他批次进行训练。使用 Pearson 相关系数比较每个测试批次中每个 CHIR 剂量的预测偏差分数和真实 ΔCHIR 浓度。运行 Jupyter Notebook "cross_batch_validation.ipynb" 以实现跨批次验证(图 3F)。
注意:对于选定的 CHIR 持续时间 h,在持续时间 h 下训练的 logistic 回归模型中具有浓度的所有井的预测将转换为偏差分数,反映 CHIR 浓度 c 的最优性。接近 0 的偏差评分表明浓度 c 在持续时间 h 下预测为最佳,而负或阳性偏差评分表明剂量不足或过量,这可以作为以后调整 CHIR 剂量的信号。请注意,由于预测是提前(12 小时)做出的,因此仍然可以调整第 I 阶段剩余时间的最终 CHIR 持续时间和 CHIR 浓度。 - 应用经过训练的 logistic 回归模型来评估新批次中的 CHIR 剂量。对于给定的 CHIR 浓度,计算具有浓度的平行孔的 0-12 小时图像流特征,将特征传递给在不同 CHIR 持续时间下训练的逻辑回归模型,并相应地获得每个孔的预测标签。根据模型的预测,计算偏差分数以评估不同 CHIR 持续时间下的每种 CHIR 浓度。
- 通过基于模型的 CHIR 剂量评估,通过在 48 小时之前将其 CHIR 持续时间或浓度调整为最佳浓度,相应地在每个次优 CHIR 浓度下抢救井。
- 调整持续时间:根据模型对最佳 CHIR 持续时间的预测,调整这些孔中含 CHIR 的培养基切换到 CM 分化培养基的时间点。如果错过了最佳时间点,请丢弃这些样本。
- 调整浓度:在预设的 CHIR 持续时间下,如果预测当前浓度为"低",则直接向培养基中加入高浓度的 CHIR 以提高 CHIR 浓度。需要时用 PBS 在 DMSO (50 mM) 中稀释 CHIR,加入适量液体,然后轻轻摇动板。如果预测当前浓度为"高",则吸出一定量的含 CHIR 的培养基,并补充相同体积的 CM 分化培养基。
注意:在分化的前 48 小时内,细胞会分泌因子以改善细胞生长和分化,因此不要完全改变培养基。此外,在区分中必须保持介质体积恒定。因此,在调整 CHIR 浓度时,请确保添加的液体体积尽可能小。
- 第二阶段的 ML 策略:认可 CM 承诺的 CPC
数据集:第 6 天的明场图像对和 CM 承诺的 CPC 的手动注释掩码
模型:ResNeSt38 和 Grad-CAM36 ,用于弱监督学习- 在第 6 天准备一个由明场图像组成的数据集。通过跟踪从第 12 天到第 6 天的图像流中的 cTnT + 细胞,在明场图像中手动注释 CM 提交的 CPC。
- 将 CM 提交的 CPC 的明场图像和相应的手动注释(称为掩码)裁剪成补丁,每个补丁的大小为整个孔图像大小的 1/5.5。以重叠的方式提取补丁,其中相邻补丁在训练集中共享 50% 的重叠,在测试集中共享 75% 的重叠。将 ≥30% CM 承诺 CPC 的补丁标记为"阳性"。将没有 CM 承诺的 CPC 的补丁标记为"负"。将工作目录切换到 "stage_II"。运行 "./Crop_and_Reconstruct/mask_crop.m" 以裁剪图像。运行"./Crop_and_Reconstruct/classification.m"将标签(0:负;1:正)分配给明场斑块(图 3G)。
- 将标记的图像块随机分为训练集和测试集。
- 训练深度卷积神经网络 ResNeSt38 以学习对这些补丁进行分类。在测试集上评估经过训练的 ResNeSt,以确认模型表现出较高的分类准确性。分别运行 "train.py" 和 "test.py" 进行训练和测试(图 3H)。
- 使用 Grad-CAM36 突出显示对 ResNeSt 推理贡献最大的区域,由热图表示。对热图进行二值化(使用阈值,例如,最大值为 10%),×获取预测的 CM 承诺的 CPC 区域,这些区域被命名为成像识别的 CPC (IR-CPC) 区域。
- 使用准确率、F1 分数、精度、召回率、特异性和交并比 (IoU) 将 IR-CPC 区域与测试集上的手动注释掩码进行比较。使用 Pearson 相关系数将 IR-CPC 区域的比例与分化效率指数(根据最终的 cTnT 荧光图像计算)进行比较。运行 "./Evaluation/index_calculate.m" 和 "./Evaluation/Pearson_correlation.m" 进行性能评估(图 3I)。
- 应用经过训练的模型来识别阶段 II 新批次的 CPC。将全井明场图像裁剪成补丁,将补丁传递给经过训练的 ResNeSt,并获得补丁级别的 IR-CPC 区域,然后将其合并以产生全井 IR-CPC 区域。计算 IR-CPC 区域的分数作为最终分化效率的指标。纯化 IR-CPC 以减少细胞污染(如下所述)。
注:由于 CPC 识别模型可以对最终分化效率进行早期评估,因此它也可以作为筛选有利于分化过程的小分子的高通量读数,这可能会进一步优化分化方案35。
- 成像识别的 CPC 的区域选择性纯化
注意:光活化实验基于由电动载物台辅助的倒置荧光显微镜。- 根据训练模型预测的 IR-CPC 区域,丢弃无法生成任何 IR-CPC 的井。为了纯化这些 CM 定型的 CPC,使用非细胞毒性光激活探针 Dual-Activatable Cell Tracker 1 (DACT-1) 区域选择性标记非 CPC39。
- 将 DACT-1 (10 mM) 溶解在 DMSO 中并储存在 -20 °C。 将第 6 天细胞与 1 μM DACT-1 在 CM 分化培养基中在 37 °C 下孵育 30 分钟。
- 在显微镜下检查活细胞,并在 MATLAB 中选择感兴趣区域 (ROI) 作为多边形。在多边形内绘制间距为 20 μm 的平行线,并计算交点的坐标。
- 根据轨迹线设置电动载物台的移动轨迹,然后载物台以 0.12 mm/s 的速度移动,以便组装在载物台上的 405nm 激光线(直径 20 μm)可以全局扫描 ROI 以限制性地光活化非 CPC。照射后,使用 561 nm 激光线检测 DACT-1 标记的细胞。
- 使用 0.05% 胰蛋白酶-EDTA(经 PBS 稀释)在 37 °C 下解离细胞 5-7 分钟,并在培养箱中轻轻摇动板 2 分钟。通过 40 μm 细胞过滤器过滤细胞,并以 500 × g 离心 3 分钟。将细胞重悬于 0.5% BSA 中,并将试管保持在冰上直至分选。还要准备一个未照射的样品作为分选的阴性对照。
- 启动流式细胞术分选系统并设置分选仪软件。选择 561 nm 波长的激光器,并使用 610/20 的带通滤光片进行 DACT1+ 分选。对于每个样品,使用多边形门控工具在前向散射 (FSC) 和侧向散射 (SSC) 的双变量图中圈出细胞群,以选择单个细胞。然后,运行阴性对照以确认背景荧光,运行区域选择性照射的样品,并为 DACT1 + 群体绘制门。
- 在含有 10% FBS 和 5 μM Y27632 的 CM 维持培养基中收集并重悬分选的细胞,并将分选的细胞接种在基质胶包被的 96 孔板中。
- 第二天,将培养基更换为 CM 维持培养基,并等待收获高纯度 CM。
- 阶段 III 的 ML 策略:识别 CM
数据集:第 12 天的明场图像对和最终的 cTnT 荧光图像
型号: pix2pix37- 准备一个由 CMs 的明场图像和相应的 cTnT 荧光图像组成的数据集。为了增加多样性,请确保数据集包括具有不同分化效率和细胞系的孔。
- 将数据集随机划分为训练集和测试集。将工作目录切换到 "stage_III"。将明场图像和相应的 cTnT 荧光图像分别保存在"./pix2pix/datasets/(CM|CM_new_cell_lines)/A/(train|test)/*.png"和".../B/(train|test)/*.png"。运行以下命令:
cd pix2pix/数据集
python combine_A_and_B.py --fold_A ./CM/A --fold_B ./CM/B --fold_AB ./CM/ --no_multiprocessing
CD .. - 在训练集上训练 pix2pix 模型37 。运行以下命令(图 3J):
python train.py --dataroot ./datasets/CM --name brightfield2fluorescence --model pix2pix --input_nc 1 --output_nc 1 --load_size 1536 --crop_size 256 --lr 2e-4 --n_epochs 1000 --n_epochs_decay 0 --norm 实例 --netD n_layers --n_layers_D 1 --batch_size 16 --方向 AtoB --save_epoch_freq 100 --dataset_mode 对齐 --use_resize_conv --种子 1234 - 将经过训练的 pix2pix 模型应用于测试集。运行以下命令:
python test.py --dataroot ./datasets/CM --name brightfield2fluorescence --model pix2pix --direction AtoB --input_nc 1 --output_nc 1 --load_size 1536 --crop_size 1536 --use_resize_conv --eval --num_test 1000 - 将模型预测的荧光图像与真实(即实验获得的)荧光图像进行比较。在像素级别,使用 Pearson 相关系数比较预测的强度和真实强度。此外,在图像级别,使用 Pearson 相关系数将预测的分化效率指数与真实分化效率指数进行比较。运行"stage_III/evaluation/pixel_correlation.m"和"image_correlation.ipynb"进行这些比较(图 3K,L)。
- 应用经过训练的模型在阶段 III 识别新批次中的 CM。将 CM 的新明场图像传递给经过训练的 pix2pix 模型,并获得荧光图像和区分效率的计算机 模拟 预测。
注:为了减少细胞污染,基于 DACT-1 的光激活细胞分选也适用于纯化模型识别的 CM。
Access restricted. Please log in or start a trial to view this content.
结果
基于明场成像和 ML,可以智能监控和优化整个差异化过程。在 PSC 阶段,我们开发了一个 ML 模型,可以根据初始 PSC 集落的形态学特征预测最终分化效率,以确定开始分化的最合适或合适的时间点(图 4A、B)。随机森林模型预测的分化效率与真正的分化效率高度相关 (Pearson's r = 0.76, P < 0.0001) (图 4B
Access restricted. Please log in or start a trial to view this content.
讨论
在这里,我们描述了一个详细的方案,以克服当前 PSC 应用和翻译中的主要问题之一——细胞分化的可变性。通过利用活细胞明场成像和 ML,我们迭代优化了 PSC 分化,以实现跨细胞系和批次的一致高效率。然而,在上述分化过程中,方案中的几个关键步骤对分化是否成功具有决定性影响。由于 PSC 阶段和 I 期的细胞状态至关重要,因此实验人员需要特别注意早期细胞形态...
Access restricted. Please log in or start a trial to view this content.
披露声明
Yang Zhao、Jue Zhang、Xiaochun Yang、Yao Wang 和 Daichao Chen 正在为本文报道的 PSC 分化策略申请专利 (202210525166.X)。
致谢
我们感谢孙秋诗、王瑶、夏宇、杨金宇、林畅、岑子木、梁冬冬、魏荣、徐泽、习光银、薛刚、叶灿、王立鹏、邹鹏、王世强、Pablo Rivera-Fuentes、Salome Püntener、陈志兴、刘毅和张珏,为这一战略奠定了基础。这项工作得到了国家重点研发计划(2018YFA0800504、2019YFA0110000)和中国载人航天计划空间医学实验项目(HYZHXM01020)对赵阳的支持。 图 1 是使用 BioRender.com 创建的。
Access restricted. Please log in or start a trial to view this content.
材料
| Name | Company | Catalog Number | Comments |
| 0.25% Trypsin-EDTA | Gibco | 25200056 | Diluted digests were used for CPC and CM digestion |
| 4% Paraformaldehyde in PBS | KeyGEN BioTECH | KGIHC016 | |
| 6-well Cell Culture Plate | NEST | 703001 | |
| 96-well Cell Culture Plate | NEST | 701001 | |
| B27 Supplement | Gibco | 17504044 | |
| B27 Supplement Minus Insulin | Gibco | A1895601 | |
| Bovine serum albumin (BSA) | GPC BIOTECH | AA904-100G | |
| Celldiscoverer 7 | Zeiss | Instruments used to take bright-field images throughout differentiation and final cTnT images | |
| CHIR99021 | Selleck | S1263 | |
| DMEM/F12 | Gibco | 12634010 | |
| Donkey anti-Mouse IgG (H+L) Highly Cross-Adsorbed Secondary Antibody, Alexa Fluor 488 | Thermo | A-21202 | Secondary Antibody |
| FACSAria III | BD Biosciences | Flow cytometry sorter | |
| Fetal Bovine Serum (FBS) | VISTECH | SE100-B | |
| Hoechst 33342 | YEASEN | 40732ES03 | |
| Human Pluripotent Stem Cell Chemical-defined Medium | Cauliscell Inc | 400105 | Basal medium of PSC preparation medium |
| iPS-18 | TaKaRa | Y00300 | |
| iPS-B1 | Cellapy | CA4025106 | |
| iPS-F | Nuwacell | RC01001-B | |
| iPS-M | Nuwacell | RC01001-A | |
| IWR1-1-endo | Selleck | S7086 | IWR1 |
| Jupyter Notebook | N/A | Version 6.4.0 | https://jupyter.org/ |
| MATLAB | MathWorks | Version R2020a | Software for scientific computation and image annotation |
| Matrigel Matrix | Corning | 354230 | Matrigel |
| Mouse monoclonal IgG1 anti-cTnT | Thermo | MA5-12960 | cTnT primary antibody |
| Normal Donkey Serum | Jackson | 017-000-121 | |
| ORCA-Flash 4.0 V3 digital CMOS camera | Hamamatsu | C13440-20CU | The digital camera assembled on Celldiscoverer7 |
| PBS | NEB | 21-040-CVR | |
| Penicillin-Streptomycin | Gibco | 15140-122 | |
| Pluripotency Growth Mater 1 basal medium | Cellapy | CA1007500-1 | Basal medium of PSC culture medium |
| Pluripotency Growth Mater 1 supplement | Cellapy | CA1007500-2 | Supplement of PSC culture medium |
| Prism | Graphpad | Version 8/9 | Statistical software for statistical analysis and plotting |
| Python | N/A | version 3.6 | Python 3 environment for scientific computation, with packages pytorch (1.9.0), numpy, scipy, pandas, visdom, scikit-learn, scikit-image, opencv-python, and matplotlib software for scientific computation and image annotation. |
| RPMI 1640 | Gibco | 11875176 | |
| Supplement hPSC-CDM (500x) | Cauliscell Inc | 00015 | Supplement of PSC preparation medium |
| TiE | Nikon | An inverted fluorescence microscope (with modification) for region-selevtive purification | |
| Triton X-100 | Amresco | 9002-93-1 | |
| Versene Solution | Thermo | 15040066 | EDTA solution for PSC digestion |
| Y27632 | Selleck | S6390 | |
| Zen | Zeiss | Version 3.1 | A supporting software of Celldiscoverer7 for image acquisition, processing and analysis |
参考文献
- Yoshida, Y., Yamanaka, S. Induced pluripotent stem cells 10 years later: for cardiac applications. Circ Res. 120 (12), 1958-1968 (2017).
- Shi, Y., Inoue, H., Wu, J. C., Yamanaka, S. Induced pluripotent stem cell technology: a decade of progress. Nat Rev Drug Discov. 16 (2), 115-130 (2017).
- Takahashi, K., et al. Induction of pluripotent stem cells from adult human fibroblasts by defined factors. Cell. 131 (5), 861-872 (2007).
- Burridge, P. W., et al. Chemically defined generation of human cardiomyocytes. Nat Methods. 11 (8), 855-860 (2014).
- Mordwinkin, N. M., Burridge, P. W., Wu, J. C. A review of human pluripotent stem cell-derived cardiomyocytes for high-throughput drug discovery, cardiotoxicity screening, and publication standards. J Cardiovasc Transl Res. 6 (1), 22-30 (2013).
- Vanden Berg, C. W., Elliott, D. A., Braam, S. R., Mummery, C. L., Davis, R. P. Differentiation of human pluripotent stem cells to cardiomyocytes under defined conditions. Methods Mol Biol. 1353, 163-180 (2016).
- Burridge, P. W., Holmström, A., Wu, J. C. Chemically defined culture and cardiomyocyte differentiation of human pluripotent stem cells. Curr Protoc Hum Genet. 87, 1-15 (2015).
- Li, J., et al. Human pluripotent stem cell-derived cardiac tissue-like constructs for repairing the infarcted myocardium. Stem Cell Rep. 9 (5), 1546-1559 (2017).
- Smith, A. S. T., Macadangdang, J., Leung, W., Laflamme, M. A., Kim, D. -H. Human iPSC-derived cardiomyocytes and tissue engineering strategies for disease modeling and drug screening. Biotechnol Adv. 35 (1), 77-94 (2017).
- Sharma, A., et al. Use of human induced pluripotent stem cell-derived cardiomyocytes to assess drug cardiotoxicity. Nat Protoc. 13 (12), 3018-3041 (2018).
- Mehta, A., et al. Identification of a targeted and testable antiarrhythmic therapy for long-QT syndrome type 2 using a patient-specific cellular model. Eur Heart J. 39 (16), 1446-1455 (2018).
- Cahan, P., Daley, G. Q. Origins and implications of pluripotent stem cell variability and heterogeneity. Nat Rev Mol Cell Biol. 14 (6), 357-368 (2013).
- Kim, K., et al. Epigenetic memory in induced pluripotent stem cells. Nature. 467 (7313), 285-290 (2010).
- Rouhani, F., et al. Genetic background drives transcriptional variation in human induced pluripotent stem cells. PLoS Genet. 10 (6), e1004432(2014).
- Kim, K., et al. Donor cell type can influence the epigenome and differentiation potential of human induced pluripotent stem cells. Nat Biotechnol. 29 (12), 1117-1119 (2011).
- Garitaonandia, I., et al. Increased risk of genetic and epigenetic instability in human embryonic stem cells associated with specific culture conditions. PLoS One. 10 (2), e0118307(2015).
- Engler, A. J., Sen, S., Sweeney, H. L., Discher, D. E. Matrix elasticity directs stem cell lineage specification. Cell. 126 (4), 677-689 (2006).
- Matoba, N., Yamashita, T., Takayama, K., Sakurai, F., Mizuguchi, H. Optimal human iPS cell culture method for efficient hepatic differentiation. Differentiation. 104, 13-21 (2018).
- Volpato, V., et al. Reproducibility of molecular phenotypes after long-term differentiation to human iPSC-derived neurons: A multi-site omics study. Stem Cell Rep. 11 (4), 897-911 (2018).
- Anderson, N. C., et al. Balancing serendipity and reproducibility: Pluripotent stem cells as experimental systems for intellectual and developmental disorders. Stem Cell Rep. 16 (6), 1446-1457 (2021).
- Sepac, A., et al. Comparison of cardiomyogenic potential among human ESC and iPSC lines. Cell Transplant. 21 (11), 2523-2530 (2012).
- Laco, F., et al. Unraveling the inconsistencies of cardiac differentiation efficiency induced by the GSK3β inhibitor CHIR99021 in human pluripotent stem cells. Stem Cell Rep. 10 (6), 1851-1866 (2018).
- Volpato, V., Webber, C. Addressing variability in iPSC-derived models of human disease: guidelines to promote reproducibility. Dis Model Mech. 13 (1), 042317(2020).
- Hu, B. -Y., et al. Neural differentiation of human induced pluripotent stem cells follows developmental principles but with variable potency. Proc Natl Acad Sci USA. 107 (9), 4335-4340 (2010).
- Huo, J., et al. Evaluation of batch variations in induced pluripotent stem cell-derived human cardiomyocytes from 2 major suppliers. Toxicol Sci. 156 (1), 25-38 (2017).
- Rostam, H. M., Reynolds, P. M., Alexander, M. R., Gadegaard, N., Ghaemmaghami, A. M. Image based machine learning for identification of macrophage subsets. Sci Rep. 7 (1), 3521(2017).
- Christiansen, E. M., et al. In silico labeling: Predicting fluorescent labels in unlabeled images. Cell. 173 (3), 792-803 (2018).
- Buggenthin, F., et al. Prospective identification of hematopoietic lineage choice by deep learning. Nat Methods. 14 (4), 403-406 (2017).
- Smith, Z. D., Nachman, I., Regev, A., Meissner, A. Dynamic single-cell imaging of direct reprogramming reveals an early specifying event. Nat Biotechnol. 28 (5), 521-526 (2010).
- Fan, K., Zhang, S., Zhang, Y., Lu, J., Holcombe, M., Zhang, X. A machine learning assisted, label-free, non-invasive approach for somatic reprogramming in induced pluripotent stem cell colony formation detection and prediction. Sci Rep. 7 (1), 13496(2017).
- Kusumoto, D., et al. Automated deep learning-based system to identify endothelial cells derived from induced pluripotent stem cells. Stem Cell Rep. 10 (6), 1687-1695 (2018).
- Zhu, Y., et al. Deep learning-based predictive identification of neural stem cell differentiation. Nat Commun. 12 (1), 2614(2021).
- Qian, T., et al. Label-free imaging for quality control of cardiomyocyte differentiation. Nat Commun. 12 (1), 4580(2021).
- Ounkomol, C., Seshamani, S., Maleckar, M. M., Collman, F., Johnson, G. R. Label-free prediction of three-dimensional fluorescence images from transmitted-light microscopy. Nat Methods. 15 (11), 917-920 (2018).
- Yang, X., et al. A live-cell image-based machine learning strategy for reducing variability in PSC differentiation systems. Cell Discovery. 9 (1), 53(2023).
- Selvaraju, R. R., et al. Grad-CAM: Visual explanations from deep networks via gradient-based localization. Proc IEEE Int Conf Computer Vision (ICCV). , 618-626 (2017).
- Isola, P., Zhu, J. -Y., Zhou, T., Efros, A. A. Image-to-image translation with conditional adversarial networks. Proc IEEE Conf Computer Vision Pattern Recognition (CVPR). , 5967-5976 (2017).
- Zhang, H., et al. ResNeSt: Split-attention networks. Proc IEEE/CVF Conf Computer Vision Pattern Recognition Workshops (CVPRW). , 2735-2745 (2022).
- Halabi, E. A., et al. Dual-activatable cell tracker for controlled and prolonged single-cell labeling). ACS Chem. Biol. 15 (6), 1613-1620 (2020).
- Hannan, N. R. F., Segeritz, C. -P., Touboul, T., Vallier, L. Production of hepatocyte-like cells from human pluripotent stem cells. Nat Protoc. 8 (2), 430-437 (2013).
- Zhang, H., Shen, M., Wu, J. C. Generation of quiescent cardiac fibroblasts derived from human induced pluripotent stem cells. Methods Mol Biol. 2454, 109-115 (2022).
- Harding, M. J., McGraw, H. F., Nechiporuk, A. The roles and regulation of multicellular rosette structures during morphogenesis. Development. 141 (13), 2549-2558 (2014).
- Najm, F. J., et al. Rapid and robust generation of functional oligodendrocyte progenitor cells from epiblast stem cells. Nat Methods. 8 (11), 957-962 (2011).
- Hogrebe, N. J., Maxwell, K. G., Augsornworawat, P., Millman, J. R. Generation of insulin-producing pancreatic β cells from multiple human stem cell lines. Nat Protoc. 16 (9), 4109-4143 (2021).
Access restricted. Please log in or start a trial to view this content.
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。