Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Eine auf Lebendzellen basierende Strategie für maschinelles Lernen zur Überwachung der Differenzierung pluripotenter Stammzellen
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Die verfügbaren Systeme zur Differenzierung von pluripotenten Stammzellen (PSC) zu funktionellen Zellen werden derzeit durch Probleme mit starker Variabilität von Linie zu Linie und von Charge zu Charge behindert. Hier stellen wir am Beispiel der kardialen Differenzierung ein Protokoll vor, um den Prozess der PSC-Differenzierung auf Basis von bildbasiertem maschinellem Lernen intelligent zu überwachen und zu modulieren.
Zusammenfassung
Technologien für pluripotente Stammzellen (PSC) sind in der Arzneimittelforschung, in der Krankheitsmodellierung und in der regenerativen Medizin weit verbreitet. Verfügbare PSC-zu-funktionelle Zelldifferenzierungssysteme werden jedoch durch Probleme mit starker Variabilität von Linie zu Linie und von Charge zu Charge behindert. Eine präzise Steuerung der Zelldifferenzierung in Echtzeit ist daher wichtig. In diesem Protokoll beschreiben wir eine nicht-invasive und intelligente Strategie, die die Variabilität in der Zelldifferenzierung durch den Einsatz von bildbasiertem maschinellem Lernen überwindet. Am Beispiel der PSC-zu-Kardiomyozyten-Differenzierung liefert diese Methodik detaillierte Informationen für die Kontrolle des PSC-Anfangszustands, die frühzeitige Beurteilung und Intervention in Differenzierungsbedingungen sowie die Eliminierung der falsch differenzierten Zellkontamination, wodurch eine gleichbleibend hochwertige Differenzierung von PSCs zu funktionellen Zellen realisiert wird. Prinzipiell kann diese Strategie auf andere Zelldifferenzierungs- oder Reprogrammierungssysteme mit mehreren Schritten ausgeweitet werden, um die Zellherstellung zu unterstützen und unser Verständnis der Mechanismen während der Zellschicksalsumwandlung zu verbessern.
Einleitung
Pluripotente Stammzellen (PSCs) besitzen die bemerkenswerte Fähigkeit, sich in vitro in viele Zelltypen zu differenzieren. Diese differenzierten funktionellen Zellen könnten für die Zelltherapie, die Modellierung von Krankheiten und die Entwicklung von Medikamenten verwendet werden, die alle für die Forschung oder klinische Anwendungen wertvoll sind 1,2,3. So wurde beispielsweise eine Vielzahl von Methoden entwickelt, um PSCs in Kardiomyozyten (CMs) zu differenzieren4,5,6,7. Diese CMs können für Kardiotoxizitätstests von Arzneimitteln, die Modellierung von Herzerkrankungen und die Zelltransplantation eingesetzt werden 8,9,10,11.
Die Umwandlung von PSC in die terminalen differenzierten Zellen ist jedoch ein schrittweiser Prozess, und multiple Störungen während des Differenzierungsprozesses können zu unterschiedlichen Zellschicksalen führen. Unterschiedliche genetische Hintergründe und epigenetische Markierungen von PSC-Linien beeinflussen das Potenzial zur Differenzierung zu einer bestimmten Linie 12,13,14,15; die Anzahl der PSC-Passagen und akkumulierte Genmutationen sind ebenfalls Ursachen für die PSC-Heterogenität; Unterschiede in den experimentellen Abläufen, die von verschiedenen Experimentatoren angewandt werden, können auch zu völlig unterschiedlichen Differenzierungsergebnissen führen 16,17,18,19,20. Daher ist derzeit eines der Hauptprobleme bei der Herstellung von PSC-abgeleiteten Zellen die Instabilität zwischen den Zelllinien und -chargen 21,22,23,24,25. Instabilität bei der PSC-Differenzierung führt oft zu mehreren wiederholten Experimenten, die viel Zeit und Arbeitsressourcen in Anspruch nehmen. Um dieses Problem anzugehen, ist es entscheidend, eine Strategie zu entwickeln, die die Variabilität zwischen Zelllinien und Chargen minimiert und so die Stabilität und Robustheit der Differenzierung erhöht.
In jüngster Zeit haben Fortschritte in der hochauflösenden Mikroskopie und im maschinellen Lernen (ML) die Anwendung der ML-basierten quantitativen Bildanalyse in der Zellbiologie erleichtert und es ermöglicht, wertvolle Informationen in der Zellbildgebung zu nutzen 26,27,28,29,30,31,32,33,34. In unserer früheren Arbeit haben wir eine auf Lebendzellbildern basierende ML-Strategie vorgeschlagen, um den PSC-Differenzierungsstatus in Echtzeit zu überwachen und in ihn einzugreifen, um die Stabilität und Effizienz der PSC-Differenzierung zu verbessern (Abbildung 1)35. Am Beispiel der PSC-zu-Kardiomyozyten-Differenzierung bewerteten wir den anfänglichen PSC-Zustand mit Random-Forest-Modellen, sagten die optimale Differenzierungsbedingung mit logistischen Regressionsmodellen voraus und erkannten erfolgreich differenzierte Zellen mit den Deep-Learning-basierten Grad-CAM36 und pix2pix37. ML-Modelle lernten, Zelllinien aus einer Reihe von morphologischen Hellfeldmerkmalen zu identifizieren, einschließlich Merkmalen über Fläche, Umfang, Konvexität, Festigkeit, Helligkeit, Bewegungsgeschwindigkeit und andere implizite Merkmale, die von tiefen neuronalen Faltungsnetzen extrahiert wurden. Basierend auf der Schlussfolgerung aus diesen etablierten ML-Modellen konnten wir die Kontrolle des anfänglichen PSC-Zustands, die frühzeitige Bewertung und Intervention in die Differenzierungsbedingungen sowie die Beseitigung der falsch differenzierten Zellkontamination realisieren, was zusammen eine umfassende und genaue Modulation des kardialen Differenzierungsprozesses ermöglichte. Hier stellen wir ein Schritt-für-Schritt-Protokoll für die Entwicklung der Strategie zur Verfügung.
Protokoll
1. Zelldifferenzierung und -charakterisierung
- Vorbereitung von Kulturreagenzien und Kulturplatten
- Bereiten Sie das PSC-Kulturmedium vor, indem Sie 2 ml Nahrungsergänzungsmittel und 0,2 % Penicillin-Streptomycin zu 48 ml Basalmedium hinzufügen. Aliquotieren Sie das Präparat und lagern Sie es bei -20 °C. Lagern Sie dieses Medium bis zu 4 Wochen bei 4 °C.
- Bereiten Sie PSC-Präparationsmedium vor, indem Sie 1 ml Nahrungsergänzungsmittel und 0,2 % Penicillin-Streptomycin zu 500 ml Basalmedium hinzufügen. Bei der Verwendung das Medium für den einmaligen Gebrauch vorheizen und das Medium bis zu 3 Wochen bei 4 °C lagern.
- Bereiten Sie das CM-Differenzierungsmedium vor, indem Sie 1x B27-Supplement minus Insulin und 1% Penicillin-Streptomycin in RPMI 1640 geben. Bereiten Sie das CM-Erhaltungsmedium vor, indem Sie 1x B27-Supplement und 1% Penicillin-Streptomycin in RPMI 1640 geben. Lagern Sie diese beiden Arten von Medien 2 Wochen lang bei 4 °C.
- Tauchen Sie das Matrigel in Eis und halten Sie es über Nacht bei 4 °C. Stellen Sie sicher, dass Matrigel vollständig aufgetaut ist. aliquotieren Sie dann in 1 mL Aliquots, die bei -80 °C gelagert werden, für die spätere Verwendung, und fügen Sie 1 mL Matrigel in 49 mL DMEM/F12 bei 4 °C hinzu, um die Matrigel-Arbeitslösung herzustellen. Geben Sie 850 μl dieser Matrigel-Arbeitslösung pro Vertiefung einer 6-Well-Kulturplatte hinzu und inkubieren Sie die Platte mindestens 30 Minuten lang bei 37 °C. Für die 96-Well-Kulturplatte, die für die CM-Differenzierung verwendet wird, fügen Sie 35 μl Matrigel-Arbeitslösung pro Well hinzu.
HINWEIS: Halten Sie Matrigel immer auf Eis; Die Pipettenspitzen und -röhrchen sollten bei 4 °C vorgekühlt werden. Die Matrigel-Arbeitslösung kann 2 Wochen lang bei 4 °C gelagert werden. Achten Sie darauf, Blasen zu vermeiden, wenn Sie die Matrigel-Arbeitslösung pipettieren.
- PSC-Wartung und -Durchgang
- Bewahren Sie PSCs in 6-Well-Kulturplatten auf. Bestätigen Sie die Zelldichte unter einem Mikroskop und bereiten Sie sich auf die Passage vor, wenn die Konfluenz ~70 % erreicht (Abbildung 2A).
- Verwenden Sie EDTA, um die Zellen für die Passage zu verdauen. PSC-Kulturmedium, PBS und EDTA in einem 37 °C warmen Wasserbad erwärmen und Y27632 in das Medium bis zu einer Endkonzentration von 5 μM geben.
- Waschen Sie die PSCs mit 1 mL PBS, fügen Sie 1 mL EDTA pro Vertiefung hinzu und inkubieren Sie die Platte 3 Minuten lang in einem 37 °C Inkubator.
- Aspirieren Sie das EDTA, bevor sich die Zellen von der Kulturplatte lösen, und spülen Sie dann mit dem 1 ml PSC-Kulturmedium den Boden der Vertiefung vorsichtig 3-4x ab, um die Zellen zu resuspendieren.
HINWEIS: Dies ermöglicht es uns, den Schritt der Zentrifugation zu überspringen und Zellschäden bei Passiervorgängen zu reduzieren. - Aspirieren Sie das DMEM/F12 aus einer Matrigel-beschichteten Vertiefung einer 6-Well-Kulturplatte und fügen Sie 2 ml frisches PSC-Kulturmedium pro Vertiefung hinzu (Y27632 hinzugefügt). Geben Sie die Zellsuspension mit dem Split-Verhältnis von 1:6 bis 1:12 in die Wells. Stellen Sie die Platte in den 37 °C heißen Inkubator mit 5 % CO2.
- Wechseln Sie das PSC-Kulturmedium (ohne Y27632) 12 bis 24 Stunden nach der Übergabe; Wechseln Sie dann das Medium und überprüfen Sie jeden Tag den Zellzustand und die Konfluenz.
- Kardiale Differenzierung von PSCs
- Für die CM-Differenzierung werden PSCs in eine 96-Well-Kulturplatte in PSC-Vorbereitungsmedium eingepflanzt (Y27632 hinzugefügt). Für die CM-Differenzierung führen Sie die gleichen Schritte wie oben (Schritte 1.2.2-1.2.6) aus, um PSCs in eine 96-Well-Kulturplatte in PSC-Vorbereitungsmedium zu säen (Y27632 hinzugefügt).
HINWEIS: In diesem Stadium kann die Dauer der EDTA-Behandlung angemessen verlängert werden (5 Minuten oder länger), um die PSCs ausreichend zu dissoziieren, so dass die Klone nach der Passage kleiner sein können. Unsere Studie hat gezeigt, dass innerhalb desselben Gebiets kleinere Kolonien der Differenzierung förderlich sind. - Stufe I: Wenn PSCs eine Konfluenz von 80-90 % erreichen, schalten Sie das Medium auf ein CM-Differenzierungsmedium mit 2-20 μM CHIR99021 (CHIR) um (Abbildung 2B). Der Zeitpunkt der Mediumsumschaltung wird als Tag 0 bezeichnet. Nach 24-48 h CHIR-Behandlung wechseln Sie das Medium auf frisches CM-Differenzierungsmedium.
HINWEIS: CHIR ist ein GSK3beta-Inhibitor für die Mesoderm-Induktion. Die optimale CHIR-Dosis für die CM-Differenzierung schwankt je nach Zelllinie und Charge. Die optimale CHIR-Dosis beträgt beispielsweise 8-10 μM, 48 h für iPS-B1. Um verschiedene Hellfeldbilder für das Training des ML-Modells zu sammeln, titrierten wir hier die CHIR-Konzentrationen für jede Experimentcharge für unterschiedliche Differenzierungseffizienzen. - Stufe II: Bei 72 h (Tag 3) das Medium durch das CM-Differenzierungsmedium ersetzen, ergänzt mit 5 μM IWR1 und Kultur für 48 h (Tag 5). Wechseln Sie das Medium mit dem CM-Differenzierungsmedium für 1-2 Tage (bis zu den Tagen 6-7). Zu diesem Zeitpunkt differenzieren sich PSCs zu kardialen Vorläuferzellen (CPC).
- Stufe III: Ersetzen Sie das Medium durch das CM-Erhaltungsmedium und tauschen Sie das Medium alle 3 Tage aus. Ernten Sie den CM an Tag 10 oder Tag 12 für die weitere Analyse.
- Für die CM-Differenzierung werden PSCs in eine 96-Well-Kulturplatte in PSC-Vorbereitungsmedium eingepflanzt (Y27632 hinzugefügt). Für die CM-Differenzierung führen Sie die gleichen Schritte wie oben (Schritte 1.2.2-1.2.6) aus, um PSCs in eine 96-Well-Kulturplatte in PSC-Vorbereitungsmedium zu säen (Y27632 hinzugefügt).
- Immunfluoreszenzfärbung von CM
- Waschen Sie die Zellen an Tag 10 oder Tag 12 mit PBS und fixieren Sie sie mit 4% Paraformaldehyd in PBS für 15 min bei Raumtemperatur. Waschen Sie die CMs 2x mit PBS und lagern Sie die Platte bei 4 °C bis zu ~1 Woche.
HINWEIS: Paraformaldehyd ist ein giftiges Reagenz mit einem stechenden Geruch. Arbeiten Sie in einer chemischen Haube und schützen Sie Augen und Hände. - Behandeln Sie die Zellen zum Zeitpunkt der Färbung 15 Minuten lang bei Raumtemperatur mit permeabilisierender Lösung (0,1 % Triton X-100 in PBS). dann inkubieren Sie die Zellen in einer Blockierungslösung (0,1 % Triton X-100 mit 3 % Eselserum in PBS) für 30 Minuten bei Raumtemperatur.
- Die Probe wird mit einem kardialen Troponin T (cTnT)-Primärantikörper (1:300) inkubiert, der über Nacht bei 4 °C in einer Blockierungslösung verdünnt wurde, um die CMs zu identifizieren.
- Sammeln Sie den primären Antikörper und waschen Sie die Zellen mit PBS 3x. Die Probe wird mit Sekundärantikörpern in PBS mit 1 % Rinderserumalbumin für 1 h bei 37 °C in dunkler Umgebung inkubiert.
HINWEIS: Der gesammelte Primärantikörper kann für 2-3x in 1 Woche wiederverwendet werden.
- Sammeln Sie den primären Antikörper und waschen Sie die Zellen mit PBS 3x. Die Probe wird mit Sekundärantikörpern in PBS mit 1 % Rinderserumalbumin für 1 h bei 37 °C in dunkler Umgebung inkubiert.
- Entfernen Sie den Sekundärantikörper aus den Zellen und waschen Sie die Zellen 3x mit PBS. Verwenden Sie Hoechst 33342 (1:1.000 in PBS), um Zellkerne 5 Minuten lang bei Raumtemperatur zu färben. Spülen Sie die Zelle 3x mit PBS und fügen Sie 100 μl PBS pro Vertiefung hinzu, um ein Austrocknen zu vermeiden. Lagern Sie die Platte bei 4 °C an einem dunklen Ort und nehmen Sie innerhalb von 1 Woche Fluoreszenzbilder auf.
- Waschen Sie die Zellen an Tag 10 oder Tag 12 mit PBS und fixieren Sie sie mit 4% Paraformaldehyd in PBS für 15 min bei Raumtemperatur. Waschen Sie die CMs 2x mit PBS und lagern Sie die Platte bei 4 °C bis zu ~1 Woche.
2. Erfassung von Bildströmen während des gesamten Differenzierungsprozesses
- Aufbau eines Versuchsaufbaus für das Sammeln von Bildern
- Verwenden Sie ein automatisiertes Mikroskop, das Lebendzellkulturen und Bildgebung unterstützt, um Hellfeldbilder verschiedener Stadien der CM-Differenzierung und cTnT-Immunfluoreszenzbilder des Differenzierungsergebnisses zu sammeln (Abbildung 2A-E). Verwenden Sie die mikroskopunterstützende Software, um das Experimentierprogramm zu entwerfen und das Mikroskop zu steuern.
- Öffnen Sie die Software und erstellen Sie einen neuen Versuchsplan. Wählen Sie ein 5-fach-Objektiv und eine 2-fach-Tubuslinse für die Bildgebung. Aktivieren Sie die Optionen Kacheln und Z-Stapel.
- Fügen Sie einen TL-Hellfeldkanal für Hellfeld-Bildgebung im Menü Kanäle hinzu. Fügen Sie AF488- und H3342-Kanäle für die Immunfluoreszenzbildgebung hinzu. Ändern Sie den Strahlengang im Menü "Imaging Setup" für die Imaging mit der externen digitalen CMOS-Kamera.
- Öffnen Sie das Menü "Erfassungsmodus" und stellen Sie 2 x 2 Binning ein, um das Signal-Rausch-Verhältnis zu erhöhen.
- Öffnen Sie das Menü Z-Stapel, und wählen Sie das Modell Mitte aus. Legen Sie die Anzahl der Slices und die Intervalle beim Scannen fest. Wählen Sie 3-5 Schichten mit Intervallen von 3-6 μm für scharfe Bilder in der Analyse.
- Richten Sie im Fenster Navigation & Kacheln die Kachelregionen nach Carrier ein und legen Sie 25 Kacheln (5 Spalten x 5 Zeilen) für ein Feld fest. Wählen Sie im Menü Kacheln die Vorlage Multiwell 96 in Sample Carrier aus, und legen Sie eine Überlappung von 5 % bis 15 % für benachbarte Regionen fest.
- Wenn ein kontinuierliches Imaging erforderlich ist, aktivieren Sie die Option Time series und legen Sie die Dauer und das Intervall im Menü Zeitreihe fest. Legen Sie für das Sammeln des Bildstroms in Phase I eine Dauer von 12 Stunden ohne Intervalle fest.
- Aufnahme von Hellfeld- und Immunfluoreszenzbildern
- Um Bilder aufzunehmen, legen Sie zuerst die Zellkulturplatte in die Probenschale und legen Sie die Probe in das Mikroskop. Wählen Sie die Vorlage Multiwell 96 aus, die dem Versuchsplan entspricht. Besteht die Probe aus lebenden Zellen, öffnen Sie das Heizsystem und die CO2 -Pumpe, um die für die Kultur geeignete Bedingung (37 °C, 5 % CO2) aufrechtzuerhalten.
- Öffnen Sie das experimentelle Vorgabenprojekt, und speichern Sie es als neues. Öffnen Sie das Menü Kacheln und kalibrieren Sie die Position der Platte manuell. Wählen Sie im Fenster Navigation & Kacheln die gewünschten Vertiefungen aus und klicken Sie auf Erstellen, um Kachelregionen für diese Vertiefungen zu erstellen.
- Klicken Sie im Menü Kacheln auf Kachelbereiche überprüfen, und führen Sie den Autofokus aus, um alle Wells zu überprüfen. Korrigieren Sie dann manuell die Fokussierung der einzelnen Vertiefungen unter dem Hellfeld, indem Sie mit dem Mausrad scrollen.
- Markieren Sie die gewünschten Kanäle und stellen Sie die Belichtungszeiten im Menü Kanäle ein. Die empfohlenen Belichtungszeiten betragen 1,625 ms für Hell, 100 ms für H3342 und 550 ms für AF488. Führen Sie einen einzigen Snap aus, um die Bilder aller Kanäle zu überprüfen.
- Klicken Sie abschließend auf die Schaltfläche Experiment starten, und warten Sie auf die automatische Bildgebung. In der Regel dauert es ~1,2 Stunden, um das Scannen einer ganzen 96-Well-Kulturplatte abzuschließen (96 Wells, drei Schichten, 25 Kacheln für jede Well unter dem Hellfeld). Wählen Sie im Verarbeitungsframework Image Export aus, wählen Sie den Dateityp des unkomprimierten TIFF-Formats oder PNG-Formats aus und wenden Sie es an. Verwenden Sie Bilder von 9 Kacheln (3 x 3) in der Mitte des Bohrlochs für die weitere Analyse (Abbildung 2A-E).
3. Etablierung der image-based ML-Strategie in jeder Phase des Differenzierungsprozesses
- Software-Vorbereitung
- Installieren Sie die Python-Umgebung mit den Paketen pytorch (1.9.0), numpy, scipy, scikit-learn, pandas, visdom, scikit-image, opencv-python und matplotlib.
- Installieren Sie Jupyter Notebook.
- Installieren Sie MATLAB.
- Laden Sie den Code und die Beispieldatasets von https://github.com/zhaoyanglab/ML-for-PSC-differentiation herunter.
HINWEIS: Die Leistung von ML hängt von der Qualität der Datensätze und der Auswahl der Modelle und Hyperparameter ab. Es ist nicht garantiert, dass die Beispielimplementierung für alle Umstände optimal ist. ML-Ingenieure sollten den Code anpassen und das Modell und die Hyperparameter an ihre spezifischen Bedürfnisse anpassen.
- ML-Strategie in der PSC-Phase: Kontrolle der anfänglichen PSC-Koloniezustände
Datensatz: Paare von Hellfeldbildern bei 0 h (vor der CHIR-Behandlung) und finale Differenzierungseffizienz
Modell: Random Forest- Bereiten Sie einen Datensatz vor, der aus Hellfeldbildern bei 0 h (vor der CHIR-Behandlung) und den endgültigen cTnT-Fluoreszenzbildern besteht. Um die Vielfalt der morphologischen Profile von 0-Stunden-Hellfeldbildern zu erhöhen, stellen Sie sicher, dass der Datensatz PSC-Kolonien aus verschiedenen Zelllinien enthält, wobei die Differenzierung über ein Zeitintervall nach der Passage eingeleitet wird. Behandeln Sie die Wells im Datensatz mit den optimalen CHIR-Bedingungen in Phase I.
HINWEIS: Um sicherzustellen, dass die Bienenvölker unter den optimalen CHIR-Bedingungen behandelt werden, können Vertiefungen innerhalb derselben Charge mit titrierten CHIR-Dosen behandelt werden. Nur Bohrlöcher mit hocheffizienten CHIR-Bedingungen sind im Datensatz enthalten. - Quantifizieren Sie die Differenzierungseffizienz jedes Wells durch den Differenzierungseffizienzindex, der aus seinen cTnT-Fluoreszenzbildern berechnet wird, der definiert ist durch
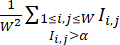 (wobei
(wobei 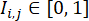 die Fluoreszenzintensität an der Pixelposition (i, j) im W × W Fluoreszenzbild darstellt, und die Summe wird über alle Pixelpositionen mit einer Intensität über einem Schwellenwert α, in der Regel auf 0,5 festgelegt). Berechnen Sie innerhalb jeder Zelllinie den maximalen Differenzierungseffizienzindex für alle Vertiefungen in dieser Zelllinie, und normalisieren Sie deren Differenzierungseffizienzindizes um den Maximalwert.
die Fluoreszenzintensität an der Pixelposition (i, j) im W × W Fluoreszenzbild darstellt, und die Summe wird über alle Pixelpositionen mit einer Intensität über einem Schwellenwert α, in der Regel auf 0,5 festgelegt). Berechnen Sie innerhalb jeder Zelllinie den maximalen Differenzierungseffizienzindex für alle Vertiefungen in dieser Zelllinie, und normalisieren Sie deren Differenzierungseffizienzindizes um den Maximalwert.
HINWEIS: Da das Differenzierungspotenzial jeder Zelllinie von Natur aus unterschiedlich ist, wird eine solche Normalisierung mehr auf die morphologischen Merkmale der Kolonie konzentrieren, die verschiedenen Zelllinien gemeinsam sind und zum Differenzierungsergebnis beitragen. Die Normalisierung macht PSC-Kolonien aus verschiedenen Zelllinien vergleichbar und mildert die durch Zelllinien induzierte Verzerrung. - Quantifizieren Sie die morphologischen Profile von 0-h-Hellfeldbildern durch hochdimensionale Merkmale, die die Eigenschaften der Kolonieform offenbaren. Um Koloniebereiche in Hellfeldbildern zu umreißen, berechnen Sie die lokale Entropie (mit dem Radius r); Pixel mit einer lokalen Entropie, die größer als ein Schwellenwert σ ist, werden durch abgedeckte Zellen gekennzeichnet. Verwenden Sie eine Parametereinstellung von r = 10 Pixel und σ = 3. Schalten Sie das Arbeitsverzeichnis auf "./colony_control"; Speichern Sie die Hellfeldbilder, Binärbilder von Zellregionen und cTnT-Fluoreszenzbilder unter "colony_control/ image_data/CD00-*/[brightfield|cell_region|ctnt]/S*.png"; und führen Sie das Jupyter Notebook "Compute_features.ipynb" aus, um Features zu berechnen, die sich unter anderem auf Koloniefläche, Umfang, Solidität, Konvexität, Zirkularität und Schwerpunktkonturentfernungen beziehen.
- Unterteilen Sie das Dataset nach dem Zufallsprinzip in einen Trainingssatz und einen Testsatz, wobei der Trainingssatz zwischen 50 % und 80 % der Gesamtdaten ausmacht. Führen Sie den letzten Block aus: Teilen Sie das Dataset in Jupyter Notebook, um das Dataset zu teilen (Abbildung 3A).
- Trainieren Sie ein Random-Forest-Regressionsmodell mit dem Trainingssatz, um die Differenzierungseffizienz von 0-Stunden-Hellfeldbild-Features vorherzusagen. Führen Sie das Jupyter Notebook "Machine_learning.ipynb" aus, um das Modell zu trainieren und zu testen.
- Werten Sie das trainierte Random Forest-Modell im Testsatz aus. Bestätigen Sie, dass die vorhergesagte Differenzierungseffizienz stark mit der wahren korreliert, indem Sie den Pearson-Korrelationskoeffizienten verwenden (Abbildung 3B).
HINWEIS: Da der Testsatz nicht in die Trainingsphase einbezogen wird, deutet eine hohe Konsistenz zwischen der vorhergesagten und der wahren Differenzierungseffizienz im Testsatz darauf hin, dass das Modell gut auf unsichtbare Stichproben verallgemeinern kann. - Ermitteln Sie die Wichtigkeit von Merkmalen aus dem trainierten Random-Forest-Modell, und wählen Sie 8 Merkmale mit der größten Wichtigkeit aus. Visualisieren Sie ihre Beziehung zur endgültigen Differenzierungseffizienz, indem Sie den Bereich der Features in Klassen unterteilen und die mittlere Differenzierungseffizienz in jedem Abschnitt berechnen. Führen Sie das Jupyter Notebook "Feature_importance.ipynb" aus, um die Visualisierung durchzuführen (Abbildung 3C).
HINWEIS: Die Visualisierung kann helfen zu verstehen, wie Änderungen in der Form der PSC-Kolonie zur Differenzierung beitragen. - Wenden Sie das trainierte Modell in neuen Batches an, um den PSC-Koloniestatus in Echtzeit zu überwachen. Erfassen Sie das Hellfeldbild zu jeder vollen Stunde, übergeben Sie das Bild an das trainierte Modell und erhalten Sie die vorhergesagte Differenzierungseffizienz. Sobald der prognostizierte Wirkungsgrad einen Schwellenwert (z. B. 50 %) überschreitet, starten Sie den Differenzierungsprozess. Wenn der prognostizierte Wirkungsgrad den Schwellenwert nicht erreichen konnte, verwerfen Sie diese Stichproben.
HINWEIS: Während der PSC-Phase wird das Differenzierungspotenzial der PSC-Völker zunächst steigen und später abnehmen. Die Echtzeitüberwachung ermöglicht es den Experimentatoren, den günstigsten Zeitpunkt zu identifizieren, um die Differenzierung einzuleiten.
- Bereiten Sie einen Datensatz vor, der aus Hellfeldbildern bei 0 h (vor der CHIR-Behandlung) und den endgültigen cTnT-Fluoreszenzbildern besteht. Um die Vielfalt der morphologischen Profile von 0-Stunden-Hellfeldbildern zu erhöhen, stellen Sie sicher, dass der Datensatz PSC-Kolonien aus verschiedenen Zelllinien enthält, wobei die Differenzierung über ein Zeitintervall nach der Passage eingeleitet wird. Behandeln Sie die Wells im Datensatz mit den optimalen CHIR-Bedingungen in Phase I.
- ML-Strategie in Phase I: Frühzeitige Bewertung und Anpassung der CHIR-Dosen
Datensatz: Paare von Hellfeld-Bildströmen bei 0-12 h und CHIR-Konzentrationsmarkierungen (niedrig/optimal/hoch)
Modell: logistische Regression- Bereiten Sie ein Dataset vor, das aus Whole-Well-Hellfeld-Bildströmen besteht, wobei die Bilder stündlich zwischen 0 h und 12 h aufgenommen werden. Notieren Sie die CHIR-Dosen (d. h. Kombinationen aus CHIR-Konzentrationen und -Dauer) jeder Vertiefung. Stellen Sie sicher, dass der Datensatz verschiedene Chargen und Wells mit titrierten CHIR-Dosen enthält (z. B. Konzentration = 2, 4, 6, 8, 10 und 12 μM; Dauer = 24, 36 und 48 h), um die Vielfalt zu erhöhen. Schalten Sie das Arbeitsverzeichnis auf "./stage_I"; Speichern Sie die Bilder unter dem Namen "./data/image/CD01-*/S*/T*.png", wobei CD01-* (CD01-1, CD01-2, CD01-3, CD01-4) der Chargenname ist, S* (S1, S2, ..., S96) der Index von well und T*.png (T1.png, T2.png, ..., T10.png) das vorverarbeitete Hellfeldbild des Wells in verschiedenen Zeitschritten des Bildstroms ist.
- Berechnen Sie in jeder Charge den Prozentsatz der cTnT+ -Zellen für jede Vertiefung, der berechnet wird, indem die Anzahl der cTnT+ -Zellen durch die Gesamtzahl der Hoechst+ -Zellen im Fluoreszenzbild dividiert wird. Mittelwerte den Prozentsatz der cTnT+ -Zellen über parallele Wells mit der gleichen CHIR-Dosisbedingung. Speichern Sie die CHIR-Dosisbedingung und den Prozentsatz der cTnT+ -Zellen für jede Vertiefung in einer CSV-Datei "./data/image/CD01-*/CD01-?_exp_config.csv".
- Bestimmen Sie in jeder Charge den "niedrigen", "optimalen" und "hohen" CHIR-Konzentrationsbereich unter jeder CHIR-Dauer nach den folgenden Kriterien. CHIR-Konzentrationen mit einem durchschnittlichen Prozentsatz an cTnT+ -Zellen über einem Schwellenwert (z. B. 20 %) werden als "optimal" bezeichnet. CHIR-Konzentrationen außerhalb des optimalen Bereichs werden als "niedrig" oder "hoch" bezeichnet. Beschriften Sie die Vertiefungen unter jeder CHIR-Dauer entsprechend den entsprechenden CHIR-Konzentrationen als "niedrig", "optimal" und "hoch".
HINWEIS: Empirisch gibt es eine negative Korrelation zwischen den CHIR-Konzentrationen und der Dauer von hocheffizienten Wells innerhalb einer Charge: Bei einer höheren CHIR-Dauer sinken die optimalen Konzentrationen. Mit anderen Worten, die Optimalität der CHIR-Konzentrationen hängt von der CHIR-Dauer ab, und daher unterscheiden sich die Markierungen der Wells zwischen den verschiedenen CHIR-Dauern. - Berechnen Sie in jeder Charge unter jeder CHIR-Dauer die ΔCHIR-Konzentration für jede Konzentration c, um ihre Abweichung vom Optimum zu quantifizieren. Bezeichnen Sie den optimalen CHIR-Konzentrationsbereich mit [c1, c2] und dann ΔCHIR-Konzentration(c) = c - c 1 (wenn c < c1), 0 (wenn c1 ≤ c ≤ c2) oder c - c2 (wenn c > c2).
- Extrahieren Sie Merkmale der Bildströme im Datensatz und stellen Sie sicher, dass sie die morphologischen Veränderungen der Zellen während der Phase I widerspiegeln. Um Bildmerkmale über lokale Entropie, Fläche, Umfänge, Helligkeit, optischen Fluss und fraktale Dimension zu berechnen, führen Sie
cd ./data
Python-compute_features.py
CD..
HINWEIS: Um die morphologischen Veränderungen hervorzuheben, können die Werte einiger Merkmale (z. B. Fläche, Umfang und optischer Fluss) anhand ihrer anfänglichen Basiswerte in den Bildströmen normalisiert werden. - Unterteilen Sie das Dataset nach dem Zufallsprinzip in einen Trainingssatz und einen Testsatz. Führen Sie das Jupyter Notebook "./data/prepare_dataset.ipynb" aus, um das Dataset zu teilen und die Datasets als Datenrahmen zu organisieren (Abbildung 3D).
- Trainieren Sie für jede CHIR-Dauer ein logistisches Regressionsmodell, um das CHIR-Konzentrationslabel (niedrig/optimal/hoch) aus den Bildstrom-Features im Trainingssatz vorherzusagen. Bewerten Sie die Klassifizierungsleistung des trainierten logistischen Regressionsmodells auf dem Testsatz anhand von Genauigkeit, Präzision, Abruf, F1-Wert und Fläche unter der Kurve (AUC). Führen Sie das Jupyter Notebook "machine_learning.ipynb" zu Trainings- und Testzwecken aus (Abbildung 3E).
- Bewerten Sie die Modellleistung bei der CHIR-Dosisbewertung. Führen Sie im Testsatz die vorhergesagten Beschriftungen von parallelen Wells mit derselben CHIR-Konzentration mithilfe von Abweichungswerten (im Bereich von -1 bis 1) zusammen, die durch definiert sind
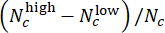 , wobei Nc die Anzahl der Wells mit der Konzentration c angibt und
, wobei Nc die Anzahl der Wells mit der Konzentration c angibt und 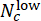
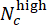 die Wells zwischen ihnen als "niedrig" bzw. "hoch" vorhergesagt werden. Vergewissern Sie sich, dass die vorhergesagten Abweichungswerte stark mit der wahren ΔCHIR-Konzentration für jede CHIR-Dosis korrelieren, indem Sie den Pearson-Korrelationskoeffizienten verwenden.
die Wells zwischen ihnen als "niedrig" bzw. "hoch" vorhergesagt werden. Vergewissern Sie sich, dass die vorhergesagten Abweichungswerte stark mit der wahren ΔCHIR-Konzentration für jede CHIR-Dosis korrelieren, indem Sie den Pearson-Korrelationskoeffizienten verwenden. - Führen Sie eine batchübergreifende Validierung durch, um die Generalisierungsleistung des Modells zu testen. Lassen Sie in jeder Runde eine Charge zum Testen übrig, während Sie andere zum Training verwenden. Vergleichen Sie die vorhergesagten Abweichungswerte und die tatsächliche ΔCHIR-Konzentration für jede CHIR-Dosis in jeder Testcharge unter Verwendung des Pearson-Korrelationskoeffizienten. Führen Sie das Jupyter Notebook "cross_batch_validation.ipynb" aus, um die batchübergreifende Überprüfung zu implementieren (Abbildung 3F).
HINWEIS: Für eine ausgewählte CHIR-Dauer h wird die Vorhersage für alle Bohrlöcher mit Konzentration aus dem logistischen Regressionsmodell, das unter der Dauer h trainiert wurde, in einen Abweichungswert umgewandelt, der die Optimalität der CHIR-Konzentration c widerspiegelt. Ein Abweichungswert nahe 0 deutet darauf hin, dass die Konzentration c unter der Dauer h als optimal vorhergesagt wird, während ein negativer oder positiver Abweichungswert auf eine Unter- oder Überdosierung hindeutet, was als Signal für eine spätere Anpassung der CHIR-Dosen dienen kann. Da die Vorhersage früh (12 h) erfolgt, können die endgültige CHIR-Dauer und die CHIR-Konzentration während des restlichen Stadiums I noch angepasst werden. - Wenden Sie die trainierten logistischen Regressionsmodelle an, um CHIR-Dosen in neuen Chargen zu bewerten. Berechnen Sie für eine gegebene CHIR-Konzentration 0-12 h Bildstrom-Features für parallele Bohrlöcher mit Konzentration, übergeben Sie die Merkmale an die logistischen Regressionsmodelle, die unter unterschiedlichen CHIR-Dauern trainiert wurden, und erhalten Sie dementsprechend die vorhergesagten Beschriftungen für jedes Bohrloch. Berechnen Sie basierend auf der Vorhersage der Modelle Abweichungswerte, um jede CHIR-Konzentration unter verschiedenen CHIR-Dauern zu bewerten.
- Mit der modellbasierten CHIR-Dosisabschätzung werden die Vertiefungen unter jeder suboptimalen CHIR-Konzentration entsprechend gerettet, indem ihre CHIR-Dauer oder -Konzentration vor 48 h in Richtung des Optimums angepasst wird.
- Anpassen der Dauer: Entsprechend der Modellvorhersage über die optimale CHIR-Dauer wird der Zeitpunkt angepasst, zu dem das CHIR-haltige Medium in diesen Vertiefungen auf das CM-Differenzierungsmedium umgeschaltet wird. Wenn der optimale Zeitpunkt verpasst wird, verwerfen Sie diese Stichproben.
- Anpassen der Konzentration: Wenn unter der voreingestellten CHIR-Dauer die aktuelle Konzentration als "niedrig" vorhergesagt wird, fügen Sie direkt eine hohe Konzentration von CHIR in das Medium hinzu, um die CHIR-Konzentration zu erhöhen. Verdünnen Sie CHIR bei Bedarf in DMSO (50 mM) mit PBS, fügen Sie eine angemessene Menge Flüssigkeit hinzu und schütteln Sie die Platte vorsichtig. Wenn die aktuelle Konzentration als "hoch" vorhergesagt wird, aspirieren Sie ein bestimmtes Volumen des CHIR-haltigen Mediums und ergänzen Sie es mit dem gleichen Volumen des CM-Differenzierungsmediums.
HINWEIS: In den ersten 48 Stunden der Differenzierung sezernieren die Zellen Faktoren, um das Zellwachstum und die Differenzierung zu verbessern, wechseln Sie also das Medium nicht vollständig. Darüber hinaus ist es notwendig, das mittlere Volumen in der Differenzierung konstant zu halten. Stellen Sie daher sicher, dass das Volumen der zugegebenen Flüssigkeit so gering wie möglich ist, wenn Sie die CHIR-Konzentration anpassen.
- ML-Strategie in Phase II: Anerkennung von CM-gebundenen CPCs
Datensatz: Paare von Hellfeldbildern an Tag 6 und manuell annotierte Masken von CM-gebundenen CPCs
Modell: ResNeSt38 mit Grad-CAM36 für schwach überwachtes Lernen- Bereiten Sie an Tag 6 einen Datensatz bestehend aus Hellfeldbildern vor. Kommentieren Sie CM-committed CPCs in den Hellfeldbildern manuell, indem Sie die cTnT+ -Zellen in den Bildströmen von Tag 12 bis Tag 6 verfolgen.
- Schneiden Sie die Hellfeldbilder und die entsprechenden manuellen Anmerkungen (als Masken bezeichnet) von CM-gebundenen CPCs in Patches zu, wobei jeder Patch bei 1/5,5 der Whole-Well-Bildgröße groß ist. Extrahieren Sie Patches auf eine überlappende Weise, bei der benachbarte Patches eine Überlappung von 50 % im Trainingssatz und eine Überlappung von 75 % im Testsatz aufweisen. Kennzeichnen Sie Patches mit ≥30 % CM-zugesicherten CPCs als "positiv". Kennzeichnen Sie Patches ohne CM-zugesicherte CPCs als "negativ". Schalten Sie das Arbeitsverzeichnis auf "stage_II". Führen Sie "./Crop_and_Reconstruct/mask_crop.m" aus, um die Bilder zuzuschneiden. Führen Sie "./Crop_and_Reconstruct/classification.m" aus, um Hellfeld-Patches Beschriftungen (0: negativ; 1: positiv) zuzuweisen (Abbildung 3G).
- Unterteilen Sie die beschrifteten Image-Patches nach dem Zufallsprinzip in einen Trainingssatz und einen Testsatz.
- Trainieren Sie ein tiefes neuronales Faltungsnetz, ResNeSt38 , um zu lernen, diese Patches zu klassifizieren. Werten Sie das trainierte ResNeSt auf dem Testsatz aus, um zu bestätigen, dass das Modell eine hohe Klassifizierungsgenauigkeit aufweist. Führen Sie "train.py" und "test.py" zum Trainieren bzw. Testen aus (Abbildung 3H).
- Verwenden Sie Grad-CAM36 , um die Regionen hervorzuheben, die am meisten zur Inferenz von ResNeSt beitragen, dargestellt durch Heatmaps. Binarisieren Sie die Heatmaps (unter Verwendung eines Schwellenwerts, z. B. maximal 10 % ×), um die vorhergesagten CM-zugesicherten CPC-Regionen zu erhalten, die als Imaged-Recognized CPC-REGIONEN (IR-CPC) bezeichnet werden.
- Vergleichen Sie die IR-CPC-Regionen mit manuell annotierten Masken im Testsatz anhand von Genauigkeit, F1-Score, Präzision, Erinnerung, Spezifität und Intersection-over-Union (IoU). Vergleichen Sie den Anteil der IR-CPC-Regionen mit den Differenzierungseffizienzindizes (berechnet aus den endgültigen cTnT-Fluoreszenzbildern) unter Verwendung des Pearson-Korrelationskoeffizienten. Führen Sie "./Evaluation/index_calculate.m" und "./Evaluation/Pearson_correlation.m" für die Leistungsbewertung aus (Abbildung 3I).
- Wenden Sie das trainierte Modell an, um CPCs für neue Chargen in Phase II zu erkennen. Schneiden Sie Whole-Well-Hellfeldbilder in Patches zu, übergeben Sie die Patches an den trainierten ResNeSt und erhalten Sie die IR-CPC-Regionen auf Patch-Ebene, die dann zusammengeführt werden, um die Whole-Well-IR-CPC-Regionen zu erhalten. Berechnen Sie den Anteil der IR-CPC-Regionen als Indikator für die endgültige Differenzierungseffizienz. Reinigen Sie die IR-CPCs, um die Zellkontamination zu reduzieren (siehe unten).
ANMERKUNG: Da das CPC-Erkennungsmodell eine frühe Bewertung der endgültigen Differenzierungseffizienz vornehmen kann, kann es auch als Hochdurchsatz-Auslesung für das Screening kleiner Moleküle dienen, die für den Differenzierungsprozess förderlich sind, was das Differenzierungsprotokollweiter optimieren kann 35.
- Regionsselektive Aufreinigung von abgebildet erkannten CPCs
HINWEIS: Die Photoaktivierungsexperimente basieren auf einem inversen Fluoreszenzmikroskop, das mit einem motorisierten Tisch ausgestattet ist.- Entsprechend den IR-CPC-Regionen, die vom trainierten Modell vorhergesagt werden, verwerfen Sie die Bohrlöcher, die keine IR-CPCs erzeugen konnten. Um diese CM-gebundenen CPCs aufzureinigen, verwenden Sie eine nicht-zytotoxische photoaktivierbare Sonde, Dual-Activatable Cell Tracker 1 (DACT-1), um nicht-CPC39 regionsselektiv zu markieren.
- Lösen Sie den DACT-1 (10 mM) in DMSO auf und lagern Sie ihn bei -20 °C. Inkubieren Sie Tag-6-Zellen mit 1 μM DACT-1 in CM-Differenzierungsmedium für 30 min bei 37 °C.
- Untersuchen Sie die lebenden Zellen unter einem Mikroskop und wählen Sie die Region of Interest (ROI) als Polygone in MATLAB aus. Zeichnen Sie parallele Linien mit einem Abstand von 20 μm innerhalb des Polygons und berechnen Sie die Koordinaten der Schnittpunkte.
- Stellen Sie die bewegliche Spur des motorisierten Tisches entsprechend den Leiterbahnen ein, dann würde sich der Tisch mit 0,12 mm/s bewegen, so dass eine auf dem Tisch montierte 405-nm-Laserlinie (20 μm Durchmesser) den ROI global scannen kann, um Nicht-CPCs eingeschränkt zu photoaktivieren. Nach der Bestrahlung werden die DACT-1-markierten Zellen mit einer 561-nm-Laserlinie detektiert.
- Dissoziieren Sie die Zellen mit 0,05 % Trypsin-EDTA (verdünnt durch PBS) bei 37 °C für 5-7 Minuten und schütteln Sie die Platte vorsichtig 2 Minuten lang in einem Inkubator. Die Zellen werden durch ein 40 μm Zellsieb filtriert und bei 500 × g für 3 min zentrifugiert. Resuspendieren Sie die Zellen in 0,5 % BSA und halten Sie das Röhrchen bis zum Sortieren auf Eis. Bereiten Sie auch eine unbestrahlte Probe als Negativkontrolle für die Sortierung vor.
- Starten Sie das Durchflusszytometrie-Sortiersystem und richten Sie die Sortiersoftware ein. Wählen Sie die Laser mit einer Wellenlänge von 561 nm und verwenden Sie einen Bandpassfilter von 610/20 für die DACT1+-Sortierung. Verwenden Sie für jede Stichprobe ein polygonales Anschnittwerkzeug, um die Zellpopulation in einem bivariaten Diagramm aus Vorwärtsstreuung (FSC) und Seitenstreuung (SSC) einzukreisen, um die einzelne Zelle auszuwählen. Führen Sie dann die Negativkontrolle aus, um die Hintergrundfluoreszenz zu bestätigen, führen Sie die regionenselektiv bestrahlte Probe aus und zeichnen Sie das Gate für die DACT1+ -Population.
- Sammeln und resuspendieren Sie die sortierten Zellen in CM-Erhaltungsmedium mit 10 % FBS und 5 μM Y27632 und säen Sie die sortierten Zellen in einer Matrigel-beschichteten 96-Well-Platte.
- Wechseln Sie am nächsten Tag das Medium auf CM-Erhaltungsmedium und warten Sie, bis Sie hochreine CMs ernten.
- ML-Strategie in Phase III: Anerkennung von CMs
Datensatz: Paare von Hellfeldbildern am Tag 12 und die endgültigen cTnT-Fluoreszenzbilder
Modell: pix2pix37- Bereiten Sie einen Datensatz vor, der aus Hellfeldbildern von CMs und den entsprechenden cTnT-Fluoreszenzbildern besteht. Um die Vielfalt zu erhöhen, stellen Sie sicher, dass der Datensatz Vertiefungen mit unterschiedlichen Differenzierungseffizienzen und Zelllinien enthält.
- Unterteilen Sie das Dataset nach dem Zufallsprinzip in einen Trainingssatz und einen Testsatz. Schalten Sie das Arbeitsverzeichnis auf "stage_III". Speichern Sie die Hellfeldbilder und die entsprechenden cTnT-Fluoreszenzbilder unter "./pix2pix/datasets/(CM|CM_new_cell_lines)/A/(train|test)/*.png" bzw. ".../B/(train|test)/*.png". Führen Sie den folgenden Befehl aus:
cd pix2pix/Datensätze
python combine_A_and_B.py --fold_A ./CM/A --fold_B ./CM/B --fold_AB ./CM/ --no_multiprocessing
CD.. - Trainieren Sie das pix2pix Model37 auf dem Trainingsset. Führen Sie den folgenden Befehl aus (Abbildung 3J):
python train.py --dataroot ./datasets/CM --name brightfield2fluorescence --model pix2pix --input_nc 1 --output_nc 1 --load_size 1536 --crop_size 256 --lr 2e-4 --n_epochs 1000 --n_epochs_decay 0 --norm instance --netD n_layers --n_layers_D 1 --batch_size 16 --direction AtoB --save_epoch_freq 100 --dataset_mode ausgerichtet --use_resize_conv --seed 1234 - Wenden Sie das trainierte pix2pix-Modell auf das Testgerät an. Führen Sie den folgenden Befehl aus:
python test.py --dataroot ./datasets/CM --name brightfield2fluorescence --model pix2pix --direction AtoB --input_nc 1 --output_nc 1 --load_size 1536 --crop_size 1536 --use_resize_conv --eval --num_test 1000 - Vergleichen Sie die vom Modell vorhergesagten Fluoreszenzbilder mit den echten (d.h. experimentell erhaltenen) Bildern. Vergleichen Sie auf Pixelebene die vorhergesagten Intensitäten und die tatsächlichen Intensitäten mithilfe des Pearson-Korrelationskoeffizienten. Vergleichen Sie außerdem auf Bildebene die vorhergesagten Differenzierungseffizienzindizes mit den tatsächlichen Indizes unter Verwendung des Pearson-Korrelationskoeffizienten. Führen Sie für diese Vergleiche "stage_III/evaluation/pixel_correlation.m" und "image_correlation.ipynb" aus (Abbildung 3K,L).
- Wenden Sie das trainierte Modell an, um CMs in neuen Chargen in Phase III zu erkennen. Geben Sie neue Hellfeldbilder von CMs an das trainierte pix2pix-Modell weiter und erhalten Sie eine In-silico-Vorhersage der Fluoreszenzbilder und der Differenzierungseffizienz.
HINWEIS: Um die Zellkontamination zu reduzieren, ist die DACT-1-basierte photoaktivierte Zellsortierung auch für die Reinigung von CMs geeignet, die vom Modell erkannt werden.
Ergebnisse
Basierend auf Hellfeld-Imaging und ML kann der gesamte Differenzierungsprozess intelligent überwacht und optimiert werden. In der PSC-Phase entwickelten wir ein ML-Modell, das die endgültige Differenzierungseffizienz entsprechend den morphologischen Merkmalen der anfänglichen PSC-Kolonien vorhersagen konnte, um den am besten geeigneten oder geeigneten Zeitpunkt für die Einleitung der Differenzierung zu bestimmen (Abbildung 4A,B). Die vom...
Diskussion
Hier haben wir ein detailliertes Protokoll beschrieben, um eines der Hauptprobleme bei der aktuellen PSC-Anwendung und -Translation zu überwinden – die Variabilität in der Zelldifferenzierung. Durch die Nutzung von Hellfeld-Imaging und ML für lebende Zellen haben wir die PSC-Differenzierung iterativ optimiert, um eine gleichbleibend hohe Effizienz über Zelllinien und Chargen hinweg zu erreichen. Im obigen Differenzierungsprozess haben jedoch mehrere kritische Schritte im Protokoll ...
Offenlegungen
Yang Zhao, Jue Zhang, Xiaochun Yang, Yao Wang und Daichao Chen melden ein Patent für die PSC-Differenzierungsstrategie an, über die in diesem Artikel berichtet wird (202210525166.X).
Danksagungen
Wir danken Qiushi Sun, Yao Wang, Yu Xia, Jinyu Yang, Chang Lin, Zimu Cen, Dongdong Liang, Rong Wei, Ze Xu, Guangyin Xi, Gang Xue, Can Ye, Li-Peng Wang, Peng Zou, Shi-Qiang Wang, Pablo Rivera-Fuentes, Salome Püntener, Zhixing Chen, Yi Liu und Jue Zhang dafür, dass sie den Grundstein für diese Strategie gelegt haben. Diese Arbeit wurde vom National Key R&D Program of China (2018YFA0800504, 2019YFA0110000) und dem Space Medical Experiment Project des China Manned Space Program (HYZHXM01020) an Yang Zhao unterstützt. Abbildung 1 wurde mit BioRender.com erstellt.
Materialien
| Name | Company | Catalog Number | Comments |
| 0.25% Trypsin-EDTA | Gibco | 25200056 | Diluted digests were used for CPC and CM digestion |
| 4% Paraformaldehyde in PBS | KeyGEN BioTECH | KGIHC016 | |
| 6-well Cell Culture Plate | NEST | 703001 | |
| 96-well Cell Culture Plate | NEST | 701001 | |
| B27 Supplement | Gibco | 17504044 | |
| B27 Supplement Minus Insulin | Gibco | A1895601 | |
| Bovine serum albumin (BSA) | GPC BIOTECH | AA904-100G | |
| Celldiscoverer 7 | Zeiss | Instruments used to take bright-field images throughout differentiation and final cTnT images | |
| CHIR99021 | Selleck | S1263 | |
| DMEM/F12 | Gibco | 12634010 | |
| Donkey anti-Mouse IgG (H+L) Highly Cross-Adsorbed Secondary Antibody, Alexa Fluor 488 | Thermo | A-21202 | Secondary Antibody |
| FACSAria III | BD Biosciences | Flow cytometry sorter | |
| Fetal Bovine Serum (FBS) | VISTECH | SE100-B | |
| Hoechst 33342 | YEASEN | 40732ES03 | |
| Human Pluripotent Stem Cell Chemical-defined Medium | Cauliscell Inc | 400105 | Basal medium of PSC preparation medium |
| iPS-18 | TaKaRa | Y00300 | |
| iPS-B1 | Cellapy | CA4025106 | |
| iPS-F | Nuwacell | RC01001-B | |
| iPS-M | Nuwacell | RC01001-A | |
| IWR1-1-endo | Selleck | S7086 | IWR1 |
| Jupyter Notebook | N/A | Version 6.4.0 | https://jupyter.org/ |
| MATLAB | MathWorks | Version R2020a | Software for scientific computation and image annotation |
| Matrigel Matrix | Corning | 354230 | Matrigel |
| Mouse monoclonal IgG1 anti-cTnT | Thermo | MA5-12960 | cTnT primary antibody |
| Normal Donkey Serum | Jackson | 017-000-121 | |
| ORCA-Flash 4.0 V3 digital CMOS camera | Hamamatsu | C13440-20CU | The digital camera assembled on Celldiscoverer7 |
| PBS | NEB | 21-040-CVR | |
| Penicillin-Streptomycin | Gibco | 15140-122 | |
| Pluripotency Growth Mater 1 basal medium | Cellapy | CA1007500-1 | Basal medium of PSC culture medium |
| Pluripotency Growth Mater 1 supplement | Cellapy | CA1007500-2 | Supplement of PSC culture medium |
| Prism | Graphpad | Version 8/9 | Statistical software for statistical analysis and plotting |
| Python | N/A | version 3.6 | Python 3 environment for scientific computation, with packages pytorch (1.9.0), numpy, scipy, pandas, visdom, scikit-learn, scikit-image, opencv-python, and matplotlib software for scientific computation and image annotation. |
| RPMI 1640 | Gibco | 11875176 | |
| Supplement hPSC-CDM (500x) | Cauliscell Inc | 00015 | Supplement of PSC preparation medium |
| TiE | Nikon | An inverted fluorescence microscope (with modification) for region-selevtive purification | |
| Triton X-100 | Amresco | 9002-93-1 | |
| Versene Solution | Thermo | 15040066 | EDTA solution for PSC digestion |
| Y27632 | Selleck | S6390 | |
| Zen | Zeiss | Version 3.1 | A supporting software of Celldiscoverer7 for image acquisition, processing and analysis |
Referenzen
- Yoshida, Y., Yamanaka, S. Induced pluripotent stem cells 10 years later: for cardiac applications. Circ Res. 120 (12), 1958-1968 (2017).
- Shi, Y., Inoue, H., Wu, J. C., Yamanaka, S. Induced pluripotent stem cell technology: a decade of progress. Nat Rev Drug Discov. 16 (2), 115-130 (2017).
- Takahashi, K., et al. Induction of pluripotent stem cells from adult human fibroblasts by defined factors. Cell. 131 (5), 861-872 (2007).
- Burridge, P. W., et al. Chemically defined generation of human cardiomyocytes. Nat Methods. 11 (8), 855-860 (2014).
- Mordwinkin, N. M., Burridge, P. W., Wu, J. C. A review of human pluripotent stem cell-derived cardiomyocytes for high-throughput drug discovery, cardiotoxicity screening, and publication standards. J Cardiovasc Transl Res. 6 (1), 22-30 (2013).
- Vanden Berg, C. W., Elliott, D. A., Braam, S. R., Mummery, C. L., Davis, R. P. Differentiation of human pluripotent stem cells to cardiomyocytes under defined conditions. Methods Mol Biol. 1353, 163-180 (2016).
- Burridge, P. W., Holmström, A., Wu, J. C. Chemically defined culture and cardiomyocyte differentiation of human pluripotent stem cells. Curr Protoc Hum Genet. 87, 1-15 (2015).
- Li, J., et al. Human pluripotent stem cell-derived cardiac tissue-like constructs for repairing the infarcted myocardium. Stem Cell Rep. 9 (5), 1546-1559 (2017).
- Smith, A. S. T., Macadangdang, J., Leung, W., Laflamme, M. A., Kim, D. -. H. Human iPSC-derived cardiomyocytes and tissue engineering strategies for disease modeling and drug screening. Biotechnol Adv. 35 (1), 77-94 (2017).
- Sharma, A., et al. Use of human induced pluripotent stem cell-derived cardiomyocytes to assess drug cardiotoxicity. Nat Protoc. 13 (12), 3018-3041 (2018).
- Mehta, A., et al. Identification of a targeted and testable antiarrhythmic therapy for long-QT syndrome type 2 using a patient-specific cellular model. Eur Heart J. 39 (16), 1446-1455 (2018).
- Cahan, P., Daley, G. Q. Origins and implications of pluripotent stem cell variability and heterogeneity. Nat Rev Mol Cell Biol. 14 (6), 357-368 (2013).
- Kim, K., et al. Epigenetic memory in induced pluripotent stem cells. Nature. 467 (7313), 285-290 (2010).
- Rouhani, F., et al. Genetic background drives transcriptional variation in human induced pluripotent stem cells. PLoS Genet. 10 (6), e1004432 (2014).
- Kim, K., et al. Donor cell type can influence the epigenome and differentiation potential of human induced pluripotent stem cells. Nat Biotechnol. 29 (12), 1117-1119 (2011).
- Garitaonandia, I., et al. Increased risk of genetic and epigenetic instability in human embryonic stem cells associated with specific culture conditions. PLoS One. 10 (2), e0118307 (2015).
- Engler, A. J., Sen, S., Sweeney, H. L., Discher, D. E. Matrix elasticity directs stem cell lineage specification. Cell. 126 (4), 677-689 (2006).
- Matoba, N., Yamashita, T., Takayama, K., Sakurai, F., Mizuguchi, H. Optimal human iPS cell culture method for efficient hepatic differentiation. Differentiation. 104, 13-21 (2018).
- Volpato, V., et al. Reproducibility of molecular phenotypes after long-term differentiation to human iPSC-derived neurons: A multi-site omics study. Stem Cell Rep. 11 (4), 897-911 (2018).
- Anderson, N. C., et al. Balancing serendipity and reproducibility: Pluripotent stem cells as experimental systems for intellectual and developmental disorders. Stem Cell Rep. 16 (6), 1446-1457 (2021).
- Sepac, A., et al. Comparison of cardiomyogenic potential among human ESC and iPSC lines. Cell Transplant. 21 (11), 2523-2530 (2012).
- Laco, F., et al. Unraveling the inconsistencies of cardiac differentiation efficiency induced by the GSK3β inhibitor CHIR99021 in human pluripotent stem cells. Stem Cell Rep. 10 (6), 1851-1866 (2018).
- Volpato, V., Webber, C. Addressing variability in iPSC-derived models of human disease: guidelines to promote reproducibility. Dis Model Mech. 13 (1), 042317 (2020).
- Hu, B. -. Y., et al. Neural differentiation of human induced pluripotent stem cells follows developmental principles but with variable potency. Proc Natl Acad Sci USA. 107 (9), 4335-4340 (2010).
- Huo, J., et al. Evaluation of batch variations in induced pluripotent stem cell-derived human cardiomyocytes from 2 major suppliers. Toxicol Sci. 156 (1), 25-38 (2017).
- Rostam, H. M., Reynolds, P. M., Alexander, M. R., Gadegaard, N., Ghaemmaghami, A. M. Image based machine learning for identification of macrophage subsets. Sci Rep. 7 (1), 3521 (2017).
- Christiansen, E. M., et al. In silico labeling: Predicting fluorescent labels in unlabeled images. Cell. 173 (3), 792-803 (2018).
- Buggenthin, F., et al. Prospective identification of hematopoietic lineage choice by deep learning. Nat Methods. 14 (4), 403-406 (2017).
- Smith, Z. D., Nachman, I., Regev, A., Meissner, A. Dynamic single-cell imaging of direct reprogramming reveals an early specifying event. Nat Biotechnol. 28 (5), 521-526 (2010).
- Fan, K., Zhang, S., Zhang, Y., Lu, J., Holcombe, M., Zhang, X. A machine learning assisted, label-free, non-invasive approach for somatic reprogramming in induced pluripotent stem cell colony formation detection and prediction. Sci Rep. 7 (1), 13496 (2017).
- Kusumoto, D., et al. Automated deep learning-based system to identify endothelial cells derived from induced pluripotent stem cells. Stem Cell Rep. 10 (6), 1687-1695 (2018).
- Zhu, Y., et al. Deep learning-based predictive identification of neural stem cell differentiation. Nat Commun. 12 (1), 2614 (2021).
- Qian, T., et al. Label-free imaging for quality control of cardiomyocyte differentiation. Nat Commun. 12 (1), 4580 (2021).
- Ounkomol, C., Seshamani, S., Maleckar, M. M., Collman, F., Johnson, G. R. Label-free prediction of three-dimensional fluorescence images from transmitted-light microscopy. Nat Methods. 15 (11), 917-920 (2018).
- Yang, X., et al. A live-cell image-based machine learning strategy for reducing variability in PSC differentiation systems. Cell Discovery. 9 (1), 53 (2023).
- Selvaraju, R. R., et al. Grad-CAM: Visual explanations from deep networks via gradient-based localization. Proc IEEE Int Conf Computer Vision (ICCV). , 618-626 (2017).
- Isola, P., Zhu, J. -. Y., Zhou, T., Efros, A. A. Image-to-image translation with conditional adversarial networks. Proc IEEE Conf Computer Vision Pattern Recognition (CVPR). , 5967-5976 (2017).
- Zhang, H., et al. ResNeSt: Split-attention networks. Proc IEEE/CVF Conf Computer Vision Pattern Recognition Workshops (CVPRW). , 2735-2745 (2022).
- Halabi, E. A., et al. Dual-activatable cell tracker for controlled and prolonged single-cell labeling). ACS Chem. Biol. 15 (6), 1613-1620 (2020).
- Hannan, N. R. F., Segeritz, C. -. P., Touboul, T., Vallier, L. Production of hepatocyte-like cells from human pluripotent stem cells. Nat Protoc. 8 (2), 430-437 (2013).
- Zhang, H., Shen, M., Wu, J. C. Generation of quiescent cardiac fibroblasts derived from human induced pluripotent stem cells. Methods Mol Biol. 2454, 109-115 (2022).
- Harding, M. J., McGraw, H. F., Nechiporuk, A. The roles and regulation of multicellular rosette structures during morphogenesis. Development. 141 (13), 2549-2558 (2014).
- Najm, F. J., et al. Rapid and robust generation of functional oligodendrocyte progenitor cells from epiblast stem cells. Nat Methods. 8 (11), 957-962 (2011).
- Hogrebe, N. J., Maxwell, K. G., Augsornworawat, P., Millman, J. R. Generation of insulin-producing pancreatic β cells from multiple human stem cell lines. Nat Protoc. 16 (9), 4109-4143 (2021).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten