Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Una estrategia de aprendizaje automático basada en imágenes de células vivas para monitorear la diferenciación de células madre pluripotentes
* Estos autores han contribuido por igual
En este artículo
Resumen
Los sistemas de diferenciación de células madre pluripotentes (PSC) a células funcionales disponibles se ven obstaculizados actualmente por problemas de variabilidad severa de línea a línea y de lote a lote. Aquí, utilizando la diferenciación cardíaca como ejemplo principal, presentamos un protocolo para monitorizar y modular de forma inteligente el proceso de diferenciación de PSC basado en el aprendizaje automático basado en imágenes.
Resumen
Las tecnologías de células madre pluripotentes (PSC) se han utilizado ampliamente en el descubrimiento de fármacos, el modelado de enfermedades y la medicina regenerativa. Sin embargo, los sistemas de diferenciación de PSC a células funcionales disponibles se ven obstaculizados por problemas de variabilidad severa de línea a línea y de lote a lote. Por lo tanto, es importante un control preciso de la diferenciación celular en tiempo real. En este protocolo, describimos una estrategia no invasiva e inteligente que supera la variabilidad en la diferenciación celular mediante el uso de aprendizaje automático basado en imágenes de campo claro. Tomando como ejemplo la diferenciación de PSC a cardiomiocitos, esta metodología proporciona información detallada para el control del estado inicial de PSC, la evaluación temprana y la intervención en las condiciones de diferenciación, y la eliminación de la contaminación celular mal diferenciada, logrando juntos una diferenciación consistente de alta calidad de PSC a células funcionales. En principio, esta estrategia puede extenderse a otros sistemas de diferenciación o reprogramación celular con múltiples pasos para apoyar la fabricación de células, así como para mejorar nuestra comprensión de los mecanismos durante la conversión del destino celular.
Introducción
Las células madre pluripotentes (PSC) poseen la notable capacidad de diferenciarse en muchos tipos de células in vitro. Estas células funcionales diferenciadas podrían utilizarse para la terapia celular, el modelado de enfermedades y el desarrollo de fármacos, todas ellas valiosas para la investigación o las aplicaciones clínicas 1,2,3. Por ejemplo, se han desarrollado una variedad de métodos para diferenciar las PSC en cardiomiocitos (CM)4,5,6,7. Estos CM se pueden aplicar para pruebas de cardiotoxicidad de fármacos, modelado de enfermedades cardíacas y trasplante de células 8,9,10,11.
Sin embargo, la conversión de PSC a las células terminales diferenciadas es un proceso escalonado, y múltiples perturbaciones durante el proceso de diferenciación pueden llevar a las células a destinos celulares divergentes. Los diferentes antecedentes genéticos y las marcas epigenéticas de las líneas PSC influyen en el potencial de diferenciación hacia un linaje específico 12,13,14,15; el número de pasajes de PSC y las mutaciones genéticas acumuladas también son fuentes de heterogeneidad de PSC; Las diferencias en las operaciones experimentales empleadas por diferentes experimentadores también pueden conducir a resultados de diferenciación completamente diferentes 16,17,18,19,20. Por lo tanto, actualmente uno de los principales problemas en la producción de células derivadas de PSC es la inestabilidad entre líneas celulares y lotes 21,22,23,24,25. La inestabilidad en la diferenciación de PSC a menudo conduce a múltiples experimentos repetidos, lo que consume importantes recursos de tiempo y mano de obra. Para abordar este problema, es crucial desarrollar una estrategia que minimice la variabilidad entre líneas celulares y lotes, mejorando así la estabilidad y robustez de la diferenciación.
Recientemente, los avances en microscopía de alta resolución y aprendizaje automático (ML) han facilitado la aplicación del análisis cuantitativo de imágenes basado en ML en biología celular, lo que permite utilizar información valiosa en características de imágenes celulares 26,27,28,29,30,31,32,33,34. En nuestro trabajo anterior, propusimos una estrategia de ML basada en imágenes de células vivas para monitorear e intervenir en el estado de diferenciación de PSC en tiempo real para mejorar la estabilidad y eficiencia de la diferenciación de PSC (Figura 1)35. Tomando como ejemplo la diferenciación de PSC a cardiomiocitos, evaluamos el estado inicial de PSC utilizando modelos de bosque aleatorio, predijimos la condición óptima de diferenciación utilizando modelos de regresión logística y reconocimos células diferenciadas con éxito utilizando Grad-CAM36 y pix2pix37 basados en aprendizaje profundo. Los modelos de ML aprendieron a identificar linajes celulares a partir de una variedad de características morfológicas de campo claro, incluidas características sobre el área, la circunferencia, la convexidad, la solidez, el brillo, la velocidad de movimiento y otras características implícitas extraídas por redes neuronales convolucionales profundas. Sobre la base de la inferencia de estos modelos de ML establecidos, logramos el control del estado inicial de PSC, la evaluación temprana y la intervención en las condiciones de diferenciación y la eliminación de la contaminación celular mal diferenciada, proporcionando en conjunto una modulación completa y precisa del proceso de diferenciación cardíaca. Aquí proporcionamos un protocolo paso a paso para desarrollar la estrategia.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Diferenciación y caracterización celular
- Preparación de reactivos de cultivo y placas de cultivo
- Prepare el medio de cultivo de PSC agregando 2 mL de suplemento y Penicilina-Estreptomicina al 0,2% a 48 mL de medio basal. Alícuota y almacene el suplemento a -20 °C. Almacene este medio a 4 °C durante un máximo de 4 semanas.
- Prepare el medio de preparación de PSC agregando 1 mL de suplemento y 0.2% de penicilina-estreptomicina a 500 mL de medio basal. Cuando lo use, precaliente el medio para un solo uso y guárdelo a 4 °C durante un máximo de 3 semanas.
- Prepare el medio de diferenciación de CM agregando 1x suplemento de B27 menos insulina y 1% de penicilina-estreptomicina en RPMI 1640. Prepare el medio de mantenimiento CM agregando 1x suplemento de B27 y 1% de penicilina-estreptomicina en RPMI 1640. Almacene estos dos tipos de medio a 4 °C durante 2 semanas.
- Sumerja el Matrigel en hielo y manténgalo a 4 °C durante la noche. Asegúrese de que Matrigel se descongele por completo; a continuación, alícuota en alícuotas de 1 mL almacenadas a -80 °C para su uso posterior, y añadir 1 mL de Matrigel en 49 mL de DMEM/F12 a 4 °C para preparar la solución de trabajo de Matrigel. Añadir 850 μL de esta solución de trabajo de Matrigel por pocillo de una placa de cultivo de 6 pocillos, e incubar la placa a 37 °C durante al menos 30 min. Para la placa de cultivo de 96 pocillos utilizada en la diferenciación de CM, agregue 35 μL de solución de trabajo Matrigel por pocillo.
NOTA: Mantenga Matrigel en hielo en todo momento; las puntas y los tubos de pipeta deben enfriarse previamente a 4 °C. La solución de trabajo de Matrigel puede almacenarse a 4 °C durante 2 semanas. Tenga cuidado de evitar burbujas al pipetear la solución de trabajo Matrigel .
- Mantenimiento y paso de PSC
- Mantenga las PSC en placas de cultivo de 6 pocillos. Confirme la densidad celular bajo un microscopio y prepárese para el paso cuando la confluencia alcance ~70% (Figura 2A).
- Use EDTA para digerir las células para el paso. Calentar el medio de cultivo PSC, PBS y EDTA en un baño de agua a 37 °C y añadir Y27632 en el medio hasta una concentración final de 5 μM.
- Lave las PSC con 1 mL de PBS, agregue 1 mL de EDTA por pocillo e incube la placa en una incubadora a 37 °C durante 3 min.
- Aspire el EDTA antes de que las células se desprendan de la placa de cultivo, luego use 1 mL de medio de cultivo PSC para enjuagar suavemente el fondo del pocillo 3-4 veces para resuspender las células.
NOTA: Esto nos permite omitir el paso de centrifugación y reducir el daño celular en las operaciones de paso. - Aspire el DMEM/F12 de un pocillo recubierto de Matrigel de una placa de cultivo de 6 pocillos y añada 2 mL de medio de cultivo PSC fresco por pocillo (Y27632 añadido). Agregue la suspensión de celdas a los pozos con una relación de división de 1:6 a 1:12. Coloque la placa en la incubadora a 37 °C con 5% de CO2.
- Cambiar el medio de cultivo PSC (sin Y27632) de 12 a 24 h después del paso; Luego, cambie el medio y verifique el estado de la celda y la confluencia todos los días.
- Diferenciación cardíaca de las PSC
- Para la diferenciación de CM, siembre las PSC en una placa de cultivo de 96 pocillos en un medio de preparación de PSC (Y27632 agregado). Para la diferenciación de CM, siga los mismos pasos anteriores (pasos 1.2.2-1.2.6) para sembrar PSC en una placa de cultivo de 96 pocillos en medio de preparación de PSC (Y27632 agregado).
NOTA: En esta etapa, la duración del tratamiento con EDTA podría extenderse adecuadamente (5 minutos o más) para disociar suficientemente las PSC de modo que los clones pudieran ser más pequeños después del paso. Nuestro estudio ha demostrado que dentro de una misma área, las colonias más pequeñas son propicias para la diferenciación. - Etapa I: Cuando las PSC alcancen el 80-90% de confluencia, cambie el medio a medio de diferenciación CM con 2-20 μM CHIR99021 (CHIR) (Figura 2B). El punto de tiempo de la conmutación del medio se denomina día 0. Después de 24-48 h de tratamiento CHIR, cambie el medio a medio de diferenciación de CM fresco.
NOTA: CHIR es un inhibidor de GSK3beta para la inducción del mesodermo. La dosis óptima de CHIR para la diferenciación de CM fluctúa con diferentes líneas celulares y lotes. Por ejemplo, la dosis óptima de CHIR es de 8-10 μM, 48 h para iPS-B1. Para recopilar diversas imágenes de campo claro para entrenar el modelo de ML, aquí valoramos las concentraciones de CHIR para cada lote de experimentos para diferentes eficiencias de diferenciación. - Estadio II: A las 72 h (día 3), sustituir el medio por el medio de diferenciación CM suplementado con 5 μM de IWR1 y cultivo durante 48 h (día 5). Cambie el medio con el medio de diferenciación CM durante 1-2 días (a los días 6-7). En este punto, las PSC se diferencian en células progenitoras cardíacas (CPC).
- Etapa III: Reemplace el medio con medio de mantenimiento CM y reemplace el medio cada 3 días. El día 10 o el día 12, coseche el CM para su posterior análisis.
- Para la diferenciación de CM, siembre las PSC en una placa de cultivo de 96 pocillos en un medio de preparación de PSC (Y27632 agregado). Para la diferenciación de CM, siga los mismos pasos anteriores (pasos 1.2.2-1.2.6) para sembrar PSC en una placa de cultivo de 96 pocillos en medio de preparación de PSC (Y27632 agregado).
- Tinción por inmunofluorescencia de MC
- El día 10 o el día 12, lavar las células con PBS y fijarlas con paraformaldehído al 4% en PBS durante 15 min a temperatura ambiente. Lave los CM 2 veces con PBS y guarde la placa a 4 °C hasta ~1 semana.
NOTA: El paraformaldehído es un reactivo tóxico con un olor acre. Opere en una capucha química y proteja los ojos y las manos. - En el momento de la tinción, tratar las células con una solución permeable (Triton X-100 al 0,1% en PBS) durante 15 min a temperatura ambiente; luego, incube las células en una solución de bloqueo (Triton X-100 al 0,1% que contiene suero de burro al 3% en PBS) durante 30 minutos a temperatura ambiente.
- Incubar la muestra con el anticuerpo primario de troponina T cardíaca (cTnT) (1:300) diluido en solución bloqueante durante la noche a 4 °C para identificar las CM.
- Recoja el anticuerpo primario y lave las células con PBS 3x. Incubar la muestra con anticuerpos secundarios en PBS con albúmina sérica bovina al 1% durante 1 h a 37 °C en un ambiente oscuro.
NOTA: El anticuerpo primario recolectado se puede reutilizar 2-3 veces en 1 semana.
- Recoja el anticuerpo primario y lave las células con PBS 3x. Incubar la muestra con anticuerpos secundarios en PBS con albúmina sérica bovina al 1% durante 1 h a 37 °C en un ambiente oscuro.
- Retire el anticuerpo secundario de las células y lave las células 3 veces con PBS. Utilice Hoechst 33342 (1:1.000 en PBS) para teñir los núcleos durante 5 minutos a temperatura ambiente. Enjuague la celda 3 veces con PBS y agregue 100 μL de PBS por pocillo para evitar que se seque. Guarde la placa a 4 °C en un lugar oscuro y adquiera imágenes fluorescentes en el plazo de 1 semana.
- El día 10 o el día 12, lavar las células con PBS y fijarlas con paraformaldehído al 4% en PBS durante 15 min a temperatura ambiente. Lave los CM 2 veces con PBS y guarde la placa a 4 °C hasta ~1 semana.
2. Adquisición del flujo de imágenes durante todo el proceso de diferenciación
- Montaje del diseño experimental para la recogida de imágenes
- Utilice un microscopio automatizado que apoye el cultivo de células vivas y la obtención de imágenes para recopilar imágenes de campo claro de las diferentes etapas de la diferenciación de la CM e imágenes inmunofluorescentes de cTnT del resultado de la diferenciación (Figura 2A-E). Utilice el software de soporte del microscopio para diseñar el programa del experimento y controlar el microscopio.
- Abra el software y cree un nuevo diseño experimental. Elija un objetivo de 5x y una lente de tubo de 2x para la toma de imágenes. Compruebe las opciones Mosaicos y Apilamiento Z.
- Agregue el canal de campo claro TL para imágenes de campo claro en el menú Canales. Agregue los canales AF488 y H3342 para la obtención de imágenes de inmunofluorescencia. Modifique la trayectoria de la luz en el menú Configuración de imágenes para obtener imágenes con la cámara CMOS digital externa.
- Abra el menú Modo de adquisición y configure la agrupación en binning 2 x 2 para aumentar la relación señal-ruido.
- Abra el menú Z-stack y elija el modelo Centro. Establezca el número de segmentos e intervalos al escanear. Elija de 3 a 5 cortes con intervalos de 3 a 6 μm para imágenes enfocadas en el análisis.
- En la ventana Navegación y mosaicos, configure las regiones de mosaico por portador y establezca 25 mosaicos (5 columnas x 5 filas) para un pozo. En el menú Mosaicos, seleccione la plantilla Multiwell 96 en Sample Carrier y establezca una superposición del 5 % al 15 % en las regiones adyacentes.
- Si se necesitan imágenes continuas, marque la opción Seriede imágenes y establezca la duración y el intervalo en el menú Series temporales. Para recoger el flujo de imágenes en la etapa I, establezca una duración de 12 h sin intervalos.
- Adquisición de imágenes de campo claro e inmunofluorescentes
- Para adquirir imágenes, primero coloque la placa de cultivo celular en la bandeja de muestras y cargue la muestra dentro del microscopio. Seleccione la plantilla Multiwell 96 para que coincida con el diseño experimental. Si la muestra consta de células vivas, abra el sistema de calefacción y la bomba de CO2 para mantener las condiciones adecuadas para el cultivo (37 °C, 5% de CO2).
- Abra el proyecto experimental preestablecido y guárdelo como uno nuevo. Abra el menú Mosaicos y calibre la posición de la placa manualmente. En la ventana Navegación y teselas, seleccione los pozos necesarios y haga clic en Crear para construir regiones de teselas para estos pozos.
- Haga clic en Verificar regiones de mosaico en el menú Mosaicos y ejecute el enfoque automático para verificar todos los pozos. A continuación, corrija manualmente el enfoque de cada pozo bajo el campo claro desplazando la rueda del ratón.
- Marque los canales necesarios y establezca los tiempos de exposición en el menú Canales. Los tiempos de exposición recomendados son 1,625 ms para Bright, 100 ms para H3342 y 550 ms para AF488. Ejecute un solo snap para comprobar las imágenes de todos los canales.
- Por último, haga clic en el botón Iniciar experimento y espere a que se generen imágenes automáticas. Por lo general, se tarda ~ 1,2 h en finalizar el escaneo de una placa de cultivo completa de 96 pocillos (96 pocillos, tres capas, 25 mosaicos para cada pocillo bajo el campo claro). En el marco de procesamiento, elija Exportación de imagen, seleccione el tipo de archivo del formato TIFF sin comprimir o el formato PNG y aplique. Utilice imágenes de 9 baldosas (3 x 3) en el centro del pozo para el análisis posterior (Figura 2A-E).
3. Establecimiento de la estrategia de ML basada en imágenes en cada etapa del proceso de diferenciación
- Preparación del software
- Instale el entorno de Python, con los paquetes pytorch (1.9.0), numpy, scipy, scikit-learn, pandas, visdom, scikit-image, opencv-python y matplotlib.
- Instale Jupyter Notebook.
- Instale MATLAB.
- Descargue el código y los conjuntos de datos de ejemplo de https://github.com/zhaoyanglab/ML-for-PSC-differentiation.
NOTA: El rendimiento del aprendizaje automático depende de la calidad de los conjuntos de datos y de la elección de los modelos y los hiperparámetros. No se garantiza que la implementación de ejemplo sea óptima para todas las circunstancias. Los ingenieros de ML deben personalizar el código y adaptar el modelo y los hiperparámetros a sus necesidades específicas.
- Estrategia de ML en la etapa de PSC: Control de los estados iniciales de las colonias de PSC
Conjunto de datos: pares de imágenes de campo claro a 0 h (antes del tratamiento CHIR) y eficiencia de diferenciación final
Modelo: bosque aleatorio- Prepare un conjunto de datos que conste de imágenes de campo claro a 0 h (antes del tratamiento CHIR) y las imágenes finales de fluorescencia cTnT. Para aumentar la diversidad de los perfiles morfológicos de las imágenes de campo claro de 0 h, asegúrese de que el conjunto de datos incluya colonias de PSC de diferentes líneas celulares, con la diferenciación iniciada durante un intervalo de tiempo después del paso. Tratar los pozos del conjunto de datos con las condiciones CHIR óptimas en la etapa I.
NOTA: Para garantizar que las colonias se traten con las condiciones óptimas de CHIR, los pocillos dentro del mismo lote se pueden tratar con dosis de CHIR tituladas. Solo se incluyen en el conjunto de datos los pozos con condiciones CHIR de alta eficiencia. - Cuantifique la eficiencia de diferenciación de cada pocillo mediante el Índice de Eficiencia de Diferenciación calculado a partir de sus imágenes de fluorescencia cTnT, que se define por
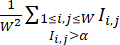 (donde
(donde 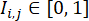 representa la intensidad de fluorescencia en la ubicación del píxel (i, j) en la imagen de fluorescencia W × W, y la suma se toma sobre todas las ubicaciones de píxeles con intensidad por encima de un umbral α, normalmente se establece en 0,5). Dentro de cada línea celular, calcule el Índice de Eficiencia de Diferenciación máximo en todos los pozos de esta línea celular y normalice sus Índices de Eficiencia de Diferenciación por el valor máximo.
representa la intensidad de fluorescencia en la ubicación del píxel (i, j) en la imagen de fluorescencia W × W, y la suma se toma sobre todas las ubicaciones de píxeles con intensidad por encima de un umbral α, normalmente se establece en 0,5). Dentro de cada línea celular, calcule el Índice de Eficiencia de Diferenciación máximo en todos los pozos de esta línea celular y normalice sus Índices de Eficiencia de Diferenciación por el valor máximo.
NOTA: Dado que el potencial de diferenciación de cada línea celular es inherentemente diferente, dicha normalización hará más hincapié en las características morfológicas de la colonia compartidas entre varias líneas celulares que contribuyen al resultado de la diferenciación. De hecho, la normalización hace que las colonias de PSC de diferentes líneas celulares sean comparables y mitiga el sesgo inducido por las líneas celulares. - Cuantificar los perfiles morfológicos de imágenes de campo claro de 0 h mediante características de alta dimensión que revelan las propiedades de la forma de la colonia. Para delinear las regiones de colonias en imágenes de campo claro, calcule la entropía local (con radio r); Los píxeles con una entropía local mayor que un umbral σ se identifican mediante celdas cubiertas. Utilice una configuración de parámetros de r = 10 píxeles y σ = 3. Cambie el directorio de trabajo a "./colony_control"; guarde las imágenes de campo claro, las imágenes binarias de las regiones celulares y las imágenes de fluorescencia cTnT en "colony_control/ image_data/CD00-*/[brightfield|cell_region|ctnt]/S*.png"; y ejecute Jupyter Notebook "Compute_features.ipynb" para calcular características relacionadas con el área de colonia, la circunferencia, la solidez, la convexidad, la circularidad y las distancias de contorno de centroide, entre otras.
- Divida aleatoriamente el conjunto de datos en un conjunto de entrenamiento y un conjunto de prueba, donde el conjunto de entrenamiento comprende entre el 50 % y el 80 % de los datos totales. Ejecute el bloque final Divida el conjunto de datos en Jupyter Notebook para dividir el conjunto de datos (figura 3A).
- Entrene un modelo de regresión de bosque aleatorio en el conjunto de entrenamiento para predecir la eficiencia de la diferenciación a partir de características de imagen de campo claro de 0 h. Ejecute Jupyter Notebook "Machine_learning.ipynb" para entrenar y probar el modelo.
- Evalúe el modelo de bosque aleatorio entrenado en el conjunto de pruebas. Confirme que la eficiencia de diferenciación predicha se correlaciona altamente con la verdadera utilizando el coeficiente de correlación de Pearson (Figura 3B).
NOTA: Dado que el conjunto de prueba no está involucrado en la fase de entrenamiento, una alta consistencia entre la eficiencia de diferenciación predicha y la verdadera en el conjunto de prueba indica que el modelo puede generalizar bien en muestras no vistas. - Obtenga la importancia de las características del modelo de bosque aleatorio entrenado y seleccione las 8 características con la mayor importancia. Visualice su relación con la eficiencia de diferenciación final dividiendo el rango de características en bins y calculando la eficiencia de diferenciación media en cada bin. Ejecute Jupyter Notebook "Feature_importance.ipynb" para realizar la visualización (figura 3C).
NOTA: La visualización puede ayudar a comprender cómo los cambios en la forma de la colonia de PSC contribuyen a la diferenciación. - Aplique el modelo entrenado en nuevos lotes para supervisar los estados de las colonias de PSC en tiempo real. Capture la imagen de campo claro cada hora, pase la imagen al modelo entrenado y obtenga la eficiencia de diferenciación predicha. Una vez que la eficiencia prevista supera un umbral (por ejemplo, 50%), comienza el proceso de diferenciación. Si la eficiencia prevista no pudo alcanzar el umbral, deseche estas muestras.
NOTA: Durante la etapa de PSC, el potencial de diferenciación de las colonias de PSC primero aumentará y luego disminuirá. El monitoreo en tiempo real permite a los experimentadores identificar el punto de tiempo más propicio para iniciar la diferenciación.
- Prepare un conjunto de datos que conste de imágenes de campo claro a 0 h (antes del tratamiento CHIR) y las imágenes finales de fluorescencia cTnT. Para aumentar la diversidad de los perfiles morfológicos de las imágenes de campo claro de 0 h, asegúrese de que el conjunto de datos incluya colonias de PSC de diferentes líneas celulares, con la diferenciación iniciada durante un intervalo de tiempo después del paso. Tratar los pozos del conjunto de datos con las condiciones CHIR óptimas en la etapa I.
- Estrategia de ML en la etapa I: evaluación temprana y ajuste de las dosis de CHIR
Conjunto de datos: pares de flujos de imágenes de campo claro a 0-12 h y etiquetas de concentración CHIR (baja/óptima/alta)
Modelo: regresión logística- Prepare un conjunto de datos que conste de secuencias de imágenes de campo claro de pozo completo, donde las imágenes se adquieren cada hora entre 0 h y 12 h. Registre las dosis de CHIR (es decir, las combinaciones de concentraciones y duración de CHIR) de cada pocillo. Asegúrese de que el conjunto de datos incluya diferentes lotes y pocillos con dosis CHIR tituladas (por ejemplo, concentración = 2, 4, 6, 8, 10 y 12 μM; duración = 24, 36 y 48 h) para aumentar la diversidad. Cambie el directorio de trabajo a "./stage_I"; guarde las imágenes como "./data/image/CD01-*/S*/T*.png", donde CD01-* (CD01-1, CD01-2, CD01-3, CD01-4) es el nombre del lote, S* (S1, S2, ..., S96) es el índice del pocillo y T*.png (T1.png, T2.png, ..., T10.png) es la imagen de campo claro preprocesada del pocillo en diferentes pasos de tiempo del flujo de imágenes.
- En cada lote, calcule el porcentaje de células cTnT+ para cada pocillo, que se calcula dividiendo el número de células cTnT+ por el número total de células Hoechst+ en la imagen de fluorescencia. Promedie el porcentaje de células cTnT+ en pocillos paralelos con la misma condición de dosis CHIR. Guarde la condición de dosis CHIR y el porcentaje de células cTnT+ para cada pocillo en un archivo csv "./data/image/CD01-*/CD01-?_exp_config.csv".
- En cada lote, determine el rango de concentración de CHIR "bajo", "óptimo" y "alto" bajo cada duración de CHIR de acuerdo con los siguientes criterios. Las concentraciones de CHIR con un porcentaje medio de células cTnT+ por encima de un umbral (por ejemplo, 20%) se identifican como "óptimas". Las concentraciones de CHIR más allá del rango óptimo se identifican como "bajas" o "altas". Debajo de cada duración de CHIR, etiquete los pozos como "bajo", "óptimo" y "alto" de acuerdo con sus concentraciones de CHIR correspondientes.
NOTA: Empíricamente, existe una correlación negativa entre las concentraciones de CHIR y la duración de los pozos de alta eficiencia dentro de un lote: para una mayor duración de CHIR, las concentraciones óptimas disminuyen. En otras palabras, la optimalidad de las concentraciones de CHIR depende de la duración del CHIR y, por lo tanto, las etiquetas de los pozos difieren entre las diferentes duraciones de CHIR. - En cada lote, bajo cada duración de CHIR, calcule la concentración de ΔCHIR para cada concentración c para cuantificar su desviación del óptimo. Denote el rango óptimo de concentración CHIR por [c1, c2] y, a continuación, la concentración ΔCHIR (c) = c - c1 (si c < c1), 0 (si c1 ≤ c ≤ c2) o c - c 2 (si c > c2).
- Extraiga las características de los flujos de imágenes en el conjunto de datos, asegurándose de que reflejen los cambios morfológicos de las células durante la etapa I. Para calcular las características de la imagen sobre la entropía local, el área, las circunferencias, el brillo, el flujo óptico y la dimensión fractal, ejecute
cd ./datos
compute_features.py Python
CD..
NOTA: Para resaltar los cambios morfológicos, los valores de algunas características (por ejemplo, área, circunferencias y flujo óptico) se pueden normalizar mediante sus valores de referencia iniciales en los flujos de imágenes. - Divida aleatoriamente el conjunto de datos en un conjunto de entrenamiento y un conjunto de prueba. Ejecute Jupyter Notebook "./data/prepare_dataset.ipynb" para dividir el conjunto de datos y organizar los conjuntos de datos como tramas de datos (figura 3D).
- Para cada duración de CHIR, entrene un modelo de regresión logística para predecir la etiqueta de concentración de CHIR (baja/óptima/alta) a partir de las características del flujo de imágenes en el conjunto de entrenamiento. Evalúe el rendimiento de la clasificación del modelo de regresión logística entrenado en el conjunto de pruebas mediante la exactitud, la precisión, la recuperación, la puntuación F1 y el área bajo la curva (AUC). Ejecute Jupyter Notebook "machine_learning.ipynb" para el entrenamiento y las pruebas (figura 3E).
- Evalúe el rendimiento del modelo en la evaluación de la dosis de CHIR. En el conjunto de pruebas, combine las etiquetas predichas de pozos paralelos con la misma concentración CHIR utilizando puntuaciones de desviación (que van de -1 a 1), definidas por
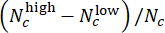 , donde Nc denota el número de pozos con concentración c y
, donde Nc denota el número de pozos con concentración c y 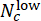 los
los 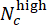 pozos entre ellos se predicen como "bajo" y "alto", respectivamente. Confirme que las puntuaciones de desviación predichas se correlacionan en gran medida con la concentración real de ΔCHIR para cada dosis de CHIR utilizando el coeficiente de correlación de Pearson.
pozos entre ellos se predicen como "bajo" y "alto", respectivamente. Confirme que las puntuaciones de desviación predichas se correlacionan en gran medida con la concentración real de ΔCHIR para cada dosis de CHIR utilizando el coeficiente de correlación de Pearson. - Realice una validación entre lotes para probar el rendimiento de generalización del modelo. En cada ronda, deje un lote para probar mientras usa otros para entrenar. Compare las puntuaciones de desviación predichas y la concentración real de ΔCHIR para cada dosis de CHIR en cada lote de prueba utilizando el coeficiente de correlación de Pearson. Ejecute Jupyter Notebook "cross_batch_validation.ipynb" para implementar la validación entre lotes (figura 3F).
NOTA: Para una duración CHIR seleccionada h, la predicción para todos los pozos con concentración del modelo de regresión logística entrenado bajo la duración h se convertirá en una puntuación de desviación, que refleja la optimalidad de la concentración CHIR c. Una puntuación de desviación cercana a 0 sugiere que se predice que la concentración c es óptima durante la duración h, mientras que una puntuación de desviación negativa o positiva sugiere una condición de dosis insuficiente o sobredosis, que puede servir como señal para el ajuste posterior de las dosis de CHIR. Tenga en cuenta que, dado que la predicción se realiza temprano (12 h), la duración final de CHIR y la concentración de CHIR durante el resto de la etapa I aún se pueden ajustar. - Aplique los modelos de regresión logística entrenados para evaluar las dosis de CHIR en nuevos lotes. Para una concentración CHIR determinada, calcule las características del flujo de imágenes de 0 a 12 h para pozos paralelos con concentración, pase las características a los modelos de regresión logística entrenados bajo diferentes duraciones CHIR y, en consecuencia, obtenga las etiquetas predichas para cada pozo. Sobre la base de la predicción de los modelos, calcule las puntuaciones de desviación para evaluar cada concentración de CHIR en diferentes duraciones de CHIR.
- Con la evaluación de la dosis de CHIR basada en modelos, rescata los pozos bajo cada concentración de CHIR subóptima en consecuencia, ajustando su duración o concentración de CHIR hacia la óptima antes de las 48 h.
- Ajuste de la duración: de acuerdo con la predicción del modelo sobre la duración óptima de CHIR, ajuste el momento en que el medio que contiene CHIR en estos pozos se cambia al medio de diferenciación CM. Si no se alcanza el punto de tiempo óptimo, deseche estas muestras.
- Ajuste de la concentración: bajo la duración preestablecida de CHIR, si se predice que la concentración actual es "baja", agregue directamente una alta concentración de CHIR en el medio para aumentar la concentración de CHIR. Diluya CHIR en DMSO (50 mM) con PBS cuando sea necesario, agregue un volumen adecuado de líquido y agite suavemente la placa. Si se prevé que la concentración de corriente es "alta", aspire un cierto volumen de medio que contenga CHIR y complemente con el mismo volumen de medio de diferenciación de CM.
NOTA: En las primeras 48 h de diferenciación, las células segregarán factores para mejorar el crecimiento y la diferenciación celular, por lo que no cambie completamente el medio. Además, es necesario mantener constante el volumen del medio en la diferenciación. Por lo tanto, asegúrese de que el volumen de líquido agregado sea lo más pequeño posible al ajustar la concentración de CHIR.
- Estrategia de ML en la etapa II: reconocimiento de los CPC comprometidos con el CM
Conjunto de datos: pares de imágenes de campo claro en el día 6 y máscaras anotadas manualmente de CPC confirmados por CM
Modelo: ResNeSt38 con Grad-CAM36 para el aprendizaje débilmente supervisado- Prepare un conjunto de datos que conste de imágenes de campo claro el día 6. Anote manualmente los CPC confirmados por CM en las imágenes de campo claro mediante el seguimiento de las celdas cTnT+ en los flujos de imágenes desde el día 12 hasta el día 6.
- Recorte las imágenes de campo claro y la anotación manual correspondiente (denominada máscaras) de los CPC confirmados por CM en parches, con cada parche con un tamaño de 1/5,5 del tamaño de la imagen de pocillo completo. Extraiga parches de forma superpuesta en los que los parches adyacentes comparten una superposición del 50 % en el conjunto de entrenamiento y una superposición del 75 % en el conjunto de pruebas. Etiquete los parches con CPC comprometidos con el ≥30 % de CM como "positivos". Etiquete los parches sin CPC confirmados por CM como "negativos". Cambie el directorio de trabajo a "stage_II". Ejecute "./Crop_and_Reconstruct/mask_crop.m" para recortar las imágenes. Ejecute "./Crop_and_Reconstruct/classification.m" para asignar etiquetas (0: negativas; 1: positivas) a los parches de campo claro (Figura 3G).
- Divida aleatoriamente los parches de imagen etiquetados en un conjunto de entrenamiento y un conjunto de prueba.
- Entrene una red neuronal convolucional profunda, ResNeSt38 , para aprender a clasificar estos parches. Evalúe el ResNeSt entrenado en el conjunto de pruebas para confirmar que el modelo demuestra una alta precisión de clasificación. Ejecute "train.py" y "test.py" para el entrenamiento y las pruebas, respectivamente (Figura 3H).
- Utilice Grad-CAM36 para resaltar las regiones que más contribuyen a la inferencia de ResNeSt, representadas por mapas de calor. Binarice los mapas de calor (mediante un umbral, por ejemplo, 10 % × máximo) para obtener las regiones CPC confirmadas por CM predichas, que se denominan regiones CPC (IR-CPC) reconocidas por imágenes.
- Compare las regiones IR-CPC con máscaras anotadas manualmente en el conjunto de pruebas utilizando la exactitud, la puntuación F1, la precisión, la recuperación, la especificidad y la intersección sobre unión (IoU). Compare la proporción de regiones IR-CPC con los índices de eficiencia de diferenciación (calculados a partir de las imágenes finales de fluorescencia de cTnT) utilizando el coeficiente de correlación de Pearson. Ejecute "./Evaluation/index_calculate.m" y "./Evaluation/Pearson_correlation.m" para la evaluación del rendimiento (Figura 3I).
- Aplique el modelo entrenado para reconocer los CPC de los nuevos lotes en la etapa II. Recorte imágenes de campo claro de pocillo completo en parches, pase los parches al ResNeSt, entrenado y obtenga las regiones IR-CPC en el nivel de parche, que luego se fusionan para producir las regiones IR-CPC de pocillo completo. Calcule la fracción de regiones IR-CPC como indicador de la eficiencia de diferenciación final. Purifique los IR-CPC para reducir la contaminación celular (descrito a continuación).
NOTA: Dado que el modelo de reconocimiento CPC puede realizar una evaluación temprana de la eficiencia de diferenciación final, también puede servir como una lectura de alto rendimiento para el cribado de moléculas pequeñas que conducen al proceso de diferenciación, lo que puede optimizar aún más el protocolo de diferenciación35.
- Purificación selectiva por región de CPC con reconocimiento de imagen
NOTA: Los experimentos de fotoactivación se basan en un microscopio de fluorescencia invertida facilitado con una platina motorizada.- De acuerdo con las regiones IR-CPC predichas por el modelo entrenado, descarte los pozos que no pudieron generar ningún IR-CPC. Para purificar estos CPC comprometidos con CM, utilice una sonda fotoactivable no citotóxica, Dual-Activatable Cell Tracker 1 (DACT-1) para etiquetar selectivamente la región que no es CPC39.
- Disuelva el DACT-1 (10 mM) en DMSO y guárdelo a -20 °C. Incubar las células del día 6 con 1 μM de DACT-1 en medio de diferenciación CM durante 30 min a 37 °C.
- Inspeccione las células vivas bajo un microscopio y seleccione la región de interés (ROI) como polígonos en MATLAB. Dibuje líneas paralelas con un espaciado de 20 μm dentro del polígono y calcule las coordenadas de los puntos de intersección.
- Establezca la pista móvil de la etapa motorizada de acuerdo con las líneas de trazado, luego la etapa se movería a 0,12 mm/s para que una línea láser de 405 nm (20 μm de diámetro) ensamblada en la plataforma pudiera escanear globalmente el ROI para fotoactivar los no CPC de manera restringida. Después de la irradiación, detecte las células marcadas con DACT-1 utilizando una línea láser de 561 nm.
- Disociar las células utilizando tripsina-EDTA al 0,05% (diluido por PBS) a 37 °C durante 5-7 min, y agitar suavemente la placa en una incubadora durante 2 min. Filtrar las células a través de un colador de células de 40 μm y centrifugar a 500 × g durante 3 min. Vuelva a suspender las células en BSA al 0,5% y mantenga el tubo en hielo hasta la clasificación. Prepare también una muestra no irradiada como control negativo para la clasificación.
- Ponga en marcha el sistema de clasificación por citometría de flujo y configure el software de clasificación. Elija los láseres de longitud de onda de 561 nm y utilice un filtro de paso de banda de 610/20 para la clasificación DACT1+. Para cada muestra, utilice una herramienta de compuerta poligonal para rodear la población de células en un gráfico bivariado de dispersión directa (FSC) y dispersión lateral (SSC) para seleccionar la celda única. A continuación, ejecute el control negativo para confirmar la fluorescencia de fondo, ejecute la muestra irradiada selectivamente por región y dibuje la puerta para la población DACT1+ .
- Recoja y resuspenda las células clasificadas en un medio de mantenimiento de CM con 10% de FBS y 5 μM Y27632, y siembre las células clasificadas en una placa de 96 pocillos recubierta de Matrigel.
- Al día siguiente, cambie el medio a medio de mantenimiento de CM y espere para cosechar CM de alta pureza.
- Estrategia de ML en la etapa III: reconocimiento de los MC
Conjunto de datos: pares de imágenes de campo claro en el día 12 y las imágenes finales de fluorescencia de cTnT
Modelo: pix2pix37- Prepare un conjunto de datos que conste de imágenes de campo claro de CM y las correspondientes imágenes de fluorescencia cTnT. Para aumentar la diversidad, asegúrese de que el conjunto de datos incluya pozos con varias eficiencias de diferenciación y líneas celulares.
- Divida aleatoriamente el conjunto de datos en un conjunto de entrenamiento y un conjunto de prueba. Cambie el directorio de trabajo a "stage_III". Guarde las imágenes de campo claro y las imágenes de fluorescencia cTnT correspondientes en "./pix2pix/datasets/(CM|CM_new_cell_lines)/A/(train|test)/*.png" y ".../B/(train|test)/*.png", respectivamente. Ejecute el siguiente comando:
CD PIX2PIX2/Conjuntos de datos
python combine_A_and_B.py --fold_A ./CM/A --fold_B ./CM/B --fold_AB ./CM/ --no_multiprocessing
CD.. - Entrena el pix2pix model37 en el set de entrenamiento. Ejecute el siguiente comando (Figura 3J):
python train.py --dataroot ./datasets/CM --name brightfield2fluorescence --model pix2pix --input_nc 1 --output_nc 1 --load_size 1536 --crop_size 256 --lr 2e-4 --n_epochs 1000 --n_epochs_decay 0 --norm instance --netD n_layers --n_layers_D 1 --batch_size 16 --direction AtoB --save_epoch_freq 100 --dataset_mode alineado --use_resize_conv --seed 1234 - Aplique el modelo pix2pix entrenado en el conjunto de prueba. Ejecute el siguiente comando:
python test.py --dataroot ./datasets/CM --name brightfield2fluorescence --model pix2pix --direction AtoB --input_nc 1 --output_nc 1 --load_size 1536 --crop_size 1536 --use_resize_conv --eval --num_test 1000 - Compare las imágenes de fluorescencia predichas por el modelo con las verdaderas (es decir, obtenidas experimentalmente). A nivel de píxel, compare las intensidades predichas y las intensidades verdaderas utilizando el coeficiente de correlación de Pearson. Además, a nivel de imagen, compare los Índices de Eficiencia de Diferenciación previstos con los verdaderos utilizando el coeficiente de correlación de Pearson. Ejecute "stage_III/evaluation/pixel_correlation.m" y "image_correlation.ipynb" para estas comparaciones (Figura 3K,L).
- Aplique el modelo entrenado para reconocer CM en nuevos lotes en la etapa III. Pase nuevas imágenes de campo claro de CM al modelo pix2pix entrenado y obtenga una predicción in silico de las imágenes de fluorescencia y la eficiencia de diferenciación.
NOTA: Para reducir la contaminación celular, la clasificación de células fotoactivadas basada en DACT-1 también es aplicable para purificar CM reconocidos por el modelo.
Access restricted. Please log in or start a trial to view this content.
Resultados
Sobre la base de imágenes de campo claro y ML, el proceso de diferenciación general se puede monitorear y optimizar de manera inteligente. En la etapa de PSC, desarrollamos un modelo de ML que pudo predecir la eficiencia de diferenciación final de acuerdo con las características morfológicas de las colonias iniciales de PSC, para determinar el momento más adecuado o apropiado para iniciar la diferenciación (Figura 4A,B). La eficiencia...
Access restricted. Please log in or start a trial to view this content.
Discusión
Aquí, describimos un protocolo detallado para superar uno de los principales problemas en la aplicación y traducción actual de PSC: la variabilidad en la diferenciación celular. Al aprovechar las imágenes de campo claro de células vivas y el aprendizaje automático, optimizamos de forma iterativa la diferenciación de PSC para lograr una alta eficiencia constante en todas las líneas celulares y lotes. Sin embargo, en el proceso de diferenciación anterior, varios pasos críticos e...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Yang Zhao, Jue Zhang, Xiaochun Yang, Yao Wang y Daichao Chen están presentando una patente para la estrategia de diferenciación de PSC informada en este documento (202210525166.X).
Agradecimientos
Agradecemos a Qiushi Sun, Yao Wang, Yu Xia, Jinyu Yang, Chang Lin, Zimu Cen, Dongdong Liang, Rong Wei, Ze Xu, Guangyin Xi, Gang Xue, Can Ye, Li-Peng Wang, Peng Zou, Shi-Qiang Wang, Pablo Rivera-Fuentes, Salomé Püntener, Zhixing Chen, Yi Liu y Jue Zhang, por sentar las bases de esta estrategia. Este trabajo fue apoyado por el Programa Nacional Clave de Investigación y Desarrollo de China (2018YFA0800504, 2019YFA0110000) y el Proyecto de Experimento Médico Espacial del Programa Espacial Tripulado de China (HYZHXM01020) a Yang Zhao. La Figura 1 se creó con BioRender.com.
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| 0.25% Trypsin-EDTA | Gibco | 25200056 | Diluted digests were used for CPC and CM digestion |
| 4% Paraformaldehyde in PBS | KeyGEN BioTECH | KGIHC016 | |
| 6-well Cell Culture Plate | NEST | 703001 | |
| 96-well Cell Culture Plate | NEST | 701001 | |
| B27 Supplement | Gibco | 17504044 | |
| B27 Supplement Minus Insulin | Gibco | A1895601 | |
| Bovine serum albumin (BSA) | GPC BIOTECH | AA904-100G | |
| Celldiscoverer 7 | Zeiss | Instruments used to take bright-field images throughout differentiation and final cTnT images | |
| CHIR99021 | Selleck | S1263 | |
| DMEM/F12 | Gibco | 12634010 | |
| Donkey anti-Mouse IgG (H+L) Highly Cross-Adsorbed Secondary Antibody, Alexa Fluor 488 | Thermo | A-21202 | Secondary Antibody |
| FACSAria III | BD Biosciences | Flow cytometry sorter | |
| Fetal Bovine Serum (FBS) | VISTECH | SE100-B | |
| Hoechst 33342 | YEASEN | 40732ES03 | |
| Human Pluripotent Stem Cell Chemical-defined Medium | Cauliscell Inc | 400105 | Basal medium of PSC preparation medium |
| iPS-18 | TaKaRa | Y00300 | |
| iPS-B1 | Cellapy | CA4025106 | |
| iPS-F | Nuwacell | RC01001-B | |
| iPS-M | Nuwacell | RC01001-A | |
| IWR1-1-endo | Selleck | S7086 | IWR1 |
| Jupyter Notebook | N/A | Version 6.4.0 | https://jupyter.org/ |
| MATLAB | MathWorks | Version R2020a | Software for scientific computation and image annotation |
| Matrigel Matrix | Corning | 354230 | Matrigel |
| Mouse monoclonal IgG1 anti-cTnT | Thermo | MA5-12960 | cTnT primary antibody |
| Normal Donkey Serum | Jackson | 017-000-121 | |
| ORCA-Flash 4.0 V3 digital CMOS camera | Hamamatsu | C13440-20CU | The digital camera assembled on Celldiscoverer7 |
| PBS | NEB | 21-040-CVR | |
| Penicillin-Streptomycin | Gibco | 15140-122 | |
| Pluripotency Growth Mater 1 basal medium | Cellapy | CA1007500-1 | Basal medium of PSC culture medium |
| Pluripotency Growth Mater 1 supplement | Cellapy | CA1007500-2 | Supplement of PSC culture medium |
| Prism | Graphpad | Version 8/9 | Statistical software for statistical analysis and plotting |
| Python | N/A | version 3.6 | Python 3 environment for scientific computation, with packages pytorch (1.9.0), numpy, scipy, pandas, visdom, scikit-learn, scikit-image, opencv-python, and matplotlib software for scientific computation and image annotation. |
| RPMI 1640 | Gibco | 11875176 | |
| Supplement hPSC-CDM (500x) | Cauliscell Inc | 00015 | Supplement of PSC preparation medium |
| TiE | Nikon | An inverted fluorescence microscope (with modification) for region-selevtive purification | |
| Triton X-100 | Amresco | 9002-93-1 | |
| Versene Solution | Thermo | 15040066 | EDTA solution for PSC digestion |
| Y27632 | Selleck | S6390 | |
| Zen | Zeiss | Version 3.1 | A supporting software of Celldiscoverer7 for image acquisition, processing and analysis |
Referencias
- Yoshida, Y., Yamanaka, S. Induced pluripotent stem cells 10 years later: for cardiac applications. Circ Res. 120 (12), 1958-1968 (2017).
- Shi, Y., Inoue, H., Wu, J. C., Yamanaka, S. Induced pluripotent stem cell technology: a decade of progress. Nat Rev Drug Discov. 16 (2), 115-130 (2017).
- Takahashi, K., et al. Induction of pluripotent stem cells from adult human fibroblasts by defined factors. Cell. 131 (5), 861-872 (2007).
- Burridge, P. W., et al. Chemically defined generation of human cardiomyocytes. Nat Methods. 11 (8), 855-860 (2014).
- Mordwinkin, N. M., Burridge, P. W., Wu, J. C. A review of human pluripotent stem cell-derived cardiomyocytes for high-throughput drug discovery, cardiotoxicity screening, and publication standards. J Cardiovasc Transl Res. 6 (1), 22-30 (2013).
- Vanden Berg, C. W., Elliott, D. A., Braam, S. R., Mummery, C. L., Davis, R. P. Differentiation of human pluripotent stem cells to cardiomyocytes under defined conditions. Methods Mol Biol. 1353, 163-180 (2016).
- Burridge, P. W., Holmström, A., Wu, J. C. Chemically defined culture and cardiomyocyte differentiation of human pluripotent stem cells. Curr Protoc Hum Genet. 87, 1-15 (2015).
- Li, J., et al. Human pluripotent stem cell-derived cardiac tissue-like constructs for repairing the infarcted myocardium. Stem Cell Rep. 9 (5), 1546-1559 (2017).
- Smith, A. S. T., Macadangdang, J., Leung, W., Laflamme, M. A., Kim, D. -H. Human iPSC-derived cardiomyocytes and tissue engineering strategies for disease modeling and drug screening. Biotechnol Adv. 35 (1), 77-94 (2017).
- Sharma, A., et al. Use of human induced pluripotent stem cell-derived cardiomyocytes to assess drug cardiotoxicity. Nat Protoc. 13 (12), 3018-3041 (2018).
- Mehta, A., et al. Identification of a targeted and testable antiarrhythmic therapy for long-QT syndrome type 2 using a patient-specific cellular model. Eur Heart J. 39 (16), 1446-1455 (2018).
- Cahan, P., Daley, G. Q. Origins and implications of pluripotent stem cell variability and heterogeneity. Nat Rev Mol Cell Biol. 14 (6), 357-368 (2013).
- Kim, K., et al. Epigenetic memory in induced pluripotent stem cells. Nature. 467 (7313), 285-290 (2010).
- Rouhani, F., et al. Genetic background drives transcriptional variation in human induced pluripotent stem cells. PLoS Genet. 10 (6), e1004432(2014).
- Kim, K., et al. Donor cell type can influence the epigenome and differentiation potential of human induced pluripotent stem cells. Nat Biotechnol. 29 (12), 1117-1119 (2011).
- Garitaonandia, I., et al. Increased risk of genetic and epigenetic instability in human embryonic stem cells associated with specific culture conditions. PLoS One. 10 (2), e0118307(2015).
- Engler, A. J., Sen, S., Sweeney, H. L., Discher, D. E. Matrix elasticity directs stem cell lineage specification. Cell. 126 (4), 677-689 (2006).
- Matoba, N., Yamashita, T., Takayama, K., Sakurai, F., Mizuguchi, H. Optimal human iPS cell culture method for efficient hepatic differentiation. Differentiation. 104, 13-21 (2018).
- Volpato, V., et al. Reproducibility of molecular phenotypes after long-term differentiation to human iPSC-derived neurons: A multi-site omics study. Stem Cell Rep. 11 (4), 897-911 (2018).
- Anderson, N. C., et al. Balancing serendipity and reproducibility: Pluripotent stem cells as experimental systems for intellectual and developmental disorders. Stem Cell Rep. 16 (6), 1446-1457 (2021).
- Sepac, A., et al. Comparison of cardiomyogenic potential among human ESC and iPSC lines. Cell Transplant. 21 (11), 2523-2530 (2012).
- Laco, F., et al. Unraveling the inconsistencies of cardiac differentiation efficiency induced by the GSK3β inhibitor CHIR99021 in human pluripotent stem cells. Stem Cell Rep. 10 (6), 1851-1866 (2018).
- Volpato, V., Webber, C. Addressing variability in iPSC-derived models of human disease: guidelines to promote reproducibility. Dis Model Mech. 13 (1), 042317(2020).
- Hu, B. -Y., et al. Neural differentiation of human induced pluripotent stem cells follows developmental principles but with variable potency. Proc Natl Acad Sci USA. 107 (9), 4335-4340 (2010).
- Huo, J., et al. Evaluation of batch variations in induced pluripotent stem cell-derived human cardiomyocytes from 2 major suppliers. Toxicol Sci. 156 (1), 25-38 (2017).
- Rostam, H. M., Reynolds, P. M., Alexander, M. R., Gadegaard, N., Ghaemmaghami, A. M. Image based machine learning for identification of macrophage subsets. Sci Rep. 7 (1), 3521(2017).
- Christiansen, E. M., et al. In silico labeling: Predicting fluorescent labels in unlabeled images. Cell. 173 (3), 792-803 (2018).
- Buggenthin, F., et al. Prospective identification of hematopoietic lineage choice by deep learning. Nat Methods. 14 (4), 403-406 (2017).
- Smith, Z. D., Nachman, I., Regev, A., Meissner, A. Dynamic single-cell imaging of direct reprogramming reveals an early specifying event. Nat Biotechnol. 28 (5), 521-526 (2010).
- Fan, K., Zhang, S., Zhang, Y., Lu, J., Holcombe, M., Zhang, X. A machine learning assisted, label-free, non-invasive approach for somatic reprogramming in induced pluripotent stem cell colony formation detection and prediction. Sci Rep. 7 (1), 13496(2017).
- Kusumoto, D., et al. Automated deep learning-based system to identify endothelial cells derived from induced pluripotent stem cells. Stem Cell Rep. 10 (6), 1687-1695 (2018).
- Zhu, Y., et al. Deep learning-based predictive identification of neural stem cell differentiation. Nat Commun. 12 (1), 2614(2021).
- Qian, T., et al. Label-free imaging for quality control of cardiomyocyte differentiation. Nat Commun. 12 (1), 4580(2021).
- Ounkomol, C., Seshamani, S., Maleckar, M. M., Collman, F., Johnson, G. R. Label-free prediction of three-dimensional fluorescence images from transmitted-light microscopy. Nat Methods. 15 (11), 917-920 (2018).
- Yang, X., et al. A live-cell image-based machine learning strategy for reducing variability in PSC differentiation systems. Cell Discovery. 9 (1), 53(2023).
- Selvaraju, R. R., et al. Grad-CAM: Visual explanations from deep networks via gradient-based localization. Proc IEEE Int Conf Computer Vision (ICCV). , 618-626 (2017).
- Isola, P., Zhu, J. -Y., Zhou, T., Efros, A. A. Image-to-image translation with conditional adversarial networks. Proc IEEE Conf Computer Vision Pattern Recognition (CVPR). , 5967-5976 (2017).
- Zhang, H., et al. ResNeSt: Split-attention networks. Proc IEEE/CVF Conf Computer Vision Pattern Recognition Workshops (CVPRW). , 2735-2745 (2022).
- Halabi, E. A., et al. Dual-activatable cell tracker for controlled and prolonged single-cell labeling). ACS Chem. Biol. 15 (6), 1613-1620 (2020).
- Hannan, N. R. F., Segeritz, C. -P., Touboul, T., Vallier, L. Production of hepatocyte-like cells from human pluripotent stem cells. Nat Protoc. 8 (2), 430-437 (2013).
- Zhang, H., Shen, M., Wu, J. C. Generation of quiescent cardiac fibroblasts derived from human induced pluripotent stem cells. Methods Mol Biol. 2454, 109-115 (2022).
- Harding, M. J., McGraw, H. F., Nechiporuk, A. The roles and regulation of multicellular rosette structures during morphogenesis. Development. 141 (13), 2549-2558 (2014).
- Najm, F. J., et al. Rapid and robust generation of functional oligodendrocyte progenitor cells from epiblast stem cells. Nat Methods. 8 (11), 957-962 (2011).
- Hogrebe, N. J., Maxwell, K. G., Augsornworawat, P., Millman, J. R. Generation of insulin-producing pancreatic β cells from multiple human stem cell lines. Nat Protoc. 16 (9), 4109-4143 (2021).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados