Method Article
使用基因组-规模CRISPR/Cas9基因屏幕进行细胞表面受体识别
摘要
本手稿描述了一种基于基因组规模的细胞筛选方法,用于识别细胞外受体-配体相互作用。
摘要
膜嵌入细胞表面受体之间的直接相互作用促进的细胞间通信对多细胞生物的正常发育和功能至关重要。然而,检测这些交互在技术上仍然具有挑战性。本手稿描述了一种系统基因组级CRISPR/Cas9敲除基因筛选方法,揭示了特定细胞表面识别事件所需的细胞通路。该测定利用哺乳动物蛋白表达系统中产生的重组蛋白作为狂热的结合探针,在基于细胞的基因筛选中识别相互作用伙伴。该方法可用于识别与膜嵌入受体的分面相对应的重组结合探针检测到的细胞表面相互作用所需的基因。重要的是,鉴于这种方法的基因组规模性质,它不仅具有识别直接受体的优点,而且还可以识别在细胞表面呈现受体所需的细胞成分,从而为受体生物学提供有价值的见解。
引言
细胞表面受体蛋白的细胞外相互作用直接重要的生物过程,如组织组织、宿主病原体识别和免疫调节。研究这些相互作用是更广泛的生物医学界感兴趣的,因为膜受体是系统传递的治疗,如单克隆抗体的可操作的目标。尽管这些互动很重要,但研究这些互动在技术上仍然具有挑战性。这主要是因为膜嵌入受体是两栖的,使它们难以从生物膜分离进行生化操作,并且它们的相互作用被弱相互作用的亲和力(μM-mM范围内的KDs)1典型化。1因此,许多常用的方法不适合检测这种类蛋白质相互作用11,2。2
已经开发出一系列方法,专门研究细胞外受体-配体相互作用,考虑其独特的生化特性3。其中一些方法包括将受体的整个异域作为哺乳动物或昆虫细胞基系统中的可溶性重组蛋白表达,以确保这些蛋白质含有在结构上很重要的翻译后修饰,如甘油和二硫化物键。为了克服低亲和力绑定,异域经常被寡头化,以增加其绑定狂热。Avid蛋白异域已成功地用作结合探针,以识别直接重组蛋白-蛋白质相互作用屏幕44、5、6、75,6,7中的相互作用伙伴。虽然广泛成功,但重组蛋白基方法要求将膜受体的异域作为可溶性蛋白质产生。因此,它仅适用于包含连续细胞外区域(例如单通道 I 型、II 型或 GPI 锚定)的蛋白质,通常不适用于多次跨越膜的受体复合物和膜蛋白。
表达克隆技术,其中补充DNA(cDNAs)库被转染成细胞,并测试结合型获得,也用于识别细胞外蛋白质-蛋白质相互作用8。近年来,大量克隆和测序的cDNA表达质粒的提供,为细胞系过度表达cDNA编码细胞表面受体的方法提供了便利,以筛选重组蛋白的结合,以识别相互作用9,9,10。与基于重组蛋白的方法不同,基于cDNA的过度表达方法提供了识别等离子膜中相互作用的可能性。然而,使用 cDNA 表达结构的成功取决于细胞以正确折叠的形式过度表达蛋白质的能力,但这通常需要细胞辅助因子,如运输机、护工和正确的寡聚物组装。因此,排泄单个cDNA可能不足以实现细胞表面表达。
使用cDNA结构或重组蛋白探针的筛选技术是资源密集型的,需要大量的cDNA或重组蛋白库。最近采用了专门设计的基于质谱的方法来识别不需要组装大型库的细胞外相互作用。然而,这些技术需要对细胞表面进行化学操作,这可以改变细胞表面分子的生化性质,目前仅适用于由糖基化蛋白11、12,12介导的相互作用。目前可用的大多数方法也严重关注蛋白质之间的相互作用,而在很大程度上忽略了膜微环境的贡献,包括糖甘、脂质和胆固醇等分子。
最近使用CRISPR方法开发高效双乙醚靶向,使缺乏定义基因的细胞的基因组库能够以系统和公正的方式筛选出涉及不同背景的细胞成分,包括解剖细胞信号过程、识别对药物、毒素和病原体产生抗药性的扰动,并确定抗体的特异性13、14、15、16。13,14,15,16在这里,我们描述了一种基于基因组规模的CRISPR敲除细胞筛选测定方法,它提供了替代当前生物化学方法,以识别细胞外受体-配体相互作用。这种通过遗传屏幕识别膜受体介导的相互作用的方法特别适用于对单个配体有关注力的研究人员,因为它避免了编译大量cDNA或重组蛋白库的需要。
该测定包括三个主要步骤:1) 高度狂热的重组蛋白结合探针,由感兴趣的受体的细胞外区域组成,产生并用于基于荧光的流细胞测量结合测定;2) 结合测定用于识别表示重组蛋白探针相互作用伙伴的细胞系;3) 生成与感兴趣的蛋白质相互作用的Cas9表达型细胞系,并执行基于基因组规模的CRISPR/Cas9的敲除屏幕(图1)。在这个遗传屏幕中,将重组蛋白与细胞系结合用作可测量的表型,其中,在挖空库中失去结合探针能力的细胞使用基于荧光的活性细胞分拣(FACS)和导致通过测序识别的结合表型丢失的基因进行排序。原则上,识别编码负责结合狂热探针的受体的基因及其细胞表面显示所需的基因。
该协议的第一步是生产代表膜结合受体的异域的avid重组蛋白探针。众所周知,当这些受体的异体被表达为重组可溶性蛋白1时,它们经常保持其细胞外结合功能。对于感兴趣的蛋白质,可溶性重组蛋白可以在任何合适的真核蛋白表达系统中以任何格式产生,前提是它可以进行寡聚,以增强结合性,并且它包含可用于荧光流细胞测定(例如,FLAG-tag,生物素标签)的标签。使用HEK293蛋白表达系统生产膜受体可溶性异域的详细协议,以及不同的多美化技术和用于生产五角蛋白和单体蛋白的蛋白质表达结构,此前已描述过,1,17。此处的协议将描述从单体生物微化蛋白中产生荧光avid探针的步骤,将它们结合到氟铬(例如,植物素或PE)中,这些链球菌结合在一起,可以直接用于基于细胞的结合测定,其优点是不需要二次抗体进行检测。执行基因组尺度屏幕的一般协议已经描述了20,21,因此该协议主要侧重于使用CRISPR/Cas9敲除筛选系统使用CRISPR/Cas9敲除筛选系统使用人类V1("Yusa")库18执行流细胞学重组蛋白结合屏幕的细节。20,
研究方案
1. 生产并纯化生物微化的蛋白质
- 使用哺乳动物或昆虫细胞蛋白表达系统来产生可溶性重组,他标记的生物微化蛋白(见表1中的质粒结构)。
注:Kerr等人17日描述了使用HEK293细胞表达系统生产单体生物素和他标记的蛋白质的详细协议。使用HEK293表达系统表达的蛋白质异域被分泌到培养介质中。 - 通过在3,000 x g的离心中使细胞颗粒20分钟,收集可溶性蛋白质。
- 通过0.22 μm过滤器过滤上清液,以1:1,000的比例将Ni2+-NTA琼脂珠添加到过滤蛋白上清液中(即50 μL的50%琼脂浆加入50 mL的上清液)。在旋转平台上在旋转平台上在 4°C 下孵育过夜或至少 4-5 小时。
- 通过添加 5 mL 的他净化洗涤缓冲液来清洗聚丙烯柱。有关所有缓冲区组合,请参阅表 2。
- 将整个珠蛋白上清液混合物倒入柱中。珠子会积聚在底座上。
- 用 15 mL 的洗涤缓冲液洗涤 2 倍的珠子。为避免蛋白质稀释,请用5 mL注射器小心地从柱上抽取残留洗涤缓冲液并丢弃。
- 小心地将300-500μL的他净化洗脱液缓冲液直接加入珠子,孵育至少1小时。 再次使用1 mL注射器仔细提取液体,收集洗脱的蛋白质。使用脱盐列将洗脱缓冲液交换到所需的缓冲器(例如,通常为 PBS 或 HBS)。将所有蛋白质储存在4°C,直到进一步使用。
2. 单聚物生物微化蛋白的定量和寡位化
注:为了增加结合性,在结合测定中使用四聚苯三甲苯单体蛋白之前,在四聚苯三甲苯酚-PE上进行寡聚。通过测试一系列生物微化单体对链球菌的固定浓度的稀释系列,并凭经验确定不检测过量生物化单体的最低稀释量,实现单体蛋白和四聚苯三酚-PE的最佳结合比率。
- 使用适当的稀释缓冲液(PBS或HBS,含有1%牛血清白蛋白[BSA])在96孔板中,对生物微化蛋白样品进行至少8次连续稀释。确保每次稀释的最终体积至少为 200 μL。
- 通过从每口井中取出 100 μL 并转移到新的 96 井板中,制作样品的重复板。始终包含控件。在这种情况下,对照是仅标记蛋白(即生物微值的他标记的Cd4域3+4蛋白)。这将用作所有绑定检测中的控制探头。
- 稀释稀释缓冲液中的链球菌-PE至0.1微克/米。
- 在其中一个板上,加入稀释性链球菌-PE的100μL。重复的板将作为控件。在控制板中加入 100 μL 稀释缓冲液,以平衡体积。
- 在室温 (RT) 下孵育 20 分钟。同时,用稀释缓冲液将链球菌涂层板的孔块15分钟。
- 将样品的总量从两个板转移到链球菌涂层板的单个孔中,并在RT孵育1小时。
- 用 200 μL 洗涤缓冲液(即 PBS 或 HBS,用 0.1% 补间-20,2% BSA)清洗板 3x。加入100 μL 2 μg/mL小鼠抗大鼠Cd4d3+4 IgG (OX68),在RT孵育1小时。
- 用洗涤缓冲液清洗板 3 倍。在RT时,在0.2微克/米线下加入100 μL的抗小鼠碱性磷酸酶偶联。
- 用洗涤缓冲液清洗板 3 倍,在稀释缓冲液中洗涤 1 倍。
- 在二乙醇胺缓冲液中以1mg/mL制备p-硝基磷酸酯。在每个井中加入100 μL,孵育15分钟。
- 在 405 nm 处进行吸水性读数。使用板上没有信号的最小稀释作为适当的稀释系数来创建四重体(图2)。
- 通过孵育4微克/mL链球菌-PE和适当的生物微化蛋白稀释30分钟,为所有样品和控制制作10倍四足联染色液。
3. 流细胞测量基细胞结合测定
- 对于粘附细胞,去除培养介质,用PBS洗涤1倍,无需二价阳离子。然后添加细胞分离解决方案(例如 EDTA)。让细胞分离5-10分钟。轻轻敲击烧瓶以释放细胞。
注:避免使用基于胰蛋白酶的产品,因为它们可以切下细胞表面的蛋白质。 - 将分离的细胞收集到管子中。对于悬浮细胞(例如HEK293细胞),直接从培养瓶中收集细胞到管中。
- 在200 x g下颗粒细胞5分钟。取出上清液,重新悬浮洗涤缓冲液中的颗粒(即PBS/1%BSA)。
- 使用血细胞计对细胞进行计数,并将浓度调整为 2.5 x 105-1 x 106细胞/mL。在 96 孔 U 或 V 底板上的制备细胞混合物的阿利quot 100 μL。在 400 x g下旋转板 5 分钟。使用多通道移液器拆下上清液。
- 加入100 μL的标准化荧光标记高度狂热的蛋白质探针和控制到以前准备的板与细胞和孵育1小时在4°C。装订 1 小时后,以 400 x g旋转板 5 分钟。
- 取出上清液,加入200μL的洗涤缓冲液(即PBS/1%BSA)。通过上下移液很好地混合。
- 在400 x g的离心下将细胞中丸5分钟。重复洗涤步骤1倍。两次下水后,完全去除上清液,并在100μL的PBS中重新悬浮细胞颗粒。
- 通过流细胞测定分析细胞。使用黄绿色激光(即 561 nm)检测 PE 荧光。
- 首先分析被控制探头染色的细胞。根据 PE 荧光的分布,为绑定总体绘制一个门,使不超过 1% 的控制单元落在此门中。
- 分析样本并确定位于绑定门中的单元格的分数。
注:基因屏幕需要显示较高结合群的细胞线,因为它们的信号噪声比较高。理想情况下,超过80%的细胞应该属于这个门。
4. 确定热拉比平类表皮和肝素硫酸盐侧链的结合贡献
注:许多蛋白质的活性是热粘结的,因此热处理后结合活性的丧失是令人鼓舞的。建议确定负电荷甘油糖,主要是肝素硫酸盐(HS)对重组蛋白的分量的贡献。这是因为HS在细胞结合测定中的结合可以添加,而不是共同依赖于其他受体19。这意味着观察到的结合可以完全由细胞表面蛋白酶的HS侧链进行调节,而不是由特定的受体调节。在细胞表面与HS结合不一定是非特异性的,而是蛋白质的特性,在进行全遗传筛查之前,了解这种特性是很有用的。
- 准备经过热处理的蛋白质样品,用于结合测定。
- 在80°C下加热正态但未结合的单体蛋白10分钟。
- 将热处理的蛋白质与链球菌-PE结合,假设与ELISA确定的未经处理的抗压蛋白具有相同的比例(参见第2节)。
- 准备肝素阻断的蛋白质样本。
- 在PBS中制备8个1:3稀释可溶性肝素,起始浓度为2mg/mL,最终体积为100μL。
- 在肝素稀释液中孵育100 μL的制备结合探头至少30分钟。
- 在第3节所述的结合测定中使用热处理蛋白和全200μL的肝素/蛋白质混合物。代表结果如图3A、B所示。
5. 选择单元格系,稳稳地表达 Cas9
注:在绑定感兴趣的探头的细胞系可用于CRISPR筛选之前,必须首先对其进行设计以表示Cas9核酸酶和选定的高活性克隆19。
- 使用以下通用的扁病毒生产协议,使用 Cas9 表达式的慢病毒构造生成扁病毒(参见表 1)。
- 在37°C和5%CO2下培养DMEM/10%FBS介质中的HEK293-FT细胞。种子HEK293-FT细胞在转染前1天,使其在转染当天的80%汇入。
注:HEK293FT细胞粘附松散;因此,当它们用于生产扁病毒时,请考虑在涂有0.1%(w/v)明胶的培养瓶上电镀,以增加依从性。 - 在早上执行转染。将转移载体、包装混合和转染试剂添加到预热转染兼容介质(例如 Opti-MEM)。通过反转管 10-15x 混合。在 RT 孵育 5 分钟。有关确切卷,请参阅表 3。
- 按制造商建议添加转染试剂。通过快速涡旋混合。在RT孵育30分钟。
- 非常小心地吸气所花的介质。将转染兼容介质添加到板中。
- 将转染试剂/DNA复合物滴到板的一侧,然后非常轻柔地旋转,缓慢地通过板展开。
- 在37°C孵育3-5小时,用D10介质代替介质。孵化过夜。
- 第二天早上,用新鲜的D10介质替换介质。孵化过夜。
- 第二天下午晚些时候,收集病毒上清。带低蛋白结合的 0.45 μm 过滤器的过滤器。可以选择添加新鲜的 D10 介质,孵育过夜,并在第二天回忆上清剂。
- 病毒超钠在4°C下稳定,只有几天。储存在-80°C,长期储存。
注:为了产生高度浓缩的慢病毒制剂,对于难以转导的细胞来说,超级亚质也可以通过在4°C的6,000 x g过夜的离心浓缩。用耐乙醇的钢笔标记半透明的病毒颗粒,并丢弃上清液。将颗粒重新悬浮在原始体积的 1/100 中,浓度增加 100 倍。
- 在37°C和5%CO2下培养DMEM/10%FBS介质中的HEK293-FT细胞。种子HEK293-FT细胞在转染前1天,使其在转染当天的80%汇入。
- 用扁病毒转导细胞。
- 板 1 x 106细胞每孔在 6 孔板与 3 mL 适当的培养介质。有些细胞比其他细胞更容易被转化。为了便于转导细胞(例如HEK细胞),将慢病毒直接添加到细胞中。对于难以转导的细胞,可能需要遵循如下所述的自旋协议。
- 在 15 mL 锥形管中,阿利quot 2 mL 的 2-5 x 106个单元/mL。
- 将扁子病毒与8μg/mL六溴二苯甲酰胺一起加入,并在RT孵育30分钟。
- 在 32 °C 下,在 800 x g下离心 100 分钟。然后,将细胞重新悬浮在同一介质中,并将细胞悬浮液加入适当的培养瓶中,并带有适当的培养素。
- 允许转导至少24小时。之后取出含有病毒的介质,并添加新鲜的介质。
- 再过24小时后,将介质更改为辅以适当抗生素的介质。Cas9 构造包含用于选择的胚砂丁电阻盒。
注:必须通过执行剂量响应杀灭曲线,针对每个细胞系优化砂精的含量。2.5-50微克/mL之间的胚砂素浓度应在转染后10天内杀死大多数未转导细胞系。
- 板 1 x 106细胞每孔在 6 孔板与 3 mL 适当的培养介质。有些细胞比其他细胞更容易被转化。为了便于转导细胞(例如HEK细胞),将慢病毒直接添加到细胞中。对于难以转导的细胞,可能需要遵循如下所述的自旋协议。
- 执行选择,直到控制板中的所有细胞(即,使用相同浓度选择抗生素治疗的非转导细胞)被杀死。
6. 选择高 Cas9 活动克隆
注:多克隆Cas9可用于成功执行遗传屏幕;然而,选择具有高Cas9活性的克隆可以提高筛选结果18。
- 使用限制稀释或单细胞排序单个抗拉他丁细胞进入三个96孔板的孔中,含有补充的培养基,补充了胚砂素。克隆将在2-4周内开始出现。选择 10-20 个克隆,并在 6 个孔板中展开。
- 使用快速评估GFP-BFP(绿色荧光蛋白-蓝色荧光蛋白)系统对Cas9活动的克隆进行测定,该系统使用外源基因敲除系统,其中细胞通过用GRNA靶向GRNA或空gRNA作为对照18的构造进行转化。
- 订购报告器质粒:GFP-BFP质粒,对照-BFP质粒(表1)。
- 使用第 5.1 节中所述的慢病毒生产协议,为 GFP-BFP 质粒和控制-BFP 质粒生产慢子病毒。
- 分别用小病毒编码GFP-BFP系统和控制-BFP,将每个Cas9表达的细胞系克隆进行转录。遵循第 5.2 节中的协议。
- 转导3天后,使用流细胞测定检查每个克隆的GFP-BFP荧光。分别使用 488 nm 激光和 405 nm 激光检测 GFP 和 BFP。
- 通过仅检查 BFP 与 GFP-BFP 双正细胞的比率来量化每个克隆中的 Cas9 活性。高活性 Cas9 单元最好具有 >95% GFP 敲除效率(图 4)。
7. 生成全基因组CRISPR-Cas9筛选淘汰库
- 对于使用人类 V1 库18进行全基因组筛选,请订购全基因组库(参见表 1),并使用制造商手册中"图书馆复制协议"下提供的协议从细菌刺中准备质粒库。
- 使用全基因组库质粒制剂,利用第5.1节所述的慢病毒生产协议,生成编码gRNA的慢病毒库,有针对性地破坏人类基因。
注:一种好的做法是生产一批针对转导进行优化的慢病毒制剂,以提高实验的一致性。 - 使用第 5.2 节中的转染协议执行小规模测试转染,以确定每个细胞系所需的病毒量,以实现 30% 的转导。使用流细胞测定来评估 BFP 荧光作为转导效率的代理。
- 要转导HEK293细胞,只需将预定的慢病毒制剂添加到正常生长培养的30-50 x 106细胞中,持续4小时。然后用扁病毒去除介质,代之以新的生长介质。
- 对于其他细胞系,使用第 5.2.1 节中的自旋协议,但规模较大,因此总共可换出 30-50 x 106个细胞。为此,在 15 mL 锥形管中,从 5 x 106个单元/mL的分配额 2 mL 进行,并按照指示继续。
- 对于粘附细胞系,通过在转导后24小时加入紫杉霉素来选择转导细胞。
注:通过执行剂量反应杀灭曲线来优化紫霉素浓度。通常浓度在1-10微克/米线之间应在3-5天内杀死非转导细胞。避免使用更高浓度的紫霉素,因为这可能增加选择由多个单一引导RNA(sgRNA)转导的细胞的机会。 - 对于悬浮细胞,使用细胞分拣机收获转导细胞(即BFP阳性)细胞3天后,并生成至少包含10 x 106个细胞的库。一旦选择使用BFP,生长在介质中的细胞补充适量的紫杉霉素。
注:避免选择仅用紫霉素进行悬浮细胞系,因为很难去除悬浮细胞培养物中可能干扰细胞分泌的死细胞和碎屑。 - 文化突变库在转染后9-16天,每2-3天定期通过一次。
8. 细胞表面结合的基因筛选
- 将突变细胞库在 200 x g下 5 分钟,重新悬浮在 PBS 中的细胞。
- 将细胞分成两个 15 mL 锥形管,每个管中至少有 50 x 106个细胞。
- 在 200 x g下旋转一个锥形管 5 分钟,去除上清液,并在 -20°C 处冷冻细胞颗粒。这是控制填充,稍后将处理。
- 以 10 mL 的 PBS/1% BSA 重新悬浮另一管中的颗粒。将 100 μL 的细胞作为 96 井板上的负控制放在一边。
- 将适当的预结合重组蛋白添加到锥形管中的细胞悬浮液中,将负对照蛋白添加到96孔板中。
- 在台式转子上以 4°C 执行细胞染色至少 1 小时,轻轻旋转 (6 rpm)。
- 在200 x g下将细胞切碎5分钟,去除上清液。执行两个洗涤步骤,然后在 5 mL PBS 中重新挂起细胞。
- 通过30μm细胞滤网对细胞进行应变,以去除细胞簇。使用流分拣机进行分析。
- 使用负控制样本对 BFP+/PE- 单元格进行门控。
- 对样品进行排序并收集 BFP+/PE- 单元格。分门取决于细胞与蛋白质的结合,但通常收集PE阴性样本的1-5%。在补充图 1中提供了分拣门的示例。
- 从所选门中收集 500,000-1,000,000 个单元格。鉴于细胞数量少,请考虑在 1.5 mL 离心管中采集样品,以尽量减少损失。
- 在500 x g上离心5分钟,将分拣的细胞切碎5分钟。小心地去除上清液并丢弃。可以在 -20°C 下储存颗粒长达 6 个月。
9. 基因组DNA提取和gRNA富集的第一个PCR
- 从高复杂性控制种群中提取基因组DNA。
- 如果控制群在-20°C下冻结,则拿出锥形管并添加PBS。保持冰上解冻颗粒。
- 使用商业套件(参见材料表),使用制造商的建议从 50 x 106 细胞中提取基因组 DNA。将DNA浓度调整到1毫克/mL。
- 对于每个样品,为PCR设置一个与72μgDNA对应的主混合物。在96口PCR板的36口井中,每口井报价50μL。所需的引底序列列在表 4中。使用表 5和6 中的指南。
- 在2%(w/v)琼脂胶凝胶上从6-12个有代表性的样品中解析5μL的PCR。应观察到 ±250 bp 的单个透明带。如果频带昏暗,重复 PCR,再进行 2-3 个循环。
- 使用多通道移液器从每口井收集 5 μL 的 PCR 产品(总计 180 μL),并将其汇集在具有 900 μL 结合缓冲液从商业套件的储液罐中(参见材料表)。
- 使用商用 PCR 净化套件净化 PCR 产品。从商业试剂盒中分离出50μL的洗脱缓冲液(参见材料表),并测量DNA浓度。
- 已为丢失绑定表型而排序的样本不太可能由大量独立克隆组成。因此,没有必要用72μg的DNA进行PCR。使用适当的商业套件分离DNA(参见材料表)。使用之前所述的协议(第 9.1.3 节)使用 100 纳克/μL DNA设置 3-4 PCR 反应。如果排序的细胞数小于 100,000,请使用细胞水清,而不是基因组 DNA 制剂。
- 在96孔PCR板中,阿利quot约10,000个细胞/孔。
- 将细胞切在板中,并小心地去除大部分上清液。颗粒不可见。
- 在每个井中加入25μL的水,在95°C下加热样品10分钟。
- 将5 μL的2mg/mL新鲜稀释的蛋白质酶K加入每个井1小时,并在56°C孵育。然后在95°C下加热样品10分钟,使蛋白酶K失去活性。
- 每次PCR反应使用10μL的细胞乳酶混合物。电解器应在 24 小时内使用。
10. 索引条码和测序的第二轮 PCR
- 将产品从第一轮 PCR 稀释到 40 皮克/μL。
- 每个样本设置一个 PCR(使用表 7和表 8中提供的指南)。使用高保真聚合酶对于最大限度地减少聚合酶在sgRNA扩增过程中引起的误差非常重要。
- 在 2% (w/v) 琼脂胶凝胶上解决 5 μL 的 PCR 产品。应观察到 ±330 bps 的单个透明带。
- 使用顺磁珠纯化PCR产品,在PCR产品中加入31.5μL(总体积为0.7倍)的重悬浮珠,混合良好,并在RT孵育5分钟。
- 将管子放在磁性机架上 3 分钟。珠子应捕获在板的一侧,溶液应清晰。小心地取出并丢弃上清液。
- 在管子中加入150μl80%新鲜制备的乙醇。孵育30s,然后小心地取出并丢弃上清液。
- 重复步骤 13.6,这一次为 180 μL。然后空气干燥珠子5分钟。
- 从磁铁上拆下管。从珠子到35μL无菌EB缓冲液的EluteDNA靶点。孵育3分钟,然后将管子放回磁架3分钟。
- 将含有洗脱的PCR产物的上清液约30μL转移到干净的管中。
- 在下一代测序平台上对样品进行排序。对于 HumanV1 gRNA 库,请使用表 4中列出的自定义底漆来序列 19 bp。
11. 生物信息学分析,以确定受体和相关途径
- 使用 MAGeCK 的计数函数将序列从排序和未排序的填充映射到参考库。该函数将生成原始计数文件(补充表 1)。
注:关于MAGeCK的安装和MAGeCK内不同功能的使用的详细说明,在王等人20日先前发布的协议中作了描述。 - 检查屏幕中使用的控制库的技术标准。
- 中位数规范化原始计数,并使用 R21或等效软件中的ggplot2包绘制质粒中计数的实证累积密度函数图并控制未排序的样本(图 5A)。
- 使用质粒计数作为"控制"运行 MAGeCK 的-测试函数,将未排序控制样本中的计数作为"测试"样本运行。函数将生成一个基因摘要文件(补充表 1)。
- 打开基因摘要文件,绘制先前分类的基本和非必需基因的日志折变化(neg_lfc列)的分布(图5B)。
- 选择显著耗尽的基因(neg_fdr < 0.05),并使用浓缩剂23包或任何等效的通路浓缩包R(图5C)进行通路浓缩分析。
- 使用默认设置运行 MAGeCK 的-test函数。使用未排序控制样本的原始计数作为"控制",并在执行分析时将排序样本计数计为"处理"。
- 打开 MAGeCK 生成的基因摘要文件,按升序对排名列进行排名。使用FDR(pos_fdr列)< 0.05 作为识别命中的截止点。 FDR受体通常排名很高,通常处于第一位置。
- 在 R 或等效软件中绘制正选择(pos_score)的强通排名算法 (RRA) 分数(图 5D)。
- 选择基因命中,并使用浓缩包或R中的任何等效通路浓缩包进行通路浓缩分析,以识别浓缩途径。
结果
提供了两个有代表性的基因组尺度敲除屏的测序数据,以识别分别在NCI-SNU-1和HEK293细胞中执行的人类TNFSF9和P.恶性疟原虫RH5的结合伙伴(补充表1)。RH5的结合行为受到硫酸盐及其已知受体BSG24(图3C)的影响,而TNFRSF9专门与其已知的受体TNFSF9结合,在预孵化时不失去与可溶性肝素的结合。图3B中的蛋白质3代表TNFRSF9。
对于两个细胞系,还提供3天(9天、14天和16天后)在对照突变库中分布gRNA(补充表1)。gRNA分布表明,整个实验过程中,库的复杂性一直保持(图5A)。TNFSF9配体鉴定的基因筛选在转染后的第14天进行,而RH5的遗传筛选则在第9天进行后转染。通过检查观察到的gRNA褶皱变化的分布情况,针对一组非必要基因的分布与基本基因参考组分布22(图5B)相比,评估了屏幕的技术质量。此外,路径级浓缩还表明,在将对照样本与原始质粒库进行比较时,在"辍学"种群中确定了预期的基本途径并显著丰富了。图 5C中描述了第 14 天 NCI-SNU-1 示例的示例。
使用MAGeCK的-测试函数(关于MAGeCK的基因摘要输出,请参阅补充表1)在对照组与排序的种群中分布gRNA用于识别表型屏幕中的受体。MAGeCK在基因级分析中报告的修正RRA分数根据按p值排列的基因进行绘制。MAGeCK 中的 RRA 分数提供了 gRNA 排名始终高于预期的度量。在 TNFRSF9 的屏幕上,点击量最大的是 TNFSF9,它是 TNFRSF9 的已知绑定伙伴(图 5D)。此外,还确定了与TP53通路有关的一些基因。在RH5的情况下,除了已知的受体(BSG) 和产生硫酸盐的 GG (SLC35B2) 所需的基因外,还确定了另一个基因 (SLC16A1) (图 5E)。SLC16A1 是将BSG贩运到细胞表面所需的陪同人员。这些结果共同证明了屏幕能够识别直接相互作用的受体以及该受体以功能形式在细胞表面表达所需的细胞成分的能力。
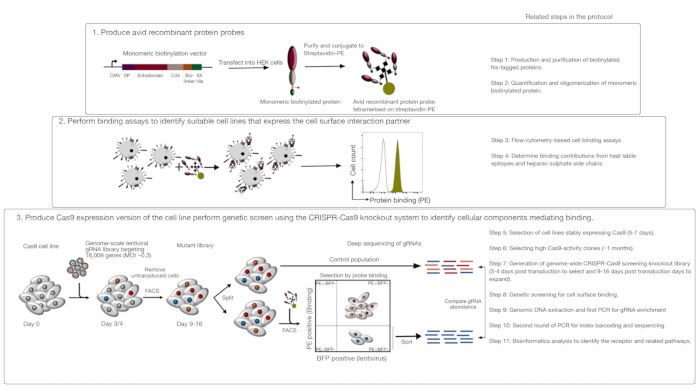
图1:识别细胞表面受体的基因筛选方法概述。此测定包括三个主要步骤:首先,代表细胞表面受体的异域的重组蛋白以细胞线表示,该细胞系可以添加结构上关键的翻译后修饰,如 HEK293 细胞。单体蛋白异域通过结合链霉菌-PE来增加其结合性。其次,这些狂热的探针用于细胞结合测定,其中PE荧光(绿色)明显变化指示的细胞系上的明亮染色与负对照蛋白(黑色)相比,表明存在细胞表面结合伙伴。第三,选择受体阳性Cas9表达细胞系,并使用针对绝大多数蛋白质编码基因的gRNA进行基因组规模筛选。在生成突变库时,通常使用 30% 的转导效率,这是基于泊松分布概率,确保每个细胞接收单个 gRNA,以便生成的表型归因于特定的敲除。传感器表示的BFP标记用于使用FACS选择含有gRNA的细胞。色情屏幕在转导后 9-16 天内执行。在屏幕的当天,突变细胞总群分为两部分。一半作为对照种群保留,另一半被选择用于重组蛋白结合。突变库中不再能够结合重组蛋白的细胞使用FACS进行排序,在排序与对照人群中的gRNA的丰富用于识别标记的avid探针细胞表面结合所需的基因。指示协议中需要大量时间的步骤。这个数字是从夏尔马等人19.请点击此处查看此图形的较大版本。
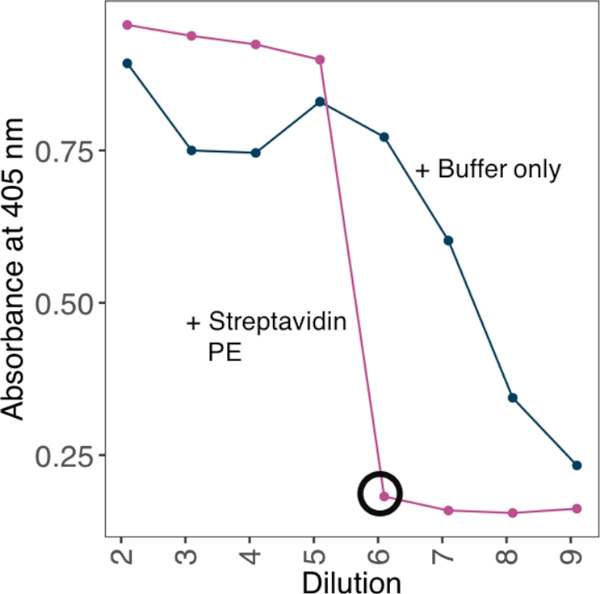
图2:使用基于ELISA的方法确定生物微化蛋白与链球菌蛋白-PE的比率。链球菌-PE结合策略的一个例子,用于从生物微化单聚蛋白中产生一个狂热的探针。一系列稀释性生物微化单体被孵育成固定浓度的链球菌。ELISA确定了无法检测到过量生物微量单体的最低稀释量。ELISA 是执行有或没有预孵一系列蛋白质稀释与 10 纳克链球菌-PE。在存在链球菌-PE时,计算了未识别信号的最小稀释度(圆形黑色)和饱和所需的蛋白质量,以生成具有4 μg/mL链球菌-PE的10倍库存溶液。请点击此处查看此图形的较大版本。
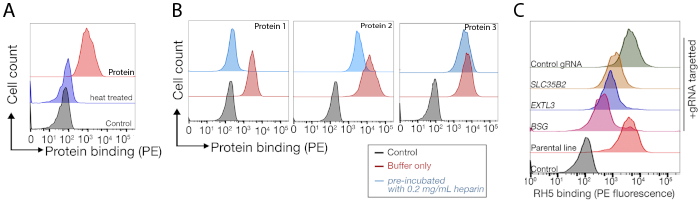
图3:蛋白质与细胞系的代表性结合。与对照样本相比,与细胞系结合的蛋白质荧光明显增加。重组蛋白的热处理(80°C,10分钟),所有结合都回对负对照,表明结合行为依赖于正确折叠的蛋白质。(B) 与细胞表面不同类别的蛋白质结合行为;依赖GG。从左到右,蛋白质可以分为三种类型:蛋白质类型1只吸附HS。这些蛋白质在预孵育后失去其结合,肝素浓度超过0.2mg/mL。蛋白质类型 2 与 HS 结合,除了特定的受体。这些蛋白质在预阻塞实验中失去部分结合。蛋白质类型 3 不绑定 HS。与亲线相比,这些蛋白质不会失去结合。(C) 以添加剂方式与 HS 和特定受体结合的蛋白质(即 RH5)的示例。靶向受体(即BSG)或HS合成所需的酶(例如SLC35B2、EXTL3),仅部分减少RH5与对照对照的细胞结合。转导多克隆线可用于此类实验,以建立结合行为。这个数字是从夏尔马等人19.请点击此处查看此图形的较大版本。
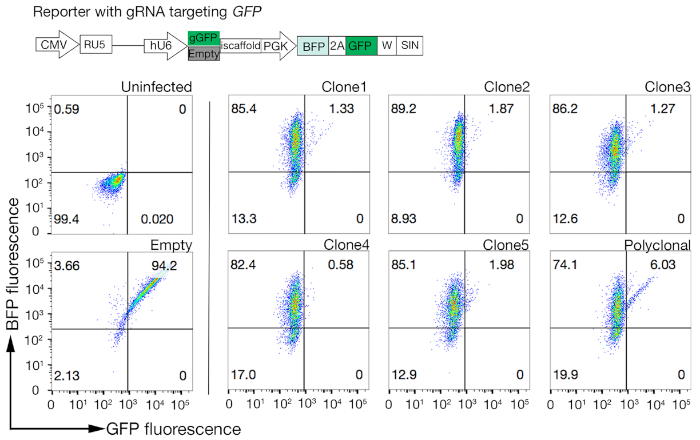
图 4:选择具有高 Cas9 活性的克隆细胞系。使用GFP-BFP报告系统评估了NCI-SNU-1细胞系多克隆线和克隆系的基因组编辑效率,其中细胞系与病毒进行转导,病毒具有gRNA靶向质粒编码的GFP或没有(即"空")。图解图被描绘出来。流细胞学用于在转导后测试BFP和GFP表达式,并将其与未感染的控制进行比较。GFP 表达式用作 Cas9 活动的代理,而 BFP 表达式标记了传感器。未感染和空受感染的细胞的配置文件看起来与所有克隆相似。代表配置文件在左侧面板中描述。与多克隆线(右面板)相比,NCI-SNU-1细胞系的五个克隆都显示GFP损失较高,克隆4具有最高的效率,耐火材料总体最少。这个数字是从夏尔马等人19.请点击此处查看此图形的较大版本。
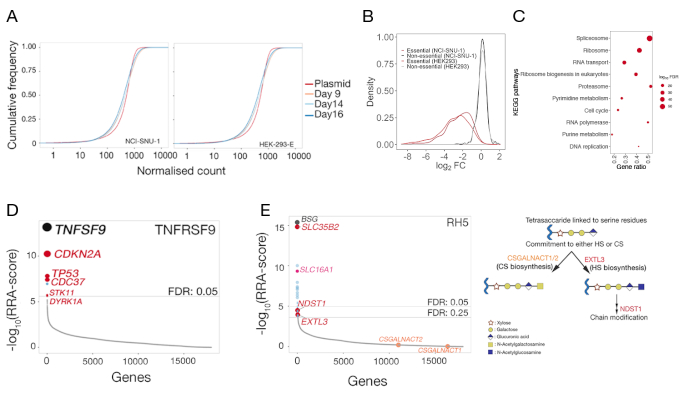
图5:用于识别细胞表面结合伙伴的基因筛选的代表性结果。(A) 累积分布函数图将质粒体库中的gRNA丰度与第9天、14日和16日HEK-293-E和NCI-SNU-1细胞的突变库进行比较。对于任何给定数字,累积密度函数报告低于该阈值的数据点的百分比。与原始质粒种群相比,突变细胞群的微小变化表示与质粒库相比,gRNA的子集的耗竭。(B) 以前在 HEK293 和 NCI-SNU-1 细胞系中被归类为必需(红色)或非必需(黑色)的基因的分数变化分布。非必需基因的折叠变化分布居于±0,而基本基因的分布则向左转向负折变化。(C) NCI-SNU-1突变对照种群在转染后14天内耗尽的基因中,有显著丰富的通路。确定了已知的已知细胞基本途径。(D) 强健的等级算法 (RRA) - 在已分类细胞中富集的基因得分,这些基因已经失去了结合 TNFRSF9 探针的能力。基因根据RRA评分进行排名。已知的相互作用伙伴TNFSF9和与TP53通路相关的基因(红色标记)在屏幕中被识别。(E) 从 GRNA 富集分析中识别的 RH5 与 HEK293 细胞(左面板)结合所需的基因的有序 RRA 分数。SLC35B2和SLC16A1是在 5% 的错误发现率 (FDR) 阈值内识别的。在HS生物合成途径中,另外两个基因(即EXTL3和NDST1)在25%的FDR内被识别。描绘一般GAG生物合成途径的原理图,相关基因映射到相应的步骤(面板2)。在屏幕中未发现硫酸软骨素生物成因所需的基因(即CSGALNACT1/2)。这个数字是从夏尔马等人19.请点击此处查看此图形的较大版本。
| 质粒名称 | 质 粒# | 使用 |
| 蛋白质表达结构:CD200RCD4d3_4-生物链接器-他 | 加金: 36153 | 生产含有CD4d3+4、生物结合和6-他标签的重组蛋白。 |
| pMD2.G | 加金: 12259 | VSV-G 信封表示质粒;生产扁病毒 |
| psPAX2 | 加金: 12260 | 伦迪病毒包装质粒,生产扁病毒 |
| Cas9 构造: pKLV2-EF1a-Cas9Bd-W | 加金: 68343 | 生产有组织表达Cas9线 |
| gRNA表达结构:pKLV2-U6gRNA5(BbsI)-PGKpuro2ABFP-W | 加金: 67974 | CRISPR gRNA 表达载体,具有改进的脚手架和紫杉/BFP 标记 |
| 人类改进的全基因组敲除CRISPR库 | 加金: 67989 | 一个gRNA库,针对18,010个人类基因,设计用于扁病毒。 |
| GFP-BFP 结构:pKLV2-U6gRNA5(gGFP)-PGKBFP2AGFP-W | 加金: 67980 | Cas9 活动报告员与 BFP 和 GFP。 |
| 空结构:pKLV2-U6gRNA5(空)-PGKBFP2AGFP-W | 加金: 67979 | Cas9 活动报告器(控制)与 BFP 和 GFP。 |
表1:此方法中使用的质粒。
| 缓冲区名称 | 组件 |
| HBS (10X) | 1.5 M NaCl 和 200 mM HEPES 在 MiliQ 水中,调整到 pH 7.4 |
| PBS (10X) | 80 g NaCl,2 g KCl,14.4 g Na2HPO4和 2.4 g KH2PO4在 MiliQ 水中,调整到 pH 7.4 |
| 磷酸钠缓冲液(80mM库存) | 7.1 g Na2HPO4.2H2O,5.55 g NaH2PO4,调整至 pH 7.4 |
| 他的净化结合缓冲器 | 20 mM 磷酸钠缓冲液,0.5 M NaCl 和 20 mM Imidazole,调整至 pH 7.4 |
| 他的净化洗脱缓冲液 | 20 mM 磷酸钠缓冲液,0.5M NaCl 和 400 mM Imidazole,调整至 pH 7.4 |
| 二乙醇胺缓冲液 | 10% 二乙醇胺和 0.5 mM MgCl2 在 MiliQ 水中,调整到 pH 9.2: |
| D10 | DMEM,1% 青霉素链霉素(100 单位/mL)和 10% 热灭活 FBS |
表 2:本研究所需的缓冲区。
| 组件 | 10厘米的菜 | 6 孔板 |
| 293FT 电池 | 70~80% 汇 | 70~80% 汇 |
| 转染兼容介质(Opti-MEM)(步骤5.1.2) | 3 mL | 500 μL |
| 转染兼容介质(Opti-MEM)(步骤5.1.4) | 5 mL | 2 mL |
| 伦迪病毒转移载体 | 3 μg | 0.5微克 |
| psPax2(见表1) | 7.4 微克 | 1.2 微克 |
| pMD2.G(见表1) | 1.6 微克 | 0.25微克 |
| 加试剂 | 12 μL | 2 μL |
| 利波霉素 LTX | 36 μL | 6 μL |
| D10 (步骤 7.1.7) | 5 mL | 1.5 mL |
| D10(步骤 7.1.8 和 7.1.10) | 8 mL | 2 mL |
表3:扁病毒包装混合物试剂的数量和体积。
表4:用于放大gRNA和NGS的引底序列。请点击此处查看此文件(右键单击以下载)。
| 试剂 | 每次反应的体积 | 主混合 (x38) |
| Q5 热启动高保真 2x | 25 μL | 950 μL |
| 底漆 (L1/U1) 混合 (每个 10 μM) | 1 μL | 38 μL |
| 基因组DNA(1毫克/mL) | 2 μL | 72 μL |
| H2O | 22 μL | 1100 μL |
| 总 | 50 μL | 1900 μL |
表5:用于从高复杂度样品中放大gRNA的PCR。
| 周期号 | 变性 | 退火 | 扩展 |
| 1 | 98 °C, 30s | ||
| 2-24 | 98 °C, 10s | 61 °C, 15s | 72 °C, 20s |
| 25 | 72 °C, 2 分钟 |
表 6:第一个 PCR 的 PCR 条件。
| 试剂 | 每次反应的体积 |
| KAPA HiFi 热启动就绪混合 | 25 μL |
| 底漆(PE1.0/指数底漆)混合(每个5μM) | 2μL |
| 第一个 PCR 产品 (40 pg/μL) | 5 μL |
| H2O | 18 μL |
| 总 | 50 μL |
表7:基因屏幕中sgRNA索引标记的PCR。
| 周期号 | 变性 | 退火 | 扩展 |
| 1 | 98 °C, 30s | ||
| 2-15 | 98 °C, 10s | 66 °C, 15s | 72 °C, 20s |
| 16 | 72 °C, 5 分钟 |
表8:第二次PCR的PCR条件。
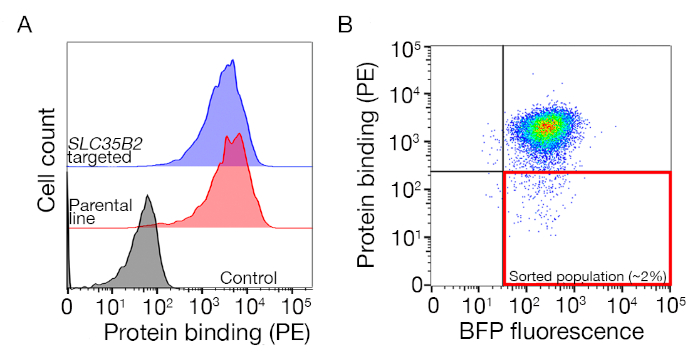
补充图 S1:绘制门的指南,用于对不具约束力的填充进行排序。(A) 与对照种群相比,理想的蛋白质候选物筛选应具有结合种群的明显变化,并且结合应保留在缺乏HS生物合成机制的细胞上。肝素阻断实验可用于代替SLC35B2靶细胞系的测试。(B) 细胞缺乏蛋白质异位的表面染色,但从扁病毒转导中表达BFP荧光的细胞被收集。显示的细胞来自一个屏幕,用于识别GABBR222的受体。这个数字是从夏尔马等人19.请点击此处查看此图形的较大版本。
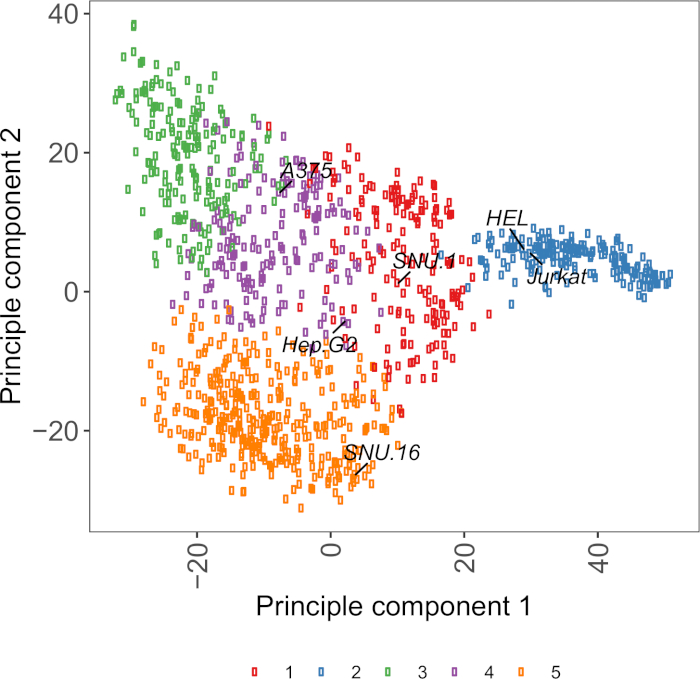
补充图S2:细胞表面糖蛋白转录组基基于PCA图,使用来自1000多个癌细胞系的RNA-seq数据。细胞模型Passport27中的细胞系根据1,500个细胞表面糖蛋白的FPKM值,使用K-手段聚类进行聚类。每个群集的代表性单元格行都已标记。第5组完全由造血源细胞系组成(另见补充表2)。请点击此处查看此图形的较大版本。
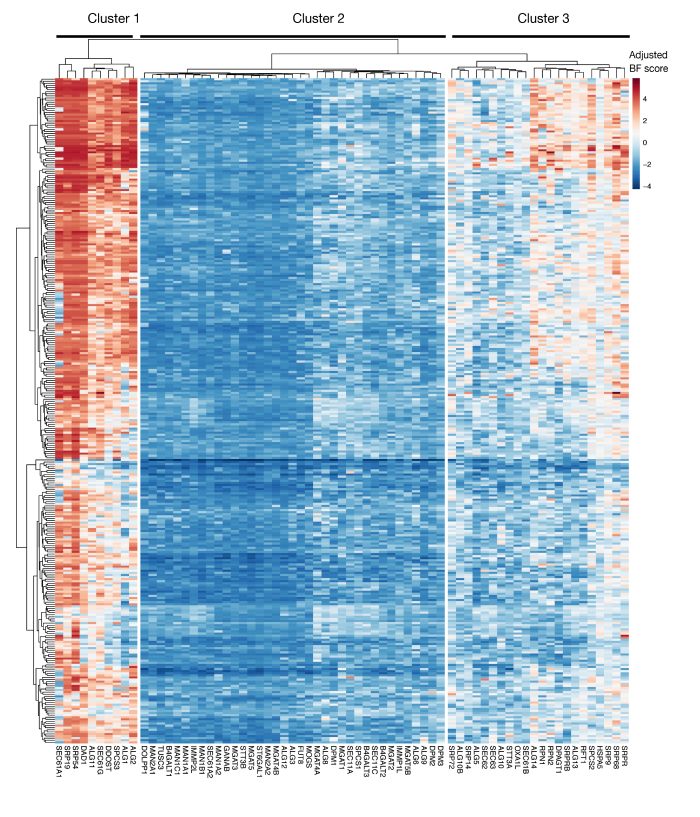
补充图S3:KEGG注释蛋白导出和项目分数N相关糖基化基因的必需性分数。为蛋白质导出和N相关糖基化通路(X轴)的基因绘制了 330 个细胞系(柱,未标记)的调整后贝叶素-基本性分数。与原始质粒库相比,分数高于 0 表示突变体严重枯竭。这些基因可以分为三个不同的簇,它们代表细胞系中不同程度的基本性。此群集可用于决定排序的当天。如果屏幕是在较晚的时间点(第 16 天)执行的,则可能无法识别已知对细胞(组 1 和 3)至关重要的基因。请点击此处查看此图形的较大版本。
补充表 1:原始计数文件和 MAGeCK 软件生成的gene_summary与具有代表性的遗传屏幕相关的文件。请点击此处查看此文件(右键单击以下载)。
补充表2:根据细胞表面受体的表达对细胞系的聚类。请点击此处查看此文件(右键单击以下载)。
讨论
介绍了基于CRISPR的筛选策略,用于识别编码细胞识别中涉及的细胞成分的基因。使用CRISPR活化的类似方法也提供了一种基因替代品,可以识别重组蛋白的直接相互作用受体,而无需生成大型蛋白质库26。然而,这种方法的一个主要优点是,它适用于由细胞上本地显示的表面分子介导的相互作用,并且不依赖于受体的过度表达,从而影响受体的结合性。因此,与其他方法不同,该技术对受体的生化性质或细胞生物学没有作任何假设,并且提供了一个机会来研究通常难以使用生化方法(如非常大的蛋白质)或多次穿过膜或与其他蛋白质形成复合物的蛋白质(如甘油、糖脂和磷脂等蛋白质)等分子介导的相互作用。鉴于该方法的基因组规模性质,这种方法不仅具有识别受体的优点,而且具有结合事件所需的额外细胞成分的优点,从而提供对受体细胞生物学的见解。
当使用这种方法来识别孤立蛋白的受体时,这种方法的主要局限性之一是首先确定与蛋白质结合的细胞系的初始要求。这并不总是容易的,识别显示结合表型的细胞行,该表型也允许遗传屏幕,这可能是部署此测定的时间限制步骤。有些细胞系往往与其他细胞系的蛋白质结合更多。这与结合 HS 的蛋白质特别相关,因为这些蛋白质倾向于与显示 HS 侧链的任何细胞系结合,而不考虑本机结合上下文。此外,我们观察到,在细胞系中提高合成酶(即含有HS的蛋白细胞)的调节,导致HS结合蛋白26的结合增加。在选择用于筛查的细胞系时,这可能是需要考虑的一个因素。然而,需要注意的还有,HS的添加剂结合不会干扰对特定受体的结合。这意味着,如果观察到绑定,则可能完全由 HS 进行中介,因为 HS 在此测定中介导的绑定是累加的,而不是共依赖19。在这种情况下,描述的肝素阻断方法可以识别此类行为,而无需执行完整的遗传筛查。
选择细胞系的有用资源是细胞模型Passport,它包含基因组学、转录组学和培养条件信息,用于+1,000个癌细胞系27。根据生物背景,可以根据细胞的表达特征选择细胞。为了帮助选择细胞系,我们根据1,500个预先批注的人类细胞表面糖蛋白28的表达,在细胞模型Passport中聚集了1,000个细胞系(补充图2;每个细胞系的簇信息以及生长条件在补充表2中提供)。测试具有未知功能的蛋白质的结合时,从每个簇中选择一组具有代表性的细胞系以增加覆盖各种受体的机会非常有用。如果可以选择一种选择,建议选择易于培养且易于转导的细胞系。由于这些细胞系将用于基因组规模筛选,因此最好能大量轻松生长,并放任扁病毒转导,因为它是在后续步骤中为CRISPR基因筛查提供sgRNA的最常用方法。
通常,表型选择以单一排序进行。但是,这是由染色细胞群与对照的亮度决定的。对于所需表型的信噪比低或屏幕的目的是识别具有强表型的突变体的情况,可以采用迭代轮选择。当对基于 FACS 的屏幕使用迭代选择方法时,必须考虑分拣过程可能导致细胞死亡,这主要是由于分拣机的纯力。因此,并非所有收集的单元格都将在下一轮排序中表示。
库的复杂性是执行成功的遗传屏幕的一个非常重要的因素,尤其是对于负选择屏幕,因为这些屏幕的耗竭程度只能通过将结果与起始库中的结果进行比较来确定。对于负选择屏幕,通常维护 500-1,000 x 复杂性的库。但是,正选择屏幕对库大小更强,因为在这样的屏幕中,预计只有少量突变体被选定为特定的表型。因此,在此描述的正选择屏幕中,库大小可以减小到 50-100 倍的复杂性,而不会影响屏幕的质量。此外,在这些屏幕中,还可以对给定的单元格行在给定的一天使用控制库,作为当天为给定单元格行排序的所有样本的"常规控制"。这将减少需要生成和排序的控制库的数量。
使用这种方法的另一个重要考虑因素是功能丧失屏幕在确定对体外细胞生长至关重要的基因方面的局限性。屏幕的时机在这方面至关重要,因为突变细胞在培养中保存的时间越长,基本基因突变的细胞变得不可行且不再在突变库中表示的可能性就越大。最近作为项目评分倡议的一部分在300多个细胞系中执行的基因筛选显示,KEGG批注蛋白分泌和N-糖基化途径中的多个基因通常被确定为对一些细胞系至关重要(补充图3)29。29如果要在细胞识别过程中研究增殖和生存能力所需的基因的影响,则可以在设计屏幕时考虑到这一点。在这种情况下,在早期时间点(例如,转染后第 9 天)进行屏幕通常适用。但是,如果该方法用于识别具有强尺寸效应而非一般细胞通路的几个目标,则在以后的时间点(例如,转染后的第 15-16 天)执行屏幕可能比较合适。
筛查结果非常强劲;在过去进行的8个重组蛋白结合屏幕中,细胞表面受体在每种情况下都是19次点击。因此,当使用这种方法识别相互作用伙伴时,人们应该期望在表面上发现导致其呈现的受体和因素,从而具有很高的统计可信度。一旦屏幕执行,并使用单个 gRNA 敲除验证命中,可以使用现有的生化方法(如 AVEXIS4)进行进一步的后续操作,并使用表面质质子共振直接对纯化蛋白进行饱和结合。此处描述的方法适用于所有可能生成可溶性重组结合探针的蛋白质。
总之,这是一种基因组级CRISPR敲除方法,用于识别由细胞表面蛋白质介导的相互作用。这种方法通常适用于识别细胞表面识别所需的细胞通路,在广泛的不同生物背景下,包括生物体自己的细胞(如神经和免疫学识别)之间,以及宿主细胞和病原体蛋白之间。这种方法为为受体识别设计的生化方法提供了一种基因替代,因为它不需要事先对受体的生化性质或细胞生物学进行任何假设,因此它有很大的潜力做出完全意想不到的发现。
披露声明
作者没有什么可透露的。
致谢
这项工作得到了授予GJW的威康信托赠款号206194的支持。我们感谢细胞学核心设施:蜂玲Ng,詹妮弗格雷厄姆,山姆汤普森和克里斯托弗霍尔帮助FACS。
材料
| Name | Company | Catalog Number | Comments |
| Anti-mouse alkaline phosphatase | Sigma | A4656 | |
| Blasticidin | Chem-Cruz | SC-204655 | |
| Blood & Cell Culture DNA Maxi Kit | Qiagen | 13362 | |
| BSA | Sigma | A9647-100G | |
| Diethanolamine | Sigma | 398179 | |
| DMEM | Gibco | 31966-021 | |
| Dneasy Blood and Tissue kit | Qiagen | 69504 | |
| DynaMag-96 Side Magnet | Invitrogen | 12331D | |
| HEK293T packaging cells | ATCC | CRL-3216 | |
| Heparin | Sigma | H4784-1G | |
| KAPA HiFi HotStart ReadyMix | Kapa | KK2602 | |
| Lipofectamine LTX with PLUS reagent | Invitrogen | 15338100 | |
| MoFlo XDP cell sorter | BD | ||
| Ni2+-NTA agarose beads | Jena Bioscience | AC-501-25 | |
| OPTI-MEM | Life Technologies | 31985-070 | |
| OX-68 antibody | AbD Serotec | MCA1022R | |
| p-nitrophenyl phosphate | Sigma | 1040-506 | |
| PD-10 desalting columns | GE healthcare | 17085101 | |
| Polybrene | Millipore | TR-1003-G | |
| Polypropylene tubes with 5 mL bed volume | Qiagen | 34964 | |
| Proteinase K, recombinant, PCR Grade | Roche | 3115879001 | |
| Puromycin | Gibco | A11138-03 | |
| Q5 Hot Start High-Fidelity 2× Master Mix | NEB | M0494L | |
| QIAquick PCR purification kit | Qiagen | 28104 | |
| SCFA filter | Nalgene | 190-2545 | |
| Sony Cell sorter | Sony | ||
| SPRI beads (Agencourt AMPure XP beads) | Beckman | A63881 | |
| Streptavidin-coated microtitre plates | Nalgene | 734-1284 | |
| Streptavidin-PE | Biolegend | 405204 |
参考文献
- Wright, G. J. Signal initiation in biological systems: the properties and detection of transient extracellular protein interactions. Molecular bioSystems. 5 (12), 1405-1412 (2009).
- van der Merwe, P. A., Barclay, A. N. Transient intercellular adhesion: the importance of weak protein-protein interactions. Trends in Biochemical Sciences. 19 (9), 354-358 (1994).
- Wood, L., Wright, G. J. Approaches to identify extracellular receptor-ligand interactions. Current Opinion in Structural Biology. 56, 28-36 (2019).
- Bushell, K. M., Söllner, C., Schuster-Boeckler, B., Bateman, A., Wright, G. J. Large-scale screening for novel low-affinity extracellular protein interactions. Genome Research. 18 (4), 622-630 (2008).
- Visser, J. J., et al. An extracellular biochemical screen reveals that FLRTs and Unc5s mediate neuronal subtype recognition in the retina. eLife. 4, e08149 (2015).
- Özkan, E., et al. An extracellular interactome of immunoglobulin and LRR proteins reveals receptor-ligand networks. Cell. 154 (1), 228-239 (2013).
- Martinez-Martin, N., et al. An Unbiased Screen for Human Cytomegalovirus Identifies Neuropilin-2 as a Central Viral Receptor. Cell. 174 (5), 1158-1171 (2018).
- Bianchi, E., Doe, B., Goulding, D., Wright, G. J. Juno is the egg Izumo receptor and is essential for mammalian fertilization. Nature. 508 (7497), 483-487 (2014).
- Mullican, S. E., et al. GFRAL is the receptor for GDF15 and the ligand promotes weight loss in mice and nonhuman primates. Nature Medicine. 23 (10), 1150-1157 (2017).
- Turner, L., et al. Severe malaria is associated with parasite binding to endothelial protein C receptor. Nature. 498 (7455), 502-505 (2013).
- Frei, A. P., et al. Direct identification of ligand-receptor interactions on living cells and tissues. Nature Biotechnology. 30 (10), 997-1001 (2012).
- Sobotzki, N., et al. HATRIC-based identification of receptors for orphan ligands. Nature Communications. 9 (1), 1519 (2018).
- Sharma, S., Petsalaki, E. Application of CRISPR-Cas9 Based Genome-Wide Screening Approaches to Study Cellular Signalling Mechanisms. International Journal of Molecular Sciences. 19 (4), (2018).
- Gebre, M., Nomburg, J. L., Gewurz, B. E. CRISPR-Cas9 Genetic Analysis of Virus-Host Interactions. Viruses. 10 (2), (2018).
- Zotova, A., Zotov, I., Filatov, A., Mazurov, D. Determining antigen specificity of a monoclonal antibody using genome-scale CRISPR-Cas9 knockout library. Journal of Immunological Methods. 439, 8-14 (2016).
- Puschnik, A. S., Majzoub, K., Ooi, Y. S., Carette, J. E. A CRISPR toolbox to study virus-host interactions. Nature Reviews. Microbiology. 15 (6), 351-364 (2017).
- Kerr, J. S., Wright, G. J. Avidity-based extracellular interaction screening (AVEXIS) for the scalable detection of low-affinity extracellular receptor-ligand interactions. Journal of Visualized Experiments. (61), e3881 (2012).
- Tzelepis, K., et al. A CRISPR Dropout Screen Identifies Genetic Vulnerabilities and Therapeutic Targets in Acute Myeloid Leukemia. Cell Reports. 17 (4), 1193-1205 (2016).
- Sharma, S., Bartholdson, S. J., Couch, A. C. M., Yusa, K., Wright, G. J. Genome-scale identification of cellular pathways required for cell surface recognition. Genome Research. 28 (9), 1372-1382 (2018).
- Wang, B., et al. Integrative analysis of pooled CRISPR genetic screens using MAGeCKFlute. Nature Protocols. 14 (3), 756-780 (2019).
- Hart, T., et al. Evaluation and Design of Genome-Wide CRISPR/SpCas9 Knockout Screens. G3. 7 (8), 2719-2727 (2017).
- Kuleshov, M. V., et al. Enrichr: a comprehensive gene set enrichment analysis web server 2016 update. Nucleic Acids Research. 44 (W1), W90-W97 (2016).
- Crosnier, C., et al. Basigin is a receptor essential for erythrocyte invasion by Plasmodium falciparum. Nature. 480 (7378), 534-537 (2011).
- Kirk, P., et al. CD147 is tightly associated with lactate transporters MCT1 and MCT4 and facilitates their cell surface expression. The EMBO Journal. 19 (15), 3896-3904 (2000).
- Chong, Z. S., Ohnishi, S., Yusa, K., Wright, G. J. Pooled extracellular receptor-ligand interaction screening using CRISPR activation. Genome Biology. 19 (1), 205 (2018).
- van der Meer, D., et al. Cell Model Passports-a hub for clinical, genetic and functional datasets of preclinical cancer models. Nucleic Acids Research. 47 (D1), D923-D929 (2019).
- Bausch-Fluck, D., et al. A mass spectrometric-derived cell surface protein atlas. PloS One. 10 (3), e0121314 (2015).
- Behan, F. M., et al. Prioritization of cancer therapeutic targets using CRISPR-Cas9 screens. Nature. 568 (7753), 511-516 (2019).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。