Method Article
Genom ÖlçeğiCRISPR/Cas9 Genetik Ekranlar Kullanılarak Hücre Yüzey Reseptör Tanımlama
Bu Makalede
Özet
Bu el yazması, hücre dışı reseptör-ligand etkileşimlerini tanımlamak için genom ölçeğinde hücre tabanlı tarama yaklaşımını açıklamaktadır.
Özet
Membrana gömülü hücre yüzey reseptörleri arasındaki doğrudan etkileşimler aracılığı ile aracılık edilen hücreler arası iletişim, çok hücreli organizmaların normal gelişimi ve işleyişi için çok önemlidir. Ancak, bu etkileşimleri algılamak teknik olarak zor olmaya devam etmektedir. Bu makale, belirli hücre yüzey tanıma olayları için gerekli hücresel yolları ortaya koyan sistematik genom ölçekli CRISPR/Cas9 nakavt genetik tarama yaklaşımını açıklamaktadır. Bu test, bir memeli protein ekspresyon sisteminde üretilen rekombinant proteinleri hücre tabanlı genetik bir ekranda etkileşim ortaklarını belirlemek için hevesli bağlayıcı problar olarak kullanır. Bu yöntem, membrana gömülü reseptörlerin ektodomains karşılık rekombinant bağlayıcı problar tarafından tespit hücre yüzeyi etkileşimleri için gerekli genleri tanımlamak için kullanılabilir. Daha da önemlisi, bu yaklaşımın genom ölçekli doğası göz önüne alındığında, aynı zamanda sadece doğrudan reseptör tespit değil, aynı zamanda hücre yüzeyinde reseptör sunumu için gerekli olan hücresel bileşenleri tespit avantajı vardır, böylece reseptörbiyolojisi içine değerli anlayışlar sağlayan.
Giriş
Hücre yüzeyreseptör proteinleri tarafından ekstrasellüler etkileşimleri doku organizasyonu, konak-patojen tanıma ve bağışıklık regülasyonu gibi önemli biyolojik süreçleri yönlendirir. Membran reseptörleri monoklonal antikorlar gibi sistematik olarak teslim terapötik eyleme hedefleri olduğundan, bu etkileşimleri soruşturma, daha geniş biyomedikal topluluk için ilgi çekicidir. Önemine rağmen, bu etkileşimleri incelemek teknik olarak zor olmaya devam etmektedir. Bunun başlıca nedeni membrana gömülü reseptörlerin amphipatik olması, biyokimyasal manipülasyon için biyolojik membranlardan izole etmelerini zorlaştırması ve etkileşimlerinin zayıf etkileşim yakınlıkları (μM-mMaralığındaki KDs)1. Sonuç olarak, birçok yaygın olarak kullanılan yöntemler protein etkileşimleri bu sınıf tespit etmek için uygun değildir1,2.
Benzersiz biyokimyasal özelliklerini dikkate alan ekstrasellüler reseptör-ligand etkileşimlerini özellikle araştırmak için çeşitli yöntemler geliştirilmiştir3. Bu yaklaşımların bir kısmı, bu proteinlerin glikanlar ve disülfür bağları gibi yapısal olarak önemli olan çeviri sonrası modifikasyonlar içermesini sağlamak için memeli veya böcek hücre tabanlı sistemlerde çözünür rekombinant protein olarak reseptörün tüm ektoetki alanını ifade etmeyi içerir. Düşük yakınlık bağlama üstesinden gelmek için, ektodomains genellikle bağlayıcı avidite artırmak için oliomerize edilir. Hırslı protein ektodomains başarıyla doğrudan rekombinant protein-protein etkileşim ekranlarında etkileşim ortakları belirlemek için bağlayıcı problar olarak kullanılmıştır4,5,6,7. Geniş başarılı olsa da, rekombinant protein bazlı yöntemler bir membran reseptörünün ektoetkialanı çözünür bir protein olarak üretilmesi gerekir. Bu nedenle, genellikle bitişik bir hücre dışı bölge içeren proteinler için geçerlidir (örneğin, tek geçişli tip I, tip II veya GPI-anchored) ve genellikle membrana birden fazla kez yayılan reseptör kompleksleri ve membran proteinleri için uygun değildir.
Tamamlayıcı DNA'lardan oluşan bir kütüphanenin hücrelere aktarıldığı ve bağlayıcı bir fenotip artışı için test edildiği ekspresyon klonlama teknikleri de hücre dışı protein-protein etkileşimlerini tanımlamak için kullanılmıştır8. Son yıllarda klonlanmış ve sıralı cDNA ekspresyonu plazmidlerinin büyük koleksiyonlarınınkullanılabilirliği,cDNA'ları kodlayan hücre yüzey reseptörlerini aşırı ifade eden hücre hatlarının etkileşimleri tanımlamak için rekombinant proteinlerin bağlanması için tarandığı yöntemleri kolaylaştırmıştır9,10. CDNA aşırı ekspresyon temelli yaklaşımlar, rekombinant protein bazlı yöntemlerin aksine, plazma membranı bağlamında etkileşimleri belirleme olanağı nı karşılar. Ancak, cDNA ekspresyonu yapılışındaki başarı, hücrelerin proteini doğru katlanmış formda aşırı ifade etme yeteneğine bağlıdır, ancak bu genellikle taşıyıcılar, şaperonlar ve doğru oligomerik montaj gibi hücresel aksesuar faktörleri gerektirir. Bu nedenle tek bir cDNA'nın aktarılması hücre yüzeyi ekspresyonunu elde etmek için yeterli olmayabilir.
CDNA yapıları veya rekombinant protein probları kullanarak tarama teknikleri kaynak yoğun dur ve cDNA veya rekombinant protein kütüphanelerinin büyük koleksiyonlarını gerektirir. Özellikle tasarlanmış kütle spektrometresi tabanlı yöntemler büyük kütüphanelerin montaj gerektirmeyen hücre dışı etkileşimleri belirlemek için son zamanlarda kullanılmıştır. Ancak, bu teknikler hücre yüzeyinde mevcut moleküllerin biyokimyasal doğasını değiştirebilir ve şu anda sadece glikozile proteinler11,12aracılık etkileşimleri için geçerli olan hücre yüzeyinin kimyasal manipülasyon gerektirir. Şu anda mevcut yöntemlerin çoğunluğu da büyük ölçüde glikan, lipidler ve kolesterol gibi moleküller de dahil olmak üzere membran mikroortamdan katkısı göz ardı ederken proteinler arasındaki etkileşimleri üzerinde yoğun olarak odaklanır.
CRISPR tabanlı yaklaşımlar kullanılarak son derece verimli biallelik hedeflemenin son zamanlarda gelişmesi, farklı bağlamlarda yer alan hücresel bileşenleri tanımlamak için sistematik ve tarafsız bir şekilde taranabilen tek bir havuzda tanımlanmış genlerden yoksun hücrelerin genom ölçeğinde kütüphanelerini sağlamıştır, hücresel sinyal süreçlerinin incelenmesi, ilaçlara, toksinlere ve patojenlere karşı direnç sağlayan tedirginliklerin belirlenmesi ve antikorların özgüllüğünün belirlenmesidahil 13,14,15,16. Burada, hücre dışı reseptör-ligand etkileşimlerini tanımlamak için mevcut biyokimyasal yaklaşımlara alternatif sağlayan genom ölçekli CRISPR tabanlı nakavt hücre tarama testini tanımlıyoruz. Membran reseptörlerinin genetik ekranlar aracılığı ile oluşturduğu etkileşimleri tanımlama yaklaşımı, özellikle cd'lerin veya rekombinant proteinlerin büyük kütüphanelerinin derlenmesine gerek duymadığı için bireysel ligandlara odaklanmış bir ilgi duyan araştırmacılar için uygundur.
Bu tahlil üç ana adımdan oluşur: 1) İlgi reseptörünün hücre dışı bölgelerinden oluşan son derece hırslı rekombinant protein bağlama probları floresan esaslı akış sitometrisi-bağlama tahlillerinde üretilir ve kullanılır; 2) Bağlayıcı tahliller rekombinant protein probu etkileşim ortağı ifade eden bir hücre hattı tanımlamak için kullanılır; 3) Etki proteini ile etkileşime girilen hücre hattının Cas9-ekspres versiyonu üretilir ve genom ölçekli CRISPR/Cas9 tabanlı nakavt ekranı yapılır (Şekil 1). Bu genetik ekranda, bir rekombinant proteinin hücre hatlarına bağlanması, sondayı bağlama yeteneğini yitirmiş nakavt kitaplığı içindeki hücrelerin floresan bazlı aktif hücre sıralama (FACS) ve sıralama ile tanımlanan bağlayıcı fenotip kaybına neden olan genler kullanılarak sıralandığı ölçülebilir bir fenotip olarak kullanılır. Prensip olarak, avid probu bağlamaktan sorumlu reseptörü kodlayan genler ve hücre yüzeyi görüntüsü için gerekli olanlar tanımlanır.
Bu protokolün ilk adımı membrana bağlı reseptörlerin ektoetkialanını temsil eden hırslı rekombinant protein problarının üretimini içerir. Bu reseptörlerin ektodomains bir rekombinant çözünür protein1olarak ifade edildiğinde sık sık ekstrasellüler bağlayıcı işlevlerikorumak bilinmektedir. İlgi çekici bir protein için, çözünür rekombinant proteinler, daha fazla bağlayıcı avidite için oliomerize edilebilen herhangi bir formatta uygun ökaryotik protein ekspresyon sisteminde üretilebilir ve floresan bazlı akış sitometrisi bazlı bağlayıcı tahlillerde (örn. HEK293 protein ekspresyonu sistemi kullanılarak membran reseptörlerinin çözünür ektodomains üretimi için ayrıntılı protokoller, hem pentamerik proteinlerin ve monomerik proteinlerin üretimi için farklı multimerizasyon teknikleri ve protein ekspresyonu yapıları daha önce tarif edilmiştir1,17. Buradaki protokol, monomerik biyotinylated proteinlerden floresan avid probları üreterek, doğrudan hücre bazlı bağlayıcı tahlillerde kullanılabilen ve algılama için ikincil bir antikor gerektirmeyen bir avantaja sahip olan bir florokroma (örneğin, phycoerythrin veya PE) konjuge streptavidin'e konjuge ederek floresan avid probları üretme adımlarını açıklayacaktır. Genom ölçekli ekranlar gerçekleştirmek için genel protokoller zaten tarif edilmiştir20,21, bu nedenle protokol esas olarak Insan V1 kullanarak CRISPR / Cas9 nakavt tarama sistemi kullanarak akış sitometri tabanlı rekombinant protein bağlama ekranları performans özellikleri odaklanmak ("Yusa") kütüphane18.
Protokol
1. Biyotinylated His-tagged proteinlerin üretimi ve saflaştırılması
- Çözünür rekombinant His-tagged biyotinylated proteinler üretmek için bir memeli veya böcek hücre tabanlı protein ekspresyonu sistemi kullanın (Tablo 1plazmid yapıları bakınız).
NOT: HEK293 hücre ekspresyonu sistemini kullanarak monomerik biyotin ve Onun etiketli proteinlerin üretimi için ayrıntılı bir protokol Kerr ve ark.17tarafından açıklanmıştır. HEK293 ifade sistemi kullanılarak ifade edilen protein ektodomainleri kültür ortamına salgılanır. - 20 dk için 3.000 x g santrifüj ile hücreleri peletleme tarafından çözünür proteinleri toplamak.
- Supernatant'ı 0,22 μm'lik bir filtreden filtreleyin ve 1:1.000 oranında filtrelenmiş protein supernatant'a Ni2+-NTA agarose boncukları ekleyin (yani 50 μL%50 agarose bulamaç 50 mL supernatant'a). Bir gecede veya en az 4-5 saat 4 °C'de dönen bir platformda kuluçkaya yatırın.
- Polipropilen kolonunu 5 mL His-arıtma yıkama tamponu ekleyerek yıkayın. Tüm arabellek kompozisyonları için Tablo 2'ye bakın.
- Sütuniçine tüm boncuk-protein supernatant karışımı dökün. Boncuklar tabanda birikir.
- Boncukları 15 mL yıkama tamponu ile 2x yıkayın. Protein seyreltilmesini önlemek için, 5 mL şırınga ile kolondan kalan yıkama tamponu dikkatlice çekin ve atın.
- Dikkatle boncuklar doğrudan 300-500 μL His-saflaştırma elüsülasyon tampon ekleyin ve en az 1 saat için kuluçka. Tuzsuzsütunları kullanarak elüsyon arabelleği istenilen arabellekle (örn. normalde PBS veya HBS) değiştirin. Tüm proteinleri daha fazla kullanılana kadar 4 °C'de saklayın.
2. Monomerik biyotinylated proteinin nicelleştirilmesi ve oliomerizasyonu
NOT: Bağlayıcı aviditeyi artırmak için tetramerik streptavidin-PE üzerindeki biyosoneomerlenmiş monomerik proteinleri bağlayıcı tahlillerde kullanmadan önce oligomerize edin. Sabit bir streptavidin konsantrasyonuna karşı bir seyreltme dizilibiyotinylated monomerleri test ederek ve aşırı biyotinylated monomerlerin tespit edilebildiği minimum seyreltme kurarak monomerik proteinlerin ve tetramerik streptavidin-PE'nin optimal konjugasyon oranlarını elde edin.
- 96 kuyu plakasında uygun bir seyreltme tamponu (%1 büyükbaş hayvan serum albumini [BSA]) ile uygun bir seyreltme tamponu (PBS veya HBS) kullanarak biyotinylated protein örneklerinin en az sekiz seri seyreltme yapın. Her seyreltmenin son hacminin en az 200 μL olduğundan emin olun.
- Her kuyudan 100 μL çıkararak ve yeni bir 96 kuyu plakasına aktararak numunelerin bir kopyasını yapın. Her zaman bir kontrol ekleyin. Bu durumda kontroller sadece etikete özel proteinlerdir (yani, biotinylated His-tagged Cd4 etki alanı 3+4 protein). Bu, tüm bağlayıcı tahlillerde bir kontrol sondası olarak kullanılacaktır.
- Seyreltme tamponunda streptavidin-PE'yi 0.1 μg/mL'ye seyreltin.
- Plakalardan sadece birine seyreltilmiş streptavidin-PE'nin 100 μL'sini ekleyin. Yinelenen plaka bir kontrol görevi göreceksiniz. Hacimleri eşitlemek için kontrol plakasına 100 μL seyreltme tamponu ekleyin.
- Oda sıcaklığında 20 dakika (RT) kuluçka. Bu arada, 15 dakika seyreltme tampon ile bir streptavidin kaplı plaka kuyuları blok.
- Numunenin toplam hacmini her iki plakadan streptavidin kaplı plakaların bireysel kuyularına aktarın ve RT'de 1 saat kuluçkaya yatırın.
- Plakayı 200 μL yıkama tamponuyla (örneğin, PBS veya HBS %0,1 Ara-20, %2 BSA) ile yıkayın. 100 μL 2 μg/mL fare anti-rat Cd4d3+4 IgG (OX68) ekleyin ve RT'de 1 saat kuluçkaya yatırın.
- Plakayı 3x yıkama tamponuile yıkayın. Rt'de 1 saat boyunca 0,2 g/mL'de 100 μL anti-fare alkalin fosfataz eşlenik ekleyin.
- Plakayı yıkama tamponu yla 3 kat, seyreltme tamponunda 1x yıkayın.
- P-nitrofenil fosfatı 1 mg/mL'de dietanolamine tamponda hazırlayın. Her kuyuya 100 μL ekleyin ve 15 dakika kuluçkaya yatırın.
- 405 nm'de emicilik okumaları alın. Tetramer oluşturmak için uygun seyreltme faktörü olarak plakaüzerinde sinyal bulunmayan minimum seyreltme kullanın (Şekil 2).
- 4 μg/mL streptavidin-PE kuluçka ve RT. Store konjuge proteinleri 4 °C'de ışık korumalı bir tüpte saklayarak tüm numuneler ve kontroller için 10x tetramer boyama solüsyonu yapın.
3. Akış sitometrisi esaslı hücre bağlama tahlilleri
- Yapışık hücreler için kültür ortamını çıkarın ve divalent katyonlar olmadan PBS ile 1x yıkayın. Ardından hücre ayırma çözümleri (örneğin, EDTA) ekleyin. Hücrelerin 5-10 dakika boyunca ayrılmasına izin verin.
NOT: Hücre yüzeyi proteinlerini yarık taslayabilecekleri için tripsin bazlı ürünler kullanmaktan kaçının. - Bir tüp içine müstakil hücreleri toplamak. Süspansiyon içinde büyüyen hücreler için (örneğin, HEK293 hücreleri), kültür şişelerinden hücreleri doğrudan bir tüpe toplar.
- 5 dk. 200 x g pelet hücreleri supernatant çıkarın ve yıkama tampon (yani, PBS/1% BSA) pelet askıya.
- Hücreleri hemositometre kullanarak sayın ve konsantrasyonu 2,5 x 105-1 x 106 hücre/mL olarak ayarlayın. Aliquot 100 μL hazırlanmış hücre karışımı 96 iyi U- veya V- dipli plaka üzerinde. 400 x g5 dakika için plaka spin . Çok kanallı pipetle süpernatant'ı çıkarın.
- Normalleştirilmiş floresan 100 μL'lik yüksek hırslı protein probları ve kontrollerini daha önce hazırlanmış hücrelerle hazırlanmış tabaklara ekleyin ve 4 °C'de 1 saat kuluçkaya yatırın. 1 saat bağlandıktan sonra plakayı 400 x g'de 5 dk çevirin.
- Supernatant çıkarın ve yıkama tampon (yani, PBS/1% BSA) 200 μL ekleyin. Yukarı ve aşağı borular oluşturarak iyice karıştırın.
- 400 x g'de 5 dk. Yıkama adımını 1x tekrarlayın. İki yıkamadan sonra, süpernatantı tamamen çıkarın ve hücre peletini 100 μL PBS'de yeniden askıya alın.
- Akış sitometrisi ile hücreleri analiz edin. PE floresansını saptamak için sarı-yeşil lazeri (yani 561 nm) kullanın.
- Önce kontrol sondasıyla boyanmış hücreleri analiz edin. PE floresan dağılımına göre, kontrol hücresinin en fazla % 1'inin bu kapıya düşmesine yol açsın diye bağlayıcı popülasyon için bir kapı çizin.
- Örneği analiz edin ve bağlama kapısına düşen hücrelerin fraksiyonunu belirleyin.
NOT: Daha yüksek bağlayıcı popülasyon gösteren hücre hatları genetik ekranlar için istenir, çünkü daha yüksek bir sinyal-gürültü oranına sahiptirler. İdeal olarak hücrelerin %80'inden fazlası bu kapıiçine düşmelidir.
4. Isı labile epitopyaları ve heparan sülfat kenar zincirlerinden bağlayıcı katkıların belirlenmesi
NOT: Birçok proteinin aktivitesi ısı lama, bu nedenle ısıl işlem sonrasında bağlayıcı aktivite kaybı cesaret vericidir. Rekombinant proteinlerin bağlanmasına aracılık eden heparan sülfat (HS) başta olmak üzere negatif yüklü glikozaminoglikanların katkısını belirlemesi tavsiye edilir. Bunun nedeni, burada açıklanan hücre bağlama sataşasında HS tarafından bağlanan bağlayıcının diğer reseptörlere bağımlı hale gelmeden daha fazla katkı maddesi olmasıdır19. Bu gözlenen bağlama tamamen hücre yüzeyi proteoglikanhların HS yan zincirleri tarafından değil, belirli bir reseptör tarafından aracılık anlamına gelir. Hücre yüzeyinde HS bağlama mutlaka nonspesifik değil, tam bir genetik ekran gerçekleştirmeden önce bilmek yararlı bir protein, bir özelliktir.
- Bağlayıcı tahlillerde kullanılmak üzere ısıl işlem görmüş protein örnekleri hazırlayın.
- Normalleştirilmiş ancak konsinye edilmemiş monomerik proteini 80 °C'de 10 dakika ısıtın.
- Isıl işlem görmüş proteini, ELISA tarafından belirlenen tedavi edilmemiş muadili ile aynı konjugasyon oranını varsayarak streptavidin-PE'ye yönlendirin (bölüm 2'ye bakın).
- Heparin bloklu protein örneklerini hazırlayın.
- PBS'de 2 mg/mL başlangıç konsantrasyonu ve 100 μL son hacimile sekiz tane 1:3 çözünür heparin seyreltme hazırlayın.
- Heparin seyreltmelerinde en az 30 dakika boyunca hazırlanmış bağlayıcı probların 100 μL'sini inkübülye edin.
- Bölüm 3'te açıklanan bağlayıcı tahlillerde ısıl işlem görmüş protein ve tam 200 μL heparin/protein karışımını kullanın. Temsil sonuçları Şekil 3A,B'degösterilmiştir.
5. Cas9'u ifade eden hücre hatlarının seçilmesi
NOT: İlgi sondasını bağlayan hücre hattı CRISPR taramasında kullanılabilmeden önce, önce Cas9 nükleazını ifade etmek için tasarlanmalıdır ve son derece aktif bir klon seçilmiş19.
- Cas9 ekspresyonu için lentiviral yapıyı kullanarak lentivirüs üretmek için aşağıdaki genel lentivirus üretim protokolünü kullanın (Tablo 1'ebakın).
- Kültür HEK293-FT hücreleri DMEM/10% FBS ortamda 37 °C ve% 5 CO2. Tohum HEK293-FT hücreleri transfeksiyon dan 1 gün önce böylece transfeksiyon gününde ~%80 konfeksiyon oluştururlar.
NOT: HEK293FT hücreler gevşek yapışıktır; bu nedenle, lentivirüslerin üretimi için kullanıldığında, yapışmayı artırmak için % 0,1 (w/v) jelatin ile kaplanmış kültür şişeleri üzerine kaplamayı düşünün. - Sabah transfeksiyonları gerçekleştirin. Önceden ısıtılmış transfeksiyon uyumlu ortama transfer vektörü, paketleme karışımı ve transfeksiyon reaktifini ekleyin (örn. Opti-MEM). Tüpü 10-15x ters çevirerek karıştırın. RT.'de 5 dk inkübat. Tam hacimler için Tablo 3'e bakın.
- Üretici tarafından önerilen transfeksiyon reaktifini ekleyin. Hızlı girdap ile karıştırın. RT'de 30 dakika kuluçka.
- Çok dikkatli bir şekilde harcanan orta aspire. Plakaya transfeksiyon uyumlu ortam ekleyin.
- Transfeksiyon reaktifi/DNA komplekslerini plakanın yan tarafına damlayan ekleyin ve yavaşça çok hafifçe girdap tarafından plaka yayılın.
- 37 °C'de 3-5 saat kuluçkaya yatın ve orta yı D10 orta ile değiştirin. Bir gecede kuluçkaya yat.
- Ertesi gün sabah, taze D10 orta ile orta değiştirin. Bir gecede kuluçkaya yat.
- Ertesi gün öğleden sonra, viral supernatant toplamak. Düşük protein bağlayıcılığı olan 0,45 μm filtre ile filtre uygulayın. İsteğe bağlı olarak, taze D10 orta ekleyin, bir gecede kuluçka ve ertesi gün supernatant hatırlamak.
- Virüs süpernattları 4 °C'de sadece birkaç gün stabildir. Uzun süreli depolama için -80 °C'de saklayın.
NOT: Hücrelerin aktarılması zor transdüksiyon için arzu edilebilir son derece konsantre lentiviral preparat oluşturmak için, supernatants da 4 °C gecede 6.000 x g santrifüj ile konsantre edilebilir. Yarı saydam viral peleti etanol dirençli kalemle işaretleyin ve süpernatant'ı atın. Toplamada 100 kat artış için orijinal hacmin 1/100'de 1/100'de peletyeniden askıya alın.
- Kültür HEK293-FT hücreleri DMEM/10% FBS ortamda 37 °C ve% 5 CO2. Tohum HEK293-FT hücreleri transfeksiyon dan 1 gün önce böylece transfeksiyon gününde ~%80 konfeksiyon oluştururlar.
- Lentivirüsler ile hücreleri iletin.
- Uygun kültür ortamının 3 mL'si ile 6 kuyulukta kuyu başına 1 x 106 hücre plakası. Bazı hücreler diğerlerinden daha kolay transdüktör. Hücreleri (örneğin, HEK hücreleri) kolayca iletmek için, doğrudan hücrelere lentivirus ekleyin. Hücreleri aktarmanın zor olması için, aşağıda açıklandığı gibi bir spinolasyon protokolüne uymak gerekebilir.
- Aliquot 2 mL 2-5 x 106 hücre/mL 15 mL konik tüp.
- 8 μg/mL hexadimethrine bromür ve 30 dakika RT inkübat ile birlikte lentivirus ekleyin.
- 32 °C'de 800 x g'de 100 dk santrifüj. Daha sonra aynı ortamda hücreleri yeniden askıya ve uygun ortam ile uygun kültür şişeleri içine hücre süspansiyon ekleyin.
- En az 24 saat transdüksiyonlara izin verin. Daha sonra virüsü içeren ortamı çıkarın ve taze ortam ekleyin.
- Başka bir 24 saat sonra, uygun antibiyotikler ile tamamlanır biri için medya değiştirin. Cas9 yapısı seçim için blasticidin direnç kaseti içerir.
NOT: Blasticidin miktarı bir doz yanıt öldürme eğrisi gerçekleştirerek her hücre hattı için optimize edilmelidir. 2.5-50 μg/mL arasındaki bir blasticidin konsantrasyonu transdüksiyondan sonraki 10 gün içinde transedinedilmemiş hücre hatlarını öldürmelidir.
- Uygun kültür ortamının 3 mL'si ile 6 kuyulukta kuyu başına 1 x 106 hücre plakası. Bazı hücreler diğerlerinden daha kolay transdüktör. Hücreleri (örneğin, HEK hücreleri) kolayca iletmek için, doğrudan hücrelere lentivirus ekleyin. Hücreleri aktarmanın zor olması için, aşağıda açıklandığı gibi bir spinolasyon protokolüne uymak gerekebilir.
- Kontrol plakasındaki tüm hücreler (yani, aynı seçim antibiyotik konsantrasyonu ile tedavi edilmiş transeneden olmayan hücreler) öldürülene kadar seçim yapın.
6. Yüksek Cas9 aktivitesi klonları seçme
NOT: Poliklonal Cas9 başarılı genetik ekranlar gerçekleştirmek için kullanılabilir; ancak, yüksek Cas9 aktivitesi ile bir klon seçerek tarama sonuçlarını artırır18.
- Sınırlayıcı seyreltme veya tek hücreli tek hücreli tek blastisdin dirençli hücreleri blasticidin ile desteklenen kültür ortamı içeren üç 96 kuyu plakası kuyularına kullanın. Klonlar 2-4 hafta arasında ortaya çıkmaya başlar. Seçin 10-20 klonlar ve 6 kuyu plakaları genişletin.
- Cas9 aktivitesi için klonlar hızlı değerlendirmek GFP-BFP (yeşil floresan protein-mavi floresan protein) sistemi kullanarak, hangi hücrelerin bir gRNA hedefleyen GFP veya bir kontrol olarak boş bir gRNA ile GFP ifade eden bir yapı ile ya transdükstre bir dışgen gen nakavt sistemi kullanır18.
- Sipariş muhabiri plazmidleri: GFP-BFP plazmid, Kontrol-BFP plazmidi (Tablo 1).
- Bölüm 5.1'de açıklanan lentivirus üretim protokolünü kullanarak Hem GFP-BFP plazmidi hem de Control-BFP plazmidi için lentivirus üretin.
- GFP-BFP sistemini ve Control-BFP'yi ayrı ayrı kodlayan lentivirüs ile her Cas9 ifade eden hücre hattı klonunu aktarın. Bölüm 5.2'deki protokolü izleyin.
- Transdüksiyon 3 gün sonra, akış sitometri kullanarak her klonun GFP-BFP floresansını inceleyin. GFP ve BFP'yi tespit etmek için sırasıyla 488 nm lazer ve 405 nm lazer kullanın.
- BFP'nin sadece GFP-BFP-çift pozitif hücrelere oranını inceleyerek her klondaki Cas9 aktivitesini ölçün. Yüksek aktivitecas9 hücreleri ideal olarak >95% GFP nakavt verimliliğine sahip olmalıdır(Şekil 4).
7. Genom çapında CRISPR-Cas9 tarama nakavt kütüphanesinin üretimi
- Human V1 library18'ikullanarak genom çapında tarama için genom çapındaki kütüphaneyi sipariş edin (Tablo 1'ebakın) ve üretici kılavuzunda "Kütüphane Çoğaltma Protokolleri" altında sağlanan protokolü kullanarak plazmid kitaplığını bakteri sapından hazırlayın.
- Bölüm 5.1'de açıklanan lentivirus üretim protokolünü kullanarak insan genlerinin hedeflenen bozulması için lentiviral kütüphane kodlama gRNA'ları üretmek için genom çapındaki kütüphane plazmid preparatını kullanın.
NOT: İyi bir uygulama deneysel tutarlılığı artırmak için transdüksiyon için optimize edilmiş lentiviral hazırlık tek bir toplu üretmektir. - %30 transdüksiyon elde etmek için her hücre hattı için gerekli virüs miktarını belirlemek için küçük ölçekli test transdüksiyonları gerçekleştirmek için bölüm 5.2'deki transdüksiyon protokolünü kullanın. BFP floresansını transdüksiyon verimliliği için bir vekil olarak değerlendirmek için akış sitometrisini kullanın.
- HEK293 hücrelerini iletmek için, önceden belirlenmiş lentiviral preparatları ~4 saat için normal büyüme ortamlarında kültürlenmiş 30-50 x 106 hücreye eklemeniz yeterlidir. Sonra lentivirus ile medya kaldırmak ve taze büyüme medya ile değiştirin.
- Diğer hücre hatları için, bölüm 5.2.1'deki spinogülasyon protokolünü kullanın, ancak daha büyük bir ölçekte, toplam 30-50 x 106 hücre nin transe olduğunu belirtin. Bunun için 15 mL konik tüpte 5 x 106 hücre/mL'den 2 mL aliquot ve belirtildiği gibi devam edin.
- Yapışık hücre hatları için, transdüksiyondan sonra puromisin 24 saat ekleyerek transdükse hücreleri seçin.
NOT: Bir doz yanıt öldürme eğrisi gerçekleştirerek püromisin konsantrasyonlarını optimize edin. Normalde 1-10 μg/mL arasındaki konsantrasyonlar transedite olmayan hücreleri 3-5 gün içinde öldürür. Bu, tek bir kılavuz RNA (sgRNA) tarafından transdüklenen hücreleri seçme şansını artırabilir, çünkü puromisin yüksek konsantrasyonlarda kullanmaktan kaçının. - Süspansiyon hücreleri için, hasat transduced (yani, BFP pozitif) hücreleri 3 gün posttransdüksiyon bir hücre ayırıcı kullanarak ve en az 10 x 106 hücreleri içeren kütüphaneler oluşturmak. BFP kullanılarak seçildikten sonra, uygun miktarda puromisin ile desteklenen ortamdaki hücreleri büyütün.
NOT: Süspansiyon hücre hatları için sadece puromisin ile seçimleri kaçının, çünkü hücre sıralamasını engelleyebilecek süspansiyon hücre kültürlerinden ölü hücreleri ve enkazları çıkarmak zordur. - Kültür mutant kütüphanesi için 9-16 gün posttransdüksiyon düzenli geçiş ile her 2-3 günde.
8. Hücre yüzeyinin bağlanması için genetik tarama
- 5 dk için 200 x g mutant hücre kütüphanesi pelet ve PBS hücreleri resuspend.
- Hücreleri her tüpte en az 50 x 106 hücre içeren iki adet 15 mL konik tüpe bölün.
- 5 dk için 200 x g bir konik tüp spin, supernatant çıkarın ve -20 °C'de hücre pelet dondurun. Bu kontrol popülasyonudur ve daha sonra işlenir.
- PBS/1% BSA 10 mL diğer tüp pelet resuspend. 96 kuyu plakası üzerinde negatif kontrol olarak 100 μL'lik hücreleri bir kenara koyun.
- Konik tüpteki hücre süspansiyonuna uygun prekonjuge rekombinant proteini ve negatif kontrol proteinlerini 96 kuyu plakasına ekleyin.
- Hücre boyamayı 4 °C'de en az 1 saat boyunca nazik bir rotasyonla (6 rpm) bir tezgah üstü rotorüzerinde gerçekleştirin.
- 5 dk için 200 x g hücreleri pelet, supernatant çıkarın. İki yıkama adımı gerçekleştirin ve 5 mL PBS'deki hücreleri yeniden askıya alın.
- Hücre kümelerini kaldırmak için 30 μm'lik bir hücre süzgeci olsa hücreleri zorlayın. Akış ayırıcıkullanarak analiz edin.
- BFP+/PE- hücreleri için negatif kontrol örneğini kapıya kullanın.
- Örneği sıralayın ve BFP+/PE- hücrelerini toplayın. Sıralama kapıları hücrelerin proteine bağlanmasına bağlıdır ancak normalde PE negatif numunelerin %1-5'i toplanır. Sıralama kapısı örneği Ek Şekil 1'deverilmiştir.
- Seçili kapıdan 500.000-1.000.000 hücre toplayın. Hücrelerin düşük sayıda göz önüne alındığında, kayıpları en aza indirmek için 1,5 mL santrifüj tüpü örnekleri toplama düşünün.
- Pelet 5 dk. 500 x g santrifüj tarafından sıralanmış hücreleri dikkatlice supernatant çıkarın ve atın. Peletin -20 °C'de 6 aya kadar saklayabilirsiniz.
9. Genomik DNA ekstraksiyonu ve gRNA zenginleştirme için ilk PCR
- Genomik DNA'yı yüksek karmaşıklıkkontrol popülasyonundan çıkar.
- Kontrol popülasyonu -20 °C'de dondurulduysa, konik tüpü çıkarın ve PBS ekleyin. Peleti eritmek için buzda tutun.
- 50 x 106 hücreden genomik DNA çıkarmak için üreticinin önerilerini kullanarak ticari bir kit (Bkz. Malzeme Tablosu)kullanın. DNA konsantrasyonu 1 mg/mL'ye ayarlayın.
- Her örnek için, 72 μg DNA'ya karşılık gelen PCR için bir ana karışım ayarlayın. Aliquot 50 μL iyi başına 36 kuyuda 96 iyi PCR plaka. Gerekli astar dizileri Tablo 4'telistelenmiştir. Kılavuzu Tablo 5 ve 6'dakullanın.
- PCR'nin 5 μL'sini %2 (w/v) agarose jel üzerinde 6-12 temsili numuneden çözün. ~250 bp'de tek bir açık bant gözlenmelidir. Bantlar soluksa, 2-3 döngü için PCR'yi tekrarlayın.
- Her kuyudan (toplam 180°L) 5 μL PCR ürün toplamak ve ticari bir kitten 900 μL bağlayıcı arabellek içeren bir rezervuarda havuza çıkarmak için çok kanallı bir pipet kullanın (Bkz. Malzeme Tablosu).
- PCR ürünlerini ticari bir PCR arıtma kiti kullanarak arındırın. DNA'yı ticari bir kitten 50 μL'lik elüsyon tamponuna dönüştürün (Bkz. Malzeme Tablosu)ve DNA konsantrasyonu ölçün.
- Bağlayıcı fenotip kaybı için sıralanmış örneklerin çok sayıda bağımsız klondan oluşması olası değildir. Bu nedenle PCR'nin 72 g DNA ile yapılması gerekli değildir. UYGUN bir ticari kit kullanarak DNA'yı izole edin (bkz. Malzemeler Tablosu). 100 ng/μL DNA ile daha önce açıklanan protokolü (bölüm 9.1.3) kullanarak 3-4 PCR reaksiyonları ayarlayın. Sıralanmış hücre sayısı 100.000'den az ise genomik DNA preparatları yerine hücre lisatları kullanın.
- Aliquot yaklaşık 10.000 hücre / iyi bir 96 iyi PCR plaka.
- Plakadaki hücreleri pelet ve dikkatle supernatant en kaldırın. Pelet görünmez.
- Her kuyuya 25°L su ekleyin ve numuneleri 95 °C'de 10 dakika ısıtın.
- 5 mg/mL taze seyreltilmiş proteinaz K'yi her kuyuya 1 saat ve inkübün 56 °C'de ekleyin. Daha sonra proteini K inaktive etmek için numuneyi 95 °C'de 10 dakika ısıtın.
- PCR reaksiyonu başına 10 μL hücre lisat karışımı kullanın. Lysates 24 saat içinde kullanılmalıdır.
10. Dizin barkodlama ve sıralama için PCR ikinci tur
- Ürünleri ilk tur PCR'den 40 pg/μL'ye seyreltin.
- Örnek başına bir PCR ayarlayın (Tablo 7 ve 8'desağlanan kılavuzu kullanın). SgRNA amplifikasyonu sırasında polimerazTarafından ortaya çıkan hataları en aza indirmek için yüksek kaliteli polimeraz kullanımı önemlidir.
- %2 (w/v) agarose jel üzerinde 5 μL PCR ürün çözün. ~330 bps'de tek bir açık bant gözlenmelidir.
- PCR ürünlerine 31,5 μL (0,7 x toplam hacim) ekleyerek, iyi karıştırarak ve RT'de 5 dakika kuluçkaya yatarak PCR ürünlerini paramanyetik boncuklar kullanarak arındırın.
- Tüpü 3 dakika boyunca manyetik bir rafa yerleştirin. Boncuklar plakanın yan tarafında yakalanmalı ve çözelti açık olmalıdır. Supernatant'ı dikkatlice çıkarın ve atın.
- Tüpe %80 taze hazırlanmış etanol 150 μl ekleyin. 30 s için kuluçka, ve sonra dikkatle kaldırmak ve supernatant atın.
- 13.6 adımtekrar, 180 μL ile bu kez. Sonra 5 dakika boyunca boncuk kuru.
- Tüpü mıknatıstan çıkarın. Boncuklardan 35 μL steril EB tamponuna kadar elute DNA hedefi. 3 dakika kuluçka, sonra 3 dakika için manyetik raf geri tüp koymak.
- Eluted PCR ürünlerini içeren süpernatantın yaklaşık 30 μL'sini temiz bir tüpe aktarın.
- Örnekleri yeni nesil sıralama platformunda sırala. HumanV1 gRNA kitaplığı için Tablo 4'te listelenen özel astarı kullanarak 19 bp sırasını kullanın.
11. Reseptör ve ilgili yolları belirlemek için biyoinformatik analizi
- MAGeCK'nin sayı işlevini kullanarak sıralanmış ve sıralanmamış popülasyondan başvuru kitaplığına kadar olan harita dizileri. İşlev ham sayım dosyası verir (Ek Tablo 1).
NOT: MAGeCK'nin kurulumu ve MAGeCK içinde farklı işlevlerin kullanımı ile ilgili ayrıntılı talimatlar Wang ve ark.20tarafından daha önce yayınlanmış bir protokolde açıklanmıştır. - Ekranda kullanılan denetim kitaplığı teknik standardını kontrol edin.
- Ham sayıları ortalamahale getirin ve ggplot2 paketini R21 veya eşdeğer yazılımda kullanarak plazmid'deki sayıların ampirik kümülatif yoğunluk fonksiyon çizimini çizer ve sıralanmamış numuneleri kontrol eder(Şekil 5A).
- Plazmid popülasyonundaki sayıları "kontrol" olarak kullanarak MAGeCK'nin test işlevini ve sıralanmamış kontrol örneklerinden gelen sayıları "test" örneği olarak çalıştırın. Fonksiyon bir gen özeti dosyası(Ek Tablo 1)verecektir.
- Gen özet dosyasını açın ve daha önce kategorize edilmiş temel ve gereksiz genler için log-fold-changes(neg|lfc sütunu) dağılımınıçizin 22 (Şekil 5B).
- Önemli ölçüde tükenmiş genleri seçin(neg|fdr < 0.05) ve zenginleştirici23 paketini veya R'deki eşdeğer yol zenginleştirme paketlerini kullanarak yol zenginleştirme analizi yapın(Şekil 5C).
- Varsayılan ayarla MAGeCK'ın -test işlevini çalıştırın. Çözümleme yaparken sıralanmamış kontrol örneğinden ham sayımları "kontrol" olarak kullanın ve sıralanmış numuneden "tedavi" olarak sayar.
- MAGeCK tarafından oluşturulan gen özet dosyasını açın ve pos|rank sütununa artan sırada sıralayın. FDR (pos|fdr sütunu) < 0,05'i isabetlerin tanımlanması için bir kesme olarak kullanın. Reseptör genellikle yüksek sırada, genellikle ilk pozisyonda.
- R veya eşdeğer bir yazılımda pozitif seçim(pos|score)için Sağlam-Sıralama Algoritması (RRA) puanlarınıçizin( Şekil 5D).
- Zenginleştirilmiş yolları belirlemek için gen isabetlerini seçin ve zenginleştirme paketini veya R'deki eşdeğer yol zenginleştirme paketlerini kullanarak yol zenginleştirme analizi yapın.
Sonuçlar
NCI-SNU-1 ve HEK293 hücrelerinde sırasıyla yapılan insan TNFSF9 ve P. falciparum RH5'in bağlayıcı partnerinin belirlenmesi için iki temsili genom ölçekli nakavt ekranlarından elde edilen verilerin sıralanması sağlanır(Ek Tablo 1). RH5'in bağlayıcı davranışı hem heparan sülfat tan hem de bilinen reseptörÜ BSG24 'den(Şekil 3C)etkilenirken, TNFRSF9 özellikle bilinen reseptörü TNFSF9'a bağlı ydı ve çözünür heparin ile preinküsasyon aciltlerini kaybetmedi. Şekil 3B'deki protein 3 TNFRSF9'u temsil eder.
Her iki hücre hattı için de 3 gün sonra kontrol mutant kütüphanesinde gRNA dağıtımı (9, 14 ve 16 gün posttransdüksiyon) sağlanır(Ek Tablo 1). GRNA dağılımı, kitaplık karmaşıklığının deney boyunca korundu(Şekil 5A). TNFSF9 için ligandın tanımlanması için genetik tarama 14. Ekranların teknik kalitesi, temel genlerin referans kümesine göre gerekli olmayan genlerin referans kümesini hedefleyen gRNA'ların gözlenen kat-değişikliklerinin dağılımı incelenerek değerlendirildi22 (Şekil 5B). Buna ek olarak, yol düzeyinde zenginleştirme de beklenen temel yollar tespit edildi ve önemli ölçüde "drop-out" nüfus içinde orijinal plazmid kütüphane ile kontrol örneği karşılaştırırken zenginleştirilmiş olduğunu ortaya koymuştur. Şekil 5C'de14 nci-SNU-1 örneğine sahip bir örnek gösterilmiştir.
KONTROLdeki gRNA'ların, MAGeCK'nin -test fonksiyonu kullanılarak sıralanmış popülasyona karşı dağılımı (bkz. MAGeCK'den gelen gen özeti çıktısı için Ek Tablo 1) henotibik ekranlardan reseptörü tanımlamak için kullanılmıştır. GEN düzeyi analizinde MAGeCK tarafından bildirilen modifiye RRA skoru p değerlerine göre sıralanan genler üzerinde çizilir. MAGeCK'deki RRA skoru, GRNA'ların sürekli olarak beklenenden daha yüksek sıralandığı bir ölçü sağlar. TNFRSF9 için ekranda, üst hit TNFSF9, TNFRSF9 bilinen bir bağlayıcı ortağıdır(Şekil 5D)oldu. Buna ek olarak, TP53 yolu ile ilgili genlerin bir dizi de tespit edilmiştir. RH5 durumunda, bilinen reseptöre ek olarak(BSG) ve sülfatlı GAGs üretimi için gerekli gen(SLC35B2),ek bir gen(SLC16A1)de tespit edilmiştir (Şekil 5E). SLC16A1 hücrelerin yüzeyine BSG ticareti için gerekli bir refakatçi25. Birlikte, bu sonuçlar doğrudan etkileşen reseptörleri ve bu reseptör için gerekli hücresel bileşenleri tanımlamak için ekranın yeteneğini fonksiyonel bir biçimde hücrelerin yüzeyinde ifade etmek göstermektedir.
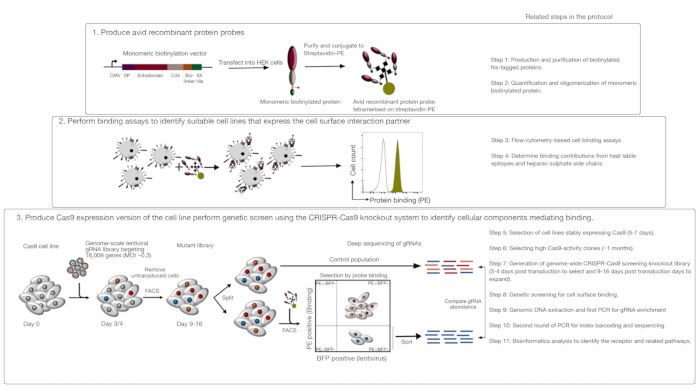
Şekil 1: Hücre yüzey reseptörlerini tanımlamak için genetik tarama yaklaşımına genel bakış. Bu tetkik üç ana adımdan oluşur: Birincisi, hücre yüzey reseptörlerinin ektoetkisini temsil eden rekombinant proteinler HEK293 hücreleri gibi yapısal olarak kritik posttranslational modifikasyonlar ekleyebilirsiniz bir hücre hattı ile ifade edilir. Monomerik protein ektodomains onların bağlayıcı avidite artırmak için streptavidin-PE konjugating tarafından oliomerize edilir. İkinci olarak, bu hevesli problar hücresel bağlayıcı tahlillerde kullanılır, hücre hatlarında PE floresansında belirgin bir kayma ile gösterilen parlak boyama (yeşil) negatif kontrol proteini (siyah) ile karşılaştırıldığında bir hücre yüzeyi bağlama ortağının varlığını gösterir. Üçüncü olarak, reseptör pozitif Cas9 ifade eden hücre hatları seçilir ve protein kodlayan genlerin büyük çoğunluğunu hedefleyen gRNA'lar kullanılarak genom ölçeğinde tarama yapılır. Mutant kütüphaneler oluştururken, her hücrenin belirli bir nakavt atfedilen tek bir gRNA alır sağlayan Poisson dağıtım olasılığı dayalı% 30 transdüksiyon verimliliği, kullanmak yaygındır. Transduced hücreleri tarafından ifade Edilen BFP işaretçisi, FACS kullanılarak gRNA içeren hücreleri seçmek için kullanılır. Phenotipik ekranlar 9-16 gün posttransdüksiyon arasında yapılır. Ekran gününde, toplam mutant hücre popülasyonu ikiye ayrılır. Bir yarısı kontrol popülasyonu olarak tutulur ve diğer yarısı rekombinant protein bağlanması için seçilir. Artık rekombinant protein bağlamak mümkün mutant kütüphane hücreleri FACS kullanılarak sıralanır ve sıralanmış karşı kontrol popülasyonunda gRNA zenginleştirme etiketli avid prob hücre yüzeyi bağlanması için gerekli genleri tanımlamak için kullanılır. Önemli ölçüde zaman gerektiren protokoldeki adımlar belirtilir. Bu rakam Sharma ve ark.19'dandeğiştirilmiştir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
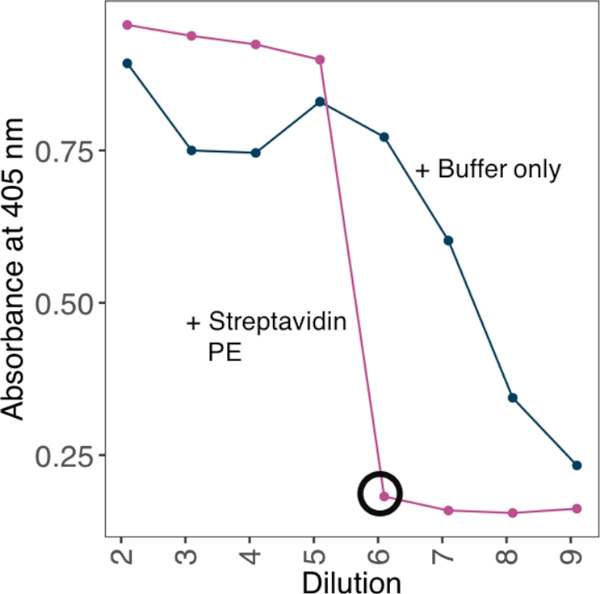
Şekil 2: ELISA tabanlı bir yöntem le biyotinylated proteinin streptavidin-PE'ye oranının belirlenmesi. Streptavidin-PE konjugasyon stratejisinin bir örneği, biyotinylated monomerik proteinden hevesli bir sonda oluşturmak için kullanılır. Biyotinylated monomerlerin seyreltme serisi streptavidin sabit bir konsantrasyon karşı inkübe edildi. Fazla biyotinylated monomerlerin tespit edilebildiği minimum seyreltme ELISA tarafından saptandı. ELISA, 10 ng streptavidin-PE ile bir dizi protein seyreltme ile veya preinktüs yapmadan yapıldı. Streptavidin-PE varlığında, sinyal inanılamayan minimum seyreltme (siyah daire içine alınmış) ve doygunluk için gerekli protein miktarı 4 μg/mL streptavidin-PE ile 10x stok çözeltisi oluşturmak için hesaplanmıştır. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
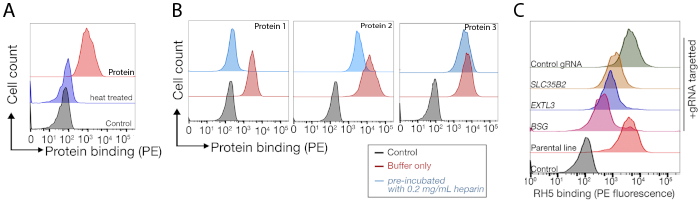
Şekil 3: Proteinlerin hücre hatlarına temsili bağlanması. (A) Hücre hatlarına protein bağlanması, kontrol örneğine göre hücre ile ilişkili floresanda belirgin bir artış göstermiştir. Isıl işlem (10 dk için 80 °C) rekombinant proteinin tüm bağlanmayı negatif kontrole bağlayarak bağlayıcı davranışın doğru katlanmış proteine bağlı olduğunu göstermiştir. (B) Hücre yüzeylerine protein bağlama davranışı farklı sınıflarda; GAGs bağımlılığı. Soldan sağa, proteinler üç tip olarak sınıflandırılabilir: Protein türü 1 sadece HS'ye adsorbs. Bu proteinler preinküsasyon dan sonra heparin konsantrasyonları 0.2 mg/mL'nin üzerinde olan bağlanmalarını kaybederler. Protein tipi 2 belirli bir reseptöre ek olarak HS bağlanır. Bu proteinler ön engelleme deneylerinde kısmi bağlanmayı kaybederler. Protein tipi 3 HS bağlamaz. Bu proteinler ebeveyn çizgileri ile karşılaştırıldığında bağlayıcı kaybetmezsiniz. (C) HS'ye ve belirli bir reseptöre katkı maddesi yle bağlanan bir protein (yani RH5) örneği. HS sentezi için gerekli olan reseptör (örn. BSG) veya enzimleri (örneğin, SLC35B2, EXTL3) hedeflemek, RH5'in kontrollere göre hücrelere bağlanmasını kısmen azaltır. Transduced poliklonal çizgiler bağlayıcı davranış kurmak için bu tür deneylerde kullanılabilir. Bu rakam Sharma ve ark.19'dandeğiştirilmiştir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
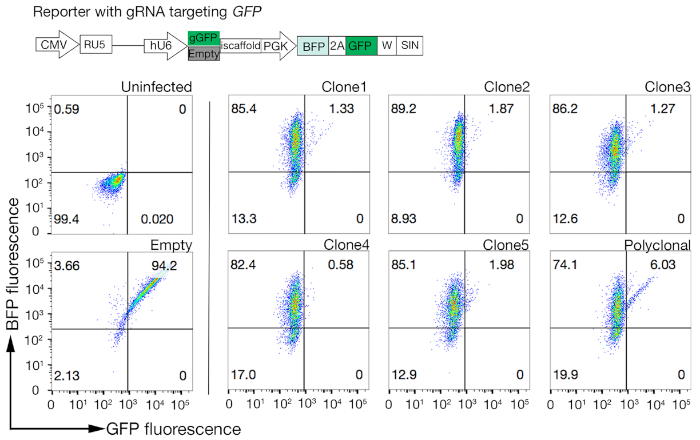
Şekil 4: Yüksek Cas9 aktivitesine sahip klonal hücre çizgileri seçilir. NCI-SNU-1 hücre hatlarının hem çokklonel hem de klonlanmış hatlarının genom düzenleme verimliliği GFP-BFP muhabir sistemi kullanılarak değerlendirildi ve hücre hatları gRNA'yı hedefleyen plazmid kodlu veya gFP'siz (yani "boş") virüslerle aktarıldı. Bir şema tasvir edilmiştir. Akış sitometrisi transdüksiyon sonrası hem BFP hem de GFP ekspresyonunu test etmek ve enfekte olmayan kontrolle karşılaştırıldığında kullanıldı. GFP ifadesi Cas9 etkinliği için proxy olarak kullanılırken, BFP ifadesi transduced hücreleri işaretledi. Enfekte olmayan ve boş enfekte hücreler için profil tüm klonlar için benzer görünüyordu. Temsil profilleri sol panelde gösterilmiştir. NCI-SNU-1 hücre hattının beş klonu da poliklonal çizgiye (sağ panel) göre daha yüksek bir GFP kaybı gösterirken, klon 4 en düşük refrakter popülasyona en yüksek verimi gösterdi. Bu rakam Sharma ve ark.19'dandeğiştirilmiştir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
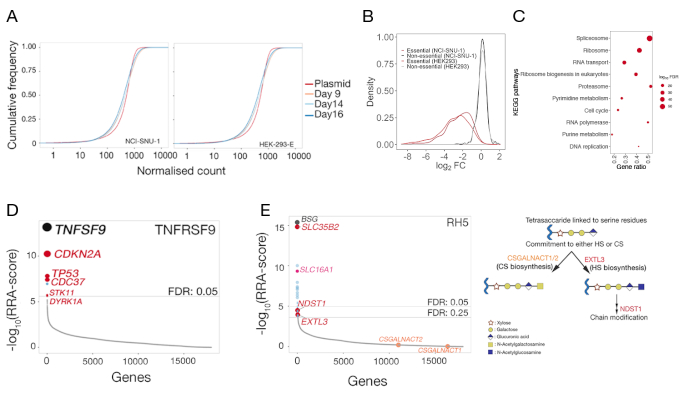
Şekil 5: Hücre yüzeyi bağlayıcı ortakların belirlenmesi için genetik ekranlardan elde edilen sonuçlar. (A) Kümülatif dağılım fonksiyonu, plazmid kütüphanesindeki gRNA bolluğunu 9, 14 ve 16 günlük posttransdüksiyon günlerinde HEK-293-E ve NCI-SNU-1 hücrelerinin mutant kütüphaneleri ile karşılaştırarak çizer. Belirli bir sayı için, kümülatif yoğunluk işlevi bu eşiğin altında olan veri noktalarının yüzdesini bildirir. Mutant hücre popülasyonunun orijinal plazmid popülasyonuna göre küçük değişimi, plazmid kitaplığına kıyasla gRNA alt kümesindeki tükenmeyi temsil eder. (B) HEK293 ve NCI-SNU-1 hücre hatlarında daha önce gerekli (kırmızı) veya gereksiz (siyah) olarak sınıflandırılan genlerdeki log-fold değişikliklerinin dağılımı. Gerekli olmayan genler için kıvrım değişimlerinin dağılımı ~0'da ortalanırken, temel genler negatif kıvrım değişikliklerine doğru sola kaymıştır. (C) NCI-SNU-1 mutant kontrol popülasyonunda 14 gün posttransdüksiyon da tükenmiş genlerdeki önemli ölçüde zenginleştirilmiş yollar. Bilinen hücre esaslı yollar tespit edildi. (D) TNFRSF9 sondasını bağlama yeteneğini kaybetmiş sıralanmış hücrelerde zenginleştirilmiş genler için Sağlam Sıralama Algoritması (RRA)-skoru. Genler RRA skoruna göre sıralandı. Bilinen etkileşim ortağı TNFSF9 ve TP53 yolu ile ilgili genler (kırmızı etiketli) ekranda tespit edildi. (E) RH5'in HEK293 hücrelerine bağlanması için gerekli gRNA zenginleştirme analizinden tanımlanan genler için sıralı RRA puanları (sol panel). SLC35B2 ve SLC16A1%5'lik bir yanlış bulma oranı (FDR) eşiği içinde tanımlanmıştır. FDR içinde HS biyosentez yolunda iki ek gen (örn. EXTL3 ve NDST1)%25 FDR içinde saptandı. İlgili genlerle genel GAG biyosentez yolunu gösteren şema ilgili adımlara eşlenmiştir (panel 2). Ekranda kondroitin sülfat biyogenezine bağlılık için gerekli genler (csgalnact1/2)tanımlanmadı. Bu rakam Sharma ve ark.19'dandeğiştirilmiştir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
| Plazmid adı | Plasmid # | Kullanın |
| Protein ekspresyonu yapısı: CD200RCD4d3+4-bio-linker-his | Ek Leyen Gen: 36153 | CD4d3+4, biotin ve 6-his etiketleri ile rekombinant Protein üretimi. |
| pMD2.G | Ek Leyen Gen: 12259 | Plazmidi ifade eden VSV-G zarfı; lentivirus üretimi |
| psPAX2 | Ek Leyen Gen: 12260 | Lentiviral ambalaj plazmid, lentivirus üretimi |
| Cas9 yapı: pKLV2-EF1a-Cas9Bsd-W | Ek Leyen: 68343 | Cas9 hattının kurucu olarak ifade ediletilmesi nin imalatı |
| gRNA ifade yapısı: pKLV2-U6gRNA5(BbsI)-PGKpuro2ABFP-W | Ek Leyen Gen: 67974 | Geliştirilmiş bir iskele ve puro/BFP belirteçleri ile CRISPR gRNA ifade vektörü |
| İnsan Geliştirilmiş Genom çapında Nakavt CRISPR Kütüphanesi | Ek Leyen Gen: 67989 | Lentivirüs'te kullanılmak üzere tasarlanmış 18.010 insan genine karşı bir gRNA kütüphanesi. |
| GFP-BFP yapısı: pKLV2-U6gRNA5(gGFP)-PGKBFP2AGFP-W | Ek Leyen: 67980 | BFP ve GFP ile Cas9 aktivite muhabiri. |
| Boş yapı: pKLV2-U6gRNA5(boş)-PGKBFP2AGFP-W | Ek Leyen Gen: 67979 | BFP ve GFP ile Cas9 aktivite muhabiri (kontrol). |
Tablo 1: Bu yaklaşımda kullanılan plazmidler.
| Arabellek adı | Bileşen |
| HBS (10X) | MiliQ suda 1,5 M NaCl ve 200 mM HEPES, pH 7,4'e ayarlayın |
| PBS (10X) | MiliQ suda 80 g NaCl, 2 g KCl, 14,4 g Na2HPO4 ve 2,4 g KH2PO4 MiliQ suda, pH 7,4'e ayarlayın |
| Sodyum Fosfat Tamponu (80mM stok) | 7.1 g Na2HPO4.2H2O, 5.55 g NaH2PO4, pH 7.4 ayarlayın |
| Onun-arıtma bağlayıcı tampon | 20 mM Sodyum Fosfat Tamponu, 0.5 M NaCl ve 20 mM Imidazol, pH 7.4'e ayarlayın |
| Onun-arıtma elüsiyon tampon | 20 mM Sodyum Fosfat Tamponu, 0.5M NaCl ve 400 mM Imidazol, pH 7.4'e ayarlayın |
| Diethanolamine tampon | MiliQ suda %10 diethanolamine ve 0,5 mM MgCl2, pH 9.2'ye ayarlayın: |
| D10 | DMEM, %1 penisilin-streptomisin (100 adet/mL) ve %10 ısı inaktive FBS |
Tablo 2: Bu çalışma için tamponlar gereklidir.
| Bileşen | 10 cm çanak | 6-iyi plaka |
| 293FT hücreler | %70-80 konfluent | %70-80 konfluent |
| Transfeksiyon uyumlu ortam (Opti-MEM) (Adım 5.1.2) | 3 mL | 500 μL |
| Transfeksiyon uyumlu ortam (Opti-MEM) (Adım 5.1.4) | 5 mL | 2 mL |
| Lentiviral transfer vektörü | 3 μg | 0,5 g |
| psPax2 (tablo 1'e bakınız) | 7.4 g | 1,2 μg |
| pMD2.G (tablo 1'e bakınız) | 1,6 g | 0,25 g |
| PLUS reaktifi | 12 μL | 2 μL |
| Lipofektamin LTX | 36 μL | 6 μL |
| D10 (Adım 7.1.7) | 5 mL | 1,5 mL |
| D10 (Adım 7.1.8 ve 7.1.10) | 8 mL | 2 mL |
Tablo 3: Lentivirus ambalaj karışımı için reaktif miktarları ve hacimleri.
Tablo 4: GRNA ve NGS'yi yükseltmek için astar dizileri. Bu dosyayı görüntülemek için lütfen buraya tıklayın (İndirmek için sağ tıklatın).
| Reaktif | Reaksiyon başına hacim | Ana karışım (x38) |
| Q5 Hot Start Yüksek Sadakat 2x | 25 μL | 950 μL |
| Astar (L1/U1) karışımı (her biri 10 μM) | 1 μL | 38 μL |
| Genomik DNA (1 mg/mL) | 2 μL | 72 μL |
| H2O | 22 μL | 1100 μL |
| Toplam | 50 μL | 1900 μL |
Tablo 5: Yüksek karmaşıklıktaki örneklerden gRNA'ların amplifikasyonu için PCR.
| Döngü numarası | Denatüre | Tavlama | Uzantısı |
| 1 | 98 °C, 30'lar | ||
| 2-24 | 98 °C, 10'lar | 61 °C, 15'ler | 72 °C, 20'li |
| 25 | 72 °C, 2 dk |
Tablo 6: İlk PCR için PCR koşulları.
| Reaktif | Reaksiyon başına hacim |
| KAPA HiFi HotStart ReadyMix | 25 μL |
| Astar (PE1.0/index astar) karışımı (her biri 5 μM) | 2μL |
| İlk PCR ürünü (40 pg/μL) | 5 μL |
| H2O | 18 μL |
| Toplam | 50 μL |
Tablo 7: Genetik ekranlardan sgRNA indeks etiketleme için PCR.
| Döngü numarası | Denatüre | Tavlama | Uzantısı |
| 1 | 98 °C, 30'lar | ||
| 2-15 | 98 °C, 10'lar | 66 °C, 15s | 72 °C, 20'li |
| 16 | 72 °C, 5 dk |
Tablo 8: İkinci PCR için PCR koşulları.
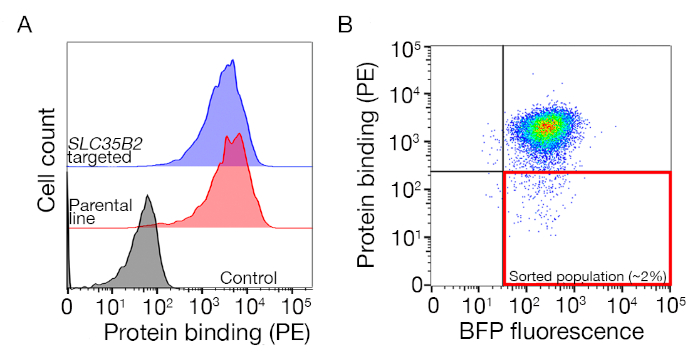
Ek Şekil S1: Bağlayıcı olmayan popülasyonu sıralamak için kapı çizimkılavuzu. (A)Tarama için ideal bir protein adayı, kontrol popülasyonuna göre bağlayıcı popülasyonda net bir değişime sahip olmalı ve HS biyosentezi için makine bulunmayan hücrelerde bağlanma tutulmalıdır. Bir heparin engelleme deneyi SLC35B2 hedefli hücre hatları üzerinde test yerine kullanılabilir. (B) Protein ektoetkialanından yüzey boyaması olmayan ancak lentiviral transdüksiyondan BFP floresansını ifade eden hücreler toplandı. Görüntülenen hücreler GABBR222için reseptör belirlenmesi için bir ekrandan vardır. Bu rakam Sharma ve ark.19'dandeğiştirilmiştir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
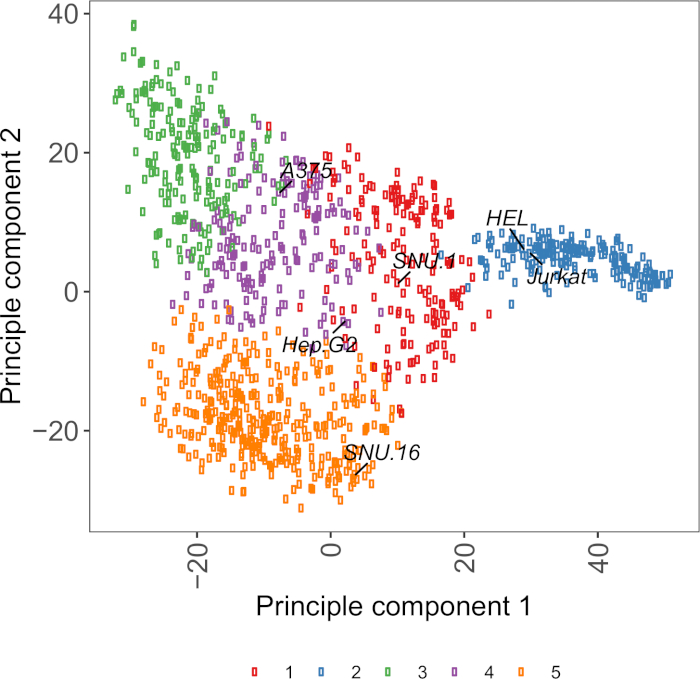
Ek Şekil S2: Hücre yüzeyglikoprotein transkripsiyon eldeki PCA çizimi 1.000'den fazla kanser hücresi hattından gelen RNA-seq verileri kullanılarak. Hücre Modeli Passport27'nin hücre hatları ~1.500 hücre yüzeyglikoproteinlerinin FPKM değerlerine göre K-araçları kümeleme kullanılarak kümelenmiştir. Her kümedeki temsili hücre çizgileri etiketlenir. Küme 5 tamamen hematopoetik kökenli hücre çizgilerinden oluşuyordu (ayrıca ek tablo 2'yebakınız). Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
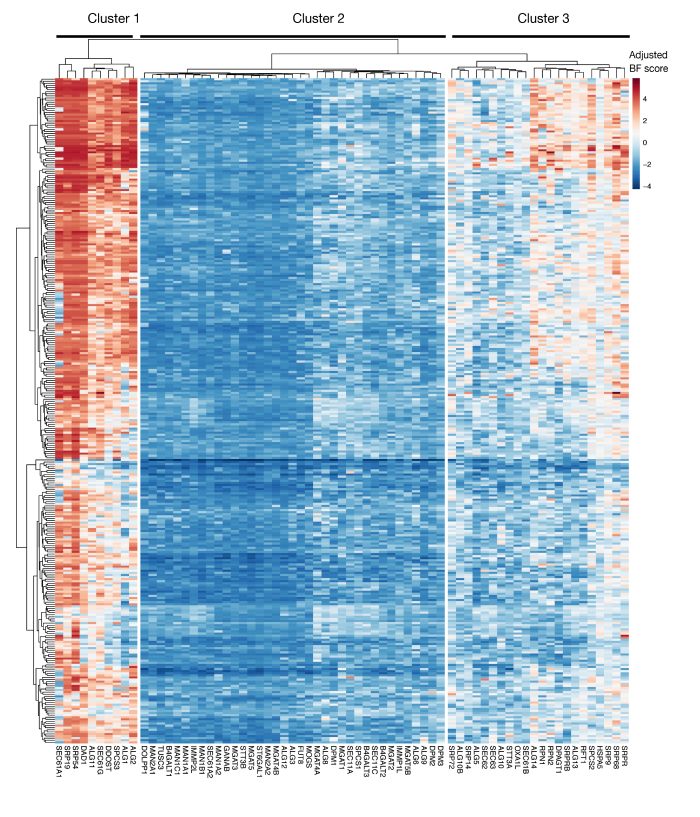
Ek Şekil S3: Proje skorlarından KEGG-ek açıklama protein ihracatı ve N'ye bağlı glikozilasyon genleri için özgünlük puanları. ~330 hücre hatları (sütunlar, etiketli değil) için ayarlanmış Bayes-özgünlük puanları protein ihracat ve N-bağlı glikozilasyon yolu (X-eksen) genleri için çizilmiştir. 0'dan yüksek puanlar, mutant popülasyonda orijinal plazmid kütüphanesine kıyasla önemli bir tükenmeyi temsil eder. Genler, hücre hatlarındaki farklı özgünlük düzeylerini temsil eden üç farklı kümeye ayrılabilir. Bu kümeleme sıralama gününe karar vermek için kullanılabilir. Ekran geç bir zaman noktasında (16. gün) yapılırsa, hücreler için gerekli olduğu bilinen genlerin (küme 1 ve 3) tanımlanmaması mümkündür. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Ek Tablo 1: Ham sayım dosyaları ve MAGeCK yazılımı, temsili genetik ekranlarla ilgili gene_summary dosyalar oluşturdu. Bu dosyayı görüntülemek için lütfen buraya tıklayın (İndirmek için sağ tıklatın).
Ek Tablo 2: Hücre yüzey reseptörlerinin ekspresyonuna göre hücre hatlarının kümelanması. Bu dosyayı görüntülemek için lütfen buraya tıklayın (İndirmek için sağ tıklatın).
Tartışmalar
Hücresel tanıma ile ilgili hücresel bileşenleri kodlayan genleri tanımlamak için CRISPR tabanlı bir tarama stratejisi tanımlanmıştır. CRISPR aktivasyonu kullanarak benzer bir yaklaşım da büyük protein kütüphaneleri oluşturmak için gerek kalmadan rekombinant proteinlerin doğrudan etkileşim reseptörlerini belirlemek için genetik bir alternatif sağlar26. Ancak, bu yaklaşımın en önemli bir avantajı, hücre üzerinde doğal olarak görüntülenen yüzey molekülleri tarafından aracılık etkileşimleri için geçerli olmasıdır ve reseptörün bağlayıcı avidite etkileyebilir reseptörlerin aşırı ekspresyonu bağlı değildir. Diğer yöntemlerin aksine, bu nedenle, bu teknik reseptörlerin biyokimyasal doğası veya hücre biyolojisi ile ilgili hiçbir varsayımda bulunmaz ve normalde çok büyük proteinler gibi biyokimyasal yaklaşımlar kullanılarak incelenmesi zor olan proteinlerin aracılık ettiği etkileşimleri veya membranı birden fazla kez geçen veya diğer proteinlerle kompleksler oluşturan ve glikan, glikolipid ve fosfolipid gibi proteinler dışındaki molekülleri inceleme fırsatı sağlar. Yöntemin genom ölçekli doğası göz önüne alındığında, bu yaklaşım aynı zamanda reseptör ütülünün tanımlanması değil, aynı zamanda bağlayıcı olay için gerekli olan ek hücresel bileşenlerin de avantajına sahiptir, böylece reseptörün hücre biyolojisi hakkında bilgi sağlar.
Bir yetim proteinin reseptörü tanımlamak için kullanırken bu yöntemin önemli sınırlamalarından biri ilk proteine bağlanan bir hücre hattı tanımlamak için ilk gereksinimdir. Bu her zaman kolay değildir ve genetik ekranlara da izin veren bağlayıcı bir fenotip gösteren bir hücre hattını tanımlamak bu taslağı dağıtmak için zaman sınırlayıcı bir adım olabilir. Bazı hücre hatları diğerlerinden daha fazla proteine bağlanma eğilimindedir. Bu özellikle HS'yi bağlayan proteinler için önemlidir, çünkü bu proteinler yerel bağlama bağlamına bakılmaksızın HS yan zincirlerini gösteren herhangi bir hücre hattına bağlanma eğilimindedirler. Ayrıca hücre hatlarında sindekanların (yani HS içeren proteoglikanlar) yükseltisinin HS bağlayıcı proteinlerin daha fazla bağlanmasına yol açtığını gözlemledik26. Bu tarama için hücre hattı seçerken dikkate alınması gereken bir faktör olabilir. Ancak, dikkat edilmesi gereken önemli olan HS'nin katkı maddesinin belirli bir reseptöre bağlanmasını engellememesidir. Bu, bağlama gözlenirse, bu teşbikte HS'nin aracılık ettiği bağlayıcının19'abağımlı değil katkı maddesi olması nedeniyle yalnızca HS tarafından aracılık edilmesinin mümkün olduğu anlamına gelir. Böyle bir senaryoda, açıklanan heparin engelleme yaklaşımı tam bir genetik tarama yapmaya gerek kalmadan bu tür davranışları belirleyebilir.
Hücre hatları seçmek için yararlı bir kaynak Hücre Modeli Pasaport, genomik içeren, transkripsiyon, ve kültür durum bilgileri ~ 1,000 kanser hücre hatları27. Biyolojik içeriğe bağlı olarak, hücreler ifade profillerine göre seçilebilir. Hücre hatlarının seçimine yardımcı olmak için, ~1.500 premaye edilmiş insan hücresi yüzey glikoproteinleri28 ifadesine göre Hücre Modeli Pasaport ~1.000 hücre hatları kümelenmiş 28 (Ek Şekil 2; büyüme koşulları ile birlikte her hücre hattı için küme bilgileri Ek Tablo 2'deverilmiştir ). Bilinmeyen fonksiyonu ile bir proteinin bağlanması test ederken, reseptörlerin geniş bir yelpazede kapsayan şansını artırmak için her kümeden temsili hücre hatları bir panel seçmek yararlıdır. Bir seçim göz önüne alındığında, kültür ve transduce kolay hücre hatları seçmek için tavsiye edilir. Bu hücre hatları genom ölçekli taramada kullanılacağı için, daha sonraki adımlarda CRISPR tabanlı genetik tarama için sgRNA'nın teslimi için en yaygın yöntem olduğu için, büyük miktarlarda kolayca yetiştirilebilen ve lentiviral transdüksiyona izin verilmesi tercih edilir.
Genellikle, fenotip seçimleri tek bir sıralamada gerçekleştirilir. Ancak, bu kontrol ile karşılaştırıldığında lekeli hücre popülasyonunun parlaklığı ile belirlenir. İstenilen fenotipin sinyal-gürültü oranının düşük olduğu veya ekranın amacının güçlü fenotiplere sahip mutantları belirlemek olduğu senaryolar için yinelemeli seçim turları benimsenebilir. FACS tabanlı ekranlar için yinelemeli bir seçim yaklaşımı kullanırken, sıralama işleminin esas olarak ayıklayıcının katıksız gücü nedeniyle hücre ölümüne neden olabileceğini göz önünde bulundurmak önemlidir. Böylece, toplanan tüm hücreler sıralama sonraki turda temsil edilecektir.
Kütüphane karmaşıklığı başarılı genetik ekranlar, özellikle negatif seçim ekranları için performans çok önemli bir faktördür, çünkü bu tükenme ölçüde sadece başlangıç kitaplığı mevcut ne sonuçları karşılaştırArak belirlenebilir. Negatif seçim ekranları için, 500-1.000 x karmaşıklıkta kitaplıklar korumak için yaygındır. Pozitif seçim ekranları, ancak, kütüphane boyutları için daha sağlam, bu tür ekranlarda mutantların sadece az sayıda belirli bir fenotip için seçilmesi bekleniyor çünkü. Bu nedenle, burada açıklanan olumlu seçim ekranında, kitaplık boyutu ekranın kalitesinden ödün vermeden 50-100x karmaşıklığa düşürülebilir. Buna ek olarak, bu ekranlarda belirli bir günde belirli bir hücre hattı için bir denetim kitaplığı kullanmak da mümkündür verilen hücre hattı için gün sıralanmış tüm örnekler için bir "genel kontrol" olarak. Bu, üretilmesi ve sıralanmış olması gereken denetim kitaplıklarının sayısını azaltır.
Bu yaklaşımın kullanımı için bir diğer önemli husus in vitro hücre büyümesi için gerekli olan genlerin belirlenmesinde fonksiyon kaybı ekranlarının sınırlamalarıdır. Bu bağlamda ekranların zamanlaması çok önemlidir, çünkü mutant hücreler kültürde ne kadar uzun süre tutulursa, temel genlerdeki mutasyona sahip hücrelerin canlı olma olasılığı da o kadar yüksektir ve artık mutant kütüphanesinde temsil edilmiyor. 300'den fazla hücre hattında Proje Puanı girişiminin bir parçası olarak gerçekleştirilen son genetik ekranlar, KEGG-açıklamalı protein salgılaması ve N-glikozilasyon yolundaki birden fazla genin genellikle bir dizi hücre hattı için gerekli olarak tanımlandığını göstermektedir (Ek Şekil 3)29. Bu durum, hücresel tanıma süreci bağlamında çoğalma ve canlılık için gerekli genlerin etkisinin araştırılamaması durumunda ekran tasarımında göz önünde bulundurulabilir. Bu durumda, ekranların erken bir zaman noktasında (örn. 9. gün posttransdüksiyon) yapılması genel olarak uygun olacaktır. Ancak, yaklaşım genel hücresel yollar yerine güçlü boyut etkileri olan birkaç hedefi tanımlamak için kullanılırsa, daha sonraki bir zaman noktasında (örneğin, gün 15-16 posttransdüksiyon) ekranlar gerçekleştirmek için uygun olabilir.
Tarama sonuçları çok sağlam; geçmişte yapılan sekiz rekombinant protein bağlama ekranlarında, hücre yüzey reseptörü her durumda en çok etkilenen19. Etkileşim ortağını tanımlamak için bu yaklaşımı kullanırken, bu nedenle reseptörün ve yüzeydeki sunumuna katkıda bulunan faktörlerin yüksek istatistiksel güvenle tanımlanması beklenmelidir. Ekran yapıldıktan ve tek bir gRNA nakavt kullanılarak bir vuruş doğrulandıktan sonra, AVEXIS4 gibi mevcut biyokimyasal yöntemler ve yüzey plazmon rezonansı kullanılarak saflaştırılmış proteinlerin doğrudan doygun bağlanması kullanılarak daha fazla takip yapılabilir. Burada açıklanan yaklaşım, çözünür rekombinant bağlayıcı probu oluşturmak mümkün olan tüm proteinler için geçerlidir.
Özetle, bu hücre yüzeyproteinleri tarafından aracılık etkileşimleri belirlemek için bir genom ölçekli CRISPR nakavt yaklaşımdır. Bu yöntem genellikle hücre yüzeyinin tanınması için gerekli hücresel yolları belirlemek için bir organizmanın kendi hücreleri (örneğin, nöral ve immünolojik tanıma) ve konak hücreleri ve patojen proteinler arasında da dahil olmak üzere çok çeşitli biyolojik bağlamlarda uygulanabilir. Bu yöntem, reseptör tanımlaması için tasarlanmış biyokimyasal yaklaşımlara genetik bir alternatif sağlar ve reseptörlerin biyokimyasal doğası veya hücre biyolojisi ile ilgili önceden varsayımlar gerektirmediği için tamamen beklenmedik keşifler yapma potansiyeline sahiptir.
Açıklamalar
Yazarların açıklayacak bir şeyi yok.
Teşekkürler
Bu çalışma, GJW'a verilen 206194 wellcome trust hibe numarası ile desteklenmiştir. Biz Cytometry Core tesisi teşekkür ederiz: Bee Ling Ng, Jennifer Graham, Sam Thompson, ve Christopher Hall FACS ile yardım için.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Anti-mouse alkaline phosphatase | Sigma | A4656 | |
| Blasticidin | Chem-Cruz | SC-204655 | |
| Blood & Cell Culture DNA Maxi Kit | Qiagen | 13362 | |
| BSA | Sigma | A9647-100G | |
| Diethanolamine | Sigma | 398179 | |
| DMEM | Gibco | 31966-021 | |
| Dneasy Blood and Tissue kit | Qiagen | 69504 | |
| DynaMag-96 Side Magnet | Invitrogen | 12331D | |
| HEK293T packaging cells | ATCC | CRL-3216 | |
| Heparin | Sigma | H4784-1G | |
| KAPA HiFi HotStart ReadyMix | Kapa | KK2602 | |
| Lipofectamine LTX with PLUS reagent | Invitrogen | 15338100 | |
| MoFlo XDP cell sorter | BD | ||
| Ni2+-NTA agarose beads | Jena Bioscience | AC-501-25 | |
| OPTI-MEM | Life Technologies | 31985-070 | |
| OX-68 antibody | AbD Serotec | MCA1022R | |
| p-nitrophenyl phosphate | Sigma | 1040-506 | |
| PD-10 desalting columns | GE healthcare | 17085101 | |
| Polybrene | Millipore | TR-1003-G | |
| Polypropylene tubes with 5 mL bed volume | Qiagen | 34964 | |
| Proteinase K, recombinant, PCR Grade | Roche | 3115879001 | |
| Puromycin | Gibco | A11138-03 | |
| Q5 Hot Start High-Fidelity 2× Master Mix | NEB | M0494L | |
| QIAquick PCR purification kit | Qiagen | 28104 | |
| SCFA filter | Nalgene | 190-2545 | |
| Sony Cell sorter | Sony | ||
| SPRI beads (Agencourt AMPure XP beads) | Beckman | A63881 | |
| Streptavidin-coated microtitre plates | Nalgene | 734-1284 | |
| Streptavidin-PE | Biolegend | 405204 |
Referanslar
- Wright, G. J. Signal initiation in biological systems: the properties and detection of transient extracellular protein interactions. Molecular bioSystems. 5 (12), 1405-1412 (2009).
- van der Merwe, P. A., Barclay, A. N. Transient intercellular adhesion: the importance of weak protein-protein interactions. Trends in Biochemical Sciences. 19 (9), 354-358 (1994).
- Wood, L., Wright, G. J. Approaches to identify extracellular receptor-ligand interactions. Current Opinion in Structural Biology. 56, 28-36 (2019).
- Bushell, K. M., Söllner, C., Schuster-Boeckler, B., Bateman, A., Wright, G. J. Large-scale screening for novel low-affinity extracellular protein interactions. Genome Research. 18 (4), 622-630 (2008).
- Visser, J. J., et al. An extracellular biochemical screen reveals that FLRTs and Unc5s mediate neuronal subtype recognition in the retina. eLife. 4, e08149 (2015).
- Özkan, E., et al. An extracellular interactome of immunoglobulin and LRR proteins reveals receptor-ligand networks. Cell. 154 (1), 228-239 (2013).
- Martinez-Martin, N., et al. An Unbiased Screen for Human Cytomegalovirus Identifies Neuropilin-2 as a Central Viral Receptor. Cell. 174 (5), 1158-1171 (2018).
- Bianchi, E., Doe, B., Goulding, D., Wright, G. J. Juno is the egg Izumo receptor and is essential for mammalian fertilization. Nature. 508 (7497), 483-487 (2014).
- Mullican, S. E., et al. GFRAL is the receptor for GDF15 and the ligand promotes weight loss in mice and nonhuman primates. Nature Medicine. 23 (10), 1150-1157 (2017).
- Turner, L., et al. Severe malaria is associated with parasite binding to endothelial protein C receptor. Nature. 498 (7455), 502-505 (2013).
- Frei, A. P., et al. Direct identification of ligand-receptor interactions on living cells and tissues. Nature Biotechnology. 30 (10), 997-1001 (2012).
- Sobotzki, N., et al. HATRIC-based identification of receptors for orphan ligands. Nature Communications. 9 (1), 1519 (2018).
- Sharma, S., Petsalaki, E. Application of CRISPR-Cas9 Based Genome-Wide Screening Approaches to Study Cellular Signalling Mechanisms. International Journal of Molecular Sciences. 19 (4), (2018).
- Gebre, M., Nomburg, J. L., Gewurz, B. E. CRISPR-Cas9 Genetic Analysis of Virus-Host Interactions. Viruses. 10 (2), (2018).
- Zotova, A., Zotov, I., Filatov, A., Mazurov, D. Determining antigen specificity of a monoclonal antibody using genome-scale CRISPR-Cas9 knockout library. Journal of Immunological Methods. 439, 8-14 (2016).
- Puschnik, A. S., Majzoub, K., Ooi, Y. S., Carette, J. E. A CRISPR toolbox to study virus-host interactions. Nature Reviews. Microbiology. 15 (6), 351-364 (2017).
- Kerr, J. S., Wright, G. J. Avidity-based extracellular interaction screening (AVEXIS) for the scalable detection of low-affinity extracellular receptor-ligand interactions. Journal of Visualized Experiments. (61), e3881 (2012).
- Tzelepis, K., et al. A CRISPR Dropout Screen Identifies Genetic Vulnerabilities and Therapeutic Targets in Acute Myeloid Leukemia. Cell Reports. 17 (4), 1193-1205 (2016).
- Sharma, S., Bartholdson, S. J., Couch, A. C. M., Yusa, K., Wright, G. J. Genome-scale identification of cellular pathways required for cell surface recognition. Genome Research. 28 (9), 1372-1382 (2018).
- Wang, B., et al. Integrative analysis of pooled CRISPR genetic screens using MAGeCKFlute. Nature Protocols. 14 (3), 756-780 (2019).
- Hart, T., et al. Evaluation and Design of Genome-Wide CRISPR/SpCas9 Knockout Screens. G3. 7 (8), 2719-2727 (2017).
- Kuleshov, M. V., et al. Enrichr: a comprehensive gene set enrichment analysis web server 2016 update. Nucleic Acids Research. 44 (W1), W90-W97 (2016).
- Crosnier, C., et al. Basigin is a receptor essential for erythrocyte invasion by Plasmodium falciparum. Nature. 480 (7378), 534-537 (2011).
- Kirk, P., et al. CD147 is tightly associated with lactate transporters MCT1 and MCT4 and facilitates their cell surface expression. The EMBO Journal. 19 (15), 3896-3904 (2000).
- Chong, Z. S., Ohnishi, S., Yusa, K., Wright, G. J. Pooled extracellular receptor-ligand interaction screening using CRISPR activation. Genome Biology. 19 (1), 205 (2018).
- van der Meer, D., et al. Cell Model Passports-a hub for clinical, genetic and functional datasets of preclinical cancer models. Nucleic Acids Research. 47 (D1), D923-D929 (2019).
- Bausch-Fluck, D., et al. A mass spectrometric-derived cell surface protein atlas. PloS One. 10 (3), e0121314 (2015).
- Behan, F. M., et al. Prioritization of cancer therapeutic targets using CRISPR-Cas9 screens. Nature. 568 (7753), 511-516 (2019).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır