Method Article
Идентификация рецепторов поверхности клетки используя Геном-Шкала CRISPR/Cas9 Генетические экраны
В этой статье
Резюме
Данная рукопись описывает подход к скринингу на основе клеток в масштабе генома для выявления внеклеточных взаимодействий рецепторов-лигандов.
Аннотация
Межклеточная коммуникация, опосредованное прямыми взаимодействиями между мембранными клеточными рецепторами, имеет решающее значение для нормального развития и функционирования многоклеточных организмов. Однако обнаружение этих взаимодействий остается технически сложным. Данная рукопись описывает систематический подход к генетическому скринингу, основанному на геноме CRISPR/Cas9, который показывает клеточные пути, необходимые для конкретных событий распознавания поверхности клеток. Этот анализ использует рекомбинантные белки, вырабатываемые в системе экспрессии белка млекопитающих в качестве заядлых связывающих зондов для выявления партнеров по взаимодействию на генетическом экране на основе клеток. Этот метод может быть использован для определения генов, необходимых для взаимодействия поверхности клеток, обнаруженных рекомбинантными связывающими зондами, соответствующими эктодоменам мембранных рецепторов. Важно отметить, что, учитывая геном масштаба природы этого подхода, он также имеет то преимущество, не только выявление прямого рецептора, но и клеточных компонентов, которые необходимы для представления рецептора на поверхности клетки, тем самым обеспечивая ценную информацию о биологии рецептора.
Введение
Внеклеточные взаимодействия белков клеточных рецепторов поверхности направляют важные биологические процессы, такие как организация тканей, распознавание хоста и патогена и иммунная регуляция. Исследование этих взаимодействий представляет интерес для более широкого биомедицинского сообщества, потому что мембранные рецепторы являются действенными целями систематически поставляемых терапевтических препаратов, таких как моноклональные антитела. Несмотря на их важность, изучение этих взаимодействий остается технически сложной задачей. Это главным образом потому, что мембранные рецепторы амфипатических, что делает их трудно изолировать от биологических мембран для биохимических манипуляций, и их взаимодействия являются олицетворяемыми слабым имитителями (KDв диапазоне мМ-ММ)1. Следовательно, многие широко используемые методы не подходят для обнаружения этого класса белковых взаимодействий1,,2.
Ряд методов был разработан специально для изучения внеклеточных рецепторов-лиганд взаимодействий, которые принимают их уникальные биохимические свойства во внимание3. Ряд из этих подходов включают выражение всего эктодона рецептора в качестве растворимого рекомбинантного белка в системах млекопитающих или насекомых, основанных на клетках, чтобы гарантировать, что эти белки содержат постпереводные изменения, которые являются структурно важными, такие как гликаны и дисульфидные связи. Чтобы преодолеть с низким содержанием связывания, эктодоменов часто олигомеризированы, чтобы увеличить их связывания алчность. Заядлый белок эктодомеменов были успешно использованы в качестве связывающих зондов для выявления партнеров взаимодействия в прямых рекомбинантных белково-белковых экранов взаимодействия4,,5,,6,7. Хотя в целом успешным, рекомбинантные методы на основе белка требуют, чтобы эктодомен мембранного рецептора быть произведены в качестве растворимого белка. Таким образом, это обычно применимо только к белкам, которые содержат смежные внеклеточной области (например, однопроходный тип I, тип II, или GPI-якорь) и, как правило, не подходит для рецепторных комплексов и мембранных белков, которые охватывают мембрану несколько раз.
Методы клонирования выражения, в которых библиотека дополнительных ДНК (cDNAs) трансфицируется в клетки и проверяется на увеличение-связывания фенотипа также были использованы для выявления внеклеточного белка-белковых взаимодействий8. Наличие больших коллекций клонированных и секвенированных плазмидов экспрессии кДНК в последние годы облегчило методы, в которых клеточные линии, перевыражающие cDNAs, кодирующие рецепторы поверхности клеток, проверяются на связывание рекомбинантных белков для определения взаимодействий9,10. Подходы, основанные на переэкспрессии кДНК, в отличие от рекомбинантных белковых методов, дают возможность выявлять взаимодействия в контексте плазменной мембраны. Тем не менее, успех использования конструкций экспрессии кДНК зависит от способности клеток переэкспрессировать белок в правильно сложенной форме, но это часто требует клеточных вспомогательных факторов, таких как транспортеры, сопровождающие, и правильное олигомерной сборки. Таким образом, трансфекции одной кДН может быть недостаточно для достижения экспрессии поверхности клетки.
Методы скрининга с использованием конструкций кДНК или рекомбинантных белковых зондов являются ресурсоемкими и требуют больших коллекций библиотек кДНК или рекомбинантных белков. Специально разработанные методы на основе масс-спектрометрии были использованы в последнее время для выявления внеклеточных взаимодействий, которые не требуют сборки больших библиотек. Тем не менее, эти методы требуют химических манипуляций поверхности клетки, которые могут изменить биохимический характер молекул, присутствующих на поверхности клеток и в настоящее время применимы только для взаимодействия опосредовано гликозилатированных белков11,12. Большинство имеющихся в настоящее время методов также в значительной степени сосредоточены на взаимодействии между белками, в то время как в значительной степени игнорируя вклад мембраны микросреды, в том числе молекул, таких как гликаны, липиды и холестерин.
Недавнее развитие высокоэффективного биалелеческого таргетинга с использованием CRISPR-подходов позволило геном масштаба библиотек клеток, не хватает определенных генов в одном пуле, которые могут быть проверены в систематическом и беспристрастным образом для выявления клеточных компонентов, участвующих в различных контекстах, в том числе вскрытие клеточных процессов сигнализации, выявление возмущений, которые придают устойчивость к наркотикам, токсинов и патогенов, и определения конкретных антител15,14,15,. Здесь мы описываем геном масштаба CRISPR основе нокаут-клеток скрининга анализ, который обеспечивает альтернативу текущим биохимических подходов для выявления внеклеточных рецепторов-лиганд взаимодействий. Такой подход к выявлению взаимодействий, опосредованных мембранными рецепторами генетическими экранами, особенно подходит для исследователей, которые имеют целенаправленный интерес к отдельным лигандам, поскольку он позволяет избежать необходимости компиляции больших библиотек cDNA или рекомбинантных белков.
Этот анализ состоит из трех основных шагов: 1) Высоко заядлые рекомбинантные протеиновые связывающие зонды, состоящие из внеклеточных областей рецептора интереса, производятся и используются в связывающих сяпись на основе флуоресценции цитометрии; 2) Связывание анализы используются для определения клеточной линии, которая выражает взаимодействие партнера рекомбинантного зонда белка; 3) Cas9-выражение версия клеточной линии, которая взаимодействует с белком интерес производится и геном масштаба CRISPR / Cas9 основе нокаут экран выполняется(Рисунок 1). В этом генетическом экране, связывание рекомбинантного белка в клеточных линиях используется в качестве измеримого фенотипа, в котором клетки в библиотеке нокаутом, которые потеряли способность связывать зонд сортируются с помощью флуоресценции на основе активированной сортировки клеток (FACS) и генов, которые вызвали потерю связывающего фенотипа, идентифицированного путем секвенирования. В принципе, гены, кодируя рецептор, ответственный за связывание алчный зонд и те, необходимые для его поверхности клетки дисплей определены.
Первый шаг этого протокола включает в себя производство алчный рекомбинантных белковых зондов, представляющих эктодомен мембранных рецепторов. Эти рецепторы, как известно, часто сохраняют свои внеклеточные связывающие функции, когда их эктодоменов выражаются в качестве рекомбинантного растворимого белка1. Для белка интерес, растворимые рекомбинантные белки могут быть произведены в любой подходящей системы экспрессии эукариотического белка в любом формате при условии, что он может быть олигомеризирован для увеличения связывания жадности, и он содержит теги, которые могут быть использованы в флуоресценции на основе потока цитометрии основе связывающей анализы (например, FLAG-тег, биоттин-тег). Подробные протоколы для производства растворимых эктодоменов мембранных рецепторов с использованием системы экспрессии белка HEK293, а также различные методы мультимеризации и построения экспрессии белка для производства как пентамериковых белков, так и мономерных белков, ранее описаны1,,17. Протокол здесь будет описывать шаги для генерации флуоресцентных алчных зондов из мономерных биоинтинилобных белков, спряжение их стрептавидин спряженный к флюорохром (например, фикоэритрин, или PE), которые могут быть использованы непосредственно в клеточной основе связывающих анализов и имеет преимущество не требует вторичного антитела для обнаружения. Общие протоколы для выполнения генома масштаба экранов уже описаны20,21, Таким образом, протокол в основном сосредоточены на специфике выполнения потока цитометрии основе рекомбинантных белков связывающих экранов с использованием CRISPR / Cas9 нокаут скрининга системы с использованием человека V1 ("Yusa") библиотека18.
протокол
1. Производство и очистка биотинилатированных белков, отмеченных его
- Используйте систему экспрессии белка на основе млекопитающих или клеток на основе клеток для производства растворимых рекомбинантных биоинфинилатированных белков (см. плазмидные конструкции в таблице 1).
ПРИМЕЧАНИЕ: Подробный протокол для производства мономерного биотина и его помеченных белков с использованием системы экспрессии клеток HEK293 описан Kerr et al.17. Белковые эктодомемы, выраженные с помощью системы экспрессии HEK293, выделяются в культурную среду. - Соберите растворимые белки путем гранулирования клеток центрифугированием при 3000 х г в течение 20 мин.
- Фильтр супернатант через фильтр 0,22 мкм и добавить Ni2 "-NTAагарозбий в фильтрованный белок супернатант в 1:1,000 соотношение (т.е., 50 л 50% агароза шлам в 50 мл супернатанта). Инкубировать на ночь или не менее 4-5 ч при 4 градусах По Цельсию на вращающейся платформе.
- Вымойте полипропиленовый столбец, добавив 5 мл буфера его очистки. Обратитесь к таблице 2 для всех буферных композиций.
- Налейте всю смесь бис-белка супернатант в колонке. Бусы будут накапливаться на базе.
- Вымойте бисер 2x с 15 мл мытьбуфера. Чтобы избежать разбавления белка, тщательно нарисуйте остаточный буфер стирки из колонны шприцем 5 мл и отбросьте.
- Аккуратно добавьте 300-500 л буфера его очищения elution непосредственно в бисер и инкубируйте не менее 1 ч. Соберите нелепое белок, снова тщательно вытягивая жидкость с помощью шприца 1 мл. Обмен буфера elution на желаемый буфер (например, обычно PBS или HBS) с помощью опреснительных столбцов. Храните все белки при 4 градусах Цельсия до дальнейшего использования.
2. Количественная и олигомеризация мономерного биоинтинилатного белка
ПРИМЕЧАНИЕ: Чтобы увеличить связывание алчность, олигомеризировать биоинтинилатированных мономерных белков на тетрамерных стрептавидин-PE, прежде чем использовать их в связывающей анализы. Достичь оптимального соотношения спряжения мономерных белков и тетрамерных стрептавидин-ПЭ путем тестирования серии разбавления биотинилатированных мономеров против фиксированной концентрации стрептавидина и эмпирически установления минимального разбавления, при котором не может быть обнаружено избыток биотинилатированных мономеров.
- Сделайте по крайней мере восемь серийных разбавлений биотинилапованных образцов белка, используя соответствующий буфер разбавления (либо PBS или HBS с 1% бычьего сывороточных альбумина »BSA) в 96 хорошо пластины. Убедитесь, что окончательный объем каждого разбавления составляет не менее 200 л.
- Сделайте дубликат пластины образцов, удалив 100 злиц из каждой скважины и переведя в новую пластину 96 скважин. Всегда включайте элемент управления. В этом случае элементы управления являются белками только для тегов (т.е. биотинилатированными его тегами Домен Cd4 3'4 белка). Это будет использоваться в качестве управляющего зонда во всех связывающих анализах.
- Разбавить стрептавидин-PE до 0,1 мкг/мл в буфере разбавления.
- Только на одной из пластин добавьте 100 л разбавленного стрептавидина-ПЭ. Дубликатпластина будет служить элементом управления. Добавьте 100 л буфера разбавления в контрольную пластину, чтобы уравнять объемы.
- Инкубировать в течение 20 минут при комнатной температуре (RT). В то же время, блокировать скважины стрептавидина покрытием пластины с буфером разбавления в течение 15 минут.
- Перенесите общий объем образца с обеих пластин в отдельные скважины стрептавидиновых пластин и инкубировать за 1 ч на РТ.
- Вымойте пластину 3x с 200 мл буфера для мытья (т.е. pbS или HBS с 0,1% Tween-20, 2% BSA). Добавьте 100 л 2 мкг/мл мыши против крысы Cd4d3'4 IgG (OX68) и инкубировать в течение 1 ч на RT.
- Вымойте тарелку 3x с буфером мытья. Добавьте 100 л антимышечной щелочной фосфатазы на 0,2 мкг/мл в течение 1 ч на RT.
- Вымойте пластину 3x с буфером мытья и 1x в буфере разбавления.
- Подготовка р-нитрофенил фосфат на 1 мг /мл в буфере дитаноламин. Добавьте 100 л в каждую скважину и инкубировать в течение 15 минут.
- Возьмите абсорбции показания на 405 нм. Используйте минимальное разбавление, при котором нет сигнала на пластине в качестве соответствующего фактора разбавления для создания тетрамеры(рисунок 2).
- Сделать 10x тетрамер окрашивания решение для всех образцов и контроля путем инкубации 4 мкг /мЛ стрептавидин-PE и соответствующие биоинтинилафтами белка разбавления в течение 30 минут на RT. Store конъюгированных белков в светозащищенной трубке при 4 градусах по Цельсию до дальнейшего использования.
3. Поток цитометрии на основе клеточной связывания анализы
- Для адептов клеток, удалить культуры средств массовой информации и мыть 1x с PBS без divalent катионов. Затем добавьте решения для отслоения клеток (например, EDTA). Разрешить клетки, чтобы отделить в течение 5-10 мин. Аккуратно нажмите колбу, чтобы освободить клетки.
ПРИМЕЧАНИЕ: Избегайте использования продуктов на основе трипсина, поскольку они могут расщеплять белки поверхности клеток. - Соберите отдельные клетки в трубку. Для клеток, растущих в суспензии (например, heK293 клетки), непосредственно собирать клетки из культуры колбы в трубку.
- Клетки гранул при 200 х г в течение 5 мин. Удалите супернатант и отдохните гранулы в буфере для мытья (т.е. PBS/1% BSA).
- Подсчитайте клетки с помощью гемоцитометра и отрегулируйте концентрацию до 2,5 х 10-1х 106 клеток/мл. Aliquot 100 л подготовленной клеточной смеси на 96 хорошо U- или V- bottomed пластины. Спин пластины в течение 5 мин на 400 х г. Удалите супернатант с помощью многоканальной пипетки.
- Добавьте 100 л/с нормализованных флуоресцентно помеченных высокозаядлых белковых зондов и элементов управления в ранее подготовленные пластины с клетками и инкубировать в течение 1 ч при 4 градусах Цельсия. После связывания в течение 1 ч, спина пластины на 400 х г в течение 5 мин.
- Удалите супернатант и добавьте 200 л буфера для мытья (т.е. PBS/1% BSA). Хорошо перемешать, пипетка вверх и вниз.
- Пелле клетки центрифугации при 400 х г в течение 5 мин. Повторите шаг стирки 1x. После двух стирнет, полностью удалить супернатант и resuspend клеточной гранулы в 100 Зл Л PBS.
- Анализ клетки по течению цитометрии. Используйте желто-зеленый лазер (т.е. 561 нм) для обнаружения флуоресценции ПЭ.
- Сначала проанализируйте клетки, которые были окрашены зондом управления. Основываясь на распределении фторесценции ПЭ, нарисуйте ворота для связывающей популяции таким образом, что в этих воротах попадает не более 1% контрольной ячейки.
- Проанализируйте образец и определите фракцию ячеек, попадающих в связывающие ворота.
ПРИМЕЧАНИЕ: Сотовые линии, которые отображают более высокую связующую популяцию, необходимы для генетических экранов, так как они имеют более высокое соотношение сигнала к шуму. В идеале более 80% клеток должны подпадать под эти ворота.
4. Определение обязательных взносов от тепловых лабильных эпитопов и гепарановых сульфатных боковых цепей
ПРИМЕЧАНИЕ: Активность многих белков является тепловой лабилой, поэтому потеря связывающей активности после тепловой обработки обнадеживает. Рекомендуется определить вклад отрицательно заряженных гликозиногликанов, в основном гепарана сульфата (ГС), в посредничество привязки рекомбинантных белков. Это потому, что связывание СС в клеточной связывания, описанной здесь, может быть добавкой, а не созависимым от других рецепторов19. Это означает, что наблюдаемые связывания могут быть полностью опосредованы HS боковые цепи протеогликанов поверхности клеток, а не конкретный рецептор. Связывание с ГС на поверхности клетки не обязательно неспецифическое, а скорее свойство белка, который полезно знать перед выполнением полного генетического экрана.
- Подготовка теплообработанных белков для использования в связывающих анализов.
- Нагрейте нормализованный, но неконъюгированный мономерный белок при температуре 80 градусов по Цельсию в течение 10 мин.
- Спрягать термообработанный белок с стрептавидином-ПЭ при таком же соотношении конъюгации, как и его необработанный аналог, определяемый ELISA (обратитесь к разделу 2).
- Приготовьте образцы белка, заблокированные гепарином.
- Приготовьте восемь 1:3 разбавления растворимого гепарина в PBS со стартовой концентрацией 2 мг/мл и конечным объемом 100 л.
- Инкубировать 100 л подготовленных связывающих зондов в разбавлениях гепарина в течение не менее 30 мин.
- Используйте термообработанный белок и полный 200 л гепарина/белковой смеси в связывающих анализах, описанных в разделе 3. Результаты представительства показаны на рисунке 3A,B.
5. Выбор клеточных линий, четко выражающих Cas9
ПРИМЕЧАНИЕ: Прежде чем клеточная линия, которая связывает зонд интерес может быть использована в скрининге CRISPR, он должен быть сначала разработан, чтобы выразить Nuclease Cas9 и высокоактивный клон выбран19.
- Используйте следующий общий протокол производства лентивирусного цитивируса для производства лентивирусной конструкции с использованием лентивирусной конструкции для выражения Cas9 (отсылай к таблице 1).
- Культура HEK293-FT клетки в DMEM/10% FBS средств массовой информации на 37 КК и 5% CO2. Семена HEK293-FT клетки за день до трансфекции, так что они являются 80% конфи в день трансфекции.
ПРИМЕЧАНИЕ: HEK293FT клетки слабо приверженцы; поэтому, когда они используются для производства лентивирусов, рассмотреть покрытие их на культуре фляги покрыты 0,1% (w/v) желатин для увеличения соблюдения. - Выполняйте трансфекции по утрам. Добавьте вектор переноса, упаковочную смесь и трансфекционный реагент в совместимые с трансфекцией средства прерывистого трансфекции (например, Opti-MEM). Смешайте путем инвертирования трубки 10-15x. Инкубировать в течение 5 минут на RT. Обратитесь к таблице 3 для точных объемов.
- Добавьте трансфекционный реагент, как это было предложено производителем. Смешайте путем быстрого вихря. Инкубировать в течение 30 минут на RT.
- Очень тщательно аспирируйте отработанное средство. Добавьте трансфекционный совместимый носители в пластину.
- Добавить трансфекционный реагент / ДНК комплексов dropwise на стороне пластины и медленно распространяется через пластину, закрученного очень осторожно.
- Инкубировать при 37 градусах по Цельсию на 3-5 ч и заменить среду со средним D10. Инкубировать на ночь.
- На следующий день утром замените носитель свежим средством D10. Инкубировать на ночь.
- На следующий день поздно вечером, собирать вирусный супернатант. Фильтр с фильтром 0,45 мкм с низким связыванием белка. Дополнительно добавьте свежий средний D10, инкубировать на ночь и вспомнить супернатант на следующий день.
- Вирус супернатанты стабильны при 4 градусах по Цельсию в течение нескольких дней. Хранить при -80 градусах по Цельсию для длительного хранения.
ПРИМЕЧАНИЕ: Для создания высококонцентрированного лецивирусного препарата, который может быть желательным для трансдукции трудно преобразующих клеток, супернатанты также могут быть сконцентрированы при центругировании при 6000 х г на ночь при 4 градусах Цельсия. Отметьте полупрозрачные вирусные гранулы с помощью пера, устойчивого к этанолу, и отбросьте супернатант. Приостановите гранулы в 1/100th первоначального объема для 100x увеличение концентрации.
- Культура HEK293-FT клетки в DMEM/10% FBS средств массовой информации на 37 КК и 5% CO2. Семена HEK293-FT клетки за день до трансфекции, так что они являются 80% конфи в день трансфекции.
- Преобразуем клетки с помощью лентивирусов.
- Плита 1 х 106 клеток на хорошо в 6 хорошо пластины с 3 мл соответствующих средств культуры. Некоторые клетки легче трансцируются, чем другие. Для легкой для преобразывания клеток (например, heK-клеток) непосредственно добавляйте лентивирус в клетки. Для трудно преобразующих клеток, это может быть необходимо следовать протоколу спинокуляции, как описано ниже.
- Aliquot 2 мл 2-5 х 106 ячеек/мл в 15 мл конической трубки.
- Добавить лентивирус вместе с 8 мкг/мЛ гексадитрина бромистого и инкубировать на RT в течение 30 минут.
- Центрифуга в течение 100 мин при 800 х г при 32 градусах По Цельсию. Затем перенакинул ячейки в те же носители и добавить подвеску клетки в соответствующие культурные фляги с соответствующими носителями.
- Разрешить трансдукции, по крайней мере 24 ч. После удаления средств, содержащих вирус и добавить свежие среды.
- После еще 24 ч, изменить средства массовой информации на тот, который дополняется соответствующими антибиотиками. Конструкция Cas9 содержит кассету сопротивления бласцидина для выбора.
ПРИМЕЧАНИЕ: Количество бласцидина должно быть оптимизировано для каждой линии клеток, выполняя кривую дозы ответа убить. Концентрация бласцидина между 2,5-50 мкг/мл должна убить большинство нетрансутированных клеточных линий в течение 10 дней после трансдукции.
- Плита 1 х 106 клеток на хорошо в 6 хорошо пластины с 3 мл соответствующих средств культуры. Некоторые клетки легче трансцируются, чем другие. Для легкой для преобразывания клеток (например, heK-клеток) непосредственно добавляйте лентивирус в клетки. Для трудно преобразующих клеток, это может быть необходимо следовать протоколу спинокуляции, как описано ниже.
- Выполните отбор до тех пор, пока все клетки в контрольной пластине (т.е. нетрансиндуцированные клетки, которые были обработаны с той же концентрацией отбора антибиотиков) будут убиты.
6. Выбор клонов высокой активности Cas9
ПРИМЕЧАНИЕ: Поликлональный Cas9 может быть использован для успешного выполнения генетических экранов; однако, выбор клона с высокой активностью Cas9 улучшает результаты скрининга18.
- Используйте ограничение разбавления или одноклеточных сортировать отдельные бласцидин-устойчивые клетки в скважины из трех 96 хорошо пластин, содержащих культуры средств массовой информации дополняется бласцидин. Клоны начнут появляться между 2-4 недель. Выберите 10-20 клонов и расширьте в 6 колодцах.
- Анализ клонов для cas9 деятельности с помощью быстрой к оценке GFP-BFP (зеленый флуоресцентный белок-голубой флуоресцентный белок) система, которая использует экзогенный ген нокаут системы, в которой клетки трансцируются либо с построением выражения GFP с gRNA-таргетинга GFP или пустой gRNA в качестве контроля18.
- Заказать репортер плазмиды: GFP-BFP плазмид, Контроль-BFP плазмид (Таблица 1).
- Производить лентивирус как для плазмида GFP-BFP, так и для плазмида Control-BFP с использованием протокола производства лентивирусов, описанного в разделе 5.1.
- Преобразуйте каждый клон линии клеток Cas9 с помощью лентивирусного кодирования системы GFP-BFP и Control-BFP отдельно. Следуйте протоколу в разделе 5.2.
- После 3 дней трансдукции, изучить GFP-BFP флуоресценции каждого клона с помощью цитометрии потока. Используйте 488 нм лазер и 405 нм лазер для обнаружения GFP и BFP соответственно.
- Извилит активность Cas9 в каждом клоне, изучив соотношение BFP только к положительным ячейкам GFP-BFP-double. Высокая активность Cas9 клетки должны в идеале иметь sgt;95% GFP нокаут эффективности(рисунок 4).
7. Поколение генома всей crispR-Cas9 скрининг нокаут библиотеки
- Для скрининга генома с помощью библиотеки Человека V118, заказать геном атакжем всей библиотеки (обратитесь к таблице 1) и подготовить плазмид библиотеки из бактериального удара с помощью протокола, предусмотренного в соответствии с "Протоколы для библиотечной репликации" в руководстве производителя.
- Используйте целую библиотеку плазмида для создания лентивирусной библиотеки, кодирующей гРНК для целенаправленного разрушения генов человека, используя протокол производства лентивирусов, описанный в разделе 5.1.
ПРИМЕЧАНИЕ: Хорошая практика заключается в том, чтобы произвести одну партию лентивирусного препарата, который оптимизирован для трансдукции для улучшения экспериментальной консистенции. - Используйте протокол трансдукции в разделе 5.2 для выполнения небольших тестовых трансдуктоций для определения необходимого количества вируса для каждой клеточной линии для достижения 30% трансдукции. Используйте цитометрию потока для оценки флуоресценции BFP в качестве прокси для эффективности трансдукции.
- Чтобы преобразовать клетки HEK293, просто добавьте заранее определяемый лентивирусный препарат до 30-50 х 106 клеток, культивированных в нормальных носителях роста в течение 4 ч. Затем удалите средства массовой информации с лентивирусом и заменить свежими средствами роста.
- Для других клеточных линий используйте протокол спинокуляции в разделе 5.2.1, но в большем масштабе, таким образом, что в общей сложности трансцируется 30-50 х 106 ячеек. Для этого, aliquot 2 мл 5 х 106 ячеек/ мл в 15 мл конической трубки и продолжить, как указано.
- Для адептовых клеточных линий выберите трансиндуцированные клетки, добавив пуромицин 24 ч после трансдукции.
ПРИМЕЧАНИЕ: Оптимизация концентрации пуромицина, выполняя кривую дозы ответ убить. Обычно концентрации между 1-10 мкг/мл должны убивать нетрансиндуцированные клетки в течение 3-5 дней. Избегайте использования более высоких концентраций пуромицина, потому что это может увеличить шансы выбора клеток, которые были транс-индуцированы более чем одним руководством РНК (sgRNA). - Для подвесных клеток, урожай транс-индуцированных (т.е. BFP положительных) клеток 3 дней послетрансдукции с помощью сортировки клеток и генерировать библиотеки, которые содержат по крайней мере 10 х 106 клеток. После выбора с помощью BFP, расти клетки в средствах массовой информации дополняется соответствующим количеством пуромицицина.
ПРИМЕЧАНИЕ: Избегайте выделения только с пуромицином для линий подвесных клеток, потому что трудно удалить мертвые клетки и мусор из подвесной клеточной культуры, которые могут помешать сортировке клеток. - Библиотека культурных мутантов в течение 9-16 дней посттрансдукции с регулярным прохождением каждые 2-3 дня.
8. Генетический скрининг на связывание поверхности клетки
- Пеллетмут библиотеку клеток мутантов на 200 x g в течение 5 минут и повторно приостанавливает клетки в PBS.
- Разделите клетки на две конические трубки 15 мл, в каждой трубке - не менее 50 х 106 ячеек.
- Закрутите одну коническую трубку при 200 х г в течение 5 мин, удалите супернатант и заморозьте клеточную гранулу при -20 градусов по Цельсию. Это контроль наяпопуляции и будет обработан позже.
- Приостановите гранулы в другой трубе в 10 мл PBS/1% BSA. Отложите 100 л клеток в качестве отрицательного контроля на пластине 96 скважин.
- Добавьте соответствующий предварительно конюгенированный рекомбинантный белок к клеточной суспензии в конической трубке и отрицательным белкам контроля к пластине 96 скважин.
- Выполните клеточные окрашивания, по крайней мере 1 ч при 4 градусах по Цельсию на скамейке ротора с нежным вращением (6 об/мин).
- Пелле клетки на 200 х г в течение 5 мин, удалить супернатант. Выполните два шага мытья, а затем повторной работы клеток в 5 мл PBS.
- Процедить клетки, хотя 30 мкм ячейки ситечко для удаления клеточных кластеров. Проанализируйте с помощью сортировки потока.
- Используйте отрицательный контрольный образец для ворот для bfP/PE-ячеек.
- Сортируйте образец и соберите ячейки BFP/PE./PE. Сортировка ворот будет зависеть от связывания клеток к белку, но, как правило, 1-5% от PE отрицательных образцов собраны. Пример сортировочной двери приведен на дополнительной рисунке 1.
- Соберите 500 000-1 000 000 ячеек из выбранных ворот. Учитывая низкое количество клеток, рассмотреть вопрос о сборе образцов в 1,5 мл центробежной трубки, чтобы свести к минимуму потери.
- Пеллети отсортированные клетки путем центрифуга при 500 х г в течение 5 мин. Тщательно удалите супернатант и отбросьте. Можно хранить гранулы при -20 градусов по Цельсию в течение 6 месяцев.
9. Геномная экстракция ДНК и первый ПЦР для обогащения гРНК
- Извлекайте геномную ДНК из высокой сложности контроля населения.
- Если контрольная популяция была заморожена при -20 градусов по Цельсию, выняйте коническую трубку и добавьте PBS. Держите на льду, чтобы разморозить гранулы.
- Используйте коммерческий комплект (см. Таблицу Материалов),используя рекомендации производителя для извлечения геномной ДНК из 50 х 106 клеток. Отрегулируйте концентрацию ДНК до 1 мг/мл.
- Для каждого образца навяжете мастер-микс для ПЦР, соответствующую 72 мкг ДНК. Aliquot 50 л на скважину в 36 скважинах 96 скважин ы ПЦР пластины. Необходимые последовательности праймера перечислены в таблице 4. Используйте руководство в таблице 5 и 6.
- Разрешить 5 ЗЛ ПЦР из 6-12 репрезентативных образцов на 2% (w/v) агарозный гель. Следует наблюдать одну четкую полосу на уровне 250 баррелей в сутки. Если полосы слабые, повторите ПЦР в течение дополнительных 2-3 циклов.
- Используйте многоканальный пипетку для сбора 5 qL продуктов ПЦР из каждой скважины (всего 180 л) и объединить их в резервуар с 900 qL связывающего буфера из коммерческого комплекта (см. Таблица материалов).
- Очистите продукты ПЦР с помощью коммерческого комплекта очистки ПЦР. Выделите ДНК в 50 л элютионного буфера из коммерческого комплекта (см. Таблица материалов)и измерить концентрацию ДНК.
- Образцы, которые были отсортированы на потерю связывающего фенотипа, вряд ли состоят из большого числа независимых клонов. Поэтому не обязательно выполнять ПЦР с 72 мкг ДНК. Изолировать ДНК с помощью соответствующего коммерческого комплекта (см. Таблица материалов). Навлажь 3-4 ПЦР-реакции с помощью описанного ранее протокола (раздел 9.1.3) с 100 нг/Л ДНК. Если отсортированное число клеток меньше 100 000 используют клеточные лисаты вместо препаратов геномной ДНК.
- Aliquot приблизительно 10.000 клеток/наилучшим образом в плите ПЦР 96 наилучшим образом.
- Пелле клетки в тарелке и тщательно удалить большую часть супернатанта. Гранулы не будут видны.
- Добавьте 25 qL воды в каждую скважину и нагрейте образцы при температуре 95 градусов по Цельсию в течение 10 мин.
- Добавьте 5 qL 2 мг/мл свежеразбавленной протеиназы К к каждому колодцу в течение 1 ч и инкубировать при 56 градусах Цельсия. Затем нагреть образец в течение 10 мин при температуре 95 градусов по Цельсию, чтобы инактивировать протеиназа K.
- Используйте 10 зЛ клеточной смеси лизата на реакцию ПЦР. Лысаты должны использоваться в течение 24 ч.
10. Второй раунд ПЦР для индексного штрихкодирования и секвенирования
- Разбавить продукты от первого раунда ПЦР до 40 пг/ Л.
- Навладивайте один ПЦР на образец (используйте руководство, представленное в таблицах 7 и 8). Использование полимеразы высокой точности важно для минимизации ошибок, введенных полимеразой во время усиления сгРН.
- Разрешить 5 ЗЛ пЦР продуктов на 2% (w/v) агарозный гель. Следует наблюдать одну четкую полосу на уровне 330 б.п.
- Очистите продукты ПЦР с использованием парамагнитных бусинок, добавив 31,5 л (0,7x общего объема) повторного бисера к продуктам ПЦР, хорошо перемешивая и инкубируя в течение 5 мин на RT.
- Поместите трубку на магнитную стойку на 3 мин. Бусинки должны быть захвачены на стороне пластины и раствор должен быть ясным. Тщательно удалите и отбросьте супернатант.
- Добавьте в трубку 150 л свежеприготовленного этанола на 80%. Инкубировать в течение 30 с, а затем осторожно удалить и отбросить супернатант.
- Повторите шаг 13.6, на этот раз с 180 Зл. Затем просушите бусинки на 5 мин.
- Снимите трубку с магнита. Выяснение ДНК цель из бисера в 35 Л стерильного буфера EB. Инкубировать в течение 3 мин, затем положить трубку обратно в магнитную стойку в течение 3 мин.
- Перенесите около 30 qL супернатанта, содержащего неочищенные продукты ПЦР, в чистую трубку.
- Секвенирование образцов на платформе секвенирования следующего поколения. Для библиотеки HumanV1 gRNA используйте пользовательский грунтовку, перечисленный в таблице 4, чтобы последовательность 19 bp.
11. Анализ биоинформатики для выявления рецепторов и связанных с ним путей
- По карте последовательности от отсортированной и несортированной популяции до справочной библиотеки, используюейтой функцией подсчета MAGeCK. Функция даст сырой файл подсчета(Дополнительная таблица 1).
ПРИМЕЧАНИЕ: Подробные инструкции по установке MAGeCK и использованию различных функций в MAGeCK описаны в ранее опубликованном протоколе Wang et al.20. - Проверьте технический стандарт библиотеки управления, используемой на экране.
- Медиана нормализует количество сырья и использовать пакет ggplot2 в R21 или эквивалентное программное обеспечение для графика эмпирического кумулятивного плотности функционального участка графов в плазмиди и управления несортированными образцами(рисунок 5A).
- Запустите функцию теста MAGeCK, используя отсчеты плазмидной популяции в качестве "контроля", а количество от несортированных контрольных образцов в качестве "тестовой" выборки. Функция даст ген резюме файл(Дополнительная таблица 1).
- Откройте файл сводки гена и нарисуйте распределение бревенчатых изменений (столбецneg'lfc) для ранее классифицированных основных и несущественных генов22 (рисунок 5B).
- Выберите значительно истощенные гены(neg'fdr ylt; 0.05) и выполняйте анализ обогащения пути с помощью обогащательногопакета 23 или любых эквивалентных пакетов обогащения пути в R(Рисунок 5C).
- Выполнить функцию теста MAGeCK с настройками по умолчанию. Используйте необработанные подсчеты из несортированного контрольного образца как«контроль»и отсчитываемые образцы как«обработка»при выполнении анализа.
- Откройте сводное файл гена, генерируемый MAGeCK, и ранжировать столбец pos'rank в порядке возрастания. Используйте FDR (колонка pos'fdr) злт; 0,05 в качестве отсечения для идентификации хитов. Рецептор обычно занимает высоко, часто в первой позиции.
- Участок Robust-Рейтинг-Алгоритм (RRA) оценки для положительного отбора (pos'score) в R или эквивалентное программное обеспечение(рисунок 5D).
- Выберите ген хитов и выполнить анализ обогащения пути с помощью обогащателя пакет или любые эквивалентные пакеты обогащения пути в R для выявления обогащенных путей.
Результаты
Секвенирование данных с двух репрезентативных экранов нокаута генома для идентификации связывающего партнера человека TNFSF9 и P. falciparum RH5, выполненных в NCI-SNU-1 и HEK293, соответственно, предоставляются(Дополнительная таблица 1). На связывающее поведение RH5 повлияли как гепарановый сульфат, так и его известный рецептор BSG24 (Рисунок 3C),в то время как TNFRSF9 специально связан со своим известным рецептором TNFSF9 и не терял привязки при преинкубации растворимым гепарином. Белок 3 на рисунке 3B представляет TNFRSF9.
Для обеих клеточных линий, распределение GRNAs в библиотеке контрольных мутантов после 3 дней (9, 14, и 16 дней посттрансдукции) также предусмотрены(Дополнительная таблица 1). Распределение gRNA показало, что сложность библиотеки сохранялась на протяжении всего эксперимента(рисунок 5А). Генетический экран для идентификации лиганда для TNFSF9 был выполнен на день 14 posttransduction, тогда как то для RH5 было выполнено день 9 posttransduction. Техническое качество экранов было оценено путем изучения распределения наблюдаемых складных изменений гРНК, нацеленных на справочный набор несущественных генов по сравнению с распределением для эталонного набора основных генов22 (рисунок 5B). Кроме того, обогащение на уровне путей также показало, что ожидаемые основные пути были выявлены и значительно обогащены в популяции "выпадения" при сравнении контрольной выборки с оригинальной плазмидной библиотекой. Пример с образцом дня 14 NCI-SNU-1 изображен на рисунке 5C.
Распределение gRNA в контрольной и отсортированной популяции с использованием -тестовой функции MAGeCK (см. Дополнительную таблицу 1 для вывода сводковых генов от MAGeCK) использовалось для идентификации рецептора из фенотипических экранов. Измененная оценка RRA, зарегистрированная MAGeCK в анализе уровня генов, построена на основе генов, ранжированных по значениям p. Оценка RRA в MAGeCK обеспечивает меру, в которой gRNAs ранжируются стабильно выше, чем ожидалось. На экране для TNFRSF9, топ хит был TNFSF9, который является известным связующим партнером TNFRSF9 (Рисунок 5D). Кроме того, был также выявлен ряд генов, связанных с пути TP53. В случае RH5, в дополнение к известным рецептором (BSG) и ген, необходимый для производства сульфатных GAGs(SLC35B2), дополнительный ген (SLC16A1) также был определен(Рисунок 5E). SLC16A1 является сопровождающим, необходимым для торговли BSG на поверхность ячеек25. Вместе эти результаты демонстрируют способность экрана идентифицировать непосредственно взаимодействующие рецепторы и клеточные компоненты, необходимые для того, чтобы этот рецептор выражался на поверхности клеток в функциональной форме.
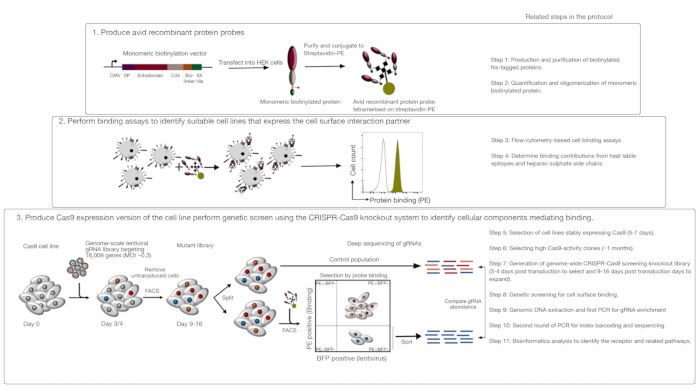
Рисунок 1: Обзор генетического подхода к скринингу для выявления рецепторов поверхности клеток. Этот анализ состоит из трех основных шагов: Во-первых, рекомбинантные белки, представляющие эктодоин рецепторов поверхности клеток, выражены в клеточной линии, которая может добавить структурно критические постпереводные изменения, такие как клетки HEK293. Мономерный белок эктодоменов олигомеризированы путем спряжения стрептавидин-PE, чтобы увеличить их связывания алчность. Во-вторых, эти алчный зонды используются в клеточной связывания анализы, где яркие окрашивания на клеточных линий, указанные видные сдвиг в PE флуоресценции (зеленый) по сравнению с отрицательным протеином контроля (в черном) демонстрирует наличие клеточной поверхности связывающего партнера. В-третьих, выбираются рецепторно-положительные клеточные линии Cas9 и проводится скрининг генома с использованием гРНК, ориентированных на подавляющее большинство генов, кодирующих белок. При генерации библиотек мутантов, обычно используется 30% эффективности трансдукции, которая основана на вероятности распределения Пуассона, которая гарантирует, что каждая клетка получает один gRNA таким образом, что результирующий фенотип приписывается конкретному нокауту. Маркер BFP, выраженный трансиндуцированными клетками, используется для выбора клеток, содержащих ГРНК с помощью FACS. Фенотипические экраны выполняются между 9-16 дней посттрансдукции. В день экрана общая популяция клеток мутантов делится на две части. Одна половина хранится в качестве контрольной популяции, а другая половина отбирается для рекомбинантного связывания белка. Клетки из библиотеки мутантов, которые больше не в состоянии связать рекомбинантный белок, сортируются с помощью FACS, и обогащение gRNA в отсортированной и контрольной популяции используется для определения генов, необходимых для связывания поверхности клетки помеченного алчного зонда. Указаны шаги в протоколе, которые требуют значительного времени. Эта цифра была изменена из Шарма и др.19. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
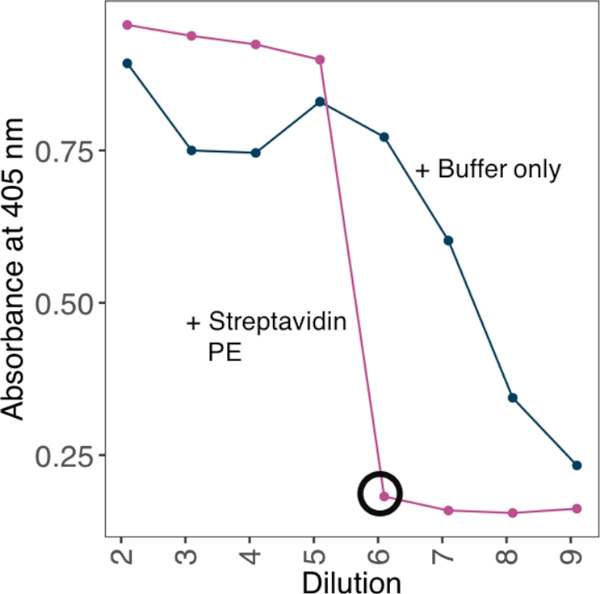
Рисунок 2: Установление соотношений биотинилапопрома белка к стрептавидину-PE с помощью метода на основе ELISA. Пример стратегии спряжения стрептавидина-ПЭ, используемой для генерации алчный зонд из биотинилапотного мономерного белка. Против фиксированной концентрации стрептавидина была инкубирована серия разбавления биотинители. Минимальное разбавление, при котором не может быть обнаружено избыток биотинители, было определено ELISA. ELISA была выполнена с или без preincubating диапазон разбавления белка с 10 нг стрептавидин-PE. При наличии стрептавидина-PE, минимальное разбавление, при котором не было выявлено сигнала (кружевной черный) и количество белка, необходимого для насыщения, было рассчитано на генерацию 10-x запасного раствора с 4 мкг/мл стрептавидина-PE. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
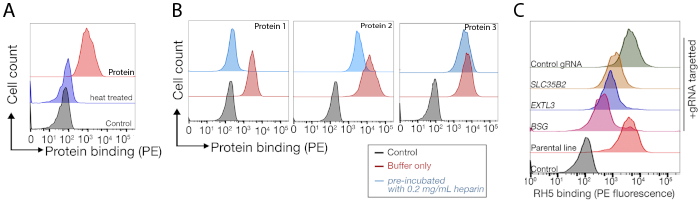
Рисунок 3: Представительная привязка белков к клеточным линиям. (A) Протеина связывания клеточных линий было четкое увеличение клеточной флуоресценции по сравнению с контрольным образцом. Тепловая обработка (80 градусов по Цельсию в течение 10 мин) рекомбинантного белка отменяла все переплеты обратно к отрицательному контролю, демонстрируя, что связывающее поведение зависит от правильно сложенного белка. (B) Различные классы поведения связывания белка к поверхностям клетки; зависимость от ГАГ. Слева направо, белки могут быть классифицированы на три типа: Белок типа 1 только адсорбы в HS. Эти белки теряют свои связывания после преинкубации с концентрацией гепарина более 0,2 мг/мл. Белок типа 2 связывается с ГС в дополнение к конкретному рецептору. Эти белки теряют частичную связывание в предблокирующих экспериментах. Белок типа 3 не связывает HS. Эти белки не теряют связывания по сравнению с родительскими линиями. (C) Пример белка (т.е., RH5), который связывается с ГС и специфическим рецептором в аддитивной манере. Ориентация либо на рецептор (т.е. BSG) или ферменты, необходимые для синтеза СГ (например, SLC35B2, EXTL3) лишь частично снижает связывание RH5 с клетками относительно контроля. Трансиндуцированные поликлональные линии могут быть использованы в таких экспериментах для установления связывающего поведения. Эта цифра была изменена из Шарма и др.19. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
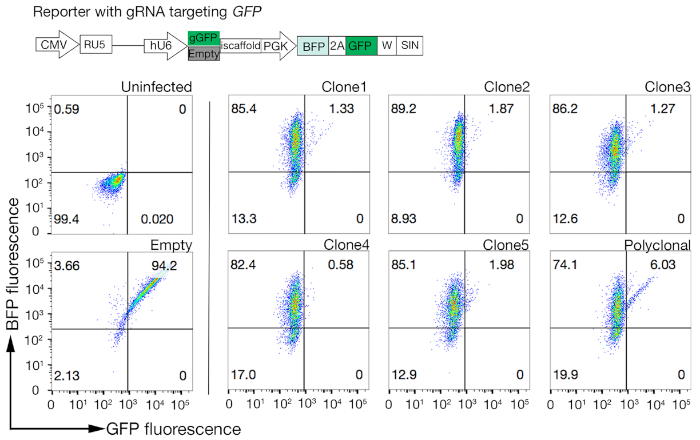
Рисунок 4: Выбор клональных клеточных линий с высокой активностью Cas9. Эффективность редактирования генома как поликлональных, так и клонированных линий клеточных линий NCI-SNU-1 оценивалась с помощью системы репортеров GFP-BFP, в которой клеточные линии переводились вирусами с помощью плазмидного плазмида, нацелившегося на гфальтари или без (т.е. «пустой»). Изображена схема. Цитометрия потока использовалась для проверки экспрессии BFP и GFP после трансдукции и сравнивалась с неинфицированным контролем. Выражение GFP использовалось в качестве прокси для деятельности Cas9, в то время как выражение BFP с пометкой трансиндуцированных ячеек. Профиль для неинфицированных и пустых инфицированных клеток выглядел одинаково для всех клонов. Профили представителей изображены на левой панели. Все пять клонов клеточной линии NCI-SNU-1 показали более высокую потерю GFP по сравнению с поликлональной линией (правая панель), при этом клон 4 показывает самую высокую эффективность с самой низкой огнеупорной популяцией. Эта цифра была изменена из Шарма и др.19. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
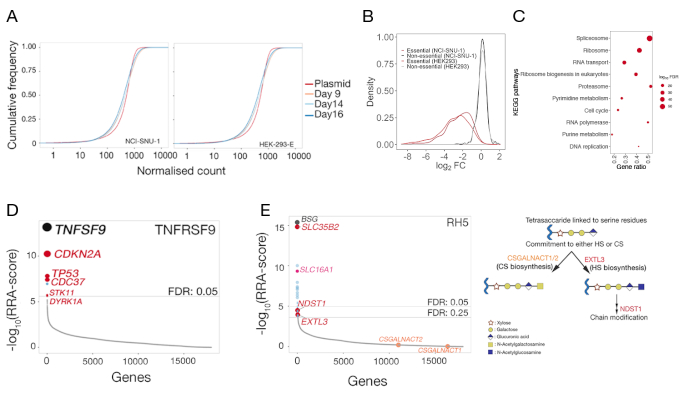
Рисунок 5: Представитель результаты генетических экранов для идентификации клеточной поверхности связывания партнеров. (A) Совокупные участки функции распределения сравнения изобилия gRNA в плазмидной библиотеке к библиотекам мутантов HEK-293-E и NCI-SNU-1 на 9, 14 и 16 дней посттрансдукции. Для любого данного числа функция кумулятивной плотности сообщает процент точек данных, которые были ниже этого порога. Небольшой сдвиг популяции мутантных клеток по сравнению с первоначальной популяцией плазмидных представляет собой истощение в подмножестве гРНК по сравнению с плазмидной библиотекой. (B) Распределение изменений в журнале, которые ранее были классифицированы как существенные (красные) или несущественные (черные) в HEK293 и NCI-SNU-1 клеточных линий. Распределение складных изменений для несущественных генов сосредоточено на 0, в то время как для основных генов сместился влево в сторону отрицательных изменений складок. (C) Значительно обогащенные пути в генах истощены в NCI-SNU-1 мутант контроля населения 14 дней послетрансдукции. Были выявлены известные клеточные пути. (D) Robust Rank Algorithm (RRA) - оценка для генов, которые были обогащены в отсортированных клетках, которые потеряли способность связывать зонд TNFRSF9. Гены были ранжированы в соответствии с RRA-счет. Известный партнер по взаимодействию TNFSF9 и гены, связанные с пути TP53 (помечены красным цветом) были определены на экране. (E) Rank-ordered RRA-оценки для генов, идентифицированных из анализа обогащения gRNA, необходимых для связывания RH5 к клеткам HEK293 (левая панель). SLC35B2 и SLC16A1 были выявлены в пределах порога ложного обнаружения (FDR) в 5%. Два дополнительных гена в пути биосинтеза HS (т.е., EXTL3 и NDST1)были определены в пределах ФДР 25%. Схематическая изображение общего пути биосинтеза GAG с соответствующими генами, отображаемыми на соответствующие шаги (панель 2). Гены, необходимые для принятия обязательств по биогене зуфору хондроитин (т.е. CSGALNACT1/2)не были идентифицированы на экране. Эта цифра была изменена из Шарма и др.19. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
| Плазмида имя | Плазмидные # | Использовать |
| Конструкция экспрессии протеина: CD200RCD4d3'4-био-linker-его | Адген: 36153 | Производство рекомбинантного белка с CD4d3'4, биотином и 6-его тегами. |
| pMD2.G | Адген: 12259 | Конверт VSV-G, выражающий плазмид; производство лентивирусной |
| psPAX2 | Аддген: 12260 | Лентивирусная упаковка плазмида, производство лентивирусного |
| Cas9-конструкция: pKLV2-EF1a-Cas9Bsd-W | Аддген: 68343 | Производство составно выражающей линии Cas9 |
| конструкция выражения gRNA: pKLV2-U6gRNA5 (BbsI)-PGKpuro2ABFP-W | Добавление: 67974 | Вектор экспрессии CRISPR gRNA с улучшенными эшафотами и маркерами puro/BFP |
| Улучшенная геномная библиотека Knockout CRISPR | Аддген: 67989 | Библиотека gRNA против 18 010 человеческих генов, предназначенных для использования в лентивирусе. |
| Конструкция GFP-BFP: pKLV2-U6gRNA5 (gGFP)-PGKBFP2AGFP-W | Аддген: 67980 | Cas9 деятельности репортера с BFP и GFP. |
| Пустая конструкция: pKLV2-U6gRNA5 (пустой)-PGKBFP2AGFP-W | Адген: 67979 | Cas9 деятельности репортера (контроль) с BFP и GFP. |
Таблица 1: Плазмиды, используемые в этом подходе.
| Имя буфера | Компоненты |
| HBS (10X) | 1.5 M NaCl и 200 мМ HEPES в воде Милич, приспособите к рН 7.4 |
| PBS (10X) | 80 г NaCl, 2 г KCl, 14,4 г Na2HPO4 и 2,4 г KH2PO4 в воде Мили, приспосабливайте к рН 7,4 |
| Фосфатный буфер натрия (80м) | 7.1 g Na2HPO4.2H2O, 5.55 g2PO4,отрегулировать к pH 7.4 |
| Его очистка связывающий буфер | 20 мМ фосфатный буфер натрия, 0,5 М NaCl и 20 мМ Имидазол, приспосабливаться к рН 7,4 |
| Буфер его очищения elution | 20 мМ фосфатный буфер натрия, 0.5M NaCl и 400 mM Имидазол, приспосабливайте к pH 7.4 |
| Дитаноламин буфер | 10% дизтанноламина и 0,5 мМ MgCl2 в воде Мили, приспосабливайся к рН 9.2: |
| D10 | DMEM, 1% пенициллин-стрептомицин (100 единиц/мл) и 10% теплоинактивированный FBS |
Таблица 2: Буферы, необходимые для этого исследования.
| Компоненты | 10-сантиметровое блюдо | 6-хорошая тарелка |
| 293FT-клетки | 70-80% слияние | 70-80% слияние |
| Трансфектии совместимые носители (Opti-MEM) (Шаг 5.1.2) | 3 мл | 500 л |
| Трансфектии совместимые носители (Opti-MEM) (Шаг 5.1.4) | 5 мл | 2 мл |
| Вектором передачи лентивир | 3 мкг | 0,5 мкг |
| psPax2 (см. таблицу 1) | 7,4 мкг | 1,2 мкг |
| pMD2.G (см. таблицу 1) | 1,6 мкг | 0,25 мкг |
| Plus реагент | 12 зл | 2 л |
| Липофектамин LTX | 36 зл | 6 зл |
| D10 (Шаг 7.1.7) | 5 мл | 1,5 мл |
| D10 (Шаг 7.1.8 и 7.1.10) | 8 мл | 2 мл |
Таблица 3: Объемы и объемы реагентов для лентивирусной упаковки.
Таблица 4: Праймер последовательности для усиления gRNA и NGS. Пожалуйста, нажмите здесь, чтобы просмотреть этот файл (Право нажмите, чтобы скачать).
| Реагента | Объем на одну реакцию | Мастер-микс (x38) |
| No 5 Горячий старт высокой верности 2x | 25 зл. | 950 л |
| Смесь праймер (L1/U1) (по 10 км) | 1 зл | 38 зл |
| Геномная ДНК (1 мг/мл) | 2 л | 72 Зл |
| H2O | 22 зл. | 1100 л |
| Общая | 50 зл | 1900 г. Л. |
Таблица 5: ПЦР для усиления ГРНК из образцов высокой сложности.
| Номер цикла | Денатурная | Отжига | Расширение |
| 1 | 98 кв.с. | ||
| 2-24 | 98 кв.с. | 61 КК, 15с | 72 КК, 20s |
| 25 | 72 КК, 2 мин |
Таблица 6: Условия ПЦР для первого ПЦР.
| Реагента | Объем на одну реакцию |
| KAPA HiFi HotStart ReadyMix | 25 зл. |
| Смесь primer (PE1.0/index primer) (по 5 мкм каждый) | 2 л |
| Первый продукт ПЦР (40 пг/л) | 5 зл |
| H2O | 18 зл |
| Общая | 50 зл |
Таблица 7: ПЦР для индексирования sgRNA с генетических экранов.
| Номер цикла | Денатурная | Отжига | Расширение |
| 1 | 98 кв.с. | ||
| 2-15 | 98 кв.с. | 66 кв.м, 15с | 72 КК, 20s |
| 16 | 72 КК, 5 мин |
Таблица 8: Условия ПЦР для второго ПЦР.
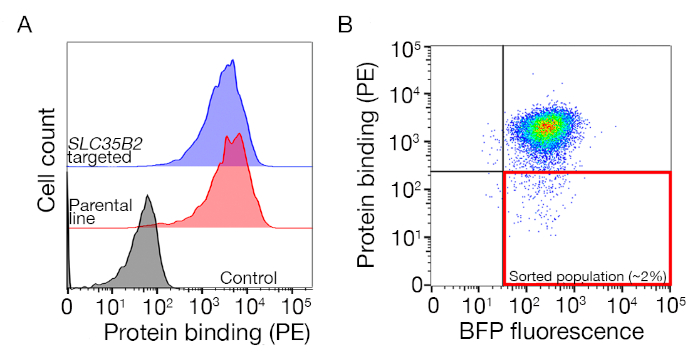
Дополнительная диаграмма S1: Руководство по рисованию ворот для сортировки неимеющей обязательной популяции. (A) Идеальный кандидат белка для скрининга должны иметь четкий сдвиг связывающей популяции по сравнению с контрольной популяции и связывание должно быть сохранено на клетках, не имеющих механизмов для биосинтеза HS. Эксперимент по блокированию гепарина может быть использован вместо тестирования на целевых клеточных линиях SLC35B2. (B) Клетки, не имеющие поверхности окрашивания из белка эктодона, но выражая BFP флуоресценции от лентивирусной трансдукции были собраны. Клетки отображаются с экрана для идентификации рецептора для GABBR222. Эта цифра была изменена из Шарма и др.19. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
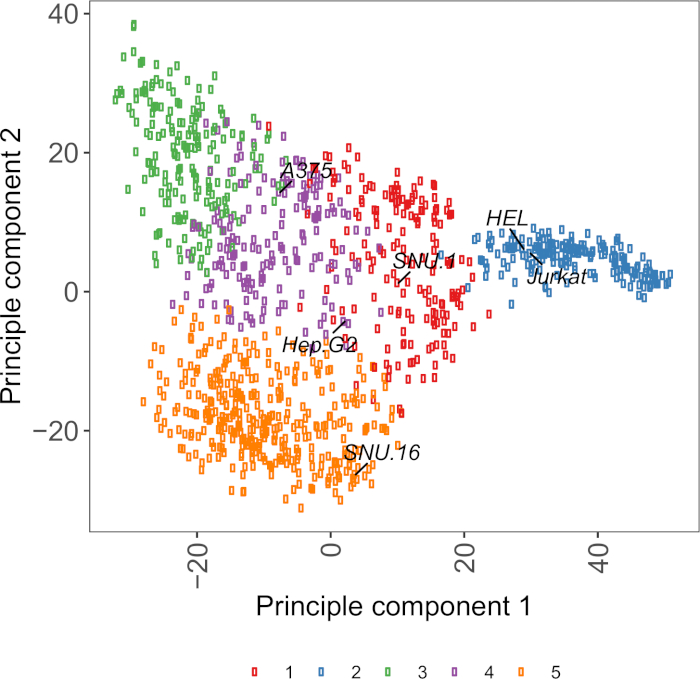
Дополнительная диаграмма S2: Клеточный поверхности гликопротеина транскриптомики на основе PCA участок с использованием РНК-сек данных из более чем 1000 раковых клеток линий. Сотовые линии из Cell Model Passport27 были сгруппированы с использованием K-средств кластеризации в соответствии с значениями FPKM 1500 клеточных гликопротеинов поверхности. Помечены представительные клеточные линии из каждого кластера. Кластер 5 полностью состоял из клеточных линий гематопоиетического происхождения (также см. Дополнительную таблицу 2). Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
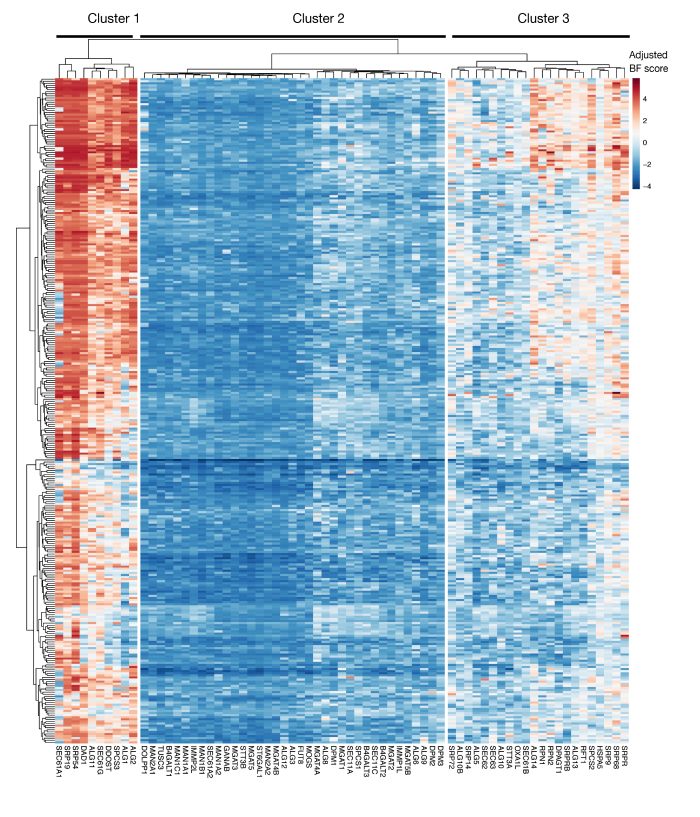
Дополнительная цифра S3: Essentiality оценки для KEGG-аннотации белка экспорта и N-связанных гликозиловых генов из результатов проекта. Скорректированные баллы Байеса-существенности для линий клеток в 330 фунтов (столбцы, не помечены) построены для генов экспорта белков и N-связанных гликозилата пути (X-оси). Результаты выше 0 представляют собой значительное истощение популяции мутантов по сравнению с оригинальной плазмидной библиотекой. Гены могут быть разделены на три различных кластера, которые представляют различные уровни существенности в клеточных линиях. Эта кластеризация может быть использована для решения дня сортировки. Если экран выполняется в конце времени (день 16), вполне возможно, что гены, которые, как известно, имеют важное значение для клеток (кластеры 1 и 3) не будут определены. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
Дополнительная таблица 1: Сырье кол-files для и MAGeCK программного обеспечения, генерируемого gene_summary файлов, связанных с репрезентативными генетическими экранами. Пожалуйста, нажмите здесь, чтобы просмотреть этот файл (Право нажмите, чтобы скачать).
Дополнительная таблица 2: Кластеризация клеточных линий в зависимости от выражения рецепторов поверхности клетки. Пожалуйста, нажмите здесь, чтобы просмотреть этот файл (Право нажмите, чтобы скачать).
Обсуждение
Описана стратегия скрининга на основе CRISPR для выявления генов, кодирующих клеточные компоненты, участвующие в распознавании клеток. Аналогичный подход с использованием активации CRISPR также обеспечивает генетическую альтернативу для выявления непосредственно взаимодействующих рецепторов рекомбинантных белков без необходимости генерировать большие белковые библиотеки26. Однако одним из основных преимуществ такого подхода является то, что он применим к взаимодействиям, опосредованным молекулами поверхности, нативно отображаемыми на клетке, и не зависит от переэкспрессии рецепторов, которые могут влиять на связывающую алчность рецептора. В отличие от других методов, таким образом, этот метод не делает никаких предположений относительно биохимической природы или клеточной биологии рецепторов и дает возможность изучать взаимодействия, опосредованные белками, которые обычно трудно изучать с помощью биохимических подходов, таких как очень большие белки, или те, которые пересекают мембрану несколько раз или образуют комплексы с другими белками, и молекулы, кроме белков, таких как гликаны, гликолипы, гликолипы. Учитывая геноммасштаба характер метода, этот подход также имеет преимущество не только выявление рецептора, но и дополнительные клеточные компоненты, которые необходимы для связывания события, тем самым обеспечивая понимание клеточной биологии рецептора.
Одним из основных ограничений этого метода при его использовании для определения рецептора белка сироты является первоначальное требование сначала определить клеточную линию, которая связывается с белком. Это не всегда легко и выявления клеточной линии, которая отображает связывание фенотипа, который также является вседозволенным для генетических экранов может быть ограничивающим время шагом для развертывания этого асссе. Некоторые клеточные линии, как правило, связываются с большим количеством белков, чем другие. Это особенно актуально для белков, которые связывают HS, потому что эти белки, как правило, связываются с любой клеточной линии, которая отображает HS боковых цепей, независимо от родного контекста связывания. Кроме того, мы заметили, что upregulation синдеканов (т.е., протеогликанов, которые содержат HS) в клеточных линиях приводит к увеличению связывания HS-связывающих белков26. Это может быть фактором, чтобы принять во внимание при выборе клеточной линии для скрининга. Однако также важно отметить, что аддитивная привязка ГС не мешает связыванию с конкретным рецептором. Это означает, что если привязка наблюдается, вполне возможно, что она опосредована исключительно СГ, потому что привязка опосредовано СС в этом ассе является добавкой, а не созависимым19. В таком сценарии описанный подход блокирования гепарина может определить такое поведение без необходимости выполнять полный генетический экран.
Полезным ресурсом для выбора клеточных линий является Cell Model Passport, который содержит геномику, транскриптомику и информацию о состоянии культуры для линий раковых клеток no 100027. В зависимости от биологического контекста, клетки могут быть выбраны на основе их профилей выражения. Для того чтобы помочь выбору линий клетки, мы сгруппировали линии клетки 1.000 в паспорте модели клетки согласно выражению q1.500 preannotated гликопротеинов поверхности клетки человека28 (Дополнительная Рисунок 2; информация кластера для каждой линии клетки вместе с условиями роста обеспечены в дополнительной таблице 2). При тестировании связывания белка с неизвестной функцией, полезно выбрать панель репрезентативных клеточных линий из каждого кластера, чтобы увеличить вероятность покрытия широкого спектра рецепторов. При выборе рекомендуется выбирать клеточные линии, которые легко культивируются и легко преобразовываются. Поскольку эти клеточные линии будут использоваться в скрининге в масштабе генома, предпочтительнее, чтобы они могли быть легко выращены в больших количествах и разрешительны для трансдукции лентивиральной инфекции, поскольку это наиболее распространенный метод доставки sgRNA для генетического скрининга на основе CRISPR в более поздних шагах.
Как правило, выделение фенотипа осуществляется в одном сорте. Однако это определяется яркостью популяции окрашенных клеток по сравнению с контролем. Итеративные раунды выделений могут быть приняты для сценариев, в которых соотношение сигнала к шуму желаемого фенотипа является низким, или когда целью экрана является выявление мутантов, которые имеют сильные фенотипы. При использовании итеративного подхода отбора для экранов на основе FACS, важно учитывать, что процесс сортировки может привести к гибели клеток, в основном из-за силы сортировки. Таким образом, не все собранные ячейки будут представлены в следующем раунде сортировки.
Сложность библиотеки является очень важным фактором при успешном выполнении генетических экранов, особенно для отрицательных экранов отбора, потому что степень истощения в них может быть определена только путем сравнения результатов с тем, что присутствовало в стартовой библиотеке. Для отрицательных экранов отбора, он является общим для поддержания библиотек 500-1000 х сложности. Положительные экраны отбора, однако, более надежны для размеров библиотеки, потому что на таких экранах ожидается, что для конкретного фенотипа будет выбрано лишь небольшое число мутантов. Таким образом, в описанном здесь положительном экране отбора размер библиотеки может быть уменьшен до 50-100-кратной сложности без ущерба для качества экрана. Кроме того, на этих экранах также можно использовать библиотеку управления для данной линии ячейки в данный день в качестве "общего контроля" для всех образцов, отсортированных в день для данной линии ячейки. Это позволит сократить количество контрольных библиотек, которые необходимо производить и секвенировать.
Другим важным соображением для использования этого подхода является ограничение потери функции экранов в выявлении генов, которые имеют важное значение для роста клеток in vitro. Сроки экранов имеет решающее значение в этом отношении, так как чем дольше клетки-мутанты хранятся в культуре, тем выше вероятность того, что клетки с мутациями в основных генах становятся нежизнеспособными и больше не представлены в библиотеке мутантов. Последние генетические экраны, выполненные в рамках инициативы Project Score в более чем 300 клеточных линиях показывают, что несколько генов в KEGG-аннотированный белок секреции и N-гликозилационный путь часто определены как необходимые для ряда клеточных линий (Дополнительная рисунок 3)29. Это может быть принято во внимание при проектировании экранов, если влияние генов, необходимых для распространения и жизнеспособности, должно быть исследовано в контексте процесса распознавания клеток. В этом случае проведение экранов в ранний момент (например, день 9 посттрансдукции) было бы в целом уместно. Однако, если этот подход используется для определения нескольких целей с сильными эффектами размера, а не общие клеточные пути, было бы целесообразно выполнять экраны на более поздний момент времени (например, день 15-16 посттрансдукции).
Результаты скрининга очень надежны; в восьми рекомбинантных экранов связывания белка выполняется в прошлом, рецептор поверхности клетки был верхний удар в каждом случае19. При использовании этого подхода для определения партнера по взаимодействию следует ожидать, что рецептор и факторы, способствующие его представлению на поверхности, будут идентифицированы с высокой статистической достоверностью. После того, как экран выполняется и хит проверяется с помощью одного gRNA нокаут, дальнейшие последующие действия могут быть выполнены с использованием существующих биохимических методов, таких как AVEXIS4 и прямой сатурируемый связывания очищенных белков с использованием поверхностного плазмонрезонанса. Описанный здесь подход применим ко всем белкам, для которых можно создать растворимый рекомбинантный связывающий зонд.
Таким образом, это геном масштаба CRISPR нокаут подход для выявления взаимодействий опосредовано клеточных белков поверхности. Этот метод, как правило, применим для выявления клеточных путей, необходимых для распознавания поверхности клеток в широком диапазоне различных биологических контекстов, в том числе между собственными клетками организма (например, нейронным и иммунологическим распознаванием), а также между клетками-хозяинами и патогенными белками. Этот метод обеспечивает генетическую альтернативу биохимическим подходам, предназначенным для идентификации рецепторов, и поскольку он не требует каких-либо предварительных предположений относительно биохимической природы или клеточной биологии рецепторов, он имеет большой потенциал, чтобы сделать совершенно неожиданные открытия.
Раскрытие информации
Авторам нечего раскрывать.
Благодарности
Эта работа была поддержана грантом Wellcome Trust No 206194, присужденным GJW. Мы благодарим Cytometry Core объекта: Би Линг Нг, Дженнифер Грэм, Сэм Томпсон, и Кристофер Холл за помощь с FACS.
Материалы
| Name | Company | Catalog Number | Comments |
| Anti-mouse alkaline phosphatase | Sigma | A4656 | |
| Blasticidin | Chem-Cruz | SC-204655 | |
| Blood & Cell Culture DNA Maxi Kit | Qiagen | 13362 | |
| BSA | Sigma | A9647-100G | |
| Diethanolamine | Sigma | 398179 | |
| DMEM | Gibco | 31966-021 | |
| Dneasy Blood and Tissue kit | Qiagen | 69504 | |
| DynaMag-96 Side Magnet | Invitrogen | 12331D | |
| HEK293T packaging cells | ATCC | CRL-3216 | |
| Heparin | Sigma | H4784-1G | |
| KAPA HiFi HotStart ReadyMix | Kapa | KK2602 | |
| Lipofectamine LTX with PLUS reagent | Invitrogen | 15338100 | |
| MoFlo XDP cell sorter | BD | ||
| Ni2+-NTA agarose beads | Jena Bioscience | AC-501-25 | |
| OPTI-MEM | Life Technologies | 31985-070 | |
| OX-68 antibody | AbD Serotec | MCA1022R | |
| p-nitrophenyl phosphate | Sigma | 1040-506 | |
| PD-10 desalting columns | GE healthcare | 17085101 | |
| Polybrene | Millipore | TR-1003-G | |
| Polypropylene tubes with 5 mL bed volume | Qiagen | 34964 | |
| Proteinase K, recombinant, PCR Grade | Roche | 3115879001 | |
| Puromycin | Gibco | A11138-03 | |
| Q5 Hot Start High-Fidelity 2× Master Mix | NEB | M0494L | |
| QIAquick PCR purification kit | Qiagen | 28104 | |
| SCFA filter | Nalgene | 190-2545 | |
| Sony Cell sorter | Sony | ||
| SPRI beads (Agencourt AMPure XP beads) | Beckman | A63881 | |
| Streptavidin-coated microtitre plates | Nalgene | 734-1284 | |
| Streptavidin-PE | Biolegend | 405204 |
Ссылки
- Wright, G. J. Signal initiation in biological systems: the properties and detection of transient extracellular protein interactions. Molecular bioSystems. 5 (12), 1405-1412 (2009).
- van der Merwe, P. A., Barclay, A. N. Transient intercellular adhesion: the importance of weak protein-protein interactions. Trends in Biochemical Sciences. 19 (9), 354-358 (1994).
- Wood, L., Wright, G. J. Approaches to identify extracellular receptor-ligand interactions. Current Opinion in Structural Biology. 56, 28-36 (2019).
- Bushell, K. M., Söllner, C., Schuster-Boeckler, B., Bateman, A., Wright, G. J. Large-scale screening for novel low-affinity extracellular protein interactions. Genome Research. 18 (4), 622-630 (2008).
- Visser, J. J., et al. An extracellular biochemical screen reveals that FLRTs and Unc5s mediate neuronal subtype recognition in the retina. eLife. 4, e08149 (2015).
- Özkan, E., et al. An extracellular interactome of immunoglobulin and LRR proteins reveals receptor-ligand networks. Cell. 154 (1), 228-239 (2013).
- Martinez-Martin, N., et al. An Unbiased Screen for Human Cytomegalovirus Identifies Neuropilin-2 as a Central Viral Receptor. Cell. 174 (5), 1158-1171 (2018).
- Bianchi, E., Doe, B., Goulding, D., Wright, G. J. Juno is the egg Izumo receptor and is essential for mammalian fertilization. Nature. 508 (7497), 483-487 (2014).
- Mullican, S. E., et al. GFRAL is the receptor for GDF15 and the ligand promotes weight loss in mice and nonhuman primates. Nature Medicine. 23 (10), 1150-1157 (2017).
- Turner, L., et al. Severe malaria is associated with parasite binding to endothelial protein C receptor. Nature. 498 (7455), 502-505 (2013).
- Frei, A. P., et al. Direct identification of ligand-receptor interactions on living cells and tissues. Nature Biotechnology. 30 (10), 997-1001 (2012).
- Sobotzki, N., et al. HATRIC-based identification of receptors for orphan ligands. Nature Communications. 9 (1), 1519 (2018).
- Sharma, S., Petsalaki, E. Application of CRISPR-Cas9 Based Genome-Wide Screening Approaches to Study Cellular Signalling Mechanisms. International Journal of Molecular Sciences. 19 (4), (2018).
- Gebre, M., Nomburg, J. L., Gewurz, B. E. CRISPR-Cas9 Genetic Analysis of Virus-Host Interactions. Viruses. 10 (2), (2018).
- Zotova, A., Zotov, I., Filatov, A., Mazurov, D. Determining antigen specificity of a monoclonal antibody using genome-scale CRISPR-Cas9 knockout library. Journal of Immunological Methods. 439, 8-14 (2016).
- Puschnik, A. S., Majzoub, K., Ooi, Y. S., Carette, J. E. A CRISPR toolbox to study virus-host interactions. Nature Reviews. Microbiology. 15 (6), 351-364 (2017).
- Kerr, J. S., Wright, G. J. Avidity-based extracellular interaction screening (AVEXIS) for the scalable detection of low-affinity extracellular receptor-ligand interactions. Journal of Visualized Experiments. (61), e3881 (2012).
- Tzelepis, K., et al. A CRISPR Dropout Screen Identifies Genetic Vulnerabilities and Therapeutic Targets in Acute Myeloid Leukemia. Cell Reports. 17 (4), 1193-1205 (2016).
- Sharma, S., Bartholdson, S. J., Couch, A. C. M., Yusa, K., Wright, G. J. Genome-scale identification of cellular pathways required for cell surface recognition. Genome Research. 28 (9), 1372-1382 (2018).
- Wang, B., et al. Integrative analysis of pooled CRISPR genetic screens using MAGeCKFlute. Nature Protocols. 14 (3), 756-780 (2019).
- Hart, T., et al. Evaluation and Design of Genome-Wide CRISPR/SpCas9 Knockout Screens. G3. 7 (8), 2719-2727 (2017).
- Kuleshov, M. V., et al. Enrichr: a comprehensive gene set enrichment analysis web server 2016 update. Nucleic Acids Research. 44 (W1), W90-W97 (2016).
- Crosnier, C., et al. Basigin is a receptor essential for erythrocyte invasion by Plasmodium falciparum. Nature. 480 (7378), 534-537 (2011).
- Kirk, P., et al. CD147 is tightly associated with lactate transporters MCT1 and MCT4 and facilitates their cell surface expression. The EMBO Journal. 19 (15), 3896-3904 (2000).
- Chong, Z. S., Ohnishi, S., Yusa, K., Wright, G. J. Pooled extracellular receptor-ligand interaction screening using CRISPR activation. Genome Biology. 19 (1), 205 (2018).
- van der Meer, D., et al. Cell Model Passports-a hub for clinical, genetic and functional datasets of preclinical cancer models. Nucleic Acids Research. 47 (D1), D923-D929 (2019).
- Bausch-Fluck, D., et al. A mass spectrometric-derived cell surface protein atlas. PloS One. 10 (3), e0121314 (2015).
- Behan, F. M., et al. Prioritization of cancer therapeutic targets using CRISPR-Cas9 screens. Nature. 568 (7753), 511-516 (2019).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены