Method Article
Identificazione del recettore della superficie cellulare utilizzando schermi genetici CRISPR/Cas9 su scala genomica
In questo articolo
Riepilogo
Questo manoscritto descrive un approccio di screening basato sulle cellule su scala genomica per identificare le interazioni recettore-ligando extracellulari.
Abstract
La comunicazione intercellulare mediata da interazioni dirette tra i recettori della superficie cellulare incorporati nella membrana è fondamentale per il normale sviluppo e funzionamento degli organismi multicellulari. Tuttavia, il rilevamento di queste interazioni rimane tecnicamente impegnativo. Questo manoscritto descrive un approccio sistematico di screening genetico CRISPR/Cas9 su scala genomica che rivela i percorsi cellulari necessari per eventi specifici di riconoscimento della superficie cellulare. Questo saggio utilizza proteine ricombinanti prodotte in un sistema di espressione proteica dei mammiferi come acconde di legame avido per identificare i partner di interazione in uno schermo genetico basato sulle cellule. Questo metodo può essere utilizzato per identificare i geni necessari per le interazioni della superficie cellulare rilevate dalle sonde di legame ricombinanti corrispondenti agli ectodomini dei recettori incorporati nella membrana. È importante sottolineare che, data la natura su scala del genoma di questo approccio, ha anche il vantaggio di identificare non solo il recettore diretto, ma anche i componenti cellulari necessari per la presentazione del recettore sulla superficie cellulare, fornendo così preziose informazioni sulla biologia del recettore.
Introduzione
Le interazioni extracellulari delle proteine del recettore della superficie cellulare dirigono importanti processi biologici come l'organizzazione dei tessuti, il riconoscimento ospite-patogeno e la regolazione immunitaria. Lo studio di queste interazioni è di interesse per la comunità biomedica più ampia, perché i recettori della membrana sono obiettivi utilizzabili di terapie sistematicamente consegnate come gli anticorpi monoclonali. Nonostante la loro importanza, lo studio di queste interazioni rimane tecnicamente impegnativo. Ciò è dovuto principalmente al fatto che i recettori incorporati nella membrana sono anfipatici, rendendoli difficili da isolare dalle membrane biologiche per la manipolazione biochimica, e le loro interazioni sono caratterizzate dalle affinità di interazione deboli (KDs nella gamma M-MM)1. Di conseguenza, molti metodi comunemente utilizzati non sono adatti a rilevare questa classe di interazioni proteiche1,2.
È stata sviluppata una serie di metodi per studiare specificamente le interazioni recettore-ligando extracellulare che prendono in considerazione le loro proprietà biochimiche uniche3 . Alcuni di questi approcci prevedono l'espressione dell'intero ectodominio di un recettore come proteina ricombinante solubile nei sistemi basati su mammiferi o cellule di insetti per garantire che queste proteine contengano modifiche post-traduzionali strutturalmente importanti, come glicani e legami disulfide. Per superare l'associazione a bassa affinità, gli ectodomains sono spesso oligomerizzati per aumentare la loro avidità vincolante. Gli ectoscopi delle proteine avide sono stati utilizzati con successo come sonde di legame per identificare i partner di interazione negli schermi di interazione proteina-proteina ricombinanti diretti4,5,6,7.7 Anche se ampiamente riusciti, i metodi a base di proteine ricombinanti richiedono che l'ectodominio di un recettore della membrana sia prodotto come proteina solubile. Pertanto, è generalmente applicabile solo alle proteine che contengono una regione extracellulare contigua (ad esempio, tipo i, tipo II o ancoraggio gpI) e non è generalmente adatto per i complessi recettori alpinisti e le proteine della membrana che si estendono più volte sulla membrana.
Tecniche di clonazione dell'espressione in cui una libreria di DNA complementari (cDANA) viene trasinfezione in cellule e testata per un fenotipo di guadagno-di-legante sono state utilizzate anche per identificare le interazioni extracellulari-proteine8. La disponibilità di grandi collezioni di plasmidi di espressione cDNA clonati e in sequenza negli ultimi anni ha facilitato metodi in cui le linee cellulari che sovraesprimono i cDNA che codificano i recettori della superficie cellulare vengono sottoposte a screening per l'associazione di proteine ricombinanti per identificare le interazioni9,10. Gli approcci basati sulla sovraespressione del cDNA, a differenza dei metodi ricombinanti basati sulle proteine, offrono la possibilità di identificare le interazioni nel contesto della membrana plasmatica. Tuttavia, il successo dell'utilizzo di costrutti di espressione cDNA dipende dalla capacità delle cellule di sovraesprimere la proteina nella forma correttamente piegata, ma questo spesso richiede fattori accessori cellulari come trasportatori, chaperones e corretto assemblaggio oligomerico. Tradurre un singolo cDNA potrebbe quindi non essere sufficiente per ottenere l'espressione della superficie cellulare.
Le tecniche di screening con costrutti cDNA o sonde proteiche ricombinanti sono ad alta intensità di risorse e richiedono grandi raccolte di cDNA o librerie proteiche ricombinanti. I metodi basati sulla spettrometria di massa specificamente progettati sono stati utilizzati di recente per identificare le interazioni extracellulari che non richiedono l'assemblaggio di grandi librerie. Tuttavia, queste tecniche richiedono la manipolazione chimica della superficie cellulare, che può alterare la natura biochimica delle molecole presenti sulla superficie delle cellule e sono attualmente applicabili solo alle interazioni mediate dalle proteine glicosillate11,12. La maggior parte dei metodi attualmente disponibili si concentra anche pesantemente sulle interazioni tra le proteine, ignorando in gran parte il contributo del microambiente della membrana, comprese molecole come glicani, lipidi e colesterolo.
Il recente sviluppo di un targeting bialleleico altamente efficiente utilizzando approcci basati su CRISPR ha permesso a librerie su scala genomica di cellule prive di geni definiti in un unico pool che possono essere esaminati in modo sistematico e imparziale per identificare i componenti cellulari coinvolti in contesti diversi, tra cui sezionare i processi di segnalazione cellulare, l'identificazione di perturbazioni che conferiscono resistenza a farmaci, tossine e patogeni, e determinare la specificità degli anticorpi13,14,15,16. Qui, descriviamo un test dello screening delle cellule a eliminazione di knockout basato su CRISPR su scala genomica che fornisce un'alternativa agli attuali approcci biochimici per identificare le interazioni recettore-ligando extracellulari. Questo approccio di identificazione delle interazioni mediate dai recettori della membrana da parte dei recettori genetici è particolarmente adatto per i ricercatori che hanno un interesse mirato sui singoli ligandi perché evita la necessità di compilare grandi librerie di cDNA o proteine ricombinanti.
Questo saggio è costituito da tre passaggi principali: 1) Le sonde di legame proteico ricombinante altamente accanite costituite dalle regioni extracellulari di un recettore di interesse sono prodotte e utilizzate nei saggi di legame basati sulla citometria del flusso a fluorescenza; 2) I saggi vincolanti vengono utilizzati per identificare una linea cellulare che esprime il partner di interazione della sonda proteica ricombinante; 3) Viene prodotta una versione che esprime Cas9 della linea cellulare che interagisce con la proteina di interesse e viene eseguita una schermata di knockout basata su genomica CRISPR/Cas9 (Figura 1). In questo screening genetico, il legame di una proteina ricombinante alle linee cellulari viene utilizzato come fenotipo misurabile in cui le cellule all'interno della libreria knockout che hanno perso la capacità di legare la sonda vengono ordinate utilizzando lo smistamento cellulare attivato basato sulla fluorescenza (FACS) e i geni che hanno causato la perdita del fenotipo legante identificato dal sequenziamento. In linea di principio, vengono identificati i geni che codificano il recettore responsabile del legame della sonda avida e quelli necessari per il suo display di superficie cellulare.
Il primo passo di questo protocollo prevede la produzione di acconde proteiche ricombinanti accanite che rappresentano l'ectodominio dei recettori legati alla membrana. Questi recettori sono noti per mantenere frequentemente le loro funzioni di legame extracellulare quando i loro ectodomini sono espressi come una proteina solubile ricombinante1. Per una proteina di interesse, le proteine ricombinanti solubili possono essere prodotte in qualsiasi sistema di espressione proteica eucariotica adatto in qualsiasi formato a condizione che possa essere oligomerizzato per una maggiore avidità vincolante e contiene tag che possono essere utilizzati nei saggi di legame basati sulla citometria basata sulla flutura (ad esempio, tag FLAG, biotina-tag). Protocolli dettagliati per la produzione di ectodomains solubili di recettori della membrana utilizzando il sistema di espressione proteica HEK293, così come diverse tecniche di multimerizzazione e costrutti di espressione proteica per la produzione di proteine penameriche e proteine monomeriche sono stati precedentemente descritti1,17. Il protocollo qui descriverà i passaggi per generare sonde avide fluorescenti da proteine biopoiriche monomeriche coniugandole a streptavidina coniugate a un fluorocro (ad esempio, ficoerythrin, o PE), che può essere utilizzato direttamente nei testdi di legame cellulare e ha il vantaggio di non richiedere un anticorpo secondario per il rilevamento. Sono già stati descritti protocolli generali per l'esecuzione di schermi su scala genomica20,21, quindi il protocollo si concentra principalmente sulle specifiche dell'esecuzione di schermi di legame proteico ricombinanti basati sulla citometria a flusso utilizzando il sistema di screening a eliminazione diretta CRISPR/Cas9 utilizzando la libreria Human V1 ("Yusa")18.
Protocollo
1. Produzione e purificazione di proteine biotinylate Con etichettatura His
- Utilizzare un sistema di espressione proteica a base di cellule di mammiferi o insetti per produrre proteine biotinylate solubili ricombinanti dell'Iconnetto (vedere costrutti di plasmide nella tabella 1).
NOTA: Un protocollo dettagliato per la produzione di biotina monomerica e proteine His-tagged utilizzando il sistema di espressione cellulare HEK293 è descritto da Kerr et al.17. Gli ectodomini proteici espressi utilizzando il sistema di espressione HEK293 sono secreti nel mezzo di coltura. - Raccogliere le proteine solubili pellendo le cellule con la centrifugazione a 3.000 x g per 20 min.
- Filtrare il supernatante attraverso un filtro da 0,22 m e aggiungere le perle di agarose Ni2o-NTA al supernatante proteico filtrato in un rapporto di 1:1.000 (cioè 50 l unli del 50% di liquami agarose in 50 mL di supernatant). Incubare durante la notte o almeno 4-5 h a 4 gradi centigradi su una piattaforma rotante.
- Lavare la colonna di polipropilene aggiungendo 5 mL di tampone di lavaggio di depurazione. Fare riferimento alla tabella 2 per tutte le composizioni di buffer.
- Versare l'intera miscela supernatante perline-proteina nella colonna. Perline si accumulano alla base.
- Lavare le perline 2x con 15 mL di tampone di lavaggio. Per evitare la diluizione proteica, disegnare con cura il buffer di lavaggio residuo dalla colonna con una siringa da 5 mL e scartare.
- Aggiungere con cura 300-500 ll buffer di eguagliazione della sua depurazione direttamente alle perline e incubare per almeno 1 h. Raccogliere la proteina elauta di nuovo estraendo con cura il liquido utilizzando una siringa da 1 mL. Scambiare il buffer di elusione nel buffer desiderato (ad esempio, normalmente PBS o HBS) utilizzando le colonne di depreventivo. Conservare tutte le proteine a 4 gradi centigradi fino a un ulteriore utilizzo.
2. Quantificazione e oligomerizzazione della proteina bioorata monomerica
NOTA: Per aumentare l'avidità vincolante, oligomerizzare le proteine monomeriche biotinylate sullo streptavidin-PE tetramerico prima di utilizzarle nei testagi vincolanti. Raggiungi rapporti di coniugazione ottimali di proteine monomeriche e tetramerici streptavidin-PE testando una serie di diluizione di monomeri biotinylati contro una concentrazione fissa di streptavidina e, stabilindo empiricamente la diluizione minima a cui non possono essere rilevati monomeri biotinylati in eccesso.
- Effettua almeno otto diluizioni seriali di campioni di proteine biotinylate utilizzando un buffer di diluizione appropriato (PBS o HBS con 1% di albumina di siero bovino [BSA]) in una piastra di 96 pozzetti. Assicurarsi che il volume finale di ogni diluizione sia di almeno 200 gradi.
- Fare una piastra duplicata dei campioni rimuovendo 100 l da ogni pozzo e trasferendolo in una nuova piastra da 96 pozzetto. Includere sempre un controllo. In questo caso i controlli sono proteine solo tag (cioè, proteina Cd4 di dominio Cd4 con etichetta Tura 3. Questo verrà utilizzato come sonda di controllo in tutti i saggi vincolanti.
- Diluire streptavidin-PE a 0,1 g/mL nel tampone di diluizione.
- Su una sola delle piastre, aggiungere 100 l dello streptavidin-PE diluito. La piastra duplicata fungerà da controllo. Aggiungere 100 L di tampone di diluizione nella piastra di controllo per equalizzare i volumi.
- Incubare per 20 min a temperatura ambiente (RT). Nel frattempo, bloccare i pozzetti di una piastra rivestita di streptavidin con il tampone di diluizione per 15 min.
- Trasferire il volume totale del campione da entrambe le piastre ai singoli pozzetti delle piastre rivestite di streptavidin e incubare per 1 h a RT.
- Lavare la piastra 3x con 200 l di tampone di lavaggio (cioè PBS o HBS con 0,1% Tween-20, 2% BSA). Aggiungete 100 l l di 2 g/mL di topo anti-ratto Cd4d3-4 IgG (OX68) e incubate per 1 h a RT.
- Lavare la piastra 3x con il tampone di lavaggio. Aggiungete 100 l di coniugato al fosfofolano antito-tono a 0,2 g/mL per 1 ora a RT.
- Lavare la piastra 3x con il tampone di lavaggio e 1x nel tampone di diluizione.
- Preparare il fosfato p-nitrophenyl a 1 mg/mL nel tampone di dietanolamina. Aggiungere 100 l in ogni pozzo e incubare per 15 min.
- Prendere le letture di assorbimento a 405 nm. Utilizzare la diluizione minima alla quale non vi è alcun segnale sulla piastra come fattore di diluizione appropriato per creare tetrameri (Figura 2).
- Fare una soluzione di colorazione 10x tetramer per tutti i campioni e controlli incubando 4 streptavidin-PE e l'appropriata diluizione proteica biotinyalata per 30 min a RT. Memorizzare proteine coniugate in un tubo protetto dalla luce a 4 gradi centigradi fino a un ulteriore utilizzo.
3. Saggi di legame cellulare basati sulla citometria di flusso
- Per le cellule aderenti, rimuovere i supporti di coltura e lavare 1x con PBS senza cazioni divalenti. Aggiungere quindi soluzioni di distacco delle celle (ad esempio, EDTA). Lasciare che le cellule si stacchino per 5-10 min.
NOTA: Evitare l'uso di prodotti a base di trypsin in quanto possono fendere le proteine della superficie cellulare. - Raccogliere le cellule staccate in un tubo. Per le cellule che crescono in sospensione (ad esempio, cellule HEK293), raccogliere direttamente le cellule dai flaconci di coltura in un tubo.
- Celle di pellet a 200 x g per 5 min. Rimuovere il supernatante e risospendere il pellet nel buffer di lavaggio (ad esempio, PBS/1% BSA).
- Contare le cellule utilizzando un emocitometro e regolare la concentrazione a 2,5 x 105-1 x 106 cellule / mL. Aliquota 100 - L di miscela di cellule preparate su una piastra con fondo U o V da 96 pozzetto. Girare la piastra per 5 min a 400 x g. Rimuovere il supernatante con una pipetta multicanale.
- Aggiungete 100 l di sonde proteiche normalizzate fluorescenti etichettate ad alta avidità e controlli nelle piastre precedentemente preparate con cellule e incubate per 1 h a 4 gradi centigradi. Dopo il legame per 1 h, ruotare la piastra a 400 x g per 5 min.
- Rimuovere il supernatante e aggiungere 200 l di buffer di lavaggio (ad esempio, PBS/1% BSA). Mescolare bene pipettando su e giù.
- Pellet le cellule per centrifugazione a 400 x g per 5 min. Ripetere la fase di lavaggio 1x. Dopo due fumi, rimuovere completamente il supernatante e risospendere il pellet cellulare in 100 gradi di PBS.
- Analizzare le cellule per citometria di flusso. Utilizzare il laser giallo-verde (cioè 561 nm) per rilevare la fluorescenza PE.
- Analizzare innanzitutto le cellule che sono state macchiate con la sonda di controllo. Sulla base della distribuzione della fluorescenza PE, disegnare un cancello per la popolazione vincolante in modo che non più dell'1% della cella di controllo cade in questo cancello.
- Analizzare il campione e determinare la frazione di celle che rientra nel cancello di rilegatura.
NOTA: le linee cellulari che mostrano una popolazione di legame superiore sono desiderate per gli schermi genetici, in quanto hanno un rapporto segnale-rumore più elevato. Idealmente oltre l'80% delle cellule dovrebbe cadere all'interno di questo cancello.
4. Determinazione dei contributi vincolanti da epitopi labili di calore e catene laterali del solfato eparano
NOTA: L'attività di molte proteine è labile termica, quindi la perdita di attività di legame dopo il trattamento termico è incoraggiante. Si consiglia di determinare il contributo da glicosaminoglicani caricati negativamente, principalmente solfato eparano (HS), nella mediazione del legame delle proteine ricombinanti. Questo perché il legame da HS nel legame cellulare assay qui descritto può essere additivo piuttosto che codipendente su altri recettori19. Ciò significa che il legame osservato potrebbe essere interamente mediato da catene laterali HS di proteoglicani di superficie cellulare e non da un recettore specifico. Legare a HS sulla superficie cellulare non è necessariamente aspecifica, ma piuttosto una proprietà di una proteina, che è utile sapere prima di eseguire un intero schermo genetico.
- Preparare campioni di proteine trattate termicamente da utilizzare nei saggi vincolanti.
- Riscaldare la proteina monomerica normalizzata ma non coniugata a 80 gradi centigradi per 10 minuti.
- Coniugare la proteina trattata termicamente alla streptavidin-PE assumendo lo stesso rapporto di coniugazione della sua controparte non trattata come determinato da ELISA (fare riferimento alla sezione 2).
- Preparare campioni di proteine bloccate dall'eparina.
- Preparare otto diluizioni 1:3 di eparina solubile in PBS con una concentrazione iniziale di 2 mg/mL e un volume finale di 100 l.
- Incubare 100 l di sonde di legame preparate nelle diluizioni dell'eparina per almeno 30 min.
- Utilizzare proteine trattate termicamente e l'intera miscela di eparina/proteina nei saggi di legame descritti nella sezione 3. I risultati rappresentativi sono riportati nella figura 3A,B.
5. Selezione delle linee cellulari che esprimono stabilmente Cas9
NOTA: Prima che la linea cellulare che lega la sonda di interesse possa essere utilizzata nello screening CRISPR, deve essere progettata per esprimere la nucleale Cas9 e un clone altamente attivo selezionato19.
- Utilizzare il seguente protocollo generale di produzione di lentivirus per produrre lentivirus utilizzando il costrutto lentivirale per l'espressione Cas9 (fare riferimento alla tabella 1).
- Coltura cellule HEK293-FT nei media DMEM/10% FBS a 37 e 5% CO2. Seme HEK293-FT cellule 1 giorno prima della trasfezione in modo che siano confluenti di 80% il giorno della trasfezione.
NOTA: le cellule HEK293FT sono vagamente aderenti; pertanto, quando vengono utilizzati per la produzione di lentivirus, prendere in considerazione la placcatura su flaconi di coltura rivestiti con 0.1% (w/v) gelatina per aumentare l'aderenza. - Eseguire trasfettazioni al mattino. Aggiungere il vettore di trasferimento, il mix di imballaggi e il reagente di trasfezione in supporti compatibili con la traduzione preriscaldata (ad esempio, Opti-MEM). Mescolare invertendo il tubo 10-15x. Incubare per 5 min a RT. Fare riferimento alla tabella 3 per i volumi esatti.
- Aggiungere il reagente di trasfezione come suggerito dal produttore. Mescolare con vortice rapido. Incubare per 30 min a RT.
- Molto attentamente aspirare il mezzo trascorso. Aggiungere supporti compatibili con la trasfezione alla piastra.
- Aggiungere i complessi reagente/DNA di trasfezione goccia sul lato della piastra e lentamente diffondere attraverso la piastra vorticoso molto delicatamente.
- Incubare a 37 oC per 3-5 h e sostituire il mezzo con il supporto D10. Incubare durante la notte.
- Il giorno dopo, al mattino, sostituire il mezzo con un nuovo supporto D10. Incubare durante la notte.
- Il giorno dopo, nel tardo pomeriggio, raccogli il supernabile virale. Filtrare con un filtro di 0,45 m con rilegatura a bassa proteina. Facoltativamente, aggiungere il nuovo supporto D10, incubare durante la notte e ricordare il supernatante il giorno successivo.
- I supernatanti a i virus sono stabili a 4 gradi centigradi solo per pochi giorni. Conservare a -80 gradi centigradi per lo stoccaggio a lungo termine.
NOTA: Per generare una preparazione lentivirale altamente concentrata, che potrebbe essere auspicabile per la trasduzione di cellule difficili da traslare, i supernatanti possono anche essere concentrati con la centrifugazione a 6.000 g durante la notte a 4 gradi centigradi. Segna il pellet virale traslucido con una penna resistente all'etanolo e scarta il supernatante. Risospendere il pellet in 1/100 del volume originale per un aumento di 100 volte la concentrazione.
- Coltura cellule HEK293-FT nei media DMEM/10% FBS a 37 e 5% CO2. Seme HEK293-FT cellule 1 giorno prima della trasfezione in modo che siano confluenti di 80% il giorno della trasfezione.
- Trasduci le cellule con lentivirus.
- Piastra 1 x 106 cellule per pozzo in una piastra 6 po' di pozzi con 3 mL di supporti di coltura appropriati. Alcune cellule sono più facilmente trasdotte di altre. Per trasducire le cellule facili da trasducio (ad es. cellule HEK), aggiungere direttamente il lentivirus alle cellule. Per le cellule difficili da trasducio, potrebbe essere necessario seguire un protocollo di spinoculazione come descritto di seguito.
- 2 mL di 2-5 x 106 celle/mL in un tubo conico da 15 mL.
- Aggiungere il lentivirus insieme a 8 brommide di esaditohrine e incubare RT per 30 min.
- Centrifuga per 100 min a 800 x g a 32 gradi centigradi. Quindi risospeso le celle nello stesso supporto e aggiungere la sospensione cellulare in flaconi di coltura appropriati con supporti appropriati.
- Consentire le trasduzioni per almeno 24 h. Successivamente rimuovere il supporto contenente il virus e aggiungere un nuovo supporto.
- Dopo un altro 24 h, cambiare il supporto a uno che è integrato con gli antibiotici appropriati. Il costrutto Cas9 contiene una cassetta di resistenza blasticidina per la selezione.
NOTA: La quantità di blasticidin deve essere ottimizzata per ogni linea cellulare eseguendo una curva di uccisione di risposta alla dose. Una concentrazione di blasticidin a una frequenza di blasticidin a 2,5-50 g/mL dovrebbe uccidere la maggior parte delle linee cellulari non tradutte entro 10 giorni dalla trasduzione.
- Piastra 1 x 106 cellule per pozzo in una piastra 6 po' di pozzi con 3 mL di supporti di coltura appropriati. Alcune cellule sono più facilmente trasdotte di altre. Per trasducire le cellule facili da trasducio (ad es. cellule HEK), aggiungere direttamente il lentivirus alle cellule. Per le cellule difficili da trasducio, potrebbe essere necessario seguire un protocollo di spinoculazione come descritto di seguito.
- Eseguire la selezione fino a quando tutte le cellule nella piastra di controllo (cioè le cellule non tradotte che sono state trattate con la stessa concentrazione di antibiotici di selezione) non vengono uccise.
6. Selezione di cloni di attività Cas9 elevati
NOTA: Polyclonal Cas9 può essere utilizzato per eseguire con successo schermi genetici; tuttavia, la selezione di un clone con un'elevata attività Cas9 migliora i risultati dello screening18.
- Utilizzare la diluizione limitante o le singole cellule resistenti alla blasticidina in pozzi di tre 96 lastre ben contenenti mezzi di coltura integrati con blasticidin. I cloni inizieranno ad emergere tra 2-4 settimane. Selezionare 10-20 cloni ed espandersi in 6 piastre di pozzo.
- Prova i cloni per l'attività di Cas9 usando il sistema GFP-BFP (proteina fluorescente verde fluorescente proteina-blu) che utilizza un sistema di knockout genico esogeno in cui le cellule vengono trasserie sia con un costrutto che esprime GFP con un GFP che punta al gRNA o un gRNA vuoto come controllo18.
- Plasmidi dei giornalisti d'ordine: plasmide GFP-BFP, plasmide Control-BFP (Tabella 1).
- Produrre lentivirus sia per il plasmide GFP-BFP che per il plasmide Control-BFP utilizzando il protocollo di produzione di lentivirus descritto nella sezione 5.1.
- Traduci ogni clone della linea cellulare che esprime Cas9 con il lentivirus che codifica il sistema GFP-BFP e Control-BFP separatamente. Seguire il protocollo nella sezione 5.2.
- Dopo 3 giorni di trasduzione, esaminare la fluorescenza GFP-BFP di ogni clone utilizzando la citometria di flusso. Utilizzare il laser a 488 nm e il laser a 405 nm per rilevare rispettivamente GFP e BFP.
- Quantitarlare l'attività di Cas9 in ogni clone esaminando il rapporto tra BFP e cellule ad alto aspetto GFP-BFP-double. Le celle Cas9 ad alta attività dovrebbero idealmente avere efficienza di eliminazione delle eliminagie >95% GFP (Figura 4).
7. Generazione di una libreria knockout di screening CRISPR-Cas9 a livello genomico
- Per lo screening a livello genomico utilizzando la libreria Human V118, ordinare la libreria a livello di genoma (fare riferimento alla tabella 1) e preparare la libreria di plasmide dalla pugnalata batterica utilizzando il protocollo fornito sotto "Protocolli per la replica della libreria" nel manuale del produttore.
- Utilizzare la preparazione del plasmide della libreria a livello di genoma per produrre una libreria lentivirale che codifica i gRNA per l'interruzione mirata dei geni umani utilizzando il protocollo di produzione del lentivirus descritto nella sezione 5.1.
NOTA: Una buona pratica è quella di produrre un singolo lotto di preparazione lentivirale che è ottimizzato per la trasduzione per migliorare la coerenza sperimentale. - Utilizzare il protocollo di trasduzione nella sezione 5.2 per eseguire trasduzioni di prova su piccola scala per determinare la quantità necessaria di virus per ogni linea cellulare per ottenere una trasduzione del 30%. Utilizzare la citometria a flusso per valutare la fluorescenza BFP come proxy per l'efficienza della trasduzione.
- Per trasdurre le cellule HEK293, è sufficiente aggiungere la preparazione lentivirale predeterminata a 30-50 x 106 cellule coltivate in normali supporti di crescita per 4 h. Quindi rimuovere i media con lentivirus e sostituirli con nuovi media di crescita.
- Per altre linee cellulari, utilizzare il protocollo di spinoculazione nella sezione 5.2.1 ma su scala più ampia, in modo che un totale di 30-50 x 106 cellule siano tradotte. Per questo, 2 mL di 5 x 106 celle /mL in un tubo conico da 15 mL e procedere come indicato.
- Per le linee cellulari aderenti, selezionare le cellule tradotte aggiungendo la panchina 24 h dopo la trasduzione.
NOTA: Ottimizzare le concentrazioni di puromycin eseguendo una curva di uccisione di risposta alla dose. Normalmente le concentrazioni tra 1-10 g/mL dovrebbero uccidere le cellule non traduttrici entro 3-5 giorni. Evitare di utilizzare concentrazioni più elevate di puromycina perché questo può aumentare le probabilità di selezionare cellule che sono state trasdotte da più di una guida singola RNA (sgRNA). - Per le cellule sospensione, raccogliere le cellule tradusse (cioè BFP positivo) 3 giorni dopo la trasduzione utilizzando una selezionatrice cellulare e generare librerie che contengono almeno 10 x 106 cellule. Una volta selezionato utilizzando BFP, far crescere le cellule nei media integrate con una quantità appropriata di puromycin.
NOTA: Evita le selezioni solo con la puromicina per le linee cellulari delle sospensioni, perché è difficile rimuovere le cellule morte e i detriti dalle colture cellulari in sospensione che possono interferire con lo smistamento delle cellule. - Cultura biblioteca mutante per 9-16 giorni posttrassduzione con passaggio regolare ogni 2-3 giorni.
8. Screening genetico per il rilegatura della superficie cellulare
- Pellet la biblioteca cellulare mutante a 200 x g per 5 min e risospendere le cellule in PBS.
- Dividere le cellule in due tubi conici da 15 mL con almeno 50 x 106 cellule in ogni tubo.
- Ruotare un tubo conico a 200 x g per 5 min, rimuovere il supernatante e congelare il pellet cellulare a -20 gradi centigradi. Questa è la popolazione di controllo e verrà elaborata in un secondo momento.
- Risospendere il pellet nell'altro tubo in 10 mL di PBS/1% BSA. Mettere da parte 100 l di cellule come controllo negativo su una piastra di 96 pozze.
- Aggiungere la proteina ricombinante preconcetta appropriata alla sospensione cellulare nel tubo conico e alle proteine di controllo negativo alla piastra del pozzo 96.
- Eseguire la colorazione cellulare per almeno 1 h a 4 gradi centigradi su un rotore della parte superiore con rotazione delicata (6 giri).
- Pellet le cellule a 200 x g per 5 min, rimuovere il supernatante. Eseguire due fasi di lavaggio, quindi risospendere le celle in 5 mL di PBS.
- Filtrare le cellule anche se un colino cellulare di 30 m per rimuovere gli ammassi di cellule. Analizzare utilizzando una sequenza di flusso.
- Utilizzare il campione di controllo negativo per eseguire il gate per le celle BFP/PE-.
- Ordinare il campione e raccogliere le celle BFP/PE-. Le porte di ordinamento dipenderanno dal legame delle cellule alla proteina, ma normalmente sono raccolti l'1-5% dei campioni negativi del PE. Un esempio di un cancello di smistamento è disponibile nella figura supplementare 1.
- Raccogli 500.000-1.000.000 di celle dal cancello selezionato. Dato il basso numero di cellule, prendere in considerazione la raccolta dei campioni in un tubo di centrifugazione da 1,5 ml per ridurre al minimo le perdite.
- Pellet le cellule ordinate per centrifugando a 500 x g per 5 min. Rimuovere con attenzione il supernatante e scartare. È possibile conservare il pellet a -20 gradi centigradi per un massimo di 6 mesi.
9. Estrazione del DNA genomico e prima PCR per l'arricchimento del gRNA
- Estrarre il DNA genomico dalla popolazione ad alta complessità.
- Se la popolazione di controllo è stata congelata a -20 gradi centigradi, estrarre il tubo conico e aggiungere PBS. Tenere sul ghiaccio per scongelare il pellet.
- Utilizzare un kit commerciale (vedi Tabella dei materiali)utilizzando le raccomandazioni del produttore per estrarre il DNA genomico da 50 x 106 cellule. Regolare la concentrazione del DNA a 1 mg/mL.
- Per ogni campione, impostare un mix master per la PCR corrispondente a 72 g di DNA. Aliquota 50 l per pozzo in 36 pozzetti di una piastra PCR 96 pozzetto. Le sequenze di primer necessarie sono elencate nella tabella 4. Utilizzare la guida nelle tabelle 5 e 6.
- Risolvere 5 - L della PCR da 6-12 campioni rappresentativi su un gel di agarose 2% (w/v). Deve essere osservata una singola banda trasparente a 250 pb. Se le bande sono deboli, ripetere la PCR per altri 2-3 cicli.
- Utilizzare una pipetta multicanale per raccogliere 5 ll l di prodotti PCR da ogni pozzo (180 ll in totale) e raggrupparli in un serbatoio con 900 - L di buffer di rilegatura da un kit commerciale (vedi Tabella dei materiali).
- Purificare i prodotti PCR utilizzando un kit di purificazione PCR commerciale. Eluso il DNA in 50 - L di tampone di eluizione da un kit commerciale (vedi Tabella dei materiali)e misura la concentrazione del DNA.
- È improbabile che i campioni ordinati per la perdita di fenotipo legante siano composti da un gran numero di cloni indipendenti. Pertanto, non è necessario eseguire la PCR con 72 g di DNA. Isolare il DNA utilizzando un kit commerciale appropriato (vedere Tabella dei materiali). Impostare 3-4 reazioni PCR utilizzando il protocollo descritto in precedenza (sezione 9.1.3) con 100 ng/L DNA. Se il numero di cellule ordinate è inferiore a 100.000 utilizzare lisati cellulari invece di preparati di DNA genomico.
- Aliquota circa 10.000 cellule / bene in una piastra PCR 96 bene.
- Pellet le cellule nella piastra e rimuovere con attenzione la maggior parte del supernatante. Il pellet non sarà visibile.
- Aggiungete 25 gradi d'acqua in ogni pozzo e scaldate i campioni a 95 gradi centigradi per 10 minuti.
- Aggiungete 5 gradi di proteineassi K appena diluite in ogni pozzo per 1 h e incubate a 56 gradi centigradi. Quindi riscaldare il campione per 10 minuti a 95 gradi centigradi per inattivare la proteinasi K.
- Utilizzare 10 l of cell lyssate mixture per reazione PCR. Lysates devono essere utilizzati entro 24 h.
10. Secondo ciclo di PCR per la codifica a barre e la sequenza degli indici
- Diluire i prodotti dal primo PCR rotondo a 40 pg/L.
- Impostare una PCR per campione (utilizzare la guida fornita nelle tabelle 7 e 8). L'uso di una polimerasi ad alta fedeltà è importante per ridurre al minimo gli errori introdotti dalla polimerasi durante l'amplificazione dello sgRNA.
- Risolvere 5 - L di prodotti PCR su un 2% (w/v) gel di agarose. Deve essere osservata una singola banda trasparente a 330 bps.
- Purificare i prodotti PCR utilizzando perline paramagnetiche aggiungendo 31,5 l'l of (0,7 volte di volume totale) di perline risospese ai prodotti PCR, mescolando bene e incubando per 5 min a RT.
- Posizionare il tubo su un rack magnetico per 3 min. Le perline devono essere catturate sul lato della piastra e la soluzione deve essere chiara. Rimuovere e scartare con attenzione il supernatante.
- Aggiungere al tubo 150 l l dell'80% di etanolo appena preparato. Incubare per 30 s, e poi rimuovere con attenzione e scartare il supernatante.
- Ripetere il passaggio 13.6, questa volta con 180 gradi. Quindi asciugare all'aria le perline per 5 min.
- Rimuovere il tubo dal magnete. Eluire il bersaglio del DNA da perline a 35 litri di tampone EB sterile. Incubare per 3 min, quindi rimettere il tubo nel rack magnetico per 3 min.
- Trasferire circa 30 ll l del supernatante contenente i prodotti PCR eluiti in un tubo pulito.
- Sequenziare i campioni su una piattaforma di sequenziamento di nuova generazione. Per la libreria gRNA HumanV1, utilizzare il primer personalizzato elencato nella tabella 4 per sequenza 19 bp.
11. Analisi bioinformatica per identificare il recettore e le vie correlate
- Mappare le sequenze dalla popolazione ordinata e non ordinata alla libreria di riferimenti utilizzando la funzione di conteggio di MAGeCK. La funzione produrrà un file di conteggio non elaborato (supplementari tabella 1).
NOTA: istruzioni dettagliate sull'installazione di MAGeCK e sull'utilizzo di diverse funzioni all'interno di MAGeCK sono descritte in un protocollo pubblicato in precedenza da Wang et al.20. - Controllare lo standard tecnico della libreria di controllo utilizzata sullo schermo.
- Mediana normalizza i conteggi grezzi e utilizza il pacchetto ggplot2 in R21 o un software equivalente per tracciare un grafico della funzione di densità cumulativa empirica dei conteggi in plasmide e controllare i campioni non ordinati (Figura 5A).
- Eseguire la funzione -test di MAGeCK usando i conteggi della popolazione plasmidcome "controllo" e i conteggi da campioni di controllo non ordinati come campione di "test". La funzione è produrrà un file di riepilogo genico (Tabella supplementare 1).
- Aprire il file di riepilogo del gene e disegnare la distribuzione delle modifiche di log-fold (colonnaneg-lfc) per i geni essenziali e non essenziali precedentemente classificati22 (Figura 5B).
- Selezionare geni significativamente impoveriti (neg-fdr < 0.05) ed eseguire l'analisi dell'arricchimento del percorso utilizzando il pacchetto enrichr23 o qualsiasi pacchetto di arricchimento del percorso equivalente in R (Figura 5C).
- Eseguire la funzione -test di MAGeCK con l'impostazione predefinita. Utilizzare i conteggi non elaborati dal campione di controllo non ordinato come "controllo" e conta dal campione ordinato come "trattamento" quando si esegue l'analisi.
- Aprire il file di riepilogo del gene generato da MAGeCK e classificare la colonna pos-rank in ordine crescente. Utilizzare FDR (colonnapos-fdr) < 0.05 come limite per l'identificazione degli hit. Il recettore è di solito classificato altamente, spesso in prima posizione.
- Tracciare i punteggi RrA (Robust-Ranking-Algorithm) per la selezione positiva (pos-score) in R o un software equivalente (Figura 5D).
- Selezionare i risultati genici ed eseguire l'analisi dell'arricchimento del percorso utilizzando il pacchetto di arricchimento o qualsiasi pacchetto di arricchimento del percorso equivalente in R per identificare i percorsi arricchiti.
Risultati
Vengono forniti i dati di sequenziamento da due schermate di eliminazione su scala genoma rappresentativa per l'identificazione del partner legante di TNFSF9 umano e P. falciparum RH5 eseguite rispettivamente nelle cellule NCI-SNU-1 e HEK293 (tabella supplementare 1). Il comportamento vincolante di RH5 è stato influenzato sia dal solfato eparano che dal suo recettore noto BSG24 (Figura 3C), mentre TNFRSF9 specificamente legato al suo recettore noto TNFSF9 e non ha perso il legame al preincubation con eparina solubile. La proteina 3 nella figura 3B rappresenta il TNFRSF9.
Per entrambe le linee cellulari, viene fornita anche la distribuzione dei gRNA nella libreria mutante di controllo dopo 3 giorni (9, 14 e 16 giorni di posttrasduzione)(tabella supplementare 1). La distribuzione di gRNA ha rivelato che la complessità della libreria è stata mantenuta nel corso dell'esperimento (Figura 5A). Lo screening genetico per l'identificazione del legamento per il TNFSF9 è stato eseguito il giorno 14 post-trasduzione, mentre quello per RH5 è stato eseguito giorno 9 posttrasduzione. La qualità tecnica degli schermi è stata valutata esaminando la distribuzione dei cambiamenti di piegatura osservati dei gRNA mirati a un insieme di riferimento di geni non essenziali rispetto alla distribuzione per serie di riferimento di geni essenziali22 (Figura 5B). Inoltre, l'arricchimento a livello di percorso ha anche rivelato che i percorsi essenziali attesi sono stati identificati e notevolmente arricchiti nella popolazione "drop-out" quando si confronta il campione di controllo con la biblioteca plasmide originale. Un esempio con il giorno 14 NCI-SNU-1 esempio è illustrato nella Figura 5C.
La distribuzione dei gRNA nel controllo rispetto alla popolazione ordinata utilizzando la funzione -test di MAGeCK (vedi tabella supplementare 1 per l'output di sintesi genica da MAGeCK) è stata utilizzata per identificare il recettore dagli schermi fenotipici. Il punteggio RRA modificato riportato da MAGeCK nell'analisi a livello genico viene tracciato rispetto ai geni classificati in base ai valori p. Il punteggio RRA in MAGeCK fornisce una misura in cui i gRNA sono classificati costantemente più in alto del previsto. Nella schermata per TNFRSF9, il primo hit è stato TNFSF9, che è un partner di associazione noto di TNFRSF9 (Figura 5D). Inoltre, è stato identificato anche un certo numero di geni correlati alla via TP53. Nel caso di RH5, oltre al recettore noto (BSG) e al gene necessario per la produzione dei GAG solfati (SLC35B2), è stato identificato anche un gene aggiuntivo (SLC16A1).E SLC16A1 è un accompagnatore necessario per il traffico bsG alla superficie delle cellule25. Insieme, questi risultati dimostrano la capacità dello schermo di identificare i recettori che interagiscono direttamente e i componenti cellulari necessari affinché tale recettore sia espresso sulla superficie delle cellule in forma funzionale.
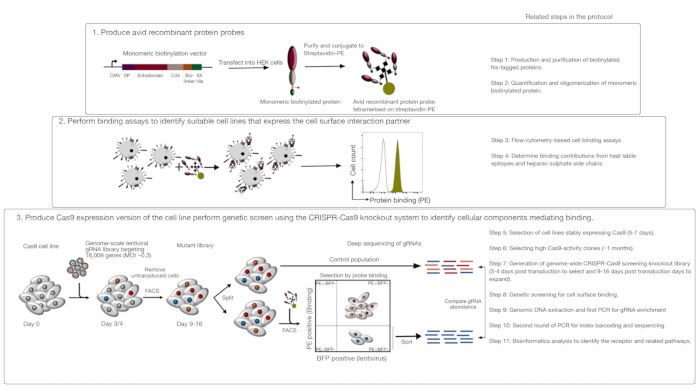
Figura 1: Panoramica dell'approccio di screening genetico per identificare i recettori della superficie cellulare. Questo saggio è costituito da tre passaggi principali: in primo luogo, le proteine ricombinanti che rappresentano l'ectodominio dei recettori della superficie cellulare sono espresse in una linea cellulare che può aggiungere modifiche post-traduzionali strutturalmente critiche come le cellule HEK293. Gli ectoidric delle proteine monomeriche sono oligomerizzati coniugando streptavidin-PE per aumentare la loro avidità vincolante. In secondo luogo, queste sonde avide sono utilizzate nei saggi di legame cellulare in cui la colorazione luminosa sulle linee cellulari indicata da un prominente spostamento nella fluorescenza PE (in verde) rispetto a una proteina di controllo negativo (in nero) dimostra la presenza di un partner di legame della superficie cellulare. In terzo luogo, vengono selezionate linee cellulari che esprimono il Cas9 positivo al recettore e viene eseguito lo screening su scala del genoma utilizzando gRNA mirati alla stragrande maggioranza dei geni codificanti delle proteine. Durante la generazione di librerie mutanti, è comune usare l'efficienza di trasduzione del 30%, che si basa sulla probabilità di distribuzione di Poisson che assicura che ogni cellula riceva un singolo gRNA in modo tale che il fenotipo risultante sia attribuito a un knockout specifico. Il marcatore BFP espresso dalle cellule trasdotte viene utilizzato per selezionare le celle contenenti gRNA utilizzando FACS. Gli schermi fenotipici vengono eseguiti tra 9-16 giorni dopo la trasduzione. Il giorno dello schermo, la popolazione totale di cellule mutanti è divisa in due. Una metà è mantenuta come la popolazione di controllo e l'altra metà è selezionata per il legame proteico ricombinante. Le cellule della biblioteca mutante che non sono più in grado di legare la proteina ricombinante vengono ordinate utilizzando FACS e l'arricchimento dei gRNA nella popolazione di smistamento e controllo viene utilizzato per identificare i geni necessari per il legame della superficie cellulare della sonda avida etichettata. Sono indicati i passaggi del protocollo che richiedono un tempo considerevole. Questa cifra è stata modificata da Sharma etal. Fare clic qui per visualizzare una versione più grande di questa figura.
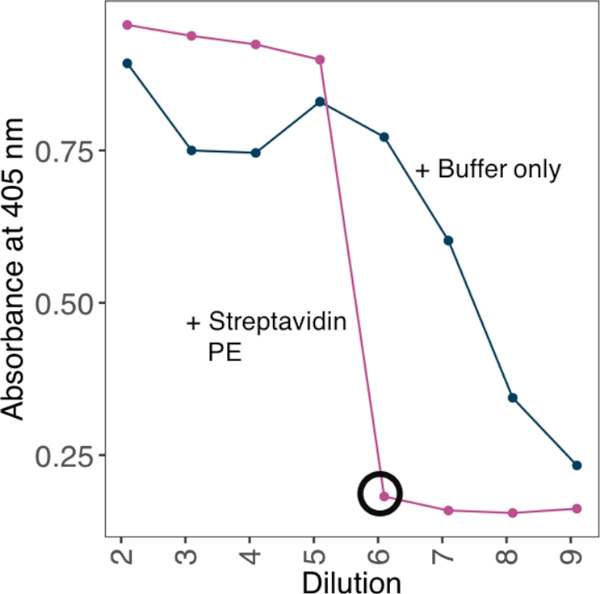
Figura 2: Stabilire i rapporti tra proteine biotinyalate e streptavidin-PE utilizzando un metodo basato su ELISA. Un esempio di strategia di coniugazione streptavidin-PE utilizzata per generare una sonda avida da una proteina monomerica biotinylata. Una serie di diluizione di monomeri biotinylati è stata incubata contro una concentrazione fissa di streptavidina. ELISA ha determinato la diluizione minima alla quale non è possibile rilevare monomeri biolettivi in eccesso. ELISA è stato eseguito con o senza preincubare una gamma di diluizioni proteiche con 10 ng di streptavidin-PE. In presenza di streptavidin-PE, la diluizione minima alla quale non è stato identificato alcun segnale (nero cerchiato) e la quantità di proteine necessarie per la saturazione è stata calcolata per generare una soluzione di stock 10x con 4 streptavidin-PE. Fare clic qui per visualizzare una versione più grande di questa figura.
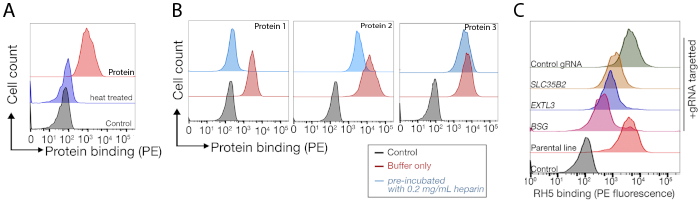
Figura 3: Legame rappresentativo delle proteine alle linee cellulari. (A) Il legame proteico alle linee cellulari ha avuto un chiaro aumento della fluorescenza associata alle cellule rispetto al campione di controllo. Il trattamento termico (80 gradi centigradi per 10 minuti) della proteina ricombinante ha abrogato tutto il legame con un controllo negativo, dimostrando che il comportamento legante dipendeva dalla proteina piegata correttamente. (B) Diverse classi di comportamento di legame proteico alle superfici cellulari; dipendenza dai GAS abase per la protezione dell'utente. Da sinistra a destra, le proteine possono essere classificate in tre tipi: Proteina di tipo 1 solo annunci a HS. Queste proteine perdono il loro legame dopo la preincubazione con concentrazioni di eparina superiori a 0,2 mg/mL. Proteina di tipo 2 si lega all'HS oltre a un recettore specifico. Queste proteine perdono un legame parziale negli esperimenti di preblocco. Proteina di tipo 3 non lega HS. Queste proteine non perdono il legame rispetto alle linee parentali. (C) Un esempio di una proteina (cioè RH5) che si lega all'HS e a un recettore specifico in modo additivo. Puntare al recettore (ad esempio, BSG) o agli enzimi necessari per la sintesi HS (ad esempio, SLC35B2, EXTL3) riduce solo parzialmente il legame di RH5 alle cellule rispetto ai controlli. Le linee policlonali trasdotte possono essere utilizzate in tali esperimenti per stabilire un comportamento di legame. Questa cifra è stata modificata da Sharma etal. Fare clic qui per visualizzare una versione più grande di questa figura.
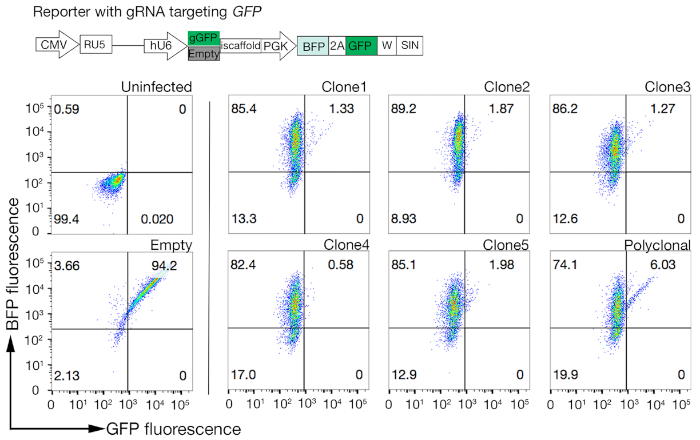
Figura 4: Selezione di linee cellulari clonali con elevata attività Cas9. L'efficienza di editing del genoma delle linee sia policlonali che clonate delle linee cellulari NCI-SNU-1 è stata valutata utilizzando il sistema di reporter GFP-BFP, in cui le linee cellulari sono state trasdotte con virus con un GFP codificato plasmido mirato a gRNA o senza (cioè "vuoto"). Viene raffigurato uno schema. La citometria di flusso è stata utilizzata per testare sia l'espressione BFP che GFP dopo la trasduzione e rispetto al controllo non infetto. L'espressione GFP è stata utilizzata come proxy per l'attività Cas9, mentre l'espressione BFP ha contrassegnato le cellule trasdotte. Il profilo per le cellule infette e vuote non si vedeva simile a tutti i cloni. I profili rappresentativi sono rappresentati nel pannello di sinistra. Tutti e cinque i cloni della linea cellulare NCI-SNU-1 hanno mostrato una perdita più elevata di GFP rispetto alla linea policlonale (pannello destro), con clone 4 che mostra la massima efficienza con la più bassa popolazione refrattaria. Questa cifra è stata modificata da Sharma etal. Fare clic qui per visualizzare una versione più grande di questa figura.
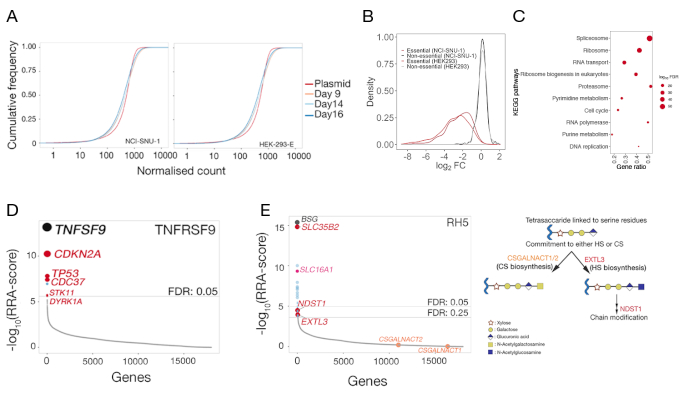
Figura 5: Risultati rappresentativi da schermi genetici per l'identificazione dei partner di legame della superficie cellulare. (A) Grafici della funzione di distribuzione cumulativa confrontando l'abbondanza di gRNA nella biblioteca di plasmide con le librerie mutanti delle cellule HEK-293-E e NCI-SNU-1 nel giorno 9, 14 e 16 giorni di posttrasduzione. Per un determinato numero, la funzione densità cumulativa segnala la percentuale di punti dati che erano al di sotto di tale soglia. Il piccolo spostamento della popolazione di cellule mutanti rispetto alla popolazione plasmide originale rappresenta l'esaurimento in un sottoinsieme di gRNA rispetto alla biblioteca plasmide. (B) Distribuzione delle modifiche di ripiegamento dei log nei geni che in precedenza erano classificati come essenziali (rosso) o non essenziali (nero) nelle linee cellulari HEK293 e NCI-SNU-1. La distribuzione dei cambi di piegatura per i geni non essenziali si è centrata a 0, mentre quella per i geni essenziali si è spostata verso sinistra verso cambiamenti negativi della piega. (C) Percorsi significativamente arricchiti nei geni esauriti nella popolazione di controllo mutato NCI-SNU-1 14 giorni dopo la trasduzione. Sono stati identificati i percorsi noti-essenziali delle cellule previsti. (D) Robust Rank Algorithm (RRA)-punteggio per i geni che sono stati arricchiti nelle cellule ordinate che avevano perso la capacità di legare la sonda TNFRSF9. I geni sono stati classificati in base al punteggio RRA. Il partner di interazione noto TNFSF9 e i geni correlati al percorso TP53 (etichettati in rosso) sono stati identificati sullo schermo. (E) Punteggi RRA ordinati per i geni identificati dall'analisi dell'arricchimento del gRNA necessari per il legame di RH5 alle cellule HEK293 (pannello sinistro). SLC35B2 e SLC16A1 sono state identificate entro una soglia di percentuale di falsi scoperti (FDR) del 5%. Nell'FDR sono stati identificati altri due geni nella via della biosintesi HS (ad esempio EXTL3 e NDST1)entro la FDR del 25%. Schema raffigurante il percorso di biosintesi GAG generale con i geni pertinenti mappati ai passi corrispondenti (pannello 2). I geni necessari per l'impegno nella biogenesi del solfato di condroitina (cioè CSGALNACT1/2) non sono stati identificati sullo schermo. Questa cifra è stata modificata da Sharma etal. Fare clic qui per visualizzare una versione più grande di questa figura.
| Nome plasmide | Plasmide # | Utilizzare |
| Costrutto di espressione proteica: CD200RCD4d3-4-bio-linker-his | Addgene: 36153 | Produzione di proteine ricombinanti con CD4d3, biotina e 6-suoi tag. |
| pMD2.G | Addgene: 12259 | Busta VSV-G che esprime plasmide; produzione di lentivirus |
| psPAX2 (in base al sistema psPAX2) | Addgene: 12260 | Plasmide di imballaggio lentivirale, produzione di lentivirus |
| Cas9-costrutto: pKLV2-EF1a-Cas9Bsd-W | Addgene: 68343 | Produzione di linea Cas9 che esprime in modo costitutivo |
| Costrutto di espressione gRNA: pKLV2-U6gRNA5(BbsI)-PGKpuro2ABFP-W | Addgene: 67974 | Vettore di espressione gRNA CRISPR con scaffold migliorato e marcatori puro/BFP |
| Libreria Knockout CRISPR a livello genomico migliorato | Addgene: 67989 | Una libreria di gRNA contro 18.010 geni umani, progettata per l'uso in lentivirus. |
| Costrutto GFP-BFP: pKLV2-U6gRNA5(gGFP)-PGKBFP2AGFP-W | Addgene: 67980 | Cas9 activity reporter con BFP e GFP. |
| Costrutto vuoto: pKLV2-U6gRNA5(vuoto)-PGKBFP2AGFP-W | Addgene: 67979 | Cas9 activity reporter (controllo) con BFP e GFP. |
Tabella 1: Plasmidi utilizzati in questo approccio.
| Nome buffer | Componenti |
| HBS (10X) | 1,5 M NaCl e 200 mM HEPES in acqua MiliQ, regolare al pH 7.4 |
| PBS (10X) | 80 g NaCl, 2 g KCl, 14,4 g Na2HPO4 e 2,4 g KH2PO4 in acqua MiliQ, regolare a pH 7.4 |
| Buffer fosfato di sodio (80mM di stock) | 7.1 g Na2HPO4.2H2O, 5,55 g NaH2PO4, regolare al pH 7.4 |
| Buffer di rilegatura per la depurazione | 20 mM Disfato buffer di sodio, 0,5 M NaCl e 20 mM Imidazole, regolare a pH 7.4 |
| Cuscinetto di elusione per la depurazione | 20 mM Disfato buffer di sodio, 0.5M NaCl e 400 mM Imidazole, regolare a pH 7.4 |
| Buffer di etanolamina | 10% di etanolamina e 0,5 mM MgCl2 in acqua MiliQ, regolare al pH 9.2: |
| D10 | DMEM, 1% penicillina-streptomycin (100 unità/mL) e 10% di calore inattivato FBS |
Tabella 2: Buffer necessari per questo studio.
| Componenti | Piatto di 10 cm | Piastra a 6 benessere |
| 293CELLULE cellule | 70-80% confluenti | 70-80% confluenti |
| Supporti compatibili con la trasfezione (Opti-MEM) (Passaggio 5.1.2) | 3 mL | 500 lL |
| Supporti compatibili con la trasfezione (Opti-MEM) (Passaggio 5.1.4) | 5 mL | 2 mL |
| Vettore di trasferimento lentivirale | 3 g | 0,5 g di g |
| psPax2 (vedi tabella 1) | 7,4 g di g | 1,2 g di g |
| pMD2.G (vedi tabella 1) | 1,6 g di g | 0,25 g di g |
| Reagente PLUS | 12 ll | 2 ll |
| Lipofectamine LTX | 36 l l | 6 ll |
| D10 (Passaggio 7.1.7) | 5 mL | 1,5 mL |
| D10 (Passaggio 7.1.8 e 7.1.10) | 8 mL | 2 mL |
Tabella 3: Quantità e volumi di reagenti per miscela di imballaggi lentivirus.
Tabella 4: Sequenze di primer per amplificare gRNA e NGS. Fare clic qui per visualizzare questo file (fare clic con il pulsante destro del mouse per scaricare).
| Reagente | Volume per reazione | Miscela master (x38) |
| Q5 Hot Start Ad alta fedeltà 2x | 25 ll | 950 ll |
| Primer (L1/U1) mix (10 M ciascuno) | 1 ll | 38 ll |
| DNA genomico (1 mg/mL) | 2 ll | 72 ll |
| H2O | 22 ll | 1100 lL |
| Totale | 50 ll | 1900 lL |
Tabella 5: PCR per l'amplificazione dei gRNA da campioni ad alta complessità.
| Numero ciclo | Denatura | Ricottura | Estensione |
| 1 | 98 gradi centigradi, 30 anni | ||
| 2-24 | 98 gradi centigradi, 10 anni | 61 s, 15s | 72 gradi centigradi, 20 anni |
| 25 | 72 gradi centigradi, 2 min |
Tabella 6: condizioni PCR per il primo PCR.
| Reagente | Volume per reazione |
| KAPA HiFi HotStart ReadyMix | 25 ll |
| Primer (PE1.0/index primer) mix (5 M ciascuno) | 2 - L |
| Primo prodotto PCR (40 pg/L) | 5 ll |
| H2O | 18 ll |
| Totale | 50 ll |
Tabella 7: PCR per l'indicizzazione degli sgRNA da schermi genetici.
| Numero ciclo | Denatura | Ricottura | Estensione |
| 1 | 98 gradi centigradi, 30 anni | ||
| 2-15 | 98 gradi centigradi, 10 anni | 66 gradi centigradi, 15 anni | 72 gradi centigradi, 20 anni |
| 16 | 72 gradi centigradi, 5 min |
Tabella 8: condizioni PCR per il secondo PCR.
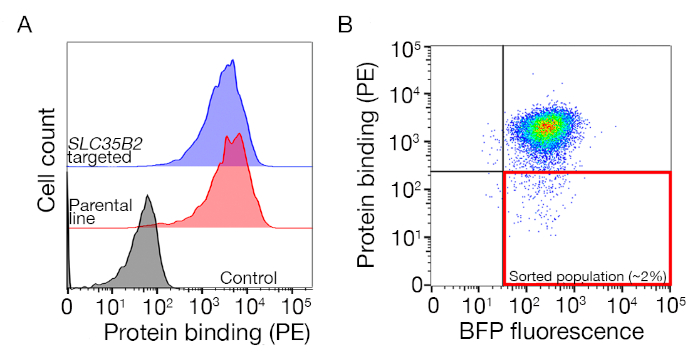
Figura supplementare S1: Una guida per disegnare cancelli per lo smistamento della popolazione non vincolante. (A) Un candidato proteico ideale per lo screening dovrebbe avere un chiaro spostamento della popolazione legante rispetto alla popolazione di controllo e il legame dovrebbe essere mantenuto su cellule prive di macchinari per la biosintesi HS. Un esperimento di blocco dell'eparina può essere utilizzato al posto dei test sulle linee cellulari mirate SLC35B2. (B) Sono state raccolte cellule prive della colorazione superficiale dall'ectodominio proteico, ma che esprimono la fluorescenza Della BFP dalla trasduzione lentivirale. Le cellule visualizzate provengono da una schermata per l'identificazione del recettore per GABBR222. Questa cifra è stata modificata da Sharma etal. Fare clic qui per visualizzare una versione più grande di questa figura.
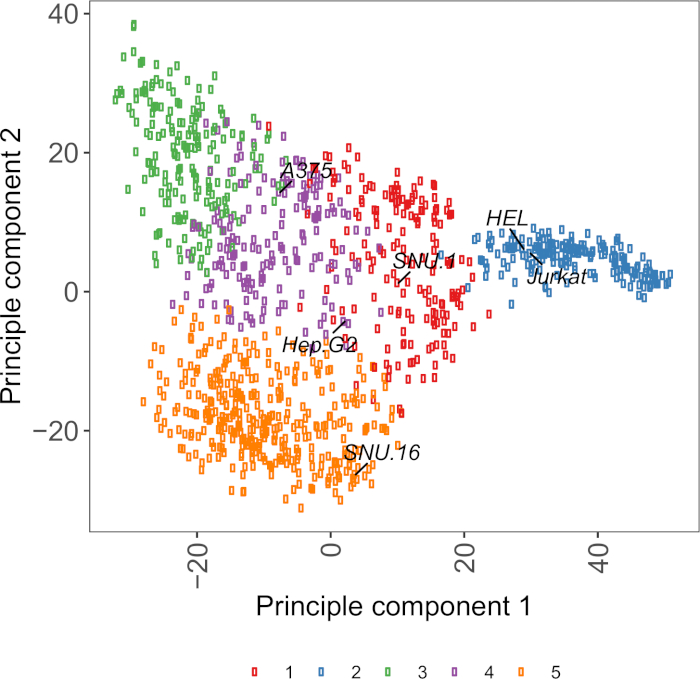
Figura supplementare S2: Trama PCA basata sulla trascrittomica della glicoproteina superficiale cellulare utilizzando dati RNA-seq da oltre 1.000 linee cellulari tumorali. Le linee cellulari di Cell Model Passport27 sono state raggruppate utilizzando il clustering K-means secondo i valori FPKM di 1.500 dollari di lecoproteine della superficie cellulare. Le linee cellulari rappresentative di ogni gruppo sono etichettate. Il cluster 5 era interamente composto da linee cellulari di origine ematopoietica (vedi anche Tabella supplementare 2). Fare clic qui per visualizzare una versione più grande di questa figura.
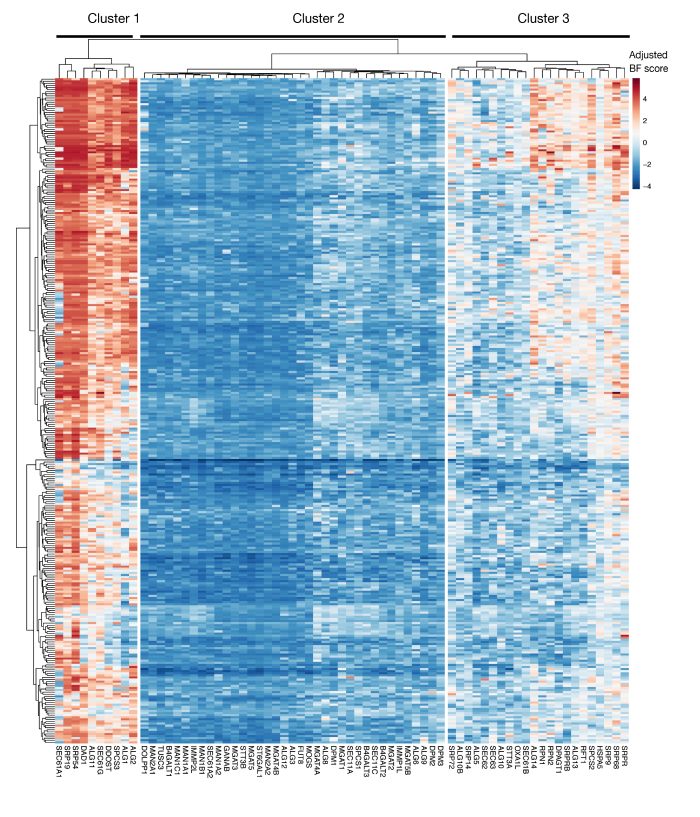
Figura supplementare S3: punteggi di essentialità per l'esportazione di proteine KEGG-annotation e geni di glicosilazione n-linked dai punteggi del progetto. I punteggi di eccellenza di Bayes rettificati per 330 linee cellulari (colonne, non etichettate) sono tracciati per i geni dell'esportazione delle proteine e del percorso di glicosilazione n (asse X). I punteggi superiori a 0 rappresentano un esaurimento significativo della popolazione mutante rispetto alla biblioteca originale del plasmide. I geni possono essere suddivisi in tre cluster distinti che rappresentano diversi livelli di essenzialità nelle linee cellulari. Questo clustering può essere utilizzato per decidere il giorno dell'ordinamento. Se lo schermo viene eseguito in un punto di tempo tardivo (giorno 16), è possibile che i geni noti per essere essenziali per le cellule (cluster 1 e 3) non saranno identificati. Fare clic qui per visualizzare una versione più grande di questa figura.
Supplementary Table 1: I file di conteggio grezzi per il software MAGeCK e i file MAGeCK hanno generato file gene_summary relativi agli schermi genetici rappresentativi. Fare clic qui per visualizzare questo file (fare clic con il pulsante destro del mouse per scaricare).
Tabella supplementare 2: Raggruppamento delle linee cellulari in base all'espressione dei recettori della superficie cellulare. Fare clic qui per visualizzare questo file (fare clic con il pulsante destro del mouse per scaricare).
Discussione
Viene descritta una strategia di screening basata su CRISPR per identificare i geni che codificano i componenti cellulari coinvolti nel riconoscimento cellulare. Un approccio simile che utilizza l'attivazione CRISPR fornisce anche un'alternativa genetica per identificare i recettori che interagiscono direttamente delle proteine ricombinanti senza la necessità di generare grandi librerie proteiche26. Tuttavia, uno dei principali vantaggi di questo approccio è che è applicabile alle interazioni mediate da molecole di superficie originariamente visualizzate sulla cellula e non dipende dalla sovraespressione dei recettori, che può influenzare l'avidità di legame del recettore. A differenza di altri metodi, quindi, questa tecnica non fa supposizioni per quanto riguarda la natura biochimica o la biologia cellulare dei recettori e offre l'opportunità di studiare le interazioni mediate da proteine normalmente difficili da studiare utilizzando approcci biochimici, come proteine molto grandi, o quelli che attraversano la membrana più volte o formano complessi con altre proteine e molecole diverse da proteine come glicani, glicolipidi e fosfoipi. Data la natura della scala genomica del metodo, questo approccio ha anche il vantaggio di identificare non solo il recettore, ma anche componenti cellulari aggiuntivi necessari per l'evento di legame, fornendo così approfondimenti sulla biologia cellulare del recettore.
Una delle principali limitazioni di questo metodo quando lo si utilizza per identificare il recettore di una proteina orfana è il requisito iniziale per identificare prima una linea cellulare che si lega alla proteina. Questo non è sempre facile e identificare una linea cellulare che visualizza un fenotipo vincolante che è anche permissivo agli schermi genetici può essere il passo che limita il tempo per l'implementazione di questo saggio. Alcune linee cellulari tendono a legarsi a più proteine rispetto ad altre. Ciò è particolarmente rilevante per le proteine che legano l'HS, perché queste proteine tendono a legarsi a qualsiasi linea cellulare che visualizza catene laterali HS, indipendentemente dal contesto di legame nativo. Inoltre, abbiamo osservato che l'upregulation dei syndecan (cioè i proteoglicani che contengono HS) nelle linee cellulari porta ad un maggiore legame delle proteine leganti HS26. Questo potrebbe essere un fattore da prendere in considerazione quando si seleziona la linea cellulare per lo screening. Tuttavia, anche importante notare è che il legame additivo di HS non interferisce con il legame a un recettore specifico. Ciò significa che se si osserva l'associazione, è possibile che sia mediata esclusivamente da HS perché l'associazione mediata da HS in questo saggio è additiva piuttosto che codipendente19. In uno scenario di questo tipo, l'approccio di blocco dell'eparina descritto può identificare tali comportamenti senza dover eseguire uno schermo genetico completo.
Una risorsa utile per la scelta delle linee cellulari è Cell Model Passport, che contiene informazioni sulla genomica, la trascrittomica e le condizioni di coltura per le linee cellulari tumorali da 1.000 dollari27. A seconda del contesto biologico, le cellule possono essere scelte in base ai loro profili di espressione. Per facilitare la selezione delle linee cellulari, abbiamo raggruppato 1.000 linee cellulari in Cell Model Passport in base all'espressione di 1.500 dollari di glicoproteine superficiali delle cellule umane preantate28 (Figura supplementare 2; le informazioni sul cluster per ogni linea cellulare insieme alle condizioni di crescita sono fornite nella tabella supplementare 2). Durante il test del legame di una proteina con funzione sconosciuta, è utile selezionare un pannello di linee cellulari rappresentative da ogni cluster per aumentare la possibilità di coprire una vasta gamma di recettori. Data una scelta, si consiglia di scegliere linee cellulari facili da coltura e facili da trasdurare. Poiché queste linee cellulari saranno utilizzate nello screening su scala del genoma, è preferibile che possano essere coltivate facilmente in grandi quantità e siano permissive alla trasduzione lentivirale, perché è il metodo più comunemente disponibile per la consegna di sgRNA per lo screening genetico basato su CRISPR nelle fasi successive.
Generalmente, le selezioni di fenotipi vengono effettuate in un unico tipo. Tuttavia, questo è determinato dalla luminosità della popolazione di cellule macchiate rispetto al controllo. I cicli iterativi delle selezioni potrebbero essere adottati per scenari in cui il rapporto segnale-rumore del fenotipo desiderato è basso, o quando lo scopo dello schermo è quello di identificare mutanti con fenotipi forti. Quando si utilizza un approccio di selezione iterativa per schermi basati su FACS, è importante considerare che il processo di ordinamento può causare la morte delle cellule, principalmente a causa della pura forza della selezionatrice. Pertanto, non tutte le celle raccolte saranno rappresentate nel prossimo ciclo di ordinamento.
La complessità della biblioteca è un fattore molto importante nell'esecuzione di schermi genetici di successo, soprattutto per schermi di selezione negativi perché l'entità dell'esaurimento in questi può essere determinata solo confrontando i risultati con quelli che erano presenti nella libreria di partenza. Per le schermate di selezione negativa, è comune mantenere librerie di complessità 500-1.000. Le schermate di selezione positive, tuttavia, sono più robuste alle dimensioni delle librerie, perché in tali schermi solo un piccolo numero di mutanti dovrebbe essere selezionato per un particolare fenotipo. Pertanto, nella schermata di selezione positiva descritta di seguito, le dimensioni della libreria possono essere ridotte a una complessità di 50-100 volte senza compromettere la qualità dello schermo. Inoltre, in queste schermate è anche possibile utilizzare una libreria di controlli per una determinata linea cellulare in un determinato giorno come "controllo generale" per tutti i campioni ordinati nel giorno per quella determinata linea cellulare. In questo modo si riduce il numero di librerie di controllo che devono essere prodotte e sequenziate.
Un'altra considerazione importante per l'utilizzo di questo approccio sono i limiti degli schermi di perdita di funzione nell'identificazione dei geni essenziali per la crescita delle cellule in vitro. La tempistica degli schermi è cruciale a questo proposito, poiché più a lungo le cellule mutanti sono mantenute in coltura, maggiore è la probabilità che le cellule con mutazioni nei geni essenziali diventino non vitali e non siano più rappresentate nella libreria dei mutanti. I recenti screening genetici eseguiti nell'ambito dell'iniziativa Project Score in oltre 300 linee cellulari mostrano che più geni nella secrezione proteica con annotato da KEGG e nel percorso di glicosilazione N sono spesso identificati come essenziali per un certo numero di linee cellulari (Supplementary Figure 3)29. Questo può essere preso in considerazione quando si progettano schermi se l'effetto dei geni necessari per la proliferazione e la vitalità deve essere studiato nel contesto del processo di riconoscimento cellulare. In questo caso, l'esecuzione di schermi in un momento precoce (ad esempio, giorno 9 posttraduzione) sarebbe generalmente appropriato. Tuttavia, se l'approccio viene utilizzato per identificare alcuni obiettivi con effetti di dimensioni forti anziché percorsi cellulari generali, potrebbe essere opportuno eseguire schermi in un momento successivo (ad esempio, giorno 15-16 posttrasduzione).
I risultati dello screening sono molto robusti; in otto schermi di legame proteico ricombinanti eseguiti in passato, il recettore della superficie cellulare è stato il colpo più alto in ogni caso19. Quando si utilizza questo approccio per identificare il partner di interazione, ci si dovrebbe quindi aspettare che il recettore e i fattori che contribuiscono alla sua presentazione sulla superficie siano identificati con un'elevata fiducia statistica. Una volta eseguito lo schermo e convalidato un colpo utilizzando un singolo knockout di gRNA, è possibile eseguire ulteriori follow-up utilizzando metodi biochimici esistenti come AVEXIS4 e legame saturable diretto di proteine purificate utilizzando la risonanza del plasmone superficiale. L'approccio qui descritto è applicabile a tutte le proteine per le quali è possibile generare una sonda di legame ricombinante solubile.
In sintesi, questo è un approccio knockout CRISPR su scala genomica per identificare le interazioni mediate dalle proteine della superficie cellulare. Questo metodo è generalmente applicabile per identificare le vie cellulari necessarie per il riconoscimento della superficie cellulare in una vasta gamma di contesti biologici diversi, tra cui le cellule proprie di un organismo (ad esempio, il riconoscimento neurale e immunologico), nonché tra le cellule ospiti e le proteine patogene. Questo metodo fornisce un'alternativa genetica agli approcci biochimici progettati per l'identificazione dei recettori, e poiché non richiede alcuna ipotesi preliminare per quanto riguarda la natura biochimica o la biologia cellulare dei recettori ha un grande potenziale per fare scoperte completamente inaspettate.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo lavoro è stato sostenuto dalla sovvenzione Wellcome Trust numero 206194 assegnata alla GJW. Ringraziamo la struttura Cytometry Core: Bee Ling Ng, Jennifer Graham, Sam Thompson e Christopher Hall per l'aiuto con FACS.
Materiali
| Name | Company | Catalog Number | Comments |
| Anti-mouse alkaline phosphatase | Sigma | A4656 | |
| Blasticidin | Chem-Cruz | SC-204655 | |
| Blood & Cell Culture DNA Maxi Kit | Qiagen | 13362 | |
| BSA | Sigma | A9647-100G | |
| Diethanolamine | Sigma | 398179 | |
| DMEM | Gibco | 31966-021 | |
| Dneasy Blood and Tissue kit | Qiagen | 69504 | |
| DynaMag-96 Side Magnet | Invitrogen | 12331D | |
| HEK293T packaging cells | ATCC | CRL-3216 | |
| Heparin | Sigma | H4784-1G | |
| KAPA HiFi HotStart ReadyMix | Kapa | KK2602 | |
| Lipofectamine LTX with PLUS reagent | Invitrogen | 15338100 | |
| MoFlo XDP cell sorter | BD | ||
| Ni2+-NTA agarose beads | Jena Bioscience | AC-501-25 | |
| OPTI-MEM | Life Technologies | 31985-070 | |
| OX-68 antibody | AbD Serotec | MCA1022R | |
| p-nitrophenyl phosphate | Sigma | 1040-506 | |
| PD-10 desalting columns | GE healthcare | 17085101 | |
| Polybrene | Millipore | TR-1003-G | |
| Polypropylene tubes with 5 mL bed volume | Qiagen | 34964 | |
| Proteinase K, recombinant, PCR Grade | Roche | 3115879001 | |
| Puromycin | Gibco | A11138-03 | |
| Q5 Hot Start High-Fidelity 2× Master Mix | NEB | M0494L | |
| QIAquick PCR purification kit | Qiagen | 28104 | |
| SCFA filter | Nalgene | 190-2545 | |
| Sony Cell sorter | Sony | ||
| SPRI beads (Agencourt AMPure XP beads) | Beckman | A63881 | |
| Streptavidin-coated microtitre plates | Nalgene | 734-1284 | |
| Streptavidin-PE | Biolegend | 405204 |
Riferimenti
- Wright, G. J. Signal initiation in biological systems: the properties and detection of transient extracellular protein interactions. Molecular bioSystems. 5 (12), 1405-1412 (2009).
- van der Merwe, P. A., Barclay, A. N. Transient intercellular adhesion: the importance of weak protein-protein interactions. Trends in Biochemical Sciences. 19 (9), 354-358 (1994).
- Wood, L., Wright, G. J. Approaches to identify extracellular receptor-ligand interactions. Current Opinion in Structural Biology. 56, 28-36 (2019).
- Bushell, K. M., Söllner, C., Schuster-Boeckler, B., Bateman, A., Wright, G. J. Large-scale screening for novel low-affinity extracellular protein interactions. Genome Research. 18 (4), 622-630 (2008).
- Visser, J. J., et al. An extracellular biochemical screen reveals that FLRTs and Unc5s mediate neuronal subtype recognition in the retina. eLife. 4, e08149(2015).
- Özkan, E., et al. An extracellular interactome of immunoglobulin and LRR proteins reveals receptor-ligand networks. Cell. 154 (1), 228-239 (2013).
- Martinez-Martin, N., et al. An Unbiased Screen for Human Cytomegalovirus Identifies Neuropilin-2 as a Central Viral Receptor. Cell. 174 (5), 1158-1171 (2018).
- Bianchi, E., Doe, B., Goulding, D., Wright, G. J. Juno is the egg Izumo receptor and is essential for mammalian fertilization. Nature. 508 (7497), 483-487 (2014).
- Mullican, S. E., et al. GFRAL is the receptor for GDF15 and the ligand promotes weight loss in mice and nonhuman primates. Nature Medicine. 23 (10), 1150-1157 (2017).
- Turner, L., et al. Severe malaria is associated with parasite binding to endothelial protein C receptor. Nature. 498 (7455), 502-505 (2013).
- Frei, A. P., et al. Direct identification of ligand-receptor interactions on living cells and tissues. Nature Biotechnology. 30 (10), 997-1001 (2012).
- Sobotzki, N., et al. HATRIC-based identification of receptors for orphan ligands. Nature Communications. 9 (1), 1519(2018).
- Sharma, S., Petsalaki, E. Application of CRISPR-Cas9 Based Genome-Wide Screening Approaches to Study Cellular Signalling Mechanisms. International Journal of Molecular Sciences. 19 (4), (2018).
- Gebre, M., Nomburg, J. L., Gewurz, B. E. CRISPR-Cas9 Genetic Analysis of Virus-Host Interactions. Viruses. 10 (2), (2018).
- Zotova, A., Zotov, I., Filatov, A., Mazurov, D. Determining antigen specificity of a monoclonal antibody using genome-scale CRISPR-Cas9 knockout library. Journal of Immunological Methods. 439, 8-14 (2016).
- Puschnik, A. S., Majzoub, K., Ooi, Y. S., Carette, J. E. A CRISPR toolbox to study virus-host interactions. Nature Reviews. Microbiology. 15 (6), 351-364 (2017).
- Kerr, J. S., Wright, G. J. Avidity-based extracellular interaction screening (AVEXIS) for the scalable detection of low-affinity extracellular receptor-ligand interactions. Journal of Visualized Experiments. (61), e3881(2012).
- Tzelepis, K., et al. A CRISPR Dropout Screen Identifies Genetic Vulnerabilities and Therapeutic Targets in Acute Myeloid Leukemia. Cell Reports. 17 (4), 1193-1205 (2016).
- Sharma, S., Bartholdson, S. J., Couch, A. C. M., Yusa, K., Wright, G. J. Genome-scale identification of cellular pathways required for cell surface recognition. Genome Research. 28 (9), 1372-1382 (2018).
- Wang, B., et al. Integrative analysis of pooled CRISPR genetic screens using MAGeCKFlute. Nature Protocols. 14 (3), 756-780 (2019).
- R Core team. A language and environment for statistical computing. , http://www.R-project.org (2013).
- Hart, T., et al. Evaluation and Design of Genome-Wide CRISPR/SpCas9 Knockout Screens. G3. 7 (8), 2719-2727 (2017).
- Kuleshov, M. V., et al. Enrichr: a comprehensive gene set enrichment analysis web server 2016 update. Nucleic Acids Research. 44 (W1), W90-W97 (2016).
- Crosnier, C., et al. Basigin is a receptor essential for erythrocyte invasion by Plasmodium falciparum. Nature. 480 (7378), 534-537 (2011).
- Kirk, P., et al. CD147 is tightly associated with lactate transporters MCT1 and MCT4 and facilitates their cell surface expression. The EMBO Journal. 19 (15), 3896-3904 (2000).
- Chong, Z. S., Ohnishi, S., Yusa, K., Wright, G. J. Pooled extracellular receptor-ligand interaction screening using CRISPR activation. Genome Biology. 19 (1), 205(2018).
- van der Meer, D., et al. Cell Model Passports-a hub for clinical, genetic and functional datasets of preclinical cancer models. Nucleic Acids Research. 47 (D1), D923-D929 (2019).
- Bausch-Fluck, D., et al. A mass spectrometric-derived cell surface protein atlas. PloS One. 10 (3), e0121314(2015).
- Behan, F. M., et al. Prioritization of cancer therapeutic targets using CRISPR-Cas9 screens. Nature. 568 (7753), 511-516 (2019).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon