需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
使用酶标仪提高细菌生长数据高通量定量的重现性和精度
摘要
这里提出了一种高通量方案来测量生长数据,包括生长曲线、生长速率和最大生长速率。该方案使用两种产生生物膜的细菌进行了验证和确认。本研究中应用的结果和方法可以扩展到使用酶标仪的其他高通量方案。
摘要
本研究旨在开发一种可重复、可靠、高通量的方案,以监测 96 孔板中的细菌生长并分析最大生长速率。测定 2 种细菌的生长曲线和最大生长速率。研究了包括 (i) 盖子冷凝、(ii) 光程校正、(iii) 接种大小、(iv) 采样时间间隔和 (v) 空间偏差在内的问题。通过三个独立的技术重复来评估方案的可重复性,运行之间的标准差为 0.03。芽 孢杆菌 和 苔原芽 孢杆菌的最大生长速率分别为 (平均值 ± SD) 0.99 h-1 ± 0.03 h-1 和 0.85 h-1 ± 0.025 h-1。这些细菌由于具有聚集在一起的亲和力,因此在光学监测中更具挑战性。这项研究证明了接种大小、光程校正、盖子升温、采样时间间隔和孔板空间偏差对于在酶标仪上获得可靠、准确和可重复的数据至关重要。开发的方案及其验证步骤可以扩展到使用酶标仪和高通量方案的其他方法,从而减少研究人员的先天误差和材料成本。
引言
对多组学作(包括细菌的机制和代谢研究)的兴趣日益浓厚,强调了高通量和自动化方法的重要性,例如记录生长数据 1,2。包含动力学参数(例如最大生长速率)的生长数据有助于表征细菌对不同物理、化学和抗菌条件的反应。生长速率数据是用于揭示潜在基因型-表型联系1 或指示食品的微生物安全性和保质期的标准响应变量 3,4。适应性实验室进化5,6,7、全基因组筛选、某些化学测定8(certain chemical assays)和各种正向遗传筛选9(forward genetic screening)等技术都依赖于生长速率来评估结果。
细菌培养物的光密度 (OD) 测量是监测细菌生长的标准微生物学方法。OD 测量值通常在 600 nm 的波长下记录,依赖于光散射和细胞密度10,11。比尔-朗伯定律解释了 OD 值与浓度(即细胞密度、细胞数量)、光程和吸收系数的相关性。分光光度计的几何形状和光学系统会影响 OD 读数11。传统的 OD 测量方法可能非常耗时耗力,并且数据可能会带有各种人为错误。在该方案中,使用酶标仪来减少分析时间12,13 和生物污染的机会。使用酶标仪进行高通量分析广泛应用于不同的微生物学领域,如筛选产生生物膜的细菌14,15、细菌生长抑制16、酵母细胞生长17、抗真菌药敏试验18 和纳米材料的毒性筛选19。
一些研究人员已经发布了使用酶标仪的细菌生长速率方案 12,20,21。然而,尚未完全建立检查所收集数据可靠性的全面协议。据报道,由于 96 孔板中的氧转移不足,物种类型 22,23,24 和密封胶带等因素会影响可重复性 25,26。Delaney 等人报道,当使用酶标仪时,生长培养基中出现了大簇的 Methylorubrum extorquens(野生型菌株),这导致了极其嘈杂的生长数据24。通过去除与生物膜产生相关的基因,解决了这个问题24。由于细胞外聚合物物质的分泌,产生生物膜的细菌具有更大的亲和力,可以聚结在一起并形成细胞簇。因此,使用光散射技术(例如,分光光度计和酶标仪)监测它们的生长更具挑战性。
该协议旨在建立使用酶标仪在高通量方法中获得可重现数据的步骤。使用 蕈样芽孢杆菌 和 苔 原芽孢杆菌是因为它们的快速生长和生物膜产生能力,这在手动和自动方法中传统上具有挑战性。研究了 (i) 光程校正、 (ii) 盖子上的冷凝、 (iii) 接种量、 (iv) 采样时间间隔和 (v) 空间偏差等因素,以评估数据的可靠性和可重复性。该协议提出了使用酶标仪准确监测细菌生长和测量特定生长速率的步骤。
研究方案
注意:必须在无菌条件下(即,在两个火焰或生物安全柜之间)遵循本协议中的所有步骤。所有材料和工具均高压灭菌 20 分钟。有关本协议中使用的所有材料、设备和软件的详细信息,请参阅 材料表 。戴手套的手进行消毒,用手部消毒剂或 70% 酒精溶液保持湿润至少 1 分钟,之后不要从安全柜中取出。否则,在将手放回安全柜之前,必须重复消毒程序。注意: 使用明火前,请确保消毒剂完全蒸发。
注意:如前所述27 从饮用水生物过滤中分离出两种细菌,因为它们能够产生生物膜。它们通过全 16S rRNA 测序鉴定并提交给 NCBI 作为 Bacillus mycoides (SAMN10518261) 和 Paenibacillus tundrae (SAMN10452279)。
1. 在甘油中制备细菌原液
- 称取 3 g 胰蛋白酶酱油汤 (TSB) 粉末,将其溶解在 100 mL 蒸馏水中。将肉汤高压灭菌 20 分钟,然后冷却至室温。向 50 mL 培养瓶中加入 50 mL TSB。
- 将细菌转移到琼脂平板27,28 中。使用环,从琼脂平板中挑选一个细菌菌落。轻轻旋转 Loop,将 Loop 的内容物添加到 Flask 中。将培养瓶在 30 °C ± 1 °C 下孵育过夜,并以 150 rpm 的转速进行轨道振荡。
注意:温度和摇晃需要根据应变类型进行调整。 - 用 47.5 mL TSB 准备另一个 50 mL 烧瓶。将 2.5 mL 过夜培养物转移到培养瓶中。孵育 5-6 小时以达到 OD600 = 0.6 0.05(以达到指数中期)。
- 将培养物转移至 50 mL 灭菌离心管中。将培养物以 2,200 × g 离心 10 分钟。轻轻倒出肉汤,以避免丢失收集在试管底部的任何细胞。
- 向同一管中加入 5 mL 磷酸盐缓冲盐水 (PBS),并以 2,200 × g 离心 10 分钟。重复此步骤 3 次。
- 通过使用移液管滴定足量的 PBS 溶液,将细胞的 OD600 调节至 0.6 0.05。准确记录添加的 PBS 量以备将来使用。
- 将甘油滴定至悬浮液中,以达到 20% v/v。盖上试管并涡旋 15 秒。
- 将 1.5 mL 悬浮液转移至 2 mL 冻存管中。将冻存管放入合适的盒子中,并储存在 −80 °C 的冰箱中。
2. 准备过夜培养物。
- 让含有细菌原液的冻存管在室温下解冻 30 分钟或直到内容物变为液体。
注意:建议不要加速解冻过程。 - 准备含有 10 mL TSB 的消毒管。将 50 μL 原液悬浮液转移到试管中。将试管在 30 °C ± 1 °C 下孵育过夜(16-18 小时),并以 150 rpm 的轨道振荡。
3. 准备接种物。
- 准备一根装有 10 mL 无菌 TSB 的试管。将 500 μL 过夜培养物转移到试管中。孵育 6-8 小时以达到 OD600 = 0.6 0.05(或达到指数中期)。
- 将培养物转移至 15 mL 离心管中。将试管以 2,200 × g 离心 10 分钟。轻轻倒出肉汤。
- 加入 5 mL PBS 并以 2,200 × g 离心 10 分钟。重复此步骤 3 次。
- 通过使用移液管滴定 PBS 溶液,将细胞的 OD600 调节至 0.6 0.05。准确记录添加的 PBS 量以备将来使用。
4. 将生长培养基转移到酶标仪中
- 选择透明、无菌、带盖的平底孔板(即 96 孔板、6 孔板、12 孔板)。
- 在运行方案之前,使用 DI 水测量微孔板的光程校正,如步骤 1.10 中所述。
- 根据所选孔板绘制参考表,以在加载前指定样品的位置。例如,为 96 孔板绘制一个 8 x 12 的表格。
注意:参考表避免了孵化和结果收集步骤中的混淆。 - 将以下物品放入生物安全柜中:带盖的微孔板、1,000 μL 和 200 μL 移液器、1,000 μL 和 200 μL 移液器吸头、几个冻存管、一个 25 mL 烧杯、生长培养基和接种物。
注意:上述物品应无菌转移到生物安全柜中。 - 向烧杯中加入 10 mL 生长培养基。将计算出的接种量转移到装有生长培养基的烧杯中。
注意:该方案中的接种比例为 1% 和 5%。例如,要达到 5% 和 1% 的接种比例,将 500 μL 和 100 μL 接种物分别添加到 9.5 mL 和 9.9 mL 生长培养基中。 - 加入接种物后,轻轻摇动烧杯以获得均匀分布。使用参考表将 200 μL 接种的培养基分配到指定的孔中。通过添加 200 μL 生长培养基(无接种物),选择至少三个孔作为对照。
注意:确保在参考表中清楚地标记了对照孔。如果在这些孔中观察到任何生长(污染),则应重复实验。 - 将 200 μL 去离子水分配到蒸发速率较高的边缘井中。从安全柜中取出 96 孔板之前,轻轻盖上盖子。小心地将 96 孔板放入读板器中,确保没有突然移动。
5. 微孔板检测仪设置
- 打开酶标仪,然后使用嵌入式软件创建自定义方案。在 Task Manager 中,打开 protocol 并选择 Create New |标准协议。打开 Procedure 并调整设置,如下所示:
- 使用 Set Temperature 将温度设置为 30 °C(或目标值)。
- 单击 Incubator On ,然后将 梯度 设置为 1 °C。
注:为避免因盖子上的冷凝而导致数据不可靠,请确保将 梯度 设置为 1 °C。 - 选择 Preheat(预热) 选项,确保沿孔板的热量分布均匀。
- 打开 Start Kinetic 并使用 运行时间 (例如,12:00:00 或 24:00:00 等 )和 00:30:00(代表 30 分钟)或 01:00:00(代表 1 小时)的读数间隔选择所需的孵育持续时间。
注:读取每个 96 孔板大约需要 1 分钟。 - 按如下方式设置 Shaking 设置:打开 Shake;将 Shake Mode 设置为 Linear;点击 Continuous Shake;,然后将 频率 调整为 567 cpm(或任何所需的值)。
- 按如下方式设置 OD 读数 设置:单击 Read |吸光度;选择 Endpoint/Kinetic 和 Monochromators;将 Wavelength (波长 ) 设置为 600 nm;,然后单击 验证 |Save 以将其另存为具有适当名称和日期的新协议。
6. 记录生长数据
- 打开 任务管理器 |立即阅读。
- 选择保存的协议,然后单击 OK 开始协议。
- 保存读数以进行数据分析。
7. 数据分析
- 将 OD600 结果(生长数据)转换为电子表格格式。在 结果 部分,单击每个孔 的图像选项以 观察最终的生长曲线。
- 打开电子表格文件,然后根据参考表重新排列记录的数据。从其他 reads 中减去空白 reads 的平均值。
注意:空白是时间零处的 OD 读数。在该协议中,考虑 OD600 值进行计算。如果 OD600 与细胞计数的校准曲线可用,则可以使用细胞密度。
8. 生长速率测定
- 在步骤 1.6 中创建的电子表格中使用传输的数据。使用下面描述的方法和公式 (1) 来评估 生长速率。
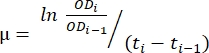 (1)
(1)
其中μ是生长速率,ODi 是每个时间点的 OD600,OD i-1 是最后一个时间点的初始 OD600 值,ti 和 ti-1 是两个提到的点之间的时间差(例如,0.5 表示 30 分钟的时间间隔,1 表示 1 小时的时间间隔)。 - 使用所需的数据分析软件计算五个连续生长速率的平均值和标准差。请注意,具有最低标准差的最大均值是 最大增长率 (μm) 12。
9. 确定空间偏差
注:微孔板检测仪和微孔板会在结果中引入偏差。评估特定微孔板读数仪中使用的板的空间偏差以确保结果的可靠性和可重复性至关重要。为此,请使用以下步骤:
- 重复步骤 1.2-1.6。
- 记录数据并根据步骤 1.7 和步骤 1.8 进行分析,以计算每个孔的生长速率。
- 创建热图以可视化 96 孔板中的生长速率变化。
- 执行统计分析(参见 补充表),确保孔之间没有显着差异。
注意:必须在所有孔中使用相同类型的细菌和相同的接种量。
10. OD 读数验证和光程校正因子
注意:确定光程校正因子对于验证数据并确保不同设备的可靠性和有效性至关重要。
- 使用步骤 1.2 和步骤 1.3 制备 B. mycoides 的过夜接种物。
- 准备三个灭菌的 50 mL 培养瓶,并向每个培养瓶中加入 49.5 mL 新鲜 TSB。将 500 μL 接种物无菌添加到安全柜中的每个培养瓶中。将 50 mL 未接种的 TSB 添加到灭菌的烧瓶中,该烧瓶将用作对照。
- 将烧瓶在 30 °C 和 150 rpm 下孵育。每 30 分钟,从每个烧瓶中无菌取样,并在分光光度计中读取和记录 OD600 。
- 将 9.9 mL TSB 添加到 25 mL 烧杯中。向烧杯中加入 100 μL 接种物并轻轻摇晃。
- 将烧杯中的 200 μL 混合物添加到 96 孔板中选定柱的 6 个孔中。将 200 μL 未接种的 TSB 添加到三个孔中作为对照。用 200 μL 去离子水填充边缘孔。
- 盖上盖子,轻轻地将 96 孔板转移到酶标仪上。按照步骤 1.5 和步骤 1.6 记录增长数据。
- 将酶标仪的 OD600 与分光光度计的 OD600 作图。请注意,斜率是两组读数之间的偏移量,称为 pathlength correction factor。
注意:培养箱和酶标仪孵育应在同一天同时进行。
结果
OD 读数验证和光程校正因子
在不同时间点采集 B. mycoides 培养物的分离样品,并使用酶标仪和分光光度计进行测量(图 1A)。采取此步骤是为了验证不同设备上的结果。OD600 数据相关但不匹配(图 1B)。相关性呈线性,斜率为 0.55 (95% 置信区间 [CI]: 0.53-0.58,R2 = 0.99),表明两种工具之间...
讨论
微孔板读板机可获得一致且可重复的生长速率。该技术可最大限度地减少人为错误并实现高通量采样。每个样品所需的少量培养物使该方法成为使用培养瓶或试管进行细胞计数的有吸引力的低成本替代方案。微孔板检测仪允许较大的样品量,从而提高统计功效,从而促进可靠的生长速率计算,同时保持较低的成本和劳动力。
本文介绍了测量和分析生?...
披露声明
作者没有需要声明的利益冲突。
致谢
这项工作由自然科学与工程研究委员会 (NSERC) / 哈利法克斯水务水务行业研究主席资助,水质和处理(批准号。IRCPJ 349838-16)。作者团队还要感谢 Anita Taylor 在审阅本文时提供的帮助。
材料
| Name | Company | Catalog Number | Comments |
| Centrifuge | Eppendorf | 5810 R | |
| Centrifuge tubes - 15 mL | ThermoFisher- Scientific | 339650 | Sterile |
| Centriguge tubes - 50 mL | ThermoFisher- Scientific | 339652 | Sterile |
| Disposable inoculating loop , 10 µL | Cole-Parmer | UZ-06231-08 | Sterile |
| Erlenmeyer flasks - 250 mL | Cole-Parmer | UZ-34502-59 | Glass |
| Isopropanol | ThermoFisher- Scientific | 396982500 | ≥99.0 |
| Phosphate Buffer Saline | Sigma-Aldrich | P4417 | |
| Pipett tips 1,000 µL | ThermoFisher- Scientific | UZ-25001-76 | |
| Pipett tips 10 mL | ThermoFisher- Scientific | UZ-25001-83 | |
| Pipett tips 200 µL | ThermoFisher- Scientific | UZ-25001-85 | |
| Pipett tips 5 mL | ThermoFisher- Scientific | UZ-25001-80 | |
| Pipettor 1,000 µL | Cole-Parmer | UZ-07909-11 | |
| Pipettor 10 mL | Cole-Parmer | UZ-07909-15 | |
| Pipettor 200 µL | Cole-Parmer | UZ-07909-09 | |
| Pipettor 5 mL | Cole-Parmer | UZ-07859-30 | |
| Tryptic Soy Broth | Millipore | 22091 | Suitable for microbiology |
参考文献
- Reuß, D. R., et al. Large-scale reduction of the Bacillus subtilis genome: Consequences for the transcriptional network, resource allocation, and metabolism. Genome Research. 27 (2), 289-299 (2017).
- Sparkes, A., et al. Towards Robot Scientists for autonomous scientific discovery. Automated Experimentation. 2 (1), (2010).
- Zwietering, M. H., Jongenburger, I., Rombouts, F. M., van t Riet, K. Modeling of the bacterial growth curve. Applied and Environmental Microbiology. 56 (6), 1875-1881 (1990).
- Pla, M., Oltra, S., Esteban, M., Andreu, S., Palop, A. Comparison of primary models to predict microbial growth by the plate count and absorbance methods. BioMed Research International. 2015 (6), 1-14 (2015).
- Choe, D., et al. Adaptive laboratory evolution of a genome-reduced Escherichia coli. Nature Communications. 10 (1), 935 (2019).
- Dykhuizen, D. E., Dean, A. M. Enzyme activity and fitness: Evolution in solution. Trends in Ecology and Evolution. 5 (8), 257-262 (1990).
- McDonald, M. J. Microbial experimental evolution - A proving ground for evolutionary theory and a tool for discovery. EMBO Reports. 20 (8), 1-14 (2019).
- Lum, P. Y., et al. Discovering modes of action for therapeutic compounds using a genome-wide screen of yeast heterozygotes. Cell. 116 (1), 121-137 (2004).
- Cagnon, C., et al. Development of a forward genetic screen to isolate oil mutants in the green microalga Chlamydomonas reinhardtii. Biotechnology for Biofuels. 6 (1), 178 (2013).
- Stevenson, K., McVey, A. F., Clark, I. B. N., Swain, P. S., Pilizota, T. General calibration of microbial growth in microplate readers. Scientific Reports. 6 (1), 38828 (2016).
- Matlock, B. C., Beringer, R. W., Ash, D. L., Page, A. F., Allen, M. W. Differences in Bacterial Optical Density Measurements between Spectrophotometers. Technical Note. ThermoScientific. , (2011).
- Hall, B. G., Acar, H., Nandipati, A., Barlow, M. Growth rates made easy. Molecular Biology and Evolution. 31 (1), 232-238 (2014).
- Rolfe, M. D., et al. Lag phase is a distinct growth phase that prepares bacteria for exponential growth and involves transient metal accumulation. Journal of Bacteriology. 194 (3), 686-701 (2012).
- Djordjevic, D., Wiedmann, M., Mclandsborough, L. A. Microtiter plate assay for assessment of Listeria monocytogenes biofilm formation. Applied and Environmental Microbiology. 68 (6), 2950-2958 (2002).
- O'Toole, G. A. Microtiter dish biofilm formation assay. Journal of Visualized Experiments. (47), e2437 (2011).
- Campbell, J. High-throughput assessment of bacterial growth inhibition by optical density measurements. Current Protocols in Chemical Biology. 3 (3), 1-20 (2012).
- Toussaint, M., Conconi, A. High-throughput and sensitive assay to measure yeast cell growth: A bench protocol for testing genotoxic agents. Nature Protocols. 1 (4), 1922-1928 (2006).
- Goughenour, K. D., Balada-Llasat, J. -. M., Rappleye, C. A. Quantitative microplate-based growth assay for determination of antifungal susceptibility of Histoplasma capsulatum yeasts. Journal of Clinical Microbiology. 53 (10), 3286-3295 (2015).
- Qiu, T. A., et al. Growth-based bacterial viability assay for interference-free and high-throughput toxicity screening of nanomaterials. Analytical Chemistry. 89 (3), 2057-2064 (2017).
- Kurokawa, M., Precise Ying, B. -. W. Precise, high-throughput analysis of bacterial growth. Journal of Visualized Experiments. (127), e56197 (2017).
- Bredit, F., Romick, T. L., Fleming, H. P. A rapid method for determination of bacterial growth kinetics. Journal of Rapid Methods and Automation in Microbiology. 3, 59-68 (1994).
- Delaney, N. F., et al. Development of an optimized medium, strain and high-throughput culturing methods for Methylobacterium extorquens. PLOS ONE. 8 (4), 62957 (2013).
- Haire, T. C., et al. Robust microplate-based methods for culturing and in vivo phenotypic screening of Chlamydomonas reinhardtii. Frontiers in Plant Science. 9, 235 (2018).
- McBirney, S. E., Trinh, K., Wong-Beringer, A., Armani, A. M. Wavelength-normalized spectroscopic analysis of Staphylococcus aureus and Pseudomonas aeruginosa growth rates. Biomedical Optics Express. 7 (10), 4034 (2016).
- Sieben, M., Giese, H., Grosch, J. H., Kauffmann, K., Büchs, J. Permeability of currently available microtiter plate sealing tapes fail to fulfil the requirements for aerobic microbial cultivation. Biotechnology Journal. 11 (12), 1525-1538 (2016).
- Zimmermann, H. F., John, G. T., Trauthwein, H., Dingerdissen, U., Huthmacher, K. Rapid evaluation of oxygen and water permeation through microplate sealing tapes. Biotechnology Progress. 19 (3), 1061-1063 (2003).
- Abkar, L., Gagnon, G. A. Biological responses to P-limitation in indigenous bacteria isolated from drinking water. AWWA Water Science. 3 (5), 1248 (2021).
- Sanders, E. R. Aseptic laboratory techniques: Plating methods. Journal of Visualized Experiments. (63), e3064 (2012).
- Hart, S. F. M., Skelding, D., Waite, A. J., Burton, J. C., Shou, W. High-throughput quantification of microbial birth and death dynamics using fluorescence microscopy. Quantitative Biology. 7 (1), 69-81 (2019).
- Periago, P. M., Abee, T., Wouters, J. A. Analysis of the heat-adaptive response of psychrotrophic Bacillus weihenstephanensis. International Journal of Food Microbiology. 79 (1-2), 17-26 (2002).
- Mira, P., Barlow, M., Meza, J. C., Hall, B. G. Statistical package for growth rates made easy. Molecular Biology and Evolution. 34 (12), 3303-3309 (2017).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。