È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Maggiore riproducibilità e precisione della quantificazione ad alto rendimento dei dati di crescita batterica utilizzando un lettore di micropiastre
In questo articolo
Riepilogo
Qui viene presentato un protocollo ad alto rendimento per misurare i dati di crescita, comprese le curve di crescita, il tasso di crescita e il tasso di crescita massimo. Il protocollo è stato verificato e convalidato utilizzando due batteri produttori di biofilm. I risultati e l'approccio applicati in questo studio possono essere estesi ad altri protocolli ad alto rendimento che utilizzano lettori di micropiastre.
Abstract
Questo studio mirava a sviluppare un protocollo ripetibile, affidabile e ad alto rendimento per monitorare la crescita batterica in piastre a 96 pozzetti e analizzare il tasso di crescita massimo. Sono state determinate le curve di crescita e i tassi di crescita massimi di due specie batteriche. Sono stati studiati problemi tra cui (i) condensazione del coperchio, (ii) correzione della lunghezza del percorso, (iii) dimensione dell'inoculazione, (iv) intervallo di tempo di campionamento e (v) distorsione spaziale. La ripetibilità del protocollo è stata valutata con tre repliche tecniche indipendenti, con una deviazione standard di 0,03 tra le esecuzioni. I tassi massimi di crescita di Bacillus mycoides e Paenibacillus tundrae sono stati determinati rispettivamente in (media ± DS) 0,99 h-1 ± 0,03 h-1 e 0,85 h-1 ± 0,025 h-1. Questi batteri sono più difficili da monitorare otticamente a causa della loro affinità a raggrupparsi. Questo studio dimostra l'importanza critica delle dimensioni dell'inoculazione, della correzione della lunghezza del percorso, del riscaldamento del coperchio, degli intervalli di tempo di campionamento e della distorsione spaziale della piastra per ottenere dati affidabili, accurati e riproducibili sui lettori di micropiastre. Il protocollo sviluppato e le sue fasi di verifica possono essere estesi ad altri metodi utilizzando lettori di micropiastre e protocolli ad alta produttività, riducendo gli errori innati dei ricercatori e i costi dei materiali.
Introduzione
Lo sviluppo dell'interesse per la manipolazione multi-omica, compresi gli studi sui meccanismi e sul metabolismo dei batteri, sottolinea l'importanza di metodi automatizzati e ad alto rendimento come la registrazione dei dati di crescita 1,2. I dati di crescita che comprendono parametri cinetici, come i tassi di crescita massimi, possono aiutare a caratterizzare le risposte batteriche a diverse condizioni fisiche, chimiche e antibatteriche. I dati sul tasso di crescita sono una variabile di risposta standard utilizzata per scoprire potenziali collegamenti genotipo-fenotipo1 o indicare la sicurezza microbica e la durata di conservazione dei prodotti alimentari 3,4. Tecniche come l'evoluzione adattiva in laboratorio 5,6,7, lo screening dell'intero genoma, alcuni saggi chimici8 e vari screening genetici in avanti9 si basano sui tassi di crescita per valutare i risultati.
Le misure di densità ottica (OD) delle colture batteriche sono un metodo microbiologico standard per monitorare la crescita batterica. Le misure OD sono spesso registrate a una lunghezza d'onda di 600 nm, basandosi sulla diffusione della luce e sulla densità delle celle10,11. La legge di Beer-Lambert spiega la dipendenza dei valori OD dalla concentrazione (cioè densità cellulare, numero di cellule), lunghezza del percorso e coefficiente di assorbività. La geometria e il sistema ottico di uno spettrofotometro influenzano le letture OD11. I metodi classici di misurazione OD possono richiedere molto tempo e lavoro e i dati possono comportare una serie di errori umani. In questo protocollo, viene utilizzato un lettore di micropiastre per ridurre il tempo dell'analista12,13 e la possibilità di contaminazione biologica. L'analisi ad alto rendimento con lettori di micropiastre è ampiamente applicata in diverse aree della microbiologia, come lo screening dei batteri produttori di biofilm14,15, l'inibizione della crescita batterica16, la crescita delle cellule di lievito17, la determinazione della suscettibilità antifungina18 e lo screening della tossicità dei nanomateriali19.
Alcuni ricercatori hanno pubblicato protocolli sul tasso di crescita batterica utilizzando un lettore di micropiastre 12,20,21. Tuttavia, non è stato completamente stabilito un protocollo approfondito che esamini l'affidabilità dei dati raccolti. È stato riportato che fattori come il tipo di specie 22,23,24 e i nastri sigillanti influiscono sulla ripetibilità a causa dell'inadeguatezza del trasferimento di ossigeno in una piastra a 96 pozzetti 25,26. Delaney et al. hanno riportato grandi ammassi di Methylorubrum extorquens (ceppo wild-type) nel terreno di crescita quando utilizzavano un lettore di micropiastre, il che causava dati di crescita estremamente rumorosi24. Il problema è stato risolto rimuovendo i geni associati alla produzione di biofilm24. A causa della secrezione di sostanze polimeriche extracellulari, i batteri produttori di biofilm hanno una maggiore affinità a fondersi insieme e creare cluster cellulari. Pertanto, è più difficile monitorare la loro crescita utilizzando tecniche di diffusione della luce (ad esempio, spettrofotometri e lettori di micropiastre).
Questo protocollo mira a stabilire i passaggi per ottenere dati riproducibili in un metodo ad alta produttività utilizzando un lettore di micropiastre. Bacillus mycoides e Paenibacillus tundrae sono stati utilizzati per la loro rapida crescita e capacità di produrre biofilm, che sono tradizionalmente difficili negli approcci manuali e automatizzati. Fattori come (i) la correzione della lunghezza del percorso, (ii) la condensa sul coperchio, (iii) la dimensione dell'inoculo, (iv) l'intervallo di tempo di campionamento e (v) la distorsione spaziale sono stati studiati per valutare l'affidabilità e la riproducibilità dei dati. Questo protocollo presenta i passaggi per monitorare accuratamente la crescita batterica e misurare tassi di crescita specifici utilizzando un lettore di micropiastre.
Protocollo
NOTA: Tutti i passaggi di questo protocollo devono essere seguiti in condizioni sterili (ad esempio, tra due fiamme o una cappa di biosicurezza). Tutti i materiali e gli strumenti vengono sterilizzati in autoclave per 20 minuti. Consulta la Tabella dei materiali per i dettagli su tutti i materiali, le attrezzature e il software utilizzati in questo protocollo. Le mani guantate vengono disinfettate, mantenute bagnate con disinfettante per le mani o soluzione alcolica al 70% per almeno 1 minuto e successivamente non rimosse dall'armadio di sicurezza. In caso contrario, la procedura di disinfezione deve essere ripetuta prima di reinserire le mani nell'armadio di sicurezza. ATTENZIONE: Assicurarsi che il disinfettante sia completamente evaporato prima di utilizzare una fiamma libera.
NOTA: Due batteri sono stati isolati dalla biofiltrazione dell'acqua potabile, come spiegato in precedenza27 per la loro capacità di produrre biofilm. Sono stati identificati mediante il sequenziamento completo dell'rRNA 16S e presentati all'NCBI come Bacillus mycoides (SAMN10518261) e Paenibacillus tundrae (SAMN10452279).
1. Preparazione del brodo batterico in glicerolo
- Pesare 3 g di brodo di soia triptico (TSB) in polvere e scioglierlo in 100 ml di acqua distillata. Sterilizzare in autoclave il brodo per 20 minuti e lasciarlo raffreddare a temperatura ambiente. Aggiungere 50 ml di TSB in un matraccio da 50 ml.
- Trasferire i batteri su piastre di agar27,28. Usando un anello, raccogli una colonia di batteri dalla piastra di agar. Aggiungere il contenuto dell'anello nel pallone facendo roteare delicatamente l'anello. Incubare il matraccio per una notte a 30 °C ± 1 °C con agitazione orbitale a 150 giri/min.
NOTA: La temperatura e l'agitazione devono essere regolate in base al tipo di deformazione. - Preparare un altro pallone da 50 ml con 47,5 ml di TSB. Trasferire 2,5 ml di coltura notturna nel pallone. Incubarlo per 5-6 ore per raggiungere OD600 = 0,6 0,05 (per arrivare alla fase esponenziale media).
- Trasferire la coltura in una provetta da centrifuga sterilizzata da 50 mL. Centrifugare la coltura a 2.200 × g per 10 minuti. Decantare delicatamente il brodo per evitare di perdere le cellule raccolte sul fondo del tubo.
- Aggiungere 5 mL di soluzione salina tamponata con fosfato (PBS) alla stessa provetta e centrifugare a 2.200 × g per 10 minuti. Ripeti questo passaggio 3 volte.
- Regolare il diametro esterno600 delle cellule a 0,6 0,05 utilizzando una pipetta per titolare una quantità adeguata di soluzione di PBS. Registra con precisione la quantità aggiunta di PBS per un uso futuro.
- Titolare il glicerolo alla sospensione fino a raggiungere il 20% v/v. Tappare il tubo e vorticare per 15 s.
- Trasferire 1,5 mL della sospensione in 2 mL di crioviali. Mettere i crioviali in un'apposita scatola e conservarli in congelatore a -80 °C.
2. Prepara la cultura durante la notte.
- Lasciare scongelare i crioviali con il ceppo batterico a temperatura ambiente per 30 minuti o fino a quando il contenuto non sarà liquido.
NOTA: Si consiglia di non accelerare il processo di scongelamento. - Preparare una provetta sterilizzata con 10 ml di TSB. Trasferire 50 μl della sospensione stock nel tubo. Incubare la provetta per una notte (16-18 h) a 30 °C ± 1 °C con agitazione orbitale a 150 giri/min.
3. Preparare l'inoculo.
- Preparare una provetta con 10 ml di TSB sterilizzato. Trasferire 500 μl della coltura notturna nella provetta. Incubarlo per 6-8 ore per raggiungere OD600 = 0,6 0,05 (o arrivare alla fase esponenziale media).
- Trasferire la coltura in una provetta da centrifuga da 15 mL. Centrifugare la provetta a 2.200 × g per 10 min. Decantare delicatamente il brodo.
- Aggiungere 5 mL di PBS e centrifugare a 2.200 × g per 10 min. Ripeti questo passaggio 3 volte.
- Regolare il diametro esterno600 delle cellule a 0,6 0,05 titolando la soluzione di PBS con una pipetta. Registra con precisione la quantità aggiunta di PBS per un uso futuro.
4. Trasferimento del terreno di coltura al lettore di micropiastre
- Scegliere una piastra a pozzetti trasparente, sterile, a fondo piatto con coperchio (ad esempio, piastra a 96 pozzetti, piastra a 6 pozzetti, piastra a 12 pozzetti).
- Misurare la correzione della lunghezza del percorso per la micropiastra utilizzando acqua deionizzata prima di eseguire il protocollo, come spiegato nel passaggio 1.10.
- Disegnare una tabella di riferimento basata sulla piastra a pozzetti selezionata per specificare le posizioni dei campioni prima del caricamento. Ad esempio, disegnare una tabella 8 x 12 per una piastra a 96 pozzetti.
NOTA: La tabella di riferimento evita confusione durante le fasi di incubazione e raccolta dei risultati. - Collocare i seguenti elementi in una cappa di biosicurezza: una micropiastra con coperchio, pipettor da 1.000 μl e 200 μl, puntali per pipette da 1.000 μl e 200 μl, diversi crioviali, un becher da 25 mL, il terreno di coltura e l'inoculo.
NOTA: Gli elementi menzionati devono essere trasferiti nella cabina di biosicurezza in modo asettico. - Aggiungere 10 mL di terreno di coltura al becher. Trasferire la quantità calcolata di inoculo nel becher con il terreno di coltura.
NOTA: I rapporti di inoculo in questo protocollo sono dell'1% e del 5%. Ad esempio, per ottenere un rapporto di inoculo del 5% e dell'1%, aggiungere 500 μl e 100 μl di inoculo rispettivamente a 9,5 mL e 9,9 mL di terreno di crescita. - Dopo aver aggiunto l'inoculo, agitare delicatamente il becher per ottenere una distribuzione uniforme. Erogare 200 μl del terreno inoculato nei pozzetti designati utilizzando la tabella di riferimento. Scegliere almeno tre pozzetti che fungano da controlli aggiungendo 200 μl di terreno di coltura (senza inoculo).
NOTA: Assicurarsi che i pozzetti di controllo siano chiaramente contrassegnati nella tabella di riferimento. Se si osserva una crescita (contaminazione) in questi pozzetti, l'esperimento deve essere ripetuto. - Erogare 200 μL di acqua deionizzata nei pozzetti del bordo dove la velocità di evaporazione è più elevata. Posizionare delicatamente il coperchio prima di rimuovere la piastra a 96 pozzetti dall'armadio di sicurezza. Posizionare con cura la piastra a 96 pozzetti nel lettore di piastre, assicurandosi che non ci siano movimenti bruschi.
5. Impostazioni del lettore di micropiastre
- Accendere il lettore di micropiastre e quindi utilizzare il software incorporato per creare il protocollo personalizzato. In Task Manager, apri il protocollo e scegli Crea nuovo | Protocollo standard. Aprire Procedura e regolare le impostazioni come segue:
- Impostare la temperatura a 30 °C (o i valori desiderati) utilizzando Set Temperature.
- Fare clic su Incubatore acceso e quindi impostare il gradiente a 1 °C.
NOTA: Per evitare dati inaffidabili dovuti alla condensa sul coperchio, assicurarsi che il gradiente sia impostato su 1 °C. - Scegliere l'opzione Preriscaldamento , che garantisce una distribuzione uniforme del calore lungo la piastra a pozzetti.
- Aprire Start Kinetic e scegliere la durata dell'incubazione desiderata utilizzando il tempo di esecuzione (ad esempio, 12:00:00 o 24:00:00, ecc.) e l'intervallo tra le letture per 00:30:00 (rappresenta 30 min) o 01:00:00 (rappresenta 1 ora).
NOTA: La lettura di ciascuna piastra a 96 pozzetti richiede circa 1 minuto. - Impostare le impostazioni di agitazione come segue: aprire Agitare; impostare la modalità di agitazione su Lineare; fare clic su Agitazione continua; e regolare la frequenza su 567 cpm (o qualsiasi valore desiderato).
- Impostare le impostazioni di lettura OD come segue: fare clic su Leggi | Assorbanza; selezionare Endpoint/Cinetici e Monocromatori; impostare la lunghezza d'onda a 600 nm; e fai clic su Convalida | Salva per salvarlo come nuovo protocollo con il nome e la data appropriati.
6. Registrazione dei dati di crescita
- Apri Task Manager | Leggi ora.
- Scegli il protocollo salvato, quindi fai clic su OK per iniziare il protocollo.
- Salva le letture per l'analisi dei dati.
7. Analisi dei dati
- Trasferisci i risultati OD600 (dati di crescita) in formato foglio di calcolo. Nella sezione dei risultati , fare clic sull'opzione dell'immagine per ciascun pozzetto per osservare le curve di crescita finali.
- Apri il file del foglio di calcolo e riordina i dati registrati in base alla tabella di riferimento. Sottrarre il valore medio delle letture vuote dalle altre letture.
NOTA: Gli spazi vuoti sono le letture OD al tempo zero. In questo protocollo, per il calcolo vengono considerati valori OD600 . La densità delle celle può essere utilizzata se è disponibile la curva di calibrazione per OD600 rispetto al numero di celle.
8. Determinazione del tasso di crescita
- Utilizzare i dati trasferiti nel foglio di calcolo creato nel passaggio 1.6. Utilizzare il metodo descritto di seguito e l'equazione (1) per valutare il tasso di crescita.
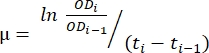 (1)
(1)
dove μ è il tasso di crescita, ODi è l'OD600 in ogni punto temporale, ODi-1 è il valore iniziale di OD600 nell'ultimo punto temporale, ti e ti-1 sono le differenze di tempo tra i due punti menzionati (ad esempio, 0,5 per intervalli di tempo di 30 minuti, 1 per intervalli di tempo di 1 ora). - Calcola la media e la deviazione standard di cinque tassi di crescita consecutivi utilizzando il software di analisi dei dati desiderato. Si noti che la media più grande con la deviazione standard più bassa è il tasso di crescita massimo (μm) 12.
9. Determinazione della distorsione spaziale
NOTA: I lettori di micropiastre e le piastre introducono distorsioni nei risultati. È fondamentale valutare le distorsioni spaziali delle piastre utilizzate in uno specifico lettore di micropiastre per garantire l'affidabilità e la riproducibilità dei risultati. Per raggiungere questo obiettivo, attenersi alla seguente procedura:
- Ripetere i passaggi 1.2-1.6.
- Registrare i dati e analizzarli secondo i passaggi 1.7 e 1.8 per calcolare il tasso di crescita per ciascun pozzo.
- Crea una mappa di calore per visualizzare le variazioni del tasso di crescita nella piastra a 96 pozzetti.
- Eseguire l'analisi statistica (vedi Tabelle Supplementari), assicurandosi che non ci siano differenze significative tra i pozzetti.
NOTA: È essenziale utilizzare lo stesso tipo di batteri e la stessa dimensione dell'inoculo in tutti i pozzetti.
10. Validazione della lettura OD e fattore di correzione della lunghezza del percorso
NOTA: Determinare il fattore di correzione della lunghezza del percorso è fondamentale per convalidare i dati e garantire l'affidabilità e la validità su diversi dispositivi.
- Utilizzare i passaggi 1.2 e 1.3 per preparare l'inoculo notturno di B. mycoides.
- Preparare tre fiasche sterilizzate da 50 ml e aggiungere 49,5 ml di cucchiaino fresco a ciascuna fiaschetta. Aggiungere asetticamente 500 μl di inoculo a ciascun pallone in una camera di sicurezza. Aggiungere 50 ml di TSB non inoculato a un pallone sterilizzato, che servirà come controllo.
- Incubare i matracci a 30 °C e 150 giri/min. Ogni 30 minuti, prelevare campioni da ciascun pallone in modo asettico e leggere e registrare OD600 nello spettrofotometro.
- Aggiungere 9,9 mL di TSB a un becher da 25 mL. Aggiungere 100 μl di inoculo al becher e agitare delicatamente.
- Aggiungere 200 μl della miscela nel becher ai sei pozzetti di una colonna selezionata in una piastra a 96 pozzetti. Aggiungere 200 μl di TSB non inoculato a tre pozzetti come controllo. Riempire i pozzetti del bordo con 200 μL di acqua deionizzata.
- Posizionare il coperchio e trasferire delicatamente la piastra a 96 pozzetti nel lettore di micropiastre. Segui i passaggi 1.5 e 1.6 per registrare i dati di crescita.
- Tracciare l'OD600 dal lettore di micropiastre rispetto all'OD600 dallo spettrofotometro. Si noti che la pendenza è l'offset tra le due serie di letture, chiamato fattore di correzione della lunghezza del percorso.
NOTA: L'incubazione dell'incubatore e del lettore di micropiastre deve essere eseguita contemporaneamente lo stesso giorno.
Risultati
Validazione della lettura OD e fattore di correzione della lunghezza del percorso
Campioni divisi di coltura di B. mycoides sono stati prelevati in diversi punti temporali e misurati utilizzando il lettore di micropiastre e lo spettrofotometro (Figura 1A). Questo passaggio è stato eseguito per convalidare i risultati su diversi dispositivi. I dati di OD600 erano correlati ma non corrispondevano (F...
Discussione
I lettori di micropiastre consentono di ottenere tassi di crescita costanti e ripetibili. Questa tecnologia riduce al minimo l'errore umano e consente il campionamento ad alto rendimento. La piccola quantità di coltura richiesta per campione rende questo approccio un'alternativa interessante e a basso costo alla conta cellulare con fiasche o provette. I lettori di micropiastre consentono di ottenere campioni di grandi dimensioni, aumentando la potenza statistica e facilitando successiva...
Divulgazioni
Gli autori non hanno conflitti di interesse da dichiarare.
Riconoscimenti
Questo lavoro è stato finanziato dal Natural Sciences and Engineering Research Council (NSERC) / Halifax Water Industrial Research Chair in Water Quality and Treatment (Grant No. IRCPJ 349838-16). Il team di autori desidera anche ringraziare l'aiuto di Anita Taylor nella revisione di questo articolo.
Materiali
| Name | Company | Catalog Number | Comments |
| Centrifuge | Eppendorf | 5810 R | |
| Centrifuge tubes - 15 mL | ThermoFisher- Scientific | 339650 | Sterile |
| Centriguge tubes - 50 mL | ThermoFisher- Scientific | 339652 | Sterile |
| Disposable inoculating loop , 10 µL | Cole-Parmer | UZ-06231-08 | Sterile |
| Erlenmeyer flasks - 250 mL | Cole-Parmer | UZ-34502-59 | Glass |
| Isopropanol | ThermoFisher- Scientific | 396982500 | ≥99.0 |
| Phosphate Buffer Saline | Sigma-Aldrich | P4417 | |
| Pipett tips 1,000 µL | ThermoFisher- Scientific | UZ-25001-76 | |
| Pipett tips 10 mL | ThermoFisher- Scientific | UZ-25001-83 | |
| Pipett tips 200 µL | ThermoFisher- Scientific | UZ-25001-85 | |
| Pipett tips 5 mL | ThermoFisher- Scientific | UZ-25001-80 | |
| Pipettor 1,000 µL | Cole-Parmer | UZ-07909-11 | |
| Pipettor 10 mL | Cole-Parmer | UZ-07909-15 | |
| Pipettor 200 µL | Cole-Parmer | UZ-07909-09 | |
| Pipettor 5 mL | Cole-Parmer | UZ-07859-30 | |
| Tryptic Soy Broth | Millipore | 22091 | Suitable for microbiology |
Riferimenti
- Reuß, D. R., et al. Large-scale reduction of the Bacillus subtilis genome: Consequences for the transcriptional network, resource allocation, and metabolism. Genome Research. 27 (2), 289-299 (2017).
- Sparkes, A., et al. Towards Robot Scientists for autonomous scientific discovery. Automated Experimentation. 2 (1), (2010).
- Zwietering, M. H., Jongenburger, I., Rombouts, F. M., van t Riet, K. Modeling of the bacterial growth curve. Applied and Environmental Microbiology. 56 (6), 1875-1881 (1990).
- Pla, M., Oltra, S., Esteban, M., Andreu, S., Palop, A. Comparison of primary models to predict microbial growth by the plate count and absorbance methods. BioMed Research International. 2015 (6), 1-14 (2015).
- Choe, D., et al. Adaptive laboratory evolution of a genome-reduced Escherichia coli. Nature Communications. 10 (1), 935 (2019).
- Dykhuizen, D. E., Dean, A. M. Enzyme activity and fitness: Evolution in solution. Trends in Ecology and Evolution. 5 (8), 257-262 (1990).
- McDonald, M. J. Microbial experimental evolution - A proving ground for evolutionary theory and a tool for discovery. EMBO Reports. 20 (8), 1-14 (2019).
- Lum, P. Y., et al. Discovering modes of action for therapeutic compounds using a genome-wide screen of yeast heterozygotes. Cell. 116 (1), 121-137 (2004).
- Cagnon, C., et al. Development of a forward genetic screen to isolate oil mutants in the green microalga Chlamydomonas reinhardtii. Biotechnology for Biofuels. 6 (1), 178 (2013).
- Stevenson, K., McVey, A. F., Clark, I. B. N., Swain, P. S., Pilizota, T. General calibration of microbial growth in microplate readers. Scientific Reports. 6 (1), 38828 (2016).
- Matlock, B. C., Beringer, R. W., Ash, D. L., Page, A. F., Allen, M. W. Differences in Bacterial Optical Density Measurements between Spectrophotometers. Technical Note. ThermoScientific. , (2011).
- Hall, B. G., Acar, H., Nandipati, A., Barlow, M. Growth rates made easy. Molecular Biology and Evolution. 31 (1), 232-238 (2014).
- Rolfe, M. D., et al. Lag phase is a distinct growth phase that prepares bacteria for exponential growth and involves transient metal accumulation. Journal of Bacteriology. 194 (3), 686-701 (2012).
- Djordjevic, D., Wiedmann, M., Mclandsborough, L. A. Microtiter plate assay for assessment of Listeria monocytogenes biofilm formation. Applied and Environmental Microbiology. 68 (6), 2950-2958 (2002).
- O'Toole, G. A. Microtiter dish biofilm formation assay. Journal of Visualized Experiments. (47), e2437 (2011).
- Campbell, J. High-throughput assessment of bacterial growth inhibition by optical density measurements. Current Protocols in Chemical Biology. 3 (3), 1-20 (2012).
- Toussaint, M., Conconi, A. High-throughput and sensitive assay to measure yeast cell growth: A bench protocol for testing genotoxic agents. Nature Protocols. 1 (4), 1922-1928 (2006).
- Goughenour, K. D., Balada-Llasat, J. -. M., Rappleye, C. A. Quantitative microplate-based growth assay for determination of antifungal susceptibility of Histoplasma capsulatum yeasts. Journal of Clinical Microbiology. 53 (10), 3286-3295 (2015).
- Qiu, T. A., et al. Growth-based bacterial viability assay for interference-free and high-throughput toxicity screening of nanomaterials. Analytical Chemistry. 89 (3), 2057-2064 (2017).
- Kurokawa, M., Precise Ying, B. -. W. Precise, high-throughput analysis of bacterial growth. Journal of Visualized Experiments. (127), e56197 (2017).
- Bredit, F., Romick, T. L., Fleming, H. P. A rapid method for determination of bacterial growth kinetics. Journal of Rapid Methods and Automation in Microbiology. 3, 59-68 (1994).
- Delaney, N. F., et al. Development of an optimized medium, strain and high-throughput culturing methods for Methylobacterium extorquens. PLOS ONE. 8 (4), 62957 (2013).
- Haire, T. C., et al. Robust microplate-based methods for culturing and in vivo phenotypic screening of Chlamydomonas reinhardtii. Frontiers in Plant Science. 9, 235 (2018).
- McBirney, S. E., Trinh, K., Wong-Beringer, A., Armani, A. M. Wavelength-normalized spectroscopic analysis of Staphylococcus aureus and Pseudomonas aeruginosa growth rates. Biomedical Optics Express. 7 (10), 4034 (2016).
- Sieben, M., Giese, H., Grosch, J. H., Kauffmann, K., Büchs, J. Permeability of currently available microtiter plate sealing tapes fail to fulfil the requirements for aerobic microbial cultivation. Biotechnology Journal. 11 (12), 1525-1538 (2016).
- Zimmermann, H. F., John, G. T., Trauthwein, H., Dingerdissen, U., Huthmacher, K. Rapid evaluation of oxygen and water permeation through microplate sealing tapes. Biotechnology Progress. 19 (3), 1061-1063 (2003).
- Abkar, L., Gagnon, G. A. Biological responses to P-limitation in indigenous bacteria isolated from drinking water. AWWA Water Science. 3 (5), 1248 (2021).
- Sanders, E. R. Aseptic laboratory techniques: Plating methods. Journal of Visualized Experiments. (63), e3064 (2012).
- Hart, S. F. M., Skelding, D., Waite, A. J., Burton, J. C., Shou, W. High-throughput quantification of microbial birth and death dynamics using fluorescence microscopy. Quantitative Biology. 7 (1), 69-81 (2019).
- Periago, P. M., Abee, T., Wouters, J. A. Analysis of the heat-adaptive response of psychrotrophic Bacillus weihenstephanensis. International Journal of Food Microbiology. 79 (1-2), 17-26 (2002).
- Mira, P., Barlow, M., Meza, J. C., Hall, B. G. Statistical package for growth rates made easy. Molecular Biology and Evolution. 34 (12), 3303-3309 (2017).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon