A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
יכולת שחזור ודיוק משופרים של כימות תפוקה גבוהה של נתוני גידול חיידקים באמצעות קורא מיקרו-פלטות
In This Article
Summary
כאן, מוצג פרוטוקול בעל תפוקה גבוהה למדידת נתוני צמיחה, כולל עקומות צמיחה, קצב צמיחה וקצב צמיחה מקסימלי. הפרוטוקול אומת ותוקף באמצעות שני חיידקים המייצרים ביופילם. ניתן להרחיב את התוצאות והגישה המיושמת במחקר זה לפרוטוקולים אחרים בעלי תפוקה גבוהה באמצעות קוראי מיקרו-פלטות.
Abstract
מחקר זה נועד לפתח פרוטוקול חוזר, אמין ובעל תפוקה גבוהה לניטור צמיחת חיידקים בצלחות של 96 בארות ולניתוח קצב הגידול המרבי. נקבעו עקומות הצמיחה ושיעורי הגידול המקסימליים של שני מיני חיידקים. נבדקו נושאים הכוללים (i) עיבוי מכסה, (ii) תיקון אורך מסלול, (iii) גודל החיסון, (iv) מרווח זמן הדגימה ו-(v) הטיה מרחבית. יכולת החזרה של הפרוטוקול הוערכה באמצעות שלושה שכפולים טכניים בלתי תלויים, עם סטיית תקן של 0.03 בין הריצות. שיעורי הגידול המקסימליים של Bacillus mycoides ו-Paenibacillus tundrae נקבעו כ±-0.99 h-1 ±-0.03 h-1 ו-0.85 h-1 ±-0.025 h-1, בהתאמה. חיידקים אלה מאתגרים יותר לניטור אופטי בשל זיקתם להתקבץ יחד. מחקר זה מדגים את החשיבות הקריטית של גודל החיסון, תיקון אורך הנתיב, התחממות המכסה, מרווחי זמן הדגימה וההטיה המרחבית של לוח הבאר כדי להשיג נתונים אמינים, מדויקים וניתנים לשחזור על קוראי מיקרו-לוחות. ניתן להרחיב את הפרוטוקול שפותח ואת שלבי האימות שלו לשיטות אחרות באמצעות קוראי מיקרו-פלטות ופרוטוקולים בעלי תפוקה גבוהה, מה שמפחית את השגיאות המולדות של החוקרים ואת עלויות החומר.
Introduction
פיתוח עניין במניפולציה מולטי-אומיקס, כולל מחקרי מנגנון ומטבוליזם של חיידקים, מדגיש את החשיבות של שיטות אוטומטיות בתפוקה גבוהה כגון רישום נתוני גדילה 1,2. נתוני גדילה הכוללים פרמטרים קינטיים, כגון שיעורי גדילה מקסימליים, יכולים לסייע באפיון תגובות חיידקים לתנאים פיזיקליים, כימיים ואנטיבקטריאליים שונים. נתוני קצב הגדילה הם משתנה תגובה סטנדרטי המשמש לחשיפת קשרים פוטנציאליים בין גנוטיפלפנוטיפ 1 או להצביע על הבטיחות המיקרוביאלית וחיי המדף של תוצרת מזון 3,4. טכניקות כגון אבולוציה מעבדתית אדפטיבית 5,6,7, סינון גנומי רחב, בדיקות כימיות מסוימות8 ומסכים גנטיים שונים קדימה9 מסתמכות על קצב הגדילה כדי להעריך את התוצאות.
מדידות צפיפות אופטית (OD) של תרביות חיידקים הן שיטה מיקרוביולוגית סטנדרטית לניטור צמיחת חיידקים. מדידות OD נרשמות לרוב באורך גל של 600 ננומטר, תוך הסתמכות על פיזור אור וצפיפות התא10,11. חוק באר-למברט מסביר את התלות של ערכי OD בריכוז (כלומר, צפיפות התא, מספר התא), אורך הנתיב ומקדם הספיגה. הגיאומטריה והמערכת האופטית של ספקטרופוטומטר משפיעות על קריאות OD11. שיטות קלאסיות של מדידות OD יכולות להיות עתירות זמן ועבודה, והנתונים יכולים לשאת מגוון טעויות אנוש. בפרוטוקול זה, קורא מיקרו-פלטות משמש כדי להפחית את זמן האנליסט12,13 ואת הסיכוי לזיהום ביולוגי. ניתוח תפוקה גבוהה באמצעות קוראי מיקרו-פלטות מיושם באופן נרחב בתחומי מיקרוביולוגיה שונים, כגון הקרנת חיידקים המייצרים ביופילם14,15, עיכוב צמיחת חיידקים16, צמיחת תאי שמרים17, קביעת רגישות אנטי פטרייתית18 והקרנת רעילות של ננו-חומרים19.
כמה חוקרים פרסמו פרוטוקולים של קצב גידול חיידקים באמצעות קורא מיקרו-פלטות 12,20,21. עם זאת, פרוטוקול יסודי הבוחן את מהימנות הנתונים שנאספו לא הוכח במלואו. מדווח כי גורמים כמו סוג המין 22,23,24 וסרטי איטום משפיעים על יכולת החזרה עקב חוסר העברת חמצן בלוח בן 96 בארות25,26. Delaney ואחרים דיווחו על אשכולות גדולים של Methylorubrum extorquens (זן מסוג בר) במדיום הגידול בעת שימוש בקורא מיקרו-פלטות, מה שגרם לנתוני צמיחה רועשים במיוחד24. הבעיה נפתרה על ידי הסרת הגנים הקשורים לייצור ביופילם24. בשל הפרשת חומרים פולימריים חוץ-תאיים, לחיידקים המייצרים ביופילם יש זיקה גדולה יותר להתלכד יחד וליצור אשכולות תאים. לכן, מאתגר יותר לעקוב אחר צמיחתם באמצעות טכניקות פיזור אור (למשל, ספקטרופוטומטרים וקוראי מיקרו-לוחות).
פרוטוקול זה נועד לקבוע צעדים להשגת נתונים הניתנים לשחזור בשיטה בעלת תפוקה גבוהה באמצעות קורא מיקרו-פלטות. נעשה שימוש ב-Bacillus mycoides ו-Paenibacillus tundrae בשל הצמיחה המהירה שלהם ויכולת ייצור הביופילם, שבאופן מסורתי מאתגרים בגישות ידניות ואוטומטיות. גורמים כגון (i) תיקון אורך מסלול, (ii) עיבוי על המכסה, (iii) גודל החיסון, (iv) מרווח זמן הדגימה ו-(v) הטיה מרחבית נחקרו כדי להעריך את המהימנות והשחזור של הנתונים. פרוטוקול זה מציג שלבים לניטור מדויק של צמיחת חיידקים ומדידת שיעורי גידול ספציפיים באמצעות קורא מיקרו-פלטות.
Protocol
הערה: יש לבצע את כל השלבים בפרוטוקול זה בתנאים סטריליים (כלומר, בין שתי להבות או ארון בטיחות ביולוגית). כל החומרים והכלים עוברים חיטוי למשך 20 דקות. עיין בטבלת החומרים לקבלת פרטים אודות כל החומרים, הציוד והתוכנה המשמשים בפרוטוקול זה. ידיים עטויות כפפות מחוטאות, נשמרות רטובות בחומר חיטוי ידיים או בתמיסת אלכוהול 70% למשך דקה אחת לפחות, ולא מוסרות מארון הבטיחות לאחר מכן. אחרת, יש לחזור על הליך החיטוי לפני הכנסת הידיים בחזרה לארון הבטיחות. זהירות: ודא שחומר החיטוי מתאדה לחלוטין לפני השימוש בלהבה פתוחה.
הערה: שני חיידקים בודדו מסינון ביולוגי של מי שתייה כפי שהוסבר קודם לכן27 בשל יכולתם לייצר ביופילם. הם זוהו על ידי ריצוף rRNA מלא של 16S והוגשו ל-NCBI כ-Bacillus mycoides (SAMN10518261) ו-Paenibacillus tundrae (SAMN10452279).
1. הכנת מלאי חיידקים בגליצרול
- שקלו 3 גרם אבקת מרק סויה טריפטי (TSB) והמיסו אותה ב-100 מ"ל מים מזוקקים. חיטוי המרק למשך 20 דקות ונותנים לו להתקרר לטמפרטורת החדר. הוסף 50 מ"ל TSB לבקבוק של 50 מ"ל.
- מעבירים את החיידקים לצלחות אגר27,28. בעזרת לולאה, בחר מושבת חיידקים מצלחת האגר. הוסף את תוכן הלולאה לבקבוק על ידי סיבוב עדין של הלולאה. דגרו את הבקבוק למשך הלילה ב-30 מעלות צלזיוס ±-1 מעלות צלזיוס עם טלטול מסלול ב-150 סל"ד.
הערה: יש לכוונן את הטמפרטורה והרעידות בהתאם לסוג המתח. - הכינו עוד בקבוק של 50 מ"ל עם 47.5 מ"ל TSB. העבירו 2.5 מ"ל מתרבות הלילה לבקבוק. דגרו אותו במשך 5-6 שעות כדי להגיע ל-OD600 = 0.6 0.05 (כדי להגיע לשלב האקספוננציאלי האמצעי).
- העבירו את התרבית לצינור צנטריפוגה מעוקר של 50 מ"ל. צנטריפוגה את התרבות ב -2,200 × גרם למשך 10 דקות. רוקנים את המרק בעדינות כדי למנוע אובדן תאים שנאספו בתחתית הצינור.
- הוסף 5 מ"ל של מי מלח עם חוצץ פוספט (PBS) לאותו צינור וצנטריפוגה ב-2,200 × גרם למשך 10 דקות. חזור על שלב זה 3 פעמים.
- התאם את ה-OD600 של התאים ל-0.6 0.05 על ידי שימוש בפיפטה כדי לטטר כמות מספקת של תמיסת PBS. רשום במדויק את הכמות הנוספת של PBS לשימוש עתידי.
- טיטראט גליצרול לתרחיף כדי להגיע ל-20% v/v. מכסה את הצינור והמערבולת למשך 15 שניות.
- העבירו 1.5 מ"ל מהמתלה ל-2 מ"ל קריוביאלים. הכניסו את הקריוביאל לקופסה מתאימה ואחסנו אותם במקפיא בחום של -80 מעלות צלזיוס.
2. הכינו תרבית לילה.
- הניחו לקריביאלים עם ציר חיידקים להפשיר בטמפרטורת החדר למשך 30 דקות או עד שהתוכן נוזלי.
הערה: מומלץ לא להאיץ את תהליך ההפשרה. - הכן צינור מעוקר עם 10 מ"ל TSB. העבירו 50 מיקרוליטר ממתלה המלאי לצינור. דגרו את הצינור למשך הלילה (16-18 שעות) ב-30 מעלות צלזיוס ±-1 מעלות צלזיוס עם טלטול מסלול ב-150 סל"ד.
3. מכינים את החיסון.
- הכן צינור עם 10 מ"ל TSB מעוקר. העבירו 500 מיקרוליטר מתרבית הלילה לצינור. דגרו אותו במשך 6-8 שעות כדי להגיע ל-OD600 = 0.6 0.05 (או להגיע לשלב האקספוננציאלי האמצעי).
- העבירו את התרבית לצינור צנטריפוגה של 15 מ"ל. צנטריפוגה את הצינור ב -2,200 × גרם למשך 10 דקות. שופכים את המרק בעדינות.
- הוסף 5 מ"ל PBS וצנטריפוגה ב -2,200 × גרם למשך 10 דקות. חזור על שלב זה 3 פעמים.
- כוונן את ה-OD600 של התאים ל-0.6 0.05 על ידי טיטרציה של תמיסת PBS באמצעות פיפטה. רשום במדויק את הכמות הנוספת של PBS לשימוש עתידי.
4. העברת מדיום גידול לקורא המיקרופלייט
- בחר צלחת באר שקופה, סטרילית, עם תחתית שטוחה עם מכסה (כלומר, צלחת 96 בארות, צלחת 6 בארות, צלחת 12 בארות).
- מדוד את תיקון אורך הנתיב עבור המיקרו-לוח באמצעות מי DI לפני הפעלת הפרוטוקול, כפי שהוסבר בשלב 1.10.
- צייר טבלת התייחסות המבוססת על לוחית הבאר שנבחרה כדי לציין את מיקומי הדגימות לפני הטעינה. לדוגמא, צייר שולחן בגודל 8X12 לצלחת של 96 בארות.
הערה: טבלת ההפניות מונעת בלבול במהלך שלבי הדגירה ואיסוף התוצאות. - הניחו את הפריטים הבאים בארון בטיחות ביולוגית: מיקרופלייט עם מכסה, פיפטור 1,000 מיקרוליטר ו-200 מיקרוליטר, קצות פיפטה של 1,000 מיקרוליטר ו-200 מיקרוליטר, מספר קריוביאלים, של 25 מ"ל, מדיום הגידול והחיסון.
הערה: יש להעביר את הפריטים שהוזכרו לארון הבטיחות הביולוגית באופן אספטי. - הוסף 10 מ"ל מצע גידול לכוס. העבירו את כמות החיסון המחושבת לכוס עם מצע הגידול.
הערה: יחסי החיסון בפרוטוקול זה הם 1% ו-5%. לדוגמה, כדי להשיג יחס חיסון של 5% ו-1%, הוסף 500 מיקרוליטר ו-100 מיקרוליטר של החיסון ל-9.5 מ"ל ו-9.9 מ"ל של מצע הגידול, בהתאמה. - לאחר הוספת החיסון, יש לנער בעדינות את הכוס לקבלת פיזור אחיד. הוצא 200 מיקרוליטר מהמדיום המחוסן לבארות המיועדות באמצעות טבלת הייחוס. בחר לפחות שלוש בארות שישמשו כבקרות על ידי הוספת 200 מיקרוליטר של מצע הגידול (ללא חיסון).
הערה: ודא שבארות הבקרה מסומנות בבירור בטבלת הייחוס. אם נצפתה גידול (זיהום) כלשהו בבארות אלה, יש לחזור על הניסוי. - הוצא 200 מיקרוליטר של מי DI לבארות הקצה שבהן קצב האידוי גבוה יותר. הנח את המכסה בעדינות לפני הוצאת צלחת 96 הבארות מארון הבטיחות. הנח את צלחת 96 הבארות בקורא הצלחות בזהירות, והבטיח שלא יהיו תנועות פתאומיות.
5. הגדרות קורא Microplate
- הפעל את קורא המיקרו-לוחות ולאחר מכן השתמש בתוכנה המוטמעת כדי ליצור את הפרוטוקול המותאם אישית. במנהל המשימות, פתח את הפרוטוקול ובחר צור חדש | פרוטוקול סטנדרטי. פתח את נוהל והתאם את ההגדרות באופן הבא:
- הגדר את הטמפרטורה ל-30 מעלות צלזיוס (או את ערכי היעד) באמצעות הגדרת טמפרטורה.
- לחץ על Incubator On ולאחר מכן הגדר את השיפוע ל-1 מעלות צלזיוס.
הערה: כדי למנוע נתונים לא אמינים עקב עיבוי על המכסה, ודא שהשיפוע מוגדר ל-1 מעלות צלזיוס. - בחר באפשרות חימום מוקדם , תוך הבטחת פיזור חום אחיד לאורך צלחת הבאר.
- פתח את התחל קינטי ובחר את משך הדגירה הרצוי באמצעות זמן הריצה (למשל, 12:00:00 או 24:00:00 וכו') ומרווח בין קריאות עבור 00:30:00 (מייצג 30 דקות) או 01:00:00 (מייצג שעה).
הערה: קריאת כל צלחת של 96 בארות אורכת כדקה אחת. - הגדר את הגדרות הרעידות באופן הבא: פתח את טלטול; הגדר את מצב טלטול לליניארי; לחץ על טלטול מתמשך; והתאם את התדר ל-567 עותקים לדקה (או כל ערך רצוי).
- הגדר את הגדרות הקריאה של OD באופן הבא: לחץ על קרא | ספיגה; בחר נקודת קצה/קינטית ומונוכרומטורים; הגדר את אורך הגל ל-600 ננומטר; ולחץ על אימות | שמור כדי לשמור אותו כפרוטוקול חדש עם השם והתאריך המתאימים.
6. רישום נתוני צמיחה
- פתח את מנהל המשימות | קרא עכשיו.
- בחר את הפרוטוקול השמור ולאחר מכן לחץ על אישור כדי להתחיל את הפרוטוקול.
- שמור את הקריאות לניתוח נתונים.
7. ניתוח נתונים
- העבר את תוצאות OD600 (נתוני צמיחה) לפורמט גיליון אלקטרוני. בקטע התוצאות , לחץ על אפשרות התמונה עבור כל באר כדי לצפות בעקומות הצמיחה הסופיות.
- פתח את קובץ הגיליון האלקטרוני וסדר מחדש את הנתונים המוקלטים על סמך טבלת ההפניה. הפחת את הערך הממוצע של הקריאות הריקות מהקריאות האחרות.
הערה: ריקים הם קריאות ה-OD בזמן אפס. בפרוטוקול זה, ערכי OD600 נלקחים בחשבון לצורך חישוב. ניתן להשתמש בצפיפות התאים אם עקומת הכיול עבור OD600 לעומת ספירת תאים זמינה.
8. קביעת קצב צמיחה
- השתמש בנתונים המועברים בגיליון האלקטרוני שנוצר בשלב 1.6. השתמש בשיטה המתוארת להלן ובמשוואה (1) כדי להעריך את קצב הגידול.
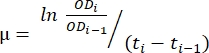 (1)
(1)
כאשר μ הוא קצב הצמיחה, ODi הוא ה-OD600 בכל נקודת זמן, ODi-1 הוא הערך הראשוני של OD600 בנקודת הזמן האחרונה, ti ו-t i-1 הם הפרשי הזמן בין שתי הנקודות שהוזכרו (למשל, 0.5 למרווחי זמן של 30 דקות, 1 למרווחי זמן של שעה). - חשב את הממוצע וסטיית התקן של חמישה שיעורי גידול עוקבים באמצעות תוכנת ניתוח הנתונים הרצויה. שימו לב שהממוצע הגדול ביותר עם סטיית התקן הנמוכה ביותר הוא קצב הגידול המקסימלי (μמ') 12.
9. קביעת הטיה מרחבית
הערה: קוראים ולוחות מיקרו-פלטות מציגים הטיה בתוצאות. חיוני להעריך את ההטיות המרחביות של הלוחות המשמשים בקורא מיקרו-פלטות ספציפי כדי להבטיח את האמינות והשחזור של התוצאות. כדי להשיג זאת, השתמש בשלבים הבאים:
- חזור על שלבים 1.2-1.6.
- רשום את הנתונים ונתח לפי שלב 1.7 ושלב 1.8 כדי לחשב את קצב הגידול עבור כל באר.
- צור מפת חום כדי לדמיין את שינויי קצב הצמיחה בצלחת 96 הבארות.
- בצע את הניתוח הסטטיסטי (ראה טבלאות משלימות), תוך הקפדה על הבדלים משמעותיים בין הבארות.
הערה: חיוני להשתמש באותו סוג חיידקים ובאותו גודל חיסון בכל הבארות.
10. אימות קריאת OD וגורם תיקון אורך נתיב
הערה: קביעת גורם תיקון אורך הנתיב היא חיונית לאימות הנתונים ולהבטיח אמינות ותוקף במכשירים שונים.
- יש להשתמש בשלב 1.2 ושלב 1.3 להכנת חיסון הלילה של B. mycoides.
- מכינים שלוש צלוחיות מעוקרות של 50 מ"ל ומוסיפים 49.5 מ"ל TSB טרי לכל בקבוק. הוסף 500 מיקרוליטר של החיסון לכל בקבוק בארון בטיחות באופן אספטי. הוסף 50 מ"ל TSB לא מחוסן לבקבוק מעוקר, שישמש כבקרה.
- דגרו את הצלוחיות ב-30 מעלות צלזיוס ו-150 סל"ד. כל 30 דקות, קח דגימות מכל בקבוק באופן אספטי וקרא ורשום OD600 בספקטרופוטומטר.
- הוסף 9.9 מ"ל TSB לכוס של 25 מ"ל. מוסיפים 100 מיקרוליטר חיסון לכוס ומנערים בעדינות.
- מוסיפים 200 מיקרוליטר מהתערובת בכוס לשש הבארות בעמודה נבחרת בצלחת של 96 בארות. הוסף 200 מיקרוליטר של TSB לא מחוסן לשלוש בארות כבקרה. מלאו את בארות הקצה ב-200 מיקרוליטר מי DI.
- הנח את המכסה והעביר בעדינות את צלחת 96 הבארות לקורא המיקרופלייט. בצע את שלב 1.5 ושלב 1.6 כדי לתעד את נתוני הצמיחה.
- התווה את ה-OD600 מקורא המיקרו-פלטות כנגד ה-OD600 מהספקטרופוטומטר. שימו לב שהשיפוע הוא ההיסט בין שתי קבוצות הקריאות, הנקרא גורם תיקון אורך המסלול.
הערה: הדגירה של החממה וקורא המיקרו-פלטות צריכה להיעשות בו זמנית באותו היום.
תוצאות
אימות קריאת OD וגורם תיקון אורך נתיב
דגימות מפוצלות של תרבית B. mycoides נלקחו בנקודות זמן שונות ונמדדו באמצעות קורא המיקרו-פלטות והספקטרופוטומטר (איור 1A). צעד זה ננקט כדי לאמת את התוצאות במכשירים שונים. נתוני OD600 היו בקורלציה אך לא תואמים ...
Discussion
קוראי מיקרו-פלטות מאפשרים להשיג שיעורי צמיחה עקביים וניתנים לחזרה. טכנולוגיה זו ממזערת טעויות אנוש ומאפשרת דגימה בתפוקה גבוהה. כמות התרבית הקטנה הנדרשת לדגימה הופכת גישה זו לחלופה אטרקטיבית ובעלות נמוכה לספירת תאים באמצעות צלוחיות או מבחנות. קוראי Microplate מאפשרים גודל מ...
Disclosures
למחברים אין ניגודי אינטרסים להצהיר עליהם.
Acknowledgements
עבודה זו מומנה על ידי מועצת המחקר למדעי הטבע וההנדסה (NSERC) / יו"ר המחקר התעשייתי של הליפקס לאיכות וטיפול במים (מענק מס. IRCPJ 349838-16). צוות המחברים רוצה גם להודות לעזרתה של אניטה טיילור בסקירת מאמר זה.
Materials
| Name | Company | Catalog Number | Comments |
| Centrifuge | Eppendorf | 5810 R | |
| Centrifuge tubes - 15 mL | ThermoFisher- Scientific | 339650 | Sterile |
| Centriguge tubes - 50 mL | ThermoFisher- Scientific | 339652 | Sterile |
| Disposable inoculating loop , 10 µL | Cole-Parmer | UZ-06231-08 | Sterile |
| Erlenmeyer flasks - 250 mL | Cole-Parmer | UZ-34502-59 | Glass |
| Isopropanol | ThermoFisher- Scientific | 396982500 | ≥99.0 |
| Phosphate Buffer Saline | Sigma-Aldrich | P4417 | |
| Pipett tips 1,000 µL | ThermoFisher- Scientific | UZ-25001-76 | |
| Pipett tips 10 mL | ThermoFisher- Scientific | UZ-25001-83 | |
| Pipett tips 200 µL | ThermoFisher- Scientific | UZ-25001-85 | |
| Pipett tips 5 mL | ThermoFisher- Scientific | UZ-25001-80 | |
| Pipettor 1,000 µL | Cole-Parmer | UZ-07909-11 | |
| Pipettor 10 mL | Cole-Parmer | UZ-07909-15 | |
| Pipettor 200 µL | Cole-Parmer | UZ-07909-09 | |
| Pipettor 5 mL | Cole-Parmer | UZ-07859-30 | |
| Tryptic Soy Broth | Millipore | 22091 | Suitable for microbiology |
References
- Reuß, D. R., et al. Large-scale reduction of the Bacillus subtilis genome: Consequences for the transcriptional network, resource allocation, and metabolism. Genome Research. 27 (2), 289-299 (2017).
- Sparkes, A., et al. Towards Robot Scientists for autonomous scientific discovery. Automated Experimentation. 2 (1), (2010).
- Zwietering, M. H., Jongenburger, I., Rombouts, F. M., van t Riet, K. Modeling of the bacterial growth curve. Applied and Environmental Microbiology. 56 (6), 1875-1881 (1990).
- Pla, M., Oltra, S., Esteban, M., Andreu, S., Palop, A. Comparison of primary models to predict microbial growth by the plate count and absorbance methods. BioMed Research International. 2015 (6), 1-14 (2015).
- Choe, D., et al. Adaptive laboratory evolution of a genome-reduced Escherichia coli. Nature Communications. 10 (1), 935 (2019).
- Dykhuizen, D. E., Dean, A. M. Enzyme activity and fitness: Evolution in solution. Trends in Ecology and Evolution. 5 (8), 257-262 (1990).
- McDonald, M. J. Microbial experimental evolution - A proving ground for evolutionary theory and a tool for discovery. EMBO Reports. 20 (8), 1-14 (2019).
- Lum, P. Y., et al. Discovering modes of action for therapeutic compounds using a genome-wide screen of yeast heterozygotes. Cell. 116 (1), 121-137 (2004).
- Cagnon, C., et al. Development of a forward genetic screen to isolate oil mutants in the green microalga Chlamydomonas reinhardtii. Biotechnology for Biofuels. 6 (1), 178 (2013).
- Stevenson, K., McVey, A. F., Clark, I. B. N., Swain, P. S., Pilizota, T. General calibration of microbial growth in microplate readers. Scientific Reports. 6 (1), 38828 (2016).
- Matlock, B. C., Beringer, R. W., Ash, D. L., Page, A. F., Allen, M. W. Differences in Bacterial Optical Density Measurements between Spectrophotometers. Technical Note. ThermoScientific. , (2011).
- Hall, B. G., Acar, H., Nandipati, A., Barlow, M. Growth rates made easy. Molecular Biology and Evolution. 31 (1), 232-238 (2014).
- Rolfe, M. D., et al. Lag phase is a distinct growth phase that prepares bacteria for exponential growth and involves transient metal accumulation. Journal of Bacteriology. 194 (3), 686-701 (2012).
- Djordjevic, D., Wiedmann, M., Mclandsborough, L. A. Microtiter plate assay for assessment of Listeria monocytogenes biofilm formation. Applied and Environmental Microbiology. 68 (6), 2950-2958 (2002).
- O'Toole, G. A. Microtiter dish biofilm formation assay. Journal of Visualized Experiments. (47), e2437 (2011).
- Campbell, J. High-throughput assessment of bacterial growth inhibition by optical density measurements. Current Protocols in Chemical Biology. 3 (3), 1-20 (2012).
- Toussaint, M., Conconi, A. High-throughput and sensitive assay to measure yeast cell growth: A bench protocol for testing genotoxic agents. Nature Protocols. 1 (4), 1922-1928 (2006).
- Goughenour, K. D., Balada-Llasat, J. -. M., Rappleye, C. A. Quantitative microplate-based growth assay for determination of antifungal susceptibility of Histoplasma capsulatum yeasts. Journal of Clinical Microbiology. 53 (10), 3286-3295 (2015).
- Qiu, T. A., et al. Growth-based bacterial viability assay for interference-free and high-throughput toxicity screening of nanomaterials. Analytical Chemistry. 89 (3), 2057-2064 (2017).
- Kurokawa, M., Precise Ying, B. -. W. Precise, high-throughput analysis of bacterial growth. Journal of Visualized Experiments. (127), e56197 (2017).
- Bredit, F., Romick, T. L., Fleming, H. P. A rapid method for determination of bacterial growth kinetics. Journal of Rapid Methods and Automation in Microbiology. 3, 59-68 (1994).
- Delaney, N. F., et al. Development of an optimized medium, strain and high-throughput culturing methods for Methylobacterium extorquens. PLOS ONE. 8 (4), 62957 (2013).
- Haire, T. C., et al. Robust microplate-based methods for culturing and in vivo phenotypic screening of Chlamydomonas reinhardtii. Frontiers in Plant Science. 9, 235 (2018).
- McBirney, S. E., Trinh, K., Wong-Beringer, A., Armani, A. M. Wavelength-normalized spectroscopic analysis of Staphylococcus aureus and Pseudomonas aeruginosa growth rates. Biomedical Optics Express. 7 (10), 4034 (2016).
- Sieben, M., Giese, H., Grosch, J. H., Kauffmann, K., Büchs, J. Permeability of currently available microtiter plate sealing tapes fail to fulfil the requirements for aerobic microbial cultivation. Biotechnology Journal. 11 (12), 1525-1538 (2016).
- Zimmermann, H. F., John, G. T., Trauthwein, H., Dingerdissen, U., Huthmacher, K. Rapid evaluation of oxygen and water permeation through microplate sealing tapes. Biotechnology Progress. 19 (3), 1061-1063 (2003).
- Abkar, L., Gagnon, G. A. Biological responses to P-limitation in indigenous bacteria isolated from drinking water. AWWA Water Science. 3 (5), 1248 (2021).
- Sanders, E. R. Aseptic laboratory techniques: Plating methods. Journal of Visualized Experiments. (63), e3064 (2012).
- Hart, S. F. M., Skelding, D., Waite, A. J., Burton, J. C., Shou, W. High-throughput quantification of microbial birth and death dynamics using fluorescence microscopy. Quantitative Biology. 7 (1), 69-81 (2019).
- Periago, P. M., Abee, T., Wouters, J. A. Analysis of the heat-adaptive response of psychrotrophic Bacillus weihenstephanensis. International Journal of Food Microbiology. 79 (1-2), 17-26 (2002).
- Mira, P., Barlow, M., Meza, J. C., Hall, B. G. Statistical package for growth rates made easy. Molecular Biology and Evolution. 34 (12), 3303-3309 (2017).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved