JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
Microplate Reader를 사용한 박테리아 성장 데이터의 고처리량 정량화의 재현성 및 정밀도 향상
요약
여기에서는 성장 곡선, 성장률 및 최대 성장률을 포함한 성장 데이터를 측정하기 위해 고처리량 프로토콜이 제공됩니다. 이 프로토콜은 두 개의 생물막 생성 박테리아를 사용하여 검증 및 검증되었습니다. 이 연구에 적용된 결과와 접근 방식은 마이크로플레이트 리더를 사용하여 다른 고처리량 프로토콜로 확장할 수 있습니다.
초록
이 연구는 96웰 플레이트에서 박테리아 성장을 모니터링하고 최대 성장률을 분석하기 위해 반복 가능하고 신뢰할 수 있는 고처리량 프로토콜을 개발하는 것을 목표로 했습니다. 두 박테리아 종의 성장 곡선과 최대 성장률을 측정했습니다. (i) 뚜껑 응결, (ii) 경로 길이 보정, (iii) 접종 크기, (iv) 샘플링 시간 간격 및 (v) 공간 편향을 포함한 문제를 조사했습니다. 프로토콜의 반복성은 실행 간 표준 편차가 0.03인 세 가지 독립적인 기술 복제로 평가되었습니다. 바실러스 마이코이데스(Bacillus mycoides )와 페니바실러스 툰드라(Paenibacillus tundrae )의 최대 성장률은 ±각각 0.99 h−1 ± 0.03 h−1 및 0.85 h−1 ± 0.025 h−1로 측정되었다. 이러한 박테리아는 서로 뭉치는 친화력 때문에 광학적으로 모니터링하기가 더 어렵습니다. 이 연구는 마이크로플레이트 리더에서 신뢰할 수 있고 정확하며 재현 가능한 데이터를 얻기 위해 접종 크기, 경로 길이 보정, 뚜껑 보온화, 샘플링 시간 간격 및 웰 플레이트 공간 편향의 중요성을 보여줍니다. 개발된 프로토콜과 검증 단계는 마이크로플레이트 리더와 고처리량 프로토콜을 사용하여 다른 방법으로 확장할 수 있어 연구자의 타고난 오류와 재료 비용을 줄일 수 있습니다.
서문
박테리아의 메커니즘 및 대사 연구를 포함한 다중 오믹스 조작에 대한 관심이 높아짐에 따라 성장 데이터 기록과 같은 고처리량 및 자동화된 방법의 중요성이 강조되고 있습니다 1,2. 최대 성장률과 같은 운동 매개변수로 구성된 성장 데이터는 다양한 물리적, 화학적, 항균 조건에 대한 박테리아 반응을 특성화하는 데 도움이 될 수 있습니다. 성장률 데이터는 잠재적인 유전자형-표현형 연관성1을 밝히거나 식품 농산물의 미생물 안전성 및 유통 기한을 나타내는 데 사용되는 표준 반응 변수입니다 3,4. 적응형 실험실 진화(adaptive laboratory evolution) 5,6,7, 게놈 전체 스크리닝(genome-wide screening), 특정 화학 분석(certain chemical assay8), 다양한 전방 유전자 스크리닝(forward genetic screening)9 등의 기술은 성장 속도에 의존하여 결과를 평가합니다.
박테리아 배양의 광학 밀도(OD) 측정은 박테리아 성장을 모니터링하기 위한 표준 미생물 학적 방법입니다. OD 측정은 종종 600nm의 파장에서 기록되며 광 산란과 셀 밀도10,11에 의존합니다. Beer-Lambert 법칙은 농도(즉, 세포 밀도, 세포 수), 경로 길이 및 흡수 계수에 대한 OD 값의 종속성을 설명합니다. 분광 광도계의 기하학과 광학 시스템은 OD 판독값에 영향을 미칩니다11. OD 측정의 고전적인 방법은 시간과 노동 집약적일 수 있으며 데이터에는 다양한 인적 오류가 있을 수 있습니다. 이 프로토콜에서는 마이크로플레이트 리더를 사용하여 분석가 시간12,13 및 생물학적 오염 가능성을 줄입니다. 마이크로플레이트 리더를 사용한 고처리량 분석은 생물막 생성 박테리아14,15, 박테리아 성장 억제16, 효모 세포 성장17, 항진균 감수성18 측정 및 나노 물질의 독성 스크리닝19와 같은 다양한 미생물학 분야에 광범위하게 적용됩니다.
몇몇 연구자들은 마이크로플레이트 리더를 사용하여 박테리아 성장률 프로토콜을 발표했습니다 12,20,21. 그러나 수집된 데이터의 신뢰성을 검토하는 철저한 프로토콜은 완전히 확립되지 않았습니다. 종유형 22,23,24 및 밀봉 테이프와 같은 요인이 96-웰 플레이트의 산소 전달 부적절성으로 인해 반복성에 영향을 미치는 것으로 보고되었습니다 25,26. Delaney 등은 마이크로플레이트 리더를 사용할 때 성장 배지에서 Methylorubrum extorquens(야생형 균주)의 큰 클러스터가 발생했다고 보고했으며, 이로 인해 매우 시끄러운 성장 데이터가 발생했다고 보고했습니다24. 이 문제는 생물막 생산과 관련된 유전자를 제거함으로써 해결되었습니다24. 세포 외 고분자 물질의 분비로 인해 생물막을 생성하는 박테리아는 서로 합쳐져 세포 클러스터를 생성하는 데 더 큰 친화력을 갖습니다. 따라서 광 산란 기술(예: 분광 광도계 및 마이크로플레이트 리더)을 사용하여 성장을 모니터링하는 것이 더 어렵습니다.
이 프로토콜은 마이크로플레이트 리더를 사용하여 고처리량 분석법으로 재현 가능한 데이터를 얻기 위한 단계를 설정하는 것을 목표로 합니다. 바실러스 마이코이데스(Bacillus mycoides )와 패니바실러스 툰드래(Paenibacillus tundrae )는 빠른 성장과 생물막 생성 능력으로 인해 사용되었는데, 이는 전통적으로 수동 및 자동 접근 방식에서 까다로웠습니다. 데이터의 신뢰성과 재현성을 평가하기 위해 (i) 경로 길이 보정, (ii) 뚜껑의 응결, (iii) 접종 크기, (iv) 샘플링 시간 간격 및 (v) 공간 편향과 같은 요인을 조사했습니다. 이 프로토콜은 마이크로플레이트 리더를 사용하여 박테리아 성장을 정확하게 모니터링하고 특정 성장률을 측정하기 위한 단계를 제시합니다.
프로토콜
참고: 이 프로토콜의 모든 단계는 멸균 상태(즉, 두 개의 화염 또는 생물 안전 캐비닛 사이)에서 따라야 합니다. 모든 재료와 도구는 20분 동안 고압멸균됩니다. 이 프로토콜에 사용되는 모든 재료, 장비 및 소프트웨어에 대한 자세한 내용은 재료 표를 참조하십시오. 장갑을 낀 손은 소독하고 손 소독제 또는 70% 알코올 용액으로 최소 1분 동안 적신 상태로 유지한 후 안전 캐비닛에서 제거하지 마십시오. 그렇지 않으면 손을 안전 캐비닛에 다시 넣기 전에 소독 절차를 반복해야 합니다. 주의 : 화염을 사용하기 전에 소독제가 완전히 증발했는지 확인하십시오.
참고: 두 개의 박테리아가 바이오필름을 생성하는 능력에 대해 앞서 설명한 바와 같이 식수 생물여과에서 분리되었습니다27 . 이들은 full-16S rRNA 염기서열분석을 통해 확인되었으며 Bacillus mycoides(SAMN10518261) 및 Paenibacillus tundrae(SAMN10452279)로 NCBI에 제출되었습니다.
1. 글리세롤에 박테리아 스톡을 준비하기
- 트립틱 소이 육수(TSB) 분말 3g의 무게를 달아 증류수 100mL에 녹입니다. 육수를 20분 동안 오토클레이브하고 실온으로 식힙니다. 50mL 플라스크에 TSB 50mL를 추가합니다.
- 박테리아를 한천 플레이트로 옮깁니다 27,28. 루프를 사용하여 한천 플레이트에서 박테리아 군체를 선택합니다. 루프를 부드럽게 소용돌이쳐 루프의 내용을 플라스크에 추가합니다. 150rpm에서 궤도 쉐이킹으로 30°C ± 1°C에서 밤새 플라스크를 배양합니다.
알림: 온도와 흔들림은 변형률 유형에 따라 조정해야 합니다. - TSB 47.5mL가 함유된 또 다른 50mL 플라스크를 준비합니다. 2.5mL의 익새 배양액을 플라스크로 옮깁니다. OD600 = 0.6 0.05 (중간 지수 단계에 도달하기 위해)에 도달하기 위해 5-6 시간 동안 배양하십시오.
- 배양물을 50mL 멸균 원심분리 튜브로 옮깁니다. 2,200 × g 에서 10분 동안 배양을 원심분리합니다. 튜브 바닥에 모인 세포가 손실되지 않도록 육수를 부드럽게 디캔팅합니다.
- 동일한 튜브에 5mL의 인산염 완충 식염수(PBS)를 추가하고 2,200×g에서 10 분 동안 원 심분리합니다. 이 단계를 3회 반복합니다.
- 피펫을 사용하여 세포의 OD600 을 0.6 0.05로 조정하여 적절한 양의 PBS 용액을 적정합니다. 나중에 사용할 수 있도록 추가된 PBS 양을 정확하게 기록합니다.
- 20% v/v에 도달하기 위하여 현탁액에 적정 글리세롤. 튜브와 소용돌이를 15초 동안 캡을 씌웁니다.
- 현탁액 1.5mL를 2mL 극저온 용액으로 옮깁니다. 냉동 식품을 적절한 상자에 넣고 -80°C의 냉동고에 보관합니다.
2. 하룻밤 문화를 준비하십시오.
- 박테리아 스톡이 포함된 냉동 식품을 실온에서 30분 동안 또는 내용물이 액체가 될 때까지 해동시킵니다.
알림: 해동 과정을 가속화하지 않는 것이 좋습니다. - 멸균된 튜브에 TSB 10mL를 준비합니다. 50μL의 스톡 현탁액을 튜브로 옮깁니다. 150rpm에서 궤도 쉐이킹으로 30°C ± 1°C에서 하룻밤(16-18시간) 튜브를 배양합니다.
3. 접종물을 준비합니다.
- 멸균된 TSB 10mL가 든 튜브를 준비합니다. 500μL의 야간 배양을 튜브로 옮깁니다. OD600 = 0.6 0.05 (또는 중간 지수 단계에 도달)에 도달하기 위해 6-8 시간 동안 배양하십시오.
- 배양액을 15mL 원심분리 튜브로 옮깁니다. 튜브를 2,200 × g 에서 10분 동안 원심분리합니다. 육수를 부드럽게 디캔팅합니다.
- PBS 5mL를 넣고 2,200×g에서 10 분 동안 원 심분리합니다. 이 단계를 3회 반복합니다.
- 피펫을 사용하여 PBS 용액을 적정하여 셀의 OD600 을 0.6 0.05로 조정합니다. 나중에 사용할 수 있도록 추가된 PBS 양을 정확하게 기록합니다.
4. 성장 배지를 마이크로플레이트 리더로 옮기기
- 뚜껑이 있는 투명하고 멸균되며 바닥이 평평한 웰 플레이트(즉, 96웰 플레이트, 6웰 플레이트, 12웰 플레이트)를 선택합니다.
- 1.10단계에서 설명한 대로 프로토콜을 실행하기 전에 DI water를 사용하여 마이크로플레이트의 경로 길이 보정을 측정합니다.
- 선택한 웰 플레이트를 기반으로 참조 테이블을 그려 로드하기 전에 샘플의 위치를 지정합니다. 예를 들어, 96웰 플레이트에 대해 8 x 12 테이블을 그립니다.
참고: 참조 테이블은 배양 및 결과 수집 단계에서 혼동을 방지합니다. - 뚜껑이 있는 마이크로플레이트, 1,000 μL 및 200 μL, 1,000 μL 및 200 μL 피펫 팁, 여러 개의 극저온 여과제, 25mL 비커, 성장 배지 및 접종물을 생물 안전 캐비닛에 넣습니다.
참고: 언급된 항목은 무균 상태로 생물 안전 캐비닛으로 옮겨야 합니다. - 비커에 성장 배지 10mL를 추가합니다. 계산된 접종량을 성장 배지와 함께 비커로 옮깁니다.
참고: 이 프로토콜의 접종 비율은 1%와 5%입니다. 예를 들어, 5% 및 1% 접종 비율을 달성하려면 500μL 및 100μL의 접종물을 성장 배지 9.5mL 및 9.9mL에 각각 첨가합니다. - 접종물을 추가한 후 비커를 부드럽게 흔들어 균일한 분포를 얻습니다. 200μL의 접종된 배지를 참조 표를 사용하여 지정된 웰에 분배합니다. 성장 배지 200μL를 추가하여 대조군 역할을 할 웰을 3개 이상 선택합니다(접종물 없음).
알림: 제어 웰이 참조 표에 명확하게 표시되어 있는지 확인하십시오. 이 우물에서 성장(오염)이 관찰되면 실험을 반복해야 합니다. - 증발 속도가 더 높은 가장자리 웰에 200μL의 DI water를 분배합니다. 안전 캐비닛에서 96웰 플레이트를 제거하기 전에 뚜껑을 부드럽게 놓으십시오. 96웰 플레이트를 플레이트 리더에 조심스럽게 놓고 갑작스러운 움직임이 없도록 합니다.
5. 마이크로플레이트 리더 설정
- 마이크로플레이트 리더를 켠 다음 임베디드 소프트웨어를 사용하여 맞춤형 프로토콜을 생성합니다. 작업 관리자에서 프로토콜을 열고 새로 만들기 | 표준 프로토콜. 절차를 열고 다음과 같이 설정을 조정합니다.
- Set Temperature를 사용하여 온도를 30°C(또는 목표 값)로 설정합니다.
- Incubator On을 클릭한 다음 Gradient를 1 °C로 설정합니다.
알림: 뚜껑의 결로 현상으로 인한 신뢰할 수 없는 데이터를 방지하려면 그라디언트 가 1°C로 설정되어 있는지 확인하십시오. - Preheat(예열) 옵션을 선택하여 웰 플레이트를 따라 균일한 열 분포를 보장합니다.
- Start Kinetic을 열고 실행 시간(예: 12:00:00 또는 24:00:00 등) 및 00:30:00(30분) 또는 01:00:00(1시간을 나타냄)에 대한 판독 간격을 사용하여 원하는 배양 기간을 선택합니다.
참고: 각 96웰 플레이트를 읽는 데 약 1분이 걸립니다. - Shaking 설정을 다음과 같이 설정하십시오 : Shake를 엽니 다. Shake Mode를 Linear로 설정합니다. Continuous Shake를 클릭하십시오. 을 클릭하고 주파수를 567cpm(또는 원하는 값)으로 조정합니다.
- OD 읽기 설정을 다음과 같이 설정합니다: Read | 흡광도; Endpoint/Kinetic 및 Monochromators를 선택합니다. 파장을 600nm로 설정하십시오. 을 클릭하고 Validate(검증) | Save(저장)를 선택하여 적절한 이름과 날짜의 새 프로토콜로 저장합니다.
6. 성장 데이터 기록
- 작업 관리자 | 지금 읽어보세요.
- 저장된 프로토콜을 선택한 다음 확인을 클릭하여 프로토콜을 시작합니다.
- 데이터 분석을 위해 판독값을 저장합니다.
7. 데이터 분석
- OD600 결과(성장 데이터)를 스프레드시트 형식으로 전송합니다. 결과 섹션에서 각 우물에 대한 이미지 옵션을 클릭하여 최종 성장 곡선을 관찰합니다.
- 스프레드시트 파일을 열고 참조 테이블을 기반으로 기록된 데이터를 재정렬합니다. 다른 읽기에서 빈 읽기의 평균값을 뺍니다.
참고: 공백은 시간 0에서의 OD 판독값입니다. 이 프로토콜에서는 OD600 값이 계산을 위해 고려됩니다. 세포 밀도는 OD600 대 세포 수에 대한 검량선을 사용할 수 있는 경우 사용할 수 있습니다.
8. 성장률 결정
- 전송된 데이터는 1.6단계에서 생성한 스프레드시트에서 사용합니다. 아래 설명된 방법과 방정식 (1)을 사용하여 성장률을 평가합니다.
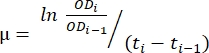 (1)
(1)
여기 서, μ은 성장률이고, ODi는 각 시점에서의 OD(600)이고, ODi-1은 마지막 시점에서의 초기 OD(600) 값이고, ti-1은 언급된 두 지점 사이의 시간 차이이다(예를 들어, 30분 시간 간격에 대한 0.5, 1h 시간 간격에 대한 1). - 원하는 데이터 분석 소프트웨어를 사용하여 5개 연속 성장률의 평균과 표준 편차를 계산합니다. 표준 편차가 가장 낮은 가장 큰 평균은 최대 성장률 (μm) 12입니다.
9. 공간 편향 판단
참고: 마이크로플레이트 리더와 플레이트는 결과에 편향을 유발합니다. 결과의 신뢰성과 재현성을 보장하기 위해 특정 마이크로플레이트 리더에 사용된 플레이트의 공간 편향을 평가하는 것이 중요합니다. 이를 위해 다음 단계를 사용하십시오.
- 1.2-1.6단계를 반복합니다.
- 데이터를 기록하고 1.7단계와 1.8단계에 따라 분석하여 각 유정의 성장률을 계산합니다.
- 96웰 플레이트의 성장률 변화를 시각화하기 위해 히트맵을 만듭니다.
- 통계 분석을 수행하여( 보충 표 참조) 우물 간에 큰 차이가 없는지 확인합니다.
참고: 모든 웰에서 동일한 유형의 박테리아와 동일한 접종물 크기를 사용하는 것이 필수적입니다.
10. OD 판독 검증 및 경로 길이 보정 계수
참고: 경로 길이 보정 계수를 결정하는 것은 데이터를 검증하고 다양한 장치에서 신뢰성과 유효성을 보장하는 데 중요합니다.
- 1.2단계 및 1.3단계를 사용하여 B. mycoides의 야간 접종을 준비합니다.
- 멸균된 50mL 플라스크 3개를 준비하고 각 플라스크에 49.5mL의 새 TSB를 추가합니다. 500μL의 접종물을 안전 캐비닛의 각 플라스크에 무균 방식으로 첨가합니다. 접종되지 않은 TSB 50mL를 대조군 역할을 하는 멸균된 플라스크에 추가합니다.
- 플라스크를 30°C 및 150rpm으로 배양합니다. 30분마다 각 플라스크에서 샘플을 무균 상태로 채취하고 분광 광도계에서 OD600 을 읽고 기록합니다.
- 25mL 비커에 9.9mL의 TSB를 추가합니다. 비커에 접종물 100μL를 넣고 부드럽게 흔듭니다.
- 비커에 있는 혼합물 200μL를 96웰 플레이트의 선택 컬럼에 있는 6개의 웰에 추가합니다. 200μL의 비접종 TSB를 대조군으로 3개의 웰에 추가합니다. 가장자리 웰에 200μL의 DI 물을 채웁니다.
- 뚜껑을 놓고 96웰 플레이트를 마이크로플레이트 리더로 부드럽게 옮깁니다. 1.5단계 및 1.6단계에 따라 성장 데이터를 기록합니다.
- 마이크로플레이트 리더의 OD600 을 분광광도계의 OD600 에 플로팅합니다. 기울기는 경로 길이 보정 계수(pathlength correction factor)라고 하는 두 판독값 세트 사이의 오프셋입니다.
참고: 인큐베이터와 마이크로플레이트 리더 인큐베이션은 같은 날에 동시에 수행해야 합니다.
결과
OD 판독 검증 및 경로 길이 보정 계수
B. mycoides 배양의 분할 샘플을 서로 다른 시점에서 채취하여 마이크로플레이트 리더와 분광 광도계를 사용하여 측정했습니다(그림 1A). 이 단계는 여러 장치에서 결과를 검증하기 위해 수행되었습니다. OD600 데이터는 상관관계가 있었지만 일치하지 않았습니다(그림 1B<...
토론
마이크로플레이트 리더를 사용하면 일관되고 반복 가능한 성장률을 얻을 수 있습니다. 이 기술은 인적 오류를 최소화하고 높은 처리량의 샘플링을 가능하게 합니다. 샘플당 배양량이 적기 때문에 이 접근 방식은 플라스크 또는 시험관을 사용한 세포 계수에 대한 매력적이고 저렴한 대안입니다. 마이크로플레이트 리더는 큰 표본 크기를 허용하여 통계적 검정력을 높이고...
공개
저자는 선언할 이해 상충이 없습니다.
감사의 말
이 작업은 자연 과학 및 공학 연구위원회 (NSERC) / 수질 및 처리 분야의 핼리팩스 물 산업 연구 의장 (보조금 번호)의 자금 지원을 받았습니다. IRCPJ 349838-16). 저자 팀은 또한 이 기사를 검토하는 데 Anita Taylor의 도움을 받은 것에 대해 감사의 뜻을 전합니다.
자료
| Name | Company | Catalog Number | Comments |
| Centrifuge | Eppendorf | 5810 R | |
| Centrifuge tubes - 15 mL | ThermoFisher- Scientific | 339650 | Sterile |
| Centriguge tubes - 50 mL | ThermoFisher- Scientific | 339652 | Sterile |
| Disposable inoculating loop , 10 µL | Cole-Parmer | UZ-06231-08 | Sterile |
| Erlenmeyer flasks - 250 mL | Cole-Parmer | UZ-34502-59 | Glass |
| Isopropanol | ThermoFisher- Scientific | 396982500 | ≥99.0 |
| Phosphate Buffer Saline | Sigma-Aldrich | P4417 | |
| Pipett tips 1,000 µL | ThermoFisher- Scientific | UZ-25001-76 | |
| Pipett tips 10 mL | ThermoFisher- Scientific | UZ-25001-83 | |
| Pipett tips 200 µL | ThermoFisher- Scientific | UZ-25001-85 | |
| Pipett tips 5 mL | ThermoFisher- Scientific | UZ-25001-80 | |
| Pipettor 1,000 µL | Cole-Parmer | UZ-07909-11 | |
| Pipettor 10 mL | Cole-Parmer | UZ-07909-15 | |
| Pipettor 200 µL | Cole-Parmer | UZ-07909-09 | |
| Pipettor 5 mL | Cole-Parmer | UZ-07859-30 | |
| Tryptic Soy Broth | Millipore | 22091 | Suitable for microbiology |
참고문헌
- Reuß, D. R., et al. Large-scale reduction of the Bacillus subtilis genome: Consequences for the transcriptional network, resource allocation, and metabolism. Genome Research. 27 (2), 289-299 (2017).
- Sparkes, A., et al. Towards Robot Scientists for autonomous scientific discovery. Automated Experimentation. 2 (1), (2010).
- Zwietering, M. H., Jongenburger, I., Rombouts, F. M., van t Riet, K. Modeling of the bacterial growth curve. Applied and Environmental Microbiology. 56 (6), 1875-1881 (1990).
- Pla, M., Oltra, S., Esteban, M., Andreu, S., Palop, A. Comparison of primary models to predict microbial growth by the plate count and absorbance methods. BioMed Research International. 2015 (6), 1-14 (2015).
- Choe, D., et al. Adaptive laboratory evolution of a genome-reduced Escherichia coli. Nature Communications. 10 (1), 935 (2019).
- Dykhuizen, D. E., Dean, A. M. Enzyme activity and fitness: Evolution in solution. Trends in Ecology and Evolution. 5 (8), 257-262 (1990).
- McDonald, M. J. Microbial experimental evolution - A proving ground for evolutionary theory and a tool for discovery. EMBO Reports. 20 (8), 1-14 (2019).
- Lum, P. Y., et al. Discovering modes of action for therapeutic compounds using a genome-wide screen of yeast heterozygotes. Cell. 116 (1), 121-137 (2004).
- Cagnon, C., et al. Development of a forward genetic screen to isolate oil mutants in the green microalga Chlamydomonas reinhardtii. Biotechnology for Biofuels. 6 (1), 178 (2013).
- Stevenson, K., McVey, A. F., Clark, I. B. N., Swain, P. S., Pilizota, T. General calibration of microbial growth in microplate readers. Scientific Reports. 6 (1), 38828 (2016).
- Matlock, B. C., Beringer, R. W., Ash, D. L., Page, A. F., Allen, M. W. Differences in Bacterial Optical Density Measurements between Spectrophotometers. Technical Note. ThermoScientific. , (2011).
- Hall, B. G., Acar, H., Nandipati, A., Barlow, M. Growth rates made easy. Molecular Biology and Evolution. 31 (1), 232-238 (2014).
- Rolfe, M. D., et al. Lag phase is a distinct growth phase that prepares bacteria for exponential growth and involves transient metal accumulation. Journal of Bacteriology. 194 (3), 686-701 (2012).
- Djordjevic, D., Wiedmann, M., Mclandsborough, L. A. Microtiter plate assay for assessment of Listeria monocytogenes biofilm formation. Applied and Environmental Microbiology. 68 (6), 2950-2958 (2002).
- O'Toole, G. A. Microtiter dish biofilm formation assay. Journal of Visualized Experiments. (47), e2437 (2011).
- Campbell, J. High-throughput assessment of bacterial growth inhibition by optical density measurements. Current Protocols in Chemical Biology. 3 (3), 1-20 (2012).
- Toussaint, M., Conconi, A. High-throughput and sensitive assay to measure yeast cell growth: A bench protocol for testing genotoxic agents. Nature Protocols. 1 (4), 1922-1928 (2006).
- Goughenour, K. D., Balada-Llasat, J. -. M., Rappleye, C. A. Quantitative microplate-based growth assay for determination of antifungal susceptibility of Histoplasma capsulatum yeasts. Journal of Clinical Microbiology. 53 (10), 3286-3295 (2015).
- Qiu, T. A., et al. Growth-based bacterial viability assay for interference-free and high-throughput toxicity screening of nanomaterials. Analytical Chemistry. 89 (3), 2057-2064 (2017).
- Kurokawa, M., Precise Ying, B. -. W. Precise, high-throughput analysis of bacterial growth. Journal of Visualized Experiments. (127), e56197 (2017).
- Bredit, F., Romick, T. L., Fleming, H. P. A rapid method for determination of bacterial growth kinetics. Journal of Rapid Methods and Automation in Microbiology. 3, 59-68 (1994).
- Delaney, N. F., et al. Development of an optimized medium, strain and high-throughput culturing methods for Methylobacterium extorquens. PLOS ONE. 8 (4), 62957 (2013).
- Haire, T. C., et al. Robust microplate-based methods for culturing and in vivo phenotypic screening of Chlamydomonas reinhardtii. Frontiers in Plant Science. 9, 235 (2018).
- McBirney, S. E., Trinh, K., Wong-Beringer, A., Armani, A. M. Wavelength-normalized spectroscopic analysis of Staphylococcus aureus and Pseudomonas aeruginosa growth rates. Biomedical Optics Express. 7 (10), 4034 (2016).
- Sieben, M., Giese, H., Grosch, J. H., Kauffmann, K., Büchs, J. Permeability of currently available microtiter plate sealing tapes fail to fulfil the requirements for aerobic microbial cultivation. Biotechnology Journal. 11 (12), 1525-1538 (2016).
- Zimmermann, H. F., John, G. T., Trauthwein, H., Dingerdissen, U., Huthmacher, K. Rapid evaluation of oxygen and water permeation through microplate sealing tapes. Biotechnology Progress. 19 (3), 1061-1063 (2003).
- Abkar, L., Gagnon, G. A. Biological responses to P-limitation in indigenous bacteria isolated from drinking water. AWWA Water Science. 3 (5), 1248 (2021).
- Sanders, E. R. Aseptic laboratory techniques: Plating methods. Journal of Visualized Experiments. (63), e3064 (2012).
- Hart, S. F. M., Skelding, D., Waite, A. J., Burton, J. C., Shou, W. High-throughput quantification of microbial birth and death dynamics using fluorescence microscopy. Quantitative Biology. 7 (1), 69-81 (2019).
- Periago, P. M., Abee, T., Wouters, J. A. Analysis of the heat-adaptive response of psychrotrophic Bacillus weihenstephanensis. International Journal of Food Microbiology. 79 (1-2), 17-26 (2002).
- Mira, P., Barlow, M., Meza, J. C., Hall, B. G. Statistical package for growth rates made easy. Molecular Biology and Evolution. 34 (12), 3303-3309 (2017).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유