Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Verbesserte Reproduzierbarkeit und Präzision der Hochdurchsatz-Quantifizierung von Daten zum Bakterienwachstum mit einem Mikroplatten-Reader
In diesem Artikel
Zusammenfassung
Hier wird ein Hochdurchsatzprotokoll vorgestellt, um Wachstumsdaten zu messen, einschließlich Wachstumskurven, Wachstumsrate und maximaler Wachstumsrate. Das Protokoll wurde anhand von zwei Biofilm-produzierenden Bakterien verifiziert und validiert. Die Ergebnisse und der in dieser Studie angewandte Ansatz können auf andere Hochdurchsatzprotokolle mit Mikroplatten-Readern ausgeweitet werden.
Zusammenfassung
Ziel dieser Studie war es, ein wiederholbares, zuverlässiges Hochdurchsatzprotokoll zu entwickeln, um das Bakterienwachstum in 96-Well-Platten zu überwachen und die maximale Wachstumsrate zu analysieren. Es wurden die Wachstumskurven und maximalen Wachstumsraten von zwei Bakterienarten bestimmt. Untersucht wurden u.a. (i) Deckelkondensation, (ii) Weglängenkorrektur, (iii) Inokulationsgröße, (iv) Abtastzeitintervall und (v) räumliche Verzerrung. Die Wiederholbarkeit des Protokolls wurde mit drei unabhängigen technischen Replikationen bewertet, mit einer Standardabweichung von 0,03 zwischen den Durchläufen. Die maximalen Wachstumsraten von Bacillus mycoides und Paenibacillus tundrae betrugen (mittlere ± SD) 0,99 h-1 ± 0,03 h-1 bzw. 0,85 h-1 ± 0,025 h-1. Diese Bakterien sind aufgrund ihrer Affinität, miteinander zu verklumpen, optisch schwieriger zu überwachen. Diese Studie zeigt, wie wichtig die Inokulationsgröße, die Weglängenkorrektur, die Deckelerwärmung, die Probenahmezeitintervalle und die räumliche Verzerrung der Well-Platte sind, um zuverlässige, genaue und reproduzierbare Daten auf Mikroplatten-Readern zu erhalten. Das entwickelte Protokoll und seine Verifizierungsschritte können mit Mikroplatten-Readern und Hochdurchsatzprotokollen auf andere Methoden erweitert werden, wodurch die inhärenten Fehler und Materialkosten der Forscher reduziert werden.
Einleitung
Das wachsende Interesse an der Multi-Omics-Manipulation, einschließlich Mechanismus- und Stoffwechselstudien von Bakterien, unterstreicht die Bedeutung von Hochdurchsatz- und automatisierten Methoden wie der Aufzeichnung von Wachstumsdaten 1,2. Wachstumsdaten, die kinetische Parameter wie maximale Wachstumsraten umfassen, können helfen, bakterielle Reaktionen auf verschiedene physikalische, chemische und antibakterielle Bedingungen zu charakterisieren. Daten zur Wachstumsrate sind eine Standard-Antwortvariable, die verwendet wird, um potenzielle Genotyp-Phänotyp-Verknüpfungenaufzudecken 1 oder die mikrobielle Sicherheit und Haltbarkeit von Lebensmitteln anzuzeigen 3,4. Techniken wie die adaptive Laborevolution 5,6,7, das genomweite Screening, bestimmte chemische Assays8 und verschiedene genetische Vorwärts-Screenings9 stützen sich auf Wachstumsraten, um die Ergebnisse zu bewerten.
Messungen der optischen Dichte (OD) von Bakterienkulturen sind eine mikrobiologische Standardmethode zur Überwachung des Bakterienwachstums. OD-Messungen werden oft bei einer Wellenlänge von 600 nm aufgezeichnet, wobei sie sich auf Lichtstreuung und die Zelldichtestützen 10,11. Das Beer-Lambert-Gesetz erklärt die Abhängigkeit der OD-Werte von der Konzentration (d. h. Zelldichte, Zellzahl), der Weglänge und dem Absorptionskoeffizienten. Die Geometrie und das optische System eines Spektralphotometers beeinflussen die OD-Messwerte11. Klassische Methoden der Außendurchmessermessung können sehr zeit- und arbeitsintensiv sein, und die Daten können eine Vielzahl menschlicher Fehler enthalten. In diesem Protokoll wird ein Mikroplatten-Reader verwendet, um die Analysezeit12,13 und die Wahrscheinlichkeit einer biologischen Kontamination zu verringern. Die Hochdurchsatzanalyse mit Mikroplatten-Readern wird in verschiedenen Bereichen der Mikrobiologie eingesetzt, z. B. beim Screening von Biofilm-produzierenden Bakterien14,15, bei der Hemmung des bakteriellen Wachstums16, beim Wachstum von Hefezellen17, bei der Bestimmung der Empfindlichkeit gegenüber Antimykotika18 und beim Toxizitätsscreening von Nanomaterialien19.
Einige Forscher haben Protokolle zur bakteriellen Wachstumsrate mit einem Mikroplatten-Reader veröffentlicht 12,20,21. Ein gründliches Protokoll, das die Zuverlässigkeit der gesammelten Daten untersucht, ist jedoch noch nicht vollständig etabliert. Es wird berichtet, dass Faktoren wie die Art der Spezies 22,23,24 und die Dichtungsbänder die Wiederholgenauigkeit aufgrund der Unzulänglichkeit des Sauerstofftransfers in einer 96-Well-Platte beeinflussen 25,26. Delaney et al. berichteten über große Cluster von Methylorubrum extorquens (Wildtyp-Stamm) im Wachstumsmedium bei Verwendung eines Mikroplatten-Readers, was zu extrem verrauschten Wachstumsdaten führte24. Das Problem wurde gelöst, indem die Gene entfernt wurden, die mit der Biofilmproduktion verbunden sind24. Aufgrund der Sekretion extrazellulärer polymerer Substanzen haben Biofilm-produzierende Bakterien eine größere Affinität, miteinander zu verschmelzen und Zellcluster zu bilden. Daher ist es schwieriger, ihr Wachstum mit Hilfe von Lichtstreutechniken (z. B. Spektralphotometer und Mikroplatten-Reader) zu überwachen.
Dieses Protokoll zielt darauf ab, Schritte zu etablieren, um reproduzierbare Daten in einem Hochdurchsatzverfahren mit einem Mikroplatten-Reader zu erhalten. Bacillus mycoides und Paenibacillus tundrae wurden aufgrund ihres schnellen Wachstums und ihrer Fähigkeit, Biofilme zu produzieren, verwendet, die bei manuellen und automatisierten Ansätzen traditionell eine Herausforderung darstellen. Faktoren wie (i) Weglängenkorrektur, (ii) Kondensation auf dem Deckel, (iii) Inokulumgröße, (iv) Abtastzeitintervall und (v) räumliche Verzerrung wurden untersucht, um die Zuverlässigkeit und Reproduzierbarkeit der Daten zu bewerten. Dieses Protokoll enthält Schritte zur genauen Überwachung des Bakterienwachstums und zur Messung spezifischer Wachstumsraten mit einem Mikroplatten-Reader.
Protokoll
HINWEIS: Alle Schritte in diesem Protokoll müssen unter sterilen Bedingungen (d. h. zwischen zwei Flammen oder einer Biosicherheitswerkbank) durchgeführt werden. Alle Materialien und Werkzeuge werden für 20 min autoklaviert. In der Materialtabelle finden Sie Details zu allen Materialien, Geräten und Software, die in diesem Protokoll verwendet werden. Behandschuhte Hände werden desinfiziert, mindestens 1 Minute lang mit Händedesinfektionsmittel oder 70%iger Alkohollösung feucht gehalten und danach nicht aus dem Sicherheitsschrank genommen. Andernfalls muss der Desinfektionsvorgang wiederholt werden, bevor die Hände wieder in den Sicherheitsschrank eingeführt werden. ACHTUNG: Stellen Sie sicher, dass das Desinfektionsmittel vollständig verdunstet ist, bevor Sie eine offene Flamme verwenden.
HINWEIS: Zwei Bakterien wurden, wie zuvor erläutert27 , aus der Trinkwasserbiofiltration auf ihre Fähigkeit zur Bildung von Biofilm isoliert. Sie wurden durch die vollständige 16S-rRNA-Sequenzierung identifiziert und als Bacillus mycoides (SAMN10518261) und Paenibacillus tundrae (SAMN10452279) an das NCBI übermittelt.
1. Aufbereitung der Bakterienbrühe in Glycerin
- Wiegen Sie 3 g Tryptische Sojabrühe (TSB) Pulver ab und lösen Sie es in 100 ml destilliertem Wasser auf. Die Brühe 20 min autoklavieren und auf Zimmertemperatur abkühlen lassen. 50 ml TSB in einen 50-ml-Kolben geben.
- Die Bakterien werden auf Agarplattenübertragen 27,28. Entnehmen Sie mit einer Schlaufe eine Bakterienkolonie von der Agarplatte. Geben Sie den Inhalt der Schlaufe in den Kolben, indem Sie die Schlaufe vorsichtig schwenken. Der Kolben wird über Nacht bei 30 °C ± 1 °C unter Orbitalschütteln bei 150 U/min inkubiert.
HINWEIS: Temperatur und Schütteln müssen je nach Dehnungstyp angepasst werden. - Bereiten Sie einen weiteren 50-ml-Kolben mit 47,5 mL TSB vor. 2,5 ml der Übernachtkultur werden in den Kolben überführt. Inkubieren Sie es 5-6 Stunden lang, um OD600 = 0,6 0,05 zu erreichen (um zur mittleren exponentiellen Phase zu gelangen).
- Übertragen Sie die Kultur in ein sterilisiertes 50-ml-Zentrifugenröhrchen. Zentrifugieren Sie die Kultur bei 2.200 × g für 10 min. Dekantieren Sie die Brühe vorsichtig, um zu vermeiden, dass die am Boden der Tube angesammelten Zellen verloren gehen.
- Geben Sie 5 ml phosphatgepufferte Kochsalzlösung (PBS) in dasselbe Röhrchen und zentrifugieren Sie sie 10 Minuten lang bei 2.200 × g . Wiederholen Sie diesen Schritt 3x.
- Der OD600 der Zellen wird auf 0,6 0,05 eingestellt, indem eine Pipette verwendet wird, um eine ausreichende Menge PBS-Lösung zu titrieren. Zeichnen Sie die hinzugefügte Menge an PBS für die zukünftige Verwendung genau auf.
- Titrieren Sie Glycerin in die Suspension, um 20 % v/v zu erreichen. Verschließen Sie das Rohr und den Wirbel für 15 s.
- 1,5 mL der Suspension in 2 mL Kryoröhrchen überführen. Legen Sie die Kryoröhrchen in eine geeignete Schachtel und lagern Sie sie in einem Gefrierschrank bei -80 °C.
2. Bereiten Sie die Kultur über Nacht vor.
- Lassen Sie die Kryofläschchen mit Bakterienfond 30 Minuten lang bei Raumtemperatur auftauen oder bis der Inhalt flüssig ist.
HINWEIS: Es wird empfohlen, den Auftauvorgang nicht zu beschleunigen. - Bereiten Sie ein sterilisiertes Röhrchen mit 10 mL TSB vor. Übertragen Sie 50 μl der Stammsuspension in das Röhrchen. Inkubieren Sie das Röhrchen über Nacht (16-18 h) bei 30 °C ± 1 °C und Orbitalschütteln bei 150 U/min.
3. Bereiten Sie das Inokulum vor.
- Bereiten Sie ein Röhrchen mit 10 mL sterilisiertem TSB vor. Übertragen Sie 500 μl der Nachtkultur in das Röhrchen. Inkubieren Sie es 6-8 Stunden lang, um OD600 = 0,6 0,05 zu erreichen (oder in die mittlere exponentielle Phase zu gelangen).
- Übertragen Sie die Kultur in ein 15-ml-Zentrifugenröhrchen. Zentrifugieren Sie das Röhrchen bei 2.200 × g für 10 min. Die Brühe vorsichtig dekantieren.
- Fügen Sie 5 mL PBS hinzu und zentrifugieren Sie 10 Minuten lang bei 2.200 × g . Wiederholen Sie diesen Schritt 3x.
- Der OD600 der Zellen wird auf 0,6 0,05 eingestellt, indem die PBS-Lösung mit einer Pipette titriert wird. Zeichnen Sie die hinzugefügte Menge an PBS für die zukünftige Verwendung genau auf.
4. Übertragen des Wachstumsmediums auf den Mikroplatten-Reader
- Wählen Sie eine transparente, sterile Well-Platte mit flachem Boden und Deckel (z. B. 96-Well-Platte, 6-Well-Platte, 12-Well-Platte).
- Messen Sie die Weglängenkorrektur für die Mikroplatte mit DI-Wasser, bevor Sie das Protokoll ausführen, wie in Schritt 1.10 beschrieben.
- Zeichnen Sie eine Referenztabelle basierend auf der ausgewählten Well-Platte, um die Positionen der Proben vor dem Laden anzugeben. Zeichnen Sie z. B. eine 8 x 12-Tabelle für eine 96-Well-Platte.
HINWEIS: Die Referenztabelle vermeidet Verwirrung während der Inkubations- und Ergebniserfassungsschritte. - Legen Sie folgende Gegenstände in eine Biosicherheitswerkbank: eine Mikroplatte mit Deckel, einen Pipettierer mit 1.000 μl und 200 μl, Pipettenspitzen mit 1.000 μl und 200 μl, mehrere Kryoröhrchen, ein 25-ml-Becherglas, das Wachstumsmedium und das Inokulum.
HINWEIS: Die genannten Gegenstände sollten aseptisch in die Biosicherheitswerkbank überführt werden. - Gib 10 ml Wachstumsmedium in das Becherglas. Übertragen Sie die berechnete Menge Inokulum mit dem Wachstumsmedium in das Becherglas.
HINWEIS: Die Inokulumverhältnisse in diesem Protokoll betragen 1 % und 5 %. Um beispielsweise ein Inokulumverhältnis von 5 % und 1 % zu erreichen, fügen Sie 500 μl und 100 μl des Inokulums zu 9,5 mL bzw. 9,9 mL des Wachstumsmediums hinzu. - Nachdem Sie das Inokulum hinzugefügt haben, schütteln Sie das Becherglas vorsichtig, um eine gleichmäßige Verteilung zu erhalten. Dispensieren Sie 200 μl des inokulierten Mediums in die dafür vorgesehenen Vertiefungen unter Verwendung der Referenztabelle. Wählen Sie mindestens drei Vertiefungen als Kontrollen, indem Sie 200 μl des Wachstumsmediums (ohne Inokulum) hinzufügen.
HINWEIS: Stellen Sie sicher, dass die Kontrollwells in der Referenztabelle deutlich gekennzeichnet sind. Wenn in diesen Vertiefungen ein Wachstum (Kontamination) beobachtet wird, sollte der Versuch wiederholt werden. - Geben Sie 200 μl DI-Wasser in die Randvertiefungen, wo die Verdampfungsrate höher ist. Setzen Sie den Deckel vorsichtig auf, bevor Sie die 96-Well-Platte aus dem Sicherheitsschrank nehmen. Legen Sie die 96-Well-Platte vorsichtig in den Plattenleser und achten Sie darauf, dass keine plötzlichen Bewegungen auftreten.
5. Einstellungen des Mikroplatten-Readers
- Schalten Sie den Mikroplatten-Reader ein und verwenden Sie dann die eingebettete Software, um das benutzerdefinierte Protokoll zu erstellen. Öffnen Sie im Task-Manager das Protokoll und wählen Sie Create New | Standard-Protokoll. Öffnen Sie Prozedur und passen Sie die Einstellungen wie folgt an:
- Stellen Sie die Temperatur mit "Temperatur einstellen" auf 30 °C (oder die Zielwerte) ein.
- Klicken Sie auf Inkubator Ein und stellen Sie dann den Gradienten auf 1 °C ein.
HINWEIS: Um unzuverlässige Daten aufgrund von Kondensation auf dem Deckel zu vermeiden, stellen Sie sicher, dass der Gradient auf 1 °C eingestellt ist. - Wählen Sie die Option Vorheizen , um eine gleichmäßige Wärmeverteilung entlang der Well-Platte zu gewährleisten.
- Öffnen Sie Start Kinetic und wählen Sie die gewünschte Inkubationsdauer anhand der Laufzeit (z. B. 12:00:00 oder 24:00:00 usw.) und des Intervalls zwischen den Messwerten für 00:30:00 (entspricht 30 min) oder 01:00:00 (entspricht 1 h).
HINWEIS: Das Ablesen jeder 96-Well-Platte dauert ca. 1 min. - Stellen Sie die Schütteleinstellungen wie folgt ein: Öffnen Sie Shake; Stellen Sie den Schüttelmodus auf Linear ein. Klicken Sie auf Kontinuierliches Schütteln; und stellen Sie die Frequenz auf 567 cpm (oder einen beliebigen Wert) ein.
- Stellen Sie die Einstellungen für den OD-Messwert wie folgt ein: Klicken Sie auf Lesen | Absorption; Wählen Sie Endpunkt/Kinetik und Monochromatoren; Stellen Sie die Wellenlänge auf 600 nm ein. und klicken Sie auf Validieren | Speichern , um es als neues Protokoll mit dem entsprechenden Namen und Datum zu speichern.
6. Aufzeichnung von Wachstumsdaten
- Öffnen Sie den Task-Manager | Jetzt lesen.
- Wählen Sie das gespeicherte Protokoll aus und klicken Sie dann auf OK , um das Protokoll zu starten.
- Speichern Sie die Messwerte für die Datenanalyse.
7. Datenanalyse
- Übertragen Sie die Ergebnisse des OD600 (Wachstumsdaten) in das Tabellenformat. Klicken Sie im Ergebnisbereich auf die Bildoption für jedes Bohrloch, um die endgültigen Wachstumskurven zu beobachten.
- Öffnen Sie die Tabellenkalkulationsdatei und ordnen Sie die aufgezeichneten Daten basierend auf der Referenztabelle neu an. Subtrahieren Sie den Mittelwert der leeren Lesevorgänge von den anderen Lesevorgängen.
HINWEIS: Leerzeichen sind die OD-Messwerte zum Zeitpunkt Null. In diesem Protokoll werden die Werte des OD600 für die Berechnung berücksichtigt. Die Zelldichte kann verwendet werden, wenn die Kalibrierkurve für OD600 vs. Zellzahl verfügbar ist.
8. Bestimmung der Wachstumsrate
- Verwenden Sie die übertragenen Daten in der in Schritt 1.6 erstellten Tabelle. Verwenden Sie die unten beschriebene Methode und Gleichung (1), um die Wachstumsrate zu bewerten.
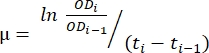 (1)
(1)
Dabei ist μ die Wachstumsrate, ODi ist der OD600 in jedem Zeitpunkt, ODi-1 ist der anfängliche OD600-Wert im letzten Zeitpunkt, ti und ti-1 sind die Zeitdifferenzen zwischen den beiden genannten Punkten (z. B. 0,5 für 30-Minuten-Zeitintervalle, 1 für 1-Stunden-Zeitintervalle). - Berechnen Sie den Mittelwert und die Standardabweichung von fünf aufeinanderfolgenden Wachstumsraten mit der gewünschten Datenanalysesoftware. Beachten Sie, dass der größte Mittelwert mit der geringsten Standardabweichung die maximale Wachstumsrate (μm) 12 ist.
9. Bestimmung der räumlichen Verzerrung
HINWEIS: Mikroplatten-Reader und -Platten führen zu Verzerrungen in den Ergebnissen. Es ist von entscheidender Bedeutung, die räumlichen Verzerrungen der Platten zu bewerten, die in einem bestimmten Mikroplatten-Reader verwendet werden, um die Zuverlässigkeit und Reproduzierbarkeit der Ergebnisse zu gewährleisten. Um dies zu erreichen, gehen Sie wie folgt vor:
- Wiederholen Sie die Schritte 1.2-1.6.
- Zeichnen Sie die Daten auf und analysieren Sie sie gemäß Schritt 1.7 und Schritt 1.8, um die Wachstumsrate für jedes Bohrloch zu berechnen.
- Erstellen Sie eine Heatmap, um die Änderungen der Wachstumsrate in der 96-Well-Platte zu visualisieren.
- Führen Sie die statistische Analyse durch (siehe ergänzende Tabellen), und stellen Sie sicher, dass keine signifikanten Unterschiede zwischen den Bohrlöchern auftreten.
HINWEIS: Es ist wichtig, in allen Vertiefungen die gleiche Art von Bakterien und die gleiche Inokulumgröße zu verwenden.
10. Validierung der OD-Messwerte und Faktor für die Weglängenkorrektur
HINWEIS: Die Bestimmung des Weglängenkorrekturfaktors ist entscheidend, um die Daten zu validieren und die Zuverlässigkeit und Gültigkeit auf verschiedenen Geräten sicherzustellen.
- Verwenden Sie die Schritte 1.2 und 1.3, um das Inokulum über Nacht von B. mycoides vorzubereiten.
- Bereiten Sie drei sterilisierte 50-ml-Kolben vor und geben Sie 49,5 ml frisches TSB in jeden Kolben. 500 μl des Inokulums werden aseptisch in jeden Kolben in einer Sicherheitswerkbank gegeben. Geben Sie 50 ml nicht geimpftes TSB in einen sterilisierten Kolben, der als Kontrolle dient.
- Die Kolben werden bei 30 °C und 150 U/min inkubiert. Alle 30 min wird aseptisch Proben aus jedem Kolben entnommen und der OD600 im Spektralphotometer abgelesen und aufgezeichnet.
- Gib 9,9 ml TSB in ein 25 ml-Becherglas. 100 μl Inokulum in das Becherglas geben und leicht schütteln.
- Geben Sie 200 μl der Mischung aus dem Becherglas in die sechs Vertiefungen in einer ausgewählten Spalte in einer 96-Well-Platte. Geben Sie 200 μl nicht geimpftes TSB als Kontrolle in drei Vertiefungen. Füllen Sie die Randvertiefungen mit 200 μl DI-Wasser.
- Setzen Sie den Deckel auf und übertragen Sie die 96-Well-Platte vorsichtig in den Mikroplatten-Reader. Führen Sie die Schritte 1.5 und 1.6 aus, um die Wachstumsdaten aufzuzeichnen.
- Plotten Sie den OD600 aus dem Mikroplatten-Reader mit dem OD600 aus dem Spektralphotometer. Beachten Sie, dass die Steigung der Versatz zwischen den beiden Messwerten ist, der als Weglängenkorrekturfaktor bezeichnet wird.
HINWEIS: Die Inkubation des Inkubators und des Mikroplatten-Readers sollte gleichzeitig am selben Tag durchgeführt werden.
Ergebnisse
Validierung des OD-Messwerts und Faktor für die Weglängenkorrektur
Geteilte Proben der B . mycoides-Kultur wurden zu unterschiedlichen Zeitpunkten entnommen und mit dem Mikroplatten-Reader und dem Spektralphotometer gemessen (Abbildung 1A). Dieser Schritt wurde unternommen, um die Ergebnisse auf verschiedenen Geräten zu validieren. Die Daten von OD600 korrelierten, stimmten aber nicht überein (...
Diskussion
Mikroplatten-Reader ermöglichen es, konsistente und wiederholbare Wachstumsraten zu erzielen. Diese Technologie minimiert menschliche Fehler und ermöglicht eine Probenahme mit hohem Durchsatz. Die geringe Menge an Kultur, die pro Probe benötigt wird, macht diesen Ansatz zu einer attraktiven und kostengünstigen Alternative zur Zellzählung mit Kolben oder Reagenzgläsern. Mikroplatten-Reader ermöglichen eine große Stichprobengröße, erhöhen die statistische Aussagekraft und erleic...
Offenlegungen
Die Autoren haben keine Interessenkonflikte anzugeben.
Danksagungen
Diese Arbeit wurde vom Natural Sciences and Engineering Research Council (NSERC) / Halifax Water Industrial Research Chair in Water Quality and Treatment (Grant No. IRCPJ 349838-16). Das Autorenteam möchte sich auch bei Anita Taylor für die Hilfe bei der Durchsicht dieses Artikels bedanken.
Materialien
| Name | Company | Catalog Number | Comments |
| Centrifuge | Eppendorf | 5810 R | |
| Centrifuge tubes - 15 mL | ThermoFisher- Scientific | 339650 | Sterile |
| Centriguge tubes - 50 mL | ThermoFisher- Scientific | 339652 | Sterile |
| Disposable inoculating loop , 10 µL | Cole-Parmer | UZ-06231-08 | Sterile |
| Erlenmeyer flasks - 250 mL | Cole-Parmer | UZ-34502-59 | Glass |
| Isopropanol | ThermoFisher- Scientific | 396982500 | ≥99.0 |
| Phosphate Buffer Saline | Sigma-Aldrich | P4417 | |
| Pipett tips 1,000 µL | ThermoFisher- Scientific | UZ-25001-76 | |
| Pipett tips 10 mL | ThermoFisher- Scientific | UZ-25001-83 | |
| Pipett tips 200 µL | ThermoFisher- Scientific | UZ-25001-85 | |
| Pipett tips 5 mL | ThermoFisher- Scientific | UZ-25001-80 | |
| Pipettor 1,000 µL | Cole-Parmer | UZ-07909-11 | |
| Pipettor 10 mL | Cole-Parmer | UZ-07909-15 | |
| Pipettor 200 µL | Cole-Parmer | UZ-07909-09 | |
| Pipettor 5 mL | Cole-Parmer | UZ-07859-30 | |
| Tryptic Soy Broth | Millipore | 22091 | Suitable for microbiology |
Referenzen
- Reuß, D. R., et al. Large-scale reduction of the Bacillus subtilis genome: Consequences for the transcriptional network, resource allocation, and metabolism. Genome Research. 27 (2), 289-299 (2017).
- Sparkes, A., et al. Towards Robot Scientists for autonomous scientific discovery. Automated Experimentation. 2 (1), (2010).
- Zwietering, M. H., Jongenburger, I., Rombouts, F. M., van t Riet, K. Modeling of the bacterial growth curve. Applied and Environmental Microbiology. 56 (6), 1875-1881 (1990).
- Pla, M., Oltra, S., Esteban, M., Andreu, S., Palop, A. Comparison of primary models to predict microbial growth by the plate count and absorbance methods. BioMed Research International. 2015 (6), 1-14 (2015).
- Choe, D., et al. Adaptive laboratory evolution of a genome-reduced Escherichia coli. Nature Communications. 10 (1), 935 (2019).
- Dykhuizen, D. E., Dean, A. M. Enzyme activity and fitness: Evolution in solution. Trends in Ecology and Evolution. 5 (8), 257-262 (1990).
- McDonald, M. J. Microbial experimental evolution - A proving ground for evolutionary theory and a tool for discovery. EMBO Reports. 20 (8), 1-14 (2019).
- Lum, P. Y., et al. Discovering modes of action for therapeutic compounds using a genome-wide screen of yeast heterozygotes. Cell. 116 (1), 121-137 (2004).
- Cagnon, C., et al. Development of a forward genetic screen to isolate oil mutants in the green microalga Chlamydomonas reinhardtii. Biotechnology for Biofuels. 6 (1), 178 (2013).
- Stevenson, K., McVey, A. F., Clark, I. B. N., Swain, P. S., Pilizota, T. General calibration of microbial growth in microplate readers. Scientific Reports. 6 (1), 38828 (2016).
- Matlock, B. C., Beringer, R. W., Ash, D. L., Page, A. F., Allen, M. W. Differences in Bacterial Optical Density Measurements between Spectrophotometers. Technical Note. ThermoScientific. , (2011).
- Hall, B. G., Acar, H., Nandipati, A., Barlow, M. Growth rates made easy. Molecular Biology and Evolution. 31 (1), 232-238 (2014).
- Rolfe, M. D., et al. Lag phase is a distinct growth phase that prepares bacteria for exponential growth and involves transient metal accumulation. Journal of Bacteriology. 194 (3), 686-701 (2012).
- Djordjevic, D., Wiedmann, M., Mclandsborough, L. A. Microtiter plate assay for assessment of Listeria monocytogenes biofilm formation. Applied and Environmental Microbiology. 68 (6), 2950-2958 (2002).
- O'Toole, G. A. Microtiter dish biofilm formation assay. Journal of Visualized Experiments. (47), e2437 (2011).
- Campbell, J. High-throughput assessment of bacterial growth inhibition by optical density measurements. Current Protocols in Chemical Biology. 3 (3), 1-20 (2012).
- Toussaint, M., Conconi, A. High-throughput and sensitive assay to measure yeast cell growth: A bench protocol for testing genotoxic agents. Nature Protocols. 1 (4), 1922-1928 (2006).
- Goughenour, K. D., Balada-Llasat, J. -. M., Rappleye, C. A. Quantitative microplate-based growth assay for determination of antifungal susceptibility of Histoplasma capsulatum yeasts. Journal of Clinical Microbiology. 53 (10), 3286-3295 (2015).
- Qiu, T. A., et al. Growth-based bacterial viability assay for interference-free and high-throughput toxicity screening of nanomaterials. Analytical Chemistry. 89 (3), 2057-2064 (2017).
- Kurokawa, M., Precise Ying, B. -. W. Precise, high-throughput analysis of bacterial growth. Journal of Visualized Experiments. (127), e56197 (2017).
- Bredit, F., Romick, T. L., Fleming, H. P. A rapid method for determination of bacterial growth kinetics. Journal of Rapid Methods and Automation in Microbiology. 3, 59-68 (1994).
- Delaney, N. F., et al. Development of an optimized medium, strain and high-throughput culturing methods for Methylobacterium extorquens. PLOS ONE. 8 (4), 62957 (2013).
- Haire, T. C., et al. Robust microplate-based methods for culturing and in vivo phenotypic screening of Chlamydomonas reinhardtii. Frontiers in Plant Science. 9, 235 (2018).
- McBirney, S. E., Trinh, K., Wong-Beringer, A., Armani, A. M. Wavelength-normalized spectroscopic analysis of Staphylococcus aureus and Pseudomonas aeruginosa growth rates. Biomedical Optics Express. 7 (10), 4034 (2016).
- Sieben, M., Giese, H., Grosch, J. H., Kauffmann, K., Büchs, J. Permeability of currently available microtiter plate sealing tapes fail to fulfil the requirements for aerobic microbial cultivation. Biotechnology Journal. 11 (12), 1525-1538 (2016).
- Zimmermann, H. F., John, G. T., Trauthwein, H., Dingerdissen, U., Huthmacher, K. Rapid evaluation of oxygen and water permeation through microplate sealing tapes. Biotechnology Progress. 19 (3), 1061-1063 (2003).
- Abkar, L., Gagnon, G. A. Biological responses to P-limitation in indigenous bacteria isolated from drinking water. AWWA Water Science. 3 (5), 1248 (2021).
- Sanders, E. R. Aseptic laboratory techniques: Plating methods. Journal of Visualized Experiments. (63), e3064 (2012).
- Hart, S. F. M., Skelding, D., Waite, A. J., Burton, J. C., Shou, W. High-throughput quantification of microbial birth and death dynamics using fluorescence microscopy. Quantitative Biology. 7 (1), 69-81 (2019).
- Periago, P. M., Abee, T., Wouters, J. A. Analysis of the heat-adaptive response of psychrotrophic Bacillus weihenstephanensis. International Journal of Food Microbiology. 79 (1-2), 17-26 (2002).
- Mira, P., Barlow, M., Meza, J. C., Hall, B. G. Statistical package for growth rates made easy. Molecular Biology and Evolution. 34 (12), 3303-3309 (2017).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten