このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
マイクロプレートリーダーを使用した細菌増殖データのハイスループット定量の再現性と精度の向上
要約
ここでは、成長曲線、成長率、最大成長率などの成長データを測定するためのハイスループットプロトコルを紹介します。このプロトコルは、2つのバイオフィルム産生細菌を使用して検証および検証されました。この研究で適用された結果とアプローチは、マイクロプレートリーダーを使用して他のハイスループットプロトコルに拡張できます。
要約
この研究は、96ウェルプレートでの細菌の増殖をモニタリングし、最大増殖速度を分析するための、再現性と信頼性に優れたハイスループットプロトコルを開発することを目的としています。2つの細菌種の成長曲線と最大成長率を決定しました。(i)まぶたの結露、(ii)光路長補正、(iii)接種サイズ、(iv)サンプリング時間間隔、(v)空間バイアスなどの問題を調査しました。プロトコルの再現性は、3 つの独立した技術的複製によって評価され、実行間の標準偏差は 0.03 でした。 Bacillus mycoides および Paenibacillus tundrae の最大成長率は、それぞれ(平均±SD)0.99 h-1 ± 0.03 h-1 および0.85 h-1 ± 0.025 h-1であると決定されました。これらの細菌は、凝集する親和性があるため、光学的に監視するのがより困難です。この研究は、マイクロプレートリーダーで信頼性、正確性、再現性のあるデータを得るためには、接種サイズ、経路長補正、蓋の温暖化、サンプリング時間間隔、ウェルプレートの空間バイアスが非常に重要であることを示しています。開発されたプロトコルとその検証ステップは、マイクロプレートリーダーやハイスループットプロトコルを使用して他の方法に拡張できるため、研究者の生来のエラーと材料コストを削減できます。
概要
細菌のメカニズムや代謝の研究など、マルチオミクス操作への関心が高まるにつれ、成長データの記録など、ハイスループットで自動化された方法の重要性が強調されています1,2。最大成長率などの速度論的パラメータを含む成長データは、さまざまな物理的、化学的、および抗菌的条件に対する細菌の応答を特徴付けるのに役立ちます。成長率データは、潜在的な遺伝子型と表現型の連鎖1を明らかにしたり、食品生産物3,4の微生物安全性と貯蔵寿命を示すために利用される標準的な応答変数です。適応的実験室進化5,6,7、ゲノムワイドスクリーニング、特定の化学アッセイ8、およびさまざまな前方遺伝スクリーニング9などの技術は、結果を評価するために成長率に依存しています。
細菌培養物の光学密度(OD)測定は、細菌の増殖をモニタリングするための標準的な微生物学的方法です。OD測定は、多くの場合、光散乱と細胞密度10,11に依存して、600nmの波長で記録されます。ランベルトベールの法則は、OD値の濃度(細胞密度、細胞数)、光路長、および吸収率係数への依存性を説明しています。分光光度計の形状と光学系は、外径の読み取り値11に影響を与えます。従来のOD測定方法は、非常に時間と労力がかかる場合があり、データにはさまざまな人為的エラーが伴う可能性があります。このプロトコルでは、マイクロプレートリーダーを使用して、分析時間12,13および生物学的汚染の可能性を減少させる。マイクロプレートリーダーを用いたハイスループット解析は、バイオフィルム産生細菌のスクリーニング14,15、細菌増殖阻害16、酵母細胞増殖17、抗真菌感受性の決定18、ナノマテリアルの毒性スクリーニング19など、さまざまな微生物学分野で広く適用されています。
数人の研究者が、マイクロプレートリーダー12,20,21を使用した細菌増殖速度プロトコルを公開しています。しかし、収集されたデータの信頼性を徹底的に検証するプロトコルは、完全には確立されていません。種22,23,24の種類やシーリングテープなどの要因が、96ウェルプレート25,26の酸素移動が不十分であるため、再現性に影響を与えると報告されている。Delaneyらは、マイクロプレートリーダーを使用した場合、増殖培地中のMethylorubrum extorquens(野生型株)の大きなクラスターを報告し、これにより非常にノイズの多い成長データを引き起こした24。この問題は、バイオフィルム産生に関連する遺伝子を除去することによって解決された24。細胞外高分子物質の分泌により、バイオフィルム産生細菌は、合体して細胞クラスターを形成するための親和性が高くなります。したがって、光散乱技術(分光光度計やマイクロプレートリーダーなど)を使用してそれらの成長を監視することはより困難です。
このプロトコルは、マイクロプレートリーダーを使用して、再現性のあるデータをハイスループットな方法で取得する手順を確立することを目的としています。 Bacillus mycoides と Paenibacillus tundrae は、その急速な成長とバイオフィルム生成能力のために使用されましたが、これらは従来、手動および自動アプローチでは困難でした。データの信頼性と再現性を評価するために、(i)光路長の補正、(ii)蓋の結露、(iii)接種サイズ、(iv)サンプリング時間間隔、(v)空間バイアスなどの要因を調査しました。このプロトコールは、マイクロプレートリーダーを使用して細菌の増殖を正確にモニタリングし、特定の増殖速度を測定するための手順を示しています。
プロトコル
注:このプロトコルのすべての手順は、無菌状態(つまり、2つの炎またはバイオセーフティキャビネットの間)で実行する必要があります。すべての材料とツールは20分間オートクレーブ処理されます。このプロトコルで使用されるすべての材料、機器、およびソフトウェアの詳細については、 資料の表 を参照してください。手袋をはめた手は消毒し、手指消毒剤または70%アルコール溶液で少なくとも1分間濡らし、その後安全キャビネットから取り出さないでください。そうでない場合は、安全キャビネットに手を戻す前に消毒手順を繰り返す必要があります。注意: 直火を使用する前に、消毒剤が完全に蒸発していることを確認してください。
注:2つの細菌は、バイオフィルムを生成する能力について前に説明したように、飲料水の生物濾過から分離されました27 。これらは、full-16S rRNAシーケンシングによって同定され、 Bacillus mycoides(SAMN10518261) および Paenibacillus tundrae(SAMN10452279)としてNCBIに提出されました。
1. グリセロール中のバクテリアストックの調製
- 3 gのトリプシン大豆ブロス(TSB)粉末を秤量し、100 mLの蒸留水に溶解します。ブロスを20分間オートクレーブし、室温まで冷まします。50 mLのフラスコに50 mLのTSBを加えます。.
- 細菌を寒天プレート27,28に移す。ループを使用して、寒天プレートから細菌のコロニーを選びます。ループを静かに渦巻かせて、ループの内容物をフラスコに追加します。フラスコを30°C±1°Cで一晩インキュベートし、150rpmでオービタルシェイクを行います。
注:温度と揺れは、ひずみの種類に応じて調整する必要があります。 - 47.5 mLのTSBを含む別の50 mLフラスコを準備します。.2.5mLの一晩培養液をフラスコに移します。OD600 = 0.6 0.05に達するまで5〜6時間インキュベートします(指数関数的な中期に達するため)。
- 培養物を50mLの滅菌済み遠心チューブに移します。培養物を2,200 × g で10分間遠心分離します。チューブの底に集まった細胞を失わないように、ブロスを優しくデカントします。
- 5 mLのリン酸緩衝生理食塩水(PBS)を同じチューブに加え、2,200 × g で10分間遠心分離します。この手順を3回繰り返します。
- ピペットを使用して細胞のOD600 を0.6 0.05に調整し、適切な量のPBS溶液を滴定します。将来の使用のために、追加されたPBSの量を正確に記録します。
- グリセロールを懸濁液に滴定して、20%v/vに到達します。チューブとボルテックスを15秒間キャップします。
- 懸濁液1.5 mLを2 mLのクライオバイアルに移します。クライオバイアルを適切な箱に入れ、-80°Cの冷凍庫で保存します。
2.オーバーナイトカルチャーを準備します。
- バクテリアストックを含むクライオバイアルを室温で30分間、または内容物が液体になるまで解凍します。
注意: 解凍プロセスを加速しないことをお勧めします。 - 滅菌したチューブにTSB10mLを充填します。ストックサスペンション50μLをチューブに移します。チューブを30°C±1°Cで一晩(16-18時間)インキュベートし、150rpmでオービタル振とうを行います。
3.接種物を準備します。
- 滅菌したTSB10mLのチューブを調製します。500 μLの一晩培養液をチューブに移します。OD600 = 0.6 0.05に達する(または指数関数的な中期に達する)まで6〜8時間インキュベートします。
- 培養物を15 mLの遠心チューブに移します。チューブを2,200 × g で10分間遠心分離します。スープをやさしくデカントします。
- PBS5 mLを加え、2,200 × g で10分間遠心分離します。この手順を3回繰り返します。
- ピペットを使用してPBS溶液を滴定することにより、細胞のOD600 を0.6 0.05に調整します。将来の使用のために、追加されたPBSの量を正確に記録します。
4. 増殖培地のマイクロプレートリーダーへの移し替え
- 蓋付きの透明で滅菌済みの平底ウェルプレート(96ウェルプレート、6ウェルプレート、12ウェルプレート)を選択します。
- ステップ1.10で説明したように、プロトコルを実行する前に、DI水を使用してマイクロプレートの光路長補正を測定します。
- 選択したウェルプレートに基づいて参照テーブルを描画し、ロード前にサンプルの位置を指定します。例えば、96 ウェルプレートの 8 x 12 テーブルを描画します。
注:リファレンステーブルは、インキュベーションおよび結果収集ステップ中の混乱を回避します。 - バイオセーフティキャビネットに、蓋付きマイクロプレート、ピペッター1,000 μLおよび200 μL、1,000 μLおよび200 μLのピペットチップ、複数のクライオバイアル、25 mLビーカー、増殖培地、および接種材料を入れます。
注:上記のアイテムは、バイオセーフティキャビネットに無菌的に移す必要があります。 - ビーカーに10mLの増殖培地を加えます。計算された量の接種物を成長培地でビーカーに移します。
注:このプロトコルの接種率は1%と5%です。例えば、接種比を5%および1%にするには、500 μLおよび100 μLの接種材料を、増殖培地9.5 mLおよび9.9 mLにそれぞれ加えます。 - 接種物を追加した後、ビーカーを静かに振って均一に分布させます。リファレンステーブルを使用して、接種した培地200μLを指定されたウェルに分注します。コントロールとして機能するウェルを少なくとも 3 つ選択し、200 μL の増殖培地 (接種物なし) を添加します。
注:コントロールウェルが参照テーブルに明確にマークされていることを確認してください。これらのウェルで成長(汚染)が観察された場合は、実験を繰り返す必要があります。 - 200 μLのDI水を、蒸発速度が高いエッジウェルに分注します。蓋を静かに置いてから、96ウェルプレートを安全キャビネットから取り外します。96ウェルプレートをプレートリーダーに慎重に置き、急な動きがないことを確認します。
5. マイクロプレートリーダーの設定
- マイクロプレートリーダーの電源を入れ、組み込みソフトウェアを使用してカスタマイズされたプロトコルを作成します。 タスク マネージャーで、 プロトコル を開き、[ 新規作成] |標準プロトコル。 「Procedure 」を開き、次のように設定を調整します。
- Set Temperatureを使用して、温度を30°C(または目標値)に設定します。
- [Incubator On]をクリックし、[Gradient]を1°Cに設定します。
注意: 蓋の結露による信頼性の低いデータを避けるために、 勾配 が 1°Cに設定されていることを確認してください。 - Preheat(予熱)オプションを選択し、ウェルプレートに沿って均一な熱分布を確保します。
- Start Kineticを開き、Run time(実行時間)(例:12:00:00または24:00:00など)とInterval between readings(00:30:00を表)または01:00:00(1時間を表)を使用して、希望のインキュベーション期間を選択します。
注:各96ウェルプレートの読み取りには約1分かかります。 - シェイク設定を次のように設定します:シェイクを開きます。シェイクモードをリニアに設定します。[連続シェイク]をクリックします。をクリックし、[Frequency] を 567 cpm (または任意の値) に調整します。
- OD読み取り設定を次のように設定します:[読み取り]をクリックします|吸光度;エンドポイント/キネティックとモノクロメーターを選択します。Wavelengthを600nmに設定します。をクリックし、[検証] |[保存] は、適切な名前と日付で新しいプロトコルとして保存します。
6. 成長データの記録
- タスクマネージャーを開く |今すぐ読む。
- 保存したプロトコルを選択し、[ OK ]をクリックしてプロトコルを開始します。
- データ分析のために読み取り値を保存します。
7. データ分析
- OD600 の結果(成長データ)をスプレッドシート形式に転送します。 結果 セクションで、各ウェルの 画像 オプションをクリックして、最終的な成長曲線を観察します。
- スプレッドシートファイルを開き、参照テーブルに基づいて記録されたデータを再配置します。ブランク読み取りの平均値を他の読み取りから減算します。
注: ブランクは、タイム 0 での OD の読み取り値です。このプロトコルでは、OD600 値が計算に考慮されます。細胞密度は、OD600 対 Cell Count の検量線が利用可能な場合に使用できます。
8. 成長率の決定
- 転送したデータを、手順1.6で作成したスプレッドシートで使用します。以下で説明する方法と式(1)を使用して 、成長率を評価します。
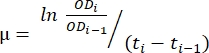 (1)
(1)
ここで、μは成長率、ODiは各時点のOD600、ODi-1は最後の時点におけるOD600の初期値、tiおよびti-1は、言及された2つの点間の時間差である(例えば、30分の時間間隔では0.5、1時間の時間間隔では1)。 - 目的のデータ分析ソフトウェアを使用して、5つの連続する成長率の平均と標準偏差を計算します。標準偏差が最も小さい最大平均は、 最大成長率 (μm) 12であることに注意してください。
9. 空間バイアスの決定
注:マイクロプレートリーダーとプレートは、結果にバイアスをもたらします。特定のマイクロプレートリーダーで使用されるプレートの空間バイアスを評価して、結果の信頼性と再現性を確保することが重要です。これを実現するには、次の手順を使用します。
- 手順1.2〜1.6を繰り返します。
- データを記録し、ステップ1.7とステップ1.8に従って分析し、各ウェルの成長率を計算します。
- ヒートマップを作成して、96ウェルプレートの成長率の変化を視覚化します。
- 統計解析を実行し( 補足表を参照)、ウェル間で有意差がないことを確認します。
注:すべてのウェルで同じ種類の細菌と同じ接種材料サイズを使用することが不可欠です。
10. OD読み取り検証とパス長補正係数
注:パス長補正係数を決定することは、データを検証し、さまざまなデバイス間で信頼性と有効性を確保するために重要です。
- ステップ1.2とステップ1.3を使用して、 B.mycoidesの一晩の接種物を準備します。
- 滅菌した50 mLフラスコを3つ調製し、各フラスコに49.5 mLの新鮮なTSBを加えます。500 μLの接種材料を安全キャビネットの各フラスコに無菌的に加えます。滅菌したフラスコに50mLの非接種TSBを加え、これがコントロールとして機能します。
- フラスコを30°C、150rpmでインキュベートします。30分ごとに、各フラスコからサンプルを無菌的に採取し、分光光度計でOD600 を読み取り、記録します。
- 25 mLのビーカーに9.9 mLのTSBを加えます。.ビーカーに100μLの接種物を加え、軽く振ってください。
- ビーカー中の混合物200 μLを、96ウェルプレートのセレクトカラムの6つのウェルに加えます。200 μLの非接種TSBをコントロールとして3つのウェルに加えます。エッジウェルに200μLのDI水を入れます。
- 蓋をして、96ウェルプレートをマイクロプレートリーダーにゆっくりと移します。ステップ 1.5 とステップ 1.6 に従って、成長データを記録します。
- マイクロプレートリーダーのOD600 と分光光度計のOD600 をプロットします。傾きは、パス長補正係数と呼ばれる 2 つの読み取りセット間のオフセットであることに注意してください。
注:インキュベーターとマイクロプレートリーダーのインキュベーションは、同じ日に同時に行う必要があります。
結果
OD読み取り検証とパス長補正係数
B. mycoides培養物の分割サンプルを異なる時点で採取し、マイクロプレートリーダーと分光光度計を使用して測定しました(図1A)。この手順は、さまざまなデバイス間で結果を検証するために行われました。OD600 のデータは相関していましたが、一致しませんでした(図 ...
ディスカッション
マイクロプレートリーダーは、一貫した再現性のある成長率を得ることができます。この技術により、人為的ミスが最小限に抑えられ、ハイスループットなサンプリングが可能になります。サンプルあたりに必要な培養量が少ないため、このアプローチは、フラスコや試験管を使用した細胞数に代わる魅力的で低コストの代替手段となります。マイクロプレートリー...
開示事項
著者は、宣言する利益相反を持っていません。
謝辞
この研究は、Natural Sciences and Engineering Research Council(NSERC)/Halifax Water Industrial Research Chair in Water Quality and Treatment(助成金番号)によって資金提供されました。IRCPJ 349838-16)。著者チームはまた、この記事のレビューにAnita Taylorの助けに感謝したいと思います。
資料
| Name | Company | Catalog Number | Comments |
| Centrifuge | Eppendorf | 5810 R | |
| Centrifuge tubes - 15 mL | ThermoFisher- Scientific | 339650 | Sterile |
| Centriguge tubes - 50 mL | ThermoFisher- Scientific | 339652 | Sterile |
| Disposable inoculating loop , 10 µL | Cole-Parmer | UZ-06231-08 | Sterile |
| Erlenmeyer flasks - 250 mL | Cole-Parmer | UZ-34502-59 | Glass |
| Isopropanol | ThermoFisher- Scientific | 396982500 | ≥99.0 |
| Phosphate Buffer Saline | Sigma-Aldrich | P4417 | |
| Pipett tips 1,000 µL | ThermoFisher- Scientific | UZ-25001-76 | |
| Pipett tips 10 mL | ThermoFisher- Scientific | UZ-25001-83 | |
| Pipett tips 200 µL | ThermoFisher- Scientific | UZ-25001-85 | |
| Pipett tips 5 mL | ThermoFisher- Scientific | UZ-25001-80 | |
| Pipettor 1,000 µL | Cole-Parmer | UZ-07909-11 | |
| Pipettor 10 mL | Cole-Parmer | UZ-07909-15 | |
| Pipettor 200 µL | Cole-Parmer | UZ-07909-09 | |
| Pipettor 5 mL | Cole-Parmer | UZ-07859-30 | |
| Tryptic Soy Broth | Millipore | 22091 | Suitable for microbiology |
参考文献
- Reuß, D. R., et al. Large-scale reduction of the Bacillus subtilis genome: Consequences for the transcriptional network, resource allocation, and metabolism. Genome Research. 27 (2), 289-299 (2017).
- Sparkes, A., et al. Towards Robot Scientists for autonomous scientific discovery. Automated Experimentation. 2 (1), (2010).
- Zwietering, M. H., Jongenburger, I., Rombouts, F. M., van t Riet, K. Modeling of the bacterial growth curve. Applied and Environmental Microbiology. 56 (6), 1875-1881 (1990).
- Pla, M., Oltra, S., Esteban, M., Andreu, S., Palop, A. Comparison of primary models to predict microbial growth by the plate count and absorbance methods. BioMed Research International. 2015 (6), 1-14 (2015).
- Choe, D., et al. Adaptive laboratory evolution of a genome-reduced Escherichia coli. Nature Communications. 10 (1), 935 (2019).
- Dykhuizen, D. E., Dean, A. M. Enzyme activity and fitness: Evolution in solution. Trends in Ecology and Evolution. 5 (8), 257-262 (1990).
- McDonald, M. J. Microbial experimental evolution - A proving ground for evolutionary theory and a tool for discovery. EMBO Reports. 20 (8), 1-14 (2019).
- Lum, P. Y., et al. Discovering modes of action for therapeutic compounds using a genome-wide screen of yeast heterozygotes. Cell. 116 (1), 121-137 (2004).
- Cagnon, C., et al. Development of a forward genetic screen to isolate oil mutants in the green microalga Chlamydomonas reinhardtii. Biotechnology for Biofuels. 6 (1), 178 (2013).
- Stevenson, K., McVey, A. F., Clark, I. B. N., Swain, P. S., Pilizota, T. General calibration of microbial growth in microplate readers. Scientific Reports. 6 (1), 38828 (2016).
- Matlock, B. C., Beringer, R. W., Ash, D. L., Page, A. F., Allen, M. W. Differences in Bacterial Optical Density Measurements between Spectrophotometers. Technical Note. ThermoScientific. , (2011).
- Hall, B. G., Acar, H., Nandipati, A., Barlow, M. Growth rates made easy. Molecular Biology and Evolution. 31 (1), 232-238 (2014).
- Rolfe, M. D., et al. Lag phase is a distinct growth phase that prepares bacteria for exponential growth and involves transient metal accumulation. Journal of Bacteriology. 194 (3), 686-701 (2012).
- Djordjevic, D., Wiedmann, M., Mclandsborough, L. A. Microtiter plate assay for assessment of Listeria monocytogenes biofilm formation. Applied and Environmental Microbiology. 68 (6), 2950-2958 (2002).
- O'Toole, G. A. Microtiter dish biofilm formation assay. Journal of Visualized Experiments. (47), e2437 (2011).
- Campbell, J. High-throughput assessment of bacterial growth inhibition by optical density measurements. Current Protocols in Chemical Biology. 3 (3), 1-20 (2012).
- Toussaint, M., Conconi, A. High-throughput and sensitive assay to measure yeast cell growth: A bench protocol for testing genotoxic agents. Nature Protocols. 1 (4), 1922-1928 (2006).
- Goughenour, K. D., Balada-Llasat, J. -. M., Rappleye, C. A. Quantitative microplate-based growth assay for determination of antifungal susceptibility of Histoplasma capsulatum yeasts. Journal of Clinical Microbiology. 53 (10), 3286-3295 (2015).
- Qiu, T. A., et al. Growth-based bacterial viability assay for interference-free and high-throughput toxicity screening of nanomaterials. Analytical Chemistry. 89 (3), 2057-2064 (2017).
- Kurokawa, M., Precise Ying, B. -. W. Precise, high-throughput analysis of bacterial growth. Journal of Visualized Experiments. (127), e56197 (2017).
- Bredit, F., Romick, T. L., Fleming, H. P. A rapid method for determination of bacterial growth kinetics. Journal of Rapid Methods and Automation in Microbiology. 3, 59-68 (1994).
- Delaney, N. F., et al. Development of an optimized medium, strain and high-throughput culturing methods for Methylobacterium extorquens. PLOS ONE. 8 (4), 62957 (2013).
- Haire, T. C., et al. Robust microplate-based methods for culturing and in vivo phenotypic screening of Chlamydomonas reinhardtii. Frontiers in Plant Science. 9, 235 (2018).
- McBirney, S. E., Trinh, K., Wong-Beringer, A., Armani, A. M. Wavelength-normalized spectroscopic analysis of Staphylococcus aureus and Pseudomonas aeruginosa growth rates. Biomedical Optics Express. 7 (10), 4034 (2016).
- Sieben, M., Giese, H., Grosch, J. H., Kauffmann, K., Büchs, J. Permeability of currently available microtiter plate sealing tapes fail to fulfil the requirements for aerobic microbial cultivation. Biotechnology Journal. 11 (12), 1525-1538 (2016).
- Zimmermann, H. F., John, G. T., Trauthwein, H., Dingerdissen, U., Huthmacher, K. Rapid evaluation of oxygen and water permeation through microplate sealing tapes. Biotechnology Progress. 19 (3), 1061-1063 (2003).
- Abkar, L., Gagnon, G. A. Biological responses to P-limitation in indigenous bacteria isolated from drinking water. AWWA Water Science. 3 (5), 1248 (2021).
- Sanders, E. R. Aseptic laboratory techniques: Plating methods. Journal of Visualized Experiments. (63), e3064 (2012).
- Hart, S. F. M., Skelding, D., Waite, A. J., Burton, J. C., Shou, W. High-throughput quantification of microbial birth and death dynamics using fluorescence microscopy. Quantitative Biology. 7 (1), 69-81 (2019).
- Periago, P. M., Abee, T., Wouters, J. A. Analysis of the heat-adaptive response of psychrotrophic Bacillus weihenstephanensis. International Journal of Food Microbiology. 79 (1-2), 17-26 (2002).
- Mira, P., Barlow, M., Meza, J. C., Hall, B. G. Statistical package for growth rates made easy. Molecular Biology and Evolution. 34 (12), 3303-3309 (2017).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved